2
1.
RNAi: PRINCIPI E MECCANISMI
1.1.
Introduzione
L’interferenza del RNA è un meccanismo regolatorio attivato da molte cellule eucariotiche mediante piccole molecole a doppio filamento di RNA che inducono un controllo omologia-dipendente dell’attività genetica. Dalla sua prima descrizione, una decina di anni fa (1998), negli animali, il principio dell’interferenza da parte dell’RNA (RNAi) ha avuto un notevole sviluppo in relazione ad un suo possibile uso come modalità terapeutica nel trattamento di patologie umane (1). In particolare, la sua espansione si è osservata da quando ne è stata effettuata la caratterizzazione nel nematode Caenorhabditis elegans (2, 3). Successivamente, fu realizzato un silenziamento stabile attraverso l’espressione forzata di piccoli RNA a forcina (shRNA) in differenti sistemi modello, come Drosophila e piante (3).
In seguito, l’RNAi fu riscontrato anche in cellule di mammifero in risposta a piccoli RNA interferenti a doppio filamento (siRNA) della lunghezza di 21 nucleotidi, utilizzati come molecole effettrici per il silenziamento di un gene in maniera sequenza-specifica (2).
3
Numerosi studi successivi hanno fatto accrescere la speranza che i meccanismi alla base di RNAi potessero essere impiegati terapeuticamente nel trattamento di alcune patologie umane. L’uso di RNAi in terapia si basa sulla sua abilità di reprimere in maniera specifica ed efficace l’espressione di una sequenza conosciuta che provoca un determinato stato patologico.
Numerose ricerche si sono quindi sviluppate in tempi brevi con lo scopo di individuare potenziali impieghi terapeutici per RNAi in un’ampia gamma di patologie; molti di questi studi recenti hanno infatti evidenziato la sua efficacia in interessanti settori terapeutici, portando i ricercatori a manifestare ottimismo, seppur ancora cauto, riguardo al trattamento con RNAi.
Le prime applicazioni cliniche si sono rivolte al trattamento della degenerazione maculare dell’anziano (AMD) (4, 5) e dell’infezione respiratoria da virus sinciziale (RSV) (6). Sebbene sia ancora necessario approfondire il suo potenziale impiego terapeutico, è interessante sottolineare che molte terapie basate sull’RNAi sono oggi in fase di sviluppo preclinico soprattutto per patologie di natura genetica e virale, per disordini neurodegenerativi e per il trattamento del cancro.
Una dettagliata comprensione dei meccanismi di base dell’RNAi è possibile ottenerla attraverso lo sviluppo di molecole dotate di migliore potenza ed efficacia. Ad esempio, risulta interessante l’espressione di
4
piccoli frammenti di siRNA e di shRNA che, presentando sia vantaggi che svantaggi specifici, devono essere significativamente presi in considerazione nel trattamento terapeutico di alcune malattie.
Da evidenziare tuttavia che, sebbene da un lato, numerosi studi in
vivo abbiano mostrato la potenziale efficacia di svariate strategie
terapeutiche basate sull’RNAi, dall’altro molti studi hanno messo in evidenza obiettivi diversi, come ad esempio l’utilizzo di un meccanismo cellulare endogeno per ottenere un beneficio terapeutico.
Da non sottovalutare anche l’insorgenza di effetti collaterali indesiderati che comprendono ad esempio l’induzione delle risposte dell’interferone di tipo I (IFN) (7) e la saturazione dei componenti endogeni. Questi fenomeni fanno parte del meccanismo dell’RNAi, ed evidenziano dunque la necessità di essere prudenti quando sono coinvolte molecole effettrici rilasciate all’interno del target cellulare.
Il problema del rilascio cellulo-specifico o tessuto-specifico costituisce un altro elemento su cui discutere per quanto riguarda l’impiego terapeutico dell’RNAi. Svariate strategie che si basano su un processo di attivazione legate al rilascio di RNAi di natura virale e non, sembrano essere efficaci per la cura di alcune malattie e gli studi in corso fanno ben sperare per il futuro, ampliando così il range di patologie che possono essere trattate con questo tipo di approccio terapeutico.
5
1.2.
Meccanismi di silenziamento di un gene mediati da
RNAi
I meccanismi dell’RNAi sono guidati da piccoli RNA che comprendono sia siRNA che microRNA (miRNA) e che derivano dall’imperfetto accoppiamento di strutture a forcina di RNA codificate naturalmente nel genoma (2).
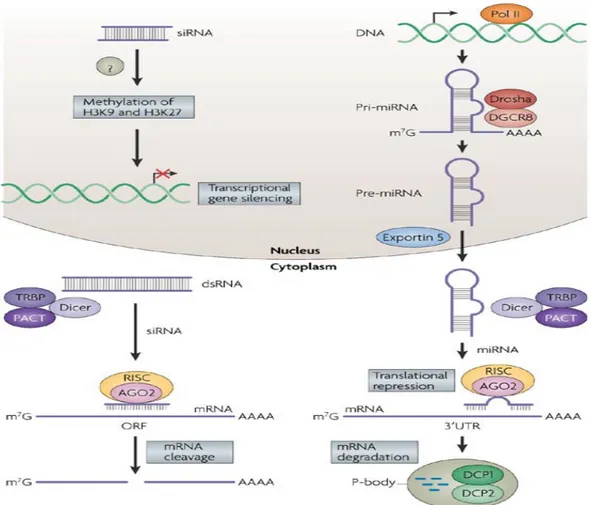
Molecole effettrici di RNAi inducono il silenziamento di un gene in diverse maniere: tali RNAi possono dirigere una scissione sequenza specifica di mRNA perfettamente complementari e/o provocare repressione nella traduzione e nella degradazione del trascritto nel caso di una non perfetta complementarietà. I meccanismi di RNAi possono poi essere volti al silenziamento di un gene nel processo di trascrizione (TGS) a livello nucleare, sebbene i dettagli meccanicistici non siano ancora ben noti a livello delle cellule di mammifero (Figura 1).
6
Figura 1. Meccanismi dell’interferenza ad RNA nelle cellule di mammifero.
Come mostra il disegno in basso a sinistra, RNA a doppi filamenti (dsRNAs) citoplasmatici sono processati da un complesso che comprende Dicer, TAR proteina di legame dell’RNA (TRBP) e una proteina attivatrice della protein chinasi PKR (PACT), all’interno del piccolo RNA interferente (siRNAs), e sono caricati nell’Argonauta (AGO2) e nel complesso di silenziamento indotto dall’RNA (RISC). Il filamento guida siRNA riconosce il sito bersaglio per dirigere il taglio dell’mRNA che è condotto dal dominio catalitico di AGO2. I siRNA che sono complementari a regioni del promotore, dirigono il silenziamento genico trascrizionale nel nucleo attraverso cambiamenti nella cromatina che coinvolgono la metilazione degli istoni (in alto a sinistra); non sono attualmente ben chiari i precisi dettagli molecolari nella cellule di mammifero. Come mostra la sequenza a destra, i trascritti primari di microRNA (pri-miRNAs) codificati endogeneamente sono trascitti dell’RNA polimerasi II (Pol II) e inizialmente processati da Drosha-DGCR8 ( gene 8 regione critica collegata allo sviluppo della sindrome si DiGeorge) per generare i precursori di miRNA (pre-miRNAs). Questi precursori sono trasportati nel citoplasma dall’esportina 5 e successivamente legati al complesso Dicer-TRBP-PACT che processa i pre-miRNA per il loro caricamento su AGO2 e RISC. Il miRNA maturo riconosce i siti bersaglio nella regione 3’ non tradotta (3’UTR) degli mRNA per dirigere l’inibizione traslazionale e la degradazione dell’ mRNA nei corpi (P) di processamento che contengono enzimi per rimuovere il cappuccio DCP1 e DCP2.
7
1.2.1. Silenziamento di un gene a livello post-trascrizionale tramite siRNA
siRNA (“short interfering RNA”) esogeni hanno come bersagli gli mRNA complementari con degradazione del trascritto primario in un processo noto come silenziamento genico post-trascrizionale (PTGS) (2). Nei nematodi, nelle piante e negli insetti, questi processi rappresentano meccanismi innati di difesa naturale antivirale, in cui l’RNA virale a doppio filamento viene processato a siRNA che media la risposta dell’RNAi; l’RNA virale è processato ad opera di un enzima Dicer (RNAase III). Per quanto riguarda le cellule di mammifero, non è ancora ben chiaro se esiste o no un meccanismo mediato dal siRNA di PTGS per l’immunità intrinseca contro le infezioni virali; rimane dunque, per il momento, un tema da approfondire ulteriormente con la ricerca.
Un efficace PTGS richiede un perfetto o quasi perfetto accoppiamento delle basi tipo Watson - Crick tra il trascritto di mRNA ed il filamento di siRNA antisenso (o guida) e comporta una scissione dell’mRNA ad opera del complesso di silenziamento indotto dall’RNA (RISC). L’Endonucleasi Argonauta 2 (AGO2) è responsabile del meccanismo di scissione del RISC ed AGO2 è l’unico membro della sottofamiglia delle proteine Argonaute dotato di attività catalitica nelle cellule di mammifero.
8
Secondo studi recenti l’attivazione del RISC è un fenomeno che coinvolge inizialmente la degradazione mediata da AGO2 del filamento senso (“passenger”) dell’siRNA a doppio filamento, generando così il filamento singolo antisenso che serve per guidare il RISC verso le sequenze complementari a livello dell’mRNA bersaglio (Figura 2). Il filamento guida è legato all’interno del dominio catalitico Rnasi H-like PIWI di AGO2 all’estremità 5′; ed un dominio PIWI-Argonauta-Zwille (PAZ) riconosce l’estremità 3′ del siRNA.
9 Figura 2. Caricamento nel RISC ed attivazione.
Gli RNA a doppio filamento (dsRNAs) e i precursori pre-miRNA sono processati da un complesso che comprende Dicer, TAR proteina di legame dell’RNA (TRBP) e una proteina attivatrice della protein chinasi PKR (PACT), che facilita il caricamento dei piccoli RNA interferenti (siRNA) o micro-RNA a doppio filamento sull’Argonauta 2 (AGO2) e il complesso di silenziamento indotto dall’RNA (RISC). Quando l’RNA a doppio filamento caricato nel RISC ha una perfetta complementarietà di sequenza, AGO2 taglia il filamento passenger producendo il RISC attivo che contiene il filamento guida complementare alla sequenza bersaglio. Quando l’RNA a doppio filamento caricato nel RISC ha una complementarietà di sequenza imperfetta viene impiegato un meccanismo “bypass” nel quale si richiede un’attività elicasica per svolgere il filamento passenger dal filamento guida e per generare un filamento miRNA maturo, producendo un RISC attivo.
10
La scissione dell’mRNA target si verifica tra le basi 10 ed 11 relative all’estremità 5′ del filamento guida di siRNA, portando alla successiva degradazione dell’mRNA trascritto, scissione provocata dalle esonucleasi cellulari. Una volta scatenata l’attivazione dal filamento guida di siRNA, RISC può innescare cicli multipli di scissione dell’mRNA allo scopo di mediare una consistente risposta di PTGS nei confronti del gene bersaglio.
Il silenziamento genico post-trascrizionale per tale motivo è stato sfruttato come metodo di elezione in potenziali applicazioni terapeutiche di RNAi, grazie alla potenza manifestata da tale via catalitica.
1.2.2. La via dei microRNA
Una funzione significativa è svolta senza dubbio dalla via del miRNA (micro- RNA) endogeno, in quanto agisce come reostato cellulare, modulando la fine regolazione dell’espressione genica durante lo sviluppo e la differenziazione. Le regioni non tradotte (3′UTR) degli mRNA rappresentano il bersaglio dei miRNA, condividendo una parziale complementarietà nella sequenza.
Questi piccoli RNA endogeni di 22 nucleotidi di lunghezza inducono un silenziamento genico post-trascrizionale attraverso una
11
repressione della traduzione. Questo processo, come precedentemente accennato, è spesso accompagnato da una successiva degradazione dell’mRNA che si verifica nei compartimenti citoplasmatici noti come corpi che processano (“P-bodies”).
Infatti, quando un miRNA presenta una totale complementarietà di sequenza con un mRNA target, questi dirige la scissione dell’mRNA attraverso l’attività RISC. Ad esempio è stato osservato che la scissione mi-196 diretta di Hoxb8 (homeobox 8) si verifica nelle cellule di mammifero indicando un livello di sovrapposizione funzionale tra siRNA ed i processi di silenziamento di un gene diretto da miRNA.
I trascritti di miRNA primari (pri-miRNA) sono generalmente trascritti dalla RNA polimerasi II (pol II) nel nucleo (sebbene una recente ricerca affermi che gli miRNA sono trascritti da RNA pol III) e sono processati dall’enzima Drosha Rnasi III in strutture di circa 70 nucleotidi a forma di stelo-loop note come miRNA precursore (pre-miRNA). L’enzima Drosha svolge la propria attività grazie all’interazione con la proteina del gene 8 che lega l’RNA a doppio filamento formando un complesso noto come microprocessore; questa proteina rappresenta una punto cruciale ed è correlata con lo sviluppo della sindrome di DiGeorge (DGCR8).
La proteina esportina 5, che lega l’RNA a doppio filamento, trasporta il pre-miRNA all’interno del citoplasma in un modo Ran-GTP dipendente, dove Dicer e le sue proteine di legame dell’RNA a doppio
12
filamento, la proteina di legame dell’RNA TAR HIV-1 (TRBP), e la proteina attivatrice della proteina chinasi PKR (PACT), processano il pre-miRNA e caricano l’pre-miRNA maturo di 22 nucleotidi nel RISC (Figura 1).
Tale via di caricamento del miRNA nel RISC non sembra essere coinvolta nella scissione del filamento “passenger” del miRNA; è stato invece ipotizzato un meccanismo di “bypass” che richiede l’attività di una elicasi necessaria a svolgere e rilasciare il filamento “passenger”. Dunque, l’imperfetta omologia di sequenza tra il filamento di miRNA maturo ed il filamento “passenger” complementare dovrebbe prevenire l’azione di AGO2 ovvero la scissione del filamento “passenger”.
Una volta che tale filamento è stato disavvolto ed allontanato e che il miRNA maturo si lega all’estremità 3′UTR dell’mRNA, il RISC può indurre la repressione traslazionale e conseguentemente favorire la successiva degradazione dell’mRNA per silenziare l’espressione di un gene (Figura 2).
La sequenza iniziale dell’miRNA maturo che circonda i primi 2-7 o 2-8 nucleotidi a livello dell’estremità 5′, deve essere totalmente complementare al suo “target”; risultano invece maggiormente tollerati nucleotidi errati a livello dell’estremità 3′ del filamento di miRNA.
Sebbene gli stadi effettori di questi meccanismi endogeni non siano stati ancora utilizzati a scopo terapeutico, delle perfette sequenze “duplex” di siRNA sono state introdotte nell’ossatura di pri-miRNA e pre-miRNA
13
consentendo dunque la produzione di miRNA mimetici. Questi possono, quindi, essere processati tramite il meccanismo dell’miRNA, innescando conseguentemente il più potente meccanismo del PTGS di degradazione dell’mRNA una volta caricato all’interno del RISC (2).
1.2.3. Silenziamento di un gene a livello trascrizionale ad opera di siRNA
Il silenziamento dell’espressione di un gene (TGS) a livello della trascrizione è stato inizialmente sperimentato a livello dei nuclei di cellule di piante o di funghi.
Il TGS regola l’espressione genica attraverso cambiamenti nella cromatina, alterazioni mediate da siRNA e attraverso il complesso meccanismo del RNAi (Figura 1).
È stato evidenziato in cellule di mammifero sia il silenziamento a livello trascrizionale sia la metilazione degli istoni, in seguito all’azione di siRNA esogeni che hanno come “target” il promotore. Il meccanismo esatto con cui il processo avviene tuttavia non è ancora ben noto.
Il TGS rappresenta pertanto una tecnica interessante e potenziale in future applicazioni terapeutiche dell’RNAi per il silenziamento prolungato, epigenetico di un gene. Infatti, risultano attualmente in corso numerose applicazioni in modelli preclinici.
14
1.3.
Progettazione di potenti inneschi di RNAi
1.3.1. Veicolazione di siRNA
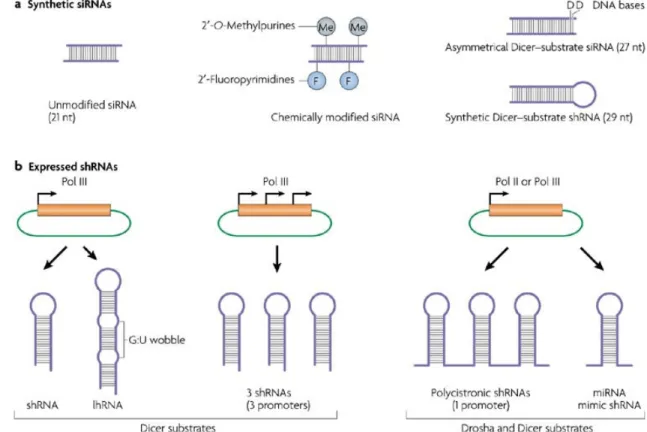
Attualmente, la maggior parte dei RNAi proposti in diverse applicazioni cliniche, derivano dall’incorporazione di siRNA a doppio filamento di 21 nucleotidi, sintetizzati chimicamente, caratterizzati da due nucleotidi sporgenti all’estremità 3′ (Figura 3a). Ciò consente indubbiamente, da un lato la sintesi su larga scala e dall’altro la produzione di molecole di siRNA uniformi e che successivamente possono essere sottoposte ad alterazioni chimiche con il fondamentale obiettivo di aumentarne la stabilità (Fig 3).
15
Figura 3. Le molecole effettrici nell’interferenza ad RNA.
a) Piccoli RNA interferenti (siRNAs) sintetici vengono somministrati in vivo. Questi possono essere prodotti contenendo modificazioni chimiche come le 2’-O-metilpurine o 2’-fluoropirimidine, che possono essere aggiunte per aumentare la stabilità. Possono essere anche prodotti siRNA substrato per Dicer asimmetrici. Questi hanno un’estremità netta che include 2 basi del DNA, mentre l’altra estremità 3’ ha 2 nucleotidi che sporgono. Questo assicura che una singola specie di siRNA venga generata dal Dicer che processa l’estremità non sfalzata. RNA sintetici a forcina più lunghi (shRNAs) sono anche processati come substrati del Dicer.
b) Vettori di espressione guidano alti livelli di espressione di shRNA mediante promotori della polimerasi III (Pol III). Lunghi RNA a forcina (lhRNAs) generano multiple specie di siRNA processati da Dicer, questo suggerisce che il Dicer dei mammiferi è processivo. Promotori multipli separati della Pol III possono essere usati in un singolo vettore per guidare l’espressione di diversi shRNA. Vettori che hanno promotori per la Pol II o Pol III generano RNA precursori più lunghi inclusi i trascritti di shRNA policistronici e micro-RNA (miRNA) mimetici che sono processati da Drosha e da Dicer.
16
Il silenziamento dell’espressione di un gene viene ottenuta progettando sequenze di siRNA che hanno come obiettivo regioni codificanti e non codificanti degli mRNA con perfetta complementarietà per l’induzione di silenziamento genico post-trascrizionale.
Svariate entità commerciali coinvolte nella realizzazione di siRNA forniscono “on-line” efficaci algoritmi di progettazione, che sono basati sulla combinazione di sequenze “target” di mRNA e strutture di tipo secondario, siRNA duplex con estremità stabili. Queste hanno come obiettivo principale quello di minimizzare potenziali “off-target effects” (OTEs) dipendenti dalla sequenza.
È interessante sottolineare come siRNA più lunghi (27 mers) e shRNA (29 nucleotidi) sintetizzati chimicamente vengano impiegati sottoforma di substrati per la processazione Dicer, manifestando la capacità di incrementare la potenza del PTGS (Figura 3a).
Inoltre, è stato dimostrato che Dicer e TRBP-PACT sono in grado di includere una piattaforma di caricamento per la formazione di RISC, e proprio l’inserimento di questo passaggio di caricamento nella via del RNAi, attraverso l’uso di substrati del Dicer, ha permesso di ottenere un silenziamento genico più potente.
La progettazione di tali siRNA lunghi 27 mers ha tenuto conto della necessità di ricreare strutture asimmetriche caratterizzate da due nucleotidi che sporgono in corrispondenza dell’estremità 3′ e da un’estremità piatta.
17
Dunque, risulta fondamentale la capacità di Dicer di riconoscere, ai fini della processazione, proprio i due nucleotidi sporgenti in posizione 3′, aspetto che garantisce la produzione di siRNA a singolo filamento. La presenza invece dell’estremità piatta, che comprende basi di DNA, potrebbe attivare l’induzione di bassi livelli di interferone; tuttavia le basse concentrazioni dei siRNA-27 mers richieste per rendere silente l’espressione genica, sembrano essere in grado di evitare o quanto meno minimizzare la risposta da parte dell’interferone.
1.3.2. shRNA espressi
E’ stato tuttavia evidenziato che se da un lato i siRNA silenziano solo transitoriamente l’espressione di un gene, poiché la loro concentrazione intracellulare viene via via diluita durante le successive divisioni cellulari, dall’altro lato, i shRNA espressi sono in grado di mediare e produrre uno stabile silenziamento a lungo termine dei loro trascritti bersaglio, cioè finché si verifica la trascrizione dei shRNA (Figura 3b). I promotori dell’RNA polimerasi II e III sono usati per guidare l’espressione dei costrutti di shRNA, in relazione al tipo di espressione richiesta.
18
In conformità con i loro normali ruoli cellulari nella produzione di abbondanti, piccoli RNA endogeni (come U6 e H1), i promotori della polimerasi III guidano l’espressione di elevati livelli di shRNA costitutivi. Risultano inoltre ben definiti sia i punti di inizio sia i segnali di fine (4-6 timidine) della trascrizione.
Gli shRNA guidati dal promotore della polimerasi II, possono essere espressi in modo tessuto-specifico e vengono trascritti sottoforma di precursori più lunghi che mimano pri-miRNA. Presentano, inoltre, segnali del cappuccio e di poliA che devono essere processati.
Tali miRNA/shRNA artificiali sono efficientemente incorporati all’interno del RISC: questo garantisce un significativo contributo per una più potente inibizione dell’espressione del gene bersaglio. Questo aspetto appare particolarmente interessante perché permette di ottenere livelli più bassi di espressione del shRNA, con conseguente possibilità di prevenire la saturazione dei componenti nella via del RNAi. Infine, un ulteriore vantaggio nell’uso di promotori della polimerasi II è rappresentato dal fatto che un singolo trascritto può contemporaneamente esprimere più miRNA ed shRNA mimetici(Figura 3b).
In conclusione, questa strategia risulta, da un punto di vista applicativo, particolarmente utile per sopprimere contemporaneamente l’espressione di due o più “targets” terapeutici, oppure per “bersagliare” più siti di un singolo prodotto genico.