Capitolo 6
Valutazioni sperimentali
In questo capitolo mostriamo le modalità di conduzione degli esperimenti effettuati per stabilire l’efficienza del metodo proposto in questa tesi.
Le valutazioni sull’efficienza del metodo vengono fatte in base a un data set reale di espressioni regolari e di query string. Infatti, vengono considerate le espressioni regolari utilizzate per consultare le informazioni contenute nel database PROSITE [2].
I risultati sono stati ottenuti in due fasi consecutive e si basano sulla scelta di alcuni parametri fondamentali che, come vedremo, inevitabilmente vanno ad influenzare la precisione con cui si esprime una determinata espressione r ∈ R attraverso uno o più intervalli di stringhe. Quindi, spiegheremo i criteri secondo cui sono state effettuate determinate scelte e mostreremo come, al variare di queste scelte, si ottengono esiti diversi, visti in termini di trade-off spazio- precisione.
6.1
Data set di espressioni regolari
Il data set R di espressioni, preso in considerazione per sperimentare il metodo proposto, è quello contenuto nel database PROSITE [2].
Il numero di pattern (circa 1300) che costituiscono il data set è limitato poiché, come abbiamo già detto in precedenza, i pattern a cui ci riferiamo non sono pattern generati casualmente da un generatore di espressioni regolari, come considerato in [21], ma sono pattern reali, che solo un utente esperto in materia di proteine può immettere per interrogare il database PROSITE.
I pattern presi in considerazione sono di lunghezza variabile e sono specificati con la sintassi esposta nel capitolo 1.
Come vedremo nella sezione che segue, nella prima fase dell’esperimento utilizziamo tutti i pattern a disposizione. Nella seconda fase, l’intero dataset viene suddiviso in sottoinsiemi contenenti pattern scelti casualmente tra i 1300 possibili. La dimensione di tali sottoinsiemi aumenta con una taglia di 100 pattern alla volta, cioè si genera un primo sottoinsieme contenente 100 pattern, un secondo sottoinsieme contenente 200 pattern, un terzo sottoinsieme contenente 300 pattern e così via fino ad ottenere un tredicesimo sottoinsieme contenente 1300.
6.2
Data set di query string
I pattern che vengono specificati per interrogare il database PROSITE rappresentano un insieme implicito di sottosequenze di aminoacidi di lunghezza finita presenti nelle proteine. Infatti, in generale, una proteina viene vista come una stringa di lunghezza finita (a volte costituita anche da moltissimi caratteri) composta da diverse sottosequenze di aminoacidi. Per condurre gli esperimenti e quindi utilizzare un insieme di query string reale, abbiamo implementato un algoritmo (esposto in fig. 6.1) con cui abbiamo estratto, da una generica proteina, tutte le sottosequenze di aminoacidi aventi corrispondenza con i pattern presenti nel dataset. In pratica, in una prima fase abbiamo prodotto un file contenente
circa 2500 proteine (prese in considerazione in [2]), ciascuna di lunghezza variabile, alcune costituite anche da 4096 caratteri, e in una seconda fase abbiamo prodotto due file Qsuccesso e Qfallimento.
Il primo file, Qsuccesso, contiene tutte le sottosequenze di aminoacidi (stringhe), contenute nelle proteine, che fanno parte del linguaggio definito da almeno un’espressione presente nel data set preso in considerazione; Qfallimento contiene alcune delle stringhe, contenute nelle proteine, per cui non c’è corrispondenza con i pattern specificati nel dataset, ovvero non esiste alcuna espressione il cui linguaggio contenga le stringhe appartenenti a Qfallimento.
Algoritmo RicercaSottoSeq (FileProteine, FilePattern)
Input: FileProteine file che contiene le proteine da
scandire;
FilePattern file che contiene i pattern.
Output: Qsuccesso file che contiene sottosequenze di
proteine che hanno almeno una espressione, in FilePattern, il cui linguaggio le include; Qfallimento file che contiene sottosequenze di proteine che non hanno alcuna espressione, in FilePattern, il cui linguaggio le include; 1. Qsuccesso := ∅ ; Qfallimento := ∅ ;
2. for each espressione r ∈ FilePattern do
3. for each stringa p ∈ FileProteine do
4. repeat
5. if (∃ sottosequenza p′ di p t.c. p′∈L(r)) then 6. Qsuccesso := Qsuccesso ∪ {p′};
7. else
8. Qfallimento := Qfallimento ∪ {p′}; 9. until (fine stringa di p);
10. return Qsuccesso, Qfallimento; Fig. 6.1 Algoritmo per la ricerca di
Le stringhe contenute rispettivamente in Qsuccesso e Qfallimento, ottenute a partire da un insieme di 2500 proteine, sono più di 4000 e costituiscono il data set di query string su cui vengono fatti gli esperimenti. Indicheremo la dimensione (in termini di numero di stringhe presenti) di questi due insiemi con il simbolo Ns .
6.3
Scelte effettuate e risultati degli esperimenti
In questa sezione mostriamo i risultati ottenuti al variare di determinate scelte effettuate.
La tabella 6.1 mostra i parametri, coinvolti nella sperimentazione, con i relativi valori.
Con il parametro k indichiamo il numero di espressioni di alternativa da espandere, presenti in un generico pattern, con DR la dimensione (espressa in numero di pattern) dell’intero data set a disposizione e con Dnum un sottoinsieme di pattern, di R, contenente num pattern.
In generale, il numero degli intervalli con cui si rappresenta una generica espressione r ∈ R varia in base al tipo di approssimazione che si sceglie (come visto nel capitolo 5). Infatti, se ad esempio un pattern contiene 5 sottoespressioni
PARAMETRI SIGNIFICATO VALORE
| |
DR
k Dnum
Cardinalità alfabeto
Dimensione data set R Numero espressioni OR Sottoinsiemi di num pattern 20 1300 100, 200, …1300 0, 1, 2, …, 8
Tabella 6.1 Parametri sperimentali e corrispondenti valori
di alternativa e il valore di k viene inizializzato a 3, allora vorrà dire che solo le prime tre espressioni di alternativa saranno espanse e il numero di intervalli, che rappresenteranno il pattern considerato, sarà in funzione del numero delle lettere racchiuse tra le parentesi [] delle prime tre sottoespressioni di alternativa. In questo caso il pattern sarà espresso come l’unione disgiunta di più sottointervalli. Le rimanenti due espressioni di alternativa verranno approssimate secondo la regola esposta nel capitolo 5.
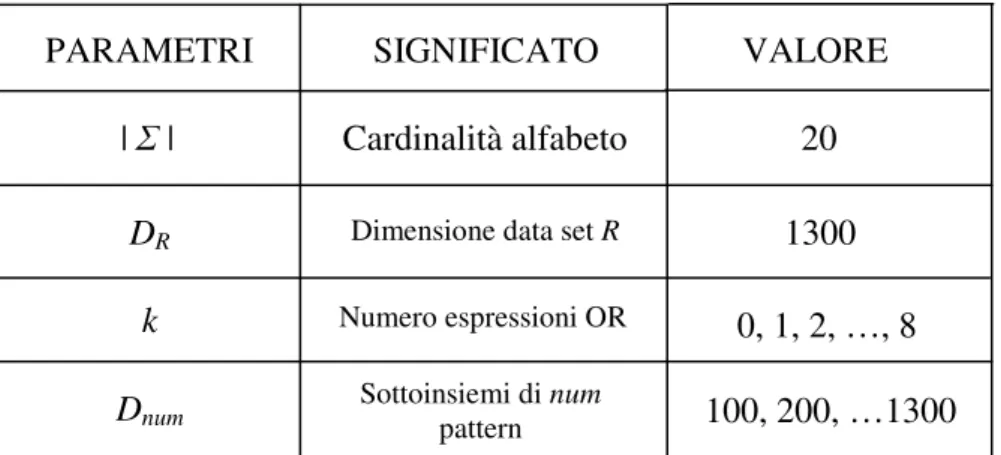
Come si può ben intuire, il numero totale di intervalli è destinato a crescere in funzione dei valori di k scelti. Di seguito mostriamo un grafico che riporta il numero totale degli intervalli (calcolati considerando il data set di dimensione DR) che si ottengono al variare del parametro k.
Più i valori di k crescono e maggiore è la precisione con cui si rappresenta l’insieme R di espressioni preso in considerazione, ovvero migliora la densità dell’insieme di intervalli IR. Infatti, per valori alti del parametro k, il rapporto che definisce la densità è sempre più tendente a 1. Però allo stesso tempo, la scelta di
Numero totale di intervalli al variare del parametro k
0 10000 20000 30000 40000 50000 60000 70000 80000 90000 1 2 3 4 5 6 7 8 9 Valori di k N um er o di in te rv al li
valori alti di k va a scapito dello spazio di memoria occupato. Infatti, c’è un trade-off spazio- precisione coinvolto nella scelta del valore di k.
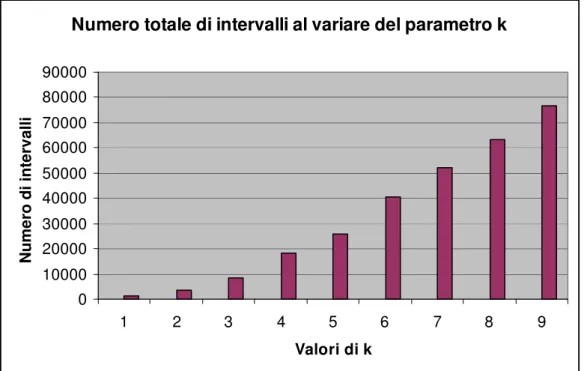
Per una generica query string qx contenuta in Qsuccesso, abbiamo calcolato, al variare del parametro k, il seguente rapporto:
dove, il numeratore, NI, indica il numero totale di intervalli in cui è contenuta qx e il denominatore, NO, indica il numero di pattern il cui linguaggio contiene la stringa qx. Per ciascun valore di k abbiamo calcolato il rapporto
e abbiamo costruito il grafico mostrato in fig. 6.3. NI NO (1)
Σ
qx ∈ Qsuccesso NIΣ
qx ∈ Qsuccesso NO (2)Valori del rapporto (2) al variare del parametro k
0 2 4 6 8 10 1 2 3 4 5 6 7 8 9 Valori di k V al or i d el r ap or to (2 )
Fig. 6.3 Valori del rapporto (2) relativi a Qsuccesso al variare di k
Dal grafico si può notare come il rapporto migliori, ovvero diminuisca, al crescere del valore di k. Questo accade perché all’aumentare di k aumenta la precisione con cui si rappresenta la generica espressione presente nel dataset.
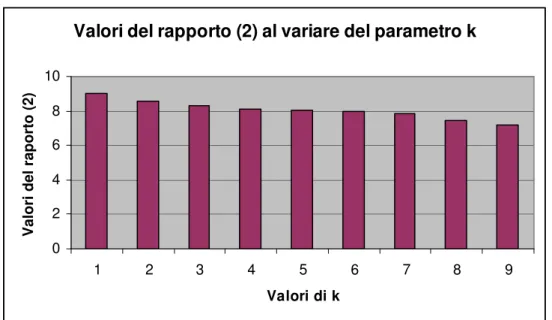
Similmente a quanto fatto per le stringhe appartenenti al file Qsuccesso, per ogni stringa appartenente a Qfallimento calcoliamo il valore del parametro NI. Quindi calcoliamo il rapporto espresso in (3) per k = 0, 1, 2, …, 8.
Il grafico esposto in fig. 6.4 mostra l’andamento di questi rapporti al variare del parametro k.
Anche per Qfallimento il rapporto migliora, ovvero diminuisce, al crescere del valore di k. Questo accade perché all’aumentare di k aumenta la precisione con cui si rappresenta la generica espressione presente nel dataset, di conseguenza diminuisce il numero di falsi positivi, appartenenti a Qfallimento, contenuti negli intervalli.
NI Ns
(3)
Valori del rapporto (3) al variare del parametro k
0 5 10 15 20 25 30 35 40 45 1 2 3 4 5 6 7 8 9 Valori di k V al or i d el r ap po rt o (3 )
Fig. 6.4 Valori del rapporto (3) relativi a Qfallimento al variare di k
Si può osservare dagli ultimi due grafici come l’idea di raffinare la ricerca esprimendo una espressione attraverso l’unione disgiunta di più intervalli non risulti proprio vincente, soprattutto se si prende in considerazione il valore di k=9. Infatti, a parità di crescita del numero di intervalli, entrambi i rapporti (2) e (3) migliorano di pochissimo.
Tutti i passi fatti sino a questo momento costituiscono la prima fase dell’esperimento. La seconda fase consiste nel considerare i valori mostrati nei grafici di fig. 6.3 e 6.4 e stabilire i valori k′ (per Qsuccesso) e k′′ (per
Qfallimento) in base al quale ripetere gli esperimenti, facendo variare però, questa volta, il numero di espressioni che costituiscono il data set.
La scelta del valore k′ viene effettuata sia tenendo conto dei valori esposti in fig. 6.3 sia tenendo conto dei valori esposti in fig. 6.2, col fine di individuare un buon compromesso tra spazio e precisione. Il valore di questo parametro viene stabilito empiricamente, guardando i dati a disposizione. Per cui, al fine di proseguire con la conduzione degli esperimenti, abbiamo stabilito un valore di k′ pari a 3.
La scelta del valore k′′ viene effettuata tenendo conto dei valori esposti in fig. 6.2 e fig. 6.4. Anche in questo caso il valore di k′′ viene stabilito empiricamente. Il valore che abbiamo scelto per proseguire gli esperimenti è k′′ = 7.
Come esposto nella sezione 6.1, consideriamo tredici sottoinsiemi Dnum di R. Ciascun sottoinsieme è costituito da num pattern generati in modo del tutto casuale. Si ottengono quindi i seguenti sottoinsiemi: D100, D200, D300,..., D1300.
Naturalmente al variare dei sottoinsiemi variano anche le stringhe contenute in Qsuccesso.
Fissato il valore k′, per ogni query string qx, contenuta in Qsuccesso, ricalcoliamo, al variare di Dnum, il rapporto esposto nella (1).
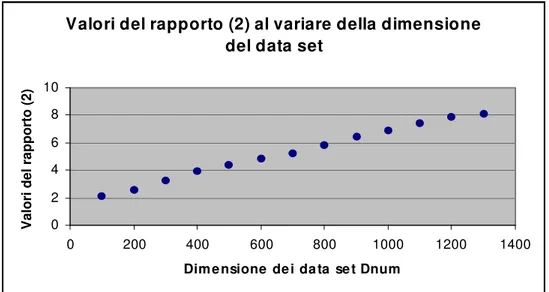
Quindi, mostriamo in fig. 6.5, il grafico contenente l’andamento dei rapporti calcolati (secondo la formula (2)) al variare di Dnum.
Fissato il valore k′′, per ogni query string qx, contenuta in Qfallimento, ricalcoliamo, al variare di Dnum, il rapporto esposto nella (3). In fig. 6.6 mostriamo il grafico contenente l’andamento dei rapporti calcolati al variare di Dnum.
Valori del rapporto (3) al variare della dimensione del data set 0 5 10 15 20 25 30 35 0 200 400 600 800 1000 1200 1400
Dimensione del data set
V al or i d el r ap po rt o (3 )
Fig.6.6 Valori del rapporto (3) relativi a Qfallimento al variare di Dnum
Valori del rapporto (2) al variare della dimensione del data set
0 2 4 6 8 10 0 200 400 600 800 1000 1200 1400
Dimensione dei data set Dnum
V al or i d el r ap po rt o (2 )
Si può notare come la crescita dei rapporti calcolati in fig.6.5 e 6.6 non sia proprio costante, ma abbastanza lenta. Comunque, guardando attentamente gli ultimi due grafici proposti, si può notare come i due rapporti si stabilizzino intorno a data set contenenti tra le 1100 e 1300 espressioni.
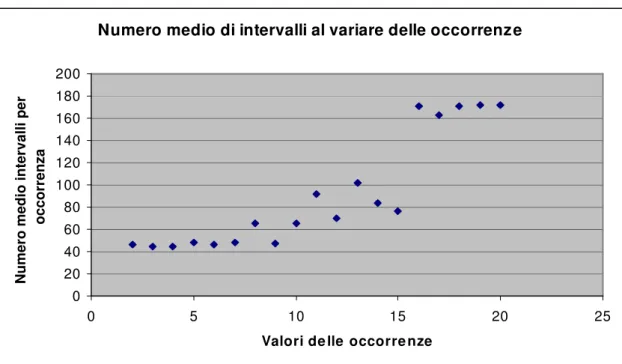
Nel grafico che segue mostriamo il numero medio di intervalli, in cui una query string qx (appartenente a Qsuccesso) è contenuta, calcolati al variare del numero delle occorrenze ottenute.
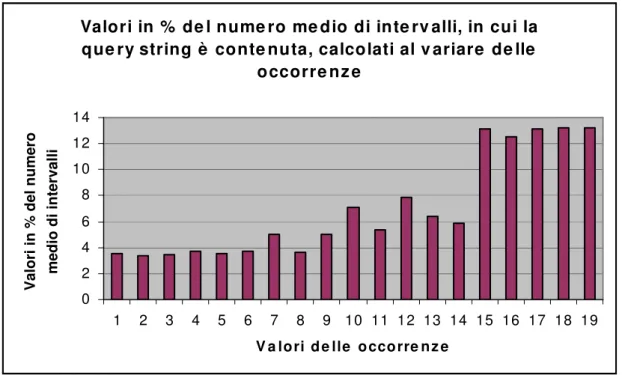
Nella pagina che segue mostriamo lo stesso grafico solo che il valore del numero medio di intervalli è espresso in percentuale.
(Per motivi di spazio il grafico è riportato nella pagina seguente)
Numero medio di intervalli al variare delle occorrenze
0 20 40 60 80 100 120 140 160 180 200 0 5 10 15 20 25
Valori delle occorrenze
N um er o m ed io in te rv al li pe r oc co rr en za
Fig. 6.7 Valori del numero medio di intervalli in cui la query string in ingresso è contenuta, calcolati al variare del numero delle occorrenze
Dal grafico si può notare come un approccio basato sulla ricerca in intervalli convenga poiché rispetto alla scansione sequenziale, che prevede la scansione totale dell’insieme R, non si esaminano mai tutti gli intervalli appartenenti all’insieme IR (ovvero il 100%), infatti il grafico mostra come la percentuale del numero medio di intervalli visitati sia in alcuni casi inferiore all’8% e in altri inferiore al 14%.
Valori in % de l nume ro me dio di inte rv alli, in cui la que ry string è conte nuta, calcolati al v ariare de lle
occorre nze 0 2 4 6 8 10 12 14 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19
V a lori de lle occorre nz e
V al or i i n % d el n um er o m ed io d i i nt er va lli
Fig. 6.8 Valori del numero medio di intervalli (espresso in %) in cui la query string in ingresso è contenuta, calcolati al variare del numero