4. RISULTATI
4.1 Costruzione delle librerie sottrattive
Nel nostro lavoro ci siamo avvalsi della tecnica dell’SSH. Questo metodo permette di ottenere banche di cDNA arricchite di trascritti differenzialmente espressi, presenti nei due campioni comparati. La caratteristica di questa tecnica è la particolare efficienza nell’isolare sequenze rare poco espresse. Per la costruzione della banca abbiamo utilizzato il PCR-Select
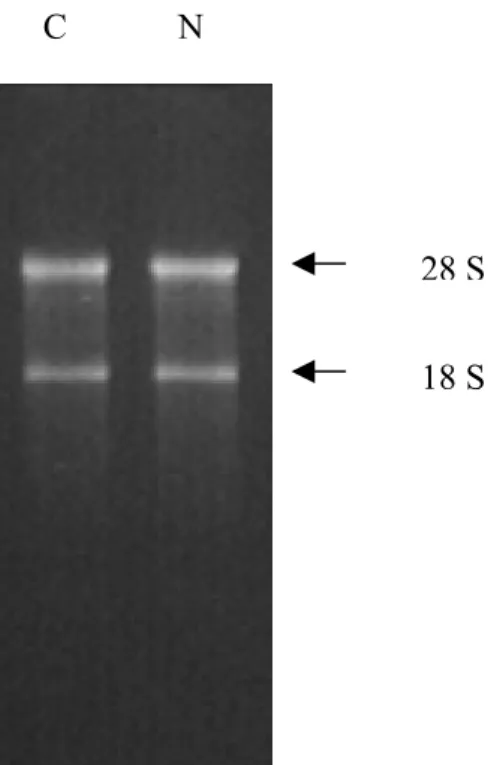
cDNA Subtraction Kit (BD Biosciences). Dopo aver estratto l’RNA totale dai nostri campioni e valutato la sua qualità su gel denaturante (fig. 8).C N
Fig. 8 : Analisi elettroforetica
su gel denaturante dell’RNA tot estratto dalle porzioni medio-temporali dei cervelli dei ratti condizionati (C) e dei ratti di controllo (N).
28 S 18 S
Dopo l’isolamentodell’mRNA poliA+ (vedere par. 3.4- Materiali e Metodi) abbiamo effettuato un controllo qualitativo e quantititativo dei nostri campioni, tramite RT-PCR, amplificando sia i geni ribosomali 5.8, 18 e 28 S (risultati non mostrati) che il gene G3PDH. Come stampo abbiamo utilizzato il cDNA ottenuto dalla retrotrascrizione sia dell’RNA totale che del poliA+.
Come si può vedere dalla fig. 9 la quantità dei nostri campioni è paragonabile e quindi si può procedere con la costruzione delle banche sottrattive.
C N
L 1 2 3 4 5 6 7 8
Fig.9: Controllo quantititativo del cDNA ottenuto tramite RT-PCR utilizzando i primer
della G3PDH e come stampo, il poliA+ del condizionato (C) e del controllo (N); lane 1-4 prodotto di amplificazione del campione condizionato;lane 5-8 prodotto di amplificazione del campione di controllo; lane 1-4 cicli 18; lane 2-5 cicli 22; Lane 3-7 cicli 26; Lane 4-8 cicli 30
Tramite l’SSH è stato quindi possibile ottenere due librerie sottrattive di cDNA: la libreria Forward, contenente i trascritti dei geni modulati positivamente dal condizionamento alla paura contestuale e la libreria reverse, costituita invece dai trascritti dei geni modulati negativamente.
Parallelamente alla costruzione delle due librerie sottrattive è stata effettuata una libreria sottrattiva di riferimento, utilizzando come driver il cDNA ottenuto da mRNA poliA+ di muscolo scheletrico
muscolo scheletrico digerito e da DNA di ΦX174 digerito con l’enzima HaeIII (forniti dal kit) .
Nella fig. 10 sono mostrati rispettivamente gli amplificati della libreria di controllo e delle due librerie sottrattive, forward e reverse. Come si può apprezzare, la sottrazione effettuata nei campioni di controllo è avvenuta in modo corretto in quanto la libreria in questione risulta costituita da bande discrete che coincidono con quelle del DNA di ΦX174 digerito con l’enzima HaeIII (marker IX).
1 2 3 4 5 6 7
Fig.10: Elettroforesi su gel di agarosio (2%) delle librerie e dei non sottratti ottenuti.
Lane 1 libreria forward sottrata; Lane 2 libreria non sottrato forward; Lane 3 libreria di
controllo del muscolo scheletrico sottratto; Lane 4 libreria di controllo del muscolo scheletrico non sottrato; Lane 5 libreria riverse; Lane 6 libreria reverse non sottratto; Lane 7 marker IX.
Terminata la costruzione delle due librerie sottrattive è stata valutata l’efficienza della sottrazione, come suggerito dal protocollo adottato (vedere par. 3.7-Materiali e Metodi).
Abbiamo effettuato una PCR con i primer della G3PDH aumentando progressivamente il numero dei cicli, e utilizzando come stampo in un caso la libreria sottrattiva e nell’altro il corrispondente non sottratto. La sottrazione di entrambe le librerie è avvenuta correttamente visto
che i segnali del gene costitutivo sono sempre presenti nei campioni non sottratti mentre appaiono solo a cicli elevati nelle librerie; questo è indice di assenza o forte riduzione dello stampo rispetto al non sottratto. Nella figura 11 è riportata come esempio la prova di efficienza di sottrazione della libreria forward .
L 1 2 3 4 5 6 7 8 M
Fig.11: Valutazione della prova di efficienza di sottrazione per la libreria forward:
prodotti di PCR utilizzando come primer 5’G3PDH e 3’G3PDH forniti dal kit e come stampo la II PCR della libreria forward (lane 1,2,3,4)e la II PCR del non sottrato della libreria forward (lane 5,6,7,8);lane 1-5 18 cicli; lane 2-6 23 cicli; lane 3-7 28 cicli; lane 4-8 33 cicli;lane M marker
I cDNA della libreria forward sono stati clonati in un vettore di clonaggio T/A (TOPO TA Cloning® kit , Invitrogen; e trasferiti in microtetraplates da 96 pozzetti; complessivamente sono stati raccolti circa 600 cloni positivi.
La tecnica della SSH è un metodo molto efficiente per ottenere librerie sottrattive e garantisce un notevole arricchimento di trascritti differenzialmente espressi; anche se i campioni sottratti possono ancora contenere alcuni cDNA comuni al tester e al driver. Questo implica che alcuni cloni delle due librerie non siano differenzialmente espressi. Lo screening primario permette di eliminare questi falsi positivi.Questo studio riporta dati relativi allo screening primario di circa 400 cloni.
4.2 Risultati screening primario
Per verificare che la libreria in questione fosse rappresentativa i nostri inserti sono stati amplificati tramite una PCR da colonia, utilizzando primers complementari alle regioni presenti ai due lati del sito di clonaggio (vedi par. 3.7- Materiali e metodi). I prodotti sono stati analizzati su gel di agarosio (2%). Il risultato ottenuto, come può essere osservato dalla fig. 12, mostra che il clonaggio è avvenuto in modo corretto. Inoltre, dal momento che i frammenti amplificati presentano tutti pesi molecolari diversi possiamo affermare che la maggior parte dei cloni non è ridondante e che la libreria forward è altamente rappresentativa.
Lane 1 20 M Fig.12: Verifica della qualità dei cloni della libreria forward; lane 1-20
amplificazione con PCR da colonia dei cloni; lane M: marker IX (Roche).
Per il momento sono stati selezionati solo i cloni che presentano un amplificato unico. Dopo denaturazione, 1 µl del prodotto di PCR è stato trasferito su due membrane Hybond N+. Le membrane sono state ibridate utilizzando come sonde i cDNA delle librerie sottrattive marcate non radioattivamente (vedere par. 3.8.3.- Materiali e metodi). Nella fig. 13 sono mostrati i risultati che abbiamo ottenuto dall’ibridazione di due membrane su cui era stato trasferito il prodotto della PCR di cloni della libreria forward. La membrana 1 è stata
ibridata con la sonda costituita dal cDNA della libreria forward (T-C), mentre la membrana 2 è stata ibridata con la sonda costituita dal cDNA della libreria reverse (C-T). Sono stati selezionati i cloni che mostrano un segnale positivo con una sonda ma non con l’altra, o che, comunque, mostrano un segnale differente nell’ibridazione con le due sonde. Questi, infatti, sono i cloni che contengono trascritti di geni candidati ad essere differenzialmente espressi. I risultati sono interpretati secondo il protocollo descritto nel PCR-Select Differential Screening Kit User Manual (BD Biosciences).
Fig. 13: Screening differenziale di cloni appartenenti alla libreria forward; la membrana 1 è stata ibridata con la sonda C-N; la membrana 2 è stata ibridata con la sonda N-C. Sono evidenziati alcuni cloni candidati ad essere differenzialmente espressi 1 .
4.3 ANALISI DELLE SEQUENZE
Nella tabella www sono mostrate alcune delle sequenze emerse dallo screening primario, identificate mediante comparazione con le sequenze depositate in banca dati (GenBank).
METABOLISMO MITOCONDRIALE
1VA6 ATP synthase subunit 6
1VE2 cytochrome oxidase subunit I
1VE9 NADH dehydrogenase subunit 5-6
1vF6 Rattus norvegicus gene for 12S
2VF2 cytochrome b mRNA,
2VG3 cytochrome c oxidase subunit I
TRASPORTO E RILASCIO DELLE VESCICOLE
1VH5 complexin 1
1VC1 coatomer protein complex, subunit beta 2
2VA6 amphiphysin (Amph)
2VB3 reg I binding protein I (Rbp1)
2VB10 ARP3 actin-related protein 3 homolog (Actr3)
2VC7 N-ethylmaleimide sensitive fusion protein attachment protein alpha
2VE11 GTP-binding protein SAR1b
TURNOVER PROTEICO
1VA11 tripartite motif protein 32 (Trim32)
2vH1 cullin 3
1VB12 proteasome maturation factor
TRASDUZIONE DEL SEGNALE INTRACELLULARE 1VB9 phosphoprotein enriched in astrocytes 15 (PEA-15)
1VC10 Type-Ia regulatory subunit of cAMP dependent protein kinase
1VB4 KIAA0974 protein
2VG1 tyrosine 3-monooxygenase/tryptophan
5-monooxygenase activation protein, zeta polypeptide Tab.4: tabella riassuntiva cloni isolati.
I geni individuati sono stati suddivisi in base alle funzioni svolte dai loro prodotti. In questo studio in particolare sono stati analizzati i cloni 1VA11, 1VB12 e 1VC10.
4.3.1 CLONE 1VA11
La sequenza del clone 1VA11 è:
5’….ACTTTGTCCCACGTAGAATAGATGATGCTCTTAGCAGAAGTTGC TTAGAAGTCAGGCACTTCAAGGCTTTAGTAGAGAGAACCACTTTAGC CCTTTGCCCTTGTGTTTGAACTTTGTATTGGTATAGAGTAGTCATCGA TCTTTCCCTTTGGACTCTGAGGTCCCTAGTCTACTGTCCTCTTCCTGTT ACAGTGTGCTTCATCTGTGGCTTTAACTCTGGCTGCATTCTGTGTGCA TGCTCCAGTGGGATCTGCTCAACCCGTCAGTGATATTATATCTCTGTA GCATGGTGTCTCCTTTGATCACCTTTACCAGTGTGAGACTGTATCATT AAGTGTCACTAAGGTGACACAAAGTCCCTTTGGGAAGCAGGACCTCT TGACTGGGAATTTCTGCCAACATAGATTGTCTTCTCTTGCAGATGTGT GAGAGGATGGTTTGACTGACCCTAGTTGGATAGTGTTGAACACTTGA GTCTGAAATCACTTGCCTTTTAAAGGAGTTTCTAGGTTAGAATTATGC CATAGCCAAGGCAGATAGGAACAGGTAAGCCTTGCTAACATATCCAG CAAACCTGGATATTTTTTAGGGCTCAGAATGTTTCATACTCCTTCCAT TGACCTATGCTAAGAGTTTTTGGAAGAATTGGATTATGTGGGCTTCG ACCAACAATGCGGGGGAAGGTCGACGTATCCATTTTCATTACGGAAG AACAAACCATTAGGTAGCTAACTCTGTATTTTCTTCAAGTGAGTGCAT ATTGTTTGACTTTGAGATTTCTTTCTGTTCTATGAAGCATTAAACTAA TAAAACTAGTAACTTCCAAAAAA….3’
Fig.11: Sequenza nucleotidica del clone 1VA11.
La sequenza ottenuta è stata confrontata con le sequenze depositate in banca dati (GenBank), utilizzando programmi come FASTA e BLASTN. Di seguito sono riportati i dati ottenuti con BLASTN.
gi|51858706|gb|BC081980.1|Rattus norvegicus tripartite motif protein 32, mRNA, complete cds
gi|58865775|ref|NM_001012103.1|Rattus norvegicus tripartite motif protein 32 (Trim32), mRNA
La sequenza del clone in analisi mostra una similarità del 99% con le sequenze BC081980.1 e NM_001012103.1 .La sequenza con più alta similarità con il nostro campione è rappresentata da una porzione della regione 3’-UTR dell’mRNA codificante per la Tripartite Motif 32 protein. La sequenza codificante del gene in questione è infatti
compresa fra i nucleotidi 137 e 2104. Di seguito è mostrato l’allineamento con una delle sequenze nucleotidiche:
4.2.3 CLONE 1VB12 La sequenza del clone 1VB12 è:
gi|58865775|ref|NM_001012103.1| Rattus norvegicus tripartite motif protein 32 (Trim32), mRNA
Score = 1584 bits (799), Expect = 0.0
Identities = 818/823 (99%), Gaps = 1/823 (0%) Strand=Plus/Plus 1VA11 1 ACTTTGTCCCACGTAGAATAGATGATGCTCTTAGCAGAAGTTGCTTAGAAGTCAGGCACT 60 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2333 ACTTTGTCCCACGTAGAATAGATGATGCTCTTAGCAGAAGTTGCTTAGAAGTCAGGCACT 2392 1VA11 61 TCAAGGCTTTAGTAGAGAGAACCACTTTAGCCCTTTGCCCTTGTGTTTGAACTTTGTATT 120 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2393 TCAAGGCTTTAGTAGAGAGAACCACTTTAGCCCTTTGCCCTTGTGTTTGAACTTTGTATT 2452 1VA11 121 GGTATAGAGTAGTCATCGATCTTTCCCTTTGGACTCTGAGGTCCCTAGTCTACTGTCCTC 180 |||||||||||||||| ||||||||||||||||||||||||||||||||||||||||||| Sbjct 2453 GGTATAGAGTAGTCATTGATCTTTCCCTTTGGACTCTGAGGTCCCTAGTCTACTGTCCTC 2512 1VA11 181 TTCCTGTTACAGTGTGCTTCATCTGTGGCTTTAACTCTGGCTGCATTCTGTGTGCATGCT 240 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2513 TTCCTGTTACAGTGTGCTTCATCTGTGGCTTTAACTCTGGCTGCATTCTGTGTGCATGCT 2572 1VA11 241 CCAGTGGGATCTGCTCAACCCGTCAGTGATATTATATCTCTGTAGCATGGTGTCTCCTTT 300 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2573 CCAGTGGGATCTGCTCAACCCGTCAGTGATATTATATCTCTGTAGCATGGTGTCTCCTTT 2632 1VA11 301 GATCACCTTTACCAGTGTGAGACTGTATCATTAAGTGTCACTAAGGTGACACAAAGTCCC 360 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2633 GATCACCTTTACCAGTGTGAGACTGTATCATTAAGTGTCACTAAGGTGACACAAAGTCCC 2692 1VA11 361 TTTGGGAAGCAGGACCTCTTGACTGGGAATTTCTGCCAACATAGATTGTCTTCTCTTGCA 420 ||||||||||||||||||||||||||||||||||| |||||||||||||||||||||||| Sbjct 2693 TTTGGGAAGCAGGACCTCTTGACTGGGAATTTCTGACAACATAGATTGTCTTCTCTTGCA 2752 1VA11 421 GATGTGTGAGAGGATGGTTTGACTGACCCTAGTTGGATAGTGTTGAACACTTGAGTCT-G 479 |||||| ||||||||||||||||||||||||||||||||||||||||||||||||||| | Sbjct 2753 GATGTGCGAGAGGATGGTTTGACTGACCCTAGTTGGATAGTGTTGAACACTTGAGTCTAG 2812 1VA11 480 AAATCACTTGCCTTTTAAAGGAGTTTCTAGGTTAGAATTATGCCATAGCCAAGGCAGATA 539 ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2813 AAATCACTTGCCTTTTAAAGGAGTTTCTAGGTTAGAATTATGCCATAGCCAAGGCAGATG 2872 1VA11 540 GGAACAGGTAAGCCTTGCTAACATATCCAGCAAACCTGGATATTTTTTAGGGCTCAGAAT 599 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2873 GGAACAGGTAAGCCTTGCTAACATATCCAGCAAACCTGGATATTTTTTAGGGCTCAGAAT 2932 1VA11 600 GTTTCATACTCCTTCCATTGACCTATGCTAAGAGTTTTTGGAAGAATTGGATTATGTGGG 659 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2933 GTTTCATACTCCTTCCATTGACCTATGCTAAGAGTTTTTGGAAGAATTGGATTATGTGGG 2992 1VA11 660 CTTCGACCAACAATGCGGGGGAAGGTCGACGTATCCATTTTCATTACGGAAGAACAAACC 719 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 2993 CTTCGACCAACAATGCGGGGGAAGGTCGACGTATCCATTTTCATTACGGAAGAACAAACC 3052 1VA11 720 ATTAGGTAGCTAACTCTGTATTTTCTTCAAGTGAGTGCATATTGTTTGACTTTGAGATTT 779 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 3053 ATTAGGTAGCTAACTCTGTATTTTCTTCAAGTGAGTGCATATTGTTTGACTTTGAGATTT 3112 1VA11 780 CTTTCTGTTCTATGAAGCATTAAACTAATAAAACTAGTAACTT 822 ||||||||||||||||||||||||||||||||||||||||||| Sbjct 3113 CTTTCTGTTCTATGAAGCATTAAACTAATAAAACTAGTAACTT 3155
4.3.2 CLONE 1VB12
La sequenza del clone 1VB12 è:
Fig.: : sequenza nucleotidica del clone 1VB12
5’….CGCAGAGCTGTTTCCAAGATGAACGCCAGAGGCCTTGGGTCGCAG CTGAAGGACAGTATTCCAGTTACAGAACTCTCAGCCAGTGGGCCTTTC GAGAGTCATGATCTTCTCCGGAAAGGGTTTTCTTGTGTGAAAAATGAA CTTTTGCCCAGTCACCCTCTTGAATTATCAGAAAAAAATTTCCAGCTCA ACCAGGACAAGATGAACTTTTCCACCCTGAGGAACATCCAGGGCCTGT TTGCTCCCCTGAAGTTACAGATGGAGTTCAAGGCTGTGCAGCAGGTTC AGCGTCTTCCGTTTCTTCCGAGCTCAAACCTCTCACTGGATATTTTGAG GGGCAACGACGAGACCATTGGTTTTGAGGATATTCTTAATGATCCATC ACAAAGTGAACTGATGGGCGAGCCACACATGATGGTGGAGTATAAGCT GGGCTTGCTGTGACGCAGGGCGCCGTGCAGGAACCACGGATGTGCTTC TCGTAGTTGCCTCTGCCGAGGTCTGATGTTCACAGCATTAAAGT ….3’
Fig.12: Sequenza del clone 1VB12.
La sequenza ottenuta è stata confrontata con le sequenze depositate in banca dati (GenBank), utilizzando programmi come FASTA e BLASTN. Di seguito sono riportati i dati ottenuti con BLASTN.
gi|13385085|ref|NM_025624.1| Mus musculus proteasome maturation protein (Pomp), mRNA
Length=735
La sequenza del clone in analisi mostra una similarità del 94% con la sequenza sopra mostrata. La sequenza con più alta similarità con il nostro campione è rappresentata dalla regione codificante e una porzione della regione 3’ UTR del mRNA codificante per la proteasome maturation protein.
La sequenza codificante del gene in questione è infatti compresa fra i nucleotidi 63 e 486.
Di seguito è mostrato l’allineamento con la sequenze nucleotidica sopra riportata:
gi|13385085|ref|NM_025624.1| Mus musculus proteasome maturation protein (Pomp), mRNA
Length=735
Score = 632 bits (319), Expect = 3e-178 Identities = 415/440 (94%), Gaps = 0/440 (0%) Strand=Plus/Plus 1VB12 3 CAGAGCTGTTTCCAAGATGAACGCCAGAGGCCTTGGGTCGCAGCTGAAGGACAGTATTCC 62 |||||||||||| ||||||||||||||||| ||||||||| ||||||||||||||||||| Sbjct 47 CAGAGCTGTTTCAAAGATGAACGCCAGAGGTCTTGGGTCGGAGCTGAAGGACAGTATTCC 106 1VB12 63 AGTTACAGAACTCTCAGCCAGTGGGCCTTTCGAGAGTCATGATCTTCTCCGGAAAGGGTT 122 ||| | || ||||||||||| ||||||||||||||||| |||||||||||||||||||| Sbjct 107 AGTCGCGGAGCTCTCAGCCAGCGGGCCTTTCGAGAGTCACGATCTTCTCCGGAAAGGGTT 166 1VB12 123 TTCTTGTGTGAAAAATGAACTTTTGCCCAGTCACCCTCTTGAATTATCAGaaaaaaaTTT 182 ||||||||||||||||||||||||||||||||||||||| || |||||||||||||| || Sbjct 167 TTCTTGTGTGAAAAATGAACTTTTGCCCAGTCACCCTCTCGAGTTATCAGAAAAAAACTT 226 1VB12 183 CCAGCTCAACCAGGACAAGATGAACTTTTCCACCCTGAGGAACATCCAGGGCCTGTTTGC 242 ||||||||||||||||||||||||||||||||| ||||||||||||||||| |||||||| Sbjct 227 CCAGCTCAACCAGGACAAGATGAACTTTTCCACGCTGAGGAACATCCAGGGTCTGTTTGC 286 1VB12 243 TCCCCTGAAGTTACAGATGGAGTTCAAGGCTGTGCAGCAGGTTCAGCGTCTTCCGTTTCT 302 ||||||||||||||||||||| |||||||| |||||||||||||| ||||| |||||||| Sbjct 287 TCCCCTGAAGTTACAGATGGAATTCAAGGCAGTGCAGCAGGTTCACCGTCTCCCGTTTCT 346 1VB12 303 TCCGAGCTCAAACCTCTCACTGGATATTTTGAGGGGCAACGACGAGACCATTGGTTTTGA 362 ||||||||||||||||||||||||||||||||||||||||| ||||||||||||||||| Sbjct 347 CCCGAGCTCAAACCTCTCACTGGATATTTTGAGGGGCAACGATGAGACCATTGGTTTTGA 406 1VB12 363 GGATATTCTTAATGATCCATCACAAAGTGAACTGATGGGCGAGCCACACATGATGGTGGA 422 ||||||||||||||||||||||||||||||||||||||| ||||| ||| |||||||||| Sbjct 407 GGATATTCTTAATGATCCATCACAAAGTGAACTGATGGGTGAGCCCCACGTGATGGTGGA 466 1VB12 423 GTATAAGCTGGGCTTGCTGT 442 |||||||||||||||||| Sbjct 467 ACATAAGCTGGGCTTGCTGT 486
4.2.4 CLONE 1VC10
La sequenza del clone 1VC10 è:
5’… CGGACGTTGGAGTTACTTAGACAACAGGCTCCAGAGAGAAGGGAA TGGAGGCAGTGTTGCTAAGGTGGATGGAGTGTAGTGTCCAGAAAC AGGAAGCTGTGTGTCTGGGTTTATGTTACCAGGTGGGTAGGGCGTG GCTGTGCTGTGGTCTACTTTCTTTATTGTTTTTTGTTTGTTTGTGTGT GTGTGTGTTTAAAGTAAACTTGTGTTCTCAACTGTATTCCAGTATTT TCCACCCTTATGTGTTACATTATTCCAATGATAACCCAACAGTTTAT TTTTATTACTGTTATTTTTTAAACAAAATTCACAGTTCGTAACTAGG CATGTTTATCTTGTGACTTATGT … 3’
Fig.13: sequenza nucleotidica del clone 1VC10
La sequenza ottenuta è stata confrontata con le sequenze depositate in banca dati (GenBank), utilizzando programmi come FASTA e BLASTN. Di seguito sono riportati i dati ottenuti con BLASTN.
gi|74181583|dbj|AK151040.1| Mus musculus bone marrow macrophage cDNA, RIKEN full-length enriched library, clone:I830020P13
product:protein kinase, cAMP dependent regulatory, type I, alpha, full insert sequence
gi|74181378|dbj|AK150927.1| Mus musculus bone marrow macrophage cDNA, RIKEN full-length enriched library, clone:I830018G14
product:protein kinase, cAMP dependent regulatory, type I, alpha, full insert sequence
La sequenza del clone in analisi mostra una similarità del 94% sia con la sequenza AK151040.1 che con la sequenza AK150927.1..
La sequenza con più alta similarità con il nostro campione è rappresentata da una porzione della regione 3’UTR dell’mRNA
codificante per la proteina Type-I
α
Regulatory Subunit Of cAMP Dependent Protein Kinase.La sequenza codificante del gene in questione è infatti compresa fra i nucleotidi 137 e 910 della sequenza AK151040.1. Di seguito è mostrato l’allineamento con questa sequenza
gi|74181583|dbj|AK151040.1| Mus musculus bone marrow macrophage cDNA, RIKEN full-length enriched
library, clone:I830020P13 product:protein kinase, cAMP dependent regulatory, type I, alpha, full insert sequence Length=2764
Score = 228 bits (115), Expect = 1e-56 Identities = 139/147 (94%), Gaps = 0/147 (0%) Strand=Plus/Plus Query 195 AAAGTAAACTTGTGTTCTCAACTGTATTCCAGTATTTTCCACCCTTATGTGTTACATTAT 254 |||||||||||||||||| |||||||| |||||||||||| |||||||||||||||||| Sbjct 1818 AAAGTAAACTTGTGTTCTTGACTGTATTTCAGTATTTTCCAGCCTTATGTGTTACATTAT 1877 Query 255 TCCAATGATAACCCAACAGTTTATTTTTATTACTGTTATTTTTTAAACAAAATTCACAGT 314 |||| ||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 1878 TCCAGTGATAACCCAACAGTTTATTTTTATTACTGTTATTTTTTAAACAAAATTCACAGT 1937 Query 315 TCGTAACTAGGCATGTTTATCTTGTGA 341 ||||||| ||||| |||||||||||| Sbjct 1938 TCGTAACGAGGCACATTTATCTTGTGA 1964
Score = 60.0 bits (30), Expect = 5e-06 Identities = 43/46 (93%), Gaps = 1/46 (2%) Strand=Plus/Plus
Query 28 GCTCCAGAGAGAAGGGAATGGAGGCAGTGTTGCT-AAGGTGGATGG 72 |||||||||||||||| |||||||||||| |||| ||||||||||| Sbjct 1591 GCTCCAGAGAGAAGGGGATGGAGGCAGTGCTGCTGAAGGTGGATGG 1636
4.3 RISULTATI DELL’ANALISI DI ESPRESSIONE TRAMITERT-PCR RELATIVA
L’analisi di espressione dei cloni 1VA11, 1VB12 e 1VC10 è stata effettuata tramite la tecnica della RT-PCR relativa, utilizzando come controllo interno il gene costitutivo G3PDH. In particolare è emerso che il consolidamento del CFC è associato ad una modulazione positiva della trascrizione dei geni della Trim motif protein 32 (TRIM 32) fig. 14, della proteasome maturation protein (POMP) fig. 15 e della Type-I
α
Regulatory Subunit Of cAMP Dependent Protein Kinase (PKAr) fig. 16.C N IX C N 0.00 0.25 0.50 0.75 1.00 1.25 1 V A 11/G 3 P D H G3PDH TRIM32 A B
Fig.14 A) RT-PCR relativa e B) Livelli di espressione relativa del trascritto della Tripartite Motif 32; il gene costitutivo non ha mostrato differenze significative tra controllo e trattato; i segnali gene-specifico sono stati normalizzati con i segnali corrispondenti del gene costitutivo (Mann-Whitney, P <0.05).
Fig. 15:A) RT-PCR relativa e B) Livelli di espressione relativa del trascritto di
proteasome maturation factor; il gene costitutivo non ha mostrato differenze
significative tra controllo e trattato; i segnali gene-specifico sono stati normalizzati con i segnali corrispondenti del gene costitutivo. La differenza è significativa (T test P< 0,05). A B C N IX G3PDH PKA G3PDH POMP C N 0.0 0.1 0.2 0.3 0.4 0.5 0.6 0.7 1V C 1 0 /G 3 P D H C N 0.0 0.1 0.2 0.3 0.4 0.5 0.6 1V B 1 2 /G 3 P D H A B
Fig.16:A) RT-PCR relativa e B) Livelli di espressione relativa del trascritto della Type-Iα Regulatory Subunit Of cAMP Dependent Protein Kinase; il gene
costitutivo non ha mostrato differenze significative tra controllo e trattato; i segnali gene-specifico sono stati normalizzati con i segnali corrispondenti del gene costitutivo. La differenza è significativa (T test, P< 0,05).