61
5. PARTE SPERIMENTALE E RISULTATI
5.1 Amplificazione
La resa e la specificità dell’amplificazione dei codoni 12 e 13 del gene KRas e dell’esone 15 del gene BRAF sono state verificate su un gel di agarosio all’1,5%. Oltre ai campioni in esame sullo stesso gel sono stati caricati un controllo di reazione negativo ed il marker di peso molecolare da 100pb.
Il gel di agarosio relativo all’amplificazione dei codoni 12 e 13 del gene KRas di alcuni campioni è mostrato in figura 15, dove si possono osservare le corse elettroforetiche relative ai prelievi analizzati di alcuni casi di carcinoma colorettale della casistica in esame. Il controllo negativo non presenta tracce di amplificazione a confermare che la PCR è priva di contaminazioni; mentre gli altri campioni presentano un’unica banda da 186 bp, lunghezza dell’amplicone di KRas, ad indicare che l’amplificazione è avvenuta in maniera specifica.
Tutte le corse elettroforetiche eseguite per la verifica dell’amplificazione dei codoni 12 e 13 di KRas hanno mostrato gli stessi risultati.
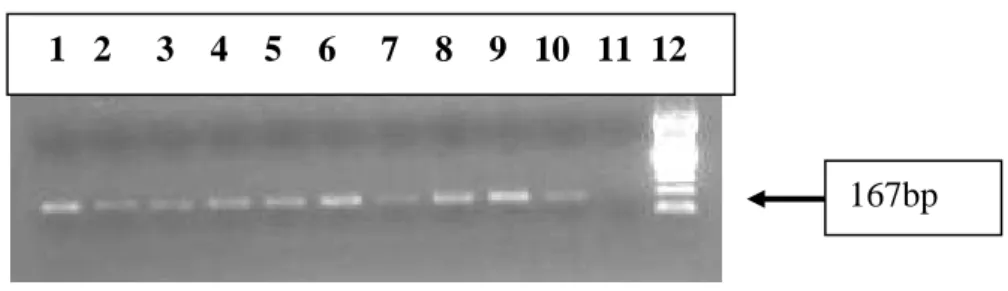
Figura 15: Esempio di corsa elettroforetica relativa ai codoni 12 e 13 di Kras. Dal pozzetto 1 al pozzetto 11 sono stati caricati i campioni da analizzare; nel pozzetto 12 è stato caricato il controllo negativo; mentre nel pozzetto 13 è stato caricato il marker da 100bp.
1 2 3 4 5 6 7 8 9 10 1112 13
62 Nella figura 16 è riportato il gel di agarosio dei campioni amplificati per l’esone 15
del gene BRAF.
Le bande ottenute mostrano che l’amplificazione dell’esone di interesse è avvenuta in maniera specifica e che non c’è stata contaminazione. Infatti si evidenzia un’unica banda che, confrontata con le bande del marker, risulta avere una lunghezza di 167bp, lunghezza dell’amplicone dell’esone 15 di BRAF, mentre in corrispondenza del controllo negativo non si nota alcuna traccia di amplificazione.
In tutte le corse elettroforetiche eseguite per la verifica dell’amplificazione dell’esone 15 del gene BRAF è stato osservato lo stesso profilo di bande.
Figura 16: Esempio di corsa elettroforetica relativa all’esone 15 di BRAF. Nei pozzetti dall’1 al 10 sono stati caricati i campioni da analizzare; nel pozzetto 11 è stato caricato il controllo negativo; mentre nel pozzetto 12 è stato caricato il marker da 100 bp.
1 2 3 4 5 6 7 8 9 10 11 12
63
5.2 Risultati di KRas
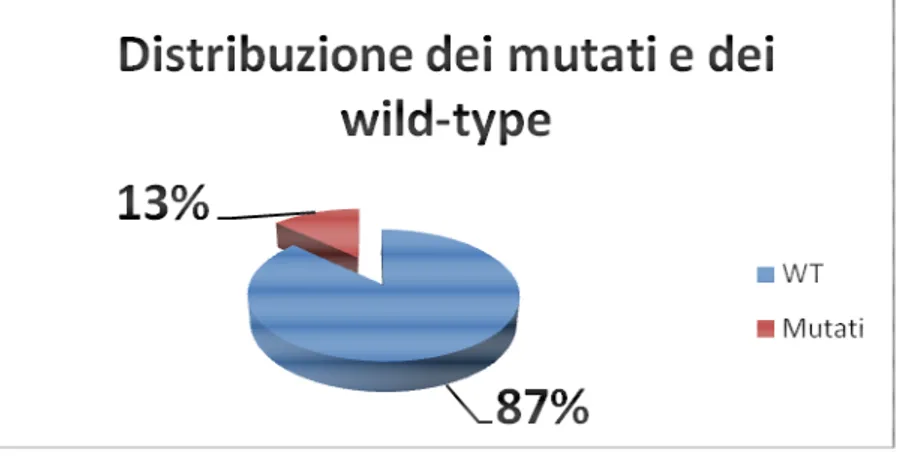
L’analisi mutazionale dei codoni 12 e 13 del gene KRas è stata eseguita, su 70 tumori, mediante la tecnica del sequenziamento genomico diretto, dei quali, 20 (29%, 20/70) sono risultati essere mutati e 50 (71%, 50/70) sono risultati essere wild-type. Considerando la distribuzione delle mutazioni dei codoni 12 e 13 del gene KRas, cioè considerando la presenza/assenza di mutazione, nei 31 pazienti arruolati nello studio, 14 pazienti sono risultati wild-type in tutti i prelievi analizzati (45%, 14/31), e 17 pazienti (55%, 17/31) sono risultati essere mutati in almeno uno dei tumori presi in esame (grafico1).
Grafico1: Distribuzione dei casi mutati e wild-type nella casistica presa in esame.
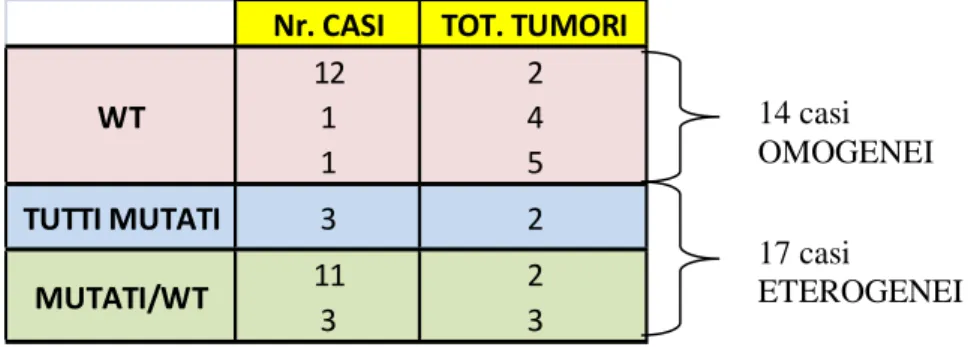
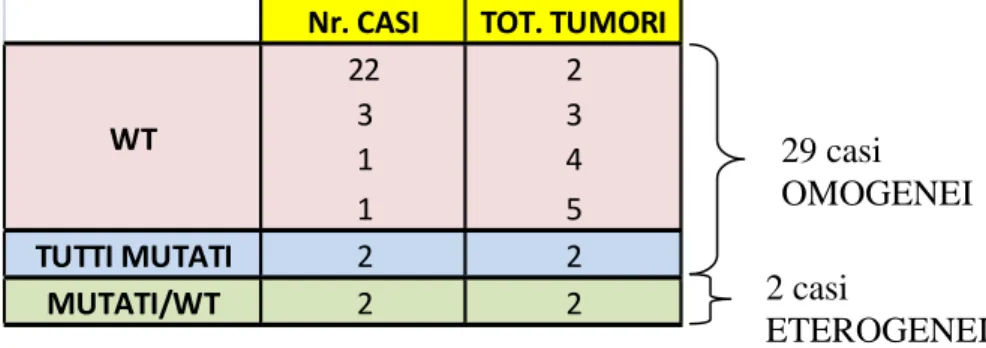
All’interno dell’intera casistica si è osservata una distribuzione di mutazione omogenea nei 14 casi in cui tutti i tumori all’analisi mutazionale sono risultati wild-type, mentre si è osservata una distribuzione di mutazione eterogenea nei 17 casi dei quali, 14 hanno riportato la presenza contemporanea di tumori wild-type e tumori mutati e 3 casi in cui, i tumori presenti in ciascun paziente riportavano mutazioni diverse tra loro (tabella1).
64
Nr. CASI TOT. TUMORI
12 2 1 4 1 5 TUTTI MUTATI 3 2 11 2 3 3 WT MUTATI/WT
Tabella 1: Casi omegenei ed eterogenei per lo stato mutazionale dei codoni 12 e 13 dell’esone 2 del gene Kras. 14 casi sono risultati omogenei, in particolare questi casi sono risultati essere wild-type in tutti i tumori analizzati (12 casi che presentavano 2 tumori, 1 caso che presentava 4 tumori e 1 caso che presentava 5 tumori); 17 casi sono risultati eterogenei o per la presenza di tumori wt e mut (14 casi) o per la presenza di tumori con mutazioni diverse (3 casi) nello stesso paziente (3 casi che presentavano 2 tumori; 11 casi che presentavano 2 tumori e 3 casi che presentavano 3 tumori).
I 3 pazienti che sono risultati essere mutati in tutti i tumori che presentavano al momento della diagnosi, sebbene fossero omogenei per la presenza di mutazione, sono però risultati eterogenei per il tipo di mutazione (tabella 2).
ID PAZIENTE KRas
neoplasia asp mucinosi G12S
neoplasia G12V adeno colon mucinoso G12V
adeno colon G13D
adeno sigma retto G12V
adeno sigma retto G12D
3
6
7
Tabella 2: Risultati dell’analisi mutazionale dei codoni 12 e 13 dell’esone 2 del gene Kras. Sono riportati i 3 casi in cui i tumori sincroni all’analisi mutazionale sono risultati tutti mutati, riportando tutti mutazioni diverse.
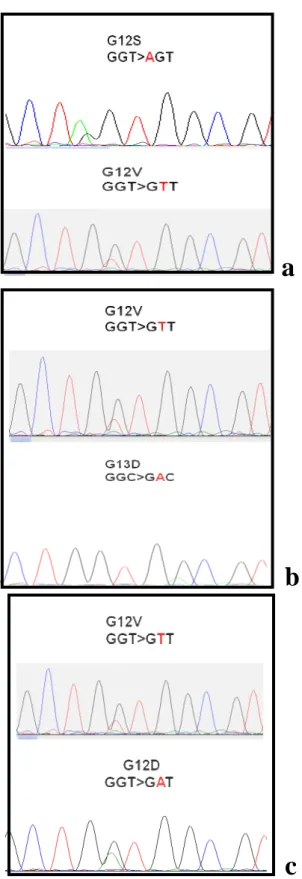
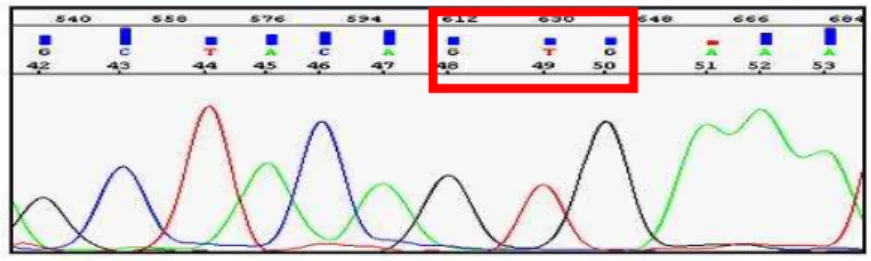
In figura 17, inoltre, sono riportati gli elettroferogrammi dei tre casi in esame, in cui si osservano le diverse mutazioni riscontrate in ciascun paziente.
14 casi OMOGENEI
17 casi
65
a
b
c
Figura 17: Elettroferogrammi dei tre pazienti (a, b, c) che hanno riportato tutti i tumori sincroni mutati.
66 Considerando nel dettaglio le mutazioni riscontrate in tutti i tumori analizzati, dei 20
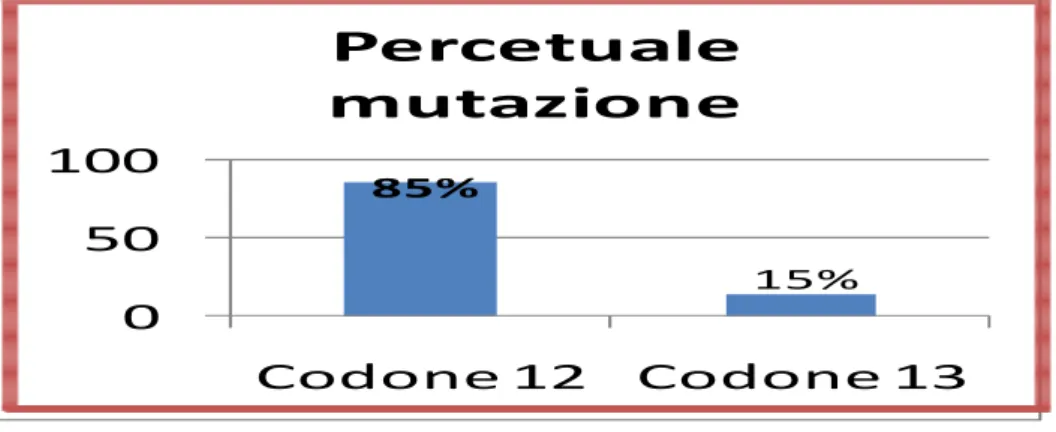
mutati, 17 (85%, 17/20) presentavano la mutazione a livello del codone 12 e 3 (15%, 3/20) a livello del codone 13 (grafico2); in tutti i casi le mutazioni trovate erano presenti in eterozigosi.
Grafico 2: Percentuali della distribuzione delle mutazioni a carico dei codoni 12 e 13 dell’esone 2 del gene KRas.
In particolare, nel codone 13 è stata riscontrata solamente la mutazione G13D, che consiste nella transversione di una guanina con un’adenina (G>A) a livello della seconda base del codone 13, che, a livello della proteina, determina la sostituzione di un residuo di glicina con uno di acido aspartico (figura18);
G13D
Figura 18: Elettroferogramma della mutazione G13D. Consiste nella transversione di una guanina con un’adenina (G>A) a livello della seconda base del codone 13, che, a livello della proteina, determina la sostituzione di un residuo di glicina con uno di acido aspartico. 85% 15% 0 50 100 Codone 12 Codone 13
Percetuale
mutazione
67 Mentre, nel codone 12, sono stati osservati diversi tipi di mutazioni, delle quali le più
frequenti sono la mutazione G12V e la mutazione G12D, riscontrate rispettivamente nel 25% e nel 30% dei casi. Entrambi le mutazioni consistono nella sostituzione del nucleotide guanina (G) in seconda posizione del codone 12 con una timina (T) e una adenina (A) rispettivamente (figura 19), con conseguente sostituzione a livello proteico di un residuo aminoacidico di glicina con uno di valina (V) e uno di acido aspartico (D) rispettivamente.
G12V
G12D
Figura 19: Elettroferogrammi delle mutazioni G12V e G12D. Entrambi consistono nella sostituzione del nucleotide guanina (G) in seconda posizione del codone 12 con una timina (T) e una adenina (A) rispettivamente, con conseguente sostituzione a livello proteico di un residuo aminoacidico di glicina con uno di valina (V) e uno di acido aspartico (D) rispettivamente.
Le altre mutazioni riscontrate sono: la mutazione G12A nel 10% dei casi, che consiste nella transversione di una guanina con una citosina (G>C) a livello della seconda base del codone 12, la quale determina, a livello della proteina, la sostituzione di un residuo aminoacidico di glicina con uno di alanina; la mutazione G12S nel 15% dei casi, che consiste nella transversione di una guanina con una adenina (G>A) a livello della prima base del codone 12, la quale determina, a livello della proteina, la sostituzione di un residuo aminoacidico di glicina con uno di serina; ed infine in un caso (5%) è stata osservata la mutazione G12C, che consiste
68 nella transversione di una guanina con una timina (G>T) a livello della prima base
del codone 12, la quale determina, a livello della proteina, la sostituzione di un residuo di glicina con un residuo di cisteina (grafico 3).
Grafico 3: Distribuzione delle mutazioni a carico del codone 12 del gene KRas.
Nell’istogramma è riportata la frequenza delle mutazioni G12A, G12S, G12V, G12D e G12C riscontrate nella casistica analizzata.
69
5.3 Risultati BRAF
L’analisi mutazionale dell’esone 15 del gene BRAF, è stata eseguita mediante un’analisi mutazionale di primo livello utilizzando la tecnica SSCP, che, mediante l’impiego di due campioni di riferimento rappresentati dalle linee cellulari di tiroide TPC ed ARO, rispettivamente wild-type e mutata V600E per l’esone 15 di BRAF, ci ha permesso di identificare i campioni wild-type e mutati.
In figura 20 è riportata un’SSCP nella quale è possibile osservare campioni con differenti pattern di migrazione, alcuni identici a quello del controllo negativo per la mutazione V600E di BRAF (cellule TPC), perciò ad una prima analisi considerati wild-type, (in figura i campioni 1,2,3,4 e 8), altri, invece, identici a quello del controllo positivo della mutazione V600E di BRAF (cellule della linea ARO) considerati, dunque, mutati (in figura 20 i campioni 5,6 e 7).
70
Figura 20: SSCP. I campioni 1, 2, 3, 4 e 8 hanno un pattern di migrazione identico al controllo negativo per la mutazione V600E di BRAF (campione 10, freccia gialla); i campioni 5, 6 e 7 riportano un pattern di migrazione uguale al controllo positivo per la mutazione V600E di BRAF (campione 9, freccia verde).
I campioni 1 e 2 sono due tumori di uno stesso caso, che, come si osserva, è risultato essere wild-type; i campioni 3 e 4 rappresentano sempre due prelievi di uno stesso caso, che riporta lo stesso risultato del precedente caso; i campioni 5 e 6 rappresentano anch’essi due prelievi di uno stesso caso, che, come si osserva è risultato essere mutato in maniera omogenea, in quanto entrambi i campioni mostrano un pattern di migrazione alterato; infine i campioni 7 e 8 rappresentano sempre due prelievi di uno stesso caso, risultato, come si osserva, avere una distribuzione eterogenea della mutazione V600E, in quanto un campione mostra un pattern di migrazione wild-type (campione 8) e l’altro mostra un pattern di migrazione alterato (campione 7).
Comunque, i risultati ottenuti dallo screening mutazionale eseguito con la tecnica di SSCP sono stati confermati mediante sequenziamento genomico diretto, eseguito per tutti i campioni che presentavano un pattern di migrazione alterato e per alcuni di quelli con pattern di migrazione identico al controllo wild-type. I risultati dell’SSCP sono stati confermati in quanto, quelli con pattern di migrazione alterato sono risultati tutti mutati V600E, mentre gli altri sono risultati tutti wild-type.
Dei 70 tumori analizzati, 64 (91%, 64/70) sono risultati wild-type e 6 (9%, 6/70) mutati.
71 Dei 31 casi analizzati, 27 casi (87%, 27/31) sono risultati essere wild-type, mentre 4
casi (13%, 4/31) sono risultati essere mutati (V600E) (grafico4).
Grafico 4: Distribuzione dei casi mutati e wild-type nell’analisi mutazionale dell’esone 15 del gene BRAF.
Considerando la distribuzione della mutazione V600E, dei 31 pazienti analizzati, essa è risultata essere eterogenea in 2 casi (6%, 2/31), che presentavano sia tumori mutati (V600E) che tumori wild-type (tabella, 6), ed omogenea in 29 casi (94%, 29/31) (tabella 7) dei quali 27 sono risultati wild-type e 2 mutati in tutti i tumori.
ID PAZIENTE BRAF
adeno sigma retto WT
adeno colon V600E
adeno colon V600E
adeno cieco WT
8
25
Tabella 6: Risultati analisi mutazionale dell’esone 15 del gene BRAF. Sono riportati tutti i casi che presentavano almeno uno dei tumori mutato a carico dell’esone 15 del gene BRAF.
72
Nr. CASI TOT. TUMORI
22 2 3 3 1 4 1 5 TUTTI MUTATI 2 2 MUTATI/WT 2 2 WT
Tabella 7: Risultati dell’analisi mutazionale dell’esone 15 del gene BRAF. 29 casi sono risultati omogenei, di cui 27 wild-type (22 casi presentavano 2 tumori, 3 casi 3 tumori, 1 caso 4 tumori e 1 caso 5 tumori) e 2 mutati per ciascun tumore presente in ogni singolo paziente; 2 casi sono risultati eterogenei per la presenza contemporanea di tumori wild-type e mutati (2 pazienti ciascuno con 2 tumori)
In figura 21 è riportata una porzione di sequenza dell’esone 15 del gene BRAF, dove nel riquadro rosso è stata evidenziata la sequenza mutata.
Tra le mutazioni a carico dell’esone 15 di BRAF, in questo studio, l’unica riscontrata è stata la mutazione V600E. La mutazione consiste nella transversione di un timina con un’adenina (T>A) a livello del nucleotide 1799 (1799) che determina, a livello della proteina, la sostituzione di una valina con un residuo di acido glutammico in posizione 600 della sequenza aminoacidica (V600E).
Figura 21: Elettroferogramma. In rosso è evidenziato il nucleotide mutato a livello del codone 600, evidenziato dal riquadro rosso. La mutazione è caratterizzata dalla presenza contemporanea, a livello della seconda base del codone 600, di due picchi, uno rosso che corrisponde al nucleotide wild-type, la timina, e l’altro verde che indica la presenza del nucleotide mutato, l’adenina.
29 casi OMOGENEI 2 casi
73 In figura 22 è riportata, invece, una porzione di sequenza dell’esone 15 del gene
BRAF, dove è stata evidenziata, nel riquadro rosso, la sequenza wild-type.
Figura 22: Elettroferogrammi: In figura è stata riportata la sequenza wild-type del codone 600 dell’esone 15 del gene BRAF.
74
5.4 Risultati KRas e BRAF
5.4.1 Mutazioni mutuamente esclusive
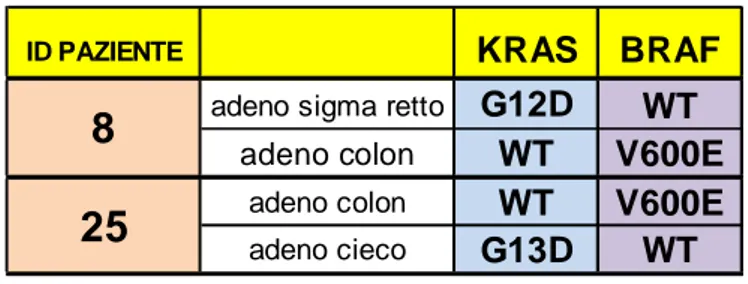
Come riportato in letteratura, la maggior parte dei casi mutati per il gene BRAF non presentano mutazioni a carico del gene KRas e, precisamente, meno dell’1% dei casi mutati per BRAF risultano essere mutati anche per KRas, infatti, in molti tumori le mutazioni di KRas e di BRAF sono eventi mutualmente esclusivi (24, 26). Tale evento è stato possibile osservarlo anche nel mio studio. Infatti, in tabella 8 sono riportati due casi riscontrati nell’intera casistica presa in esame, dove si può osservare la mutuale esclusività delle mutazioni a carico dei geni KRas e BRAF.
ID PAZIENTE KRAS BRAF
adeno sigma retto G12D WT
adeno colon WT V600E
adeno colon WT V600E
adeno cieco G13D WT
8
25
Tabella 8: Risultati KRas e BRAF. Sono riportati i 2 casi in cui si osserva come le mutazioni a carico di tali geni sono mutuamente esclusive, quando un tumore sincrono risulta essere mutato a carico di uno dei due geni, non lo è a carico dell’altro gene.
75
5.4.2 Lo stato mutazionale delle metastasi
In questo studio, 17 pazienti presentavano metastasi linfonodali e/o a distanza, sulle quali è stato possibile eseguire l’analisi mutazionale, da cui è risultato che esse riportavano uno stato mutazionale identico a quello dell’adenocarcinoma da cui probabilmente derivavano.
Dunque, tale risultato, già riportato in altri studi, non è altro che un ulteriore conferma del fatto che le metastasi riportano lo stesso stato mutazionale del tumore primitivo dal quale si presume derivino.
Nello schema sottostante sono raggruppati nei riquadri le diverse situazioni riscontrate in questo studio:
riquadro I è riportata la situazioni, riscontrata in 5 casi, in cui sono presenti due adenocarcinomi e una metastasi che segue lo stato mutazionale dell’adenocarcinoma mutato;
riquadro II è riportata la situazione, verificatasi in 8 casi, in cui sono presenti due adenocarcinomi e una metastasi che qui però segue lo stato mutazionale dell’adenocarcinoma wild-type;
riquadro III è riportata la situazione, verificatasi in un solo caso, in cui sono presenti cinque adenocarcinomi e una metastasi che segue lo stato mutazionale di uno dei cinque adenocarcinomi;
riquadro IV è riportata la situazione, verificatasi in un solo caso, in cui sono presenti 2 adenocarcinomi che presentano mutazioni diverse e una metastasi che segue lo stato mutazionale di uno dei due;
riquadro V è riportata la situazione, verificatasi in 2 casi, in cui sono presenti tre adenocarcinomi e una metastasi che segue lo stato mutazionale di uno dei due adenocarcinomi wild-type;
riquadro VI è riportata la situazione, verificatasi in un solo caso, in cui sono presenti due adenocarcinomi e due metastasi che seguono lo stato mutazionale di uno dei due adenocarcinomi.
76
Schema rappresentativo dell’andamento della stato mutazionale delle metastasi. riquadro I è riportata la situazioni, riscontrata in 5 casi, in cui sono presenti due adenocarcinomi e una metastasi che segue lo stato mutazionale dell’adenocarcinoma mutato; riquadro II è riportata la situazione, verificatasi in 8 casi, in cui sono presenti due adenocarcinomi e una metastasi che qui però segue lo stato mutazionale dell’adenocarcinoma wild-type; riquadro III è riportata la situazione, verificatasi in un solo caso, in cui sono presenti cinque adenocarcinomi e una metastasi che segue lo stato mutazionale di uno dei cinque adenocarcinomi; riquadro IV è riportata la situazione,
WT WT WT WT WT WT WT WT WT WT WT WT WT WT WT WT M M M M M M M 5 CASI 8 CASI 1 CASO 2 CASI 1 CASO 1 CASO I VI V III II IV
77
verificatasi in un solo caso, in cui sono presenti 2 adenocarcinomi che presentano mutazioni diverse e una metastasi che segue lo stato mutazionale di uno dei due;
riquadro V è riportata la situazione, verificatasi in 2 casi, in cui sono presenti tre adenocarcinomi e una metastasi che segue lo stato mutazionale di uno dei due adenocarcinomi wild-type; riquadro VI è riportata la situazione, verificatasi in un solo caso, in cui sono presenti due adenocarcinomi e due metastasi che seguono lo stato mutazionale di uno dei due adenocarcinomi.
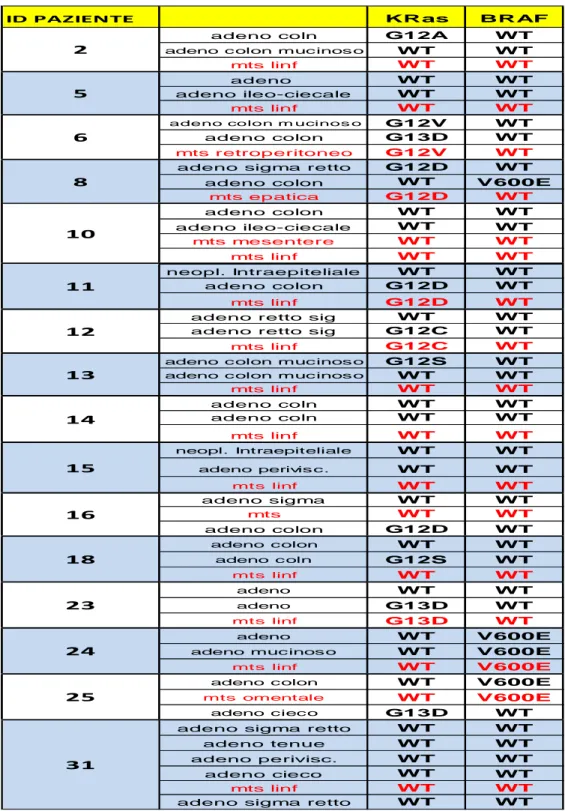
Nella tabella 9 sono riportati nel dettaglio tutti i casi che presentavano le metastasi. In rosso sono evidenziate le metastasi e il relativo stato mutazionale dei geni KRas e BRAF. Si osserva come lo stato mutazionale delle metastasi segue quello dell’adenocarcinoma dalla quale si presume derivi.
78
ID PAZIENTE KRas BRAF
adeno coln G12A WT adeno c olon muc inos o WT WT mts linf WT WT
adeno WT WT
adeno ileo-ciecale WT WT mts linf WT WT
adeno colon m ucinos o G12V WT
adeno colon G13D WT mts retroperitoneo G12V WT
adeno sigma retto G12D WT
adeno colon WT V600E mts epatica G12D WT adeno colon WT WT adeno ileo-ciecale WT WT mts mesentere WT WT mts linf WT WT neopl. Intraepiteliale WT WT adeno colon G12D WT mts linf G12D WT
adeno retto sig WT WT
adeno retto sig G12C WT mts linf G12C WT
adeno c olon muc inos o G12S WT adeno c olon muc inos o WT WT mts linf WT WT adeno coln WT WT adeno coln WT WT mts linf WT WT neopl. Intraepiteliale WT WT adeno perivis c. WT WT mts linf WT WT adeno sigma WT WT mts WT WT adeno colon G12D WT adeno colon WT WT adeno c oln G12S WT mts linf WT WT adeno WT WT adeno G13D WT mts linf G13D WT adeno WT V600E adeno muc inos o WT V600E
mts linf WT V600E
adeno colon WT V600E
mts omentale WT V600E
adeno ciec o G13D WT
adeno sigma retto WT WT
adeno tenue WT WT
adeno perivisc. WT WT
adeno cieco WT WT
mts linf WT WT
adeno sigma retto WT WT 31 16 18 23 24 25 11 12 13 14 15 2 5 6 8 10
Tabella 9: Sono riportati nel dettaglio tutti i casi che presentavano le metastasi.