1 INTRODUZIONE
1.1 Sviluppo del sistema nervoso nei vertebrati
Le prime fasi dello sviluppo del sistema nervoso dei vertebrati sono la conseguenza di un fenomeno di induzione neurale che avviene al momento della gastrulazione quando l’endo-mesoderma dorsale rilasciano molecole secrete che inducono a diventare neuroectoderma.
In seguito all’induzione neurale, avviene una fase di morfogenesi che prevede cambiamenti nella struttura del foglietto embrionale associati a movimenti cellulari, riarrangiamenti e cambiamenti nella forma delle cellule: questa fase prende il nome di neurulazione e nei vertebrati superiori è divisa in primaria e secondaria.
La neurulazione primaria prevede un ispessimento delle cellule dell’ectoderma dorsale, che diventano colonnari e formano la piastra neurale. In seguito la piastra neurale si flette e forma la “doccia neurale” al di sopra della notocorda, che deriva dal mesoderma dorsale. I margini della doccia neurale sono detti pliche neurali e si avvicinano progressivamente l’una all’altra fino a fondersi e formare il tubo neurale che sprofonda all’interno dell’embrione e rimane dorsalmente coperto dall’ectoderma non neurale (futura epidermide). La neurulazione secondaria è caratteristica della parte posteriore del tubo neurale di mammiferi, uccelli e consiste nella formazione di un cordone solido di cellule che viene cavitato in un secondo momento. La neurulazione nei pesci è simile alla neurulazione secondaria dei tetrapodi.
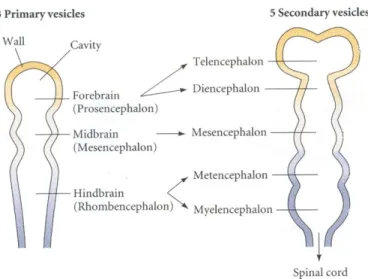
La piastra neurale precoce assomiglia ad uno strato omogeneo di cellule epiteliali, ma quando si chiude su se stessa a formare il tubo neurale una serie di costrizioni ne suddividono la parte più anteriore in vescicole che rappresentano i primordi di proencefalo, mesencefalo e romboencefalo, in senso rostro-caudale (stadio a tre vescicole). Posteriormente al romboencefalo il tubo neurale rimane un cilindro cavo precursore del midollo spinale.
Una successiva evoluzione del sistema nervoso anteriore è lo stadio a cinque vescicole in cui il proencefalo è ulteriormente regionalizzato in telencefalo e diencefalo e il romboencefalo è suddiviso in metencefalo e mielencefalo ( vedi Fig. 1.1).
Fig. 1.1- Rappresentazione schematica degli stadi di sviluppo del SNC detti a tre e cinque vescicole Gli esperimenti storici che hanno iniziato a spiegare i meccanismi dell’induzione neurale sono stati condotti su embrioni di anfibi, prima di Triturus e poi di Xenopus. Negli embrioni di anfibio, fin dai primi stadi di sviluppo (blastula), sono distinguibili un polo animale composto da piccole cellule e uno vegetativo composto da cellule più grandi, ricche di vitello. Al polo animale si trovano le cellule destinate a diventare ectoderma, al polo vegetativo quelle destinate a diventare endoderma e tra queste due regioni si trova una regione marginale che darà mesoderma. L’ectoderma dorsale è destinato a diventare il sistema nervoso. Alla gastrulazione, nella regione dell’endoderma immediatamente sotto il mesoderma dorsale, si ha la formazione del labbro dorsale del blastoporo, attraverso cui le cellule di mesoderma ed endoderma iniziano ad entrare nell’embrione per involuzione, mentre le cellule di ectoderma ne ricoprono la superficie.
Hans Spemann e Hilde Mangold nel 1924 dimostrarono come il trapianto della regione corrispondente al labbro dorsale del blastoporo, prelevato da un embrione di tritone allo stadio di gastrula precoce, in posizione ventrale in un altro embrione ospite allo stesso stadio, producesse lo sviluppo di una larva con due assi dorsali completi. In questo esperimento, la regione trapiantata si sviluppava in mesoderma dorsale, ma le altre componenti dorsali nell’asse secondario, come il sistema nervoso, erano indotte nell’ospite dall’azione del frammento trapiantato. Quest’ultima conclusione fu resa possibile dall’uso di due specie di tritone con diversa pigmentazione.
Il labbro dorsale del blastoporo, per le sue capacità dorsalizzanti ed induttive, fu chiamata “organizzatore primario” e poi “organizzatore di Spemann”. Per diverse decine di anni non si riuscirono ad individuare gli effettori molecolari dell’organizzatore, ma tra la fine degli anni ’80 e la metà degli anni ’90, grazie all’avvento delle tecniche di biologia molecolare e al cambiamento di sistema modello dagli urodeli (Triturus) agli anuri (Xenopus) si è iniziato a fare chiarezza. Nel 1989 Grunz e Tacke si accorsero che cellule prelevate dal polo animale di embrioni di Xenopus se messe in coltura dopo essere state dissociate differenziano in cellule di tipo nervoso, mentre gli animal caps non dissociati differenziano in cellule
epidermiche. Questo fece pensare che le cellule ectodermiche fossero programmate per default a dare neuroectoderma e che questo fosse permesso all’ectoderma dorsale e impedito a quello ventrale. Anni dopo, Hemmati-Brivanlou e Melton (1992), in uno studio sulla formazione del mesoderma, usarono un recettore tronco di tipo II per i fattori TGF-constatando che agiva da dominante negativo, e osservarono che bloccando la via di segnale dell’activina, oltre alla prevista inibizione dell’induzione mesodermica, si otteneva anche l’espressione di marcatori neurali.
Questo fece pensare che la via che bloccava la spontanea neuralizzazione dell’ectoderma ventrale dovesse dipendere da un fattore TGFche condivideva con l'activina il recettore di tipo II mutato nell’esperimento. Questo fattore epidermizzante era Bone Morphogenetic Protein (BMP) -4. Ci sono più di trenta proteine nella famiglia delle BMP; BMP-4 è la più coinvolta nell’inibizione neurale. Negli stessi anni furono scoperte tre molecole che erano espresse a livello dell’Organizzatore di Spemann e ne mimavano l’attività: noggin, chordin e follistatin. Ognuna di queste molecole ha effetto neuralizzante in quanto blocca la via di segnale di BMP: ad esempio Chordin è una proteina secreta che lega BMP-4 nello spazio extracellulare impedendogli di interagire coi suoi recettori (Piccolo et al. 1996).
La ridondanza di questi inibitori di BMP è, come spesso accade in biologia, prova indiretta dell’importanza dell’azione svolta.
Il doppio knock out di noggin e chordin nel topo porta a formazione di proboscide, ciclopia e assenza di mascelle; in alcuni embrioni alla mancata formazione delle strutture più anteriori del sistema nervoso centrale ( SNC) . Ben prima di queste scoperte di ordine molecolare era stato proposto un modello per lo sviluppo del sistema nervoso da Peter Nieuwkoop (1952): il modello di attivazione e trasformazione ( Fig 1.2).
Questo modello prevede un’ondata di attivazione che induce tutto l’ectoderma dorsale ad un destino neurale anteriore, i cui effettori sono appunto gli inibitori di BMP sopra menzionati, seguita da una trasformazione/ posteriorizzazione compiuta da molecole con gradiente di concentrazione postero-anteriore.
Fig. 1.2- Schema del modello di attivazione-trasformazione in Xenopus
Molecole posteriorizzanti sono: alcuni componenti della famiglia dei Fibroblast
Grouth Factor (FGF), l’acido retinoico e le proteine Wnt, omologhe di Wingless di Drosophila.
E’ stato anche dimostrato che, in Xenopus, FGF ha azione di induttore neurale diretto nelle fasi successive alla gastrulazione, ma di tipo posteriorizzante
La combinazione di inibitori di BMP (Follistatin) e FGF produce in espianti ectodermici l’accensione di geni del patterning neurale dai più anteriori a quelli del midollo spinale (Cox e Hemmati-Brivanlou 1995).
L’attività di induttore neurale posteriore di FGF è stata dimostrata anche nel pollo (Alvarez et al. 1998).
Altro agente posteriorizzante è l’acido retinoico (AR) molecola lipofilica che attraversa la membrana plasmatica ed interagisce con i suoi recettori citoplasmatici; questi sono fattori di trascrizione che in seguito al legame con l’AR divengono capaci di modulare l’espressione genica agendo su degli specifici enhancer RARE (Retinoic Acid Responsive Element).
L’acido retinoico agisce su geni importanti nel patterning rostro-caudale del SN principalmente su geni espressi posteriormente a partire dal romboencefalo: i geni
Hox ( Neiderreither et al. 2000).
L’ultima classe di fattori posteriorizzanti è quella delle proteine Wnt, che in fasi precoci dello sviluppo hanno effetto dorsalizzante sull’ectoderma (e altri foglietti) e solo dopo la gastrulazione hanno un azione posteriorizzante
Perché si sviluppino le porzioni più anteriori del sistema nervoso è necessario che tali regioni siano protette dai fattori posteriorizzanti: anteriormente al romboencefalo è espresso il gene del Citocromo P 26, che degrada l’acido retinoico, e una serie di inibitori di Wnt (Dkk, Frzb, Tlc ed altri) (Nihers et al. 1999 ), oltre a Cerberus, che contemporaneamente inibisce: BMP, Nodal e Wnt (Piccolo et al., 1999).
1.2 Patterning antero-posteriore del sistema nervoso centrale dei vertebrati
A seguito dell’induzione neurale si forma dunque già allo stadio di piastra neurale un
pattern antero-posteriore del SNC rappresentato dall’espressione di geni in domini
ristretti. Questi geni, alcuni dei quali ho già nominato, sono contemporaneamente causa ed effetto della regionalizzazione del sistema nervoso. La loro presenza in domini specifici è un effetto dell’espressione di altri geni che si trovano “a monte” nella gerarchia genica dello sviluppo del SNC (ad esempio gli inibitori di BMP e i fattori posteriorizzanti), ma è causa della successiva specificazione dei domini in cui sono espressi fino a diventare regioni distinte del SNC definiti i neuromeri ( Shimamura et al. 1995).
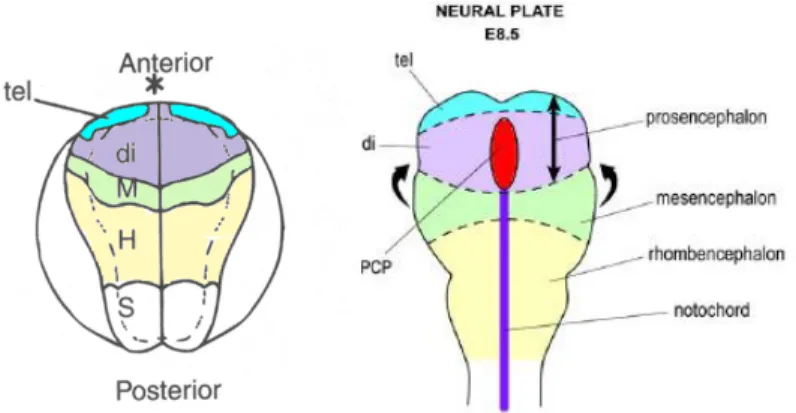
Nei vertebrati è possibile ricavare una mappa del destino delle principali regioni del SNC allo stadio di piastra neurale
Fig. 1.3 - Mappe del destino dei territori della piastra neurale di Xenopus e topo
In questo paragrafo descriverò come i geni espressi precocemente a livello di piastra neurale interagiscono per regionalizzare il SN in sviluppo, stabilendo regioni di confine tra i neuromeri e gli organizzatori secondari.
A partire dallo stadio di piastra neurale tardiva, nelle regioni più anteriori si accende il gene Bf1 a delineare le regioni del telencefalo da quelle del diencefalo, prima che si stabilisca un confine morfologico.
Tanto nei pesci quanto nei mammiferi c’è una regione dell’ectoderma non neurale, direttamente anteriore alla piastra neurale, che ha la funzione di indurre l’espressione di marcatori del telencefalo, “le cellule row1” e la anterior neural ridge ANR rispettivamente.
Le cellule della row 1 producono sia Fgf8 che Tlc,, un inibitore di Wnt che protegge le regioni più anteriori dall’effetto posteriorizzante.Otx2 è omologo al gene
Orthodenticle (Otd ) di Drosophila e codifica per un fattore di trascrizione con
omeodominio. E’ espresso nel proencefalo e nel mesencefalo dei vertebrati. La delezione di entrambi gli alleli di questo gene nel topo porta alla scomparsa delle strutture anteriori (Acampora et al. 1995), che possono essere recuperate se il gene deleto è sostituito con un costrutto in cui la coding region dell’ortologo di Drosophila è inserita tra le sequenze UTR, di Xotx2 che ne regolano la traducibilità del
messaggero (Acampora et al. 2001).
Il prodotto di Otx2 contribuisce, insieme a Gbx2, a posizionare uno degli organizzatori secondari del SN, posto al confine tra mesencefalo e romboencefalo: l’istmo.
Fgf8 oltre ad essere espresso nell’ectoderma anteriore al telencefalo è espresso anche
nel romboencefalo anteriore e media una parte dell’attività induttiva dell’istmo: se è espresso ectopicamente nel diencefalo caudale induce l’espressione di Fgf8, Wnt1 e
En2, creando così un secondo organizzatore dell’istmo e facendo acquisire al
diencefalo posteriore le caratteristiche di mesencefalo, ma con orientamento opposto rispetto alla normale polarità anteroposteriore ( Crossley et al.1996).
En2 è omologo al gene Engrailed di Drosophila; esso codifica per un fattore di
trascrizione con omeodominio. Nei vertebrati ci sono due geni engrailed En1 ed En2 entrambi espressi a “cavallo” della regione dell’istmo. La differenza tra i due geni è il timing dell’espressione, con En1 che è espresso più precocemente rispetto a En2.
Anche i geni Pax2, Pax5 e Pax8 sono necessari per mantenere l’identità dell’istmo: l’istmo è assente in topi knock out per Pax5 e nel mutante noi (no isthmus) di zebrafish in cui non è funzionante il gene pax2.1 che è l’ortologo dei tre geni pax appena nominati. L’espressione di En2 e Wnt1 è attivamente regolata dai geni Pax. Il gene Gbx2 è espresso nella porzione più anteriore del romboencefalo; il topo Gbx2 -/- manca del cervelletto, che si origina dalla porzione dorsale dei primi tre rombomeri, e il suo mesencefalo si estende posteriormente (Wassarman et al. 1997). I rombomeri sono gli otto compartimenti (r1-r8) in cui è suddiviso il romboencefalo, l’organizzazione segmentale del romboencefalo non si limita alla struttura anatomica esterna, ma anche alla sua organizzazione citologica. Infatti, neuroni che innervano i diversi archi branchiali hanno i loro corpi cellulari all’interno di questi rombomeri, distribuiti con una precisa periodicità: in particolare, i corpi cellulari dei neuroni branchiomotori presentano periodicità alternata, quelli degli interneuroni periodicità in registro coi singoli rombomeri.
Esperimenti di trapianti di rombomeri hanno dimostrato che i confini si creano tra rombomeri pari e dispari. Questo accade perché ci sono proprietà adesive diverse tra rombomeri alterni.
I rombomeri non si formano contemporaneamente: prima si formano i confini tra r3/r4 e tra r5/r6, e successivamente i restanti confini.
Sia i geni Hox che geni non Hox (come krox20 e Kreisler) sono coinvolti nella suddivisione del romboencefalo. I geni Hox, inoltre, sono coinvolti anche nello stabilire l’identità dei vari rombomeri. Krox 20 è espresso già allo stadio di piastra neurale in due aree corrispondenti ai futuri r3 e r5.
In mutanti in cui il gene Krox20 è mutato in entrambi gli alleli mancano r3 e r5 e si forma un territorio in cui i rombomeri pari adiacenti si fondono.
Alla base della formazione dei diversi compartimenti ci sono proprietà adesive e di
cell sorting diverse: nei rombomeri si alternano l’espressione di efrine (nei
rombomeri pari), e dei loro recettori (nei rombomeri dispari).
L’interazione ligando recettore fa iniziare, in entrambe le cellule, una via di segnale che porta alla repulsione delle stesse per cui si creano compartimenti separati.
Krox20 induce l’espressione di EphA4 un recettore dell’efrina B2.
L’identità dei rombomeri è specificata dai geni Hox così come i loro ortologhi nella
Drosophila ne specificano i segmenti.
I geni Hox nei vertebrati contribuiscono anche a creare i segmenti, non solo a attribuirgli un identità.
I geni Hox sono espressi nel SN dei vertebrati , a partire da r2, secondo il principio della colinearità spaziale.
In generale, i domini di espressione dei diversi geni Hox si spostano con una periodicità di due rombomeri: hoxb2 è espresso fino al confine r2/r3, Hoxb3 fino al confine r4/r5, Hoxb4 fino al confine r6/r7. Ci sono però delle eccezioni: ad esempio,
Hoxb1 è espresso solo in r4.
E’ stato osservato nel topo che la soppressione di Hoxb1 fa si che r4 assuma l’identità segmentale di r2 cioè le proiezioni dei neuroni brachimotori vanno ad innervare il primo arco branchiale invece del secondo (Studer et al. 1996) ; nel pollo invece è
stato fatto l’esperimento di guadagno di funzione infettando le cellule di r2 con un retrovirus contenente la sequenza di Hoxb1 in modo che il gene fosse costitutivamente attivo: come conseguenza le cellule di r2 si comportavano come quelle di r4 cioè le proiezioni dei neuroni brachimotori innervano il secondo arco branchiale invece del primo (Bell et al. 1999). Un guadagno di funzione di un gene
Hox porta in genere ad un cambiamento di identità in senso posteriore. I geni Hox
sono modulati dall’acido retinoico, di cui mediano l’effetto posteriorizzant (Marshall et al. 1996).
1.3 Xbh1 e il l suo pattern di espressione in Xenopus laevis
Xbh1 è ortologo, di Xenopus laevis, dei geni BarH1 e BarH2 di Drosophila. Questi
geni hanno diversi ruoli nello sviluppo del corpo dell’insetto: nell’occhio composito regolano la formazione delle cellule pigmentate dei fotorecettori R1 R6 (Higashijima et al. 1992) e dei fotorecettori R8 (Jarman et al. 1995); la formazione di microacheti, setole che vanno a comporre, insieme ai macroacheti, un organo di sensocomposto il
notum costituito da piccolissimi organi di senso, che possono assumere la forma di
peli e setole , microacheti e macroacheti. Questi sensilla sono organizzati in filari lungo i fianchi dell'animale, e sembrano fungere da organi tattili o da chemiorecettori. ; e specificano i segmenti distali della zampa (Sato et al. 1999)
Nei vertebrati gli omologhi di questi geni sono chiamati BarH-like ( Barhl) e sono divisi in due gruppi di paraloghi : Barhl1 e Barhl2, ma non è chiara l’esatta corrispondenza tra i due geni dei vertebrati e i geni BarH di Drosophila. Xbh1 appartiene, in base all’analisi della sequenza amminoacidica dedotta, al gruppo
Barhl2.
Xenopus laevis ha due paraloghi di questo gene: Xbh1 e Xbh2.
Xbh1 è un gene codificante per un fattore di trascrizione con omeodominio, altre
proteine con omeodominio sono le proteine Hox. L’omeodominio è un motivo di legame al DNA di 60 ammminoacidi molto conservato composto da tre -eliche che si organizzano tridimensionalmente per dare una struttura terziaria “ helix-turn-helix” in cui la terza elica prende contatto con specifiche sequenze di DNA.
Tutti i geni BarH, degli artropodi come dei vertebrati, appartengono ad un gruppo conservato di omeoproteine-Q50 che hanno un residuo di glutammina (Q) in posizione 50 dell’omeodomio.
A questo gruppo appartengono altri fattori di trascrizione di Drosophila: Engrailed,
Even-skipped e Antennapedia.
Un altro residuo amminoacidico caratteristico solo dell’omeodominio di BarH è una tirosina (Y) in posizione 49 al posto della più comune fenilalanina (F) .
Un'altro aspetto conservato tra geni BarH e Barhl è la presenza di domini FIL , ricchi in fenilalanina, isoleucina e leucina, che sono essenziali per l’attività di repressore trascrizionale, che sembra essere la principale caratteristica di questa proteina; a livello di questi domini avviene l’interazione con il co-repressore Groucho (Jimenez et al. 1999). Il numero di questi domini è variabile: in entrambi i geni di Drosophila
c’è un solo dominio FIL, mentre i geni Barhl ne possono contenere uno o due. Xbh1 ne ha due.
Mentre i geni BarH di Drosophila mostrano domini di espressione sovrapposti i paraloghi nei vertebrati mostrano domini di espressione distinti. Il pattern di espressione è variabile nei diversi organismi modello presi in esame. In generale, Barhl1 non è mai espresso a livello del telencefalo mentre Barhl2 non sembra avere delimitazioni in senso AP: nel topo, Mus musculus, Barhl2 si trova espresso dal telencefalo al midollo spinale (Reig et al 2007), anche se l’espressione a livello del diencefalo e più precisamente del prosomero 2, è conservata in pesci anfibi e mammiferi.
Un dominio di espressione che è prerogativa di Barhl2 è la retina, anche se il tipo di cellule della retina che specifica e il meccanismo attraverso cui agisce varia nei diversi organismi (Poggi et al. 2004; Mo et al. 2004).
Per quanto riguarda Xenopus laevis, il nostro modello sperimentale, il dominio di espressione di Xbh1 allo stadio di neurula (st 17) coincide con la regione di piastra neurale destinata a dare diencefalo posteriore. Allo stadio di bottone caudale (tailbud) (st. 27/28) continua ad essere espresso nel diencefalo posteriore (futuro talamo dorsale) ed inizia ad essere espresso nella zona dorsale interna della coppa ottica (retina neurale presuntiva). Da stadio 31 comincia ad essere espresso anche nel rombencefalo, nel mesencefalo a livello del futuro tetto ottico, e nella retina dove lo si trova anche a stadi successivi, ma nella quale la localizzazione dell’mRNA si restringe progressivamente allo strato delle cellule gangliari (Patterson et al. 2000; Poggi et al. 2004).
Successivamente all’analisi di Patterson sulla espressione di Xbh1 e Xbh2, è stato identificato, a stadi di neurula precoce, un dominio di espressione dorsale posteriore a livello mesodermico in prossimità della linea mediana (Offner et al. 2005).
1.4 Ruolo di Xbh1 nella formazione del sistema nervoso e nella retinogenesi di X.
laevis
E’ stato dimostrato da Offner et al. (2005) che il dominio di espressione mesodermico di Xbh1 è correlato con fenomeni di EPCD (early programmed cell death,) ovvero di morte cellulare programmata precoce, un meccanismo fisiologico che coinvolge le cellule del neuroectoderma per regolarne il numero.
La morte cellulare programmata o apoptosi è un meccanismo di controllo del numero e della qualità delle cellule nell’organismo in sviluppo presente nei diversi phyla di metazoi e dipende da una cascata proteolitica intracellulare mediata dalle caspasi che porta alla frammentazione del DNA, alla condensazione della cromatina e infine alla formazione di corpi apoptotici che espongono segnali di fagocitosi così da essere eliminati dai macrofagi. La morte cellulare programmata durante lo sviluppo non coinvolge solo il sistema nervoso, ma agisce in svariate parti dell’organismo ad esempio porta alla scomparsa delle membrane interdigitali.
Nel caso di Xbh1 è stato ipotizzato che il suo ruolo di repressore trascrizionale attivi la caspasi 3 innescando il programma di morte cellulare;non c’è ancora certezza se il meccanismo di attivazione della EPCD sia diretto o indiretto, ma c’è sicuramente una relazione non casuale. Ciò è stato dimostrato attraverso microiniezione dell’mRNA di Xbh1 in blastomeri dorsali di embrioni di Xenopus laevis e saggio TUNEL che permette di marcare le estremità dei frammenti di DNA degradati dalle caspasi.
Le cellule che vanno incontro a EPCD mediata da Xbh1 sono cellule del mesoderma che esprimono Chordin e Sonic hedgehog quindi gli effetti sul patterning della piastra neurale sono dovuti alla regolazione dei pathways di BMP e Shh, e cellule del diencefalo presuntivo l’altro dominio di espressione di Xbh1.
Un lavoro recente (Juraver-Geslin et al. 2010) propone Xbh1 come regolatore, a stadio di tailbud (st. 26/27) delle dimensioni del diencefalo, che è il principale dominio di espressione di Xbh1. La proteina agirebbe sempre come regolatore della Caspasi 3 che però in questo caso non opererebbe per indurre morte cellulare programmata, ma opererebbe una inibizione della -catenina. L’inattivazione del gene Casp3 nel topo causa una crescita eccessiva e disorganizzata del cervello, che appare principalmente nelle pareti del diencefalo (Kuida et al. 1996), un fenotipo simile a quello degli embrioni di Xenopus in cui la Caspasi 3 è eliminata attraverso l’uso di morpholino. Questo suggerisce che il ruolo della Caspasi 3 nel regolare la proliferazione delle cellule neuro epiteliali durante lo sviluppo del diencefalo, indipendentemente dal suo ruolo nell’apoptosi,sia conservato nella maggior parte dei vertebrati. Juraver-Geslin et al. ( 2010) affermano, in base ai dati ottenuti, che durante lo sviluppo del proencefalo la Caspasi3, direttamente o indirettamente, riduca l’attivazione del pathway canonico di Wnt. Il pathway canonico di Wnt ha come effetto il blocco della degradazione della catenina e quindi un suo accumulo intracellulare. E’ stato visto che un alterato livello, sia in aumento che in diminuzione della β-catenina intracellulare interferisce con la corretta formazione di giunzioni aderenti localizzate sull’apice delle cellule neuro epiteliali e disturbi nella formazione di queste giunzioni altera la struttura interna del neuro epitelio (Kadowaki et al. 2007).
Per quanto riguarda il ruolo di Xbh1 nella retina studi funzionali mediante lipofezione hanno dimostrato che Xbh1 indirizza i RPCs (Retinal Progenitor Cells) a diventare cellule gangliari e, contemporaneamente, ne reprime il differenziamento in fotorecettori ( Poggi et al., 2004).
Dagli studi effettuati si è arrivati a supporre che Xbh1 permetta il differenziamento in cellule gangliari agendo come repressore trascrizionale di un altro repressore. Più precisamente si ritiene che il fattore di trascrizione bHLH Xath5 inizi ad indurre il differenziamento dei progenitori retinici in cellule ganglionari attivando Xbh1 ed
Brn3d. Quest’ultimo è coinvolto nel regolare il differenziamento delle cellule
ganglionari e sarebbe mantenuto attivo da Xbh1, che reprimerebbe un repressore, ancora sconosciuto, di Brn3d (Poggi et al., 2004). Xath5 è l’ortologo del gene pro neurale della Drosophila atonal.
L’omologo di Xbh1 nel topo, Mbarhl2, promuove il differenziamento di RPCs in cellule amacrine a spese della popolazione di cellule di Müller con un meccanismo di
attivazione trascrizionale (Mo et al. 2004) inoltre Mbarhl2 agisce a valle di Atho7 e
Pou4f2 per controllare il differenziamento delle cellule gangliari ( Ding et al. 2009).
La relazione gerarchica tra i geni proneurali bHLH e BarH/barhl è conservata evolutivamente dalla Drosophila ai vertebrati; in alcuni casi sono i geni proneurali ad indurre l’espressione dei geni Bar come nella retina di Xenopus ( Poggi et al. 2004) e nei neuroni commissurali del midollo spinale del topo ( Saba t al. 2005), in altri casi i geni Bar regolano l’espressione dei geni bHLH attivandone o reprimendone la trascrizione come nel caso della formazione dei microacheti in Drosophila.
1.5 Sumoilazione di Xbh1
In uno screening col saggio di doppio ibrido, è stato isolato come possibile interattore della proteina Xbh1 il prodotto del gene SUMO-1. L’isolamento di SUMO-1 col doppio ibrido riflette probabilmente il fatto che Xbh1 è soggetta a sumoilazione. La sumoilazione consiste nella formazione di un legame covalente tra specifici residui di lisina di una proteina e la proteina Sumo. Sumo è una proteina presente in tutti gli eucarioti; nei mammiferi si conoscono 4 geni codificanti per forme diverse di questa proteina: Sumo-1, Sumo-2, Sumo-3 e Sumo-4, ma solo dei primi tre si è isolato il prodotto proteico.
Per quanto riguarda X.laevis si è trovato in banca dati sequenze annotate corrispondenti a Sumo-1 e Sumo-2 e diverse EST con un alto grado di omologia con Sumo-3.
Il nome Sumo è un acronimo di small ubiquitin-related modifier, dato che questo polipeptide assomiglia all’ubiquitina nella struttura tridimensionale e per il macchinario composto da tre enzimi che consentono la formazione del legame covalente, o isopeptidico, con il gruppo -amminico di residui di lisina.
La sequenza consensus riconosciuta, più frequentemente, dall’apparato di sumoilazione è KXE dove: con si intende un aminoacido con un residuo idrofobico ingombrante, ad esempio prolina; con K un residuo di lisina, a cui si attacca Sumo; con X un aminoacido qualunque; con E un residuo di acido glutammico.
Tuttavia questa sequenza è frequente in molte proteine non sumoilate ed inoltre è stato rilevato che Sumo si può legare a lisine non appartenenti a questo consensus. A differenza dell’ubiquitina, Sumo ha una tendenza minore a formare catene (mentre le catene di 4 o più residui di ubiquitina sono un forte segnale di degradazione proteica).
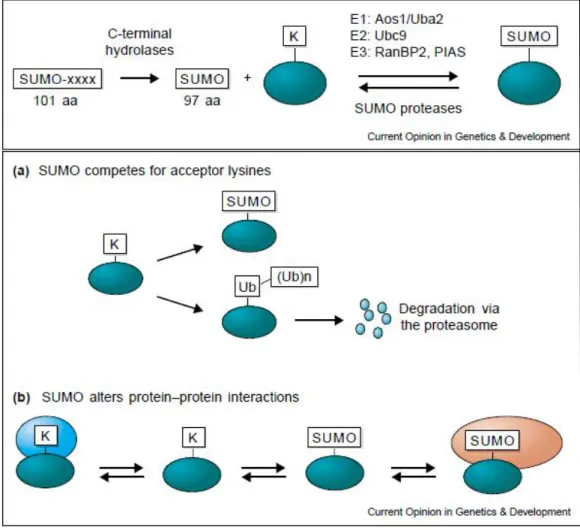
La sumoilazione può avere diversi effetti sulla struttura della proteina che ne è soggetta (Fig. 1.4):
- Può bloccare un sito di attacco per un’altra proteina, in particolare competere con l’ubiquitina preservando la proteina dalla degradazione
- Può creare un nuovo sito di legame
Da questi cambiamenti nella struttura possono dipendere cambiamenti nella funzione e nella localizzazione della proteina sumoilata.
Fig. 1.4 - Possibili conseguenze delle interazioni di Sumo con una proteina. Da Gill 2003.
Risultati sperimentali indicano che in C. elegans la sumoilazione della proteina SOP è necessaria per la sua localizzazione nucleare e per la sua azione di repressore di geni Hox (Zhang et al., 2004), così come è necessaria all’attività trascrizionale di p53 in Drosophila e quindi alla sua attività proapoptotica (Mauri et al. 2007).
Dati recenti ( Terada et al. 2010) indicano come la sumoilazione regoli la proliferazione dei progenitori retinici in Xenopus laevis impedendone l’uscita dal ciclo cellulare.
Nel caso specifico di Xbh1 si è ipotizzato che la sumoilazione possa essere necessaria per la sua attività biologica; in particolare è stato dimostrato che la mutagenesi sui due siti canonici di sumoilazione PKLE di Xbh1(v. sotto) impedisce la sua attività di specificatore di cellule gangliari nella retina di X. laevis (nostri dati non pubblicati). Dall’analisi con il programma SUMO-plot è emerso che la sequenza proteica di Xbh1 presenta due siti canonici di sumoilazione, la cui sequenza è PKLE, e altri due siti potenzialmente sumoilabili (GKVD e TKEE).
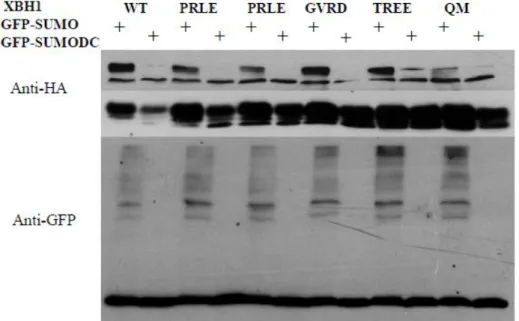
Sono stati creati costrutti mutanti in cui la lisina (K) dei siti canonici era convertita in arginina (R). La lipofezione dei costrutti singoli mutanti (K→ R 148 e K→ R 161 mettere aa) e del doppio mutante (K→ R 148,161) a livello di retina presuntiva ha portato come effetto una perdita della capacità di Xbh1 di indurre cellule gangliari; nel caso dei singoli mutanti questa riduzione non sembra significativa, mentre il doppio mutante perde completamente la capacità di indurre il destino gangliare nei progenitori retinici. Anche se la funzione biologica del doppio mutante era nulla, la presenza di altri due siti rende Xbh1 potenzialmente sumoilabile su altre posizioni non canoniche. In effetti solo nel quadruplo mutante di Xbh1, saggi di trasfezione in cellule HEK293 mostrano la totale o quasi totale assenza di sumoilazione della proteina Xbh1 (Fig. 1.5).
Per questi saggi, costrutti sono stati da me generati nel corso del tirocinio della laurea triennale e sono stati inviati presso il Dipartimento di Biochimica, Biofisica e Chimica delle Macromolecole, Universita` degli Studi di Trieste dove il Dr Licio Collavin e i suoi collaboratori hanno effettuato saggi biochimici di sumoilazione. I costrutti con le sequenze di Xbh1 wt e mutanti sono stati trasfettati insieme a un costrutto GFP-SUMO in cellule HEK293 per poi fare un saggio di “shift”. elettroforetico. Se avviene sumoilazione la proteina di fusione GFP-SUMO viene coniugata alla proteina XBH1-HA, diminuendone la mobilità elettroforetica, come osservato, dopo SDS_PAGE e western blot, rilevando la migrazione della proteina di fusione con un anticorpo anti-HA ( Fig. 1.5)
Fig. 1.5: risultato di western blot La forma GFP-Sumo DC , delta C, ha una delezione al C terminale che è l’estremità che lega la lisina bersaglio per cui non può interagire con nessuna proteina, è un controllo. Nella prima linea si trova la frazione di proteina Xbh1, nelle varie forme, ritardata nella corsa su gel dal legame con GFP-Sumo, con la mutazione di tutti e quattro i siti sumoilabili la proteina Xbh1 è quasi totalmente non sumoilata.Nella seconda la frazione di proteina Xbh1 che ha migrato alla sua altezza normale. Nella parte inferiore della figura c’è una corsa di controllo in cui è stato usato l’anticorpo anti-GFP per far vedere che dove sumo non può legarsi ad alcun bersaglio ,cioè la forma con la delezione, migra in basso. Nel controllo anti-GFP per il quadruplo mutante si ha lo stesso un andamento a ladder perché Sumo ha interagito con tutte le altre proteine sumoilabili presenti nelle cellule.
1.6 Scopo della tesi
Visto l’effetto di Xbh1 di indurre i progenitori retinici a diventare cellule gangliari ( Poggi et al. 2004) e la sua dipendenza dalla sumoilazione nello svolgere questa azione biologica la tesi è stata incentrata sul verificare il ruolo di Xbh1 in un momento precoce dello sviluppo cioè sul patterning della piastra neurale, osservando l’effetto della sovraespressione del gene, sia wild type sia mutato nei siti 148 e 161, su diversi marcatori del SNC.
La sovraespressione è stata realizzata mediante la microiniezione, dei messaggeri dei diversi costrutti, in embrioni di Xenopus leavis.