1 INTRODUZIONE
1.1 Transgenesi
1.1.1 Aspetti generali
L’uso degli animali da esperimento ha permesso il progresso sostanziale della ricerca biomedica. Importanti informazioni in campo fisiologico, biochimico e genetico sono state ottenute dallo studio di modelli animali che possiedono un genoma di dimensioni modeste, come ad esempio il moscerino della frutta Drosophila melanogaster. Le disponibilità di approfondite conoscenze di genetica classica relative a questo organismo, assieme alla possibilità di effettuare mutagenesi indotta da agenti chimici o raggi X, hanno permesso di gettare le basi per la comprensione di come i geni regolano i processi di sviluppo di un organismo (Lewis, 1978). Un limite a questo approccio sperimentale consiste nell’impossibilità di agire in maniera selettiva su un preciso gene d’interesse, in quanto questa tecnica permette di ottenere mutazioni puntiformi in maniera “random” all’interno del genoma o delezioni e traslocazioni di ampie regioni cromosomiche. Dopo l’avvento delle tecniche del DNA ricombinante, si è inaugurata all’inizio degli anni ’80 una nuova era nel campo della ricerca scientifica basata sullo sviluppo della transgenesi, la tecnica che consiste nell’inserimento di DNA esogeno nel genoma di un organismo. Anche altri settori più applicativi come l’allevamento e l’agricoltura, seppur non senza polemiche più o meno giustificate, hanno fatto largo uso della transgenesi per migliorare, sia da un punto di vista quantitativo sia qualitativo, la produzione degli animali da allevamento ed i raccolti, ottenendo come conseguenza principale un aumento economicamente vantaggioso della produttività. Un’altra applicazione della transgenesi, che ha avuto un largo e vantaggiosissimo impiego, è quello per la generazione di bioreattori, grazie ai quali è possibile ottenere in grandi quantità ed a bassi costi di produzione sostanze come l’insulina, l’interferone o l’ormone della crescita per il trattamento di importanti patologie umane.
L’uso di animali transgenici nella ricerca sia di base che applicata permette di poter studiare la funzione di un determinato gene in vivo e di analizzare le conseguenze della sua presenza nel contesto dell’organismo transgenico impiegato, allo scopo di associare
un’alterata funzione genica, dovuta a mutazioni nella sequenza dello stesso gene, all’insorgenza di una determinata malattia.
La transgenesi è stata utilizzata per modificare il genoma di diverse specie animali tra cui
Drosophila e zebrafish, ma grazie alle sue peculiari caratteristiche il topo rappresenta il
modello più adeguato. Essendo un mammifero, è molto più vicino all’uomo: infatti il genoma di topo è stato completamente sequenziato e dall’analisi genica è stata riscontrata una elevatissima omologia di sequenza, circa del 99%, tra i geni murini e quelli umani. Ciò significa che le proteine dei due organismi sono molto simili tra loro sia come struttura che funzione. Per questo motivo studiare e conoscere un meccanismo molecolare/cellulare/fisiologico nel topo permette facilmente di estrapolare all’uomo le nuove conoscenze acquisite nel modello animale transgenico. Inoltre rispetto ad altri mammiferi risulta vantaggioso come sistema di studio in quanto presenta un’alta prolificità, per cui è possibile ottenere in poco tempo un buon numero di animali su cui poter effettuare esperimenti, ed è di piccole dimensioni che permettono l’allevamento in spazi relativamente ridotti.
1.1.2 Transgenesi convenzionale nel topo
Una linea di topo transgenica può essere generata grazie al fatto che un frammento di DNA esogeno (il transgene), se iniettato nel pronucleo maschile di uno zigote di topo, può integrarsi nel genoma prima che i due pronuclei si fondano. Uno zigote così trattato può essere reimpiantato nell’utero di una femmina pseudogravida così da rendere possibile il proseguimento dello sviluppo dello zigote iniettato fino alla nascita (Gordon et al., 1980). In questo modo si potrà ottenere un individuo che verosimilmente porterà integrato nel genoma di tutte le sue cellule il DNA transgenico, comprese quelle della linea germinale. La modificazione della linea germinale permetterà di trasmettere il transgene alla progenie e quindi ottenere topi transgenici con lo stesso tipo di modificazione a livello del loro genoma. Il DNA esogeno che viene iniettato è formato da un costrutto che comprende la regione codificante del gene d’interesse preceduta da un promotore che ne regola la trascrizione. Le regioni cromosomiche in cui si integra il transgene sono in genere indipendenti sia dalla sequenza del DNA genomico che da quella del transgene stesso: l’integrazione del DNA transgenico è infatti del tutto casuale e per questo motivo soggetto agli effetti di posizione: esso può inserirsi sia in zone di eterocromatina in cui il DNA genomico è fortemente compattato e la sua trascrizione repressa, sia nelle vicinanze di sequenze regolative per il controllo trascrizionale in regioni eucromatiche. Nel primo caso,
nonostante la presenza di una sequenza promotrice in grado di attivare la trascrizione del transgene, la regione genomica eterocromatica è in grado di avere un effetto dominante sull’attivazione del transgene e quindi non si esprimerà. Nel secondo caso sarà possibile riscontrare situazioni variabili in dipendenza del contesto in cui si ha inserzione: da un controllo esclusivo da parte del promotore del transgene a situazioni in cui via via si ha una regolazione dell’espressione del transgene da parte degli elementi regolativi (“enhancers”) presenti nelle regioni limitrofe al sito di inserzione del transgene. Questa variabilità è funzione sia della distanza che intercorre sul filamento di DNA genomico tra il sito a cui il transgene si inserisce e gli elementi regolativi della trascrizione, sia della forza con cui tali elementi sono in grado di attivare la trascrizione. Un altro aspetto che può modulare l’attivazione di un transgene consiste nel fatto che il frammento di DNA transgenico iniettato può inserirsi nel DNA genomico in forma di concatenameri. Questo aspetto è di grande importanza in quanto il numero di copie presenti nel concatenamero ha un effetto considerevole sul controllo trascrizionale del transgene in quanto un alto numero di copie può indurre, nel genoma, l’organizzazione in eterocromatina che ha come effetto il silenziamento del gene (Dobie et al.,1997; Garrick et al., 1998). Quindi, la possibilità che si verifichino effetti di posizione o inserzione di concatenameri rende necessaria un’accurata analisi delle varie linee transgeniche generate, al fine di selezionare la linea in cui il transgene presenti il “pattern” di espressione atteso.
Nel campo della ricerca di base, i topi transgenici forniscono un ottimo modello per lo studio della funzione e del meccanismo d’azione di un gene. Se il gene endogeno d’interesse si esprime in una linea cellulare specifica è possibile vedere il fenotipo di una sua sovraespressione in tutte le cellule, introducendo nello zigote di topo la regione codificante del gene sotto il controllo trascrizionale di un promotore ubiquitario; oppure è possibile studiare il fenotipo di un’espressione ectopica utilizzando nel transgene un promotore tessuto-specifico per un’altra linea cellulare. Se, invece, il gene endogeno è espresso in maniera ubiquitaria è possibile ottenere la sua sovraespressione in una specifica popolazione cellulare utilizzando un transgene costituito dalla regione codificante del gene d’interesse regolata da un promotore tessuto-specifico per quella data linea cellulare.
L’uso di un promotore forte ed ubiquitario, come ad esempio un promotore virale come quello di citomegalovirus (CMV) o SV40, ha il vantaggio di permettere l’espressione costitutiva del transgene e la conseguente produzione della proteina ad alti livelli. Alcuni promotori virali però non sono pan-specifici ma correlati ai tessuti normalmente infettati dal virus risultando un fattore limitante nel caso in cui il virus non infetti la linea cellulare
desiderata (Baskar et al.,1996). Invece l’uso di promotori tessuto-specifici, che sono attivi solo in una data linea cellulare, permette di studiare l’effetto e il ruolo di un gene in una specifica popolazione di cellule. Inoltre, iniettando costrutti formati da un promotore tessuto-specifico che controlla l’attività trascrizionale di un gene “reporter”, come ad esempio EGFP e LacZ, è possibile effettuare studi di “fate mapping” visualizzando una specifica linea cellulare e seguendone il destino differenziativo durante lo sviluppo. In alcuni casi l’incompleta caratterizzazione del promotore tessuto-specifico come anche la mancata identificazione di un promotore che sia specifico per il tipo cellulare desiderato rendono questo tipo di approccio sperimentale profondamente limitante (Takahashi et
al.,1997). Inoltre, dato che durante lo sviluppo alcuni geni si esprimono in maniera
transiente, sarà possibile visualizzare la proteina codificata dal gene “reporter” solo quando il promotore che ne regola la trascrizione è attivo. Un’ulteriore limitazione si ha nel caso in cui la sovraespressione costitutiva del gene di interesse risulti letale durante l’embriogenesi.
Con l’avvento della tecnica della transgenesi condizionale è stato possibile ovviare ai limiti dati dall’attivazione transiente dei promotori tessuto-specifici e dalla sovraespressione genica ottenendo un più fine controllo spaziale e temporale dell’espressione genica rispetto ai sistemi convenzionali
1.1.3 Transgenesi condizionale
Uno dei primi sistemi di transgenesi condizionale prevede l’uso di promotori inducibili per guidare l’espressione del transgene nel topo, allo scopo di agire sull’espressione genica in maniera temporale. I promotori inducibili sono sequenze di regolazione localizzate nei promotori di geni bersaglio, alle quali si lega un fattore che attiva la loro trascrizione. Il fattore di trascrizione, generalmente inattivo, viene attivato in risposta a stimoli ambientali, come la variazione di concentrazione di certi ioni e di certe piccole molecole nutritive, della temperatura, di determinati shock. Un esempio di sistema inducibile prevede l’utilizzo del promotore del gene della metallotioneina-1 di topo. La metallotioneina-1 è una proteina a basso peso molecolare, ricca di zolfo, che serve a bloccare in forma inerte i metalli pesanti, rendendoli meno tossici. Il suo promotore è indotto da metalli pesanti, come lo zinco, ed è stato utilizzato nella generazione di topi transgenici che sovraesprimono Ets2, un fattore di trascrizione che inibisce l’apoptosi. Questi topi presentano uno sviluppo anormale a carico dello scheletro, del neurocranio e del viscerocranio che ricordano le anomalie presenti nei malati affetti da sindrome di Down
(Sumarsono et al., 1996). Questo approccio sperimentale ha permesso di comprendere il ruolo di Ets2 e di poter effettuare una correlazione con una patologia umana.
Tuttavia esistono degli svantaggi per l’uso di questo sistema inducibile che sono dovuti alla tossicità dei metalli pesanti per gli organismi da una parte, ed al fatto che si ha la presenza di una espressione di base del transgene sotto il controllo del promotore della metallotioneina1 anche nel caso in cui non si effettui somministrazione di metalli pesanti (Sumarsono et al., 1996).
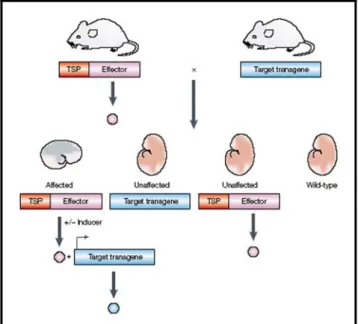
L’idea di controllare nel tempo l’espressione genica ha avuto, in seguito, un ampio successo con l’avvento del sistema di transgenesi binaria, con il quale è stato possibile introdurre anche un controllo spaziale dell’espressione genica. In questo sistema l’espressione del gene è controllata dall’interazione di due componenti: il transgene “effettore”, il cui prodotto va ad agire sul secondo componente, il transgene “target”. Il solo transgene “effettore” non influisce sull’espressione di geni endogeni, poichè il suo prodotto va ad agire soltanto sul transgene “target” con il risultato di attivarne o inattivarne la trascrizione. Dall’altra parte l’espressione del transgene “target” dipende interamente dalla presenza dell’effettore. In questo modo, incrociando due linee transgeniche di topo, una per l’“effettore” e l’altra per il “target”, l’espressione del transgene “target” si avrà solo nella progenie che presenta entrambi i transgeni, ed in presenza di un induttore esogeno che permette un’appropriata interazione dell’“effettore” sul “target” (Fig. 1).
Figura 1. Transgenesi binaria nel topo. L’interazione dei due componenti determina l’attivazione
del transgene “target” ma, in alcuni casi (per esempio quando l’“effettore” codifica per una ricombinasi sito specifica), l’“effettore” può anche inattivare il “target”. In molti sistemi binari, l’interazione dei due transgeni dipende dalla presenza di un induttore esogeno che può agire sia attivando che inibendo l’attività dell’“effettore”. TSP, “tissue specific promoter”, promotore tessuto specifico.
La transgenesi binaria può essere suddivisa in due categorie. Nella prima l’“effettore” è un fattore di trascrizione in grado di attivare in maniera specifica la trascrizione del transgene “target”, mentre nella seconda l’“effettore” è una ricombinasi sito-specifica che agisce sul gene “target” promuovendo una ricombinazione sito specifica che ha come risultato l’attivazione o il silenziamento del “target” stesso (Lewandoski, 2001).
In uno dei primi sistemi di transgenesi binaria, facente parte della prima categoria, l’“effettore” è una proteina virale, come VP16 del virus Herpes simplex, che agisce sul transgene “target” che contiene un elemento di risposta per essere attivato (Khillan et al., 1988; Bieberich et al., 1993). Questo sistema non è molto utilizzato perché presenta due grossi limiti: il transgene “target” può esprimersi ad alti livelli in assenza del transgene “effettore” e può causare tumorigenesi (Nerenberg et al., 1987; Khillan et al., 1988).
Uno dei sistemi di transgenesi binaria tra i più ampiamente utilizzati è quello regolato dalla tetraciclina (Gossen et al.,1992). In questo sistema il costrutto del transgene “effettore” è costituito da un promotore tessuto-specifico che regola la trascrizione della regione codificante di una proteina chimerica, composta dal repressore della tetraciclina di
E. coli e dal dominio di transattivazione di VP16, proteina del virus herpes simplex, che è
in grado di promuove molto efficacemente la trascrizione. La proteina repressore della tetraciclina (TetR) ha due domini, uno lega la tetraciclina, mentre l’altro lega una sequenza di 19 nucleotidi contenuta nell’operone per la tetraciclina (tetO) nel transgene “target”, attivandolo (Fig. 2).
Figura 2. Il sistema di transgenesi binaria regolato dalla tetraciclina. a) L’effettore è un transattivatore
tetR (viola) e dal dominio di transattivazione di VP16 (arancione). In assenza dell’induttore, doxiciclina (Dox), tTA lega la sequenza di 19 nucleotidi di tetO provocando l’attivazione della trascrizione da parte del promotore TATA e, di conseguenza, l’espressione del transgene “target”. In presenza di Dox, tTA cambia conformazione e non può più legare la sequenza di tetO. b) Nel sistema inverso (rtTA), il gene tetR è stato mutato in modo che leghi la sequenza di tetO ed attivi la trascrizione solo in presenza di Dox.
Ci sono due tipologie di questo sistema. Nel sistema originale, il transattivatore regolato dalla tetraciclina (tTA) non può legare il DNA quando è presente l’induttore (“tet-off”) (FIG.2a), mentre nel sistema inverso (rtTA) tTA lega il DNA solo quando è presente l’induttore (“tet-on”) (FIG.2b) (Gossen et al., 1995). L’induttore utilizzato è la doxiciclina (Dox), scelta per il suo basso costo, per la sua ampia disponibilità commerciale e perché attiva rtTA e inattiva tTA in maniera efficiente a dosi al di sotto dei livelli citotossici.
Poiché la Dox può attraversare la placenta nel topo transgenico e regolare efficientemente l’espressione genica durante l’embriogenesi, questi sistemi vengono impiegati per evitare l’espressione di un transgene letale nell’embrione. In uno studio, il sistema tTA è stato utilizzato per controllare l’espressione di un transgene “target” che codifica per la subunità A della tossina difterica (DTA). Le linee transgeniche di topo così ottenute hanno rivelato che l’induzione specifica del sistema tTA a livello del cuore negli individui adulti causa cardiomiopatie, che ricordano quelle osservate nell’uomo (Lee et al., 1998).
Questo sistema di transgenesi condizionale, come quello indotto dai metalli pesanti, sono sistemi reversibili, in quanto è possibile attivare o inattivare la trascrizione del transgene semplicemente fornendo o togliendo al topo l’induttore.
In un altro sistema di transgenesi binaria, facente parte della seconda categoria, quella in cui l’“effettore” è una ricombinasi sito-specifica, è possibile regolare l’espressione del transgene “target” in maniera irreversibile: un esempio è il sistema Cre/loxP.
1.2 I sistemi Cre/loxP e Flp/FRT
1.2.1 Aspetti generali
Le ricombinasi Cre e Flp sono enzimi che fanno parte della famiglia delle integrasi ricombinasi sito-specifiche. La ricombinasi Cre è presente in natura nel batteriofago P1 (Sauer e Henderson, 1989) ed ha un ruolo nel ciclo lisogenico, in quanto serve per l’integrazione del genoma fagico in quello batterico; mentre la ricombinasi Flp deriva dal
lievito Saccharomyces cerevisiae (O’Gorman et al., 1991). Le due ricombinasi sono in grado di riconoscere specifiche sequenze di DNA chiamate rispettivamente sito loxP e sito
FRT. Filamenti di DNA fiancheggiati da una coppia di tale siti vengono definiti “floxed” o
“fliped”. I siti loxP e FRT sono costituiti da due distinte sequenze palindromiche di 13 nucleotidi ciascuna interrotte da una sequenza asimmetrica di 8 nucleotidi detta “spacer” che conferisce direzionalità al sito. In base alla direzione ed alla posizione dei siti loxP (oppure FRT) su un filamento di DNA la ricombinasi Cre (oppure Flp) è in grado di catalizzare reazioni irreversibili di tre diversi tipi:
1) Inversione: i siti loxP/FRT si trovano sullo stesso filamento di DNA ed hanno direzioni opposte. Il prodotto della reazione è un’inversione del frammento di DNA “floxed”/“fliped” (Fig.3A).
2) Traslocazione: i siti loxP/FRT si trovano su due filamenti diversi di DNA. Il prodotto della reazione è una traslocazione cromosomica (Fig.3B).
3) Excisione: i siti loxP/FRT si trovano sullo stesso filamento ed hanno la stessa direzione. Il prodotto della reazione passa attraverso la formazione transiente di un “loop” tra i due siti, e la successiva excisione del frammento di DNA fiancheggiato dai due siti. Il frammento exciso all’interno di una cellula viene rapidamente degradato rendendo il processo di excisione irreversibile (Fig.3C).
Grazie a quest’ultimo tipo di reazione le ricombinasi Cre e Flp possono essere utilizzate per controllare l’espressione genica in modo condizionale per mezzo della ricombinazione somatica sito-specifica del DNA, catalizzando un evento di ricombinazione conservativa del DNA.
Figura 3. Rappresentazione schematica dei meccanismi d’azione della ricombinasi Cre. (A) inversione;
(B) traslocazione; (C) excisione. Queste reazioni sono catalizzate anche dalla ricombinasi Flp nel sistema Flp/FRT.
1.2.2 Applicazioni
L’evento di excisione promosso dalle ricombinasi Cre e Flp può essere utilizzato nei sistemi di transgenesi binaria per seguire, sia durante lo sviluppo embrionale sia nell’adulto, l’espressione del gene di interesse permettendo di effettuare analisi di “lineage” cellulare. Le ricombinasi Cre e Flp possono anche essere impiegate per effettuare il knock-out condizionale che permette di modulare l’espressione di un gene e di poterne studiare il ruolo all’interno di un determinato tessuto e in un preciso momento della vita dell’animale in esame. Quest’ultima applicazione sarà analizzata in dettaglio successivamente.
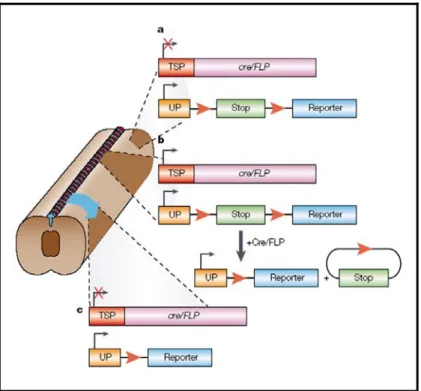
L’analisi di “lineage” cellulare è resa possibile dall’utilizzo di due linee transgeniche: la prima esprime il gene cre/flp sotto il controllo di un promotore tessuto-specifico per una data popolazione cellulare, la seconda esprime un gene “reporter” sotto il controllo trascrizionale di un promotore costitutivo. La presenza di una sequenza di DNA contenente un segnale di stop della trascrizione e fiancheggiata da due siti loxP (o FRT), comunemente indicata come “cassetta di stop”, posizionata tra il promotore costitutivo e la regione codificante del gene “reporter” previene la trascrizione di quest’ultimo. L’incrocio delle due linee determina l’excisione della cassetta di stop e quindi l’attivazione della trascrizione in maniera costitutiva del gene “reporter” nelle cellule in cui la ricombinasi Cre/Flp è stata espressa. L’irreversibilità dell’evento di excisione Cre/Flp-mediato fa sì che il gene “reporter” si esprima anche nelle cellule figlie, per cui la visualizzazione della proteina “reporter” permette di seguire una specifica linea cellulare anche quando il promotore tessuto specifico non è più attivo (Fig.4) (Lewandoski, 2001).
Figura 4. “Fate-mapping” di una linea cellulare specifica. In questa immagine sono mostrate tre possibili
situazioni che possono verificarsi:
a) nella regione in cui Cre/Flp non è espressa non si ha espressione del gene “reporter”
b) nella regione in cui Cre/Flp è espressa si ha l’excisione della cassetta di STOP e la conseguente espressione del gene “reporter”
c) la ricombinasi non è espressa ma le cellule derivano da un territorio dove la Cre/Flp era espressa ed ha promosso l’excisione della cassetta di STOP portando all’espressione del gene “reporter” che persiste anche quando il promotore non è più attivo. TSP: “tissue specific promoter” promotore tessuto specifico; UP: “ubiquitous promoter” promotore ubiquitario.
Prima di incrociare le due linee transgeniche si rendono necessarie accurate verifiche della corretta espressione dei transgeni. Effetti di posizione nella linea che esprime la ricombinasi Cre/Flp possono alterare la tessuto-specificità degli elementi regolatori, provocando l’espressione dell’enzima in una linea cellulare diversa da quella di interesse. Un altro inconveniente riguarda l’inserzione di più copie del transgene in diverse regioni del genoma: incrociando la linea transgenica che esprime cre/flp con una in cui il transgene floxato/flippato è presente in più copie nel genoma si potrebbero verificare gravi instabilità cromosomiche, quali l’aneuploidia, durante l’evento di excisione Cre/Flp-mediato.
1.3 “Knock-out” convenzionale e “knock-out” condizionale
1.3.1 Generazione di un “knock-out” convenzionale
Un metodo per introdurre transgeni nella linea germinale di topo, alternativo all’iniezione di DNA esogeno nel pronucleo maschile dello zigote, prevede l’uso della ricombinazione omologa nelle cellule staminali embrionali (ES). La ricombinazione omologa è il processo di scambio di sequenze di DNA tra due filamenti per mezzo di regioni con elevato grado di omologia in cui le sequenze omologhe si riconoscono e si appaiano, permettendo lo scambio tra i due filamenti di DNA. In questo modo sono assicurati lo scambio ed il legame tra due filamenti di DNA con i limiti dati dalle omologie tra le molecole (Court et
al, 2002).
Le cellule staminali embrionali (ES), che derivano dalla massa cellulare interna della blastocisti di topo, sono cellule totipotenti, quindi in grado di dare origine a tutti i tipi cellulari presenti in un organismo, compresa la linea germinale. Presentano il vantaggio di poter essere mantenute in coltura e la totipotenza preservata grazie alla presenza, nel mezzo di coltura, del fattore LIF (Leukemia Inhibitor Factor). In queste condizioni le cellule ES possono essere manipolate geneticamente e, grazie alla ricombinazione omologa, un frammento di DNA esogeno può essere integrato in un preciso sito del genoma (Jasin et al., 1996). Si possono così evitare gli effetti di silenziamento del gene dovuto all’inserzione in alto numero di copie così come gli effetti di posizione potendo inserire il transgene in regioni di cromatina compatibili con la desiderata espressione tessuto-specifica (Bronson et al.,1996).
Un’importante applicazione di questa tecnica prevede l’introduzione di mutazioni in geni endogeni specifici. L’uso di appropriati vettori a DNA ha permesso di sfruttare la ricombinazione omologa per introdurre frammenti di DNA esogeno all’interno di specifiche regioni del genoma (Capecchi, 1989).
Il vettore per la ricombinazione omologa è costituito da un plasmide contenente sequenze omologhe al gene bersaglio di interesse interrotte da un marcatore che permette una selezione, ad esempio il gene neo, che conferisce resistenza alla neomicina. Il vettore viene inserito mediante elettroporazione nelle cellule ES (Thomas e Capecchi, 1987). Il marcatore presente nelle sequenze ha un duplice ruolo: da un lato, la presenza del gene neo permette di selezionare i cloni delle cellule ES che hanno integrato il vettore, dall’altro,
interrompe la sequenza codificante inattivando il gene (“knock-out” convenzionale). Successivamente, possono essere selezionati ed isolati i cloni di cellule ES che hanno integrato il frammento nel genoma tramite ricombinazione omologa (Thomas e Capecchi, 1987). I cloni ES positivi alla ricombinazione omologa possono successivamente essere reintrodotti nella blastocisti di topo dove vengono incorporati dalla massa cellulare interna, e quindi nell’embrione, dove daranno origine alle cellule della linea germinale. In questo modo si ottengono ceppi di topi eterozigoti e omozigoti per il gene alterato, nei quali è possibile esaminare la completa inattivazione del gene.
1.3.2 Applicazioni
Topi “knock-out” vengono ampiamente utilizzati in ambito scientifico, ad esempio risultano essere un ottimo modello per le malattie genetiche umane fornendo la prova che l’alterata espressione di un gene può causare una determinata patologia. Inoltre dall’incrocio di topi eterozigoti o omozigoti per distinte mutazioni geniche si ottengono importanti informazioni su malattie umane dovute all’alterazione di più geni. L’uso di topi “knock-out” si rivela vantaggioso anche per stabilire nuove diagnosi e terapie; ad esempio topi “knock-out” per i geni dell’apolipoproteina E (proteina che trasporta il colesterolo nel sangue) e dell’interleuchina I (proteina con funzione di mediatore chimico tra le cellule del sistema immunitario) si sono mostrati significativamente meno sensibili all’Alzheimer e quindi utili per valutare gli effetti terapeutici di prodotti farmaceutici (Chapman et al., 2001). Nell’ambito della ricerca di base, linee “knock-out” permettono di poter studiare in dettaglio la funzione di un certo gene. Ad esempio, nel caso in cui un “knock-out” genico mantenga un fenotipo normale, è possibile dedurre la presenza di altre proteine che ne compensano la perdita di funzione. Invece, in altre circostanze, il fenotipo del topo “knock-out” non appare finché l’animale non è esposto ad un particolare stimolo indicando che la funzione genica è correlata a stress ambientali o a eventi patologici. E’ questo il caso di topi “knock-out” per un componente del recettore dell’interferone di tipo I (proteina prodotta dalle cellule animali come risposta all’infezione di un virus e capace di inibirne la replicazione) che appaiono fenotipicamente normali, ma mostrano un’estrema suscettibilità ad un’infezione virale acuta rispetto ai controlli (Hwang et al., 1995; Muller
et al., 1994).
Purtroppo non tutti i processi biologici possono essere studiati mediante inattivazione genica convenzionale. Ad esempio, alcuni geni che sono espressi nell’adulto possono avere un ruolo fondamentale durante lo sviluppo embrionale per cui la loro mutazione
porta ad un fenotipo letale che riflette un ruolo precoce non ridondante del gene. In questi casi è preclusa l’analisi della sua funzione nell’adulto con tecniche convenzionali. Per ovviare a questi limiti, è necessario poter silenziare un gene quando e dove il ricercatore vuole: ciò è possibile grazie alla tecnica del “knock-out” condizionale (Lewandoski, 2001).
1.3.3 “Knock-out” condizionale
Il “knock-out” condizionale si basa sulla possibilità di generare un allele condizionale in cui vengono collocati, all’interno del DNA genomico, due siti loxP posizionati rispettivamente a monte ed a valle della sequenza codificante del gene endogeno in analisi. In questo caso, così come abbiamo visto poco sopra per la cassetta di stop, è possibile promuovere un evento di ricombinazione somatica sito-specifica mediata dalla ricombinasi Cre che ha come risultato l’excisione della regione codificante del gene endogeno. La progenie, che risulta dall’incrocio dell’allele condizionale con una linea transgenica di topo che esprime la ricombinasi cre in maniera tessuto-specifica, presenta il gene endogeno inattivato solo in quelle cellule che esprimono cre. Per interpretare correttamente il fenotipo causato dall’inattivazione Cre-mediata di un gene è spesso essenziale conoscere in quali cellule è avvenuta l’excisione. Questo è reso possibile dalla generazione di una progenie che deriva dalla combinazione dell’allele condizionale, della linea cre tessuto-specifica e di una linea che presenta un gene “reporter” (LacZ, EGFP) inducibile. In queste condizioni la ricombinasi Cre contemporaneamente inattiva il gene endogeno ed attiva la trascrizione del gene “reporter” così da permettere di individuare le cellule in cui è avvenuto il “knock-out” genico.
Il successo ed al tempo stesso il limite di questo tipo di applicazioni dipende dalla disponibilità di elementi regolatori della trascrizione che siano in grado di attivare l’espressione della ricombinasi cre in tutta una gamma di tessuti ed a stadi diversi dello sviluppo. In questo senso è cruciale avere la possibilità di controllare sia da un punto di vista spaziale che temporale l’attivazione dell’espressione della ricombinasi Cre.
Per la risoluzione di questo problema si sfruttano le conoscenze relative al dominio spazio-temporale di espressione di numerosi geni di cui talvolta sono noti e ben caratterizzati gli elementi che ne regolano la trascrizione (enhancers). Molto spesso, tuttavia, nonostante venga individuato un gene con un profilo di espressione ad hoc, non si hanno sufficienti informazioni relative alle sue regioni regolative. In questi casi è possibile utilizzare come promotore tessuto specifico un frammento di DNA genomico delle
dimensioni di alcune centinaia di migliaia di basi (Kb) attorno alla regione codificante del gene di interesse supponendo che in esso siano contenuti tutti gli elementi regolativi che ne caratterizzano suo il profilo di espressione. Gli unici vettori capaci di contenere frammenti di DNA di tali dimensioni sono i cromosomi artificiali.
1.4 I cromosomi artificiali: BACs, PACs e YACs
Gli elementi regolativi o “enhancers” che contribuiscono al controllo trascrizionale di un gene sono distribuiti attorno alla sua regione codificante in maniera sparsa e spesso lontano dal promotore del gene stesso. Talvolta, per questo motivo con la transgenesi è possibile ricapitolare completamente le caratteristiche spaziali e temporali dell’espressione di alcuni geni solamente se si utilizzano ampie regioni di DNA genomico. Così facendo si aumenta la probabilità che possano essere presenti tutti gli elementi regolativi del gene endogeno e che l’espressione del transgene risulti essere ad hoc. Inoltre l’impiego di grandi frammenti di DNA per la generazione di transgeni consente di ridurre enormemente la suscettibilità del transgene agli effetti posizionali, indotti dalla presenza di eventuali elementi regolativi, presenti nella regione in cui il transgene si inserisce nel genoma dell’ospite. Questo è possibile grazie all’uso dei cromosomi artificiali batterici (BAC), quelli derivati dal fago P1 (PAC) e quelli di lievito (YAC).
I YAC sono particolarmente utili per clonare frammenti di DNA genomico dell’ordine di alcune megabasi, mentre i BAC e i PAC sono propagati in E. coli e possono contenere al massimo circa 300 kb. Questi ultimi sono più facilmente manipolabili rispetto agli YAC, che possono essere propagati in cellule di lievito (Copeland et al., 2001).
Il BAC è un vettore di circa una decina di Kb, contiene il gene che conferisce resistenza al cloramfenicolo ed un’origine di replicazione batterica, necessaria per la sua replicazione nelle cellule di E. coli. Il vantaggio principale dell’uso di questo vettore per l’introduzione di grandi frammenti di DNA genomico è la possibilità di ottenere grandi quantità di DNA genomico, in quanto si replica facilmente in E. coli. Spesso il BAC contiene l’intero locus di un gene eucariotico e viene utilizzato per generare topi transgenici, mediante microiniezione nel pronucleo maschile dello zigote o per trasfezione nelle cellule ES (Peterson et al., 1993). Il transgenico BAC può essere utilizzato per far esprimere ectopicamente il gene d’interesse, ossia in una popolazione cellulare diversa, allo scopo di studiare la sua funzione genica in un’altra linea cellulare specifica. Inoltre è possibile far esprimere un gene “reporter” sotto il controllo trascrizionale di tutti gli elementi regolatori
di un gene specifico per una data popolazione cellulare, in modo da poter seguire in maniera accurata tale linea durante lo sviluppo. L’applicazione più importante del transgenico BAC è il suo utilizzo nella transgenesi binaria ed, in particolare, nel sistema Cre/loxP. E’, infatti, possibile far esprimere fedelmente e in maniera strettamente specifica la ricombinasi Cre in una certa linea cellulare e, una volta generato il transgenico, consente di effettuare accuratamente esperimenti di “fate-mapping” e di “knock-out” condizionale.
1.5 Ricombinazione omologa in ceppi di E. coli
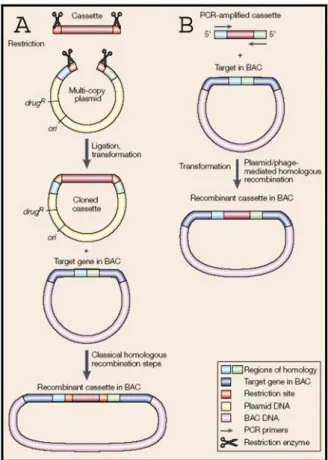
I metodi di clonaggio classici, che prevedono l’uso di enzimi di restrizione e della DNA ligasi non possono essere impiegati per modificare un vettore BAC contenente un inserto di DNA delle dimensioni di centinaia di Kb. Questo perché in grandi molecole di DNA, come i BACs, è difficile trovare un sito di restrizione unico che permetta il clonaggio. I problemi associati all’approccio tradizionale sono stati superati grazie allo sviluppo di una nuova tecnologia, la ricombinazione omologa nei batteri (Copeland et al., 2001). La ricombinazione omologa è il processo di scambio di DNA tra due molecole per mezzo di regioni con sequenza identica. In questo modo, è assicurato uno scambio ed un legame precisi tra due filamenti di DNA con i limiti dati dalle omologie tra le molecole (Court et al., 2002). Questa è una tecnica in vivo, effettuata in ceppi di E.coli, che sono stati modificati geneticamente per produrre in maniera efficiente e controllata le ricombinasi, enzimi-chiave nell’evento di ricombinazione omologa, poiché il sistema originale del batterio presentava delle limitazioni, in quanto il DNA lineare veniva rapidamente degradato ed erano necessarie regioni con sequenza identica molto ampie (Muyrers et al., 2000). Poiché la ricombinazione omologa, in questi ceppi di E. coli ingegnerizzati, è molto efficiente, le porzioni di omologia posso essere brevi di 30-50 bp e non sono necessari geni selettivi da aggiungere a valle del gene d’interesse per incrementare l’efficienza della ricombinazione omologa (Fig. 5) (Yu et al., 2000; Ellis et
Figura 5. Tecniche di clonaggio classica e di ricombinazione a confronto. A) Metodo di clonaggio
classico. B) ricombinazione omologa.
Così, come già visto nel paragrafo in cui ho trattato la ricombinazione omologa in cellule ES, con questa tecnica è possibile ottenere l’inserzione di una sequenza di DNA esogeno, come ad esempio la regione codificante della ricombinasi Cre, in un punto preciso della regione genomica presente nel BAC. Per questo è necessario collocare alle estremità della regione codificante della ricombinasi due sequenze denominate braccia di omologia corrispondenti alla sequenza del locus genico in cui si vuole inserire la ricombinasi. L’applicazione più interessante di questa tecnica è la sostituzione della regione codificante di un qualunque gene, il cui “pattern” di espressione risulti interessante per le sue caratteristiche spaziali e temporali, il cui locus è clonato nel BAC, con quella della ricombinasi Cre o di un gene “reporter” (lacZ, GFP). In questo modo è possibile ottenere un costrutto di DNA in cui il gene cre (o lacZ, GFP) risulta essere sotto il controllo trascrizionale di tutti gli elementi regolatori necessari per riprodurre in transgenesi quello stesso “pattern”.
Particolarmente utile è la possibilità di introdurre cre nei BACs per la generazione di linee transgeniche in grado di esprimere in maniera accurata la ricombinasi che possono essere utilizzate negli studi di “fate-mapping” e di “knock-out” condizionale. Ad esempio, sono stati generate topi transgenici Pax2-Cre, mediante ricombinazione omologa nel BAC
contenente l’intero locus di pax2 (fattore di trascrizione), sostituendo la regione codificante del gene con quella di cre. Incrociando questo transgenico con linee “reporter” inducibili, è stato visto che pax2 si esprime nel mesencefalo, nel cervelletto, nel bulbo olfattivo e nel rene (Ohyama e Groves, 2004).
1.6 Il sistema serotoninergico
Durante lo sviluppo embrionale dei vertebrati, i neuroni che sintetizzano la serotonina (5-HT) sono tra i primi ad essere generati e sono localizzati nella regione ventrale del rombencefalo (Wallace e Lauder, 1983). I neuroni serotoninergici sono organizzati in nuclei distinti denominati nuclei del raphe B1-9. Questi nuclei possono essere raggruppati in due classi: una caudale, B1-5 che comprende i nuclei del raphe magno, pallido ed oscuro, ed una rostrale, B6-9 che comprende i nuclei del raphe dorsale e mediano. Dai gruppi serotoninergici più caudali prendono origine le proiezioni serotoninergiche discendenti destinate al midollo spinale, mentre da quelli rostrali originano le fibre che raggiungono le strutture del sistema nervoso centrale anteriore quali la corteccia cerebrale, l’ippocampo, il diencefalo, i nuclei della base ed il cervelletto (Fig. 6).
Figura 6. Rappresentazione schematica del sistema serotoninergico in una sezione medio-sagittale di encefalo umano. In rosso sono rappresentati i nuclei del raphe rostrale e caudale, e le aree del SNC che
ricevono proiezioni serotoninergiche.
Questi neuroni sono generati a stadi precoci dello sviluppo embrionale e nel topo compaiono a partire dallo stadio E10.5 (10.5 giorni di gestazione) (Hynes e Rosenthal, 1999). La specificazione neuronale è determinata da una combinazione di fattori di trascrizione e molecole di secrezione quali: Nkx2.2, Lmx1b, Fgf4, Fgf8, Shh, GATA3 e
Pet1. Nkx2.2, Lmx1b, Fgf4, Fgf8, Shh e GATA3 specificano alcuni dei neuroni
serotoninergici, ma anche altri tipi cellulari. Pet1, invece, è strettamente specifico per la formazione del sistema serotoninergico ed è il “marker” più precoce del sistema seotoninergico che si conosce fino ad ora (Fig. 7) (Ye et al., 1998; Briscoe et al., 1999; Van Doorninck et al., 1999; Hendricks et al., 2003; Pfaar et al., 2002).
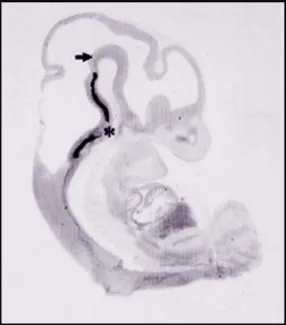
Figura 7. Espressione di Pet1 nel rombencefalo. Sezione sagittale di un embrione di topo a stadio E14.0 su
cui è stata effettuata un’ibridazione in situ contro Pet1. Pet1 si esprime in entrambi i gruppi dei nuclei del raphe. Uno caudale al plesso mesencefalico (freccia), l’altro caudale al plesso pontino (asterisco).
1.7 Scopo della tesi
Durante il mio internato di tesi ho ingegnerizzato un BAC contenente la regione genomica di Pet1 allo scopo di generare una linea transgenica di topo Pet1/Cre, in cui la regione codificante della ricombinasi Cre risulta essere sotto il controllo trascrizionale di
Pet1.
Pet1 è stato scelto per guidare l’espressione della ricombinasi Cre in una popolazione neuronale specifica, ossia i neuroni serotoninergici.
Il topo transgenico Pet1/Cre verrà utilizzato per studiare e seguire lo sviluppo del sistema serotoninergico mediante esperimenti di “fate mapping”, in cui linee transgeniche per geni “reporter” inducibili verranno incrociate con la linea transgenica Pet1/Cre, che sarà possibile generare con il BAC da me ingegnerizzato. Inoltre sarà possibile impiegare la linea transgenica Pet1/Cre in combinazione con alleli condizionali per ottenere il “knock-out” condizionale all’interno delle cellule del sistema serotoninergico.