75
La sansa vergine d’oliva è il principale residuo solido dell’estrazione olearia e a causa della sua complessità e refrattarietà biologica è difficile da smaltire. Il compostaggio, potenziato da un inoculo microbico, potrebbe essere una soluzione al problema dello smaltimento; infatti l’inoculo iniziale, con ceppi idonei alla degradazione delle sanse, permette una notevole accelerazione del processo.
Nell’ambito della presente tesi, le sanse vergini prodotte dall’estrazione dell’olio d’oliva della Cooperativa Olivicola di Arnasco (SV), sono state compostate in loco, al fine di ottenere un prodotto che potrà essere utilizzato come ammendante sulle colture di vite o di olivo della Cooperativa stessa.
In particolare sono state inoculate con uno starter microbico specifico, costituito principalmente da microrganismi indigeni delle sanse vergini precedentemente selezionati sulla base della loro capacità di innescare il processo di compostaggio delle sanse d'oliva, e sulla base di caratteristiche quali: la velocità di moltiplicazione, la termo-tolleranza, la biocompatibilità e la capacità di controllo biologico contro i più comuni microrganismi fitopatogeni del suolo, tra cui Venturia spp., Alternaria spp., Fusarium spp. e Rhizoctonia spp. (Echeverria
et al., 2011b). Successivamente tali sanse sono state compostate e infine, per
incrementare le proprietà soppressive del compost, sono state inoculate con il microrganismo antagonista Trichoderma atroviride. Tale fungo, infatti, è noto per la sua attività come antagonista nei confronti di molti patogeni delle piante, inoltre la sua presenza nel terreno di coltura induce lo sviluppo di meccanismi di resistenza nella pianta e ne promuove la crescita.
Allo scopo di seguire l’evoluzione della diversità microbica, sia batterica che fungina, durante le diverse fasi del processo di compostaggio, finissaggio e maturazione sono state utilizzate sia tecniche microbiologiche coltura-dipendenti che tecniche molecolari coltura-indipendenti.
76
3.1.
C
ONTROLLO DELLA TEMPERATURANell’ambito del compostaggio, la temperatura è un parametro “spia” che indica il passaggio da una fase del processo alla successiva. In condizioni fisico-chimiche ottimali, tale processo procede attraverso tre stadi: una breve fase mesofila di attivazione, una fase termofila di degradazione attiva e una fase di raffreddamento e maturazione caratterizzata dai processi di umificazione. I cambiamenti nel tasso e nel tipo di decomposizione, che corrispondono a ciascuno stadio, riflettono le variazioni delle popolazioni microbiche presenti nel substrato in compostaggio. Infatti, la trasformazione della materia organica è ampiamente dovuta alla rapida successione di popolazioni microbiche specializzate che esprimono una vasta serie di attività enzimatiche (Ryckeboer et al., 2003; Tuomela et al., 2000).
Nel compostaggio delle sanse d’oliva non sono stati evidenziati chiaramente i tre stadi che di norma caratterizzano il processo di compostaggio, poiché da un lato il controllo di processo e dall’altro la complessità della matrice e la presenza di sostanze anti-microbiche, come sostanze grasse e fenoli, hanno rallentato lo sviluppo dei microrganismi coinvolti nel processo di degradazione della sostanza organica. Di conseguenza, l’aumento di temperatura a seguito dei fenomeni di auto-riscaldamento microbico è risultato decisamente modesto, non evidenziando una vera e propria fase termofila. Questo tipo di processo è definito “compostaggio freddo”.
Le variazioni di temperatura misurate durante il compostaggio delle sanse d’oliva Taggiasca e Pignola della Cooperativa Olivicola di Arnasco sono riportate in Figura 20.
Il profilo di temperatura ottenuto evidenzia la presenza delle tre fasi che caratterizzano il processo di compostaggio delle sanse d’oliva. In particolare, durante la fase iniziale la temperatura è aumentata nell’arco di 20 giorni, fino a raggiungere un massimo di 45°C. Tale temperatura è rimasta costante per circa 80 giorni, dopodiché ha iniziato a decrescere e si è stabilizzata intorno a 35°C, suggerendo l’inizio della fase di raffreddamento e maturazione.
Dall’andamento della temperatura è quindi possibile stimare la durata del processo di compostaggio e finissaggio, che nel caso delle sanse della Cooperativa
77
Olivicola di Arnasco è di circa 7 mesi. Un periodo di compostaggio lungo è tipico per i residui dell’estrazione olearia, in quanto tali matrici contengono composti anti-microbici che rallentano lo sviluppo e la crescita dei microrganismi. Anche se in condizioni di processo ottimali tale processo può avvenire nell’arco di due mesi (Echeverria et al., 2011b).
Figura 1: Evoluzione della temperatura delle sanse d'oliva in compostaggio presso la Cooperativa
Olivicola di Arnasco
L’incremento iniziale di temperatura è, probabilmente, frutto del precoce rivoltamento effettuato per favorire l’omogeneizzazione della matrice. Infatti, il rivoltamento meccanico del cumulo in compostaggio apporta ossigeno nel substrato, che favorisce l’attività microbica.
La temperatura non ha mai superato i 45°C, perciò la matrice non ha raggiunto una vera e propria fase termofila, come previsto durante il compostaggio di queste matrici. Nell’ambito della presente ogni volta che faremo riferimento a questa fase, sarà utilizzato il termine “fase pseudo-termofila”.
Un risultato simile è stato ottenuto anche da Alfano et al. (2008) durante il compostaggio di sansa d’oliva areata meccanicamente. Tuttavia questi valori sono leggermente inferiori rispetto a quelli riportati in letteratura (50-60°C). Baeta-Hall
et al. (2004) hanno osservato che la sansa umida compostata con raspi d’uva e
78
termofilia. Tale differenza, seppur in minima parte, potrebbe essere collegata alla temperatura ambientale; infatti nel loro lavoro la temperatura ambiente era di circa 20°C mentre ad Arnasco era intorno a 13°C.
Baeta-Hall et al. (2004) hanno inoltre osservato che il compostaggio delle sanse d’oliva in cumuli statici con aerazione forzata aveva una fase mesofila molto espansa nel tempo, e l’incremento di temperatura è stato ottenuto solo in seguito a rivoltamento meccanico. Ciò suggerisce che l’apporto di ossigeno dall’esterno, insieme al rimescolamento della matrice durante la fase bio-ossidativa, ristabiliscono le condizioni idonee per lo sviluppo della biomassa microbica (Paredes et al., 2002; Garcia-Gomez et al., 2003). Anche Principi et al. (2003), durante il compostaggio di sanse d’oliva in cumuli statici, hanno attribuito alla scarsa presenza di ossigeno nel substrato, la causa del modesto incremento di temperatura. Infatti l’assenza di rivoltamento meccanico della matrice limita l’apporto di ossigeno e quindi lo sviluppo dei microrganismi aerobi che degradano la sostanza organica.
Pertanto, dato che la sansa d’oliva è una matrice complessa, ricca di sostanze che inibiscono la crescita di un numero elevato di microrganismi, è ipotizzabile che la popolazione microbica delle sanse in compostaggio presso la Cooperativa Olivicola di Arnasco, si sia sviluppata degradando le sostanze più semplici, ma la crescita microbica abbia poi subìto un rallentamento a causa della carenza di ossigeno. Infatti, la biomassa non avendo mai superato i 50°C è stata rivoltata sporadicamente.
79
3.2.
A
NALISI MICROBIOLOGICHE COLTURA-DIPENDENTI
Le analisi microbiologiche consentono di valutare la quantità e le diverse tipologie microbiche coltivabili presenti in una matrice.
I risultati delle analisi microbiologiche effettuate sui campioni prelevati dalle sanse d’oliva in compostaggio nell’ambito della sperimentazione svolta presso la Cooperativa Olivicola di Arnasco sono riportati in Tabella 11.
Tabella 1: Risultati delle analisi microbiologiche svolte durante la sperimentazione ad Arnasco T0 (UFC/mL) T35 (UFC/mL) T100 (UFC/mL) T145 (UFC/mL) T200 (UFC/mL) Batteri aerobi coltivabili 1,09±0,12·10 8 1,17±0,40·107 1,68±0,10·107 5,13±0,57·107 8,97±0,30·107 Attinobatteri 2,67±0,33·106 1,33±0,33·105 5,93±0,73·106 1,42±0,23·108 5,43±0,23·107 Eumiceti unicellulari 1,67±0,33·10 4 1,00±0,02·106 9,33±0,67·105 4,67±088·105 2,0±0,58·105 Funghi filamentosi 1,47±0,23·10 5 3,60±0,10·105 3,40±0,11·105 9,33±0,33·105 1,83±0,19·106 Trichoderma spp. - - - 6,67±0,67·104 1,33±0,33·104
I batteri aerobi coltivabili sono risultati il gruppo microbico più abbondante all’inizio del processo e la loro presenza è rimasta pressoché costante fino alla fine dello stesso. Questi microrganismi sono decompositori veloci, capaci di utilizzare rapidamente le sostanze più degradabili e, perciò, sono i protagonisti della fase iniziale del processo. Inoltre dispongono di una variegata dotazione di enzimi necessari per la degradazione di numerosi composti che gli consente di svilupparsi anche su substrati più recalcitranti la degradazione e di essere coinvolti nelle reazioni di umificazione durante la fase di maturazione.
In generale, molti batteri aerobi sono capaci di degradare efficacemente alcuni composti fenolici, ma sono relativamente inefficienti contro i più complessi polifenoli caratteristici dei residui dell’estrazione olearia (Mc Namara et al., 2008). È noto che i funghi sono molto più efficienti rispetto ai batteri nella degradazione di tali composti. I risultati dell’analisi microbiologica evidenziano un aumento graduale dei micro-funghi coltivabili durante il processo. Solitamente durante lo stadio mesofilo del compostaggio i funghi filamentosi competono con i
80
batteri per i composti facilmente assimilabili, ma dato che i massimi tassi di crescita specifica dei batteri superano quelli dei funghi di un ordine di grandezza (Griffin, 1985), le muffe sono presenti in quantità inferiori rispetto ai batteri. Nella fase di raffreddamento e maturazione si osserva un leggero incremento di questi microrganismi, capaci di degradare i polimeri complessi, come l'amido, le cellulose e le lignine presenti nel substrato, contribuendo anche alla formazione delle sostanze umiche.
Gli attinobatteri coltivabili hanno avuto una leggera diminuzione dopo un mese, per aumentare fino a valori di circa 108 UFC/mL alla fine del processo. É accettato che gli attinobatteri giochino un ruolo fondamentale nella fase termofila del compostaggio, poiché possono tollerare alte temperature e pH alcalini (Ryckeboer et al., 2003; Toumela et al., 2000). Inoltre, sono dotati di enzimi che degradano composti recalcitranti relativamente complessi (Goodfellow e Williams, 1983), come chitina, lignina, proteine, emicellulose e cellulosa, contribuendo alla formazione di sostanze umiche ed alla completa maturazione del substrato.
I lieviti, dopo un leggero incremento iniziale, sono costantemente diminuiti in piccola misura fino alla fine del processo. È noto che tali microrganismi prosperano sui composti facilmente assimilabili presenti nelle fasi iniziali del compostaggio, per poi ricoprire un ruolo marginale nel resto del processo. Nei residui solidi dell’estrazione olearia in compostaggio prosperano microrganismi come Geotrichum candidum (Assas et al., 2000; Giannoutsou et al., 2004),
Candida tropicalis (Fadil et al., 2003), e Saccharomyces sp. (Giannoutsou et al.,
2004) capaci di utilizzare i composti fenolici.
In letteratura la presenza di microrganismi del genere Bacillus nelle sanse in compostaggio è nota, ed è correlata alla loro capacità di produrre tannasi. Nell’ambito della presente tesi le sanse d’oliva compostate sono state inoculate con Bacillus amyloliquefaciens subsp. plantarum ceppo - M51/II, che è un ceppo batterico particolare, in quanto è termo-tollerante e presenta una doppia resistenza agli antibiotici streptomicina e rifampicina. È stato quindi possibile ricercarlo nella matrice mediante i campionamenti effettuati sulla stessa durante l’arco del processo. I risultati ottenuti sono discontinui, in quanto tale microrganismo è stato
81
rilevato solo al tempo 0 giorni (2,33±0,88·102 UFC/mL) e dopo 100 giorni dall’inizio del processo (8,50±0,50·103 UFC/mL). È quindi probabile che questo microrganismo fosse presente ma non rilevabile anche negli altri stadi del processo. Questa ipotesi è supportata dai risultati ottenuti mediante le analisi molecolari PCR-DGGE, in cui la presenza del frammento ascrivibile al batterio
Bacillus amyloliquefaciens subsp. plantarum è stata ritrovata durante tutte le
diverse fasi del processo (vedi paragrafo 3.3.1).
Al fine di incrementare il contenuto in Trichoderma spp. del compost, durante la fase di maturazione, la biomassa è stata inoculata con lo “starter B”, in cui la presenza di Trichoderma spp. era di 1,57±0,12·106 (UFC/mL). Tale microrganismo è un fungo comune della rizosfera, da tempo riconosciuto come un valido agente di biocontrollo dei patogeni fungini delle piante (Chet, 1987). Infatti svolge attività antagonistica poiché limita fortemente la crescita di funghi patogeni sfruttando vari meccanismi, quali: la produzione di sostanze antibiotiche, la competizione per gli elementi nutritivi, il mico-parassitismo e l’induzione nella pianta di resistenze sistemiche o localizzate. Inoltre è in grado di instaurare un'associazione con la pianta che promuove la crescita della stessa, accresce la disponibilità di sostanze nutritive e in generale migliora le capacità produttive delle colture (Dennis e Webster, 1971; Harman et al., 2004; Marra et al., 2006).
Per queste caratteristiche è auspicabile che Trichoderma spp. sia presente nel compost finito. Tuttavia, i risultati dell'analisi microbiologica evidenziano che il micro-fungo inoculato nella biomassa in fase di maturazione e raffreddamento non si è sviluppato ulteriormente. Infatti la densità di Trichoderma atroviride nel compost ottenuto dalle sanse d'oliva Pignola e Taggiasca della Cooperativa Olivicola di Arnasco è risultata inferiore, in proporzione, rispetto a quella ottenuta nello “starter B”. Si può ipotizzare che il fungo non abbia trovato le condizioni ottimali per la crescita a causa delle elevate temperature della stagione estiva che favoriscono l’evaporazione dell’acqua presente nella matrice e a causa del basso apporto di ossigeno in seguito alla sporadica movimentazione che richiede la biomassa in questa fase del compostaggio.
82
3.2.1. Isolamento di attinobatteri
Gli attinobatteri, seppur presenti durante l’intero processo di compostaggio, rivestono un ruolo fondamentale soprattutto nella fase di maturazione e raffreddamento del compostaggio. Infatti, tali microrganismi sono dotati di una serie di enzimi capaci di solubilizzare la lignina, quindi, insieme ai funghi, giocano un ruolo significativo nella degradazione dei composti ligno-cellulosici e nella formazione delle sostanze umiche.
Recentemente Federici et al. (2011) hanno isolato, da sanse d’oliva in compostaggio, ceppi di batteri capaci di degradare composti come tannini e polifenoli, solitamente presenti in questa matrice. Tra questi molti appartenevano a specie diverse di attinobatteri, confermando il ruolo fondamentale che rivestono nel compostaggio dei residui dell’estrazione olearia.
Nell’ambito della presente tesi sono state isolate in coltura pura colonie di attinobatteri presenti nei campioni di sansa d'oliva analizzati. In particolare, sono state isolate 31 colonie, scelte sulla base di differenze morfologiche, quali il colore e la forma, e sono state inserite nella collezione del DBPA (Dipartimento di Biologia delle Piante Agrarie) dell'Università di Pisa. Alcune delle colonie isolate durante l’analisi microbiologica, riferibili ad attinobatteri, sono indicate a titolo di esempio con frecce in colore in Figura 21.
Figura 2: Colonie di attinobatteri (alcuni esempi sono indicati dalle frecce) cresciuti su
83
Le caratteristiche dei ceppi batterici isolati, capaci di accrescersi su sanse d'oliva Taggiasca e Pignola, potranno essere indagate successivamente, allo scopo di valutare la presenza di ceppi che favoriscono la degradazione di questa matrice, come l’attività tannasica. Le sanse vergini, infatti, a causa delle loro caratteristiche particolari, forniscono una fonte di “nuovi” microrganismi con un potenziale biotecnologico diverso, come il batterio Paenibacillus jamilae produttore di esopolisaccaridi estratto dai residui dell’estrazione olearia (Aguilera et al., 2001). È quindi probabile che gli attinobatteri isolati dalle sanse vergini di Arnasco, essendo indigeni di questa matrice, abbiano sviluppato meccanismi di difesa e/o di degradazione dei composti anti-microbici presenti nelle sanse vergini prodotte dalla Cooperativa Olivicola di Arnasco e potrebbero essere utilizzati anch’essi come starter per velocizzare il processo di compostaggio delle stesse.
84
3.3.
A
NALISI MOLECOLARI COLTURA-INDIPENDENTI
Attraverso le tecniche coltura-dipendenti, la conoscenza e la comprensione delle dinamiche ecologiche che si instaurano tra le comunità microbiche rimangono limitate. Infatti tali tecniche non consentono un adeguato studio dell’immensa estensione della diversità microbica nell’ambiente naturale (Nübel
et al., 1999) soprattutto alla luce della coltivabilità dei microrganismi che risulta
essere molto bassa in quasi tutti gli habitat (<1%; Amann et al., 1995).
Al contrario, i metodi molecolari basati sull’amplificazione dei geni ribosomiali mediante PCR hanno permesso di ottenere informazioni sulla diversità eliminando i problemi associati alla coltivazione (Wagner and Loy, 2002). Alcuni studi hanno dimostrato che taxa con numero di cellule ≥ 1% rispetto al totale possono essere analizzati con le tecniche dipendenti dalla PCR (Muyzer et al., 1993; Murray et al., 1998), e solo taxa < 0,1% sono difficilmente rintracciabili.
Tra i metodi di fingerprinting genetico la DGGE permette la visualizzazione diretta della diversità microbica del campione ambientale, la comparazione rapida delle comunità e l’identificazione di popolazioni mediante l’analisi di sequenza.
L’elettroforesi su gel di poliacrilammide con gradiente di denaturanti chimici si basa sul principio per cui ogni frammento amplificato, ottenuto utilizzando primers specifici per il 16S rDNA e per il 26S rDNA delle popolazioni batteriche e fungine rispettivamente, ha un suo punto specifico di denaturazione. Pertanto è possibile distinguere i frammenti, caratterizzati dalla stessa lunghezza ma da una differente sequenza in basi azotate (Muyzer et al., 1993). Dopo la colorazione è possibile visualizzare i frammenti presenti sul gel, ciascuno dei quali rappresenta una specifica sequenza di 16S rDNA o 26S rDNA appartenente ad una specifica specie microbica. La posizione del frammento sul gel permette di identificare la specie (Stamper et al., 2003; Hoefel et al., 2005; Miura et al., 2007), e la sua concentrazione è determinata dall’intensità del frammento (Dar et al., 2005; Connaughton et al., 2006). Tuttavia, frammenti di DNA di sequenza diversa possono avere caratteristiche di mobilità simili nel gel
85
di poliacrilammide e co-migrare nella stessa posizione (Gelsomino et al., 1999; Sekiguchi et al., 2001; Speksnijder et al., 2001). Inoltre, una singola specie microbica può dare origine a bande multiple, dato che i microrganismi possiedono più operoni per i geni ribosomiali, le cui sequenze possono essere lievemente differenti tra di loro (Brosius et al., 1981; Nübel et al., 1997). Infine, l’intensità dei frammenti può non riflettere la reale abbondanza delle varie popolazioni all’interno della comunità, poiché tale parametro è dipendente dal numero totale di popolazioni presente nel campione.
In ecologia microbica il concetto di specie, intesa come unità tassonomica su cui si basano i processi ecologici, è stato associato all’unità tassonomica operazionale (Operational Taxonomic Unit, OTU). Le OTUs corrispondono per esempio alle bande in un gel elettroforetico.
È possibile stimare la diversità delle specie batteriche o fungine nei diversi campioni attraverso l’utilizzo di vari indici di diversità, quali:
• Richness (S): numero di frammenti in ogni lane, ovvero il numero delle differenti OTU in ciascun campione
• Indice di Shannon-Weaver (H): indice di diversità generale • Indice di Simpson (D): indice di dominanza
• Evenness (E): importanza relativa delle diverse OTUs in ogni campione
Tali indici sono calcolati usando l’altezza ovvero l’intensità del picco che si assume rappresenti la relativa abbondanza di ciascuna specie.
3.3.1. Diversità della comunità batterica durante le fasi del processo di compostaggio
I profili ottenuti dalla separazione elettroforetica su gel denaturante di poliacrilammide degli amplificati della regione V3-V5 del DNA ribosomiale
estratto dai campioni analizzati sono riportati in Figura 22.
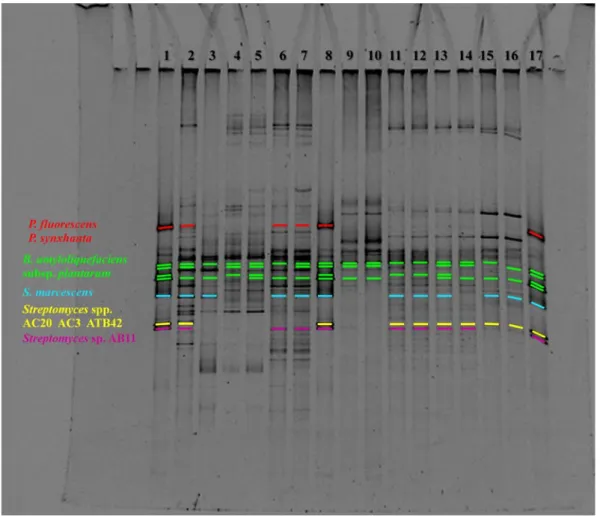
Utilizzando la matrice elaborata dal software Image Master Elite (Pharmacia Biotech) è stato possibile individuare i generi e le specie microbiche utilizzate come riferimento (lane 1, 8, 17).
86
Figura 3: Profili DGGE dei prodotti PCR delle regioni V3-V5 del 16S rDNA dei campioni di
compost analizzati. 1, 8, 17 Marker: Pseudomonas synxanta 2/3, Bacillus amyloliquefaciens subsp. plantarum M51/II, Serratia marcescens B2, Streptomyces sp. AB11, Streptomyces sp. AC20; 2, 3: biomassa al tempo 0 gg; 4, 5: biomassa al tempo 35 gg; 6, 7: biomassa al tempo 100 gg; 9, 10: “starter B”; 11, 12: biomassa al tempo 145 gg; 13, 14: biomassa al tempo 200 gg; 15, 16: campione di argilla da biomassa al tempo 200 gg
Gruppi batterici appartenenti al genere Bacillus e Pseudomonas sono tipici delle matrici in compostaggio, e diversi autori ne hanno evidenziato differenti specie durante il processo biologico (Tuomela et al., 2000; Hassen et al., 2001; Baeta-Hall et al., 2004). Baeta-Hall et al. (2004) hanno isolato, nella fase iniziale del processo di compostaggio di sansa, due ceppi di Pseudomonas capaci di degradare i composti fenolici.
Nell’ambito di questa tesi Pseudomonas spp. risulta presente nei campioni iniziali e al termine della fase pseudo-termofila, ovvero nel periodo di attiva decomposizione. Anche alcune specie del genere Bacillus sono capaci di degradare i composti fenolici. Ramos-Cormenzana et al. (1996) hanno osservato che Bacillus pumilis è capace di degradare fino al 50% dei composti fenolici
87
presenti nelle acque di vegetazione derivate dall’estrazione olearia. I risultati ottenuti evidenziano la presenza di Bacillus amyloliquefaciens subsp. plantarum durante tutto il processo di compostaggio. In particolare l’intensità dei frammenti ascrivibili a tale batterio nei profili DGGE al tempo 0 e 100 giorni, dove era stato rilevato anche mediante analisi microbiologica, risulta maggiore.
La presenza di Serratia marcescens è stata rilevata durante tutto l’arco del processo, eccetto che al tempo 35 giorni. Recentemente Pepi et al. (2010) hanno isolato specie appartenenti al genere Serratia direttamente da sanse vergini ed hanno dimostrato la loro capacità di utilizzare come unica fonte di carbonio tannini idrolizzabili e di degradarli ad acido gallico e glucosio.
Infine, le specie appartenenti al genere Streptomyces risultano praticamente assenti all’inizio del processo, cominciano a svilupparsi alla fine della fase pseudo-termofila per essere presenti durante tutto l’arco della fase di maturazione e finissaggio. Tale risultato è in accordo con i risultati dell’analisi microbiologica, che evidenzia un incremento del numero di attinobatteri nelle sanse in fase di maturazione rispetto a quelle in fase bio-ossidativa.
I profili DGGE evidenziano la dominanza di specifici gruppi microbici, corrispondenti ai frammenti più intensi, nei diversi stadi del processo. Potrebbe essere interessante determinare a quale specie batterica appartengano tali frammenti, mediante eluizione del loro DNA, amplificazione, sequenziamento e confronto delle sequenze ottenute con quelle depositate in banca dati.
L’evoluzione della diversità delle popolazioni batteriche presenti nelle sanse d’oliva Pignola e Taggiasca durante il processo di compostaggio è stata valutata utilizzando gli indici di diversità, quali: la Richness (S), l’indice di
Shannon-Weaver (H), l’indice di Simpson (D) e la Evenness (E). Tali indici stimano la
diversità strutturale dei campioni basandosi sia sul numero che sull’intensità dei frammenti ottenuti. I dati elaborati sono riportati in Tabella 12.
88
Tabella 2: Richness (S), indice di Shannon-Weaver (H), indice di Simpson (D) ed Evenness (E)
calcolati dai profili DGGE della comunità batterica ottenuti dai campioni prelevati durante il compostaggio delle sanse vergini d’oliva Taggiasca e Pignola. 2, 3: biomassa al tempo 0 gg; 4, 5: biomassa al tempo 35 gg; 6, 7: biomassa al tempo 100 gg; 11, 12: biomassa al tempo 145 gg; 13, 14: biomassa al tempo 200 gg. T0 T35 T100 T145 T200 Lane 2 3 4 5 6 7 11 12 13 14 S 30 18 22 23 36 38 29 28 26 27 H 3,3920 2,8784 3,0865 3,1290 3,5738 3,6280 3,3616 3,3269 3,2551 3,2921 D 0,0340 0,0570 0,0459 0,0441 0,0283 0,0268 0,0349 0,0361 0,0387 0,0373 E 0,9973 0,9958 0,9985 0,9979 0,9973 0,9974 0,9983 0,9984 0,9991 0,9989
I risultati ottenuti indicano la presenza di un’alta diversità microbica nella matrice in compostaggio e la rapida successione di popolazioni batteriche durante il processo, come evidenziato dagli elevati valori di Evenness (E).
Gli alti valori di Richness (S) e dell’indice di Shannon-Weaver (H) e i bassi valori dell’indice di Simpson (D) indicano un marcato aumento della diversità microbica al termine della fase pseudo-termofila (T100 gg). Seppur con una leggera riduzione, la diversità batterica rimane elevata anche nei campioni successivi (T145 gg e T200 gg). I valori dell’indice di Simpson ottenuti in tali campioni, evidenziano una maggiore uniformità tra le OTUs presenti, suggerendo una graduale stabilizzazione della struttura della popolazione batterica nel tempo.
Tale risultato, ottenuto anche da Federici et al. (2011), è coerente con il processo di stabilizzazione della sostanza organica, inizialmente caratterizzato da una fase di bio-degradazione attiva, a cui segue la stabilizzazione del substrato.
La graduale stabilizzazione delle popolazioni microbiche nel tempo è confermata dall’analisi del dendrogramma (Figura 23) generato dalla matrice elaborata con il software ImageMaster 1D Elite v3.00 (Pharmacia Biotech), dei profili DGGE riportati nella Figura 22. La similarità dei profili è stata calcolata utilizzando il coefficiente Dice basato sulla presenza-assenza delle diverse OTUs e, sulla base dei coefficienti ottenuti, i diversi profili sono stati raggruppati utilizzando il metodo di clustering UPGMA (Unweighted Pair Group Method Using Arithmetic Average).
89
Figura 4: Dendrogramma generato dalla matrice di similarità ottenuto con metodo di clustering
UPGMA (Unweighted Pair Group Method Using Arithmetic Average) basato sui profili DGGE. Le relazioni tra i campioni sono basati sulla similarità, valutata utilizzando il coefficiente Dice. 1, 8, 17 Marker: Pseudomonas synxanta 2/3, Bacillus amyloliquefaciens subsp. plantarum M51/II,
Serratia marcescens B2, Streptomyces sp. AB11, Streptomyces sp. AC20; 2, 3: biomassa al tempo
0 gg; 4, 5: biomassa al tempo 35 gg; 6, 7: biomassa al tempo 100 gg; 9, 10: “starter B”; 11, 12: biomassa al tempo 145 gg; 13, 14: biomassa al tempo 200 gg; 15, 16: campione di argilla da biomassa al tempo 200 gg
In particolare, è possibile individuare un cluster principale separato dallo “starter B”, col quale ha un grado di similarità del 40%. Anche se la matrice in esame è la medesima, è probabile che l’aggiunta di Trichoderma atroviride su crusca abbia influenzato lo sviluppo della comunità batterica del compost in maturazione. Trichoderma spp. è un microrganismo che produce sostanze attive che possono aver inibito la crescita di alcune specie microbiche, in accordo con il ridotto numero di frammenti ottenuti nei campioni di “starter B”.
All’interno del cluster principale sono presenti tutti i campioni di sansa in compostaggio. Al 47% di similarità troviamo l’argilla mescolata al compost
90
maturo, che evidentemente ha incorporato parte della microflora presente nel compost. Seguono al 56% di similarità tutti i campioni di compost. All’interno un primo sotto-cluster è costituito dai campioni al tempo 35 giorni, che sono fra loro più simili dei campioni di compost a livelli di maturazione superiore, per i quali si è avuta, in seguito al processo, un’elevata diversificazione delle popolazioni microbiche. L’altro sotto-cluster comprende i campioni a più elevata maturazione: i campioni a tempo 100 giorni, simili al 60%, si staccano dai campioni relativi al tempo 145 giorni e 200 giorni, che si raggruppano ad un livello di similarità leggermente superiore (71%), i tempi corrispondono infatti alle fasi finali di maturazione e finissaggio, caratterizzati dalla stabilizzazione del substrato e delle popolazioni microbiche.
Tutte le repliche oscillano a livelli elevati di similarità, compresi tra l’80% e il 97%, con l’unica eccezione dei campioni al tempo 0 giorni, che risultano dissimili.
3.3.2. Diversità della comunità fungina durante le fasi del processo di compostaggio
I profili ottenuti dalla separazione elettroforetica su gel denaturante di poliacrilammide degli amplificati della regione D1-D2 del DNA ribosomiale
91
Figura 5: Profili DGGE dei prodotti PCR delle regioni D1-D2 del 26S rDNA dei campioni di
compost analizzati. 1, 8, 17 Marker: Sporopachydermia lactativora 2(3), Candida butyri 8(4),
Phanerochaete chrysosporium ATCC42538, Trichoderma atroviride T14, Chaetomium globosum
ChT3; 2, 3: biomassa al tempo 0 gg; 4, 5: biomassa al tempo 35 gg; 6, 7: biomassa al tempo 100 gg; 9, 10: “starter B”; 11, 12: biomassa al tempo 145 gg; 13, 14: biomassa al tempo 200 gg; 15, 16: argilla.
Utilizzando la matrice elaborata dal software Image Master Elite (Pharmacia Biotech) è stato possibile individuare i generi e le specie microbiche utilizzate come riferimento (lane 1, 8, 17).
In particolare, la presenza delle specie Rhodotorula mucillaginosa, Candida
butyri e Trichoderma atroviride, è stata evidenziata durante la fase di maturazione
e raffreddamento della biomassa. In questa fase, successiva all’inoculo dello “starter B”, la presenza di Trichoderma spp. era attesa, ed ha confermato i risultati ottenuti dall’analisi microbiologica.
Alcune specie di Candida sono capaci di ridurre il contenuto fenolico dei residui dell’estrazione oleari (Fadil et al., 2003; Giannoutsou et al., 2004), ma
92
essendo microrganismi mesofili solitamente sono presenti nella fase iniziale del processo di compostaggio. I profili DGGE dello “starter B” evidenziano, oltre alla presenza di Trichoderma spp., anche quella di Candida butyri.
Chaetomium globosum, Phanerochaete chrysosporium e Arthrobotrys oligospora erano presenti nella matrice al tempo 0 giorni e successivamente non
sono stati rilevati. È possibile che la crescita di queste specie fungine sia stata limitata dall’innalzamento della temperatura e dalla carenza d’ossigeno del substrato.
Come detto a riguardo dei batteri, al fine di identificare ceppi fungini specifici per questa matrice, potrebbe essere interessante studiare a quale specie appartengano i frammenti dominanti nei profili DGGE.
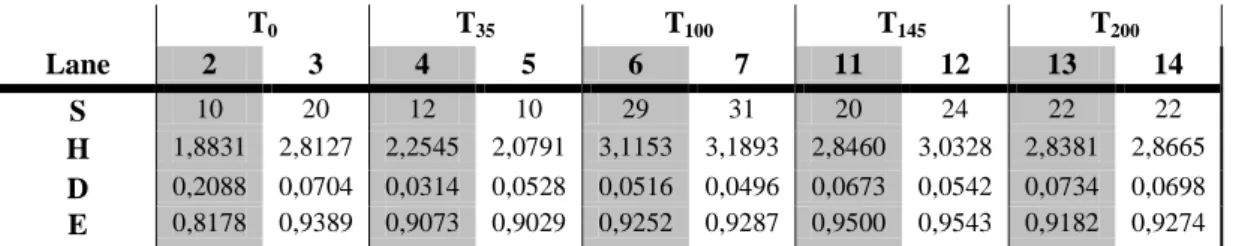
L’evoluzione della diversità delle popolazioni fungine presenti nelle sanse d’oliva Pignola e Taggiasca durante il processo di compostaggio è stata valutata utilizzando gli indici di diversità, quali: la Richness (S), l’indice di
Shannon-Weaver (H), l’indice di Simpson (D) e la Evenness (E). Tali indici stimano la
diversità strutturale dei campioni considerando sia il numero che l’intensità dei frammenti ottenuti. I dati elaborati sono riportati in Tabella 13.
Tabella 3: Richness (S), indice di Shannon-Weaver (H), indice di Simpson (D) ed Evenness (E)
calcolati dai profili DGGE della comunità fungina ottenuti dai campioni prelevati durante il compostaggio delle sanse vergini d’oliva Taggiasca e Pignola. 2, 3: biomassa al tempo 0 gg; 4, 5: biomassa al tempo 35 gg; 6, 7: biomassa al tempo 100 gg; 11, 12: biomassa al tempo 145 gg; 13, 14: biomassa al tempo 200 gg. T0 T35 T100 T145 T200 Lane 2 3 4 5 6 7 11 12 13 14 S 10 20 12 10 29 31 20 24 22 22 H 1,8831 2,8127 2,2545 2,0791 3,1153 3,1893 2,8460 3,0328 2,8381 2,8665 D 0,2088 0,0704 0,0314 0,0528 0,0516 0,0496 0,0673 0,0542 0,0734 0,0698 E 0,8178 0,9389 0,9073 0,9029 0,9252 0,9287 0,9500 0,9543 0,9182 0,9274
I risultati ottenuti evidenziano che la comunità fungina è altamente differenziata, anche se tale diversità è inferiore a quella della popolazione batterica, come indicato dai valori di Evenness (E) stimati.
Nei primi stadi del processo di compostaggio (T0 gg e T35gg) la diversità fungina è abbastanza ridotta e, come nei procarioti, aumenta fortemente alla fine della fase pseudo-termofila (T100 gg), caratterizzata da alti valori degli indici di
93
Richness (S) e Shannon-Weaver (H) e da ridotti valori dell’indice di Simpson (D).
Infine, nella fase di raffreddamento e maturazione (T145 gg e T200 gg), dove la matrice in compostaggio è stabilizzata, la diversità microbica diminuisce leggermente. I valori dell’indice di Simpson ottenuti in tali campioni, evidenziano una maggiore uniformità tra le OTUs presenti, suggerendo una graduale stabilizzazione della struttura della popolazione fungina nel tempo.
Dall’osservazione dei profili DGGE si evidenzia, inoltre, la dominanza di alcune specie microbiche in determinati stadi del processo, indicando il ruolo fondamentale svolto da microrganismi specializzati durante la bio-trasformazione della sansa vergine a compost maturo.
Al fine di valutare la similarità della comunità fungina presente nei campioni analizzati durante il processo di compostaggio è stato elaborato il dendrogramma (Figura 25), generato dalla matrice ottenuta mediante l’analisi con il software ImageMaster 1D Elite v3.00 (Pharmacia Biotech), dei profili DGGE riportati in Figura 24. La similarità dei profili è stata calcolata utilizzando il coefficiente Dice, basato sulla presenza-assenza delle diverse OTUs e, sulla base dei coefficienti ottenuti, i diversi profili sono stati raggruppati utilizzando il metodo di clustering UPGMA (Unweighted Pair Group Method Using Arithmetic Average).
94
Figura 6: Dendrogramma generato dalla matrice di similarità ottenuto con metodo di clustering
UPGMA (Unweighted Pair Group Method Using Arithmetic Average) basato sui profili DGGE. Le relazioni tra i campioni sono basati sulla similarità, valutata utilizzando il coefficiente Dice. 1, 8, 17 Marker: Sporopachydermia lactativora 2(3), Candida butyri 8(4), Phanerochaete
chrysosporium ATCC42538, Trichoderma atroviride T14, Chaetomium globosum ChT3; 2, 3:
biomassa al tempo 0 gg; 4, 5: biomassa al tempo 35 gg; 6, 7: biomassa al tempo 100 gg; 9, 10: “starter B”; 11, 12: biomassa al tempo 145 gg; 13, 14: biomassa al tempo 200 gg; 15, 16: argilla.
Dall’analisi del dendrogramma è possibile individuare un primo cluster costituito dai campioni al tempo 0 giorni e al tempo 35 giorni, che presentano una bassa similarità rispetto a tutti gli altri campioni, 27% e 31% rispettivamente. Al 37% di similarità troviamo l’argilla mescolata al compost maturo, che come per la comunità batterica, ha incorporato parte della microflora presente nella biomassa processata. Lo “starter B” presenta una similarità del 54% con i campioni di compost a livelli di maturazione superiore, evidenziando che tale inoculo ha un’influenza relativamente bassa sulle popolazioni fungine, rispetto a quella che si è potuta notare nella comunità batterica. Infine, al 58% di similarità, si evidenzia un sotto-cluster costituito dai campioni relativi alle fasi più avanzate del processo
95
di compostaggio. In particolare i campioni al tempo 100 giorni si differenziano dai campioni al tempo 145 giorni e 200 giorni, in cui gli alti valori di similarità (66%) suggeriscono una stabilizzazione nella struttura della comunità fungina. Come per la comunità batterica, è evidenziabile un’elevata diversificazione delle popolazioni fungine, parallela alle fasi evolutive del processo di compostaggio, che conferma i risultati evidenziati dagli indici di diversità (Tabella 13).
Tutte le replicazioni oscillano a livelli elevati di similarità, compresi tra l’87% e il 100%. A valori leggermente inferiori troviamo le repliche dello “starter B” (77%) e i campioni relativi al tempo 0 giorni (67%).
La tecnica PCR-DGGE è stata utilizzata per analizzare la comunità microbica durante il processo di compostaggio di varie matrici agricole (Takaku
et al., 2006; Novinscak et al., 2009; Zhang et al., 2010). In accordo con i risultati
ottenuti, questi autori hanno riportato che la struttura della comunità variava ampiamente durante le diverse fasi del processo e tendeva a stabilizzarsi in corrispondenza della maturità raggiunta dal compost. Infatti, a causa della complessità dei substrati e dei prodotti intermedi che possono formarsi durante il processo, la diversità microbica e la successione di popolazioni specializzate sono prerequisiti che assicurano la bio-degradazione dei residui organici e la maturazione del compost.
Risultati contrastanti sono riportati dagli stessi autori in merito al periodo di massima variazione della diversità microbica. I risultati ottenuti in questo lavoro sono in accordo con quelli ottenuti da Novinscak et al. (2009), dove un aumento della diversità microbica è stato osservato alla fine della fase termofila.
Gli studi sviluppati al fine di analizzare la variazione della diversità microbica durante il processo di compostaggio delle sanse d’oliva, mediante tecniche coltura-indipendenti sono molto pochi. Recentemente Federici et al. (2011) hanno analizzato la dinamica della struttura della comunità batterica durante le diverse fasi del processo di compostaggio dei residui solidi dell’estrazione olearia, utilizzando la tecnica PCR-DGGE. Essi hanno evidenziato una variazione sia quantitativa che qualitativa della comunità batterica associata alle diverse fasi del processo, con un picco di massima variazione osservato durante la fase termofila. Ad oggi, invece, non sono disponibili in letteratura
96
lavori che riguardano lo studio della popolazione fungina durante il compostaggio delle sanse d’oliva effettuato mediante la tecnica PCR-DGGE.