3. RISULTATI E DISCUSSIONE
3.1 BOZA
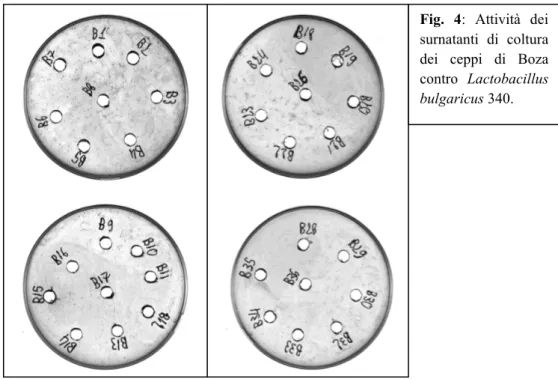
Nell’ambito della ricerca di batteri lattici produttori di batteriocine nel Boza, nessuno dei 33 ceppi analizzati ha mostrato attività contro i microrganismi indicatori. In Figura 4 sono riportati i risultati delle analisi finalizzate alla ricerca di batteriocine, che hanno visto l’impiego di Lactobacillus Bulgaricus 340 come microrganismo indicatore.
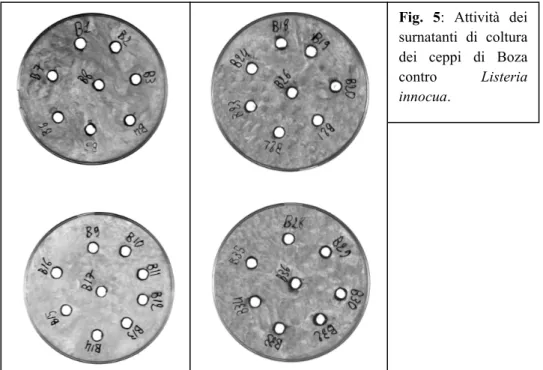
In Figura 5 sono riportati i risultati della ricerca di batteriocine attive contro
Listeria innocua.
Fig. 4: Attività dei surnatanti di coltura dei ceppi di Boza contro Lactobacillus
Come microrganismo indicatore è stato utilizzato anche Lactobacillus brevis (Fig. 6). In questo caso le batteriocine contenute nel surnatante di coltura di
Enterococcus durans A51 sono state utilizzate come controllo positivo.
Fig. 5: Attività dei surnatanti di coltura dei ceppi di Boza contro Listeria innocua.
Fig. 6: Attività dei surnatanti di coltura dei ceppi di Boza contro
Lactobacillus brevis B33 B32 B30 B36 B35 B34 A51 B29 B28 B1 B2 B3 B4 B5 B6 B15 B19 B18 B14 B16 B17 B13 B11 B12 B7 B8 B9 B10 B22 B23 B24 B26 B21 B20
La presenza di una zona più chiara intorno al pozzetto contenente il campione di controllo A51 indica la mancata crescita del microrganismo indicatore e quindi la possibile presenza di batteriocine. Questa zona non è individuabile intorno ai ceppi isolati dal Boza.
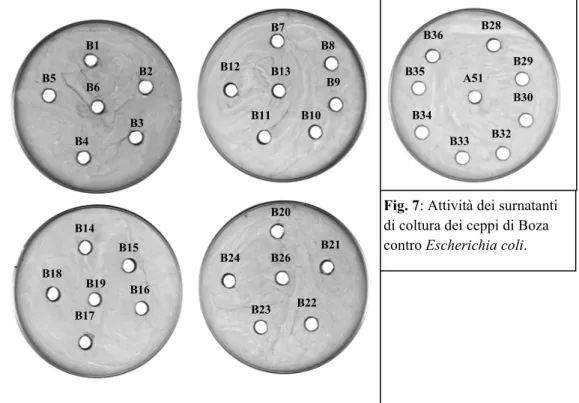
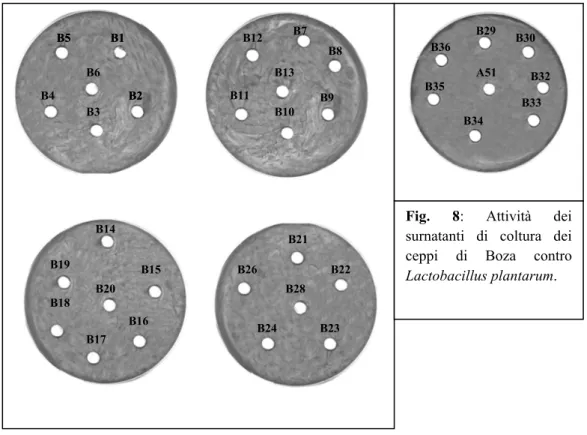
Microrganismi produttori di batteriocine sono stati infine ricercati utilizzando come microrganismi indicatori Escherichia coli (Fig. 7) e Lactobacillus
plantarum (Fig. 8). B28 B36 A51 B35 B34 B33 B32 B30 B29
Fig. 7: Attività dei surnatanti di coltura dei ceppi di Boza contro Escherichia coli.
B1 B2 B3 B4 B5 B6 B13 B11 B12 B7 B8 B9 B10 B15 B19 B18 B14 B16 B17 B23 B22 B24 B26 B21 B20
In entrambi i casi, non sono stati rilevati ceppi produttori di batteriocine. Anche le batteriocine prodotte da Enterococcus durans A5-11 si sono dimostrate inattive.
I microrganismi utilizzati come indicatori (Lactobacillus bulgaricus 340, Listeria
innocua, Lactobacillus brevis, Escherichia coli e Lactobacillus plantarum) sono,
generalmente, ceppi sensibili alle batteriocine e sono stati utilizzati, nel presente elaborato, come campione rappresentativo dei microrganismi sensibili a queste sostanze proteiche. Pertanto, in base ai risultati ottenuti, è possibile escludere la presenza, tra i 33 ceppi isolati dal Boza, di microrganismi produttori di batteriocine.
Considerando che, il Boza è una bevanda che varia molto nella composizione della flora batterica a seconda delle zone di produzione e del substrato di partenza e che, generalmente, la produzione di batteriocine è caratteristica di circa il 5% dei batteri lattici (Geis et al., 1983; Todorov et al., 2005) non sorprende il fatto di non aver identificato microrganismi produttori di batteriocine nel Boza.
B1 B5 B6 B2 B3 B4 B7 B8 B9 B11 B10 B12 B13 B28 B26 B24 B23 B22 B21 B14 B19 B20 B16 B15 B17 B18 B1 B5 B2 B29 A51 B36 B30 B35 B33 B34 B32 B23
Fig. 8: Attività dei surnatanti di coltura dei ceppi di Boza contro
3.2 PROFILO ANTIBATTERICO DI ENTEROCOCCUS DURANS
Le due batteriocine prodotte da Enterococcus durans A5-11 hanno mostrato attività antibatterica nei confronti di Lactobacillus bulgaricus 340, Listeria
innocua e Lactobacillus brevis. Non è stata invece individuata attività nei
confronti di Escherichia coli e Lactobacillus plantarum (Fig. 9).
Fig. 9: profilo antibatterico delle batteriocine prodotte da Enterococcus durans.
Osservando la zona di inibizione nei confronti dei microrganismi indicatori, è possibile concludere che i microrganismi Lactobacillus brevis e Lactobacillus
bulgaricus 340, sono maggiormente sensibili alle due batteriocine. E’ però la
presenza di inibizione nella piastra contenente Listeria innocua ad interessare maggiormente, poiché, test futuri, potrebbero rilevare la sensibilità alle batteriocine prodotte da Enterococcus durans, anche da parte di ceppi patogeni di
Listeria.
3.2.1 Cromatografia a scambio cationico
La cromatografia a scambio cationico è stata utilizzata per purificare le batteriocine prodotte da Enterococcus durans. Dall’analisi del grafico (Fig. 10) è osservabile che le due batteriocine sono state eluite tra 40–60 mS, corrispondente ad una concentrazione di NaCl tra 0,4 e 0,6 M (ad indicare che le due batteriocine sono dei composti cationici altamente caricati).
A5-11 contro Lb340 A5-11 contro Listeria A5-11 contro Lb brevis A5-11 contro Lb plantarum A5-11 contro E. coli
Fig. 10: cromatografia a scambio cationico.
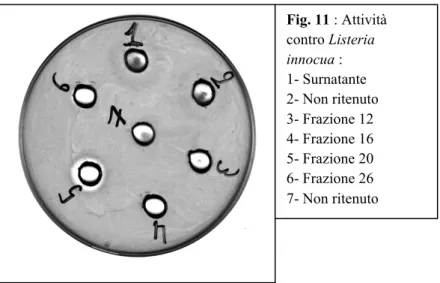
Nelle due figure che seguono (Fig. 11 e 12) sono riportati i risultati dell’analisi dell’attività antibatterica di alcune frazioni collezionate durante la cromatografia. Attività maggiore si ha nelle frazioni eluite tra 40–60 mS, ed in particolare in presenza del picco rilevato in corrispondenza della frazione 20. In questa frazione sono, infatti, maggiormente contenute le due batteriocine.
Fig. 11 : Attività contro Listeria innocua : 1- Surnatante 2- Non ritenuto 3- Frazione 12 4- Frazione 16 5- Frazione 20 6- Frazione 26 7- Non ritenuto
(s)
mS conduttività ionica 220 nm Assorbanza 12 16 20 26 mS 220 nm tempo (s)3.2.2 Cromatografia a fase inversa
Le frazioni contenenti attività antibatterica, collezionate tramite cromatografia a scambio cationico, sono state sottoposte a cromatografia a fase inversa grazie alla quale le due batteriocine sono state purificate e concentrate. L’analisi del grafico (Fig. 13) dimostra che i picchi relativi alle due batteriocine (frazioni 19 e 20) sono in parte sovrapposti e pertanto non è stato possibile separarle.
Fig. 13: cromatografia a fase inversa.
Fig. 12 : Attività contro
Lb340 : 1- Non ritenuto 2- Surnatante 3- Frazione 12 4- Frazione 16 5- Frazione 20 6- Frazione 26 7- Non ritenuto mS 220 nm
(s)
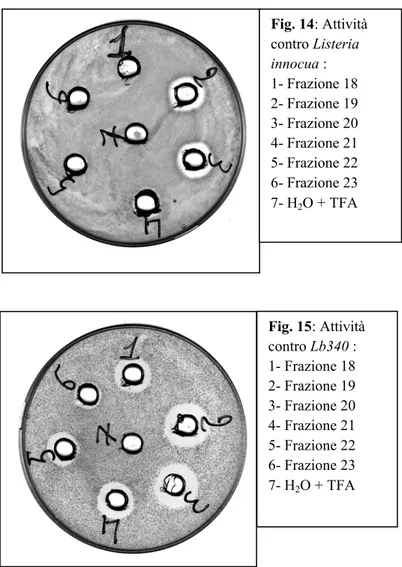
220 nm AssorbanzamS conduttività ionica 19 21 23 18 20 22 220 nm tempo (s) mSMediante il metodo della diffusione in pozzetti è stato dimostrato che l’attività antibatterica è maggiore in corrispondenza dei due picchi, per poi diminuire agli estremi (Fig. 14 e 15).
Dall’analisi della dimensione delle zone di inibizione è possibile dedurre come, a seguito delle due cromatografie, l’attività antibatterica delle due batteriocine risulti maggiore.
3.3 ATTIVITÀ DI ENTEROCOCCUS DURANS CONTRO IL FUNGO FUSARIUM GRAMINEARUM
Il surnatante di coltura ottenuto tramite centrifugazione di una coltura di
Enterococcus durans A5-11 è stato testato, assieme alle batteriocine da lui
Fig. 14: Attività contro Listeria innocua : 1- Frazione 18 2- Frazione 19 3- Frazione 20 4- Frazione 21 5- Frazione 22 6- Frazione 23 7- H2O + TFA Fig. 15: Attività contro Lb340 : 1- Frazione 18 2- Frazione 19 3- Frazione 20 4- Frazione 21 5- Frazione 22 6- Frazione 23 7- H2O + TFA
prodotte e purificate a seguito di una cromatografia a fase inversa, contro il fungo
Fusarium graminearum. I risultati riportati in Figura 16 indicano che né il
microrganismo tal quale né il surnatante di coltura sono attivi contro il fungo produttore di micotossine. Al contrario, le batteriocine purificate e quindi concentrate mostrano elevata attività contro il suddetto fungo.
L’attività di queste due batteriocine contro il fungo Fusarium graminearum è di particolare interesse in quanto questo fungo è responsabile della produzione di micotossine. Il fatto che le batteriocine diluite nel surnatante di coltura non risultano attive, dimostra l’importanza di effettuare tappe di purificazione per poter concentrare queste sostanze. Come è stato osservato, infatti, una volta concentrate, le due batteriocine hanno forte attività antifungina. E’ stato così dimostrato, che, da una parte, lo spettro d’azione delle due sostanze proteiche copre anche un fungo, dall’altra, che le tappe di purificazione e di concentrazione permettono di amplificare la capacità antibatterica e antifungina.
BATT
SURN A51
A51
Fig. 16:
Batt: batteriocine purificate a seguito di cromatografia a fase inversa
Surn A51: surnatante di coltura di Enterococcus durans neutralizzato
A51: coltura pura di
3.4 ESPRESSIONE ETEROLOGA
3.4.1 Inserimento del plasmide modificato pGEM®-T Easy Vector nelle cellule di E. coli DH 5α
Il risultato dell’amplificazione delle sequenze che codificano per le batteriocine tramite PCR sono riportate in Figura 17.
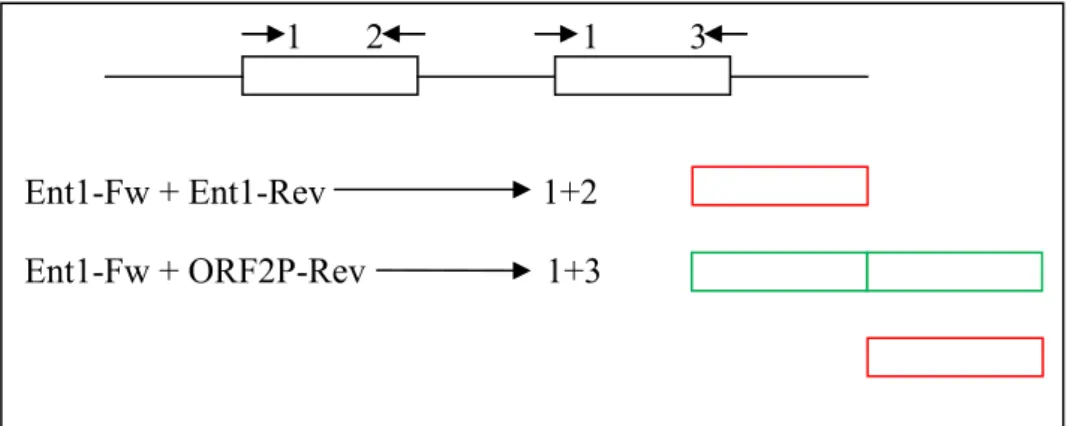
Le bande corrispondenti ai geni di interesse (135 bp e 132 bp1) sono state evidenziate con un cerchio rosso. Per il campione contenuto nella linea 2 (A5-11B), è stata osservata una seconda banda di taglia superiore (cerchio verde). Questa banda corrisponde all’amplificazione di un frammento contenente entrambi i geni delle batteriocine. Questo accade in quanto i geni sono contigui. La Figura 18 mostra l’azione dei primers.
Fig.17 : gel d’agarosio dopo PCR M- Marker 1 - A5-11A 2 - A5-11B 1 2 M 100 200
Fig. 18: siti d’azione dei primers usati per la PCR
A seguito della ligazione con il plasmide pGEM®-T Easy Vector e la trasformazione di cellule di Escherichia coli DH5α, sono state prelevate delle colonie bianche dalla capsula Petri2(Fig. 19) con lo scopo di estrarne il plasmide e verificare l’integrazione del gene della batteriocina .
Per meglio identificare il gene che codifica per la batteriocina è stata eseguita un’amplificazione tramite PCR. Il gel della Figura 20 riporta il risultato dell’amplificazione, grazie alla quale, per alcuni campioni (1.3, 1.4, 2.3 e 2.10, evidenziati con un cerchio rosso), è stato possibile amplificare un frammento con la taglia desiderata. Il plasmide contiene dunque il gene d’interesse. Anche altri
2
Vedere “Materiali e metodi – Trasformazione di cellule di Escherichia coli competeneti” 1 2 1 3 Ent1-Fw + Ent1-Rev 1+2 Ent1-Fw + ORF2P-Rev 1+3
. .
.
.
.
.
.
.
.
.
.
.
Fig. 19: capsula Petri con le colonie blu e bianche (nere nella foto)
frammenti, non interessanti dal punto di vista del lavoro svolto, sono stati ugualmente amplificati.
Fig. 20: gel d’agarosio dopo PCR per la verifica dell’inserimento del plasmide
Per avere la certezza dell’inserimento del gene codificante per la batteriocina nel plasmide, è stata realizzata un’estrazione dei plasmidi con il kit Mini Prep e i plasmidi così estratti sono stati inviati a sequenziare. In Figura 21 è riportato il gel d’agarosio realizzato per verificare l’estrazione dei plasmidi. Mentre i campioni 1.4, 2.3 e 2.10 hanno una banda uguale, il campione 1.3 presenta una banda diversa dagli altri.
100 200
1.3
Questa differenza è stata evidenziata con il sequenziamento, che ha mostrato (Fig. 22) come solo i campioni 1.4, 2.3 e 2.10 presentano la sequenza corretta. Per la sequenza nucleotidica clonata del campione 1.4 è stata rilevata una mutazione puntiforme (in figura evidenziata in giallo) nei confronti della sequenza di riferimento. Questa non modifica, però, la sequenza proteica.
1.3 1.4 2.3 2.10 Fig. 21: gel d’agarosio dopo
Fig. 22: confronto tra la sequenza di riferimento e le sequenze delle batteriocine
La costruzione del vettore di clonaggio pGEM®-T Easy Vector inserito in cellule di Escherichia coli DH 5α è stata necessaria (nonostante che né il vettore né il microrganismo permettano l’espressione) in quanto, inserendo direttamente il gene codificante per le due batteriocine nel vettore e nel microrganismo d’espressione, avrebbero potuto verificarsi dei problemi a livello di riconoscimento della sequenza e quindi di espressione.
3.4.2 Trasformazione del plasmide pET DP-LTP e inserimento in cellule di E. coli DH 5α
Dopo aver verificato la corretta costruzione dei plasmidi, è stato scelto di lavorare CAMPIONE 1.4 Enterocin ---ATGGGAGCAATCGCAAAATTAGT 23 TO1.4_M13R-IC CCGGCCGCCATGGCGGCCGCGGGAATTCGATTAAACCATGGGAGCAATCGCAAAATTAGT 180 T01.4_2_M13F CCGGCCGCCATGGCGGCCGCGGGAATTCGATTAAACCATGGGAGCAATCGCAAAATTAGT 97 *********************** Enterocin AGCAAAGTTTGGGTGGCCTATTGTTAAAAAGTATTACAAACAAATTATGCAGTTTATTGG 83 TO1.4_M13R-IC AGCAAAGTTTGGGTGGCCTATTGTTAAAAAATATTACAAACAAATTATGCAGTTTATTGG 240 T01.4_2_M13F AGCAAAGTTTGGGTGGCCTATTGTTAAAAAATATTACAAACAAATTATGCAGTTTATTGG 157 ****************************** ***************************** Enterocin AGAAGGATGGGCAATTAACAAAATCATTGAGTGGATTAAAAAACATATTTAA--- 135 TO1.4_M13R-IC AGAAGGATGGGCAATTAACAAAATCATTGAGTGGATTAAAAAACATATTTAAGCTTGTCA 300 T01.4_2_M13F AGAAGGATGGGCAATTAACAAAATCATTGAGTGGATTAAAAAACATATTTAAGCTTGTCA 217 **************************************************** CAMPIONI 2.3 et 2.10 TO2.3_M13R TGATTGTGACCATGGGAGCAATCGCAAAACTAGTGACAAAGTTTGGGTGGCCACTAATCA 159 TO2.10_M13R TGATTCTATCCATGGGAGCAATCGCAAAACTAGTGACAAAGTTTGGGTGGCCACTAATCA 157 ORF2 ---ATGGGAGCAATCGCAAAACTAGTGACAAAGTTTGGGTGGCCACTAATCA 49 T02.3_2M13FIC TGATTGTGACCATGGGAGCAATCGCAAAACTAGTGACAAAGTTTGGGTGGCCACTAATCA 239 T02.10_2M13FIC TGATTCTATCCATGGGAGCAATCGCAAAACTAGTGACAAAGTTTGGGTGGCCACTAATCA 240 ************************************************* TO2.3_M13R AAAAATTCTACAAACAAATCATGCAATTTATTGGACAAGGATGGACAATAGATCAAATTG 219 TO2.10_M13R AAAAATTCTACAAACAAATCATGCAATTTATTGGACAAGGATGGACAATAGATCAAATTG 217 ORF2 AAAAATTCTACAAACAAATCATGCAATTTATTGGACAAGGATGGACAATAGATCAAATTG 109 T02.3_2M13FIC AAAAATTCTACAAACAAATCATGCAATTTATTGGACAAGGATGGACAATAGATCAAATTG 299 T02.10_2M13FIC AAAAATTCTACAAACAAATCATGCAATTTATTGGACAAGGATGGACAATAGATCAAATTG 300 ************************************************************ TO2.3_M13R AAAAATGGCTAAAAAGACATTAAGCTTCATTAATCGAATTCCCGCGGCCGCCATGGCGGC 279 TO2.10_M13R AAAAATGGCTAAAAAGACATTAAGCTTGTAGAATCGAATTCCCGCGGCCGCCATGGCGGC 277 ORF2 AAAAATGGCTAAAAAGACATTAA--- 132 T02.3_2M13FIC AAAAATGGCTAAAAAGACATTAAGCTTCATTNNNNNAATTCCCGCGGCCGCCATGGCGGC 359 T02.10_2M13FIC AAAAATGGCTAAAAAGACATTAAGCTTGTAGNNNNNNATTCCCGCGGCCGCCATGGCGGC 360 ***********************
questi cloni batterici con l’ausilio di un kit mini Prep e questi sono stati tagliati con gli enzimi di restrizione Hind III e Nco I. La stessa cosa è stata effettuata per il vettore d’espressione pET-DP-LTP al fine di prepararlo all’inserimento dei frammenti di DNA contenuti nei campioni 1.4 e 2.3 (Fig. 23).
I frammenti di DNA separati sono stati estratti dal gel d’agarosio e, successivamente, è stata realizzata una ligazione tra i frammenti di DNA codificanti per le batteriocine e il plasmide pET-DP-LTP. Sono state, in seguito, trasformate cellule di Escherichia coli DH5α con il prodotto della ligazione. Nelle
100 200
1.4 1.4 1.4 1.4 M 2.3 2.3 2.3 2.3
pET pET pET pET pET pET Fig. 23: gel d’agarosio a seguito della digestione con Hind III e Nco I
Frammento corretto 100
capsule Petri contenenti il mezzo di coltura LB con ampicillina agarizzato al 2% sono cresciute solo le cellule trasformate contenenti l’inserto, che hanno perciò resistenza all’antibiotico.
Dopo aver prelevato delle colonie, averle fatte replicare e averne estratto il plasmide con il kit mini Prep, è stata eseguita una amplificazione tramite PCR (Figg. 24, 25) per verificare il corretto inserimento del gene d’interesse nel vettore d’espressione.
Fig. 24: gel d’agarosio dopo PCR. Il cerchio rosso indica i campioni utilizzati successivamente per la digestione.
Fig. 25: gel d’agarosio dopo PCR.
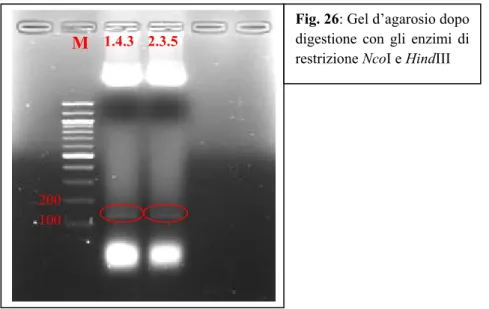
Partendo dal mini Prep realizzato su 100 ml di campione 1.4.3 e 2.3.5 è stata eseguita una digestione per mezzo degli enzimi di restrizione NcoI e HindIII al
M 2.3.1 2.3.2 2.3.3 2.3.4 2.3.5 1.4.1 1.4.2 1.4.3 1.4.4 1.4.5 100 200 100 200 1.4.15 1.4.12 1.4.13 1.4.14 1.4.10 1.4.11 1.4.9 1.4.7 1.4.8 1.4.6 M
fine di verificare il corretto inserimento del gene codificante per la batteriocina.(Fig.26).
3.4.3 Inserimento del plasmide modificato pET DP-LTP in cellule di E. coli BL 21
Il plasmide precedentemente costruito è stato infine inserito nel ceppo Escherichia
coli BL21 per l’espressione. I campioni cresciuti nelle capsule Petri contenenti il
mezzo di coltura LB con ampicillina agarizzato al 2% contengono il plasmide trasformato. Per verificare la corretta sequenza del costrutto, due campioni, contenenti uno il gene per l’espressione dell’enterocina I (1.4.3) e l’altro per la proteina ORF2 (2.3.5), sono stati inviati a sequenziare (Fig. 27).
M 1.4.3 2.3.5
Fig. 26: Gel d’agarosio dopo digestione con gli enzimi di restrizione NcoI e HindIII
100 200
Fig. 27: confronto tra le sequenze dei plasmidi e le sequenze delle batteriocine
L’analisi delle sequenze hanno permesso di verificare che i due plasmidi hanno legato correttamente i geni per l’espressione delle batteriocine.
3.4.4 Test d’espressione della proteina ricombinante
L’espressione delle batteriocine sotto forma di proteine di fusione è stata verificata mediante un test d’espressione. Dall’analisi del risultato dell’elettroforesi SDS-PAGE (Fig. 28), sono individuabili le due bande (cerchi rossi) corrispondenti alle proteine attese di massa molecolare di 22100 per l’enterocina I (campione 1.4.3) e di 22089 per ORF2P (campione 2.3.5)
ECHANTILLON 1.4.3 Ent1 ---ATGGGAGCAATCGCAAAATTAGTAGCAAAGTTTGGG 36 143-T7 GATCTGGGTACCGACGACGACCCCATGGGAGCAATCGCAAAATTAGTAGCAAAGTTTGGG 540 ************************************ Ent1 TGGCCTATTGTTAAAAAGTATTACAAACAAATTATGCAGTTTATTGGAGAAGGATGGGCA 96 143-T7 TGGCCTATTGTTAAAAAATATTACAAACAAATTATGCAGTTTATTGGAGAAGGATGGGCA 600 ***************** ****************************************** Ent1 ATTAACAAAATCATTGAGTGGATTAAAAAACATATTTAA--- 135 143-T7 ATTAACAAAATCATTGAGTGGATTAAAAAACATATTTAAGCTTGCGGCCGCACTCGAGCA 660 *************************************** ECHANTILLON 2.3.5 ORF2 ---ATGGGAGCAATC 12 235-T7 GAACGCCAGCACATGGACAGCCCAGATCTGGGTACCGACGACGACCCCATGGGAGCAATC 540 ************ ORF2 GCAAAACTAGTGACAAAGTTTGGGTGGCCACTAATCAAAAAATTCTACAAACAAATCATG 72 235-T7 GCAAAACTAGTGACAAAGTTTGGGTGGCCACTAATCAAAAAATTCTACAAACAAATCATG 600 ************************************************************ ORF2 CAATTTATTGGACAAGGATGGACAATAGATCAAATTGAAAAATGGCTAAAAAGACATTAA 132 235-T7 CAATTTATTGGACAAGGATGGACAATAGATCAAATTGAAAAATGGCTAAAAAGACATTAA 660 ************************************************************
Fig. 28: test d’espressione al tempo T33
Il gel elettroforetico ha dimostrato che, non solo i geni codificanti per le batteriocine sono stati inseriti correttamente nei ceppi Escherichia coli BL21, ma anche che queste due proteine sono espresse, permettendo così di poterle produrre separatamente ed utilizzarle per test futuri. E’ interessante notare che l’espressione delle proteine è rilevabile maggiormente dopo 3 ore dall’induzione con IPTG. Dopo 4 ore il livello diminuisce mentre è completamente assente dopo 1 e 2 ore.
3.4.5 Purificazione delle batteriocine
L’avvenuta purificazione delle batteriocine è stata verificata tramite un gel SDS – PAGE. Dal risultato riportato in Figura 29 è possibile rilevare come le bande delle due batteriocine (cerchio rosso) sono le uniche ad essere presenti sul gel. Questo ad indicare che la purificazione è avvenuta correttamente.
3
Vedere “Materiali e metodi – Test d’espressione della proteina ricombinante
M 1.4.3 2.3.5 pET BL21 1.4.3 IPTG 2.3.5 IPTG pET IPTG BL21 IPTG 66200 45000 31000 21500 14400 97400
Fig. 29: Gel SDS-PAGE per la verifica della purificazione
I primi due pozzetti rappresentano l’inizio del lavaggio con il tampone di eluizione mentre i pozzetti successivi al marker rappresentano il secondo lavaggio con il tampone di eluizione. Le batteriocine sono state rilasciate quasi completamente a seguito del secondo lavaggio.
La purificazione ha permesso di avere le due batteriocine isolate e ben separate, cosa che non era stata completamente possibile tramite HPLC (Batdorj B. et al, 2006). Questo permetterà, una volta separate in mezzo acido le batteriocine dalla tioredoxina, ulteriori analisi sull’attività antibatterica e antifungina delle due batteriocine. Infatti, potendo disporre di queste due proteine isolate e purificate, sarà più facile quantificare e qualificare queste due sostanze proteiche.
66200 45000 31000 21500 14400 97400 M 1.4.3 2.3.5 1.4.3 2.3.5