1. Introduzione
L’utilizzo di tecnologie di avanguardia per la rilevazione di specifici biomarkers presenti nel siero è attualmente importante per la conoscenza della fisiopatologia e quindi orientare in modo univoco la diagnosi precoce di molte malattie.
Le proteine del siero, infatti, sono un utile strumento di diagnosi e l’espressione modificata di alcune di esse può essere un segnale precoce di un’alterata fisiologia e quindi può essere correlata con una o più patologie [1].
L’alterazione del metabolismo delle proteine a basso peso molecolare (LMWP) e dei peptidi derivanti dalla frammentazione delle proteine più grandi è stata studiata in varie patologie utilizzando metodologie che permettono la loro identificazione come, per esempio, il RIA e l’ELISA. Questo tipo di approccio ha il vantaggio di essere altamente sensibile ma nel contempo presenta notevoli svantaggi: si può analizzare una proteina alla volta, ha costi elevati, richiede la disponibilità di anticorpi per le proteine in esame e , in special modo, permette di rilevare solo ciò che è noto non fornendo nuove informazioni.
Nello studio delle patologie renali, la frammentazione delle proteine ed i
peptidi così derivati sono stati utilizzati come biomarkers di tali anomalie
tramite analisi proteomica.
I principi fondamentali sui quali si basa l’analisi proteomica sono la separazione per punto isoelettrico e peso molecolare delle proteine e dei peptidi, che non hanno
particolari funzioni fisiologiche, attraverso le tecniche elettroforetiche quali la SDS-PAGE e la 2-DE.
Queste metodologie analitiche permettono di individuare simultaneamente più proteine e più peptidi presenti nel singolo campione e pertanto oltre al vantaggio metodologico della simultaneità prospettano la possibilità di nuove conoscenze andando a determinare la loro specificità. Questi possono diventare precisi biomarker per individuare varie patologie.
Gli esperimenti svolti in laboratorio [2] hanno avuto lo scopo di verificare se i pattern proteomici di pazienti sani (controlli) si differenziavano da quelli dei pazienti affetti da varie patologie renali. Le tecniche impiegate per tali esperimenti sono state l’SDS-PAGE e la 2-DE oltre a tecniche cromatografiche o l’utilizzo di strips con intervalli di intervalli ristretti di pH per la separazione selettiva delle proteine interessate; l’uso di queste ultime si è reso necessario per evidenziare le proteine del siero presenti in minor quantità rispetto alla grande predominanza dell’albumina.
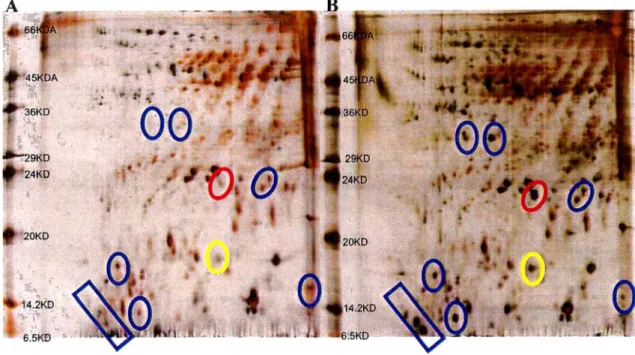
I risultati sperimentali hanno evidenziato che i pazienti nefropatici presentano, nel loro siero, una maggiore concentrazione sia di albumina che di proteine a basso peso molecolare quali, per esempio, la transtiterina (TTR) e la retinol binding protein (EBP).
molto evidentemente una maggior concentrazione delle proteine a basso peso molecolare nel campione B, che è quello relativo al paziente nefropatico.
Fig. 1- 2-DE delle proteine del siero nativo. Elettroforesi bidimensionale di un controllo (A) e di un paziente neuropatico (B), in quest’ultimo si osserva una sovraespressione di molte proteine rispetto al controllo. Sono state identificate due proteine: la transtiretina (in giallo ) e la retinol binding protein (in rosso) [2].
E’ da aggiungere, alle considerazioni sopra fatte, che i pazienti nefropatici hanno un escreto urinario nel quale la concentrazione di proteine di natura plasmatica e di frammenti da essi derivati è minore se non addirittura trascurabile rispetto a quella riferita ai pazienti sani.
2. Scopo della tesi
Nell’introduzione ho messo in evidenza i meccanismi che presiedono alla filtrazione renale delle proteine plasmatiche. In particolare è stato analizzato il ruolo di quelle a basso peso molecolare come possibili biomarkers delle malattie renali; la proteomica è il mezzo più potente d’indagine per le proteine di questo tipo. E’ stato messo bene in evidenza il ruolo del poro glomerulare e della relativa selettività della filtrazione delle macromolecole, andando a capire quale potessero essere i vari destini de tali proteine anche dopo eventuale processo di riassorbimento tubulare renale, per il controllo della concentrazione plasmatica di alcune proteine importanti, quali l’albumina. Il destino di tale proteina plasmatica è l’ argomento principale della tesi. Questa proteina, anche in dipendenza di varie patologie renali, viene filtrata, riassorbita, escreta ed è oggetto di proteolisi. Tale processo ha nelle cellule alveolari peritubulari durante il processo di riassorbimento, ad opera degli enzimi proteolitici che risiedono all’interno dei lisosomi cellulari. La frammentazione proteica procede nel plasma con i peptidi che vengono ridotti a singoli aminoacidi e immessi nel torrente sanguigno. Solamente circa il 10% dell’albumina viene eliminata con le urine, insieme agli altri prodotti del catabolismo giornaliero.
Nella parte della tesi che segue verranno discusse alcune ipotesi relative alla struttura del glomerulo, sia dal punto di vista della sua forma e grandezza sia dal quello elettrochimico. Verrà anche affrontato il problema relativo alle variazioni strutturali e di distribuzione di carica dell’albumina, correlate alle sue funzioni principali, tentando di contribuire alla spiegazione del fenomeno osservato.