U
CAPITOLO 3
MATERIALI E METODI
3.1 Generazione di una linea di fibroblasti felini
Per poter generare le linee di fibroblasti da utilizzare nel saggio, gli animali, anestetizzati e rasati, sono stati sottoposti ad una biopsia di un sottile strato di pelle sulla coscia tramite un biopsy punch del diametro di 3 mm. Per rimuovere eventuali batteri presenti, il frammento prelevato è stato lavato con terreno MEM (Minimun Essential Medium, Sigma Aldrich, St. Louis, USA) integrato con 200u/ml di penicillina (Eurobio, Francia) e 200µg/ml di streptomicina (Eurobio, Francia). A questo punto, mediante l’uso di bisturi, è stato eseguito un taglio a croce sulla biopsia per separare il derma dall’epidermide e quest’ultima è stata ulteriormente frazionata fino a generare circa 10 frammenti più piccoli. Questi sono stati trasferiti in PBS (NaCl 137mM, KCl 2,7mM, KHB 2B POB 4 B 1.5mM, NaB 2B HPOB 4 B
8.1mM) contenente tripsina all’1% e mantenuti in agitazione a 37°C in presenza di COB2B al 5% per 10 minuti per consentire all’enzima di agire. La soluzione contenente PBS, tripsina e i fibroblasti staccatisi dai frammenti è stata quindi raccolta e centrifugata a 1200 rpm per 10 minuti. Il pellet è stato risospeso in 5 ml di terreno Minimum
2mM di L-Glutammina, 100u/ml di penicillina, 100µg/ml di streptomicina e 1% di amminoacidi non essenziali (Sigma Aldrich) (MEM-α completo). Le cellule sono state quindi trasferite in un pozzetto di una piastra da sei. Il frammento di biopsia rimanente è stato sottoposto al lavaggio in PBS e tripsina altre 5 volte, fino ad ottenere 6 pozzetti contenenti fibroblasti. La crescita delle cellule è avvenuta in incubatore a 37°C e 5% di COB2B ed è stata controllata per circa 10 giorni, al termine dei quali piccole aree di fibroblasti sono risultate visibili. Le cellule hanno raggiunto la confluenza dopo circa 20 giorni (Figura 3.1).
Figura 3.1: I fibroblasti hanno raggiunto la confluenza dopo circa 20 giorni in coltura
Al raggiungimento della confluenza, il terreno è stato rimosso e le cellule, dopo essere state lavate con PBS, sono state incubate con tripsina per circa 2 minuti a 37°C; al termine dell’incubazione è stato aggiunto del terreno fresco per inattivare l’enzima. I fibroblasti sono stati raccolti e centrifugati a 1400 rpm per 6 minuti e il pellet è stato risospeso in terreno fresco. Le cellule sono state quindi seminate, in opportune diluizioni, in una nuova fiasca o nei pozzetti da trasfezione.
3.2 Trasformazione dei fibroblasti primari
Al fine di generare linee stabili e vitali in coltura per lunghi periodi di tempo i fibroblasti primari sono stati trasformati mediante trasfezione con il plasmide pACTSV2 contenente il genoma del simian virus 40 (SV40) leggermente modificato (gentile dono del Dr. Tognon, Università di Ferrara).
Ventiquattro ore prima di effettuare la trasfezione, i fibroblasti sono stati staccati come precedentemente descritto e contati; 8x10P
5
P
cellule sono state seminate in piastre da sei pozzetti contenenti MEM-α completo. Un’ora circa prima della trasfezione il terreno è stato sostituito con 4,5ml di terreno fresco.
Il protocollo usato per la trasfezione è quello del calcio fosfato. 3µg di plasmide pACTSV2 sono stati precipitati mediante aggiunta di 1/10 di volume di sodio acetato 3M pH 5,2 e 2 volumi di etanolo assoluto freddo. I campioni sono stati lasciati a -20°C overnight ed il giorno dopo, a precipitazione avvenuta, sono stati centrifugati a 12000 rpm per 30 minuti a 4°C. Il surnatante è stato quindi eliminato ed il pellet è stato lavato con 200µl di etanolo al 70%; i campioni sono stati quindi centrifugati a 12000 rpm per 10 minuti a 4°C. Il pellet ottenuto è stato separato dal surnatante ed asciugato.
I campioni precipitati sono stati risospesi in 225µl di buffer TE 0.1x a pH 8.0 (1mM TRIS-HCl, 0.1 mM EDTA). Quindi sono stati aggiunti 25µl di CaClB2B 2.5M e l’intera miscela è stata aggiunta a gocce all’HEPES 2x a pH 7 (280 mM NaCl, 10 mM KCl, 1.5 mM NaB2BPOB4B x 2HB2BO, 12 mM destrosio, 50 mM Hepes). I campioni sono stati incubati 20 minuti a temperatura ambiente e quindi la soluzione è stata aggiunta goccia a goccia in maniera uniforme sul pozzetto. La piastra è stata incubata per 6-8 ore a 37°C, dopodiché il terreno è stato rimosso e i pozzetti sono stati lavati con PBS per eliminare il DNA non incorporato dalle cellule; infine sono stati aggiunti 3 ml di terreno fresco. Le
cellule sono state mantenute in coltura per circa 3 settimane e monitorate per la comparsa di foci di trasformazione.
3.3 Produzione del vettore FIV e trasduzione dei
fibroblasti
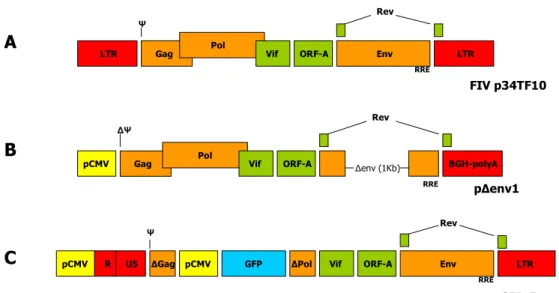
Il vettore utilizzato nelle trasduzioni è un vettore lentivirale FIV-derivato esprimente Env e GFP precedentemente prodotto e caratterizzato nel nostro laboratorio (Figura 3.2 C). Esso possiede la LTR al 5’ modificata, in quanto la regione U3 è sostituita dal promotore del cytomegalovirus (CMV); esso presenta inoltre una delezione nei geni gag e pol per favorire il clonaggio di una cassetta di espressione contenente il gene reporter GFP posto sotto il controllo di un secondo promotore CMV. Il vettore contiene infine il segnale di incapsidamento (Ψ), i geni vif, ORF-A, env, rev , la sequenza RRE e l’LTR al 3’ nella loro forma wild-type.
Il vettore viene prodotto secondo la strategia split component system precedentemente descritta mediante trasfezione sulla linea cellulare 293T, fibroblasti renali embrionali umani trasformati con l’antigene T dell’SV40. Tale linea viene mantenuta a 37°C in presenza di COB2B al 5% in terreno D-MEM (Dulbecco’s Modified Eagle’s Medium, Sigma Aldrich, S. Louis, USA) addizionato con il 10% (vol/vol) di FBS inattivato al calore, 2mM di L-glutammina, 1000U/ml di penicillina, 100g/ml di streptomicina, 1% di amminoacidi non essenziali (DMEM completo). Per l’espansione si procede nello stesso modo dei fibroblasti.
Per consentire una corretta produzione delle particelle virali, oltre che con il vettore, le cellule sono state trasfettate contemporaneamente con il costrutto di packaging pΔenv-1, il quale fornisce le proteine enzimatiche Gag e Pol necessarie per la formazione di particelle complete e funzionali (Figura 3.2 B). Infine, il vettore è stato pseudotipizzato fornendo come envelope la glicoproteina G del virus della stomatite vescicolare (VSV-G).
vGFP-Env
Δenv (1Kb)
pCMV Gag Pol Vif ORF-A BGH-polyA Rev
ΔΨ
RRE pΔenv1
Rev
pCMV R U5 ΔGag pCMV GFP ΔPol Vif ORF-A Env LTR
RRE
Ψ
LTR LTR Gag Pol Vif ORF-A Env
Rev
RRE
Ψ
FIV p34TF10
Figura 3.2:A: Genoma wild-type di FIV; B: costrutto di packaging ; C: vettore utilizzato per la trasfezione e la traduzione
Le particelle vettore da utilizzare per la trasduzione sono state prodotte mediante cotrasfezione di 10µg di vettore vGFP-Env, 8µg del costrutto pΔenv1 e 1µg del costrutto veicolante VSV-G. Il protocollo utilizzato è quello decritto per la trasformazione dei fibroblasti primari.
A
B
Dopo circa 48 ore dalla trasfezione, il surnatante delle 293T trasfettate è stato raccolto, chiarificato mediante centrifugazione a 1700 rpm per 10 minuti e filtrato con filtri del diametro di 0.45µm; esso è stato quindi aliquotato e congelato a -80°C fino al momento dell’uso.
Il giorno precedente la trasduzione, circa 3x10P
4
P
fibroblasti/pozzetto sono stati seminati in una piastra da 6 pozzetti, contenenti ciascuno 3 ml di terreno MEM-α completo. Il giorno successivo un’aliquota contenente 1 ml di particelle vettore è stata aggiunta ad ogni pozzetto. Dopo circa 6 ore, il terreno è stato rimosso e le cellule sono state lavate con PBS 1x, quindi è stato aggiunto terreno fresco.
3.4 Propagazione dei plasmidi
Tutti i costrutti utilizzati per le trasfezioni e le trasduzioni sono stati propagati in cellule batteriche del ceppo JM109 di E. Coli. Le cellule, rese competenti con il protocollo del calcio cloruro, sono state trasformate mediante aggiunta di 20ng di DNA plasmidico. Dopo una incubazione in ghiaccio di 30 minuti le cellule sono state sottoposte a shock termico a 42°C per un minuto seguito da un rapido trasferimento in ghiaccio per 3 minuti. Alle cellule è stato quindi aggiunto 1 ml di terreno SOC (2% bacto-triptone, 0.5% estratto di lievito, NaCl 10mM, KCl 25mM, MgClB2B 50mM e glucosio 20mM) e sono state lasciate crescere 1 ora a 37°C in agitazione. Al termine della crescita le cellule sono state raccolte, piastrate su terreno LB-agar (1% bacto-triptone, 0.5% estratto di lievito, 1% NaCl, 1.5% agar) addizionato di ampicillina 50µg/ml e lasciate crescere overnight a 37°C.
3.5 Estrazione dei plasmidi
Dopo la crescita overnight le singole colonie sono state inoculate in terreno liquido LB (1% triptone, 0.5% estratto di lievito, 1% NaCl) più ampicillina 50 µg/ml e cresciute in agitazione a 37°C per 16 ore. Quindi viene prelevato 1.5ml di crescita, centrifugato a 12000 rpm per 5 minuti; dal pellet viene estratto il DNA mediante lisi alcalina. Il pellet viene risospeso in 100µl di soluzione I (50mM glucosio, 25mM Tris a pH-8, 10mM EDTA a pH-8) a cui vengono aggiunti 200µl di soluzione II di lisi (0.2N NaOH, 1% SDS) e 150µl di soluzione III di neutralizzazione (3M potassio acetato e 5M acido acetico glaciale). Il lisato batterico viene quindi centrifugato per 5 minuti a 12000 rpm a 4°C ed al surnatante, contenente il DNA plasmidico, viene aggiunto un ugual volume di una miscela contenente fenolo, cloroformio e alcool isoanilico 25:24:1. In seguito alla centrifugazione a 4°C a 12000 rpm per 5 minuti si separano la fase contenente fenolo-cloroformio e proteine da quella contenente Il DNA plasmidico; quest’ultimo viene raccolto e precipitato mediante l’aggiunta di 0.7 volumi di isopropanolo e successiva centrifugazione a 12000 rpm per 20 minuti a 4°C. Il pellet ottenuto viene lavato con 200µl di etanolo al 70%, asciugato e risospeso in 20µl di HB2BO contenente 20µg/µl di RNAsi A (Sigma Aldrich); l’enzima viene lasciato agire per 30 minuti a 37°C, quindi 1µl della soluzione contenente il DNA plasmidico viene controllato mediante corsa elettroforetica su gel di agarosio all’1%.
Per ottenere preparazioni di DNA plasmidico su più larga scala da utilizzare per la trasfezione è stato utilizzato il kit di estrazione Plasmid Midi Kit (Qiagen, Hilden, Germany). Dopo una crescita overnight in 50 ml di terreno LB più ampicillina 50 µg/ml le cellule sono state centrifugate a 3000 rpm per 30 minuti a 4°C; il pellet è stato quindi separato dal surnatante. L’estrazione del
estratto è risospeso in acqua e quantificato tramite lettura spettrofotometrica; una aliquota è stata corsa su gel di agarosio all’1% per verificarne l’integrità.
Per verificare l’assenza di mutazioni, i frammenti estratti sono stati sequenziati e analizzati al sequenziatore automatico mediante reazione basata sul metodo di terminazione a catena di Sanger.
3.6 Lettura
allo
spettrofotometro
Per valutare la concentrazione ed il grado di purezza del DNA estratto è stata eseguita una lettura allo spettrofotometro UV-VIS (Beckman D.U. 640, Fullerton, CA, USA). La densità ottica (O.D.) è stata misurata alla lunghezza d’onda di 260 nm, alla quale gli acidi nucleici hanno il massimo dell’assorbimento. Per valutare la purezza è stato considerato il rapporto tra O.D.B260 Be O.D.B280B (lunghezza d’onda a cui assorbono alcuni amminoacidi); il DNA è considerato sufficientemente puro quando questo rapporto assume valori intorno a 1.8. La concentrazione del DNA viene calcolata considerando che il valore di un O.D.B260B ottenuto corrisponde ad una concentrazione di 50ng/µl; perciò la formula completa per il calcolo della concentrazione in ng/µl è: O.D.B260B x 50 x fattore di diluizione.
3.7 Elettroforesi su gel di agarosio
Per verificare l’integrità del DNA estratto, questo è stato corso su gel di agarosio. L’agar è stato sciolto nel buffer TAE 1x (242 g Tris base, 57.1 ml di EDTA 0.5M pH-8) a cui è stato aggiunto l’1% di agarosio in polvere; infine è stato aggiunto il bromuro di etidio (50ng/ml) e la miscela è stata fatta solidificare in una vasca elettroforetica. I campioni di DNA sono stati mescolati in rapporto 10:1 con loading buffer 10x (0.25% blu di bromofenolo, 0.25% xilene cianolo, 30% glicerolo in HB2BO); i campioni sono stati quindi caricati sul gel e fatti correre applicando una differenza di potenziale di 90-120 V. Le bande presenti sul gel sono state visualizzate mediante transilluminatore ad UV (Ultra Violet Products).
3.8 Valutazione dell’efficienza di trasduzione
L’efficienza di trasduzione è stata valutata sia mediante analisi al FACS, per valutare l’espressione di GFP, sia tramite western blot, per valutare l’espressione di Env. Per determinar la stabilità della trasduzione, l’analisi è stata ripetuta dopo vari passaggi in coltura ed in seguito a scongelamento.
3.8.1 Valutazione dell’espressione di GFP
Per poter effettuare l’analisi al citofluorimetro (FACScan, Becton Dickinson), i fibroblasti trasdotti sono stati lavati, tripsinizzati e centrifugati a 1400 rpm per 6 minuti. Il pellet è stato risospeso in FACS Buffer (PBS 1x, 0.1%
BSA, 0.2% NaNB3B), nuovamente centrifugato e fissato con FACS Fix (FACS Buffer, 1% paraformaldeide).
La trasduzione è stata valutata confrontando lo spostamento di cellule GFP-positive verso valori di fluorescenza più elevati rispetto a cellule di controllo (non trattate o mock-trasdotte).
3.8.2 Valutazione dell’espressione di Env
Per valutare la capacità dei fibroblasti trasdotti di esprimere Env, essi sono stati analizzati tramite western blot. Circa 10P
5
P
cellule sono state staccate e lavate con PBS 1x e quindi centrifugate a 1400 rpm per 6 minuti. Il pellet è stato risospeso in 20µl di buffer di lisi (Tris 10mM, EDTA 2mM, NaCl 0.15mM, Nonidet 0.5%) a cui sono stati aggiunti 20µl di colorante Sample buffer 2x (Tris-HCl 0.5M pH 6.8, glicerolo 10%, SDS 10%, β-mercaptoetanolo, blu di bromofenolo 0.05%). I campioni sono stati bolliti per 5 minuti e caricati sul gel. La parte superiore del gel (stacking gel) contiene 10% di mix acrilammide/bisacrilammide (Acrilamide/Bis Solution, 29:1 Bio-Rad Laboratories, Hercules, CA, USA); per preparare 10ml di gel sono stati utilizzati, oltre all’acrilammide, 2.5ml Tris-HCl 0.5 mM pH 6.8, 0.1ml di SDS al 10%, 0.1ml di APS al 10%, 4µl di TEMED (Sigma Aldrich). La parte inferiore (resolving gel) contiene invece, oltre al 30% di mix acrilammide/bisacrilammide (Bio-Rad Laboratories), 0.67ml di Tris-HCl 0.5mM pH 8.8, 40µl di SDS al 10%, 40µl di APS al 10%, 4µl di TEMED. La corsa elettroforetica è avvenuta in running buffer 1x (Tris base 25mM, 0.1% SDS, 1.44% glicina, pH 9) per circa 1 ora e 30 minuti applicando una differenza di potenziale di circa 90V.
Al termine della corsa il gel è stato sistemato su una membrana di nitrocellulosa (Amersham Pharmacia Bioscience) in grado di legare saldamente le proteine. Il gel e la membrana sono stati racchiusi in un “sandwich”, costituito da due fogli di carta da filtro, impregnati di tampone di trasferimento
(0.3% Tris base, 1.44% glicina, 20% metanolo). Il trasferimento è stato effettuato ad una temperatura di 4ºC per 1h e 30 minuti ad un voltaggio di 100V, dopodiché la membrana è stata incubata per 1h con PBS Tween-skim milk al 3% al fine di bloccare possibili siti di legame aspecifici per l’anticorpo. La membrana è stata quindi posta in agitazione overnight in presenza dell’anticorpo primario monoclonale αenv-71.2 diluito 1:500 in PBS Tween-skim milk all’1%. Dopo alcuni lavaggi in PBS-Tween è stato aggiunto l’anticorpo secondario α–mouse perossidato (BioRad Laboratories) diluito 1:1000 in skim milk allo 0.5%. La rivelazione è stata effettuata dopo circa 1 ora aggiungendo un substrato contenente diaminobenzidina (DAB) 40mg/ml, Tris-HCl 100mM pH 7.5, perossido di idrogeno, NiClB2B 80mg/ml.
3.9 Animali
utilizzati
Gli animali utilizzati per l’allestimento del saggio sono stati quattro, BR, BS, CG e BO. I primi tre gatti sono infetti dal 2001 con il ceppo Petaluma di FIV, per cui si trovano nella fase cronica dell’infezione. Essi sono stati sottoposti a 3 inoculi di DNA veicolante il gene env di FIV. La loro risposta CTL è stata valutata a 2 e 18 settimane dopo il terzo inoculo. BO, invece, è un gatto sano ed è stato utilizzato come controllo negativo.
3.10 Separazione dei PBMC da sangue intero
Agli animali anestetizzati sono stati prelevati 15 ml di sangue in presenza di 1/10 di volume di sodio citrato 3.8% per impedirne la coagulazione. Il sangue è stato quindi stratificato in una provetta contenente un ugual volume di Histopaque (Sigma Aldrich) a temperatura ambiente e centrifugato per 30 minuti a 1750 rpm senza freno. Questo consente la separazione del sangue in tre fasi: sul fondo si depositano i globuli rossi e le piastrine, al centro si trova l’anello di PBMC e nella parte superiore il plasma. I PBMC sono stati quindi raccolti e sono stati effettuati 2 lavaggi con circa 50 ml di soluzione fisiologica (NaCl 0.9%). Al termine dei lavaggi le cellule sono state risospese in 5 ml di terreno RPMI 1640 (Sigma Aldrich) addizionato con 10% di FBS, 2mM di L-glutammina, 1% di amminoacidi non essenziali, 1000U/ml di penicillina e 100µg/ml di streptomicina. I PBMC sono stati contati in turkey e tripan blu in rapporto 1:10 v/v mediante la camera di Burker per valutarne la quantità e la vitalità.
3.11 Saggio CTL
Il giorno precedente al saggio i fibroblasti trasdotti e non trasdotti di ciascun gatto sono stati tripsinizzati e contati come descritto precedentemente; una parte è stata seminata in una piastra da 48 pozzetti ad una densità di 10P
4
P
cellule per pozzetto, mentre le restanti cellule sono state ripropagate in MEM-α completo.
U
RISTIMOLAZIONE CON FIBROBLASTIU: il giorno di inizio del saggio i PBMC appena prelevati dagli animali sono stati aggiunti in rapporto 100:1 ai pozzetti contenenti i fibroblasti autologhi, sia trasdotti che non trasdotti. Le piastre sono state quindi incubate a 37°C per 5 giorni.
U
RISTIMOLAZIONE CON PEPTIDIU: i peptidi utilizzati per la ristimolazione coprono l’intera sequenza codificante di Env, hanno una lunghezza di 15 amminoacidi e si sovrappongono l’uno con l’altro di 11. Essi sono stati raccolti in un pool in modo tale che la concentrazione finale di ciascuno sia di 30µg/ml. Il giorno di inizio del saggio sono stati seminati circa 5x10P
6
P
PBMC prelevati dagli animali e sono stati incubati per 5 giorni con il pool di peptidi, la cui concentrazione finale è risultata 5µg/ml. Come controllo un ugual numero di linfociti è stato incubato con lo stesso volume di PBS.
Il quinto giorno i fibroblasti trasdotti rimasti in coltura sono stati staccati e seminati in triplicato in una nuova piastra da 48 pozzetti, ancora con una densità di 10P
4
P
cellule a pozzetto.
Il giorno successivo i PBMC ristimolati sui fibroblasti o con i peptidi sono stati raccolti e centrifugati a 1400 rpm per 5 minuti. Il pellet è stato risospeso in terreno RPMI 1640 completo e le cellule sono state contate come precedentemente descritto. I PBMC sono stati quindi seminati sui fibroblasti autologhi freschi in rapporto 10:1 o 100:1. Per ciascun gatto sono state effettuate varie prove, che saranno discusse in dettaglio nella sezione “Risultati”; come controllo sono stati utilizzati fibroblasti trasdotti non trattati.
Dopo una incubazione di circa 16 ore a 37°C i PBMC sono stati eliminati ed i fibroblasti sottostanti sono stati lavati con PBS per rimuovere completamente i linfociti e quindi sono stati raccolti e fissati come precedentemente descritto .
3.12 Valutazione della lisi cellulare
La valutazione della lisi cellulare è stata effettuata tramite analisi al FACS.
Per calibrare inizialmente lo strumento è stato utilizzato un campione di fibroblasti non trasdotti. Le cellule sono state acquisite utilizzando come parametri le loro dimensioni e la loro granulosità; questo ha consentito di identificare la popolazione dei fibroblasti vitali e di distinguerla da cellule morte ed eventuali detriti cellulari. Una volta identificati, i fibroblasti vitali sono stati racchiusi in un gate, detto G1, e per ogni successiva analisi sono state considerate soltanto le cellule al suo interno.
Successivamente le cellule sono state analizzate tenendo conto della fluorescenza emessa alla lunghezza d’onda di 508 nm, che corrisponde a quella della GFP, ed è stato effettuato un secondo gate, detto R1, il quale racchiude quindi fibroblasti vitali non fluorescenti.
Come controllo positivo di fluorescenza è stato utilizzato il campione contenente fibroblasti trasdotti ma non incubati con i PBMC. Il numero di cellule trasdotte è stato valutato considerando la percentuale di fibroblasti, racchiusi in G1, spostati verso valori più elevati di fluorescenza rispetto a quelli contenuti in R1.
Durante l’analisi dei restanti campioni è stata valutata la diminuzione di fluorescenza rispetto al controllo positivo.