1. VIRUS TORQUETENO
1.1 CENNI STORICI
Gli ultimi decenni sono stati testimoni di un grande sviluppo compiuti nel campo delle tecniche di indagine molecolare, progresso che ha condotto alla graduale identificazione di nuovi agenti virali nell’uomo. Particolare attenzione è stata rivolta alla caratterizzazione eziologica delle epatopatie fino ad allora di origine sconosciuta, con la conseguente identificazione della maggior parte degli agenti virali coinvolti nell’epatite.
Tuttavia, rimanevano ancora da associare ad uno specifico agente eziologico alcune forme di epatiti acute o croniche, non attribuibili ai virus già identificati.
In questa direzione sono stati quindi rivolti molti studi fino alla scoperta, nel 1997, di una nuova sequenza appartenente ad un virus di origine sconosciuta, nel siero di un paziente affetto da epatite criptogenetica (Nishizawa et al., 1997).
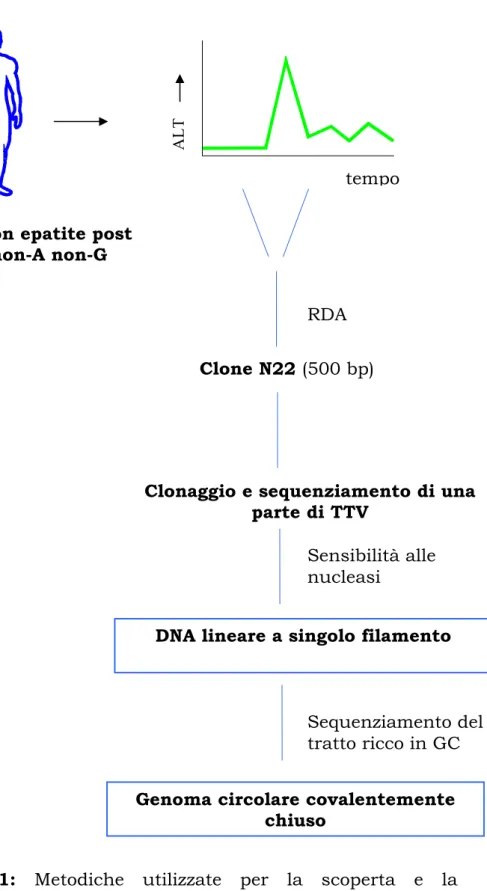
Il siero di tale paziente, che presentava un picco transitorio nei livelli di alanina aminotransferasi (ALT), fu analizzato tramite representational difference analysis (RDA); questa metodica, ideata da Lisitsyn nel 1993, permette di evidenziare sequenze di DNA selettivamente espresse nei tessuti patologici
quando paragonate con sequenze derivate dai tessuti sani degli stessi soggetti (Lisitsyn et al., 1993).
In particolare, nel paziente oggetto di studio, Nishizawa e collaboratori confrontarono il DNA plasmatico, estratto al momento del picco di ALT, con il DNA del medesimo individuo quando i livelli di ALT risultavano normali (figura I.1). Analizzando le sequenze esclusive del picco e paragonandole con quelle disponibili in banca dati, venne isolato un clone di 500 nucleotidi che non mostrava omologia con nessuna delle sequenze già note: il clone, denominato N22, fu dimostrato appartenere ad un nuovo virus a DNA a singolo filamento lineare (Nishizawa et al., 1997).
La scoperta destò subito un grande entusiasmo per la presunta identificazione dell’agente responsabile di alcune epatiti criptogenetiche e dalle iniziali del paziente il virus fu denominato TT (TTV).
Il successivo clonaggio e sequenziamento da parte di Okamoto e collaboratori portò alla caratterizzazione di un genoma virale apparentemente completo della lunghezza di 3.739 kilobasi (kb), denominato TA278 che, in considerazione delle piccole dimensioni e di altre caratteristiche del virione, fu ritenuto appartenere alla famiglia Parvoviridae (Okamoto et al., 1998).
Nel 1999 però Miyata e collaboratori, approfondendo gli studi sul prototipo del virus TA278, non riscontrarono le sequenze palindromiche terminali tipiche dei parvovirus, ma anzi identificarono, tramite PCR inversa, una regione di circa 100 nucleotidi ricca in guanine e citosine (GC), dimostrando così che TTV non aveva un genoma lineare ma circolare e che le due estremità erano unite proprio da questa regione.
I risultati ottenuti, insieme all’elevata affinità strutturale che TTV mostrava avere con il virus dell’anemia dei polli (CAV), portarono a suggerire l’inserimento del virus nella famiglia Circoviridae, come primo rappresentante dei circovirus umani (Miyata et al., 1999).
Gli studi successivi hanno permesso l’identificazione di numerose specie virali con caratteristiche strutturali simili a TTV, ma da questo geneticamente molto divergenti.
Fra le specie identificate vi sono gli isolati del gruppo PMV (Hallett et al., 2000), del gruppo SANBAN, che nella lingua giapponese significa il terzo (Hijikata et al., 1999) e i virus YONBAN (il quarto in giapponese), che mostrano meno del 50% di omologia con il prototipo TTV e un genoma di dimensioni leggermente diverse (Takahashi et al., 2000a). Emerse quindi fin dai primi studi che TTV comprendeva molte specie eterogenee dal punto di vista genetico.
Fu proprio analizzando l’eterogeneità genetica di TTV che venne identificato un altro gruppo di virus con caratteristiche intermedie tra TTV e CAV (Takahashi et al., 2000b); per questo gruppo fu coniato il termine TLMV (TTV-Like-Mini-Virus), a sottolineare le dimensioni più piccole del genoma, che risultava di 2,9 Kb, contro le 3,8 Kb tipiche di TTV.
Tutti gli studi successivi alla scoperta di TTV sono stati rivolti alla possibile correlazione del virus con patologie epatiche umane; sebbene i primi dati sembravano confermare questa ipotesi, ricerche più approfondite hanno escluso un coinvolgimento diretto di TTV nello sviluppo di tali malattie. Il possibile ruolo eziologico di TTV è stato anche indagato in altre patologie umane, tuttavia non è mai stata dimostrata una chiara associazione del virus con una manifestazione clinica.
Questi dati, insieme all’elevata prevalenza del virus nella popolazione generale, hanno anche suggerito l’ipotesi che TTV fosse un semplice commensale apatogeno dell’uomo (Griffiths, 1999; Mushahwar, 2000).
Tuttavia, nell’attesa di definire il reale ruolo patogenetico di TTV, è più corretto considerare il virus come appartenente al gruppo di virus definiti “orfani”, cioè ancora privi di una specifica associazione con una patologia.
Paziente TT con epatite post trasfusionale non-A non-G
ALT
tempo
RDA
Sensibilità alle nucleasi
Clonaggio e sequenziamento di una parte di TTV
DNA lineare a singolo filamento
Sequenziamento del tratto ricco in GC Clone N22 (500 bp)
Genoma circolare covalentemente chiuso
Figura I.1: Metodiche utilizzate per la scoperta e la caratterizzazione del virus (tratta e modificata da Bendinelli et al., 2001).
1.2 TASSONOMIA E NOMENCLATURA
Nel corso degli anni le conoscenze sulle caratteristiche strutturali e genomiche di TTV sono rapidamente aumentate e numerosi sono stati i tentativi di classificare il virus all’interno di una specifica famiglia.
Le prime osservazioni relative alle dimensioni del virione, l’assenza di involucro lipidico e la presenza di una molecola di DNA a singolo filamento, indussero gli studiosi a classificare TTV nella famiglia Parvoviridae (Okamoto et al., 1998).
Gli studi condotti da Miyata e collaboratori nel 1999 portarono però a stabilire la natura circolare del genoma e a proporre quindi l’inserimento di TTV nella famiglia Circoviridae, come unico rappresentante dei circovirus umani (Hijikata et al., 1999). Col progredire delle scoperte, infatti, TTV mostrava sempre più forti somiglianze strutturali col virus dell’anemia del pollo (CAV), un membro di questa famiglia, che comprendeva fino a quel momento tutti patogeni degli animali, tra cui anche il circovirus porcino (PCV) e il virus della malattia del becco e delle penne dei pappagalli (BFDV).
I virus appartenenti a questa famiglia sono caratterizzati da un genoma circolare a singolo filamento di DNA a polarità negativa, possiedono un capside icosaedrico probabilmente
costituito da un’unica proteina strutturale e sono privi di un rivestimento lipidico esterno (Todd et al., 1990).
Sebbene le somiglianze tra TTV e i circovirus, in particolare CAV, siano elevate, studi successivi hanno messo in luce molte differenze soprattutto a livello nucleotidico ed è stata quindi abbandonata la classificazione di TTV come membro della famiglia Circoviridae. Sia CAV che TTV sono caratterizzati da un genoma circolare a singolo filamento a polarità negativa, con dimensioni rispettivamente di circa 3200 e 3800 nucleotidi. Entrambi presentano una regione genomica codificante, (open reading frame, ORF), costituita da segmenti parzialmente sovrapposti (Iwata et al., 1998) ed una regione non tradotta (untraslated region, UTR) ricca in GC con notevole omologia tra i due virus (Miyata et al., 1999). Tuttavia, molte caratteristiche rimangono peculiari di TTV, tanto che è stata successivamente proposta anche la creazione di una nuova famiglia con TTV come unico rappresentante (Circinoviridae; Mushahwar et al. 1999) oppure la classificazione di TTV nella famiglia Paracircoviridae, insieme a CAV e TLMV (Takahashi et al., 2000b; Okamoto et Mayumi, 2001).
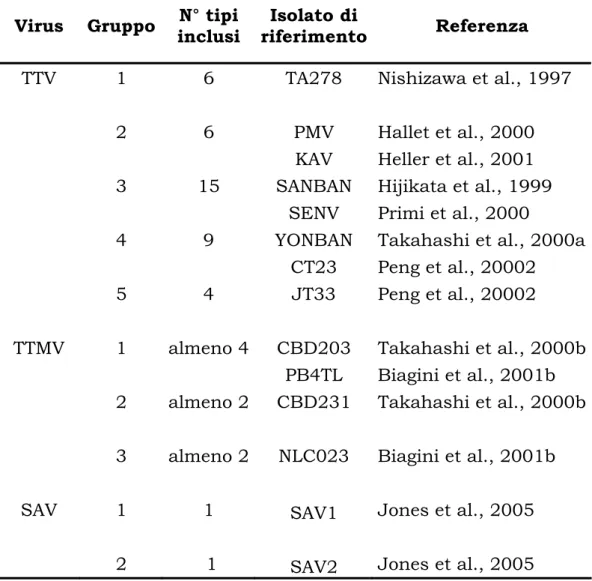
Recentemente è stato istituito dal Comitato Internazionale per la Tassonomia dei Virus (CITV) un gruppo di studio con il compito di stabilire la migliore classificazione per TTV. Nell’arduo tentativo di affidare una famiglia a TTV, il CITV ha quindi proposto di inserire il virus in un nuovo genere detto Anellovirus (dal latino “anello”), del quale farebbero parte anche TLMV e un altro gruppo di virus di più recente identificazione, denominato SAV (Small Anellovirus) (Tab. I.1); quest’ultimo gruppo di virus comprende due isolati, denominati SAV-1 e SAV-2, identificate da Jones e collaboratori nel plasma di soggetti negativi per HIV, ma con una sindrome da infezione virale acuta ad eziologia sconosciuta. Il sequenziamento degli amplificati ottenuti e l’analisi filogenetica indicavano che SAV-1 e SAV-2 mostravano una certa omologia con le sequenze di TTV e TLMV depositate in banca dati, anche se costituivano un gruppo indipendente. La differente struttura e lunghezza del genoma e la maggiore distanza genetica dagli altri anellovirus hanno portato diversi autori a proporre l’inserimento di questi virus in un terzo gruppo di anellovirus (Jones et al., 2005). Considerando le caratteristiche del genoma di TTV, il CITV ha anche proposto di cambiare il significato del vecchio acronimo: dall’inglese Transfusion Transmitted Virus al latino Torque
Teno Virus (virus a collana sottile, da torques, collana e tenuis, sottile). Per TLMV è stato invece proposto il nome di Torque Teno Mini Virus (TTMV) (Hino, 2002; Todd et al., 2002).
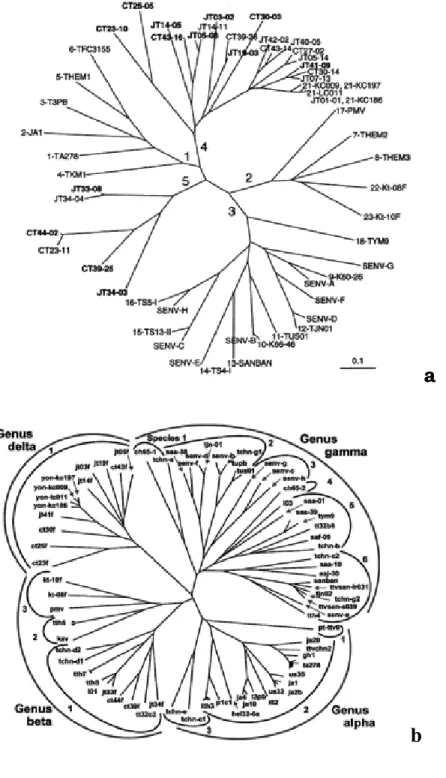
Particolarmente complessa è stata anche la classificazione degli isolati di TTV all’interno del genere anellovirus, a causa della vasta eterogeneità genetica che il virus presentava già in seguito alle analisi iniziali. I primi studi sulle relazioni filogenetiche degli isolati di TTV sono stati condotti amplificando e sequenziando brevi regioni del genoma virale; in particolare è stata utilizzata la regione N22 dell’ORF1, che ha inizialmente permesso di suddividere i diversi isolati del virus in 23 genotipi compresi in quattro maggiori raggruppamenti filogenetici (Okamoto et al., 1999; Nishizawa et al., 1999; Biagini et al., 1999; Okamoto et al., 2000c; Ukita et al., 2000; Takahashi et al., 2000a; Tanaka et al., 2000b). Questa regione si è successivamente rivelata inadatta per identificare l’intera varietà di specie di TTV, perché capace di amplificare solo determinati genotipi e quindi non idonea per una genotipizzazione “universale”.
Gli studi successivi sono stati quindi condotti sull’intero genoma e sulla regione UTR del genoma di TTV, ben più conservata in tutti gli isolati del virus. Queste analisi hanno portato all’identificazione di un numero ben maggiore di
genotipi e sottotipi (più di 40 e 70 rispettivamente), numero destinato sicuramente ad aumentare, vista la rapidità con la quale vengono identificati nuovi isolati.
Inizialmente, il criterio adottato per la classificazione prevedeva la distinzione in genotipi, identificati da numeri arabi, per isolati con divergenze nucleotidiche maggiori del 30% e in sottotipi, identificati da lettere minuscole, quando la divergenza era compresa tra il 15% e il 29%.
Seguendo queste regole, nel 2002 Peng e collaboratori proposero di raggruppare i 72 isolati fino ad allora conosciuti in 5 principali genogruppi, identificati con i numeri da 1 a 5, in base a differenze nucleotidiche inter-gruppo maggiori del 50% (Peng et al., 2002).
In questo modo, il ceppo prototipo TA278 di TTV è stato inserito insieme ai genotipi dal 2 al 6 nel genogruppo 1. L’isolato PMV, genotipo 17, (Hallet et al., 2000) è invece il prototipo del genogruppo 2, che comprende in totale 5 genotipi. Più complessa e numerosa è la distribuzione degli isolati all’interno del genogruppo 3; l’isolato prototipo è SANBAN, ma in questo genogruppo sono stati inseriti anche i ceppi SENV, per un totale di 15 genotipi. L’isolato YONBAN, genotipo 21 di TTV, è il prototipo del genogruppo 4, che include in totale 9 genotipi, mentre altri 4, rappresentati
dall’isolato JT33, sono stati classificati all’interno del genogruppo 5 (Peng et al., 2002) (fig I.2a).
Anche per TTMV è stata dimostrata una simile, se non maggiore, eterogeneità genetica, tanto che attualmente viene distinto in tre genogruppi, separati da distanze genetiche superiori al 40% (Biagini et al., 2001b; Gallian et al., 2002). Recentemente, è stato proposto da Jelcic e collaboratori un nuovo criterio di classificazione per TTV, in seguito all’identificazione e al confronto di 24 nuove sequenze con 79 isolati già presenti in banca dati. Questa classificazione prevede l’inserimento di TTV in una nuova famiglia, detta Anelloviridae, all’interno della quale gli isolati sarebbero suddivisi in 4 generi, identificati non più da numeri, ma dalle prime lettere dell’alfabeto greco. Ogni genere comprende isolati con un’omologia di sequenza minore del 50%; rispetto alla precedente classificazione, il genogruppo 1 è stato trasformato nel genere α, i genogruppi 2 e 5 riuniti nel genere β, mentre i genogruppi 3 e 4 sono stati trasformati rispettivamente nei generi γ e δ (Jelcic et al., 2004; fig. I.2b).
Secondo questo criterio, ogni genere viene poi suddiviso in specie quando l’omologia a livello nucleotidico è compresa tra il 60 e 90%, mentre si parla di tipi e sottotipi virali se l’identità di sequenza è rispettivamente minore e maggiore del 90%; per
valori invece superiori al 96% si parla di varianti dello stesso sottotipo.
Il CITV non si è però ancora pronunciato sull’approvazione di questo nuovo criterio, anche se proprio sulla base di quest’ultimo è stata già avanzata l’ipotesi di una nuova classificazione all’interno della famiglia Anelloviridae.
Secondo il nuovo metodo, ancora in corso di analisi e presentato solo in modo provvisorio, la famiglia Anelloviridae sarebbe composta non più da cinque ma da nove generi, sempre identificati con le prime lettere dell’alfabeto greco. In base a questa nuova classificazione, all’interno del genere α verrebbero inserite le cinque specie di TTV, corrispondenti ai 5 genogruppi della classificazione di Peng, il genere β comprenderebbe le 4 specie di TTMV, mentre il genere γ le 2 specie di SAV; i generi da δ a ι ospiterebbero invece i TTV degli animali.
Virus Gruppo inclusi N° tipi riferimento Isolato di Referenza TTV 1 6 TA278 Nishizawa et al., 1997
2 6 PMV Hallet et al., 2000 KAV Heller et al., 2001 3 15 SANBAN Hijikata et al., 1999
SENV Primi et al., 2000
4 9 YONBAN Takahashi et al., 2000a CT23 Peng et al., 20002 5 4 JT33 Peng et al., 20002 TTMV 1 almeno 4 CBD203 Takahashi et al., 2000b
PB4TL Biagini et al., 2001b 2 almeno 2 CBD231 Takahashi et al., 2000b 3 almeno 2 NLC023 Biagini et al., 2001b
SAV 1 1 SAV1 Jones et al., 2005
2 1 SAV2 Jones et al., 2005
Tabella I.1: Classificazione dei virus all’interno del genere Anellovirus.
a
b
Figura I.2: Alberi filogenetici costruiti sull’analisi delle distanze genetiche fra i vari isolati di TTV. In (a) quello disegnato da Peng e collaboratori (2002); i numeri indicano i 5 principali genogruppi di TTV. In (b) quello proposto da Jelcic e collaboratori (2004).
1.3 LA PARTICELLA VIRALE
Non avendo a disposizione un sistema di replicazione in vitro per TTV, le informazioni in nostro possesso sulle caratteristiche fisico-chimiche del virus derivano da studi condotti su particelle virali purificate da plasma o da feci.
Il prototipo di studio utilizzato per la caratterizzazione di TTV è stato quello appartenente al genotipo 1 (isolato TA278), perché identificato per primo e quindi più approfonditamente studiato. Non sembrano tuttavia sussistere differenze morfologiche del virione tra le diverse specie di TTV, eccetto TTMV che presenta dimensioni nettamente inferiori.
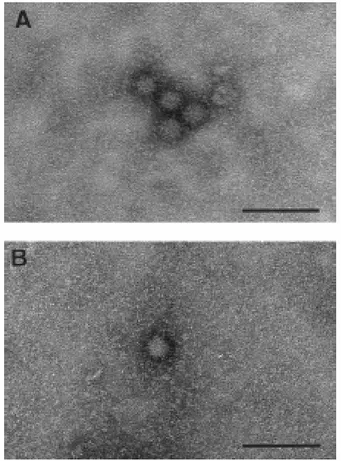
Una prima caratterizzazione morfologica di TTV è stata possibile grazie all’incubazione di sovranatante fecale, infettato da TTV di genotipo 1a, con γ-globuline purificate da plasma umano, contenente anticorpi specifici per il medesimo genotipo. Questa tecnica ha permesso la visualizzazione di TTV al microscopio elettronico (Itoh et al., 2000): il virus è apparso di forma sferica, con un capside a probabile simmetria icosaedrica, privo di involucro lipidico esterno e con un diametro di 30-32 nm, confermando quanto era stato precedentemente intuito dall’utilizzo di filtri di policarbonato a porosità decrescente (fig. I.3) (Mushahwar et al., 1999).
L’assenza di un rivestimento lipoproteico è stata ulteriormente confermata dal ritrovamento del genoma di TTV nella bile di
rcoviridae, l’eliminazione del
si ritiene che il capside sia costituito soggetti infetti (Ukita et al., 1999) e dalla resistenza del virus al trattamento con detergenti, solventi e calore secco (62°C per 96 h). Da esperimenti di ultracentrifugazione in gradiente di cloruro di cesio, la densità di TTV è risultata di 1,31-1,35 g/ml e in gradiente di saccarosio di 1,26 g/ml (Mushahwar et al., 1999; Okamoto et al., 1998).
Sin da subito TTV è apparso molto stabile e resistente; infatti, come i membri della famiglia Ci
virus in materiali infetti è stata ottenuta solo in seguito ad esposizione a temperature molto elevate o tramite purificazione per immunoaffinità (Simmonds et al., 1998; Takayama et al., 1999).
Le proteine strutturali del virus non sono ancora note ma, per la somiglianza con CAV,
da una sola proteina strutturale disposta con simmetria icosaedrica. Al momento non sono stati ancora identificati lipidi o carboidrati virali. L’esistenza nel sangue di particelle virali complessate ad immunoglobuline è stata ben documentata, mentre non si conoscono altre forme circolanti del virione (Itoh et al., 2000).
Figura I.3: Visualizzazione al microscopio elettronico di particelle di TTV isolate da sovranatante fecale.
(A) Aggregato di 4 particelle di TTV osservato in seguito ad
nte anticorpi incubazione di sovranatante fecale infettato da TTV di tipo 1a con γ-globuline di plasma umano, contene
specifici per il genotipo 1a. (B) Particella sferica singola e apparentemente senza anticorpi (da Itoh et al., 2000). La barra rappresenta una misura di 100 nm.
1.4 STRUTTURA ED ORGANIZZAZIONE DEL
GENOMA
Le informazioni sulla struttura del genoma di TTV sono state ottenute sottoponendo l’acido nucleico, estratto da plasma, all’azione di diversi enzimi: il genoma si è dimostrato sensibile alla Dnasi I e alla nucleasi Mung-bean ma resistente alla Rnasi A ed all’enzima di restrizione NdeI, evidenziando così la sua natura a singolo filamento di DNA (Okamoto et al., 1998; Mushahwar et al., 1999).
I primi studi su TTV, condotti subito dopo la sua scoperta, avevano permesso il sequenziamento di circa il 90% del genoma virale, facendo supporre che il DNA fosse lineare (Okamoto et al., 1998); analisi più approfondite hanno però portato all’identificazione di una regione ricca in GC di circa 100 nucleotidi che si è rivelata unire le due estremità del genoma (Miyata et al., 1999). Il genoma di TTV non è quindi lineare, bensì circolare.
Grazie ad esperimenti di ibridazione con molecole di RNA a polarità positiva e successiva digestione è stato possibile dimostrare che il DNA virale presenta una polarità negativa (Mushahwar et al., 1999).
Mediante clonaggio e PCR inversa, condotta a partire dalla regione N22, è stato poi possibile completare la sequenza dell’intero genoma (Mushahwar et al., 1999).
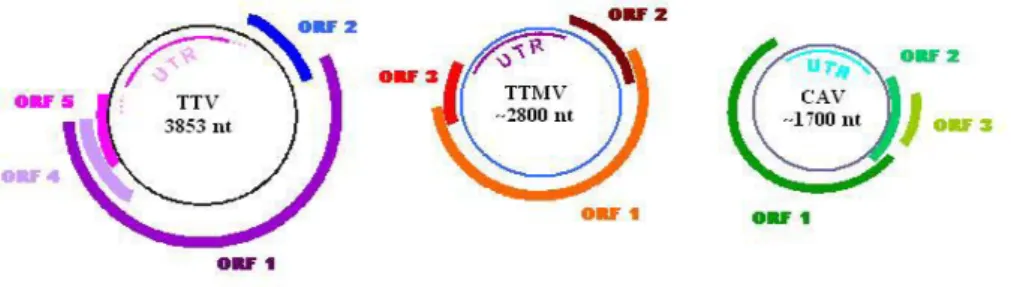
L’analisi delle sequenze di TTV e TTMV e il relativo confronto con i virus più conosciuti della famiglia Circoviridae hanno permesso di caratterizzare meglio il genoma del virus e le proteine da esso codificate. Nonostante il genoma di TTV e TTMV differisca per la rispettiva ampiezza della sequenza nucleotidica, dall’analisi strutturale di tutte le sequenze conosciute è emerso che il genoma dei due virus condivide una simile organizzazione (figura I.4).
Figura I.4. Schema e confronto del genoma di TTV (isolato TA278), TTMV e CAV. Sono evidenziate le dimensioni del genoma e le regioni codificanti.
Il genoma di TTV risulta infatti formato per due terzi da una regione codificante e per un terzo da una più piccola regione non tradotta, che sembra svolgere un ruolo molto importante nel controllo della replicazione virale e della trascrizione proteica (Kamada et al., 2004; Suzuki et al., 2004) (fig. I.5). Sul filamento positivo complementare al DNA genomico sono state individuate almeno quattro regioni codificanti, con cornici di lettura distinte ma parzialmente sovrapposte e di diversa lunghezza tra i vari isolati. Sono state avanzate delle ipotesi sulle possibili proteine codificate da questi geni, ma dati certi non sono ancora a disposizione.
Il gene ORF1 è il più ampio e codifica per una proteina di circa 770 aminoacidi (aa) in TTV e 675 in TTMV; essa rappresenta probabilmente la principale e unica componente strutturale del capside e potrebbe giocare un ruolo importante nella replicazione virale. Questa ipotesi sembra essere avvalorata dall’identificazione, all’interno della sequenza nucleotidica di tale proteina, di un motivo di circa 44 aminoacidi localizzato all’estremità N-terminale e ricco di residui basici (~40%), soprattutto arginina; per la sua natura fortemente idrofilica, questo dominio potrebbe svolgere una funzione importante nel mediare l’interazione con il DNA (Shoya et al., 1998).
A sottolineare l’affinità tra TTV e i circovirus è il ritrovamento dello stesso dominio aminoterminale della proteina codificata dal gene ORF1 anche nella proteina VP-1 di CAV.
Nella regione C-terminale sono stati invece riconosciuti i motivi aminoacidici tipici della proteina Rep, una replicasi presente nei virus a DNA che utilizzano un meccanismo a cerchio rotante per la replicazione del loro genoma.
Nella porzione centrale dell’ORF1, TTV presenta inoltre tre regioni ipervariabili, dette HVR-1, HVR-2 e HVR-3 (High Variable Regions) e potenziali siti di glicosilazione che, variabili in numero e in localizzazione nei differenti isolati, potrebbero modificare le proprietà biologiche ed antigeniche della proteina codificata da questo gene (Hijikata et al., 1999) (fig. I.5).
Il gene ORF2 codifica per una proteina strutturale di circa 120 aa in TTV e 100 aa in TTMV ed è probabilmente implicata nella replicazione del virus. A tal proposito, è stata recentemente dimostrata un’omologia di sequenza fra questa proteina, in particolare quella codificata dall’isolato SANBAN, e la proteina VP-2 di CAV, che ha una doppia funzione di fosfatasi (tirosin- e serina/treonina fosfatasi) ed è implicata nei meccanismi di trasmissione intracellulare dei segnali durante la replicazione del virus (Peters et al., 2002).
Analizzando le sequenze di molti isolati di TTV, è emerso che nell’ORF2 ci possono essere due diversi codoni di inizio trascrizione, portando così alla produzione di due proteine, indicate come ORF2a e ORF2b, uguali nella porzione carbossi-terminale ma diverse in quella amino-carbossi-terminale e le cui capacità codificanti sono ancora da definire (Tanaka et al., 2000b; Kakkola et al., 2002).
Dallo studio di transfezione in cellule COS1, mediante l’utilizzo di un costrutto ottenuto dal genoma linearizzato di TTV, posto sotto controllo di un promotore virale forte, è stata dimostrata la possibilità di eventi di splicing alternativi e la presenza di altre ORF (Kamahora et al. 2000).
In questi studi sono stati identificati tre distinti mRNA di 3,0, 1,2 e 1,0 kb con sequenze terminali comuni in 5′ e 3′.
L’analisi dei possibili siti di splicing, utilizzati per produrre questi mRNA, ha evidenziato la possibilità che esistano altre due ORF minori, dette ORF4 e ORF5, parzialmente sovrapposte alla porzione C-terminale dell’ORF1.
Il messaggero più lungo sembra contenere l’intero gene ORF1, mentre i due messaggeri più corti risulterebbero da porzioni dell’ORF2 combinate alternativamente con l’ORF4 e con l’ORF5.
L’ORF3, o ORF24, comprende circa 286 aa e l’ORF4, o ORF25, comprende circa 289 aa.
L’ORF3, di circa 286 aa, sembra codificare per due proteine (p38 e p41) dotate di un differente grado di fosforilazione e per questo simili alla proteina NS5A del virus dell’epatite C (HCV; Asabe et al., 2001). Recentemente Kooistra e collaboratori, basandosi sulla considerazione che alcune sequenze nucleotidiche di TTV mostravano similitudini con il gene dell’apoptina di CAV, hanno dimostrato che una proteina di 105 aa, codificata dalla sequenza putativa ORF3 di TTV, (Miyata et al., 1999) era in grado di provocare l’apoptosi di linee cellulari derivate da carcinoma epatocellulare umano (Kooistra et al., 2004).
Altre ORF potrebbero essere dedotte dalla sequenza nucleotidica di alcuni isolati, ma il loro significato funzionale è ancora sconosciuto (Yokoyama et al., 2002).
La rimanente porzione del genoma di TTV è costituita da una regione non tradotta di circa 1,2 Kb altamente conservata in tutti gli isolati finora conosciuti e per questo probabilmente molto importante per la vitalità del virus.
All’interno dell’UTR è possibile ritrovare in tutti gli isolati di TTV una porzione di circa cento nucleotidi ricca in
dinucleotidi GC, presente anche nella regione non tradotta di CAV.
Nell’UTR di CAV, e successivamente anche in quella di TTV, sono stati trovati elementi capaci di legare fattori di regolazione della trascrizione; in aggiunta al sito di poliadenilazione a valle dell’ORF1 e al sito di inizio trascrizione TATA BOX a monte dell’ORF2, sono state infatti identificate un certo numero di sequenze ripetute invertite, che costituiscono elementi di attivazione della trascrizione, ed altre regioni di legame per specifici fattori di trascrizione in grado di stimolare la proliferazione cellulare (SP-1, NF-κB, AP-2) (fig. I.5).
Da elaborazioni al computer è stato inoltre osservato che in tutta la regione non tradotta, soprattutto nella regione ricca in GC, si può formare una complessa struttura secondaria con motivi ad ansa, notoriamente associati a funzioni regolatorie. Studi recenti hanno mostrato che il sito di inizio della trascrizione risulta essere 113 nt a valle rispetto al promotore basale, costituito dalla TATA BOX (Kamada et al., 2004). Considerando l’elevata omologia della regione UTR, essa è stata utilizzata come bersaglio per lo sviluppo di metodiche di amplificazione potenzialmente in grado di individuare tutti i differenti genotipi di TTV.
1.5 LA REPLICAZIONE DI TTV
L’esatto meccanismo di replicazione di TTV e degli altri membri del genere anellovirus non è ancora stato definito con certezza e le ipotesi finora formulate derivano dalle conoscenze sui circovirus animali, in particolare CAV e il circovirus porcino di tipo 2 (PCV2).
Considerata la scarsa capacità codificante di tali virus, la loro replicazione è strettamente dipendente da enzimi cellulari e probabilmente anche da proteine prodotte durante la fase S del ciclo cellulare. La replicazione dei circovirus sembra quindi essere vincolata allo stato di attivazione della cellula, così come sembra accadere anche per TTV.
E’ stato infatti dimostrato in vari esperimenti come TTV necessiti, per la sua replicazione, di cellule in attiva divisione come quelle del fegato, del midollo osseo (Okamoto et al., 2000d; Okamoto e Mayumi, 2001) e del sistema linfoide; dati che indicano, almeno in parte, una localizzazione nucleare del processo replicativo (Maggi et al., 2001b; Mariscal et al., 2002).
Inoltre, l’ipotesi che TTV utilizzi enzimi di origine cellulare è rafforzata dalla mancanza nel suo genoma di sequenze codificanti per DNA polimerasi o per retrotrascrittasi note.
Analogamente ai circovirus, è stato proposto come meccanismo replicativo di TTV quello a cerchio rotante (rolling circle), con la formazione di un intermedio replicativo a doppio filamento circolare. Questa ipotesi è stata avanzata in seguito all’individuazione di alcuni motivi aminoacidici tipici della proteina Rep all’interno della regione ORF1 del virus, proteina indispensabile per replicarsi con questo meccanismo.
Inoltre, intermedi a doppio filamento circolare di DNA sono stati trovati in cellule del midollo osseo e del fegato, fattore che sembrerebbe confermare l’utilizzo di questo meccanismo. Nonostante ciò, la mancanza della sequenza nonanucleotidica, necessaria per le interazioni iniziali durante questo tipo di replicazione, fa dubitare sul fatto che TTV utilizzi realmente tale modello (Erker et al., 1999).
Considerata l’elevata eterogeneità genetica che caratterizza TTV (Umemura et al., 2001b), è inoltre probabile che la funzione di replicasi sia svolta da polimerasi di origine cellulare con scarsa attività di “correzione di bozze”.
Uno degli aspetti dell’infezione da TTV che ha destato particolare sorpresa è stato il ritrovamento del virus in un’ampia varietà di campioni, a dimostrazione della sua capacità di infettare tipi cellulari diversi.
Ciò fa supporre che i recettori utilizzati da TTV durante la fase di riconoscimento cellulare siano probabilmente molecole distribuite a livello di differenti tipi di tessuti, sebbene non sia da escludere l’utilizzo di specifici corecettori da parte dei diversi genotipi virali.
Rimane ancora da chiarire il modo in cui TTV viene rilasciato dalla cellula ospite. Tuttavia è probabile che, come per gli altri virus a DNA privi di involucro lipoproteico, la liberazione dei virioni maturi avvenga per lisi e porti di conseguenza alla morte della cellula infettata.
1.6 VARIABILITA’ GENETICA
Caratteristica peculiare di TTV è la sua elevata eterogeneità genetica, sorprendentemente elevata per un virus a DNA. A dimostrazione di ciò basta pensare all’enorme varietà di specie in cui è suddiviso il virus e alla rapidità con cui vengono continuamente identificati nuovi isolati.
La variabilità di TTV non è distribuita in modo omogeneo in tutto il genoma, ma risulta concentrata soprattutto nelle regioni codificanti con un livello massimo di divergenza all’interno delle tre sequenze HVR-1, HVR-2, HVR-3. Queste regioni sono costituite rispettivamente da 22, 47 e 31 aa nel caso del genotipo 1 e mostrano molte inserzioni e delezioni ed una divergenza amminoacidica nei differenti isolati che raggiunge in alcuni casi anche il 70%.
In uno studio condotto nel tempo, Nishizawa e collaboratori hanno analizzato la sequenza dei prodotti di PCR dedotti dall’ORF1 di due soggetti, dimostrando un’enorme variabilità delle regioni HVR nella fase acuta dell’infezione, variabilità che appariva diminuire nel periodo di remissione.
Questi dati sembrano quindi indicare un possibile coinvolgimento di queste regioni nei meccanismi di elusione del sistema immunitario (Nishizawa et al., 1999).
Le HVR sono le regioni che più contribuiscono alla formazione delle quasispecie di TTV e che mostrano un maggior livello di complessità e diversità nei soggetti con infezione cronica piuttosto che in quelli con infezione acuta autolimitante. Tuttavia sono stati evidenziati differenti gradi di variabilità all’interno dei genotipi: in un periodo compreso tra due ed otto anni, per esempio, le sequenze degli isolati di genotipo 12 e 13 in un paziente non mostravano significative mutazioni (Ukita et al., 2000).
L’elevata eterogeneità genetica di TTV non è però attribuibile solo alla presenza di HVR all’interno del genoma.
Un altro meccanismo chiamato in causa per spiegare l’elevata variabilità del virus è infatti l’utilizzo preferenziale, da parte di TTV, di polimerasi cellulari con scarsa attività di “correzione di bozze” durante la replicazione. Questa possibile spiegazione sarebbe confermata dalla frequenza con cui avvengono le sostituzioni nucleotidiche in alcune regioni del genoma (7,3x10-4 per sito per anno nelle HVR di SENV), frequenza che
risulta simile a quella di virus ad RNA e circa 10 volte maggiore di quella di virus a DNA come il virus dell’epatite B (HBV; Umemura et al., 2001b).
Un altro fattore che sembra contribuire alla determinazione dell’elevata eterogeneità genetica di TTV è la frequente presenza di infezioni miste, che favorirebbe fenomeni di ricombinazione. Spesso infatti circolano nel sangue del soggetto infettato o nello stesso distretto corporeo differenti genotipi e/o genogruppi di TTV, tra i quali possono quindi facilmente avvenire fenomeni di ricombinazione (Ball et al., 1999; Biagini et al., 1999; Mushahwar et al., 1999; Takayama et al., 1999; Niel et al., 2000; White et al., 2000). Anche per TTMV, è stata recentemente dimostrata la presenza di ben 15 differenti isolati nel sangue di un soggetto immunocompromesso (Vasconcelos et al., 2002).
La presenza di fenomeni di ricombinazione è stata anche confermata dall’identificazione di 19 siti di ricombinazione omologa, di cui 13 nella regione UTR, sia in isolati dello stesso genotipo che in quelli di genotipo diverso (Worobey, 2000; Biagini et al., 2001b).
Infine, anche la considerazione che TTV è un virus molto vecchio, che circola nella popolazione umana ormai da migliaia di anni, può giustificare il grado di variabilità genetica, che potrebbe essersi accumulata nel corso di lunghi periodi (Hijikata et al., 1999; Prescott et al., 1998).
1.7 EPIDEMIOLOGIA DI TTV
L’aspetto che forse ha destato più stupore nello studio dell’infezione da TTV è la sua elevata prevalenza nella popolazione generale e la sua ampia diffusione a livello mondiale.
I primi studi di prevalenza furono condotti utilizzando come bersaglio dell’amplificazione genomica il segmento N22 della regione ORF1; da queste analisi, circa un terzo della popolazione mondiale risultò infettato da TTV, con leggere differenze a livello geografico (Niel et al., 1999). La frequenza di individui positivi più alta fu trovata tra le popolazioni africane e sud americane, mentre in Asia il virus risultò mediamente prevalente. Nei paesi industrializzati, Stati Uniti e Nord Europa, la frequenza di TTV si dimostrò invece molto bassa. In particolare, fu notato che la diversa prevalenza del virus non era associata a particolari variabili, non dipendeva cioè dalle condizioni socio-economiche, dall’età, dal sesso o dall’appartenenza etnica.
Gli studi successivi dimostrarono però che la scelta del segmento N22 come bersaglio di amplificazione genomica non permetteva di identificare tutti gli isolati di TTV, ma solo i genotipi dall’1 al 6.
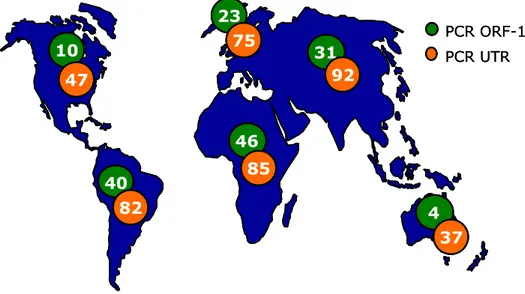
L’avvento della PCR universale, sia nella versione qualitativa che in quella quantitativa, utilizzando come bersaglio di amplificazione la regione UTR, molto più conservata, ha aumentato ulteriormente le percentuali di diffusione del virus. TTV è stato ritrovato in circa 2/3 della popolazione mondiale con modeste variazioni fra le differenti aree geografiche (Matsubara et al., 2001; Niel et Lampe, 2001); in particolare, la prevalenza di TTV tende ad essere più moderata in Nord America ed Oceania (circa 40%) e più elevata in Asia (> 90%). Nella maggior parte delle regioni analizzate, TTV è presente con una frequenza maggiore all’80%, con picchi di prevalenza che in alcuni paesi come Arabia Saudita, Singapore, Giappone possono raggiungere il 100% (fig. I.6).
40 46 85 31 23 10 PCR ORF-1PCR UTR 92 82 75 4 37 47 40 40 46 46 85 85 31 31 23 23 10 10 PCR ORF-1PCR UTR 92 92 82 82 75 75 4 4 37 37 47 47
Figura I.6: Prevalenza di TTV nella popolazione generale. Le percentuali indicate si riferiscono ai dati ottenuti amplificando regioni dell’ORF1 (verde) e dell’UTR (rosso), da Bendinelli et al., 2001.
Da alcuni studi è emerso che l’infezione da TTV sembra aumentare con l’età dei soggetti analizzati (Suzuki et al., 2000; Umemura et al., 2001a), anche se risulta altamente diffusa già nella prima infanzia (Vasconcelos et al., 2002).
E’ inoltre da sottolineare che l’utilizzo della PCR permette di rilevare solo le forme attive dell’infezione, sia croniche che acute, ma non evidenzia i casi di infezione pregressa, identificabili solo con test sierologici, attualmente non disponibili per TTV.
E’ quindi ipotizzabile che i livelli di prevalenza siano ancora più elevati di quelli osservati al momento. A conferma di ciò, il ritrovamento di sequenze virali in assenza di viremia (Maggi et al., 2001c; Pollicino et al., 2002).
La prevalenza così elevata di TTV in tutta la popolazione generale fa presupporre che il virus utilizzi molteplici modalità di trasmissione.
Quelle sicuramente già accertate sono la via parenterale e oro-fecale, sebbene molte altre siano state ipotizzate. Numerosi studi hanno infatti dimostrato che TTV e TTMV sono particolarmente frequenti in soggetti politrasfusi, talassemici, emodializzati, emofilici, tossicodipendenti e tutti coloro esposti al sangue o agli emoderivati; inoltre tali virus vengono frequentemente escreti con le feci dai soggetti infetti (Gallian et al., 2000; Luo et al., 2000; Lin et al., 2000; Nakagawa et al., 2000; Tawara et al., 2000; Azzi et al., 2001; Boysen et al., 2003).
Il ritrovamento di TTV nei neonati e in più del 50% dei campioni di sangue cordale esaminati ha fatto supporre una via di trasmissione materno-fetale del virus e ha suggerito l’ipotesi che le madri TTV positive possano infettare il figlio attraverso la placenta (Morrica et al., 2000).
TTV è stato anche identificato nel latte materno, spiegando così l’esistenza di neonati positivi con madri negative e di bambini che presentano ceppi di virus diversi da quelli trovati nel sangue materno (Davidson et al., 1999).
Un’ulteriore via di trasmissione, al momento non accertata con sicurezza, potrebbe essere quella sessuale: TTV e TTMV sono infatti stati isolati dalle secrezioni vaginali, dal seme e dalla saliva di alcuni soggetti (Gerner et al., 2000; Kazi et al., 2000; Inami et al., 2000; Liu et al., 2000; Martinez et al., 2000; Matsubara et al., 2000; Matsubara et al., 2001; Schroter et al., 2000; Stark et al., 2000; Biagini et al., 2001a; Chan et al., 2001; Fornai et al., 2001; Krekulova et al., 2001; Pirovano et al., 2002a).
Recentemente, TTV è stato ritrovato nelle secrezioni e nelle cellule nasali di bambini con malattie respiratorie acute, con titoli più elevati rispetto a quelli nel corrispondente campione di plasma. Questi dati indicano la via aerea come ulteriore possibile modalità di trasmissione, nonché l’eventualità che il tratto respiratorio possa essere un sito primario di colonizzazione e replicazione nell’ospite (Maggi et al., 2003). Sequenze simili agli isolati di TTV sono state identificate anche in animali da allevamento (bovini, suini, ovini e pollame) e da compagnia (cani e gatti), facendo quindi supporre la
possibilità di una trasmissione inter-specie dell’infezione (Leary et al., 1999; Okamoto et al., 2002).
Infine l’elevata prevalenza di TTV suggerisce che certi genotipi siano più diffusi di altri e che esistano differenze nella loro distribuzione geografica (Gallian et al., 2000; Mikuni et al., 2002). Ad esempio, il genotipo 1 ed alcuni genotipi di SENV appaiono più diffusi in Asia piuttosto che nei Paesi occidentali: in uno studio, il 10% dei donatori in Giappone è risultato positivo per SENV-D e SENV-H rispetto al 2-3% dei donatori positivi in USA ed Italia (Shibata et al., 2001; Umemura et al., 2001a). E’ stata anche dimostrata una differente prevalenza di alcuni genotipi di TTV fra paesi dello stesso continente: l’infezione con i genotipi 22 e 23 è risultata per esempio frequente in Indonesia, ma praticamente assente in Giappone (Muljono et al., 2001).
1.8 DIAGNOSI DI LABORATORIO
Sono poche le metodiche che permettono la rilevazione diagnostica di TTV, dal momento che non è attualmente possibile disporre di efficienti sistemi di coltivazione o di saggi per la ricerca di antigeni virali. L’unico approccio ad ora disponibile è quindi l’utilizzo di tecniche di amplificazione molecolare degli acidi nucleici, sia di tipo quantitativo che qualitativo (tab. I.2).
Data la vasta eterogeneità genetica che caratterizza TTV, si è rivelato di fondamentale importanza scegliere correttamente la regione bersaglio di amplificazione.
Inizialmente è stata utilizzata la porzione N22 del gene ORF1 (PCR N22); questo frammento, in seguito, non si è rivelato conservato tra tutti gli isolati di TTV e la metodica ben presto è apparsa valida solo per l’amplificazione dei genotipi 1-6 (Muljiono et al., 2001).
Studi più approfonditi sul genoma di TTV hanno poi identificato la regione UTR, che si è dimostrata molto conservata in tutti gli isolati del virus e che per questo motivo è stata scelta come bersaglio di amplificazione (PCR UTR o PCR universale). Il suo utilizzo ha infatti notevolmente
incrementato la percentuale dei soggetti risultati positivi per TTV.
La carica totale di TTV presente in un campione può essere quantificata attraverso metodiche di Real Time PCR, anch’esse dedotte dalla UTR (Kato et al., 2000; Nakagawa et al., 2000; Maggi et al., 2001a; Maggi et al.,2001c; Moen et al., 2002), e l’isolato può poi essere caratterizzato molecolarmente grazie all’utilizzo di PCR genogruppo-specifiche dedotte da diverse regioni del genoma virale (Maggi et al., 2001c).
L’appartenenza di un isolato di TTV ad un genogruppo e ad un particolare genotipo può essere successivamente confermata dal sequenziamento dei prodotti ottenuti con i saggi di PCR genogruppo-specifiche.
Virus Scopo Tipo di saggio bersaglio Regione
TTV Individuazione PCR qualitativa UTR
Quantificazione PCR Real-Time UTR
Tipizzazione PCR genogruppo-specifiche UTR / ORF1
Sequenziamento UTR/ORF1
Tabella I.2: Principali metodi molecolari usati per la diagnosi di infezione da TTV e TTMV.
Sebbene attualmente non siano disponibili, come già detto precedentemente, test sierologici per identificare anche infezioni pregresse da TTV, sono stati elaborati alcuni metodi per la determinazione di anticorpi contro il virus. Attraverso un saggio di immunoprecipitazione, nel siero di alcuni pazienti sono state rilevate IgG umane dirette contro TTV ed è stata quantificata la percentuale del virus immunocomplessato (Tsuda et al., 1999). Mediante il medesimo approccio sono state anche rilevate le IgM specifiche, utilizzando come antigene il TTV estratto da campioni fecali: le IgM trovate sono ritenute un buon marcatore dell’infezione acuta (Tsuda et al., 2001).
Utilizzando come antigeni frammenti della proteina ORF1 o ORF2 espressi in batteri, sono stati effettuati anche saggi di immunoblotting (Ott et al., 2000; Kakkola et al., 2002).
Il ritrovamento di valori diversi di prevalenza nei vari studi (dal 10 al 98% dei soggetti sani sono risultati positivi per le IgG anti-TTV), ha suggerito che il tipo di antigene adoperato è in grado di influenzare enormemente i risultati del saggio sierologico (Ott et al., 2000; Tsuda et al., 2001; Kakkola et al. 2002).
1.9 INTERAZIONI CON L’OSPITE
Le conoscenze riguardo alla storia naturale dell’infezione da TTV non sono ancora complete, ma solo alcuni aspetti sono stati dimostrati con chiarezza. Prima caratteristica tra tutte è la cronicizzazione dell’infezione, con la presenza di elevate cariche di TTV nel sangue dei soggetti infetti (Lefrere et al., 1999; Matsumoto et al., 1999; Yuki et al., 1999).
Da alcuni studi è emerso che circa l’80% dei soggetti infettati da TTV diventa portatore cronico del virus, spesso per molti anni (Takayama et al., 1999; Lefrere et al., 1999; Ali et al., 2002). Non mancano tuttavia casi in cui si assiste ad una rapida risoluzione dell’infezione, dovuta probabilmente a fattori diversi; la scomparsa del DNA virale può infatti essere attribuita alla completa eradicazione del virus, anche se sono state isolate sequenze in diversi tessuti pur in assenza di viremia (Maggi et al., 2001c; Pollicino et al., 2002).
L’assenza di TTV nel sangue dei soggetti può anche essere il risultato di una ridotta immissione in circolo del virus dovuta a fasi di latenza o ad un aumento della sua eliminazione oppure di una modificazione delle sequenze virali che non permette più la rilevazione del DNA (Yuki et al., 1999). Sembra comunque escluso che TTV possa esistere in forma episomiale
o integrata nel DNA delle cellule infettate (Tanaka et al., 2000a; Yu et al., 2002), fenomeno che invece risulta possibile per altri virus a DNA.
Una considerazione tra le più verosimili è che l’andamento dell’infezione da TTV sia diverso a seconda del genotipo infettante. A tal proposito, in uno studio condotto da Ohto e collaboratori (Ohto et al., 2002) è stata osservata la risoluzione spontanea dell’infezione in circa il 20% dei bambini infettati da TTV, ma questa percentuale differiva a seconda del genotipo virale infettante.
Sembra quindi possibile che il diverso andamento delle infezioni da TTV sia dovuto ad una differente capacità di persistenza dei genotipi del virus.
Anche i meccanismi implicati nella persistenza protratta del DNA di TTV non sono molto chiari, ma sembra giocare un ruolo fondamentale il sistema immunitario, incapace di controllare l’infezione.
In seguito all’infezione da TTV è stata osservata la comparsa di immunoglobuline di tipo M dopo circa 10-12 settimane dall’infezione, dimostrabili nel sangue per 5-11 settimane, insieme ad una più lenta, ma anche più duratura, produzione di IgG (intorno alla 15°-17° settimana), che tendono a persistere per anni (Tsuda et al., 2001).
Tuttavia rimane ancora da chiarire se questi anticorpi siano in grado di neutralizzare l’infezione; la presenza delle IgG infatti, è stata in alcuni casi associata con l’assenza o la scomparsa della viremia plasmatica, mentre altre volte tale presenza non sembra aver prodotto nessun effetto sulla persistenza del virus (Tsuda et al., 1999; Ott et al., 2000). A conferma di ciò è stato osservato che, in pazienti cronicamente infetti, TTV sembra circolare complessato ad IgG (Nishizawa et al., 1999), mentre nel sovranatante fecale è stato trovato virus libero. L’elevata frequenza con cui si verificano infezioni multiple mette comunque in evidenza la scarsa efficacia del sistema immunitario nel contrastare il virus.
Anche la risposta cellulo-mediata sembra avere un duplice ruolo nel controllo dell’infezione da TTV: le cellule del sistema immunitario possono infatti contribuire sia alla persistenza che alla disseminazione virale ai vari tessuti.
Il ritrovamento di TTV in molti tessuti e fluidi biologici, tra cui polmone, milza, pancreas, rene, linfonodi, muscolo scheletrico e tiroide, bile, saliva, seme, lacrime, fluido duodenale e secrezioni vaginali, indica che il virus è disseminato in tutto l’organismo e può stabilire un’infezione persistente in vari distretti corporei (Deng et al., 2000; Biagini et al., 2001a; Tajiri et al., 2001; Okamoto et al., 2001; Suzuki et al., 2001).
In molti di questi tessuti è stato ritrovato anche l’intermedio di replicazione di TTV, a dimostrazione che il virus non si replica solo nel fegato e nel midollo osseo, come ritenuto precedentemente, ma probabilmente in tutte le cellule che infetta (Okamoto et al., 1998; 2000b; 2000d; 2000e; Okamoto et al., 2001a; Kikuchi et al., 2000; Lopez-Alcorocho et al., 2000; Rodriguez-Inigo et al., 2000; Comar et al., 2002).
E’ tuttavia molto probabile che TTV necessiti, per la replicazione, di cellule in attiva divisione, come dimostrato nel caso dei linfo-monociti, in grado di supportare la replicazione virale solo se opportunamente stimolati (Maggi et al., 2001b; Mariscal et al., 2002).
Non è ancora chiaro se esista una sottopopolazione linfocitaria nella quale il virus si replichi e/o persista preferenzialmente, così come non è noto se i diversi genotipi possano avere tropismo tissutale diverso (Lopez-Alcorocho et al., 2000; Takahashi et al., 2002; Yu et al., 2002; Zhong et al., 2002). Quest’ultima ipotesi è in parte sostenuta dal ritrovamento, in diversi organi di un singolo soggetto, di genotipi apparentemente tessuto-specifici, in quanto diversi dalle sequenze in circolo (Okamoto et al., 2000c). Tuttavia il ritrovamento di ben 24 isolati nella milza di un paziente
affetto da tumore fa riconsiderare quanto precedentemente osservato (Jelcic et al., 2004).
Nella fase cronica dell’infezione da TTV sono stati osservati livelli virali plasmatici paragonabili a quelli di altri virus persistenti, come HCV, HIV e HBV. I titoli di TTV osservati nel plasma di diversi soggetti mostrano un ampio range di variabilità, oscillando fra 102 e 108 copie di DNA/ml (Pistello et
al., 2001); tali titoli possono rimanere stabili nel tempo in alcuni individui o variare notevolmente in altri.
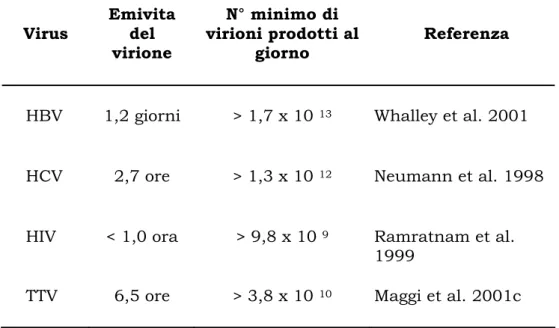
In un recente lavoro, applicando un modello matematico simile a quello utilizzato per HIV e HCV, è stato possibile studiare in pazienti positivi per HCV e cronicamente infettati da TTV, la cinetica di TTV in risposta alla terapia con interferone (IFN). Dallo studio è emerso che l’emivita di TTV nel plasma è di circa 6 ore, che ogni giorno vengono prodotti almeno 1010 virioni e che più del 90% del virus plasmatico
subisce un turnover giornaliero (Tabella I.3; Maggi et al., 2001c).
Virus Emivita del virione N° minimo di virioni prodotti al giorno Referenza
HBV 1,2 giorni > 1,7 x 10 13 Whalley et al. 2001
HCV 2,7 ore > 1,3 x 10 12 Neumann et al. 1998
HIV < 1,0 ora > 9,8 x 10 9 Ramratnam et al. 1999
TTV 6,5 ore > 3,8 x 10 10 Maggi et al. 2001c
Tabella I.3: Dinamica della viremia plasmatica di TTV confrontata con quella di altri virus persistenti.
1.9 ASSOCIAZIONE CON LA PATOLOGIA
La scoperta di TTV in un paziente affetto da epatite criptogenetica fece subito pensare di aver identificato il virus responsabile di quelle patologie epatiche di cui ancora non era nota l’origine eziologica. In realtà, anche se non è da escludere un coinvolgimento occasionale di TTV nella patologia epatica, sembra essere ormai confermato che TTV non sia la causa diretta di queste malattie del fegato (Berg et al., 1999; Mushahwar et al., 1999; Matsumoto et al., 1999; Kao et al., 2000; Shang et al., 2000; Ali et al., 2002).
È stata infatti dimostrata la presenza di TTV non solo in pazienti con epatiti criptogenetiche, ma anche in soggetti privi di alterazioni epatiche o addirittura senza alcuna manifestazione patologica (Berg et al., 1999; Viazov et al., 1998). Inoltre non è stato osservato danno epatico in scimpanzè naturalmente o sperimentalmente infettati con TTV o con virus correlati (Mushahwar et al., 1999), né è stato dimostrato un ruolo di TTV nella progressione di malattie epatiche in soggetti già infettati dai virus dell’epatite B o C. In letteratura sono comunque presenti anche studi in cui si è rilevata una possibile associazione tra danno epatico e TTV, come dimostrano le sporadiche correlazioni fra viremia ed
andamento delle transaminasi plasmatiche in alcuni soggetti (Cleavinger et al., 2000; Forns et al., 1999; Lefrère et al., 1999; Luo et al., 1999). Inoltre, in uno studio condotto su 26 casi di epatite fulminante, la mortalità registrata è stata pari al 100% in soggetti TTV positivi e del 50% in quelli negativi (Tanaka et al., 1999).
Non è quindi da escludere un possibile ruolo di TTV nell’aggravamento di queste patologie del fegato.
Bisogna tuttavia sottolineare che, vista l’enorme diffusione di TTV nella popolazione sana e la sua elevata eterogeneità genetica, e data la capacità del virus di instaurare infezioni durature, alcuni ricercatori hanno suggerito che TTV possa essere considerato un commensale della flora microbica umana o, più semplicemente, un virus completamente apatogeno (Griffiths, 1999; Mushahwar, 2000).
La difficile associazione clinica dell’infezione da TTV, e anche da TTMV, è dovuta al falso presupposto che questi virus avessero un tropismo prevalentemente epatico.
Una volta constatata l’assenza di un ruolo diretto di TTV e TTMV in questo tipo di affezioni, è stato indagato il possibile coinvolgimento di tali virus anche in altre patologie.
In particolare è stata osservata una possibile associazione dell’infezione da TTMV con gravi patologie dell’apparato renale
(Gallian et al., 2002) e la capacità di TTV d’indurre in vitro alterazioni in cellule renali simili a quelle osservate nella sindrome nefrosica (Yokoyama et al., 2002). In questo lavoro topi transgenici per varie porzioni dell’ORF1 mostravano segni patologici solo a livello del rene, tanto più gravi quanto maggiore era l’espressione del transgene, a conferma che il danno renale era indotto dalla proteina codificata.
Correlazioni interessanti sono state osservate anche tra TTV e patologie del sistema linfoide. Recenti studi hanno dimostrato che livelli più elevati del virus circolano in pazienti immunocompromessi, come quelli con infezione da HIV o con altre malattie intercorrenti (Shibayama et al., 2001; Simmonds, 2002), suggerendo che la replicazione di TTV possa essere controllata dal sistema immunitario, il cui indebolimento causerebbe un aumento dei titoli virali. Ciò è stato ulteriormente confermato dall’incremento della viremia di TTV in pazienti sottoposti a trapianto epatico (Shang et al., 2000), oltre che da una correlazione inversa fra i titoli del virus ed il numero di linfociti CD4+ in pazienti HIV positivi
(Christensen et al., 2000; Shibayama et al., 2001; Touinssi et al., 2001).
Al contrario nessuna correlazione è emersa con altre malattie come crioglobulinemia, psoriasi, lupus eritematoso sistemico (Cacoub et al., 2003; Maggi et al., 1999), mentre parziali associazioni sono state osservate in pazienti affetti da diabete, fibrosi polmonare idiopatica, artrite reumatoide in emodializzati (Gallian et al., 1999; Bando et al., 2001; Boysen et al., 2003).
Inoltre sono stati evidenziati elevati titoli di TTV in bambini con broncopolmonite (BP), piuttosto che in quelli affetti da forme meno gravi di malattie respiratorie acute (MRA); anche in questo caso, tuttavia, tale risultato non può essere considerato la dimostrazione che il virus sia la diretta causa di questo tipo di patologie (Maggi et al., 2003).
L’ampia variabilità geneticadei virus del gruppo TTV e TTMV e la loro somiglianza con i circovirus animali hanno suggerito l’ipotesi che i diversi genotipi abbiano un differente ruolo in relazione a determinate patologie, così come osservato per i circovirus porcini, dei quali solo il tipo 2 si comporta come un potente patogeno (Chae, 2004).
A favore di questa ipotesi c’è la correlazione osservata tra la presenza del genotipo 1a di TTV e il livello delle transaminasi plasmatiche in soggetti con patologie epatiche e il ritrovamento, sempre del genotipo 1, in pazienti con epatite
fulminante o con aggravamento delle condizioni cliniche (Okamura et al., 2000; Shibata et al., 2000; Moriyama et al., 2001; Tokita et al., 2001a; 2001b; 2001c; 2002).
E’ stata inoltre avanzata l’ipotesi di una possibile associazione tra genogruppo 4 di TTV e casi di broncopolmonite: a sostegno di ciò è stato infatti ritrovato tale genogruppo altamente frequente in bambini affetti da questa patologia (Maggi et al., 2003).
1.10 SENSIBILITA’ AI FARMACI ANTIVIRALI
La sensibilità di TTV ai farmaci è un altro importante aspetto che non è stato possibile analizzare direttamente, ma il cui studio è avvenuto prevalentemente in pazienti coinfettati con il virus dell’epatite C. Per questo motivo le conoscenze attualmente a disposizione derivano da analisi effettuate in corso di terapia con interferone. Le risposte osservate per TTV sono risultate paragonabili a quelle per HCV.
Dopo una o due settimane di trattamento, la terapia con IFN induce, in circa il 50% dei pazienti, una scomparsa della viremia plasmatica di TTV e livelli virali bassi o nulli per tutta la durata del trattamento (Kao et al., 2000; Dai et al., 2002). Nel rimanente 50%, si può assistere ad alcuni casi in cui la terapia con IFN non esercita particolari effetti sui livelli della viremia, mentre in altri casi il titolo virale varia notevolmente durante il periodo di osservazione, per ritornare al termine del trattamento paragonabile ai livelli basali (Akahane et al., 1999; Berg et al., 1999; Chayama et al., 1999; Maggi et al., 1999). Non è escluso comunque che in molti casi la ricomparsa di TTV dopo la terapia possa essere dovuta ad una reinfezione (Ali et al., 2002).
I motivi che causano queste diverse risposte non sono ancora chiari, anche se è stata dimostrata una migliore risposta all’interferone nel caso di bassi livelli viremici pre-trattamento e in presenza di isolati di TTV filogeneticamente distanti dal prototipo 1a (Akahane et al., 1999; Chayama et al., 1999). Inoltre, la combinazione di IFN con ribavirina mostra una maggiore efficacia nell’indurre una risposta a lungo termine verso TTV (Berg et al., 1999).
Non è chiaro se esistano differenze tra i vari genotipi di TTV nella suscettibilità all’IFN, evento comunque ritenuto probabile. Infatti, osservazioni recenti hanno mostrato, dopo il trattamento con IFN, la scomparsa del virus circolante nel 60-80% dei pazienti infettati con l’isolato SENV; quest’ultimo sembra quindi particolarmente sensibile all’azione di tale farmaco (Umemura et al., 2002; Wong et al., 2002).
La variazione dei livelli di TTV è stata studiata anche in pazienti HIV positivi trattati con farmaci antiretrovirali. Questa terapia non sembra però avere effetto sulla viremia da TTV, né la presenza del virus sembra alterare la capacità di risposta ai farmaci antiretrovirali (Watanabe et al., 2000; Kawanaka et al., 2002).