Confronto critico fra metodologie statistiche
di trattamento dei dati di espressione genica
In questo capitolo verranno presentati i risultati ottenuti dal confronto critico fra i metodi di analisi della significatività statistica, l’approccio empirico bayesiano e l’analisi della varianza per la selezione dei geni differenzialmente espressi.
Questo confronto è stato realizzato utilizzando l’insieme di dati relativi ad uno studio sull’organismo C. elegans (Golden et al., 2004), il cui scopo è stato individuare i geni potenzialmente coinvolti nel processo di crescita e invecchiamento del nematode.
Dopo aver individuato i criteri di pre-trattamento e normalizzazione dei dati grezzi, i risultati dello studio sono stati analizzati ed utilizzati per l’individuazione di un procedimento di validazione informatica incrociata.
6.1 Descrizione dell’esperimento
Il nematode C. elegans è un eccellente modello per studiare l’invecchiamento sia perché è stato ben caratterizzato da un punto di vista morfologico, sia perché sono disponibili numerosi mutanti che si distinguono significativamente per la durata della loro vita.
Nello studio preso in considerazione sono state messe a confronto, attraverso esperimenti con microarray a cDNA, l’espressione genica nel nematode “wild-type” N2 e nel mutante daf-2 , che ha una durata della vita pari al doppio di quella dell’organismo wild-type.
Sono stati analizzati cinque individui per ogni tipo e i dati di espressione genica sono stati ricavati in quattro istanti temporali differenti. In particolare, ogni individuo è stato caratterizzato a quattro stadi di crescita corrispondenti al quarto, al nono, al quattordicesimo e al diciannovesimo giorno dalla nascita. Le relative osservazioni sono state indicate con: N24d, N29d, N214d, N219d, daf24d, daf29d, daf214d, daf219d.
Per motivi non specificati, sono disponibili trentasette insiemi di osservazioni, anziché quaranta.
6.2 Caratteristiche del microarray
I microarray utilizzati in questo studio sono stati prodotti dallo stesso laboratorio che ha condotto il lavoro ed ognuno di essi è costituito da 923 sequenze, rappresentative di 921 geni, scelti in base al loro coinvolgimento in processi di risposta allo stress e agli interessi degli stessi ricercatori.
A queste sequenze sono stati aggiunti 661 controlli negativi, cioè spot sui quali non è stata depositata alcuna sequenza.
Vi sono, quindi, 1584 spot, ognuno dei quali è presente in quadruplice copia su ogni array; in totale ogni microarray è composto di 6336 spot.
6.3 Disegno sperimentale
Lo scopo principale di questo studio è stato riuscire ad identificare quali geni potessero essere responsabili della durata diversa della vita dei due nematodi, confrontando l’espressione genica dei due tipi di individui allo stesso stadio di crescita.
Un’altra questione di interesse è stata cercare di individuare i geni coinvolti nel processo di invecchiamento di ogni organismo.
Per poter dare una risposta ad entrambi i quesiti biologici è stato scelto un disegno sperimentale con riferimento, in modo da fissare una base di confronto comune per tutte le osservazioni.
Il campione di riferimento era costituito da un “pool” di RNA estratti dai nematodi N2.
6.4 Trattamento del dato
Le immagini dei microarray dopo l’ibridizzazione dei campioni sono state acquisite con uno scanner Packard Bioscience; il dato è stato poi
quantizzato utilizzando il software Genepix (Axon) senza fare alcuna selezione ulteriore dei dati.
Gli spot sono stati in seguito eliminati solo in base alla presenza di artefatti spaziali o difetti di forma dello spot, ma, poiché il livello globale di rumore non è stato giudicato alto, non è stata effettuata una correzione del backgound o una selezione in base al valore del rapporto segnale/rumore.
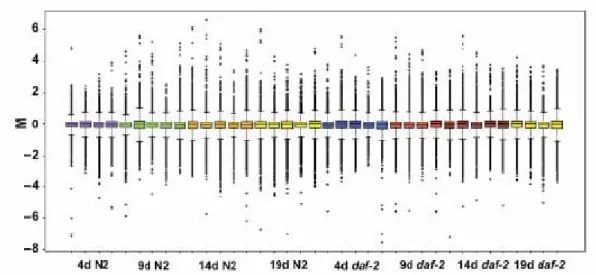
Gli spot giudicati idonei dopo questo esame sono stati normalizzati applicando una normalizzazione LOESS globale su ogni array, mentre, dopo aver osservato l’andamento dei dati normalizzati (figura 6.1), non è stata ritenuta necessaria una normalizzazione “between arrays”.
Figura 6.1: Box-plot degli array dopo la normalizzazione LOESS globale
Gli spot replicati per ogni sequenza all’interno dello stesso array sono stati utilizzati per ottenere una stima più affidabile del valore di espressione, attraverso un’interpolazione lineare ai minimi quadrati delle osservazioni intra-array. Analogamente, le repliche biologiche, cioè le osservazioni inter-array della stessa sequenza, hanno subito lo stesso processo di interpolazione allo scopo di ottenere un unico dato cumulativo dell’informazione totale disponibile per ogni sequenza ad ogni stadio di crescita.
6.5 Definizione dei contrasti per la selezione dei geni
differenzialmente espressi
I geni che presentano espressione differenziale nei diversi confronti sono stati selezionati sulla base della definizione di contrasti, cioè combinazioni lineari delle medie delle popolazioni coinvolte (Freund & Wilson, 1997).
Questi contrasti, generalmente riassunti in una matrice, sono stati poi utilizzati in un processo empirico bayesiano di stima dei parametri ed è stata ricavata la statistica B per ogni gene.
Per determinare i geni che differenziano l’organismo N2 dal mutante daf-2 allo stesso stadio di crescita, prendendo il quarto giorno come riferimento per le osservazioni relative ad ogni genotipo, sono stati selezionati tre contrasti e sono stati determinati tre insiemi di geni potenzialmente espressi in maniera significativa in base al valore assunto dalla statistica B per ogni gene identificato.
Sui dati relativi alle statistiche t di ogni gene è stato poi effettuato un F-test in modo da identificare le interazioni significative che coinvolgono sia i due diversi genotipi che lo stadio di crescita.
Per individuare i potenziali marcatori biologici del processo di invecchiamento in individui dello stesso tipo sono stati impostati dei contrasti più semplici che, prendendo come riferimento i dati ricavati al quarto giorno di crescita, hanno consentito di osservare quali geni presentano espressione differenziale per ogni stadio.
Infine, per osservare l’espressione differenziale fra genotipi diversi della stessa età, sono stati studiati i contrasti derivanti dalle osservazioni relative agli organismi N2 e daf-2 allo stesso stadio di crescita.
6.6 Definizione delle sessioni di prove
Lo scopo delle elaborazioni realizzate in questa tesi è stato osservare il comportamento dei dati sottoposti a diversi processi di selezione e normalizzazione degli spot e le potenzialità dei diversi approcci di analisi statistica nell’individuare i geni differenzialmente espressi, allo scopo di definire una metodica di validazione informatica incrociata dei dati di espressione genica.
I metodi utilizzati per la realizzazione delle prove sono l’analisi della significatività statistica, l’approccio bayesiano di stima dei parametri e l’analisi della varianza. Tali metodi sono implementati da alcuni software, disponibili in rete sui siti www.r-project.org e www.bioconductor.org nei pacchetti “siggenes”, per l’analisi della significatività statistica, “Limma” per l’approccio empirico bayesiano e “maanova” per l’analisi della varianza. Questi pacchetti sono stati utilizzati dopo averli inseriti nel software “R” di statistica biomedica come librerie.
Con ciascuno di questi metodi sono state realizzate tre sessioni di prove utilizzando i dati forniti dallo studio di invecchiamento e sottoponendoli a differenti processi di pre-trattamento e normalizzazione. In particolare:
nella Sessione I di prove sono stati utilizzati dati che non hanno subito il processo di sottrazione del background e di selezione in base al valore di SNR sottoposti ad una normalizzazione LOESS globale;
nella Sessione II di prove sono stati utilizzati dati a background sottratto, selezionati in base ad un livello accettabile di rumore e normalizzati con LOESS globale;
nella Sessione III di prove sono stati utilizzati dati a background sottratto, selezionati in base ad un livello accettabile di rumore e sottoposti ad una normalizzazione “print-tip”.
La prima sessione di prove ha avuto lo scopo di riprodurre i risultati che vengono presentati nello studio di Golden, utilizzando per l’elaborazione l’approccio empirico bayesiano, in modo da poterli poi confrontare con quelli ottenuti dalle elaborazioni realizzate con gli altri metodi.
Nella seconda sessione di prove si è cercato di mettere in evidenza il peso della sottrazione del background e della selezione degli spot in base ad un valore di soglia per il rapporto segnale/rumore, sull’identificazione dei geni differenzialmente espressi e sulla capacità dei tre metodi di elaborare un insieme di dati ridotto a causa della selezione.
Infine, la terza sessione di prove ha avuto lo scopo di valutare il grado di variabilità dei risultati a seconda del tipo di normalizzazione ad essi applicata.
6.7 Risultati delle diverse sessioni di prove
Prima di descrivere i risultati è opportuno fornire una chiave di lettura per i grafici ed illustrare i criteri che hanno portato alla realizzazione dei confronti che seguiranno.
I contrasti utilizzati nelle sessioni di prova sono stati impostati in maniera diversa a seconda del quesito biologico.
Per identificare quali geni risultano differenzialmente espressi fra i due genotipi a parità di stadio di crescita e rispetto allo stadio iniziale di riferimento sono stati utilizzati i seguenti contrasti:
(daf29d – daf24d) – (N29d – N24d); (daf214d – daf24d) – (N214d – N24d); (daf219d – daf24d) – (N219d – N24d).
L’insieme di questi contrasti è stato denominato durante le prove con il termine “full” ed ogni contrasto C è stato caratterizzato con un numero che indica la sua posizione nella lista appena descritta e con il suffisso “f” che specifica la sua appartenenza a questo insieme di contrasti. Secondo questa regola il contrasto C1f identifica il primo contrasto della lista.
I geni differenzialmente espressi nello stesso genotipo rispetto al quarto giorno di crescita sono stati estratti utilizzando i seguenti contrasti:
N29d – N24d; N214d – N24d; N219d – N24d; daf29d – daf24d; daf214d – daf24d; daf219d – daf24d.
Ognuno di questi contrasti è stato contrassegnato nelle prove con un numero che indica la sua posizione nella lista precedente, per cui, ad esempio, il contrasto daf219d – daf24d sarà identificato con C6.
Per finire, i contrasti che hanno consentito di identificare i geni differenzialmente espressi fra i due genotipi, a parità di stadio di crescita sono:
daf24d – N24d; daf29d – N29d; daf214d – N214d; daf219d – N219d.
L’insieme di questi contrasti è stato contrassegnato con il termine “age” ed ogni contrasto è identificato con un numero che indica la sua posizione nella lista precedente ed il suffisso “age” di appartenenza all’insieme dei contrasti, quindi con C3age si indicherà, ad esempio, il contrasto daf214d – N214d.
Ogni identificativo dei contrasti sarà ulteriormente caratterizzato con:
“b”, se si riferisce a dati che non hanno subito la sottrazione
del background e la selezione in base alla soglia di rumore; “bs” se è stato sottratto il background ed è stata utilizzata una
soglia di rapporto segnale/rumore per l’accettabilità dello spot;
“loess” se la normalizzazione dei dati è stata realizzata
utilizzando il metodo di interpolazione LOESS;
“p-t” quando la normalizzazione è avvenuta con il metodo
“print-tip”
“limma” o “l” se è stato utilizzato l’approccio bayesiano per
l’elaborazione. A questo proposito si è scelto di dichiarare un gene differenzialmente espresso quando la sua statistica B è maggiore di zero.
“ADS” se si riferisce ad un’elaborazione con analisi della
varianza in cui il modello per i dati è stato formulato come somma degli effetti “Array+Dye+Sample”. Nella formula del modello non sono riportati esplicitamente i termini VG e G, dal momento che qualunque applicazione dell’ANOVA nell’analisi dei microarray è imprescindibile da questi.
“ADSS” se si riferisce ad un’elaborazione con analisi della
varianza in cui il modello per i dati è stato formulato come somma degli effetti “Array+Dye+Sample+Spot”;
“sam” accompagnato da un valore in percentuale, se è stato
utilizzato il metodo di analisi della significatività statistica e la selezione dei geni differenzialmente espressi è avvenuta fissando il parametro FDR al valore indicato dalla percentuale. A questo proposito è stato deciso di utilizzare il valore 20% come soglia per questo parametro, così come suggerito dalla letteratura.
6.7.1 Sessione I di prove
Lo scopo di questa sessione di prove è stato realizzare un confronto fra le tre tecniche di elaborazione statistica a parità di metodo di normalizzazione. I dati utilizzati non hanno subito il processo di sottrazione del background e di selezione in base al rapporto segnale/rumore.
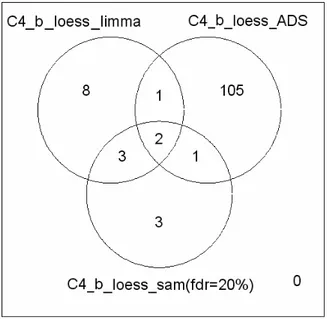
La figura 6.2 mostra per il contrasto C4age un diagramma di Venn delle intersezioni dei tre insiemi di geni differenzialmente espressi identificati con i tre metodi. Si evidenzia come l’approccio empirico bayesiano sia il più selettivo, cioè quello che individua l’insieme meno numeroso di geni.
Figura 6.2: Diagramma di Venn del contrasto C4age nella sessione I di prove
Fra i 40 geni individuati dall’approccio bayesiano sono compresi i 22 geni individuati dallo studio di Golden, che sono anche quelli in comune fra tutti e tre i metodi.
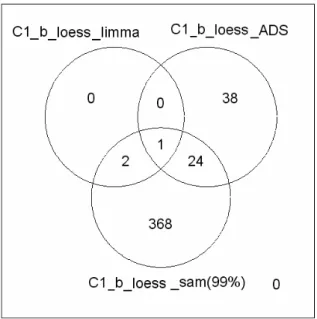
L’approccio bayesiano dimostra buone capacità di selezione anche quando i geni differenzialmente espressi rilevati sono pochi, come per il
differenzialmente espressi e l’approccio bayesiano ne identifica 3, di cui i due con statistica B più alta (>5) sono proprio quelli evidenziati nello studio, mentre solo quello con statistica B maggiore è in comune fra i tre metodi.
Figura 6.3: Diagramma di Venn del contrasto C1 nella sessione I di prove
Come si può osservare dal diagramma di Venn la differenza più evidente fra i tre metodi è che, mentre l’approccio bayesiano identifica i due geni differenzialmente espressi in un insieme di soli tre elementi, il metodo di analisi della varianza trova un solo gene dei due in un insieme di 63 elementi e l’analisi della significatività statistica, anche se identifica entrambi i geni, li trova in un insieme di 395 geni con un FDR del 99%, considerato pressochè inattendibile dal SAM.
In questa sessione di prove è stato anche osservato il comportamento del metodo di analisi della varianza al variare del modello utilizzato per interpolare i dati.
Le figure 6.4 e 6.5 mostrano due diagrammi di Venn nei quali gli insiemi identificati con il metodo ANOVA utilizzano i modelli
Figura 6.4: Diagramma di Venn del contrasto C4 con modello ADS nella sessione I di
prove
Aggiungere un effetto nel modello computato, qualora siano disponibili i gradi di libertà per stimarlo ed esso sia ortogonale agli altri, consente di migliorare la qualità dell’informazione estraibile; nelle nostre prove a tale miglioramento ha sempre fatto seguito un ampliamento dell’intersezione relativa ai geni differenzialmente espressi.
Figura 6.5: Diagramma di Venn del contrasto C4 con modello ADSS nella sessione I di
Questo ampliamento può essere giustificato dal fatto che, interpolare un modello più dettagliato, sempre che si abbiano le informazioni sufficienti per farlo, consente di poter isolare meglio l’effetto di interesse aumentando la sensibilità del metodo, ma ha come conseguenza l’inclusione di geni non considerati, in precedenza, come differenzialmente espressi.
6.7.2 Sessione II di prove
In questa sessione sono stati realizzati i confronti fra le tre tecniche di elaborazione utilizzando dati che hanno subito il processo di sottrazione del background e la selezione degli spot in base ad un adeguato livello del rapporto segnale/rumore e mantenendo come metodo per la normalizzazione l’interpolazione LOESS.
Applicando queste selezioni, l’insieme di dati disponibile per l’elaborazione si riduce di circa 1/3, cioè geni che precedentemente venivano inclusi nella normalizzazione e, quindi, nella valutazione dell’espressione differenziale, vengono ora scartati perchè giudicati non idonei.
La riduzione dell’insieme di dati ha un effetto negativo sull’elaborazione realizzata con il metodo dell’analisi della varianza: a causa dell’insufficiente quantità di informazioni il metodo non è in grado di interpolare i modelli proposti sui dati a disposizione e, quindi, non è possibile valutare l’effetto di interesse.
L’approccio empirico bayesiano non sembra essere particolarmente sensibile al processo di selezione del dato operato in questa sessione di prove, come è possibile osservare nell’esempio di figura 6.6.
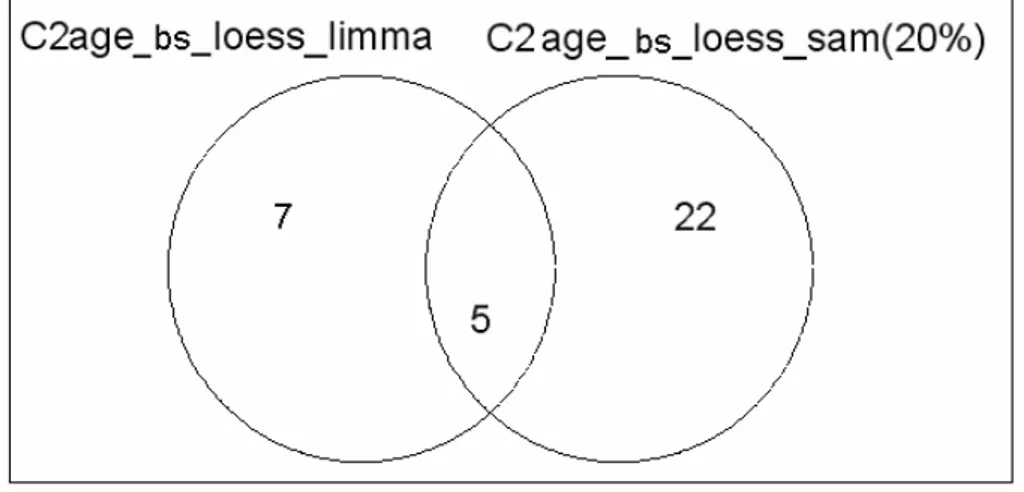
Figura 6.6: Diagramma di Venn del contrasto C2 a parità di approccio di
elaborazione e di normalizzazione sui dati della sessione I e II.
Il metodo di analisi della significatività statistica mostra, invece, un miglioramento, sia rispetto al numero di geni potenzialmente espressi in maniera significativa per un FDR=20%, sia rispetto alla quantità di geni in comune ai due metodi (figure 6.7 e 6.8).
In generale, dalle prove relative a questa sessione si può osservare un abbassamento del valore di FDR per ottenere insiemi di numerosità comparabile fra i due metodi, cioè l’analisi della significatività statistica sembra acquisire maggiore sensibilità quando i dati vengono selezionati con il procedimento descritto.
Figura 6.7:Diagramma di Venn del contrasto C2age per i dati della sessione II di prove
6.7.3 Sessione III di prove
Questa sessione è stata realizzata con dati che hanno subito il processo di sottrazione del background e la selezione degli spot in base ad un adeguato livello del rapporto segnale/rumore e per i quali è stato adottato un metodo di normalizzazione “print-tip”.
Con questo tipo di normalizzazione gli insiemi di geni differenzialmente espressi tendono ad essere meno numerosi rispetto a quelli trovati con gli stessi contrasti nella sessione II di prove.
Come è possibile osservare nell’esempio di figura 6.9, per il contrasto C4age a parità di metodo di elaborazione si selezionano 40 geni differenzialmente espressi nella sessione I, 37 nella sessione II e 23 nella sessione III.
Questo risultato può essere dovuto ad un intervento più “pesante” del processo di normalizzazione sulla distribuzione dei dati ed invita ad essere cauti nell’uso di una normalizzazione “print-tip”.
Figura 6.9: Diagramma di Venn del contrasto C4age a parità di metodo di elaborazione
per le tre sessioni di prova
6.8 Individuazione di una metodica incrociata per l’analisi
statistica dei dati di espressione genica
Dai risultati delle tre sessioni di prova realizzate è possibile individuare una metodica da utilizzare per l’analisi statistica dei dati di espressione genica.
E’ evidente che la procedura proposta necessita di essere adattata sia alla qualità delle informazioni che si hanno a disposizione che alla loro quantità. Tuttavia, la criticità dell’insieme di dati sul quale sono state testate le capacità elaborative dei tre metodi, dovuta alla bassa numerosità delle osservazioni a disposizione, ha consentito di evidenziare i loro pregi, ma anche i limiti.
L’approccio bayesiano di stima dei parametri ha dimostrato di poter gestire in maniera efficiente tutti i contrasti impostati per l’analisi dei dati
Questo metodo, che si realizza operativamente grazie all’interpolazione sui dati di modelli lineari o di polinomi di grado superiore al primo, dimostra una buona robustezza nell’adattarsi all’insieme di dati, anche se piccoli. I risultati prodotti concordano, almeno per il 50%, con quelli presentati dallo studio di invecchiamento.
E’ possibile, quindi, individuare nell’approccio bayesiano di stima dei parametri il metodo da utilizzare in prima istanza nell’analisi dei dati.
Se l’interesse dell’analista è quello di poter esprimere una maggiore confidenza nel risultato ottenuto, è necessario procedere con una validazione informatica incrociata dei risultati ottenuti con il metodo giudicato più affidabile.
La scelta dei metodi da utilizzare per questo scopo può dipendere da diversi elementi.
Un insieme di osservazioni troppo limitato non consente di utilizzare il metodo ANOVA per evidenziare il contributo delle sorgenti di variabilità che si vorrebbero quantificare.
Da questo punto di vista è importante sottolineare che, avere a disposizione i gradi di libertà sufficienti per valutare tutti gli effetti presenti nel modello utilizzato per interpolare i dati, non significa che tali effetti possono essere sempre quantificati e, se necessario, eliminati.
Infatti, nella prima sessione di prove, malgrado i gradi di libertà fossero sufficienti per la valutazione dell’effetto DG, non è stato possibile quantificarlo perché è risultato non ortogonale agli altri effetti.
Il metodo ANOVA non può, quindi, essere sempre utilizzato, ma il suo apporto, quando possibile, contribuisce ad aumentare il livello di sicurezza dei risultati.
Il metodo di analisi della significatività statistica non mostra sempre una grande capacità di produrre autonomamente risultati affidabili; ciò è stato messo in evidenza dall’elevato valore della percentuale di FDR necessaria per trovare, nell’insieme di geni potenzialmente espressi in maniera significativa, tutti i geni differenzialmente espressi identificati dall’approccio bayesiano (figura 6.3).
Nonostante questo limite evidente, utilizzare i risultati ottenuti con questo metodo in un confronto incrociato con gli altri, concorre ad una migliore definizione dell’insieme intersezione.
Per concludere, quindi, utilizzare una metodica incrociata di analisi statistica dei dati, come quella illustrata in questa tesi, consente di ottenere una maggior robustezza nel processo di elaborazione e una più alta affidabilità del risultato. Questo permette di avere a disposizione un insieme maggiormente attendibile di risultati dal quale partire per la successiva validazione dei dati con procedure biologiche alternative quali il Northern blot o la Real-Time PCR.