53 3. MATERIALI E METODI
3.1 Ceppi batterici
I ceppi di Staphylococcus epidermidis utilizzati nel presente lavoro di tesi sono stati isolati presso l’Unità Operativa (U.O.) di Microbiologia Universitaria dell’Azienda Ospedaliera Universitaria Pisana da campioni clinici pervenuti per la ricerca di germi comuni, da vari reparti del medesimo nosocomio. Tali ceppi sono stati isolati, secondo procedure standard, su comuni terreni di coltura, quali agar sangue ed agar sale-mannite. L’identificazione dei ceppi batterici isolati è stata effettuata con metodi semiautomatici utilizzando il sistema Vitek II (BioMerieux), seguendo fedelmente la procedura consigliata dalla ditta produttrice. In parallelo, è stato possibile anche effettuare l’antibiogramma di tutti i ceppi batterici identificati, cioè il profilo di suscettibilità/resistenza nei confronti di farmaci antibatterici comunemente impiegati nei protocolli terapeutici, utilizzando “card” apposite sempre del sistema Vitek II. Oltre agli isolati clinici di S. epidermidis, è stato incluso nello studio, come ceppo di controllo, il ceppo di riferimento American Type Culture Collection (ATCC) 35984 (RP62A) per la sua spiccata capacità di produrre biofilm in vitro (Gotz, 2002). I ceppi venivano considerati multi-farmaco resistenti (MDR) se presentavano una resistenza ad almeno tre distinte classi di antibiotici, con differente meccanismo d’azione.
3.2 Coltivazione e conservazione dei ceppi batterici utilizzati
Le colture liquide dei diversi ceppi di S. epidermidis erano allestite in terreno Triptone Soya Broth (TSB) (Oxoid, Basingstoke, United Kingdom) addizionato dello 0.25% di glucosio (Merck, Germany) (TSB/Glc). Per la conta delle unità formanti colonia (CFU) veniva utilizzato, invece, il terreno di coltura solido Triptone Soya Agar (TSA) (Oxoid, Basingstoke, United Kingdom). Per la preparazione degli inoculi standard, i ceppi batterici venivano fatti crescere in terreno di coltura liquido fino ad una densità ottica (OD600nm) di circa 1. Le colture venivano quindi suddivise in aliquote da 0.2 ml e conservate a –80 °C fino al momento dell’uso.
3.3 Epcidina 20 e vancomicina
L’Epcidina 20 (hep20) è stata sintetizzata dalla ditta “Peptide Specialty laboratories GmBH” (Heidelberg, Germany). L’analisi del peptide sintetico tramite “reverse-phase
54
high performance chromatography” (RP-HPLC) e spettrofotometria di massa ha rivelato una purezza maggiore del 98%. Il peptide veniva diluito in acqua milli-Q sterile per avere una concentrazione finale di 5M. La vancomicina idrocloride è stata ottenuta dalla ditta Sigma-Aldrich (St. Louis, USA) e diluita in acqua milli-Q per avere una soluzione stock di 5mM. Le soluzioni di hep20 e vancomicina venivano divise in aliquote e poste in congelatore a –80°C fino al momento dell’uso.
3.4 Saggio di formazione del biofilm
I ceppi batterici oggetto del presente studio venivano saggiati per la loro capacità di aderire e di produrre biofilm in vitro in pozzetti di piastre di polistirene da 96 sterili, con fondo piatto. A tale scopo, i batteri venivano inoculati in un volume fisso di TSB/Glc e lasciati crescere overnight a 37°C in agitazione, fino al raggiungimento della fase stazionaria di crescita. Ogni inoculo batterico veniva diluito 1:100 in terreno fresco costituito da TSB/Glc diluito a sua volta 1:1 con SPB (tampone sodio fosfato) a diversi valori di pH (7.4, 6.6, 5.8, 5). Da tale sospensione erano prelevati 90 µl che venivano trasferiti nei singoli pozzetti insieme a 10 l di SPB al pH corrispondente. Seguiva un’incubazione in condizioni stazionarie per 24 h a 37°C.
3.5 Valutazione della biomassa totale del biofilm mediante colorazione con Cristal Violetto (CV)
La capacità adesiva dei ceppi in esame alla superficie dei pozzetti e la quantità di biofilm prodotta venivano valutate mediante misurazione dell’assorbanza (OD) a 570 nm dopo colorazione con CV. Nel dettaglio, al termine del periodo di incubazione, veniva allontanato il sovranatante di ogni pozzetto e si procedeva a 3 lavaggi con 200l di PBS (Phosphate-Buffered Saline) a pH 7.4 con l’intento di eliminare tutti i batteri non adesi alla superficie. Ogni residuo di PBS veniva eliminato tamponando la piastra su carta assorbente e le cellule adese venivano fissate lasciando asciugare la piastra all’aria. Successivamente ad ogni pozzetto si aggiungeva un volume di 125 l di CV allo 0,1% (BioMerieux, Italia) e le piastre venivano incubate a temperatura ambiente per 15 min. Il colorante in eccesso veniva poi eliminato mediante 3 lavaggi con 200 l di PBS, le piastre venivano tamponate su carta assorbente e lasciate asciugare all’aria. Il CV rimasto adeso al biofilm a seguito dei lavaggi veniva, quindi, estratto con 200 l di etanolo (Sigma-Aldrich, St. Louis USA) al 98%. Dopo 15 minuti d’incubazione a
55
temperatura ambiente si trasferiva l’etanolo un nuovo pozzetto e si misurava la OD570. In ogni saggio venivano inseriti dei controlli negativi rappresentati da terreno privo di batteri. Tutti i saggi venivano effettuati in triplicato e veniva calcolata la media dei valori ottenuti e l’errore standard della media (SEM).
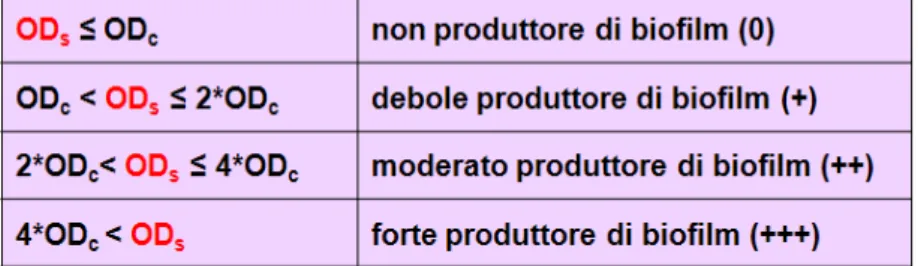
3.6 Classificazione dei ceppi batterici in base alla capacità di formare biofilm Dai valori delle OD570 ottenute, è stato possibile classificare i ceppi batterici in base alla quantità di biofilm prodotto seguendo una procedura riportata in letteratura (Stepanovic et al., 2000). Tale classificazione è basata sul confronto dei valori medi delle densità ottiche misurate per ogni ceppo batterico (ODS) con un valore di densità ottica di riferimento, definito cut-off (ODC), ottenuto come media delle OD dei controlli negativi sommata a 3 deviazioni standard. In base all’ODC si individuano 4 intervalli di classificazione (Tabella 3).
3.7 Saggio su Congo Red Agar (CRA)
I ceppi di S. epidermidis venivano valutati riguardo alla loro capacità di produrre polisaccaridi extracellulari mediante saggio su agar Congo Red (CRA) seguendo il protocollo di Freeman e collaboratori (Freeman et al., 1989) con alcune modifiche. Il CRA era composto da Muller Hinton Broth (Oxoid Ltd, England) 21 g/L, agar batteriologico (Oxoid Ltd, England) 1 %, glucosio 50 g/L e Congo Red (Sigma Aldrich, St. Louis, USA) 0.8 g/L. Il Congo Red, disciolto in acqua e sterilizzato a parte in autoclave, veniva aggiunto al terreno al momento della distribuzione nelle piastre. I ceppi batterici venivano seminati su CRA dopo crescita overnight in terreno TSB/Glc.
Tabella 3. Intervalli di classificazione di ceppi batterici in base alla produzione di biofilm. ODS: media delle O.D. del campione. ODC: OD cut-off (Stepanovic et al.,
56
Le piastre venivano così incubate per 24 ore a 37°C e per un ulteriore periodo di 24 ore a temperatura ambiente.
L’analisi della colorazione delle colonie e la loro morfologia permetteva di discriminare colonie cristalline con tonalità dal nero al grigio, tipiche dei ceppi batterici in grado di produrre polisaccaridi di matrice, da quelle rosse-bordeaux di consistenza cremosa tipiche di ceppi non produttori di polisaccaridi extracellulari.
3.8 Saggio di suscettibilità dei biofilm alle proteasi
Biofilm pre-formati di ogni ceppo, allestiti seguendo il protocollo illustrato nel paragrafo 3.4, venivano sottoposti al trattamento con proteasi. Prima di procedere al trattamento, ogni biofilm veniva lavato con 200 µl di una soluzione di NaCl (Sigma-Aldrich, St. Louis, USA) allo 0,9% e successivamente trattato per 2 h a 37°C con 50 µl di Proteinasi K (Sigma-Aldrich, St. Louis, USA) alla concentrazione di 1 mg/ml in Tris 20 mM. I pozzetti di controllo erano trattati con il solo tampone, senza enzima. Dopo il trattamento, ogni biofilm veniva lavato con 200 µl di PBS, fatto asciugare all’aria e colorato con Cristal Violetto (vedi paragrafo 3.5). Si osservavano quindi gli eventuali effetti del trattamento rispetto al controllo non trattato.
3.9 Estrazione del DNA genomico
Il DNA genomico dei quattro ceppi di S. epidermidis veniva estratto da batteri seminati su piastre di TSA e incubati a 37°C overnight. Dalle piastre, per ogni ceppo, venivano prelevate 5/6 colonie che venivano risospese in 50 µl di buffer TE (Tris-EDTA) e sottoposte a lisi a 100°C per 15 min. In seguito, ogni campione veniva centrifugato a 20000 xg per 60s a 4°C ed il sovranatante veniva recuperato in una nuova eppendorf; 5 µl del DNA estratto venivano utilizzati nelle successive reazioni di PCR.
3.10 Ricerca dei geni del complesso ica e dell’elemento trasponibile IS256 mediante reazione polimerasica a catena (PCR)
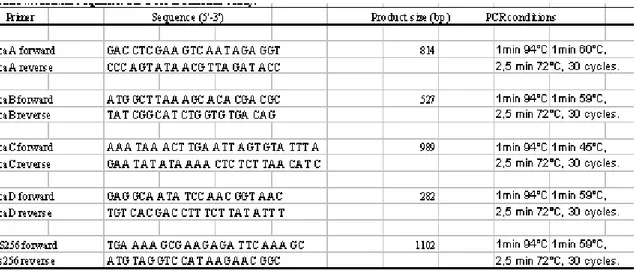
Per ogni ceppo batterico venivano allestite delle PCR per valutare la presenza dei geni A, B, C, D che compongono l’operone ica e quella dell’elemento trasponibile IS256. Le reazioni venivano allestite usando cinque coppie di primer, specifiche per ogni gene da amplificare, la cui sequenza è stata disegnata in base alle informazioni ottenute dal
57
database di sequenze GenBank dell’NCBI (National Center for Biotechnology Information).
Ogni reazione di PCR veniva condotta in 50 µl totali, costituiti da 45 µl di mix e 5 µl di DNA o 5 µl di acqua per i controlli negativi.
Le mix erano costituite da:
Conc.iniziale Conc. finale 1x (50l)
Buffer 10x 5 l
MgCl2 50 mM 1.5 mM 1.5 l
dNTPs 5 mM 0.2 mM 2 l
Taq polimerasi 5 U/l 2.5 U 0.25 l
Primer For 20 pmoli/l 0.3 pmoli/l 0.75 l Primer Rev 20 pmoli/l 0.3 pmoli/l 0.75 l
H2O 34.75 l
Le condizioni generali di amplificazione erano di 30 cicli di reazione così costituiti: 1 min per la denaturazione a 94°C;
1 min di annealing : i) 60°C per l’amplificazione del gene IcaA; ii) 59°C per IcaB, IcaD, IS256;
iii) 45°C per IcaC; 2,5 min di elongazione a 72°C;
alla fine, seguiva un’ulteriore fase di elongazione a 72°C per 10 min.
Al termine dell’amplificazione 10 µl di ogni prodotto venivano usati per l’analisi della mobilità in elettroforesi su gel d’agarosio al 2% in tampone TAE (Tris Acetate Electrophoresis Buffer). Come controllo dei pesi molecolari durante la corsa elettroforetica veniva utilizzato un DNA-marker di 100 pb. Durante ogni reazione di PCR venivano inseriti dei controlli negativi di amplificazione in cui il DNA del campione veniva sostituito con 5µl di acqua sterile e parallelamente un controllo positivo che era costituito da 5µl di DNA del ceppo di riferimento S. epidermidis ATCC 35984, positivo per la presenza dei geni ica e IS256. La sequenza dei primers utilizzati e le condizioni di amplificazione sono riportate in Tabella 4.
58
3.11 Determinazione della Minima Concentrazione Inibente (MIC) di hep20 e di vancomicina verso cellule batteriche in fase stazionaria di crescita
Al fine di valutare la MIC di hep20 e vancomicina verso cellule batteriche in condizioni fisiologiche assimilabili a quelle delle cellule in biofilm, si procedeva ad allestire le colture batteriche analogamente a quanto riportato al paragrafo 3.4. In questo caso, però, i 90 µl prelevati dalla sospensione batterica venivano trasferiti in eppendorf insieme a 10 µl di peptide per 24 ore a 37°C, in condizioni statiche. Le concentrazioni di hep20 e vancomicina utilizzate nel saggio erano comprese tra 3.12 µM e 100 µM per hep20 e da 0,5 a 64 g/ml per la vancomicina. La MIC era la più bassa concentrazione di peptide/antibiotico a cui non si osservava crescita batterica, valutata come intorbidimento del mezzo di coltura.
3.12 Saggio di inibizione della formazione di biofilm da parte di hep20
Un primo set di esperimenti è stato effettuato per stabilire il pH ottimale del mezzo di coltura da utilizzare per i successivi saggi di inibizione del biofilm. A tale scopo un ceppo di S. epidermidis è stato arbitrariamente scelto tra quelli oggetto dello studio, coltivato overnight in TSB/Glc a 37°C in agitazione e successivamente diluito 1:100 in terreno fresco rappresentato da TSB/Glc diluito al 50% con tampone SPB a pH 7.4, 6.6, 5.8 e 5. Di routine, i saggi di inibizione del biofilm sono stati effettuati a pH 6.6. A seguito della diluizione dell’inoculo, 90 µl della sospensione batterica (circa 107 CFU) e
Tabella 4. In tabella per ogni gene sono riportate le sequenze dei primer, le dimensioni attese dei frammenti di amplificazione e le condizioni di amplificazione.
59
10 µl di peptide a diverse concentrazioni venivano distribuiti in pozzetti di piastre da 96. I controlli positivi erano costituiti da 90 µl di sospensione batterica e 10 µl di SPB al pH corrispondente. Seguiva una incubazione delle piastre per vari interavlli di tempo in condizioni statiche a 37°C. Si procedeva quindi alla valutazione della biomassa totale del biofilm tramite colorazione con CV (vedi paragrafo 3.5), dell’attività metabolica cellulare tramite colorazione con alamar blu (vedi paragrafo 3.13) e del numero di cellule vitali associate al biofilm (vedi paragrafo 3.14).
3.13 Valutazione dell’attività metabolica delle cellule associate al biofilm mediante saggio con Alamar Blue
L’alamar blue (AB) (Invitrogen, Life technologies, Paisley UK), nello stato ossidato, è un composto con una colorazione blu; in presenza di cellule metabolicamente attive viene ridotto a resofurina, un composto di colorazione rosa. Venivano allestiti biofilm di ceppi rappresentativi di S. epidermidis in presenza di diverse concentrazioni di hep20, od in assenza di peptide come precedentemente descritto. Dopo incubazione, ogni piastra veniva lavata tre volte con PBS e ad ogni pozzetto venivano aggiunti 100 l di terreno fresco (TSB/Glc) e 10 l di AB. Le piastre venivano incubate a 37°C in blanda agitazione per vari tempi (da 15 min a 2h) prima di valutare l’assorbanza a 570 nm.
3.14 Determinazione del numero di batteri vitali associati al biofilm
Per valutare il numero di Unità Formanti Colonia (CFU) associate al biofilm, al termine del trattamento con hep20 si effettuavano 3 lavaggi con 200 µl di PBS sterile per pozzetto con l’intento di allontanare le cellule planctoniche presenti nel mezzo di coltura. Le cellule adese venivano distaccate meccanicamente con la punta di una micropipetta in 100 µl di PBS, sottoposte a blanda sonicazione e seminate su piastre di TSA in seguito a diluizioni scalari in PBS. Dopo incubazione a 37°C per 24-48h si effettuava la conta delle CFU.
3.15 Microscopia confocale ed analisi delle immagini
Biofilm di due ceppi rappresentativi di S. epdermidis, trattati o meno con hep20, sono stati analizzati al microscopio confocale. A tale scopo, 300 l delle sospensioni batteriche diluite, ottenute come riportato nel paragrafo 3.4, venivano distribuite in piastre da 8 pozzetti µ-Slide (Ibidi) in presenza di 6.25, 12.5 and 25 hep20 o in
60
assenza del peptide ed incubati a 37°C in condizioni statiche. Dopo 18h i biofilm venivano lavati con 300 l di SPB per tre volte. In pozzetti di piastre separate, si procedeva quindi a colorare le proteine e l’adesina intercellulare polisaccaridica (PIA) associate al biofilm mediante incubazione, rispettivamente, con 300 l di FilmTracer™ SYPRO® Ruby biofilm matrix (Molecular Probes, Life technologies) o di SPB contenente 90 g/ml di Oregon Green® 488 WGA (Molecular Probes, Life technologies) per 30 o 15 minuti, rispettivamente, al buio. La vitalità dei batteri associati al biofilm dopo trattamento con hep20 veniva determinata colorando le cellule con il “LIVE/DEAD BacLight Bacterial Viability Kit” (Molecular Probes, Life technologies) in accordo con le linee guida della casa produttrice. Dopo colorazione, le piastre venivano lavate con 300 l di SPB per tre volte, prima dell’acquisizione delle immagini. I biofilm marcati venivano esaminati con un microscopio Leica CLSM (TCS SP5, Leica, Heidelberg, Germany) usando un obbiettivo 63x ad immersione ad acqua. Il SYPRO Ruby e l’Oregon Green® 488 venivano eccitati alla lunghezza d’onda di 488 nm, ed i loro spettri di emissione venivano monitorati a 600-700 nm e 500-600 nm, rispettivamente. La lunghezza d’onda di eccitazione/emissione utilizzata per rivelare le cellule vive/morte associate al biofilm era di 488/500-540 nm per il Syto9 e di 561/570-630 nm per il propidium iodide.
Sono stati effettuati due esperimenti indipendenti in cui sono state collezionate dieci immagini (512x512 pixel) per ogni campione in posizioni selezionate casualmente. I dati acquisiti venivano processati ed analizzati con il software ImageJ versione 1.37 (NIH Image; http://rsbweb.nih.gov/ij/). Per ogni immagine veniva selezionata una regione di interesse (ROI) di 512x512 pixel per il calcolo della media di intensità di fluorescenza di SYPRO Ruby o di Oregon Green-488. Una ROI selezionata al di fuori del biofilm, veniva usata per calcolare (e quindi sottrarre) la media del segnale di background. I valori di fluorescenza media e la SEM venivano espressi in unità arbitrarie.
3.16 Valutazione del Post antimicrobial effect (PAE)
La durata dell’attività inibitoria di hep20 dopo l’allontanamento del peptide (PAE) è stata valutata verso un ceppo rappresentativo di S. epidermidis facendo aderire le cellule batteriche alle piastre di polistirene da 96 pozzetti per 3h a 37 °C in presenza di hep20 (25 and 50 M). I pozzetti delle piastre sono stati, quindi, lavati per 3 volte con PBS per
61
rimuovere il peptide e le cellule batteriche non adese; ad ogni pozzetto veniva aggiunto, infine, terreno fresco (50% TSB/Glc pH 6.6). A 3, 6, e 18 h dalla rimozione di hep20, per ogni pozzetto venivano determinate la biomassa del biofilm, l’attività metabolica cellulare ed il numero di cellule vitali, come precedentemente descritto.
3.17 Attività di vancomicina su biofilm pre-trattati con hep20
Si allestivano biofilm di due ceppi rappresentativi di S. epidermidis in presenza di hep20 alla concentrazione di 50 M per 18 h a 37°C. Dopo incubazione le piastre venivano lavate per 3 volte con PBS per rimuovere il peptide e le cellule batteriche non adese. I biofilm pre-trattati con hep20 venivano esposti a concentrazioni sub-battericide di vancomicina (32 g/ml) per ulteriori 24 h a 37°C. I controlli includevano: biofilm incubati con hep20 per 18 h e successivamente in solo terreno per ulteriori 24 h; biofilm incubati in solo terreno per 18 h e addizionati di vancomicina (32 g/ml) per le successive 24 h; biofilm incubati in terreno per 18 h e addizionati di terreno fresco per altre 24 h. Al termine del periodo di incubazione veniva valutato il numero delle CFU associate ai biofilm sottoposti ai differenti trattamenti.
3.18 Analisi statistica
La differenza tra i valori delle medie dei gruppi sperimentali veniva valutata mediante analisi della varianza ad una via (ANOVA) seguita dal test di comparazione multipla di Tukey-Kramer. Le differenze venivano considerate statisticamente significative a valori di p<0.05. L’analisi di correlazione (r) veniva effettuata utilizzando il test di correlazione di Pearson.