APPENDICE
TAVOLE RELATIVE AL PRIMO APPROCCIO SPERIMENTALE: A1-A17.
Legenda
ABBREVIAZIONE
COMUNITÀ AUTONOMA DI
PROVENIENZA DEL CAMPIONE
Andal-Ext Andalusia e Extremadura
Arag-LaRi Aragona e La Rioja
Ast -Gal Asturie e Galizia
Catal-Bale Catalogna e Isole Baleari
Murc-Vale Murcia e Valenzia
Canaria__ Isole Canarie
Casti-Man Castiglia La Mancia e Madrid
Canta-Leo Cantabria e Castiglia-León
EQUILIBRIO H-W EQUILIBRIO H-W
D8S566 D8S591
Pop P-val Pop P-val
Andal-Ext 0,1513 Andal-Ext 0,0254 Arag-LaRi 0,5824 Arag-LaRi 0,622 Ast-Gal 0,6254 Ast-Gal 0,5844 Catal-Bale 0,2313 Catal-Bale 0,027 Murc-Vale 0,794 Murc-Vale 0,1033 Canaria__ 0,2539 Canaria__ 1 Casti-Man 0,469 Casti-Man 0,2706 Canta-Leo 0,358 Canta-Leo 0,4985 Navar-Bas 0,5513 Navar-Bas 0,1148 EQUILIBRIO H-W EQUILIBRIO H-W D4S111 D1S80
Pop P-val Pop P-val
Andal-Ext 0,3302 Andal-Ext 0,1693 Arag-LaRi 0,6810 Arag-LaRi 0,0709 Ast-Gal 0,0659 Ast-Gal 0,3799 Catal-Bale 0,0390 Catal-Bale 0,0087 Murc-Vale 0,3224 Murc-Vale 0,0727 Canaria__ 1 Canaria__ 0,1293 Casti-Man 0,0200 Casti-Man 0,0007 Canta-Leo 0,0420 Canta-Leo 0,0975 Navar-Bas 0,8899 Navar-Bas 0,3287 EQUILIBRIO H-W D17S5 Pop P-val Andal-Ext 0.0000 Arag-LaRi 0.0000 Ast-Gal 0.0000 Catal-Bale 0.0000 Murc-Vale 0.0031 Canaria__ 0.0000 Casti-Man 0.0000
FREQUENZE GENOTIPICHE LOCUS D1S80 Popolazioni Geno tipi Andal-Ext Arag-LaRi Ast-Gal Catal-Bale
Murc-Vale Canaria Casti-Man
Canta-Leo Navar-Bas 15-15 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0000 15-18 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 15-22 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 17-18 0,0244 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 17-21 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0204 0,0000 0,0000 17-24 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0313 0,0000 18-18 0,0000 0,1538 0,0435 0,1071 0,2105 0,1250 0,0816 0,1250 0,0833 18-19 0,0000 0,0000 0,0000 0,0000 0,0000 0,1250 0,0000 0,0000 0,0000 18-20 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0204 0,0000 0,0000 18-21 0,0000 0,0000 0,0000 0,0000 0,0526 0,0000 0,0000 0,0313 0,0000 18-22 0,0732 0,0000 0,0000 0,0357 0,0000 0,0000 0,0408 0,0938 0,0000 18-24 0,3659 0,2308 0,1739 0,0357 0,0526 0,0000 0,1429 0,1250 0,1667 18-25 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0278 18-26 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 18-28 0,0244 0,0000 0,0435 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 18-29 0,0000 0,0000 0,0435 0,0000 0,0000 0,0000 0,0204 0,0313 0,0000 18-30 0,0000 0,0385 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 18-31 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0612 0,0313 0,0556 18-32 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0556 19-19 0,0244 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0000 19-21 0,0000 0,0000 0,0000 0,0000 0,0000 0,1250 0,0000 0,0000 0,0000 19-25 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0278 19-26 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0204 0,0000 0,0000 19-29 0,0000 0,0000 0,0435 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 19-30 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0204 0,0313 0,0000 19-33 0,0000 0,0385 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 20-22 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0278 20-24 0,0244 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0625 0,0556 20-25 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0278 20-26 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0278 20-28 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0313 0,0000 20-30 0,0000 0,0385 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 20-31 0,0000 0,0000 0,0435 0,0000 0,0000 0,0000 0,0204 0,0000 0,0278 21-21 0,0000 0,0000 0,0435 0,0000 0,0000 0,2500 0,0000 0,0000 0,0000 21-22 0,0000 0,0000 0,0000 0,0000 0,0526 0,0000 0,0000 0,0000 0,0000 21-24 0,0244 0,0000 0,0435 0,0714 0,0000 0,0000 0,0204 0,0000 0,0278 21-25 0,0000 0,0385 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 21-28 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0000 21-29 0,0000 0,0000 0,0000 0,0000 0,0000 0,1250 0,0000 0,0000 0,0000 21-30 0,0000 0,0000 0,0000 0,0000 0,0526 0,0000 0,0000 0,0000 0,0000
FREQUENZE GENOTIPICHE LOCUS D1S80 Popolazioni Geno tipi Andal-Ext Arag-LaRi Ast-Gal Catal-Bale
Murc-Vale Canaria Casti-Man
Canta-Leo Navar-Bas 23-25 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0278 23-34 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0204 0,0000 0,0000 24-24 0,1220 0,1154 0,1739 0,1429 0,0000 0,1250 0,1224 0,1563 0,0833 24-25 0,0000 0,0769 0,0870 0,0000 0,0526 0,0000 0,1020 0,0313 0,0278 24-26 0,0000 0,0385 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 24-27 0,0244 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0278 24-28 0,0488 0,0000 0,0435 0,0714 0,0000 0,0000 0,0000 0,0000 0,0000 24-29 0,0244 0,0769 0,0435 0,0000 0,0000 0,0000 0,0000 0,0313 0,0833 24-30 0,0488 0,0000 0,0000 0,0000 0,0000 0,1250 0,0000 0,0000 0,0000 24-31 0,0244 0,0385 0,0000 0,0357 0,0000 0,0000 0,0612 0,0313 0,0278 24-32 0,0244 0,0000 0,0435 0,0000 0,0000 0,0000 0,0204 0,0000 0,0278 24-33 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0313 0,0000 24-34 0,0000 0,0000 0,0435 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 24-36 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0000 24-37 0,0244 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0313 0,0000 24-38 0,0000 0,0385 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 25-25 0,0000 0,0000 0,0000 0,0357 0,0526 0,0000 0,0000 0,0000 0,0000 25-30 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0204 0,0000 0,0000 25-31 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0278 25-34 0,0000 0,0000 0,0435 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 26-26 0,0000 0,0000 0,0000 0,0000 0,0526 0,0000 0,0000 0,0313 0,0000 26-32 0,0244 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 2639 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0278 2728 0,0000 0,0000 0,0435 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 27-30 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0000 27-34 0,0000 0,0385 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 28-28 0,0244 0,0000 0,0000 0,0000 0,0526 0,0000 0,0000 0,0000 0,0000 28-29 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0000 28-31 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0313 0,0000 28-35 0,0000 0,0000 0,0000 0,0357 0,0000 0,0000 0,0000 0,0000 0,0000 29-29 0,0000 0,0000 0,0000 0,0000 0,0526 0,0000 0,0000 0,0000 0,0000 29-30 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0408 0,0000 0,0000 29-31 0,0000 0,0385 0,0000 0,0000 0,0000 0,0000 0,0408 0,0000 0,0000 30-30 0,0000 0,0000 0,0000 0,0000 0,1053 0,0000 0,0000 0,0000 0,0000 30-37 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0204 0,0000 0,0000 31-31 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0313 0,0000 34-41 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0278
FRQUENZE ALLELICHE D1S80 15 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 41 Andal-Extr 0,0000 0,0122 0,2439 0,0244 0,0122 0,0122 0,0610 0,0000 0,4634 0,0000 0,0122 0,0122 0,0610 0,0122 0,0244 0,0122 0,0244 0,0000 0,0000 0,0000 0,0000 0,0122 0,0000 0,0000 0,0000 Arag-LaRi 0,0000 0,0000 0,2885 0,0192 0,0192 0,0192 0,0000 0,0000 0,3654 0,0577 0,0192 0,0192 0,0000 0,0577 0,0385 0,0385 0,0000 0,0192 0,0192 0,0000 0,0000 0,0000 0,0192 0,0000 0,0000 Ast-Gal 0,0000 0,0000 0,1739 0,0217 0,0217 0,0652 0,0000 0,0000 0,4130 0,0652 0,0000 0,0217 0,0652 0,0652 0,0000 0,0217 0,0217 0,0000 0,0435 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 Catal-Bale 0,0357 0,0000 0,1607 0,0536 0,0000 0,0536 0,0536 0,0179 0,3036 0,0893 0,0000 0,0179 0,0893 0,0179 0,0536 0,0179 0,0000 0,0000 0,0000 0,0179 0,0179 0,0000 0,0000 0,0000 0,0000 Murc-Vale 0,0000 0,00 00 0,3947 0,0000 0,0000 0,0789 0,0789 0,0000 0,1316 0,0526 0,0263 0,0000 0,0263 0,0526 0,0526 0,0526 0,0000 0,0263 0,0000 0,0000 0,0000 0,0263 0,0000 0,0000 0,0000 Canaria___ 0,0000 0,0000 0,3125 0,1250 0,0000 0,1250 0,0000 0,0000 0,1875 0,0000 0,0000 0,0000 0,0000 0,1875 0,0625 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 0,0000 Casti-Man 0,0000 0,0102 0,2245 0,0204 0,0204 0,0204 0,0714 0,0204 0,3061 0,0612 0,0102 0,0000 0,0000 0,0612 0,0510 0,0918 0,0102 0,0000 0,0102 0,0000 0,0000 0,0102 0,0000 0,0000 0,0000 Canta-Leo 0,0000 0,0156 0,2813 0,0156 0,0469 0,0156 0,0469 0,0000 0,3438 0,0156 0,0313 0,0000 0,0313 0,0313 0,0156 0,0781 0,0000 0,0156 0,0000 0,0000 0,0000 0,0156 0,0000 0,0000 0,0000 Navar-Bas 0,0000 0,0000 0,2361 0,0139 0,0833 0,0139 0,0139 0,0139 0,3056 0,0833 0,0278 0,0139 0,0000 0,0417 0,0000 0,0694 0,0417 0,0000 0,0139 0,0000 0,0000 0,0000 0,0000 0,0139 0,0139 Totale 0,0038 0,0057 0,2462 0,0248 0,0267 0,0324 0,0420 0,0076 0,3340 0,0496 0,0153 0,0095 0,0305 0,0458 0,0305 0,0496 0,0134 0,0057 0,0095 0,0019 0,0019 0,0076 0,0019 0,0019 0,0019
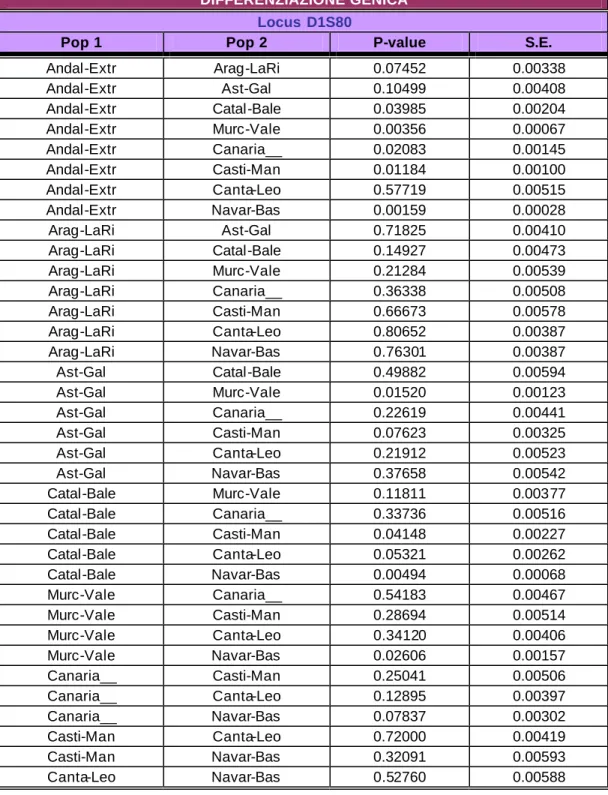
DIFFERENZIAZIONE GENICA Locus D1S80
Pop 1 Pop 2 P-value S.E.
Andal-Extr Arag-LaRi 0.07452 0.00338 Andal-Extr Ast-Gal 0.10499 0.00408 Andal-Extr Catal-Bale 0.03985 0.00204 Andal-Extr Murc-Vale 0.00356 0.00067 Andal-Extr Canaria__ 0.02083 0.00145 Andal-Extr Casti-Man 0.01184 0.00100 Andal-Extr Canta-Leo 0.57719 0.00515 Andal-Extr Navar-Bas 0.00159 0.00028 Arag-LaRi Ast-Gal 0.71825 0.00410 Arag-LaRi Catal-Bale 0.14927 0.00473 Arag-LaRi Murc-Vale 0.21284 0.00539 Arag-LaRi Canaria__ 0.36338 0.00508 Arag-LaRi Casti-Man 0.66673 0.00578 Arag-LaRi Canta-Leo 0.80652 0.00387 Arag-LaRi Navar-Bas 0.76301 0.00387 Ast-Gal Catal-Bale 0.49882 0.00594 Ast-Gal Murc-Vale 0.01520 0.00123 Ast-Gal Canaria__ 0.22619 0.00441 Ast-Gal Casti-Man 0.07623 0.00325 Ast-Gal Canta-Leo 0.21912 0.00523 Ast-Gal Navar-Bas 0.37658 0.00542 Catal-Bale Murc-Vale 0.11811 0.00377 Catal-Bale Canaria__ 0.33736 0.00516 Catal-Bale Casti-Man 0.04148 0.00227 Catal-Bale Canta-Leo 0.05321 0.00262 Catal-Bale Navar-Bas 0.00494 0.00068 Murc-Vale Canaria__ 0.54183 0.00467 Murc-Vale Casti-Man 0.28694 0.00514 Murc-Vale Canta-Leo 0.34120 0.00406 Murc-Vale Navar-Bas 0.02606 0.00157 Canaria__ Casti-Man 0.25041 0.00506 Canaria__ Canta-Leo 0.12895 0.00397 Canaria__ Navar-Bas 0.07837 0.00302 Casti-Man Canta-Leo 0.72000 0.00419 Casti-Man Navar-Bas 0.32091 0.00593 Canta-Leo Navar-Bas 0.52760 0.00588
Tabella A.4.: Approccio storico-politico: differenziazione genica a coppie di popolazioni
calcolata sulle frequenze alleliche del locus D1S80.
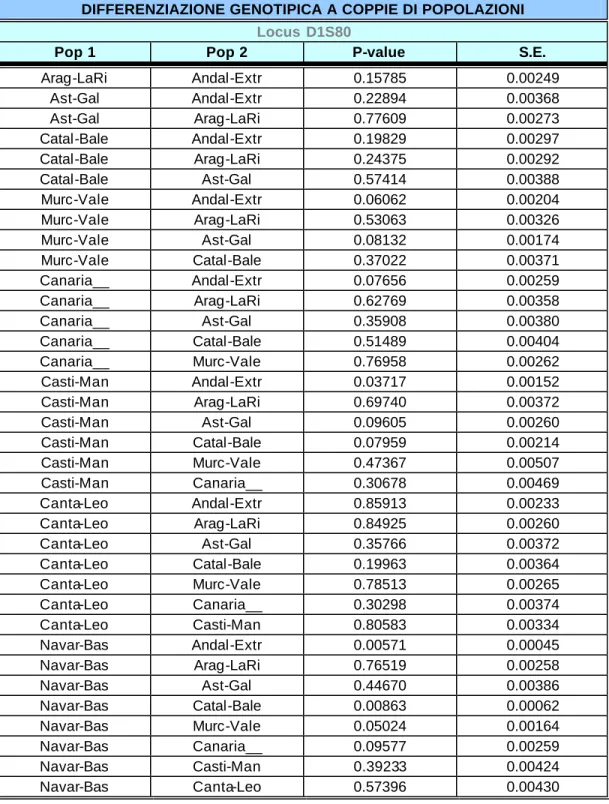
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D1S80
Pop 1 Pop 2 P-value S.E.
Arag-LaRi Andal-Extr 0.15785 0.00249 Ast-Gal Andal-Extr 0.22894 0.00368 Ast-Gal Arag-LaRi 0.77609 0.00273 Catal-Bale Andal-Extr 0.19829 0.00297 Catal-Bale Arag-LaRi 0.24375 0.00292 Catal-Bale Ast-Gal 0.57414 0.00388 Murc-Vale Andal-Extr 0.06062 0.00204 Murc-Vale Arag-LaRi 0.53063 0.00326 Murc-Vale Ast-Gal 0.08132 0.00174 Murc-Vale Catal-Bale 0.37022 0.00371 Canaria__ Andal-Extr 0.07656 0.00259 Canaria__ Arag-LaRi 0.62769 0.00358 Canaria__ Ast-Gal 0.35908 0.00380 Canaria__ Catal-Bale 0.51489 0.00404 Canaria__ Murc-Vale 0.76958 0.00262 Casti-Man Andal-Extr 0.03717 0.00152 Casti-Man Arag-LaRi 0.69740 0.00372 Casti-Man Ast-Gal 0.09605 0.00260 Casti-Man Catal-Bale 0.07959 0.00214 Casti-Man Murc-Vale 0.47367 0.00507 Casti-Man Canaria__ 0.30678 0.00469 Canta-Leo Andal-Extr 0.85913 0.00233 Canta-Leo Arag-LaRi 0.84925 0.00260 Canta-Leo Ast-Gal 0.35766 0.00372 Canta-Leo Catal-Bale 0.19963 0.00364 Canta-Leo Murc-Vale 0.78513 0.00265 Canta-Leo Canaria__ 0.30298 0.00374 Canta-Leo Casti-Man 0.80583 0.00334 Navar-Bas Andal-Extr 0.00571 0.00045 Navar-Bas Arag-LaRi 0.76519 0.00258 Navar-Bas Ast-Gal 0.44670 0.00386 Navar-Bas Catal-Bale 0.00863 0.00062 Navar-Bas Murc-Vale 0.05024 0.00164 Navar-Bas Canaria__ 0.09577 0.00259 Navar-Bas Casti-Man 0.39233 0.00424 Navar-Bas Canta-Leo 0.57396 0.00430
Tabella A.5.: Approccio storico-politico: differenziazione genotipica a coppie di
popolazioni calcolata in base alle frequenze genotipiche del locus D1S80.
FREQUENZE GENOTIPICHE LOCUS D4S111 Genotipi Pop Andal-Extr 0,023800 0,071428 0,333333 0,071428 0,428571 0,071428 Arag-LaRi 0,000000 0,185185 0,518518 0,074074 0,185185 0,037037 Ast-Gal 0,034482 0,241379 0,241379 0,000000 0,448275 0,034482 Catal-Bale 0,069767 0,186046 0,348837 0,000000 0,325581 0,069767 Murc-Vale 0,000000 0,304347 0,347826 0,000000 0,304347 0,043478 Canaria__ 0,000000 0,111111 0,333333 0,000000 0,444444 0,111111 Casti-Man 0,100000 0,260000 0,320000 0,000000 0,280000 0,040000 Canta-Leo 0,057142 0,085714 0,428571 0,114285 0,314285 0,000000 Navar-Bas 0,000000 0,205128 0,282051 0,076923 0,358974 0,076923 Totale 0,04040 0,18518 0,34680 0,04040 0,33670 0,05050
Tabella A.6.: Approccio storico-politico:
frequenze genotipiche per il locus D4S111.
FREQUENZE ALLELICHE D4S11 1 2 3 Andal-Extr 0,095238 0,583333 0,321429 Arag-LaRi 0,129630 0,703704 0,166667 Ast -Gal 0,155172 0,586207 0,258621 Catal-Bale 0,162791 0,604651 0,232558 Murc-Vale 0,152174 0,652174 0,195652 Canaria___ 0,055556 0,611111 0,333333 Casti-Man 0,230000 0,590000 0,180000 Canta-Leo 0,157143 0,628571 0,214286 Navar-Bas 0,141026 0,564103 0,294872 Totale 0,153199 0,607744 0,239057
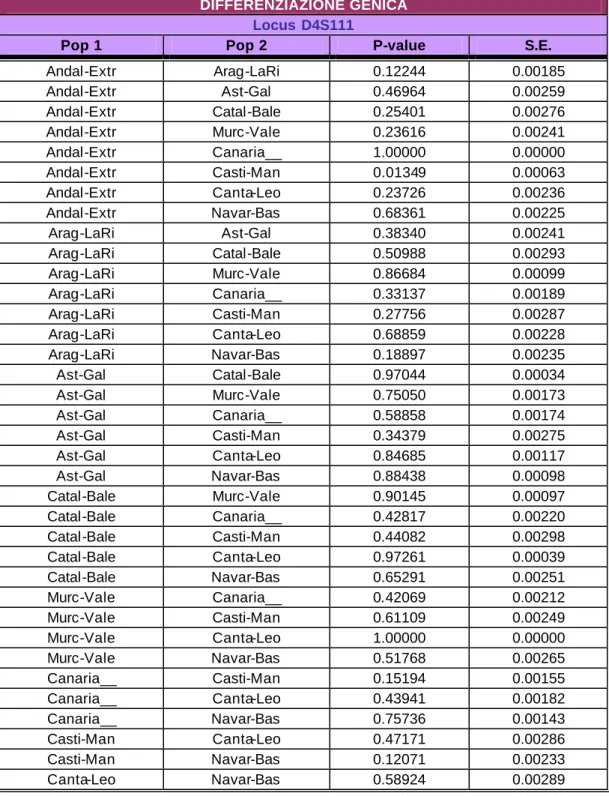
DIFFERENZIAZIONE GENICA Locus D4S111
Pop 1 Pop 2 P-value S.E.
Andal-Extr Arag-LaRi 0.12244 0.00185 Andal-Extr Ast-Gal 0.46964 0.00259 Andal-Extr Catal-Bale 0.25401 0.00276 Andal-Extr Murc-Vale 0.23616 0.00241 Andal-Extr Canaria__ 1.00000 0.00000 Andal-Extr Casti-Man 0.01349 0.00063 Andal-Extr Canta-Leo 0.23726 0.00236 Andal-Extr Navar-Bas 0.68361 0.00225 Arag-LaRi Ast-Gal 0.38340 0.00241 Arag-LaRi Catal-Bale 0.50988 0.00293 Arag-LaRi Murc-Vale 0.86684 0.00099 Arag-LaRi Canaria__ 0.33137 0.00189 Arag-LaRi Casti-Man 0.27756 0.00287 Arag-LaRi Canta-Leo 0.68859 0.00228 Arag-LaRi Navar-Bas 0.18897 0.00235 Ast-Gal Catal-Bale 0.97044 0.00034 Ast-Gal Murc-Vale 0.75050 0.00173 Ast-Gal Canaria__ 0.58858 0.00174 Ast-Gal Casti-Man 0.34379 0.00275 Ast-Gal Canta-Leo 0.84685 0.00117 Ast-Gal Navar-Bas 0.88438 0.00098 Catal-Bale Murc-Vale 0.90145 0.00097 Catal-Bale Canaria__ 0.42817 0.00220 Catal-Bale Casti-Man 0.44082 0.00298 Catal-Bale Canta-Leo 0.97261 0.00039 Catal-Bale Navar-Bas 0.65291 0.00251 Murc-Vale Canaria__ 0.42069 0.00212 Murc-Vale Casti-Man 0.61109 0.00249 Murc-Vale Canta-Leo 1.00000 0.00000 Murc-Vale Navar-Bas 0.51768 0.00265 Canaria__ Casti-Man 0.15194 0.00155 Canaria__ Canta-Leo 0.43941 0.00182 Canaria__ Navar-Bas 0.75736 0.00143 Casti-Man Canta-Leo 0.47171 0.00286 Casti-Man Navar-Bas 0.12071 0.00233 Canta-Leo Navar-Bas 0.58924 0.00289
Tabella A.8.: Approccio storico-politico : differenziazione genica a coppie di
popolazioni calcolata in base alle frequenze alleliche del locus D4S111.
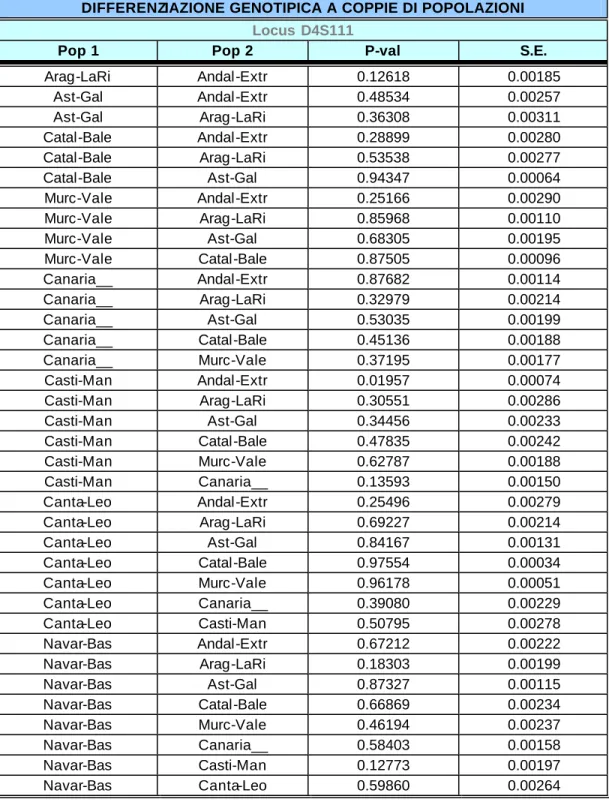
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D4S111
Pop 1 Pop 2 P-val S.E.
Arag-LaRi Andal-Extr 0.12618 0.00185 Ast-Gal Andal-Extr 0.48534 0.00257 Ast-Gal Arag-LaRi 0.36308 0.00311 Catal-Bale Andal-Extr 0.28899 0.00280 Catal-Bale Arag-LaRi 0.53538 0.00277 Catal-Bale Ast-Gal 0.94347 0.00064 Murc-Vale Andal-Extr 0.25166 0.00290 Murc-Vale Arag-LaRi 0.85968 0.00110 Murc-Vale Ast-Gal 0.68305 0.00195 Murc-Vale Catal-Bale 0.87505 0.00096 Canaria__ Andal-Extr 0.87682 0.00114 Canaria__ Arag-LaRi 0.32979 0.00214 Canaria__ Ast-Gal 0.53035 0.00199 Canaria__ Catal-Bale 0.45136 0.00188 Canaria__ Murc-Vale 0.37195 0.00177 Casti-Man Andal-Extr 0.01957 0.00074 Casti-Man Arag-LaRi 0.30551 0.00286 Casti-Man Ast-Gal 0.34456 0.00233 Casti-Man Catal-Bale 0.47835 0.00242 Casti-Man Murc-Vale 0.62787 0.00188 Casti-Man Canaria__ 0.13593 0.00150 Canta-Leo Andal-Extr 0.25496 0.00279 Canta-Leo Arag-LaRi 0.69227 0.00214 Canta-Leo Ast-Gal 0.84167 0.00131 Canta-Leo Catal-Bale 0.97554 0.00034 Canta-Leo Murc-Vale 0.96178 0.00051 Canta-Leo Canaria__ 0.39080 0.00229 Canta-Leo Casti-Man 0.50795 0.00278 Navar-Bas Andal-Extr 0.67212 0.00222 Navar-Bas Arag-LaRi 0.18303 0.00199 Navar-Bas Ast-Gal 0.87327 0.00115 Navar-Bas Catal-Bale 0.66869 0.00234 Navar-Bas Murc-Vale 0.46194 0.00237 Navar-Bas Canaria__ 0.58403 0.00158 Navar-Bas Casti-Man 0.12773 0.00197 Navar-Bas Canta-Leo 0.59860 0.00264
Tabella A.9.: Approccio storico-politico: differenziazione genotipica a coppie di
popolazioni calcolata in base alle frequenze genotipiche.
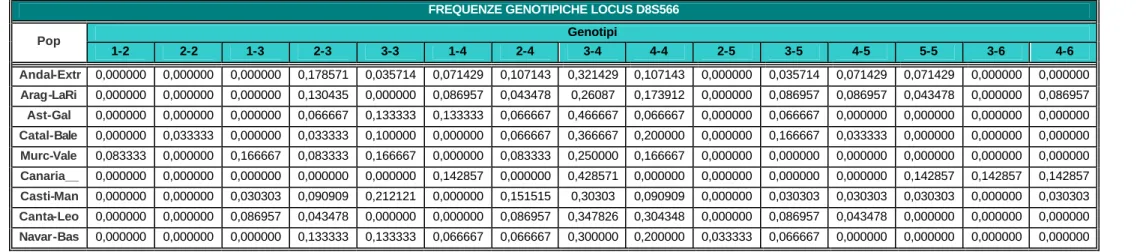
FREQUENZE GENOTIPICHE LOCUS D8S566 Genotipi Pop 1-2 2-2 1-3 2-3 3-3 1-4 2-4 3-4 4-4 2-5 3-5 4-5 5-5 3-6 4-6 Andal-Extr 0,000000 0,000000 0,000000 0,178571 0,035714 0,071429 0,107143 0,321429 0,107143 0,000000 0,035714 0,071429 0,071429 0,000000 0,000000 Arag-LaRi 0,000000 0,000000 0,000000 0,130435 0,000000 0,086957 0,043478 0,26087 0,173912 0,000000 0,086957 0,086957 0,043478 0,000000 0,086957 Ast-Gal 0,000000 0,000000 0,000000 0,066667 0,133333 0,133333 0,066667 0,466667 0,066667 0,000000 0,066667 0,000000 0,000000 0,000000 0,000000 Catal-Bale 0,000000 0,033333 0,000000 0,033333 0,100000 0,000000 0,066667 0,366667 0,200000 0,000000 0,166667 0,033333 0,000000 0,000000 0,000000 Murc-Vale 0,083333 0,000000 0,166667 0,083333 0,166667 0,000000 0,083333 0,250000 0,166667 0,000000 0,000000 0,000000 0,000000 0,000000 0,000000 Canaria__ 0,000000 0,000000 0,000000 0,000000 0,000000 0,142857 0,000000 0,428571 0,000000 0,000000 0,000000 0,000000 0,142857 0,142857 0,142857 Casti-Man 0,000000 0,000000 0,030303 0,090909 0,212121 0,000000 0,151515 0,30303 0,090909 0,000000 0,030303 0,030303 0,030303 0,000000 0,030303 Canta-Leo 0,000000 0,000000 0,086957 0,043478 0,000000 0,000000 0,086957 0,347826 0,304348 0,000000 0,086957 0,043478 0,000000 0,000000 0,000000 Navar-Bas 0,000000 0,000000 0,000000 0,133333 0,133333 0,066667 0,066667 0,300000 0,200000 0,033333 0,066667 0,000000 0,000000 0,000000 0,000000
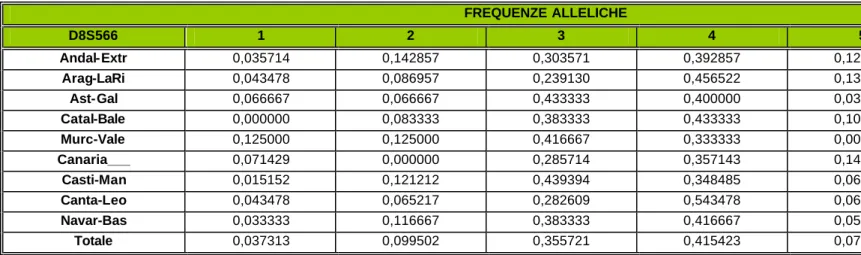
FREQUENZE ALLELICHE D8S566 1 2 3 4 5 6 Andal-Extr 0,035714 0,142857 0,303571 0,392857 0,125000 0,000000 Arag-LaRi 0,043478 0,086957 0,239130 0,456522 0,130435 0,043478 Ast-Gal 0,066667 0,066667 0,433333 0,400000 0,033333 0,000000 Catal-Bale 0,000000 0,083333 0,383333 0,433333 0,100000 0,000000 Murc-Vale 0,125000 0,125000 0,416667 0,333333 0,000000 0,000000 Canaria___ 0,071429 0,000000 0,285714 0,357143 0,142857 0,142857 Casti-Man 0,015152 0,121212 0,439394 0,348485 0,060606 0,015152 Canta-Leo 0,043478 0,065217 0,282609 0,543478 0,065217 0,000000 Navar-Bas 0,033333 0,116667 0,383333 0,416667 0,050000 0,000000 Totale 0,037313 0,099502 0,355721 0,415423 0,079602 0,012438
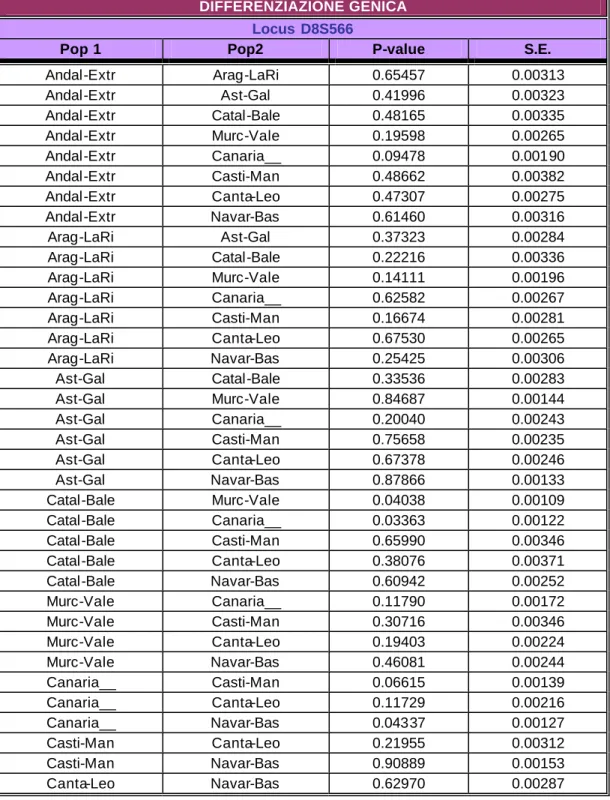
DIFFERENZIAZIONE GENICA Locus D8S566
Pop 1 Pop2 P-value S.E.
Andal-Extr Arag-LaRi 0.65457 0.00313 Andal-Extr Ast-Gal 0.41996 0.00323 Andal-Extr Catal-Bale 0.48165 0.00335 Andal-Extr Murc-Vale 0.19598 0.00265 Andal-Extr Canaria__ 0.09478 0.00190 Andal-Extr Casti-Man 0.48662 0.00382 Andal-Extr Canta-Leo 0.47307 0.00275 Andal-Extr Navar-Bas 0.61460 0.00316 Arag-LaRi Ast-Gal 0.37323 0.00284 Arag-LaRi Catal-Bale 0.22216 0.00336 Arag-LaRi Murc-Vale 0.14111 0.00196 Arag-LaRi Canaria__ 0.62582 0.00267 Arag-LaRi Casti-Man 0.16674 0.00281 Arag-LaRi Canta-Leo 0.67530 0.00265 Arag-LaRi Navar-Bas 0.25425 0.00306 Ast-Gal Catal-Bale 0.33536 0.00283 Ast-Gal Murc-Vale 0.84687 0.00144 Ast-Gal Canaria__ 0.20040 0.00243 Ast-Gal Casti-Man 0.75658 0.00235 Ast-Gal Canta-Leo 0.67378 0.00246 Ast-Gal Navar-Bas 0.87866 0.00133 Catal-Bale Murc-Vale 0.04038 0.00109 Catal-Bale Canaria__ 0.03363 0.00122 Catal-Bale Casti-Man 0.65990 0.00346 Catal-Bale Canta-Leo 0.38076 0.00371 Catal-Bale Navar-Bas 0.60942 0.00252 Murc-Vale Canaria__ 0.11790 0.00172 Murc-Vale Casti-Man 0.30716 0.00346 Murc-Vale Canta-Leo 0.19403 0.00224 Murc-Vale Navar-Bas 0.46081 0.00244 Canaria__ Casti-Man 0.06615 0.00139 Canaria__ Canta-Leo 0.11729 0.00216 Canaria__ Navar-Bas 0.04337 0.00127 Casti-Man Canta-Leo 0.21955 0.00312 Casti-Man Navar-Bas 0.90889 0.00153 Canta-Leo Navar-Bas 0.62970 0.00287
Tabella A.12.: Approccio storico-politico: differenziazione genica a coppie di
popolazioni calcolata in base alle frequenze alleliche per il locus D8S566.
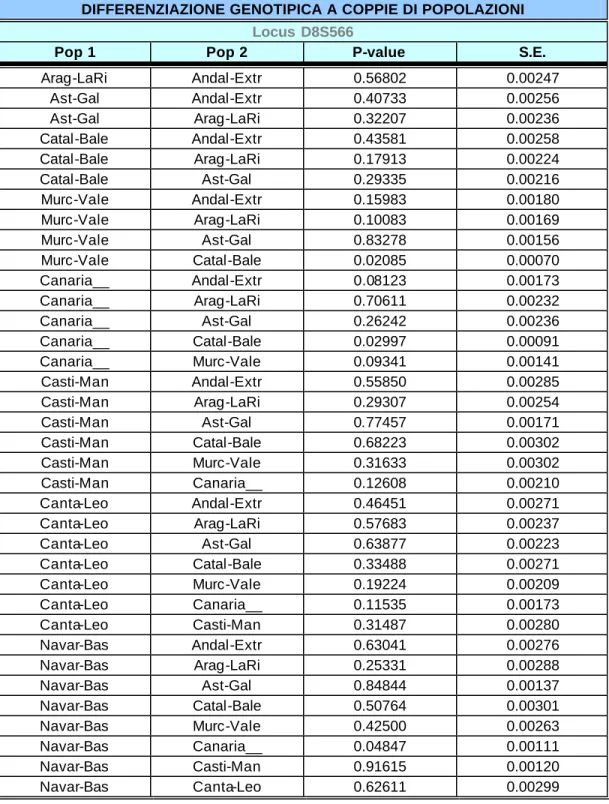
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D8S566
Pop 1 Pop 2 P-value S.E.
Arag-LaRi Andal-Extr 0.56802 0.00247 Ast-Gal Andal-Extr 0.40733 0.00256 Ast-Gal Arag-LaRi 0.32207 0.00236 Catal-Bale Andal-Extr 0.43581 0.00258 Catal-Bale Arag-LaRi 0.17913 0.00224 Catal-Bale Ast-Gal 0.29335 0.00216 Murc-Vale Andal-Extr 0.15983 0.00180 Murc-Vale Arag-LaRi 0.10083 0.00169 Murc-Vale Ast-Gal 0.83278 0.00156 Murc-Vale Catal-Bale 0.02085 0.00070 Canaria__ Andal-Extr 0.08123 0.00173 Canaria__ Arag-LaRi 0.70611 0.00232 Canaria__ Ast-Gal 0.26242 0.00236 Canaria__ Catal-Bale 0.02997 0.00091 Canaria__ Murc-Vale 0.09341 0.00141 Casti-Man Andal-Extr 0.55850 0.00285 Casti-Man Arag-LaRi 0.29307 0.00254 Casti-Man Ast-Gal 0.77457 0.00171 Casti-Man Catal-Bale 0.68223 0.00302 Casti-Man Murc-Vale 0.31633 0.00302 Casti-Man Canaria__ 0.12608 0.00210 Canta-Leo Andal-Extr 0.46451 0.00271 Canta-Leo Arag-LaRi 0.57683 0.00237 Canta-Leo Ast-Gal 0.63877 0.00223 Canta-Leo Catal-Bale 0.33488 0.00271 Canta-Leo Murc-Vale 0.19224 0.00209 Canta-Leo Canaria__ 0.11535 0.00173 Canta-Leo Casti-Man 0.31487 0.00280 Navar-Bas Andal-Extr 0.63041 0.00276 Navar-Bas Arag-LaRi 0.25331 0.00288 Navar-Bas Ast-Gal 0.84844 0.00137 Navar-Bas Catal-Bale 0.50764 0.00301 Navar-Bas Murc-Vale 0.42500 0.00263 Navar-Bas Canaria__ 0.04847 0.00111 Navar-Bas Casti-Man 0.91615 0.00120 Navar-Bas Canta-Leo 0.62611 0.00299
Tabella A.13.: Approccio storico-politico : differenziazione genotipica a coppie di
popolazioni calcolata in basa alle frequenze genotipiche per il locus D8S566
.FREQUENZE GENOTIPICHE LOCUS D8S591 Genotipi Pop 1-1 1-2 2-2 1-3 2-3 3-3 1-4 2-4 3-4 4-4 1-5 3-5 Andal-Extr 0,105263 0,184211 0,078947 0,315789 0,026316 0,157895 0,078947 0,026316 0,000000 0,026316 0,000000 0,000000 Arag-LaRi 0,074074 0,222222 0,148148 0,259259 0,111111 0,111111 0,037037 0,000000 0,037037 0,000000 0,000000 0,000000 Ast-Gal 0,153846 0,192308 0,076923 0,346154 0,153846 0,038462 0,000000 0,038462 0,000000 0,000000 0,000000 0,000000 Catal-Bale 0,125000 0,300000 0,000000 0,400000 0,050000 0,025000 0,050000 0,025000 0,000000 0,000000 0,000000 0,025000 Murc-Vale 0,130435 0,130435 0,173913 0,217391 0,086957 0,217391 0,043478 0,000000 0,000000 0,000000 0,000000 0,000000 Canaria__ 0,125000 0,250000 0,125000 0,125000 0,125000 0,125000 0,000000 0,000000 0,000000 0,000000 0,125000 0,000000 Casti-Man 0,256410 0,076923 0,051282 0,282051 0,179487 0,025641 0,076923 0,000000 0,025641 0,000000 0,025641 0,000000 Canta-Leo 0,222222 0,148148 0,000000 0,37037 0,037037 0,000000 0,074074 0,037037 0,074074 0,000000 0,000000 0,037037 Navar-Bas 0,102564 0,25641 0,051282 0,307692 0,025641 0,128205 0,051282 0,000000 0,000000 0,000000 0,025641 0,051282
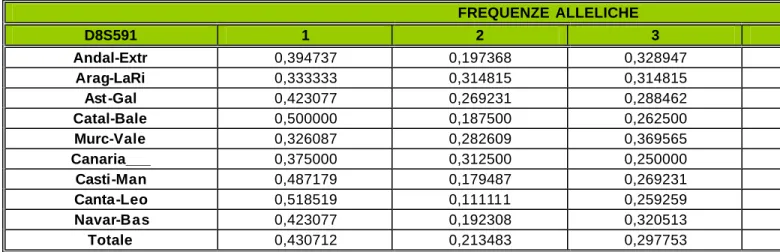
FREQUENZE ALLELICHE D8S591 1 2 3 4 5 Andal-Extr 0,394737 0,197368 0,328947 0,078947 0,000000 Arag-LaRi 0,333333 0,314815 0,314815 0,037037 0,000000 Ast -Gal 0,423077 0,269231 0,288462 0,019231 0,000000 Catal-Bale 0,500000 0,187500 0,262500 0,037500 0,012500 Murc-Vale 0,326087 0,282609 0,369565 0,021739 0,000000 Canaria___ 0,375000 0,312500 0,250000 0,000000 0,062500 Casti-Man 0,487179 0,179487 0,269231 0,051282 0,012821 Canta-Leo 0,518519 0,111111 0,259259 0,092593 0,018519 Navar-Bas 0,423077 0,192308 0,320513 0,025641 0,038462 Totale 0,430712 0,213483 0,297753 0,044944 0,013109
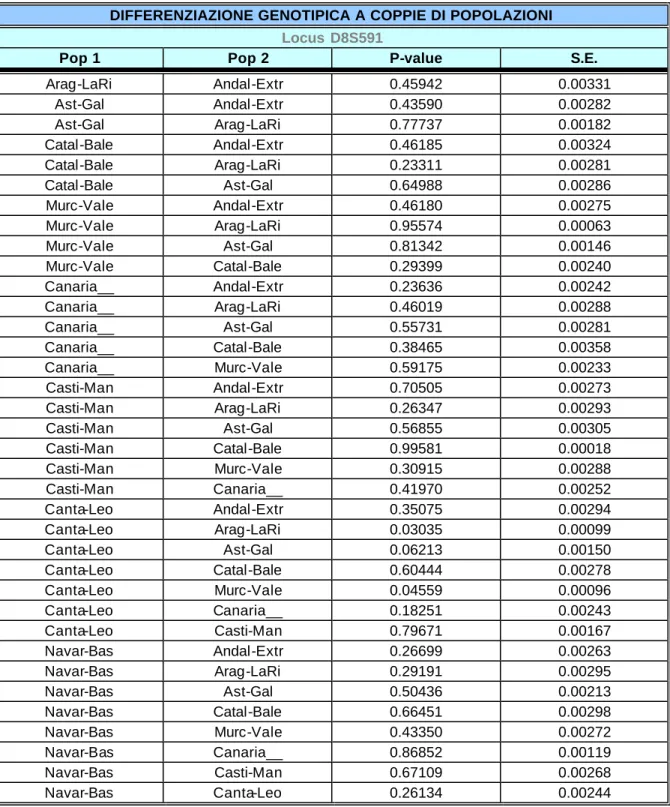
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D8S591
Pop 1 Pop 2 P-value S.E.
Arag-LaRi Andal-Extr 0.45942 0.00331 Ast-Gal Andal-Extr 0.43590 0.00282 Ast-Gal Arag-LaRi 0.77737 0.00182 Catal-Bale Andal-Extr 0.46185 0.00324 Catal-Bale Arag-LaRi 0.23311 0.00281 Catal-Bale Ast-Gal 0.64988 0.00286 Murc-Vale Andal-Extr 0.46180 0.00275 Murc-Vale Arag-LaRi 0.95574 0.00063 Murc-Vale Ast-Gal 0.81342 0.00146 Murc-Vale Catal-Bale 0.29399 0.00240 Canaria__ Andal-Extr 0.23636 0.00242 Canaria__ Arag-LaRi 0.46019 0.00288 Canaria__ Ast-Gal 0.55731 0.00281 Canaria__ Catal-Bale 0.38465 0.00358 Canaria__ Murc-Vale 0.59175 0.00233 Casti-Man Andal-Extr 0.70505 0.00273 Casti-Man Arag-LaRi 0.26347 0.00293 Casti-Man Ast-Gal 0.56855 0.00305 Casti-Man Catal-Bale 0.99581 0.00018 Casti-Man Murc-Vale 0.30915 0.00288 Casti-Man Canaria__ 0.41970 0.00252 Canta-Leo Andal-Extr 0.35075 0.00294 Canta-Leo Arag-LaRi 0.03035 0.00099 Canta-Leo Ast-Gal 0.06213 0.00150 Canta-Leo Catal-Bale 0.60444 0.00278 Canta-Leo Murc-Vale 0.04559 0.00096 Canta-Leo Canaria__ 0.18251 0.00243 Canta-Leo Casti-Man 0.79671 0.00167 Navar-Bas Andal-Extr 0.26699 0.00263 Navar-Bas Arag-LaRi 0.29191 0.00295 Navar-Bas Ast-Gal 0.50436 0.00213 Navar-Bas Catal-Bale 0.66451 0.00298 Navar-Bas Murc-Vale 0.43350 0.00272 Navar-Bas Canaria__ 0.86852 0.00119 Navar-Bas Casti-Man 0.67109 0.00268 Navar-Bas Canta-Leo 0.26134 0.00244
Tabella A.16.: Approccio storico-politico: differenziamento genotipico a coppie di popolazioni
calcolato in base alle frequenze genotipiche per il locus D8S591.
DIFFERENZIAZIONE GENICA Locus D8S591
Pop 1 Pop 2 P-value S.E.
Andal-Extr Arag-LaRi 0.42022 0.00325 Andal-Extr Ast-Gal 0.42992 0.00315 Andal-Extr Catal-Bale 0.46031 0.00411 Andal-Extr Murc-Vale 0.40068 0.00293 Andal-Extr Canaria__ 0.24148 0.00271 Andal-Extr Casti-Man 0.64374 0.00355 Andal-Extr Canta-Leo 0.32354 0.00345 Andal-Extr Navar-Bas 0.33130 0.00360 Arag-LaRi Ast-Gal 0.77524 0.00209 Arag-LaRi Catal-Bale 0.21638 0.00312 Arag-LaRi Murc-Vale 0.94825 0.00084 Arag-LaRi Canaria__ 0.49820 0.00267 Arag-LaRi Casti-Man 0.23840 0.00341 Arag-LaRi Canta-Leo 0.02866 0.00120 Arag-LaRi Navar-Bas 0.32749 0.00331 Ast-Gal Catal-Bale 0.71420 0.00277 Ast-Gal Murc-Vale 0.79563 0.00165 Ast-Gal Canaria__ 0.56645 0.00272 Ast-Gal Casti-Man 0.60061 0.00364 Ast-Gal Canta-Leo 0.09712 0.00215 Ast-Gal Navar-Bas 0.64253 0.00298 Catal-Bale Murc-Vale 0.23150 0.00359 Catal-Bale Canaria__ 0.41605 0.00278 Catal-Bale Casti-Man 0.99660 0.00015 Catal-Bale Canta-Leo 0.55438 0.00296 Catal-Bale Navar-Bas 0.72285 0.00282 Murc-Vale Canaria__ 0.51144 0.00270 Murc-Vale Casti-Man 0.23925 0.00327 Murc-Vale Canta-Leo 0.03184 0.00112 Murc-Vale Navar-Bas 0.46718 0.00356 Canaria__ Casti-Man 0.37886 0.00244 Canaria__ Canta-Leo 0.18977 0.00200 Canaria__ Navar-Bas 0.73225 0.00251 Casti-Man Canta-Leo 0.72994 0.00273 Casti-Man Navar-Bas 0.68912 0.00324 Canta-Leo Navar-Bas 0.25459 0.00340
Tabella A.17.: Approccio storico-politico : differenziazione genica a coppie di popolazioni
calcolata in base alle frequenze alleliche per il locus D8S591.
TAVOLE RELATIVE AL SECONDO APPROCCIO SPERIMENTALE: A18-A35.
LEGENDA
ABBREVIAZIONI AREE GEOGRAFICHE DI PROVENIENZA
DEL CAMPIONE
Cantabrica Zona Cantabrica, atlantica settentrionale
Pirenaica Zona dei Pirenei
Mediterranea Zona del Mediterraneo
Meseta Zona della Meseta, altipiano centrale
Andalusia Andalusia, valle del Guadalquivir
EQUILIBRIO H-W EQUILIBRIO H-W
D8S566 D8S591
Pop P-val Pop P-val
Cantabrica 0,5006 Cantabrica 0,2043 Pirenaica 0,2330 Pirenaica 0,0315 Mediterranea 0,4377 Mediterranea 0,2354 Meseta 0,9161 Meseta 0,8228 Andalusia 0,1183 Andalusia 0,0415 EQUILIBRIO H-W EQUILIBRIO H-W D4S111 D1S80
Pop P-val Pop P-val
Cantabrica 0,3793 Cantabrica 0,1709 Pirenaica 0,6823 Pirenaica 0,0057 Mediterranea 0,2995 Mediterranea 0,0151 Meseta 0,0148 Meseta 0,0018 Andalusia 0,8884 Andalusia 0,0340 EQUILIBRIO H-W D17S5 Pop P-val Cantabrica 0,0000
FREQUENZE GENOTIPICHE LOCUS D1S80 Popolazioni
Genotipi
Andalusia Meseta Zona Mediterraneanea Zona Pirenaica Zona Cantabricaa
1515 0 0 0,026316 0 0 1518 0 0,009091 0 0 0 1522 0 0 0 0 0 1718 0,035714286 0 0 0 0 1721 0 0,009091 0 0 0 1724 0 0,009091 0 0 0 1818 0 0,081818 0,131579 0,192308 0,064516 1819 0 0 0 0 0 1820 0 0,009091 0 0 0 1821 0 0 0 0 0,016129 1822 0,107142857 0,045455 0 0,038462 0 1824 0,357142857 0,163636 0,052632 0,115385 0,16129 1825 0,035714286 0 0,026316 0 0,016129 1826 0 0 0,026316 0 0 1828 0 0 0,052632 0 0,016129 1829 0 0,018182 0,026316 0 0,016129 1830 0 0,009091 0,026316 0 0 1831 0 0,036364 0,052632 0 0,032258 1832 0 0 0 0 0,032258 1919 0,035714286 0 0 0,038462 0 1921 0 0 0 0 0 1925 0 0 0 0,038462 0,016129 1926 0 0,009091 0 0 0 1929 0 0 0 0 0,016129 1930 0 0,018182 0 0 0 1933 0 0 0 0,038462 0 2021 0,009091 0 0 0 2022 0 0 0 0 0,016129 2024 0,035714286 0,018182 0 0 0,032258 2025 0 0 0 0 0,016129 2026 0 0 0 0 0,016129 2028 0 0 0 0 0,016129 2030 0 0,009091 0 0 0 2031 0 0,009091 0 0 0,032258 2121 0 0 0,026316 0 0,016129 2122 0 0 0 0 0 2124 0,035714286 0,018182 0,078947 0 0,032258 2125 0 0,009091 0 0 0 2128 0 0 0 0,038462 0 2129 0 0 0 0 0
2325 0 0 0 0 0,016129 2334 0 0,009091 0 0 0 2424 0,071428571 0,136364 0,105263 0,076923 0,112903 2425 0 0,072727 0 0 0,048387 2426 0 0,009091 0 0 0 2427 0,035714286 0 0 0 0,016129 2428 0,035714286 0,009091 0,026316 0,076923 0,016129 2429 0 0,027273 0,026316 0,038462 0,064516 2430 0,071428571 0 0 0 0 2431 0,035714286 0,036364 0 0,076923 0,016129 2432 0,035714286 0,018182 0 0 0,032258 2433 0 0,009091 0,026316 0 0 2434 0 0 0 0 0,016129 2436 0 0 0 0,038462 0 2437 0 0,009091 0,052632 0 0 2438 0 0 0 0,038462 0 2525 0 0 0,026316 0,038462 0 2530 0 0,009091 0,026316 0 0 2531 0 0 0 0 0,016129 2534 0 0 0 0 0,016129 2626 0 0,009091 0 0 0 2632 0,035714286 0 0 0 0 2639 0 0 0 0 0,016129 2728 0 0 0 0 0,016129 2730 0 0 0,026316 0 0 2734 0 0 0 0,038462 0 2828 0,035714286 0 0 0 0 2829 0 0 0 0,038462 0 2831 0 0 0 0 0,016129 2835 0 0 0,026316 0 0 2929 0 0 0 0 0 2930 0 0,018182 0 0 0 2931 0 0,018182 0 0,038462 0 3030 0 0 0 0 0 3037 0 0,009091 0 0 0 3131 0 0,009091 0 0 0 3441 0 0 0 0 0,016129
FREQUENZE ALLELICHE D1S80 Popolazioni Alleli Zona Cantabricaa Zona pirenaica Zona
Mediterraneanea Meseta Andalusia Totale
15 0,000 0,000 0,026 0,005 0,000 0,006 17 0,000 0,000 0,000 0,009 0,018 0,006 18 0,210 0,269 0,263 0,227 0,268 0,237 19 0,016 0,077 0,000 0,014 0,036 0,021 20 0,065 0,000 0,000 0,027 0,018 0,028 21 0,040 0,019 0,066 0,027 0,018 0,034 22 0,008 0,019 0,066 0,059 0,054 0,044 23 0,008 0,000 0,013 0,014 0,000 0,009 24 0,331 0,269 0,263 0,350 0,393 0,330 25 0,073 0,058 0,053 0,045 0,018 0,051 26 0,016 0,000 0,013 0,023 0,018 0,017 27 0,016 0,019 0,013 0,005 0,018 0,011 28 0,040 0,077 0,053 0,005 0,054 0,032 29 0,048 0,058 0,026 0,055 0,000 0,044 30 0,000 0,000 0,066 0,041 0,036 0,030 31 0,056 0,058 0,026 0,064 0,018 0,051 32 0,032 0,000 0,000 0,009 0,036 0,015 33 0,000 0,019 0,013 0,005 0,000 0,006 34 0,024 0,019 0,000 0,005 0,000 0,009 35 0,000 0,000 0,013 0,000 0,000 0,002 36 0,000 0,019 0,000 0,005 0,000 0,004 37 0,000 0,000 0,026 0,009 0,000 0,008 38 0,000 0,019 0,000 0,000 0,000 0,002 39 0,008 0,000 0,000 0,000 0,000 0,002 41 0,008 0,000 0,000 0,000 0,000 0,002
Tabella A.20: Approccio geografico: frequenze alleliche del locus D1S80.
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D1S80
Pop 1 Pop 2 P-value S.E.
Cantabrica Pirenaica 0.28281 0.00650 Cantabrica Mediterranea 0.00257 0.00043 Cantabrica Meseta 0.01663 0.00162 Cantabrica Andalusia 0.17707 0.00460 Pirenaica Mediterranea 0.17567 0.00445 Pirenaica Meseta 0.02197 0.00213 Pirenaica Andalusia 0.35329 0.00535 Mediterranea Meseta 0.11492 0.00485
DIFFERENZIAZIONE GENICA Locus D1S80
Pop 1 Pop 2 P-value S.E.
Cantabrica Pirenaica 0.28281 0.00650 Cantabrica Mediterranea 0.00257 0.00043 Cantabrica Meseta 0.01663 0.00162 Cantabrica Andalusia 0.17707 0.00460 Pirenaica Mediterranea 0.17567 0.00445 Pirenaica Meseta 0.02197 0.00213 Pirenaica Andalusia 0.35329 0.00535 Mediterranea Meseta 0.11492 0.00485 Mediterranea Andalusia 0.48405 0.00590 Meseta Andalusia 0.36047 0.00801
Tabella A.22.: Approccio geografico: differenziazione genica a coppie di popolazioni calcolata in
base alle frequenze alleliche del locus D1S80.
FREQUENZE GENOTIPICHE D4S111 Genotipi Pop 1-1 1-2 2-2 1-3 2-3 3-3 3-4 Cantabrica 0,0139 0,2222 0,2778 0,0556 0,3750 0,0556 0,0000 Pirenaica 0,0625 0,2500 0,3438 0,0313 0,2813 0,0313 0,0000 Mediterranea 0,0385 0,2115 0,3462 0,0192 0,3269 0,0577 0,0000 Meseta 0,0796 0,1681 0,3894 0,0442 0,2743 0,0354 0,0088 Andalusia 0,0000 0,0667 0,3667 0,0667 0,4000 0,1000 0,0000
Tabella A.23.: Approccio geografico: frequenze genotipiche del locus D4S111.
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D4S111
Pop 1 Pop 2 P-val S.E.
Cantabrica Pirenaica 0.37206 0.00286
Cantabrica Mediterranea 0.80177 0.00210
Cantabrica Meseta 0.32733 0.00569
Cantabrica Andalusia 0.22555 0.00293
FREQUENZE ALLELICHE D4S111 Alleli Pop 01 02 03 04 Zona Cantabrica 0,153 0,576 0,271 0,000 Zona Pirenaica 0,203 0,609 0,188 0,000 Zona Mediterraneanea 0,154 0,615 0,231 0,000 Meseta 0,186 0,611 0,199 0,004 Andalusia 0,067 0,600 0,333 0,000 Totale 0,162 0,602 0,234 0,002
Tabella A.24. : Approccio geografico: frequenze alleliche del locus D4S111
DIFFERENZIAZIONE GENICA Locus D4S111
Pop 1 Pop 2 P-value S.E.
Cantabrica Pirenaica 0.37206 0.00286 Cantabrica Mediterranea 0.80177 0.00210 Cantabrica Meseta 0.32733 0.00569 Cantabric a Andalusia 0.22555 0.00293 Pirenaica Mediterranea 0.62329 0.00253 Pirenaica Meseta 0.96853 0.00067 Pirenaica Andalusia 0.03557 0.00097 Mediterranea Meseta 0.78503 0.00317 Mediterranea Andalusia 0.13776 0.00227 Meseta Andalusia 0.02643 0.00115
Tabella A.26. : Approccio geografico: differenziazione genica a coppie di popolazioni calcolata in
base ai valori delle frequenze alleliche per il locus D4S111
FREQUENZE GENOTIPICHE LOCUS D8S566 Genotipi Pop 1-2 2-2 1-3 2-3 3-3 1-4 2-4 3-4 4-4 2-5 3-5 4-5 5-5 4-6 Cantabrica 0,0000 0,0000 0,0213 0,1064 0,1277 0,0851 0,0638 0,3617 0,1489 0,0213 0,0638 0,0000 0,0000 0,0000 Pirenaica 0,0000 0,0400 0,0000 0,1200 0,0400 0,0800 0,0400 0,2400 0,2000 0,0000 0,1600 0,0400 0,0400 0,0000 Mediterranea 0,0333 0,0000 0,0667 0,0333 0,1000 0,0333 0,1000 0,3333 0,1667 0,0000 0,1333 0,0000 0,0000 0,0000 Meseta 0,0000 0,0000 0,0238 0,0952 0,0952 0,0119 0,0952 0,3095 0,1786 0,0119 0,0595 0,0714 0,0119 0,0357 Andalusia 0,0000 0,0000 0,0000 0,2222 0,0556 0,0556 0,1111 0,2778 0,1111 0,0000 0,0000 0,0556 0,1111 0,0000
Tabella A.27.: Approccio geografico: frequenze genotipiche del locus D8S566.
FREQUENZE ALLELICHE LOCUS D8S566 Alleli Popolazioni 01 02 03 04 05 06 Zona Cantabrica 0,053 0,096 0,404 0,404 0,043 0,000 Zona Pirenaica 0,040 0,120 0,300 0,400 0,140 0,000 Zona Mediterraneanea 0,067 0,083 0,383 0,400 0,067 0,000 Meseta 0,018 0,101 0,339 0,440 0,083 0,018 Andalusia 0,028 0,167 0,306 0,361 0,139 0,000
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D8S566
Pop 1 Pop 2 P-value S.E.
Cantabrica Pirenaica 0.26622 0.00295 Cantabrica Mediterranea 0.96295 0.00058 Cantabrica Meseta 0.30598 0.00434 Cantabrica Andalusia 0.23001 0.00315 Pirenaica Mediterranea 0.61419 0.00299 Pirenaica Meseta 0.61649 0.00380 Pirenaica Andalusia 0.98432 0.00030 Mediterranea Meseta 0.47081 0.00405 Mediterranea Andalusia 0.47968 0.00265 Meseta Andalusia 0.56336 0.00368
DIFFERENZIAZIONE GENICA Locus D8S566
Pop 1 Pop2 P-value S.E.
Cantabrica Pirenaica 0.26622 0.00295 Cantabrica Mediterranea 0.96295 0.00058 Cantabrica Meseta 0.30598 0.00434 Cantabrica Andalusia 0.23001 0.00315 Pirenaica Mediterranea 0.61419 0.00299 Pirenaica Meseta 0.61649 0.00380 Pirenaica Andalusia 0.98432 0.00030 Mediterranea Meseta 0.47081 0.00405 Mediterranea Andalusia 0.47968 0.00265 Meseta Andalusia 0.56336 0.00368
FREQUENZE GENOTIPICHE LOCUS D8S591 Genotipi Pop 1-1 1-2 2-2 1-3 2-3 3-3 1-4 2-4 3-4 4-4 1-5 3-5 Cantabrica 0,1159 0,2464 0,0580 0,3333 0,0725 0,0870 0,0290 0,0145 0,0000 0,0000 0,0145 0,0290 Pirenaica 0,1212 0,2121 0,0303 0,4848 0,0000 0,0606 0,0606 0,0000 0,0303 0,0000 0,0000 0,0000 Mediterranea 0,1458 0,2292 0,1042 0,2500 0,0625 0,1250 0,0417 0,0208 0,0000 0,0000 0,0000 0,0208 Meseta 0,2083 0,1250 0,0625 0,2708 0,1354 0,0729 0,0521 0,0208 0,0313 0,0000 0,0104 0,0104 Andalusia 0,0357 0,2143 0,0714 0,3214 0,0357 0,1429 0,1071 0,0357 0,0000 0,0357 0,0000 0,0000
Tabella A. 31.: Approccio geografico: frequenze genotipiche del locus D8S591.
FREQUENZE ALLELICHE LOCUS D8S591 Alleli Popolazioni 01 02 03 04 05 Zona Cantabricaa 0,428 0,225 0,304 0,022 0,022 Zona Pirenaica 0,500 0,136 0,318 0,045 0,000 Zona Mediterraneanea 0,406 0,260 0,292 0,031 0,010 Meseta 0,438 0,203 0,297 0,052 0,010 Andalusia 0,357 0,214 0,321 0,107 0,000
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D8S591
Pop 1 Pop 2 P-value S.E.
Cantabrica Pirenaica 0.35880 0.00408 Cantabrica Mediterranea 0.90885 0.00177 Cantabrica Meseta 0.61417 0.00484 Cantabrica Andalusia 0.12633 0.00271 Pirenaica Mediterranea 0.28699 0.00376 Pirenaica Meseta 0.74587 0.00359 Pirenaica Andalusia 0.27790 0.00291 Mediterranea Meseta 0.80369 0.00274 Mediterranea Andalusia 0.32756 0.00394 Meseta Andalusia 0.51058 0.00384
Tabella A.33.: Approccio geografico: differenziazione gentipica a coppie di popolazioni
calcolata in base alle frequenze genotipiche per il sistema D8S591.
DIFFERENZIAZIONE GENICA Locus D8S591
Pop 1 Pop 2 P-value S.E.
Cantabrica Pirenaica 0.35880 0.00408 Cantabrica Mediterranea 0.90885 0.00177 Cantabrica Meseta 0.61417 0.00484 Cantabrica Andalusia 0.12633 0.00271 Pirenaica Mediterranea 0.28699 0.00376 Pirenaica Meseta 0.74587 0.00359 Pirenaica Andalusia 0.27790 0.00291 Mediterranea Meseta 0.80369 0.00274 Mediterranea Andalusia 0.32756 0.00394 Meseta Andalusia 0.51058 0.00384
FREQUENZE ALLELICHE LOCUS D1S80 Alleli Pop 15 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 Galiziano 0,000 0,000 0,211 0,026 0,000 0,053 0,000 0,000 0,368 0,053 0,000 0,026 0,079 0,079 0,000 0,000 0,026 0,000 0,053 0,000 0,000 0,000 0,026 0,000 Basco 0,000 0,000 0,206 0,015 0,088 0,015 0,015 0,015 0,309 0,088 0,029 0,015 0,000 0,044 0,000 0,074 0,044 0,000 0,015 0,000 0,000 0,000 0,000 0,015 Catalano 0,000 0,000 0,211 0,079 0,000 0,026 0,079 0,000 0,237 0,079 0,000 0,026 0,105 0,026 0,053 0,026 0,000 0,000 0,000 0,026 0,026 0,000 0,000 0,000 Valenciano 0,043 0,000 0,304 0,000 0,000 0,109 0,022 0,022 0,217 0,087 0,022 0,000 0,043 0,043 0,065 0,022 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 Aragonese 0,000 0,000 0,321 0,018 0,018 0,000 0,000 0,000 0,375 0,036 0,018 0,018 0,018 0,054 0,036 0,036 0,000 0,018 0,018 0,000 0,000 0,000 0,018 0,000 Castigliano 0,000 0,014 0,243 0,014 0,054 0,027 0,041 0,014 0,351 0,027 0,027 0,000 0,027 0,027 0,014 0,095 0,000 0,014 0,000 0,000 0,000 0,014 0,000 0,000 Castigliano_sud 0,000 0,000 0,224 0,000 0,026 0,000 0,066 0,013 0,395 0,039 0,013 0,000 0,013 0,079 0,026 0,092 0,013 0,000 0,000 0,000 0,000 0,000 0,000 0,000 Murciano 0,000 0,022 0,217 0,043 0,000 0,022 0,109 0,000 0,261 0,022 0,022 0,000 0,000 0,043 0,065 0,087 0,022 0,022 0,000 0,000 0,000 0,043 0,000 0,000 Andaluso 0,000 0,016 0,234 0,031 0,016 0,031 0,047 0,000 0,406 0,016 0,031 0,016 0,063 0,000 0,031 0,016 0,031 0,000 0,000 0,000 0,000 0,016 0,000 0,000 Totale 0,004 0,006 0,241 0,022 0,028 0,028 0,042 0,008 0,334 0,047 0,020 0,010 0,034 0,043 0,030 0,055 0,016 0,006 0,008 0,002 0,002 0,008 0,004 0,002
TAVOLE DEL TERZO APPROCCIO SPERIMENTALE: A36-A50.
LEGENDA
ABBREVIAZIONI AREE LINGUISTICHE DI PROVENIENZA
DEL CAMPIONE
Galiziano Lingua: Galego-Portoghese
Basco Lingua:Euskera (Basco)
Catalano Lingua:Catalano
Valenziano Valenziano (dialetto del catalano)
Aragonese dialetto Aragonese
Castigliano Lingua:Castigliano (Spagnolo)
Castigliano_sud Castigliano del sud
Murciano dialetto Murciano
Andaluso dialetto Andaluso
EQUILIBRIO H-W EQUILIBRIO H-W
D8S566 D8S591
Pop P-val Pop P-val
Galiziano 0,9165 Galiziano 0,3371 Basco 0,7301 Basco 0,2349 Catalano 0,5959 Catalano 0,0120 Valenziano 0,7496 Valenziano 0,5279 Aragonese 0,4087 Aragonese 0,3786 Castigliano 0,5372 Castigliano 0,8105 Castigliano_sud 0,9106 Castigliano_sud 0,9023 Murciano 0,5454 Murciano 0,8293 Andaluso 0,1017 Andaluso 0,0051 EQUILIBRIO H-W EQUILIBRIO H-W D4S111 D1S80
Pop P-val Pop P-val
Galiziano 0,0766 Galiziano 0,6714 Basco 0,9206 Basco 0,4469 Catalano 0,0596 Catalano 0,0102 Valenziano 0,3044 Valenziano 0,0135 Aragonese 0,8963 Aragonese 0,0961 Castigliano 0,3115 Castigliano 0,1404
EQUILIBRIO H-W D17S5 Pop P-val Galiziano 0.0000 Basco 0.0000 Catalano 0.0000 Valenziano 0.0000 Aragonese 0.0000 Castigliano 0.0000 Castigliano_sud 0.0000 Murciano 0.0000 Andaluso 0.0000
Tabella A.36.: Approccio linguistico: test Hardy-Weinberg per ciascun locus
FREQUENZE ALLELICHE LOCUS D8S566 Alleli Pop 1 2 3 4 5 6 Galiziano 0,045 0,045 0,500 0,364 0,045 0,000 Basco 0,019 0,130 0,426 0,370 0,056 0,000 Catalano 0,000 0,114 0,318 0,500 0,068 0,000 Valenzianoo 0,036 0,036 0,536 0,286 0,107 0,000 Aragonese 0,058 0,077 0,212 0,481 0,135 0,038 Castiglianono 0,037 0,074 0,315 0,519 0,056 0,000 Castiglianono_sud 0,017 0,117 0,467 0,350 0,050 0,000 Murciano 0,053 0,132 0,395 0,316 0,079 0,026 Andaluso 0,048 0,167 0,286 0,381 0,119 0,000 Totale 0,033 0,104 0,371 0,406 0,079 0,008
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D1S80
Pop 1 Pop 2 P-value S.E.
Basco Galiziano 0.15350 0.00318 Catalano Galiziano 0.56055 0.00330 Catalano Basco 0.02988 0.00097 Valenziano Galiziano 0.32992 0.00288 Valenziano Basco 0.03952 0.00150 Valenziano Catalano 0.66105 0.00382 Aragonese Galiziano 0.76482 0.00289 Aragonese Basco 0.41319 0.00432 Aragonese Catalano 0.31716 0.00332 Aragonese Valenziano 0.32828 0.00370 Castigliano Galiziano 0.12599 0.00285 Castigliano Basco 0.69926 0.00366 Castigliano Catalano 0.35507 0.00390 Castigliano Valenziano 0.37396 0.00468 Castigliano Aragonese 0.80611 0.00275 Castigliano_sud Galiziano 0.01582 0.00078 Castigliano_sud Basco 0.36459 0.00381 Castigliano_sud Catalano 0.04519 0.00154 Castigliano_sud Valenziano 0.05298 0.00174 Castigliano_sud Aragonese 0.53891 0.00398 Castigliano_sud Castigliano 0.91344 0.00201 Murciano Galiziano 0.03722 0.00117 Murciano Basco 0.06783 0.00176 Murciano Catalano 0.55221 0.00334 Murciano Valenziano 0.22266 0.00305 Murciano Aragonese 0.28050 0.00332 Murciano Castigliano 0.80082 0.00304 Murciano Castigliano_sud 0.34654 0.00416 Andaluso Galiziano 0.43528 0.00369 Andaluso Basco 0.07975 0.00179 Andaluso Catalano 0.73503 0.00326 Andaluso Valenziano 0.29314 0.00411 Andaluso Aragonese 0.51278 0.00369 Andaluso Castigliano 0.80457 0.00331 Andaluso Castigliano_sud 0.11959 0.00271 Andaluso Murciano 0.58215 0.00366
Tabella A.37.: Approccio linguistico : differenziazione genotipica a coppie di
popolazioni condotta sulla base delle frequenze genotipiche per il sistema D1S80.
Tabella A.38.: Approccio linguistico : differenziazione genica a coppie di popolazioni
basata sulle frequenze alleliche per il sistema D1S80.
DIFFERENZIAZIONE GENICA Locus D1S80
Pop 1 Pop 2 P-value S.E.
Galiziano Basco 0.16361 0.00434 Galiziano Catalano 0.45516 0.00454 Galiziano Valenziano 0.24566 0.00476 Galiziano Aragonese 0.59010 0.00521 Galiziano Castigliano 0.10492 0.00459 Galiziano Castigliano_sud 0.02121 0.00176 Galiziano Murciano 0.04060 0.00182 Galiziano Andaluso 0.40653 0.00564 Basco Catalano 0.02007 0.00144 Basco Valenziano 0.02335 0.00176 Basco Aragonese 0.37751 0.00654 Basco Castigliano 0.65495 0.00574 Basco Castigliano_sud 0.31562 0.00570 Basco Murciano 0.06405 0.00320 Basco Andaluso 0.04721 0.00235 Catalano Valenziano 0.38844 0.00485 Catalano Aragonese 0.12203 0.00376 Catalano Castigliano 0.11904 0.00411 Catalano Castigliano_sud 0.01612 0.00117 Catalano Murciano 0.48754 0.00511 Catalano Andaluso 0.46800 0.00609 Valenziano Aragonese 0.15681 0.00407 Valenziano Castigliano 0.12243 0.00400 Valenziano Castigliano_sud 0.01142 0.00078 Valenziano Murciano 0.12815 0.00361 Valenziano Andaluso 0.09184 0.00338 Aragonese Castigliano 0.75051 0.00490 Aragonese Castigliano_sud 0.46782 0.00708 Aragonese Murciano 0.16848 0.00431 Aragonese Andaluso 0.45321 0.00549 Castigliano Castigliano_sud 0.87479 0.00304 Castigliano Murciano 0.61128 0.00438 Castigliano Andaluso 0.66409 0.00548 Castigliano_sud Murciano 0.26923 0.00607 Castigliano_sud Andaluso 0.07808 0.00314 Murciano Andaluso 0.35825 0.00481
FREQUENZE GENOTIPICHE LOCUS D4S111 Genotipi Pop - - - -Galiziano 0,040000 0,240000 0,280000 0,000000 0,440000 0,000000 Basco 0,000000 0,222222 0,277778 0,083333 0,333333 0,083333 Catalano 0,096774 0,193548 0,354839 0,000000 0,290323 0,064516 Valenziano 0,000000 0,296296 0,370370 0,000000 0,296296 0,037037 Aragonese 0,000000 0,166667 0,466667 0,066667 0,266667 0,033333 Castigliano 0,050000 0,125000 0,400000 0,100000 0,300000 0,025000 Castigliano_sud 0,105263 0,184211 0,368421 0,026316 0,236842 0,052632 Murciano 0,038462 0,269231 0,230769 0,000000 0,384615 0,076923 Andaluso 0,029412 0,058824 0,323529 0,058824 0,441176 0,088235 Totale 0,041812 0,188153 0,344948 0,041812 0,327526 0,052265
Tabella A.39.: Approccio linguistico: frequenze genoripiche per il locus D4S111.
FREQUENZE ALLELICHE LOCUS D4S111 Alleli Pop 1 2 3 Galiziano 0,160 0,620 0,220 Basco 0,153 0,556 0,292 Catalano 0,194 0,597 0,210 Valenzianoo 0,148 0,667 0,185 Aragonese 0,117 0,683 0,200 Castiglianono 0,163 0,613 0,225 Castiglianono_sud 0,211 0,579 0,197 Murciano 0,173 0,558 0,269 Andaluso 0,088 0,574 0,338
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D4S111
Pop 1 Pop 2 P-val S.E.
Basco Galiziano 0.66038 0.00195 Catalano Galiziano 0.93209 0.00072 Catalano Basco 0.57688 0.00241 Valenziano Galiziano 0.90723 0.00089 Valenziano Basco 0.32199 0.00255 Valenziano Catalano 0.76439 0.00161 Aragonese Galiziano 0.76014 0.00196 Aragonese Basco 0.29711 0.00256 Aragonese Catalano 0.52990 0.00275 Aragonese Valenziano 0.91353 0.00082 Castigliano Galiziano 1 0.00000 Castigliano Basco 0.66500 0.00240 Castigliano Catalano 0.92417 0.00090 Castigliano Valenziano 0.84279 0.00116 Castigliano Aragonese 0.66233 0.00207 Castigliano_sud Galiziano 0.88071 0.00124 Castigliano_sud Basco 0.40752 0.00291 Castigliano_sud Catalano 0.98743 0.00029 Castigliano_sud Valenziano 0.70971 0.00251 Castigliano_sud Aragonese 0.40524 0.00313 Castigliano_sud Castigliano 0.73251 0.00230 Murciano Galiziano 0.80533 0.00141 Murciano Basco 0.96586 0.00045 Murciano Catalano 0.80665 0.00144 Murciano Valenziano 0.48795 0.00203 Murciano Aragonese 0.41944 0.00278 Murciano Castigliano 0.82893 0.00129 Murciano Castigliano_sud 0.78091 0.00185 Andaluso Galiziano 0.30016 0.00239 Andaluso Basco 0.50627 0.00295 Andaluso Catalano 0.14014 0.00197 Andaluso Valenziano 0.12487 0.00167 Andaluso Aragonese 0.22832 0.00216 Andaluso Castigliano 0.22583 0.00227 Andaluso Castigliano_sud 0.07620 0.00171 Andaluso Murciano 0.36654 0.00214
Tabella A.41.: Approccio linguistico: differenziazione genotipica a coppie di
popolazioni calcolata in base alle frequenze genotipiche.
DIFFERENZIAZIONE GENICA Locus D4S111
Pop 1 Pop 2 P-value S.E.
Galiziano Basco 0.68644 0.00201 Galiziano Catalano 0.92763 0.00072 Galiziano Valenziano 0.91903 0.00072 Galiziano Aragonese 0.78311 0.00150 Galiziano Castigliano 100.000 0.00000 Galiziano Castigliano_sud 0.89044 0.00112 Galiziano Murciano 0.82239 0.00135 Galiziano Andaluso 0.26763 0.00246 Basco Catalano 0.55431 0.00288 Basco Valenziano 0.37066 0.00241 Basco Aragonese 0.33735 0.00291 Basco Castigliano 0.64898 0.00253 Basco Castigliano_sud 0.38419 0.00350 Basco Murciano 0.96802 0.00037 Basco Andaluso 0.50612 0.00249 Catalano Valenziano 0.74512 0.00175 Catalano Aragonese 0.49449 0.00229 Catalano Castigliano 0.91513 0.00078 Catalano Castigliano_sud 0.98385 0.00031 Catalano Murciano 0.78465 0.00174 Catalano Andaluso 0.10037 0.00191 Valenziano Aragonese 0.91932 0.00066 Valenziano Castigliano 0.84684 0.00116 Valenziano Castigliano_sud 0.70008 0.00265 Valenziano Murciano 0.50934 0.00255 Valenziano Andaluso 0.12903 0.00200 Aragonese Castigliano 0.65506 0.00215 Aragonese Castigliano_sud 0.36456 0.00383 Aragonese Murciano 0.41146 0.00231 Aragonese Andaluso 0.22428 0.00268 Castigliano Castigliano_sud 0.70346 0.00282 Castigliano Murciano 0.79794 0.00169 Castigliano Andaluso 0.20241 0.00254 Castigliano_sud Murciano 0.76293 0.00222 Castigliano_sud Andaluso 0.04732 0.00156 Murciano Andaluso 0.32749 0.00276
Tabella A.42.: Approccio linguistico: differenziazione genica a coppie di popolazioni
basata sulle frequenze alleliche del locus D4S111.
FREQUENZE GENOTIPICHE LOCUS D8S566 Genotipi Pop 1-2 2-2 1-3 2-3 3-3 1-4 2-4 3-4 4-4 2-5 3-5 4-5 5-5 4-6 Galiziano 0,000000 0,000000 0,000000 0,090909 0,181818 0,090909 0,000000 0,454545 0,090909 0,000000 0,090909 0,000000 0,000000 0,000000 Basco 0,000000 0,000000 0,000000 0,148148 0,148148 0,037037 0,074074 0,333333 0,148148 0,037037 0,074074 0,000000 0,000000 0,000000 Catalano 0,000000 0,045455 0,000000 0,045455 0,090909 0,000000 0,090909 0,318182 0,272727 0,000000 0,090909 0,045455 0,000000 0,000000 Valenziano 0,000000 0,000000 0,071429 0,071429 0,142857 0,000000 0,000000 0,428571 0,071429 0,000000 0,214286 0,000000 0,000000 0,000000 Aragonese 0,000000 0,000000 0,000000 0,115385 0,000000 0,115385 0,038462 0,192308 0,230769 0,000000 0,115385 0,076923 0,038462 0,076923 Castigliano 0,000000 0,000000 0,074074 0,037037 0,037037 0,000000 0,111111 0,370370 0,259259 0,000000 0,074074 0,037037 0,000000 0,000000 Castigliano_sud 0,000000 0,000000 0,033333 0,100000 0,233333 0,000000 0,133333 0,300000 0,100000 0,000000 0,033333 0,066667 0,000000 0,000000 Murciano 0,052632 0,000000 0,052632 0,105263 0,157895 0,000000 0,105263 0,263158 0,105263 0,000000 0,052632 0,000000 0,052632 0,052632 Andaluso 0,000000 0,000000 0,000000 0,190476 0,047619 0,095238 0,142857 0,285714 0,095238 0,000000 0,000000 0,047619 0,095238 0,000000
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D8S566
Pop 1 Pop 2 P-value S.E.
Basco Galiziano 0.83745 0.00133 Catalano Galiziano 0.41598 0.00194 Catalano Basco 0.67624 0.00240 Valenziano Galiziano 0.90068 0.00115 Valenziano Basco 0.48880 0.00252 Valenziano Catalano 0.18777 0.00224 Aragonese Galiziano 0.26914 0.00227 Aragonese Basco 0.06976 0.00153 Aragonese Catalano 0.20628 0.00226 Aragonese Valenziano 0.11030 0.00168 Castigliano Galiziano 0.75105 0.00169 Castigliano Basco 0.54584 0.00290 Castigliano Catalano 0.69411 0.00231 Castigliano Valenziano 0.25812 0.00230 Castigliano Aragonese 0.36438 0.00296 Castigliano_sud Galiziano 0.91863 0.00104 Castigliano_sud Basco 0.98811 0.00026 Castigliano_sud Catalano 0.46796 0.00287 Castigliano_sud Valenziano 0.55518 0.00217 Castigliano_sud Aragonese 0.02417 0.00083 Castigliano_sud Castigliano 0.39975 0.00243 Murciano Galiziano 0.87931 0.00132 Murciano Basco 0.84963 0.00133 Murciano Catalano 0.38393 0.00292 Murciano Valenziano 0.71243 0.00184 Murciano Aragonese 0.46473 0.00279 Murciano Castigliano 0.47167 0.00295 Murciano Castigliano_sud 0.76170 0.00212 Andaluso Galiziano 0.44603 0.00269 Andaluso Basco 0.58031 0.00273 Andaluso Catalano 0.47808 0.00269 Andaluso Valenziano 0.25552 0.00258 Andaluso Aragonese 0.50656 0.00275 Andaluso Castigliano 0.47445 0.00280 Andaluso Castigliano_sud 0.39535 0.00261 Andaluso Murciano 0.86778 0.00159
Tabella A.45.: Approccio linguistico : differenziazione genotipica a coppie di
popolazioni basate sulle frequenze genotipiche del sistema D8S566.
Tabella A.46.: Approccio linguistico: differenziazione genica a coppie di popolazioni
basata sulle frequenze alleliche per il sistema D8S566.
DIFFERENZIAZIONE GENICA Locus D8S566
Pop 1 Pop2 P-value S.E.
Galiziano Basco 0.82592 0.00157 Galiziano Catalano 0.33862 0.00243 Galiziano Valenziano 0.91506 0.00099 Galiziano Aragonese 0.26010 0.00278 Galiziano Castigliano 0.61372 0.00240 Galiziano Castigliano_sud 0.84056 0.00149 Galiziano Murciano 0.88851 0.00134 Galiziano Andaluso 0.37989 0.00259 Basco Catalano 0.66572 0.00283 Basco Valenziano 0.47348 0.00280 Basco Aragonese 0.06119 0.00180 Basco Castigliano 0.51935 0.00351 Basco Castigliano_sud 0.98304 0.00041 Basco Murciano 0.81210 0.00217 Basco Andaluso 0.51438 0.00301 Catalano Valenziano 0.11546 0.00203 Catalano Aragonese 0.30575 0.00312 Catalano Castigliano 0.82266 0.00187 Catalano Castigliano_sud 0.42359 0.00356 Catalano Murciano 0.35150 0.00335 Catalano Andaluso 0.49543 0.00345 Valenziano Aragonese 0.09240 0.00191 Valenziano Castigliano 0.18952 0.00281 Valenziano Castigliano_sud 0.50017 0.00282 Valenziano Murciano 0.72375 0.00243 Valenziano Andaluso 0.20305 0.00238 Aragonese Castigliano 0.42262 0.00376 Aragonese Castigliano_sud 0.01761 0.00087 Aragonese Murciano 0.36098 0.00279 Aragonese Andaluso 0.56333 0.00310 Castigliano Castigliano_sud 0.32028 0.00331 Castigliano Murciano 0.37346 0.00314 Castigliano Andaluso 0.39323 0.00286 Castigliano_sud Murciano 0.67431 0.00296 Castigliano_sud Andaluso 0.28050 0.00282 Murciano Andaluso 0.83438 0.00183
FREQUENZE GENOTIPICHE LOCUS D8S591 Genotipi Pop 1-1 1-2 2-2 1-3 2-3 3-3 1-4 2-4 3-4 4-4 1-5 3-5 Galiziano 0,181818 0,181818 0,090909 0,363636 0,136364 0,000000 0,000000 0,045455 0,000000 0,000000 0,000000 0,000000 Basco 0,114286 0,228571 0,057143 0,342857 0,028571 0,114286 0,028571 0,000000 0,000000 0,000000 0,028571 0,057143 Catalano 0,071429 0,285714 0,000000 0,500000 0,035714 0,035714 0,035714 0,000000 0,000000 0,000000 0,000000 0,035714 Valenziano 0,222222 0,185185 0,111111 0,185185 0,074074 0,111111 0,074074 0,037037 0,000000 0,000000 0,000000 0,000000 Aragonese 0,062500 0,250000 0,125000 0,250000 0,09375 0,125000 0,062500 0,000000 0,031250 0,000000 0,000000 0,000000 Castigliano 0,187500 0,156250 0,000000 0,343750 0,09375 0,031250 0,062500 0,031250 0,062500 0,000000 0,000000 0,031250 Castigliano_sud 0,257143 0,171429 0,057143 0,285714 0,114286 0,028571 0,057143 0,000000 0,028571 0,000000 0,000000 0,000000 Murciano 0,166667 0,125000 0,083333 0,208333 0,208333 0,125000 0,041667 0,000000 0,000000 0,000000 0,041667 0,000000 Andaluso 0,064516 0,193548 0,096774 0,322581 0,000000 0,16129 0,096774 0,032258 0,000000 0,032258 0,000000 0,000000 Totale 0,146617 0,199248 0,067669 0,31203 0,082707 0,082707 0,052632 0,015038 0,015038 0,003759 0,007519 0,015038
FREQUENZE ALLELICHE LOCUS D8S591 Alleli Pop 1 2 3 4 5 Galiziano 0,455 0,273 0,250 0,023 0,000 Basco 0,429 0,186 0,329 0,014 0,043 Catalano 0,482 0,161 0,321 0,018 0,018 Valenzianoo 0,444 0,259 0,241 0,056 0,000 Aragonese 0,344 0,297 0,313 0,047 0,000 Castiglianono 0,469 0,141 0,297 0,078 0,016 Castiglianono_sud 0,514 0,200 0,243 0,043 0,000 Murciano 0,375 0,250 0,333 0,021 0,021 Andaluso 0,371 0,210 0,323 0,097 0,000 Totale 0,432 0,216 0,295 0,045 0,011
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI
Locus D8S591
Pop 1 Pop 2 P-value S.E.
Basco Galiziano 0.43357 0.00282 Catalano Galiziano 0.66730 0.00210 Catalano Basco 0.88940 0.00133 Valenziano Galiziano 0.89822 0.00103 Valenziano Basco 0.24148 0.00229 Valenziano Catalano 0.37418 0.00276 Aragonese Galiziano 0.64725 0.00244 Aragonese Basco 0.15884 0.00222 Aragonese Catalano 0.20890 0.00241 Aragonese Valenziano 0.72981 0.00214 Castigliano Galiziano 0.25069 0.00255 Castigliano Basco 0.32556 0.00284 Castigliano Catalano 0.69326 0.00226 Castigliano Valenziano 0.47303 0.00306 Castigliano Aragonese 0.14913 0.00193 Castigliano_sud Galiziano 0.74209 0.00241 Castigliano_sud Basco 0.22019 0.00232 Castigliano_sud Catalano 0.57239 0.00267 Castigliano_sud Valenziano 0.84463 0.00175 Castigliano_sud Aragonese 0.27464 0.00252 Castigliano_sud Castigliano 0.56922 0.00306 Murciano Galiziano 0.83871 0.00127 Murciano Basco 0.87743 0.00124 Murciano Catalano 0.84845 0.00142 Murciano Valenziano 0.62899 0.00251 Murciano Aragonese 0.78528 0.00202 Murciano Castigliano 0.45115 0.00280 Murciano Castigliano_sud 0.41643 0.00278 Andaluso Galiziano 0.34077 0.00268 Andaluso Basco 0.11755 0.00213 Andaluso Catalano 0.21843 0.00243 Andaluso Valenziano 0.64517 0.00255 Andaluso Aragonese 0.60064 0.00281 Andaluso Castigliano 0.65294 0.00274 Andaluso Castigliano_sud 0.35946 0.00292 Andaluso Murciano 0.45816 0.00248