Sezione 3 – Capitolo I
Caratterizzazione morfologica e molecolare di
Euplotes parawoodruffi Song & Bradbury, 1997
e dei simbionti ad esso associati
Parte Prima
Introduzione
I.1.1: Caratteristiche generali del gen. Euplotes
Il genere Euplotes EHRENBERG 1830 (cl. Spirotrichea BÜTSCHLI 1889, sottocl. Hypotrichia
STEIN 1859) comprende oltre 70 specie, caratterizzate da una forma ovoidale più o meno allungata e da una marcata differenziazione dorsoventrale, di lunghezza compresa fra 30 e 240 μm. Presentano distinzione strutturale e funzionale fra ciliatura orale e somatica, nonché fra ciliatura somatica dorsale, a prevalente funzione sensoriale, e ventrale, deputata principalmente al movimento.
Il genere Euplotes appartiene alla classe Spirotrichea, che comprende le specie caratterizzate da una ciliatura somatica spesso ridotta e dalla presenza di membranelle adorali (AZM) nella regione apicale del corpo cellulare, poste marginalmente alla regione peristomale. La funzione di tali membranelle è prevalentemente connessa alla nutrizione, convogliando le particelle alimentari verso il citostoma onde consentirne l’ingestione (Verni e Rosati, 1982; Verni e Rosati, 1986). Le AZM sono presenti in altre classi (es. Oligohymenophorea); altri gruppi di protozoi ciliati possano inoltre presentare strutture funzionalmente analoghe (Dragesco, 1999). All’interno del gen. Euplotes, esse costituiscono un carattere morfologico tassonomicamente rilevante. Come in tutti i generi appartenenti a questa sottoclasse, le specie appartenenti al gen. Euplotes presentano tipicamente una ciliatura somatica dorsale ridotta, consistente in file di corte cinezie con probabile funzione sensoriale. Il numero di cinezie dorsolaterali è variabile da specie a specie e costituisce anch’esso un carattere tassonomico. La ciliatura somatica ventrale è invece ben sviluppata: le cilia sono organizzate in grandi fasci a funzione locomotoria (cirri), distinguibili per la loro posizione in cirri frontali, ventrali (questi due gruppi vengono spesso accorpati nel singolo gruppo “frontoventrali”), trasversali e caudali. Il numero e la disposizione dei cirri sulla superficie
ventrale di Euplotes sono ampiamente usati come caratteri tassonomici. In particolare, il numero dei cirri frontoventrali è il marcatore più utilizzato per la determinazione delle specie in quanto, benché variabile da specie e specie, rimane notevolmente costante in ognuna di esse. La maggior parte degli Euplotes possiede nove o dieci cirri frontoventrali, la cui disposizione può variare secondo diversi pattern di distribuzione; alcune specie ne hanno
otto; una soltanto ne ha sette (E. raikovi AGAMALIEV, 1966). Caratteristica distintiva del
genere è la presenza di un apparato nucleare costituito da un unico frammento macronucleare affusolato, disposto lungo l’intero corpo cellulare e presentante una caratteristica forma a “C” rovesciata o, più raramente, a epsilon o a “T”. Il micronucleo appare solitamente piccolo e sferico, esternamente posto rispetto al macronucleo. L’argiroma dorsale, un altro carattere largamente impiegato per la classificazione delle specie di Euplotes, viene evidenziato mediante colorazioni cellulari specifiche, quali l’impregnazione argentica, e corrisponde al disegno delle giunzioni fra le varie placche alveolari al di sotto della superficie dorsale del ciliato, evidenziando quindi la disposizione delle stesse. Sono stati ad oggi descritti sei diversi tipi di argiroma dorsale.
Ciliati del genere Euplotes sono presenti e talvolta molto comuni in una grande varietà di ambienti naturali, tanto marini o salmastri che dulciacquicoli. Vivono generalmente a contatto col substrato e possono presentare un marcato tigmotattismo. Le loro dimensioni li identificano come organismi meiobentonici (Nybakken, 1988); fanno comunemente parte della fauna mesopsammica degli ambienti a sedimento grossolano. In condizioni ambientali adeguate, la maggior parte delle specie può riprodursi a ritmi elevati: per alcuni autori (Carey, 1992) esse sono da considerarsi specie r-strateghe.
- Batteri simbionti in Euplotes
La presenza di batteri endosimbionti in alcune specie di Euplotes fu segnalata per la prima volta oltre mezzo secolo fa (Fauré-Frémiet, 1952; in Görtz, 2003). Nel 1975, Klaus Heckmann descrisse il simbionte “omikron” di Euplotes aediculatus come un batterio G-, di forma allungata e dalla lunghezza di circa 3,5-5 μm, presenti in grandi quantità (900-1000 individui per cellula ospite) nel citoplasma del protozoo. Caratteristica peculiare dell’organismo era la presenza di un numero variabile (3-9) di nucleoidi elettrondensi, composti probabilmente di acidi nucleici condensati, distribuiti ad intervalli regolari nel citoplasma. Il medesimo studio dimostrò che la rimozione sperimentale di tale organismo mediante trattamento con penicillina influiva negativamente sulla crescita dell’ospite, rendendo le cellule aposimbiotiche incapaci di dividersi e, in alcuni casi, determinandone la morte. Le cellule acquisivano nuovamente la capacità di dividersi in seguito a reinfezione sperimentale, ottenuta ponendo le cellule aposimbiotiche in contatto con omogenati cellulari di E. aediculatus non trattati con antibiotico. Organismi simili sono stati in seguito descritti
in altre specie dulciacquicole di Euplotes (Heckmann et al., 1983): le specie ospitanti batteri
omikron-simili risultavano essere caratterizzate dal possedere nove cirri frontoventrali
disposti come nella specie di riferimento E. patella (si tratta del cosiddetto “9 type 1
fronto-ventral cirrus pattern”). Fra tali specie risulta compreso anche E. woodruffi (Heckmann et al., 1983). In alcuni dei ceppi analizzati era presente un simbionte diverso da omikron, a cui
si era sostituito funzionalmente (Heckmann et al., 1983). Esperimenti di aposimbiosi e reinfezione condotti sulle specie presentanti il pattern “9 type 1” hanno confermato per ciascuna di esse la dipendenza dalla presenza del simbionte (Heckmann et al., 1983). Lavori successivi (Fujishima and Heckmann, 1984) hanno inoltre dimostrato che la reinfezione con endosimbionti omikron-simili di altre specie di Euplotes (in part., cellule aposimbionti di E.
aediculatus reinfettate con simbionti di E. woodruffi acquisivano nuovamente la capacità di
dividersi). Ciò indica una identica funzione di tali organismi nelle differenti specie di
Euplotes. L’adattamento del batterio stesso alla vita simbiotica era suggerito anche dalle
ridotte dimensioni del genoma (Schmidt, 1982), cosa che si verifica in molti organismi endocitobionti obbligati. I numerosi tentativi di coltivare il batterio al di fuori dell’ospite hanno dato esito negativo (Heckmann, 1975). Ciò ha condotto all’ipotesi che il gruppo di specie di Euplotes caratterizzate da un pattern “9 type 1” condivida una deficienza metabolica in grado di influire sul meccanismo di divisione cellulare, ereditata da un antenato comune. Tale gruppo si sarebbe pertanto evoluto da un organismo ancestrale nel quale la presenza del batterio, simbionte obbligato, già suppliva a carenze metaboliche che ne avrebbero impedito la crescita (Heckmann et al., 1983, Fujishima and Heckmann, 1984); i batteri omikron-simili rappresenterebbero la progenie del simbionte ancestrale. Tali batteri
sono stati riuniti nel genere Polynucleobacter HECKMANN AND SCHMIDT 1987, collocato
all’interno della classe Betaproteobacteria. La sequenza del gene codificante per il 16S
rRNA della specie-tipo Polynucleobacter necessarius HECKMANN AND SCHMIDT 1987,
simbionte di E. aediculatus (precedentemente noto come “omikron”), è stata caratterizzata nel 1996 (Springer et al., 1996). Il genere Polynucleobacter comprende anche forme a vita libera, di recente caratterizzazione, facenti parte del bacterioplancton dulciacquicolo di acque ferme e caratterizzati da una bassa diversità genetica (Hahn et al., 2005). Batteri omikron-simili sono stati ritrovati anche in una specie di Euplotes con cirrotipo diverso dal “9 type 1” (E. harpa: Vannini et al., 2005a): la presenza di questo tipo di simbionti potrebbe pertanto rappresentare un carattere plesiomorfico condiviso da un gruppo di specie di Euplotes filogeneticamente correlate (a dispetto delle differenze di morfologia e cirrotipo, alcuni marcatori molecolari indicano una parentela fra le specie E. harpa ed E. aediculatus: Petroni
et al., 2002). All’interno del cosiddetto “Polynucleobacter cluster”, clade che comprende
attualmente oltre 180 sequenze batteriche, sono distinguibili quattro sottogruppi monofiletici (subcluster A, B, C e D): la percentuale di similarità delle sequenze di 16S rDNA all’interno
di ciascun cluster è maggiore del 97% (Hahn et al., 2005; Vannini et al., in press). Cinque delle sequenze di simbionti P. necessarius-simili ad oggi caratterizzate (da differenti ceppi di
E. aediculatus ed E. harpa) risultano collocarsi all’interno del subcluster C (Vannini et al., in press), presentando poche differenze (la più bassa è dello 0,5%) con quelle di batteri a vita
libera. Studi recenti hanno comunque confermato che i tre gruppi di specie riuniti nel
subcluster C (batteri a vita libera, simbionti obbligati di E. aediculatus, simbionti obbligati di E. harpa) rappresentano entità distinte, con ruoli ecologici differenti e nicchie ecologiche
completamente separate (Vannini et al., in press). La relazione filogenetica fra questi gruppi è al momento la più stretta mai identificata fra organismi simbionti obbligati ed organismi a vita libera, suggerendo uno sviluppo relativamente recente del rapporto simbiotico fra questi organismi e ciliati del genere Euplotes (Vannini et al., in press).
Alcune specie appartenenti al genere Euplotes e provviste del pattern “9 type 1” possiedono simbionti diversi da Polynucleobacter spp., oltre ad ospitare quest’ultimo. I dati concernenti questi simbionti secondari ad oggi disponibili sono scarsi: si tratta perlopiù di procarioti eterotrofi appartenenti a differenti classi batteriche, ma sono stati individuati anche zooclorelle (Heckmann et al., 1983) e microsporidi (Fokin et al., 2006). In certi casi è stata osservata la perdita spontanea del simbionte secondario in ceppi di Euplotes allevati in condizioni di laboratorio. La presenza di simbionti secondari è stata segnalata in varie specie di Euplotes provviste del pattern “9 type 1” (E. eurystomus, E. daidaleos, E. patella, E.
octocarinatus: Heckmann et al., 1983; Vannini, unpubl.).
Anche le specie di Euplotes che non possiedono Polynucleobacter spp. possono essere associate, talvolta in modo stabile, a batteri simbionti (Vannini et al., 2004).
Sezione 3 – Capitolo I
Parte Seconda
Risultati dell’analisi
I.2.1: Isolamento e messa in coltura di E. parawoodruffi
Esemplari di E. parawoodruffi sono stati individuati sia nei campionamenti effettuati nello Stagno 1 in data 02-10-2005 (Salinità 10‰) sia in quelli effettuati in data 16-10-2005 (Salinità 4‰). Dai campioni raccolti in quest’ultima data sono state prelevate le cellule destinate alla coltivazione. Le cellule raccolte sono state isolate in acqua di mare sintetica a salinità 4‰.
Sono stati effettuati sei tentativi di stabilire colture monoclonali di tali organismi. Alle cellule isolate è stata somministrata come alimento l’alga verde Dunaliella salina allevata in acqua di mare sintetica a salinità 5‰, in quantità proporzionali al numero di cellule presenti in coltura, a cadenza settimanale o bisettimanale. Tale procedura è quella comunemente più seguita per l’allevamento di Euplotes spp. Tutti i tentativi di coltura hanno dato esito positivo, dimostrando una relativa facilità di allevamento di tale specie. Due dei sei ceppi monoclonali così ottenuti (OS 52/1 e OS 52/3) sono stati selezionati per l’ottenimento di colture massive. E’ stato possibile ottenere in poco più di un mese colture cellulari da 800-1000 ml. Tali colture vengono mantenute in cella termostatata a 19±1°C, con alternanza luce-buio di 12 ore, e cibate con Dunaliella salina a salinità 5‰ con cadenza settimanale. Le colture così mantenute sono stabili per lunghi periodi.
I.2.2: Caratterizzazione morfologica
Cellule del ceppo OS 52/1 sono state utilizzate per le procedure di caratterizzazione morfologica volte alla determinazione della specie (cfr. Sezione 1: Introduzione - Approccio
sperimentale utilizzato per l’identificazione dell’ospite e l’individuazione di eventuali simbionti).
L’osservazione in vivo ha mostrato cellule di dimensioni medie approssimative di circa 120 x 90 μm, di forma ovoidale, leggermente slargata e squadrata nella parte anteriore. La regione citostomale si presenta di forma svasata, trapezoidale: nella parte frontale occupa l’intera larghezza della cellula e si estende per circa 4/5 del corpo lungo la superficie ventrale. Le
membranelle adorali sono in numero di 70-80. L’estremità distale delle AZM decorre parallelamente al margine della cellula, piegando poi verso il lato ventrale e prendendo rapporto con i cirri frontoventrali anteriori; l’estremità prossimale si assottiglia gradatamente e termina presso il citostoma, vicino al primo (a destra) dei cirri trasversali. Sono presenti 9 cirri frontoventrali, 5 trasversali e 4 caudali: la disposizione dei cirri frontoventrali appare simile a quella di E. patella (“9 type 1 cirrus pattern”). Il corpo cellulare si presenta appiattito dorsoventralmente; la regione dorsale appare poco convessa. Il citoplasma è di aspetto ialino, uniforme. Il vacuolo contrattile, piuttosto grande, si colloca a destra e in alto rispetto al più anteriore dei cirri trasversali.
L’apparato nucleare si compone di un unico grande frammento macronucleare ed un micronucleo. Il macronucleo è conformato in forma di tau, con la parte trasversale localizzata lungo l’estremità anteriore della cellula. Il micronucleo, più piccolo, è collocato esternamente ad esso, nella regione posteriore; in alcuni esemplari risultava debolmente colorato dalla fucsina. Tale forma e disposizione del nucleo sono caratteristiche della specie
E. woodruffi GAW 1939 e dell’affine E. parawoodruffi SONG AND BRADBURY 1997 (Inamori, 2006). In particolare, negli esemplari osservati il macronucleo presentava le due braccia distali di lunghezza significativamente diversa, con il braccio distale sinistro lungo circa il doppio del destro, e sempre inferiore alla lunghezza del braccio centrale. Questo carattere è proprio esclusivamente di E. parawoodruffi (Inamori 2006, Song and Bradbury 1997; per l’interpretazione esaustiva di tali risultati cfr. Parte Terza: Discussione dei risultati
ottenuti, presente capitolo).
Le cellule sottoposte ad impregnazione argentica hanno rivelato un argiroma dorsale tipo
“doppio-eurystomus” (cfr. E. eurystomus WRZESNIOWSKE 1870).
La specie è stata identificata come Euplotes parawoodruffi SONG AND BRADBURY 1997.
I.2.3: Caratterizzazione molecolare
E’ stata caratterizzata la sequenza del gene codificante per il 18S rRNA a partire da una estrazione di DNA condotta su cellule del ceppo OS 52/1. 10 ml di coltura sono stati centrifugati onde concentrare le cellule; l’estrazione è stata effettuata utilizzando il kit
“Nucleospin® Plant” (cfr. Sezione 2: Materiali e Metodi).
Per la reazione di PCR sono stati impiegati gli inneschi F9 (5’-CTG GTT GAT CCT GCC
AG-3’) e R1513hypo (5’-TGA TCC TTC YGC AGG TTC-3’). L’inserimento di un innesco
specifico per gli ipotrichi assieme ad uno universale ha ridotto i rischi di amplificazioni aspecifiche e/o contaminazioni. L’annealing è avvenuto a 57° C; l’estensione è stata prolungata per 2’ a 72° C.
Poiché la discriminazione morfologica fra E. woodruffi ed E. parawoodruffi presenta dei margini di incertezza, è stato deciso di caratterizzare l’intera sequenza genica, allo scopo di
visualizzare un maggior numero di regioni ipervariabili tali da permettere di distinguere le due specie. La sequenza ottenuta è stata confrontata con quelle presenti nella banca dati di riferimento (NCBI GenBank). È risultata molto simile a due sequenze precedentemente depositate: AF452710 (Chen and Song 2002), di E. woodruffi ceppo XXH (identità 1828/1830, gap assenti) ed AF452708 (Chen and Song 2002) di E. parawoodruffi (identità 1830/1832, gap assenti). Una terza sequenza di E. woodruffi (ceppo BHY) presente in banca dati (AF492707: Song 2002) ha dato un valore di identità più basso rispetto alle due precedenti (1828/1838, sei gap).
I.2.4: Individuazione e caratterizzazione del simbionte principale
E. woodruffi è una delle specie con cirrotipo “9 type 1” in cui era stata già descritta
l’associazione simbiotica con batteri del genere Polynucleobacter HECHMANN AND
SCHMIDT 1987 (Heckmann et al., 1983). La presenza di organismi simbionti era quindi attesa anche nella specie affine E. parawoodruffi. Tale specie condivide il medesimo pattern di cirri di E. woodruffi; inoltre, le due specie non sono state distinte sino al 1997: è possibile che parte dei dati disponibili in letteratura riguardo ad E. woodruffi siano in realtà da riferirsi ad E. parawoodruffi (Song and Bradbury, 1997). L’osservazione al microscopio ottico a contrasto interferenziale di cellule lisate ha confermato la presenza di endosimbionti all’interno delle cellule stesse. E’ stata pertanto eseguita una ibridazione in situ a bassa stringenza (0% f.a.) volta alla identificazione di tali organismi, impiegando sonde oligonucleotidiche a diversa specificità, fra cui una specifica per Polynucleobacter. Per confermare un eventuale risultato positivo di tale ibridazione, la procedura è stata ripetuta impiegando una sonda specifica per i β-proteobatteri, classe a cui il genere appartiene. La Tabella I.1 riassume i risultati ottenuti con tali ibridazioni.
Ibridazione Ceppo Sonda Fluorocromo Specificità n. Cellule Positive (%)
Eub338 Cy3 Eubacteria 16 100%
I OS52/1
Poly862 Fluoresceina Polynucleobacter
spp. 16 100%
(1)
II OS52/1 β23S1027 Fluoresceina Betaproteobacteria 12 100%
Note: (1): Un numero imprecisato di batteri, positivi ad Eub338, non risultavano marcati da Poly862. Tali batteri risultavano presenti nel 100% delle cellule osservate. Per la caratterizzazione di tale simbionte secondario vd. oltre.
Tabella I.1: Ibridazioni preliminari per l’identificazione del simbionte principale di E. parawoodruffi.
La penultima colonna a destra riporta il numero di cellule osservate; l’ultima la percentuale di cellule al cui interno sia stato individuato un qualsiasi numero di batteri marcati in modo specifico dalla sonda.
L’ibridazione preliminare ha confermato l’appartenenza del simbionte principale alla
sottoclasse Betaproteobacteria GARRITY, BELL AND LILBURN 2005; la positività alla sonda
genere-specifica ha reso possibile ipotizzare la sua collocazione in un genere filogeneticamente vicino a Polynucleobacter, se non all’interno del genere stesso.
- Caratterizzazione molecolare
Data l’appartenenza del simbionte alla cl. Betaproteobacteria, è stato possibile ottimizzare le procedure per l’amplificazione tramite PCR del gene per il 16S rRNA. Sono stati utilizzati gli inneschi βF19 e βR1517, entrambi specifici per tale sottoclasse. Come template è stato impiegato il DNA precedentemente estratto per la caratterizzazione molecolare dell’ospite (vd. sopra) dal ceppo OS 52/1. Onde amplificare selettivamente e in misura maggiore il gene dell’organismo bersaglio, è stata effettuata una reazione di touchdown PCR, con temperature di annealing di 63, 57 e 50° C. Il prodotto di reazione è stato sequenziato con i primer F343, R515 e F785, in modo da caratterizzare l’intero gene. Durante i tentativi di caratterizzazione del simbionte secondario presente nel medesimo ceppo (vd. oltre), è stato fatto ricorso a tecniche di clonaggio, che ha portato alla creazione di una library di cloni trasformati con inserti di 16S rDNA proveniente anche dal simbionte principale. Sono stati ottenuti, fra gli altri, 25 cloni presentanti un pattern di digestione analogo fra loro: è stato supposto che si trattasse di cloni trasformati con 16SrDNA del simbionte principale. L’inserto contenuto in quattro di questi cloni è stato sottoposto a sequenziamento, per confermare, eventualmente, il risultato ottenuto tramite il sequenziamento diretto. Due delle sequenze ottenute sono risultate chimeriche (vd. oltre); le altre due (cloni E5 ed E6) risultavano simili a quella del simbionte principale ottenuta mediante sequenziamento diretto. Sono state quindi confrontate fra di loro e con la sequenza del simbionte principale onde determinare la sequenza consensus. Le tre sequenze sono risultate identiche in tutte le posizioni.
Per effettuare l’analisi filogenetica è stata preparata una selezione di 26 specie appartenenti alla cl. Betaproteobacteria, fra le quali la sequenza consensus (ottenuta da tre diversi cloni) del 16S rRNA di un batterio, omikron-dissimile poiché privo di nucleoidi , precedentemente riconosciuto come simbionte principale di E. octocarinatus ceppo FL(12)-VI. La selezione comprende inoltre le sequenze di due organismi Polynucleobacter-simili simbionti di E.
harpa ed E. octocarinatus ceppo Fan10 (quest’ultima sequenza è una consensus ottenuta da
tre diversi cloni), quella di P. necessarius HECKMANN AND SCHMIDT 1987 simbionte di E.
aediculatus, e sei sequenze di batteri a vita libera filogeneticamente riconducibili al gen. Polynucleobacter spp., appartenenti rispettivamente ai subcluster A (Clone LWSR-76,
AY345532; Clone TLM05F05, AF534432), B (Clone 243ds10, AY212691; Clone LD17, Z99998), C (MWH HuW1, AJ550666; MWH HuK1, AJ550665) e D (MWH Cak5, AJ550655; MWH BeK4, AJ550656) (Wu Quigong and Hahn, 2005). La selezione
comprende inoltre alcune specie appartenenti alla medesima famiglia di Polynucleobacter (fam. Burkholderiaceae), comprese nei genn. Burkholderia, Ralstonia, Wautersia e
Pandoraea. Come outgroup sono state utilizzate sequenze di specie comprese nell’ordine Burchkolderiales: Comamonas terrigena (fam. Comamonadaceae), Achromobacter xyloloxydans (fam. Alcaligenaceae), Acidovorax facilis (fam. Comamonadaceae), Alcaligenes faecalis (fam. Alcaligenaceae)
Sono stati elaborati quattro alberi filogenetici con il Neighbor-joining method: per due di essi è stata attivato un filtro specifico per i β-proteobatteri; gli altri sono stati ottenuti combinando tale filtro con un altro di classe “termini”. Per ciascuna delle combinazioni di filtri sono state applicate, rispettivamente, la correzione di Felsenstein e quella di Jukes-Cantor. Tutti gli alberi realizzati con tale metodo sono risultati presentare la medesima topologia, a prescindere dalle combinazioni di filtri e correzioni adottate. Le sequenze di
Polynucleobacter spp., simbionti o a vita libera, risultano associate fra loro, formando un
gruppo monofiletico (i quattro subcluster risultano comunque distinguibili); sister group di tale clade è il cluster formato dai genn. Ralstonia e Wautersia (quest’ultimo è stato separato dal primo soltanto recentemente: Vaneechoutte et al., 2004). Il clade comprendente le sequenze del simbionte di OS 52/1 risulta basale a questo gruppo: al suo interno, la sequenza del simbionte principale di E. octocarinatus ceppo FL(12)-VI differisce da quelle di nuova caratterizzazione, collocandosi in posizione basale rispetto a queste ultime. La distanza fra le sequenze riportata sull’albero appare significativa. Sister group dei gruppi sin qui esposti è il clade comprendente i genn. Pandoraea e Burkholderia, stabilmente associati in tutti gli alberi.
Simile topologia hanno mostrato anche i due alberi calcolati secondo il criterio di
Maximum-likelihood, utilizzando la medesima selezione di specie ed applicando rispettivamente un
unico filtro specifico ed una combinazione di filtri (filtro β-proteobatteri-specifico e filtro “termini”). Nonostante la distanza evolutiva fra alcune sequenze risultasse in certi casi maggiore o minore di quanto calcolato col Neighbor-joining method, le posizioni relative di ciascun clade all’interno dell’albero sono rimaste stabili. Per quanto concerne il clade comprendente le sequenze in esame (denominato provvisoriamente “ParaPoly
cluster”), la posizione relativa e la distanza dalle altre specie sono risultate quasi invariate.
Sono stati realizzati due alberi basati sul Parsimony criterion, ottenuti sulla stessa selezione di specie. La combinazione di filtri utilizzata è la stessa riportata per il Maximum-likelihood
criterion. I valori di bootstrap sono stati calcolati sulla base di 1000 pseudorepliche. In
generale, le posizioni relative dei cladi comprendenti le varie famiglie dell’ordine
Burkholderiales sono apparse abbastanza stabili, relativamente alla selezione di specie
adottata (boostrap value maggiori del 70% per la maggior parte dei nodi). La disposizione è la stessa osservata negli alberi filognetici già descritti. L’associazione fra le sequenze del
simbionte di OS 52/1 e quella del simbionte di E. octocarinatus ceppo FL(12)-VI è risultata ben supportata (bootstrap value 100%), così come la posizione basale del clade rispetto a quello comprendente Ralstonia spp. e Polynucleobacter spp. (bootstrap value 91%-93%, variabile secondo la combinazione di filtri adottata). Nell’albero, una delle sequenze dei cloni si presenta basale rispetto all’altra e a quella ottenuta per sequenziamento diretto, che risultano associate: tale topologia è imputabile ad un artefatto del metodo di calcolo dell’albero stesso (le tre sequenze presentano infatti una identità del 100%). Questo artefatto è stato eliminato riparametrando la lunghezza dei rami in rapporto alle differenze fra le sequenze. L’albero così ottenuto è illustrato al termine del presente capitolo (Figura I.1).
- Ibridazione con sonde specie-specifiche disegnate sul 16S rRNA
Grazie alla differenza sussistente, a livello del marcatore utilizzato, fra l’organismo oggetto di indagine ed i batteri del genere Polynucleobacter precedentemente caratterizzati, è stato possibile disegnare due sonde specifiche per essi (per la procedura utilizzata, cfr. Sezione 2:
Materiali e Metodi). Le caratteristiche delle sonde sono di seguito riassunte:
-ParaPoly445: Specifica per batteri appartenenti al cosiddetto “ParaPoly cluster”, simbionti di Euplotes parawoodruffi ceppo OS 52/1 ed E. octocarinatus ceppo FL(12)-VI. Presenta almeno 1 mismatch con tutte le sequenze di Polynucleobacter depositate in banca dati.
Posizione: 445 E. coli. La posizione è stata precedentemente utilizzata per la costruzione delle sonde Polynucleobacter-specifiche PnecC e PnecABD (Hahn et al., 2005).
-ParaPoly467: Specifica per batteri appartenenti al cosiddetto “ParaPoly cluster”, simbionti di Euplotes parawoodruffi ceppo OS 52/1 ed E. octocarinatus ceppo FL(12)-VI. Presenta almeno 3 mismatch con tutte le altre sequenze di
Polynucleobacter depositate in banca dati. La sequenza differisce per 1
nucleotide da altre 34 sequenza presenti in banca dati, comprese nei gen.
Hydrogenophaga, Herbaspirilla, Aquaspirilla; differisce per 2 nucleotidi da
180 sequenze; differisce dalle altre per 3 o più nucleotidi. Posizione: 467 E. coli.
La Tabella I.2 riassume i risultati delle ibridazioni di controllo effettuate. Come sonde di controllo sono state prevalentemente impiegate Eub338 (marcata con fluoresceina), che si lega alla maggior parte degli eubatteri, e β23S_1027 (marcata con fluoresceina), specifica per i membri della sottoclasse Betaproteobacteria. E’ stato possibile effettuare le ibridazioni di controllo direttamente sul ceppo di E. parawoodruffi OS 52/1, in cui è presente un simbionte secondario appartenente alla sottoclasse Gammaproteobacteria (per la caratterizzazione di tale simbionte vd. oltre). Obiettivo dell’esperimento è stato determinare
le condizioni di stringenza necessarie al corretto funzionamento delle sonde (in questo caso, ottenendo il legame specifico con i simbionti omikron-simili evitando al contempo il legame aspecifico col simbionte secondario).
Ibridazione Ceppo Sonda Fluorocromo Specificità n. Cellule
Positive
ParaPoly467 Cy3 (vd. sopra) 10 100%(1)
I
(0% f.a.) OS52/1
Poly862 Fluoresceina Polynucleobacter
spp. 10 100%
ParaPoly467 Cy3 (vd. sopra) 7 100%(1)
II
(0% f.a.) OS52/1
β23S1027 Fluoresceina Betaproteobacteria 7 100%
ParaPoly467 Cy3 (vd. sopra) 16 100%(2)
III
(20% f.a.) OS52/1
β23S1027 Fluoresceina Betaproteobacteria 16 100%(2)
ParaPoly467 Cy3 (vd. sopra) Ca.20 100%
IV
(30% f.a.) OS52/1
β23S1027 Fluoresceina Betaproteobacteria Ca.20 0%(3)
ParaPoly467 Cy3 (vd. sopra) 4 0%(4)
V
(30% f.a.) OS52/1
β23S1027 Fluoresceina Betaproteobacteria 4 0%(4)
ParaPoly445 Fluoresceina (vd. sopra) 11 90,9%(6)
VII
(30% f.a.) OS52/1
PnecC Cy3 Polynucleobacter
subcl. “C” 11 0%
(5)
ParaPoly445 Fluoresceina (vd. sopra) 5 100%(7)
VIII
(0% f.a.) OS52/1
Eub338 Cy3 Eubacteria 5 100%(7)
ParaPoly445 Fluoresceina (vd. sopra) 8 0%(4)
IX
(30% f.a.) OS52/1
Note: (1): La sonda specie-specifica evidenziava batteri non marcati dalla sonda universale di controllo. Tali ibridazioni aspecifiche sono imputabili alle basse condizioni di stringenza utilizzata.
(2): Le due sonde hanno legato entrambe i medesimi batteri. La condizione di stringenza adottata ha impedito il legame aspecifico della sonda ParaPoly467 al simbionte secondario (vd. oltre).
(3): Il fallimento di tale ibridazione è stato imputato alle condizioni di stringenza non ottimali per la sonda impiegata. Non avendo la sonda di controllo come riferimento, l’esito della ibridazione con ParaPoly467 non può essere valutato.
(4): La condizione di stringenza adottata ha destabilizzato eccessivamente il legame della sonda.
(5): La sonda PnecC, specifica per i batteri del genere Polynucleobacter compresi all’interno del subcluster C (Hahn et al., 2005) è stata qui utilizzata come controllo negativo.
(6): Pochi batteri visibili nelle cellule positive. È probabile che le elevate condizioni di stringenza abbiano destabilizzato eccessivamente il legame.
(7): A queste condizioni è stato possibile distinguere i batteri marcati unicamente dalla sonda universale (simbionte secondario) da quelli marcati dalla sonda universale e da quella specifica (simbionte principale).
Tabella I.2: Ibridazioni di controllo effettuate per le sonde ParaPoly467 e ParaPoly445. La penultima
colonna a destra riporta il numero di cellule osservate; l’ultima la percentuale di cellule al cui interno sia stato individuato un qualsiasi numero di batteri marcati in modo specifico dalla sonda. La colonna a sinistra riporta il numero dell’ibridazione in questione e le condizioni di stringenza (espresse come percentuale di formammide nella soluzione di ibridazione) adottate.
I risultati delle ibridazioni di controllo indicano che la condizione di utilizzo ottimale per la sonda ParaPoly467 prevede la presenza del 20% di formammide nella miscela di ibridazione; la sonda ParaPoly445 ha dato buoni risultati anche in assenza di formammide. Tali sonde hanno confermato l’appartenenza della sequenza di 16S rDNA precedentemente ottenuta al simbionte procariotico principale isolato dal ceppo OS 52/1 di E. parawoodruffi; i dati fin qui raccolti sembrano indicare che esse, utilizzate alle condizioni indicate, ne permettano il riconoscimento specifico in situ. Ulteriori esperimenti di ibridazione sono però richiesti per accertare in maniera più rigorosa la specificità delle due sonde, in particolare verso batteri appartenenti al gen. Polynucleobacter.
- Indagine ultrastrutturale.
Sono stati realizzati alcuni preparati per il microscopio elettronico a trasmissione a partire da cellule del ceppo OS 52/1. È stato possibile osservare l’ultrastruttura dei simbionti presenti all’interno delle cellule. Il simbionte principale è collocato esclusivamente nello spazio cìitoplasmatico, contenuto in vacuoli rivestiti di membrana e spesso a stretto contatto con ribosomi. Non sono stato osservati batteri in divisione, né è stato possibile determinare con esattezza le dimensioni medie del batterio in quanto sono state ottenute poche sezioni longitudinali complete.
L’indagine ha invece evidenziato la completa assenza di nucleoidi all’interno del simbionte. Tali strutture sono state segnalate in tutti i batteri omikron-simili sinora caratterizzati (cfr.
Parte Terza: Discussione dei risultati ottenuti, presente capitolo). Osservazioni condotte su
più cellule ospiti, del ceppo OS 52/1 e di una aliquota del medesimo ceppo mantenuta in differenti condizioni di allevamento (risultata in seguito priva del simbionte secondario) hanno confermato tale dato.
I.2.5: Individuazione e caratterizzazione del simbionte secondario
Le ibridazioni preliminari effettuate con sonde ad ampio spettro (Eub338) in combinazione con sonde più specifiche per il simbionte primario (Poly862) hanno rivelato la presenza, oltre che del simbionte principale omikron-simile, di altri batteri endosimbionti. Questi ultimi, più piccoli e di forma meno allugata, venivano marcati positivamente dalla sonda universale per gli eubatteri, mentre non erano evidenziati dalla sonda Polynucleobacter-specifica. Un risultato analogo è stato in seguito ottenuto con la sonda ParaPoly445, specifica per le specie comprese nel “ParaPoly cluster”, utilizzata in combinazione con Eub338. La presenza di un simbionte secondario eubatterico nel ceppo OS 52/1 di E.
parawoodruffi è stata pertanto confermata. Sono state eseguite una serie di ibridazioni in situ
con sonde ad ampio spettro, finalizzate all’individuazione del gruppo tassonomico d’appartenenza dell’organismo onde poter ottimizzare le procedure per la caratterizzazione della sequenza 16S rRNA. Sono state altresì testate alcune sonde genere-specifiche precedentemente disegnate per altri batteri endosimbionti: la positività di queste ultime ibridazioni avrebbe consentito l’immediata identificazione del genere di appartenenza del simbionte secondario.
La Tabella I.3 riporta gli esiti delle ibridazioni in situ eseguite.
Ibridazione Ceppo Sonda Fluorocromo Specificità n. Cellule
Positive
α19a Cy3 Alphaproteobacteria 7 4,3%(1)
I
(0%f.a.) OS52/1
Poly862 Fluoresceina Polynucleobacter spp. 7 100%
CC23a Cy3 Caedibacter
caryophilus 9 0%
II
(0% f.a.) OS52/1
α19a Fluoresceina Alphaproteobacteria 9 0%
γ23S1027 Cy3 Gammaproteobacteria 8 100%(2) III (0% f.a.) OS52/1 β23S1027 Fluoresceina Betaproteobacteria 8 100%(2) γ23S1027 Cy3 Gammaproteobacteria 11 100%(3)
γ23S1027 Cy3 Gammaproteobacteria 11 100%(3)
IV (30% f.a.)
OS52/1
β23S1027 Fluoresceina Betaproteobacteria 11 100%(3)
Note: (1): Una sola cellula fra quelle osservate presentava alcuni batteri (tre o quattro) positivi alla sonda, localizzati alla periferia del corpo cellulare, di probabile provenienza ambientale. (2): Le sonde utilizzate competono per il medesimo sito di legame sul 16S rRNA (posizione 1027 E. coli). La sonda γ23S1027 si è tuttavia legata sia al simbionte secondario che ai batteri omikron-simili (Betaproteobacteria), correttamente marcati dall’altra sonda. Il simbionte secondario non è stato in nessun caso marcato dalla sonda β23S1027.
(3) A queste condizioni di stringenza è stato possibile discriminare fra le due sonde. Il segnale della sonda γ23S1027 risulta però estremamente indebolito.
Tabella I.3: Ibridazioni preliminari per l’identificazione del simbionte secondario di E.
parawoodruffi. La penultima colonna a destra riporta il numero di cellule osservate; l’ultima la percentuale di cellule al cui interno siano stati individuati batteri marcati in modo specifico dalla sonda. La colonna a sinistra mostra la condizione di stringenza adottata, ovvero la percentuale di formammide presente nella soluzione di ibridazione.
Le ibridazioni in situ hanno condotto all’ipotesi che il simbionte secondario di E.
parawoodruffi ceppo OS 52/1 fosse un membro della cl. Gammaproteobacteria; in
particolare, l’ibridazione competitiva con sonde specifiche rispettivamente per β- e
γ-proteobatteri ha evidenziato che l’organismo viene marcato esclusivamente da quest’ultima sonda (la cui specificità appare tuttavia scarsa a basse condizioni di stringenza). Tale organismo risulta presente in tutte le cellule osservate. Non è stato possibile stimare il numero di batteri per cellula ospite – apparentemente di poco inferiore a quello dei simbionti
omikron-simili.
- Tentativi di caratterizzazione molecolare del simbionte secondario
La presenza nei campioni del DNA-stampo del simbionte principale, un β-proteobatterio, in quantità verosimilmente comparabili a quello del simbionte secondario, non ha reso possibile procedere all’amplificazione selettiva del gene per il 16S del solo simbionte secondario, non disponendo inoltre di primer specifici per la cl. Gammaproteobacteria (che del resto differisce di pochissimo, nella sequenza del 16S rDNA, dalla cl. Betaproteobacteria, rendendo problematica l’eventuale realizzazione di simili inneschi). Si è fatto pertanto ricorso a procedure di amplificazione e clonaggio.
Al fine di aumentare la rappresentatività, nella library derivante da tale clonaggio, dei cloni trasformati con geni non appartenenti al simbionte principale (la cui sequenza del 16S rDNA era stata precedentemente caratterizzata), è stato necessario condurre la reazione di PCR in modo tale da sfavorire selettivamente l’amplificazione di tale gene, aumentando la percentuale di amplificati di altro tipo nel prodotto di reazione destinato ad essere clonato nel vettore. Sono stati pertanto selezionati inneschi universali, presentanti però almeno un
parawoodruffi, nonché con gran parte delle altre sequenze di Polynucleobacter spp.
disponibili in data base. Tali primer sono R15122b (5’-GGAGGTGATCCAACCGCA-3’) ed
R1525 (5’- CAKAAAGGAGGTGATCC-3’).
Sono state effettuate quattro reazioni di PCR, a basse condizioni di stringenza, nelle quali essi sono stati impiegati in tutte le possibili combinazioni con due inneschi forward
universali (F7a: 5’-AGAGTTTGATCCTGGCTCAG-3’; F7b:
5’-AGAGTTTGATCATGGCTCAG-3’). Delle quattro combinazioni testate, F7a + R1522b ed F7b + R1522b hanno reso le maggiori quantità di prodotto. F7b + R1525 ha avuto resa molto scarsa, mentre F7a + R1525 non ha prodotto una quantità di amplificato sufficiente ad essere visualizzata su gel. È possibile che l’innesco F7a/b interferisca negativamente con R1525: entrambe le reazioni sono state scartate. Il prodotto della reazione condotta con F7b + R1522b è stato impiegato per i successivi passaggi del clonaggio.
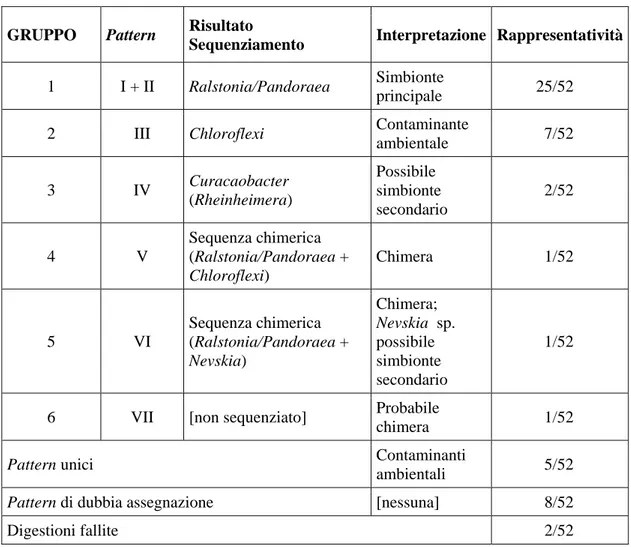
52 cloni sono stati sottoposti a digestione mediante enzimi di restrizione. L’esame dei
pattern così ottenuti ha permesso l’identificazione di dodici distinti gruppi di restrizione.
Almeno tre colonie (ove possibile) per ciascun gruppo di pattern di restrizione individuato sono state sequenziate. Sono qui riportati i risultati dell’analisi (Tabella I.4).
GRUPPO Pattern Risultato
Sequenziamento Interpretazione Rappresentatività
1 I + II Ralstonia/Pandoraea Simbionte
principale 25/52
2 III Chloroflexi Contaminante
ambientale 7/52 3 IV Curacaobacter (Rheinheimera) Possibile simbionte secondario 2/52 4 V Sequenza chimerica (Ralstonia/Pandoraea + Chloroflexi) Chimera 1/52 5 VI Sequenza chimerica (Ralstonia/Pandoraea + Nevskia) Chimera; Nevskia sp. possibile simbionte secondario 1/52
6 VII [non sequenziato] Probabile
chimera 1/52
Pattern unici Contaminanti
ambientali 5/52
Pattern di dubbia assegnazione [nessuna] 8/52
Digestioni fallite 2/52
Tabella I.4: Analisi dei pattern di restrizione ottenuti. Nella terza colonna (indicata come “Risultato
alta percentuale di similarità con la sequenza caratterizzata fra quelli presenti nella banca dati di riferimento (NCBI GenBank).
La maggior parte dei cloni (Gruppo 1: rappresentatività = 48,07%) è risultata trasformata con inserti contenenti il gene del simbionte principale, che evidentemente costituiva la gran parte dell’amplificato utilizzato nella trasformazione, nonostante l’utilizzo di primer non perfettamente complementari. All’interno della banca dati utilizzata (NCBI GenBank), tali
sequenze presentano la maggiore similarità con i generi Ralstonia (YABUUCHI, KOSAKO,
YANO, HOTTA, NISHIUCHI 1996) e Pandoraea (COENYE, FALSEN, HOSTE, OHLÉN, GORIS, GOVAN, GILLIS, VANDAMME 2000), appartenenti alla cl. Betaproteobacteria (Coenye et al.,
2000) che, assieme al gen. Burkholderia (YABUUCHI, KOSAKO, OYAIZU, YANO, HOTTA,
HASHIMOTO, EZAKI, ARAKAWA 1993) costituiscono il sister group del gen.
Polynucleobacter. Tali sequenze sono in effetti risultate identiche a quelle del simbionte
principale di E. parawoodruffi ceppo OS 52/1 precedentemente caratterizzata tramite sequenziamento diretto.
Un secondo gruppo di cloni (Gruppo 2: rappresentatività = 13,46%) è trasformato con i geni di batteri appartenente alla divisione Chloroflexi. I membri di tale raggruppamento sono perlopiù organismi fotosintetici a vita libera, reperibili in acque dolci o salmastre, formanti colonie filamentose sulla superficie di specchi d’acqua (Hanada and Pierson, 2002). Queste considerazioni, unite ai risultati della ibridazione in situ (in cui il simbionte secondario era apparso positivo all’ibridazione con sonde Gammaproteobacteria-specifiche) ha condotto all’ipotesi che si trattasse di batteri derivanti da contaminazione ambientale. Sono state successivamente individuate, all’interno dei contenitori di coltura del ceppo OS 52/1, formazioni di colonie batteriche filamentose, di colore verde, simili a quelle descritte per i
Chloroflexi. Ciò ha avvalorato ulteriormente l’ipotesi della provenienza ambientale di tali
batteri.
Dallo screening dei cloni sono emerse due sequenze (Gruppo 3: rappresentatività = 3,85%) di organismi appartenenti alla cl. Gammaproteobacteria, simili a quelle di alcuni organismi facenti parte di un genere recentemente descritto (Curacaobacter). Tale genere comprende batteri marini a vita libera, filogeneticamente vicini al gen. Rheinheimera. Data l’appartenenza alla classe cercata ed alla reciproca somiglianza delle due sequenze, è stato ipotizzato che potesse trattarsi del simbionte secondario, nonostante la bassa rappresentatività. Una ulteriore sequenza di γ-proteobatterio vicino al gen. Nevskia non è stata tenuta in considerazione in quanto poco rappresentata (comparsa unicamente come frammento all’interno di un amplicone chimerico).
- Ibridazione con sonde specie-specifiche disegnate sul 16S rRNA
Sulla base delle sequenze dei cloni A7 e B6, appartenenti al gruppo 2, è stata disegnata una sonda oligonucleotidica con cui tentare di confermare l’appartenenza della sequenza trovata
al simbionte mediante ibridazione in situ. Le caratteristiche della sonda sono di seguito riassunte:
-Rhei1245: Specifica per batteri filogeneticamente vicini al genere Rheinheimera. La sequenza non presenta mismatch con 41 delle 44 sequenze comprese in tale
cluster. Differisce per almeno 2 nucleotidi da tutte le altre sequenze presenti in
banca dati.
Posizione: 1245 E. coli.
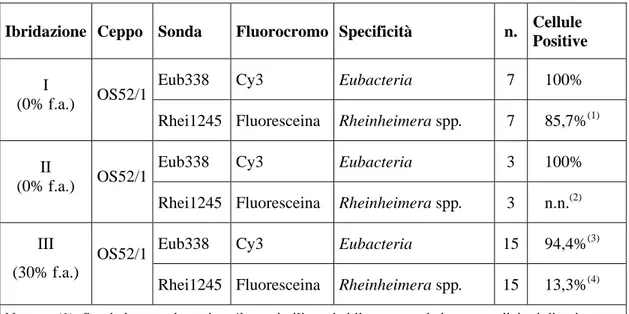
Sono state effettuate le ibridazioni di controllo di seguito riportate (cfr. Tabella I.5).
Ibridazione Ceppo Sonda Fluorocromo Specificità n. Cellule
Positive
Eub338 Cy3 Eubacteria 7 100%
I
(0% f.a.) OS52/1
Rhei1245 Fluoresceina Rheinheimera spp. 7 85,7%(1)
Eub338 Cy3 Eubacteria 3 100%
II
(0% f.a.) OS52/1
Rhei1245 Fluoresceina Rheinheimera spp. 3 n.n.(2)
Eub338 Cy3 Eubacteria 15 94,4%(3)
III (30% f.a.)
OS52/1
Rhei1245 Fluoresceina Rheinheimera spp. 15 13,3%(4)
Note: (1): Sonda legata a batteri omikron-simili, probabilmente per le basse condizioni di stringenza adottate.
(2): Sonda legatasi aspecificamente a strutture cellulari dell’ospite; non è stato possibile identificare cellule batteriche correttamente ibridate.
(3) A queste condizioni di stringenza il legame della sonda risulta indebolito, pertanto in alcune cellule neppure il simbionte principale risulta visibile.
(4): La sonda ha marcato in modo specifico alcuni batteri localizzati sul margine del corpo cellulare di alcuni degli individui ibridati. Morfologia e distribuzione osservate non coincidevano con quanto riportato per il simbionte secondario.
Tabella I.5: Ibridazioni con sonda specie-specifica (Rhei1245) per l’identificazione del simbionte
secondario di E. parawoodruffi. La penultima colonna a destra riporta il numero di cellule osservate; l’ultima la percentuale di cellule al cui interno siano stati individuati batteri specificamente marcati dalla sonda; la prima a sinistra la condizione di stringenza adottata, ovvero la percentuale di formammide presente nella soluzione di ibridazione.
Dalle ibridazioni effettuate risulta che, alla condizione di stringenza del 30% di formammide, la sonda è in grado di legarsi in modo specifico a cellule batteriche. Viceversa, la formazione di legami RNA-RNA in assenza di formammide è risultata, in molti casi, aspecifica.
In alcune cellule è stata riscontrata la presenza di un segnale positivo e specifico da parte della sonda. Tale segnale evidenziava alcuni batteri di forma coccoide, presenti in piccoli raggruppamenti distribuiti lungo il margine della cellula stessa. Non è stato possibile comprendere se essi si trovassero all’interno o all’esterno di essa. In ogni caso, la loro
distribuzione, la forma e le percentuali di diffusione nella popolazione dell’ospite non coincidono con quanto osservato per il simbionte secondario. Tali batteri sono probabilmente organismi a vita libera, appartenenti ad un genere vicino a Rheinheimera, presenti come contaminanti nelle colture cellulari di E. parawoodruffi, la cui sequenza è stata caratterizzata nel clonaggio ed è servita come base per disegnare la sonda stessa. L’approccio sperimentale utilizzato (cfr. Sezione 1: Introduzione – Approccio sperimentale utilizzato per
l’identificazione dell’ospite e l’individuazione di eventuali simbionti) ha permesso di non
attribuire erroneamente tale sequenza al simbionte secondario di E. parawoodruffi.
Successive ibridazioni in situ compiute su di un’aliquota del medesimo ceppo OS 52/1, separata dal ceppo principale da alcuni mesi, hanno dimostrato che il simbionte secondario era stato perduto. Ciò può essere imputato alle differenti condizioni sperimentali (es. cibature più frequenti) in cui tale aliquota è stata mantenuta, che possono aver determinato una maggiore velocità di crescita cellulare, con conseguente diluizione e scomparsa dei simbionti non indispensabili dalla popolazione.
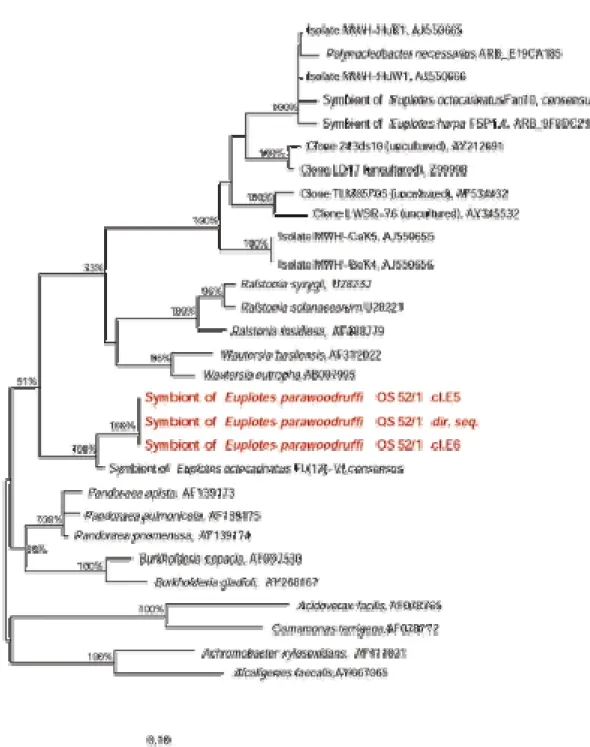
Figura I.1: Cladogramma filogenetico di una selezione di specie di β-proteobatteri comprese
nell’ordine Burkholderiales (per dettagli si rimanda al presente capitolo). È stata evidenziata la collocazione delle sequenze dei simbionti di E. parawoodruffi. I valori di bootstrap riportati sono calcolati su 1000 pseudorepliche. La lunghezza della componente orizzontale rappresenta la distanza evolutiva fra le sequenze: la barra misurante la scala corrisponde a 10 cambiamenti nucleotidici ogni 100 posizioni.
Sezione 3 – Capitolo I
Parte Terza
Discussione dei risultati ottenuti
I.3.1: Caratterizzazione morfologica e molecolare
La specie d’appartenenza degli organismi oggetto di studio è stata determinata come
Euplotes parawoodruffi SONG AND BRADBURY, 1997. Tale specie presenta numerose somiglianze con l’affine E. woodruffi, precedentemente descritto da Gaw (1939). Entrambe le specie sono descritte come salmastricole, reperibili però anche in acque dolci, nel caso di
E. woodruffi, o marine, nel caso di E. parawoodruffi; le dimensioni medie coincidono, così
come la morfologia generale del corpo cellulare, il cirrotipo (“9 type 1”) e l’argiroma. La
forma del macronucleo, a “
τ
”, rappresenta un carattere unico all’interno del genere Euplotes(in cui sono più diffusi macronuclei a forma di “C” o di “
ε
”), condiviso esclusivamente daqueste due specie (Carey, 1992).
La distinzione fra E. woodruffi ed E. parawoodruffi è stata possibile grazie al raffronto delle caratteristiche morfologiche che differiscono nelle due specie. Tali caratteri riguardano sia l’aspetto generale dell’organismo che l’organizzazione nucleare e l’infraciliatura orale.
E. woodruffi presenta un profilo più rotondeggiante rispetto ad E. parawoodruffi, le cui
estremità appaiono più squadrate; la regione peristomale di quest’ultima specie è inoltre molto più allungata di quella dell’altra, arrivando ad occupare i quattro quinti della superficie ventrale (il che corrisponde a quanto osservato negli esemplari del ceppo OS 52/1), mentre in
E. woodruffi ne occupa circa i due terzi. Inoltre, E woodruffi si presenta appiattito
dorsoventralmente, mentre la sezione trasversale di E. parawoodruffi risulta più spessa e nettamente arcuata, similmente a quanto accade nei ciliati ipotrichi del gen. Euplotidium (Inamori, 2006). Nel presente lavoro non sono state impiegate tecniche in grado di evidenziare la reale struttura della sezione trasversale della cellula: all’osservazione in vivo le cellule apparivano appiattite, e verosimilmente più sottili di cellule di Euplotidium spp. (pers. oss.). Tuttavia, non essendo disponibili esemplari di sicura attribuzione ad una delle due specie onde fare un raffronto, tale carattere non è stato considerato ai fini dell’identificazione. Un carattere tassonomico rilevante per la discriminazione è la presenza di una sottile invaginazione guttiforme inserita nella volta della regione peristomale
anteriormente al citostoma: tale struttura è assente in E. parawoodruffi. Anche questo carattere, tuttavia, è di difficile valutazione in assenza di un controllo positivo o negativo, e pertanto non è stato considerato.
Per quanto riguarda l’analisi dell’infraciliatura orale, la distribuzione della AZM osservata rappresenta un carattere che il ceppo OS 52/1 condivide con E. parawoodruffi: le AZM di E
parawoodruffi si presentano infatti con l’estremità distale nettamente piegata verso la
regione ventrale, sino ad assumere una forma a “7” (Inamori, 2006), mentre l’estremità prossimale termina molto vicino al primo dei cirri trasversali. La regione somatica delle AZM si estende, nelle cellule osservate, apparentemente per non più di 15 μm a partire dal peristoma, come riportato per E. woodruffi (Song and Bradbury, 1997; Inamori, 2006); tuttavia, non essendo state eseguite un numero di misurazioni sufficienti a supportare statisticamente tale dato, questa informazione non è stata considerata. È stato invece possibile valutare la curvatura anteriore della banda formata dalle AZM: queste piegano nettamente verso l’interno per terminare nei pressi dei cirri frontoventrali anteriori. Anche questo carattere è presente in E. parawoodruffi, mentre in E. woodruffi esse terminano relativamente lontano dai cirri frontoventrali anteriori. Inoltre, in quest’ultima specie, che possiede una regione peristomale meno estesa, la AZM decorre dritta lungo la superficie ventrale, piegando verso l’interno solo posteriormente; nella regione distale. In E.
parawoodruffi e negli esemplari osservati, invece, tale zona piega bruscamente seguendo
l’orlo della regione peristomale, terminando quasi a contatto con l’ultimo a destra dei cirri traversali.
L’apparato nucleare osservato appare simile a quanto descritto per E. parawoodruffi (braccia distali di differente lunghezza). Nel macronucleo di E. parawoodruffi il braccio distale sinistro si presenta di lunghezza doppia rispetto al destro; in virtù di tale differenza di lunghezza, la porzione centrale si unisce alle braccia nei pressi dell’angolo superiore destro del corpo cellulare, conferendo al macronucleo, a prima vista, una forma ad “S” rovesciata piuttosto che a “T” (Song and Bradbury, 1997; Inamori, 2006). Tale carattere è ben evidente nelle colorazioni nucleari delle cellule del ceppo in esame. La commessura col braccio centrale non avviene mai nei pressi dell’asse longitudinale mediano; in alcuni esemplari, tuttavia, la sottigliezza del macronucleo in quella regione non permette di osservare l’esatta posizione di aggancio dei bracci distali. Lo spessore della regione distale dell’apparato nucleare appare minore di quanto si osserva in altri ciliati del gen. Euplotes (es. E. crassus:
pers. oss.), così come in alcuni punti del braccio macronucleare mediano: tale spessore
appare inoltre variare da zona a zona. Ciò è stato interpretato come un possibile artefatto sperimentale; in alternativa, il carattere potrebbe essere ricondotto ad E. parawoodruffi, che presenta appunto un macronucleo assottigliato rispetto ad altri Euplotes, fra cui E. woodruffi (Song and Bradbury, 1997).
È stato effettuato un confronto fra fotografie di macronuclei di E. woodruffi (Song and Bradbury, 1997) ed esemplari, specificamente colorati, del ceppo OS 52/1. La struttura nucleare è apparsa dissimile da quella di E. woodruffi in tutti gli esemplari osservati. La relativa facilità di riconoscimento e misurazione di tale carattere rende minimo il rischio di interpretazioni errate. Dagli esiti di tale confronto si è stati in grado di affermare che la specie in esame non è E. woodruffi: in tale specie, infatti, il macronucleo presenta braccia di pari lunghezza; in alcuni casi tale lunghezza può differire, ma, quando ciò avviene, il braccio più lungo risulta essere invariabilmente il destro (osservazione riportata in Song and Bradbury, 1997). In E. parawooduffi la conformazione macronucleare appare simile a quella presente in OS 52/1; inoltre, corrispondono a quanto riportato in letteratura (Song and Bradbury, 1997) la disposizione del braccio distale sinistro (al di sotto della porzione distale delle AZM), la sottigliezza e l’irregolarità della forma del macronucleo e la posizione del micronucleo. Il confronto con materiale fotografico di E. parawoodruffi (Song and Bradbury, 1997) ha confermato tali osservazioni.
Per quanto riguarda l’argiroma, entrambe le specie lo possiedono del tipo “doppio-eurystomus”. Sussistono tuttavia piccole differenze che possono essere utilizzate come caratteri diagnostici (numero di corpuscoli basali per cinezia, distanza fra l’estremità anteriore di ciascuna cinezia e l’inizio della AZM). Il riconoscimento di tali caratteri è tuttavia piuttosto difficile, e non è stato ritenuto necessario considerarli ai fini dell’identificazione della specie.
La Tabella I.6 fornisce un quadro riassuntivo di quanto precedentemente esposto.
Corpo cellulare
Carattere E. woodruffi E. parawoodruffi [Ceppo OS 52/1]
Forma del corpo Ovale. Leggermente squadrata.
Leggermente squadrata.
Larghezza regione
peristomale Ca. 2/3 del corpo. Ca. 4/5 del corpo. Ca. 4/5 del corpo. Spessore de corpo Corpo appiattito. Corpo fortemente
arcuato. Apparentemente appiattito. Invaginazione peristomale Invaginazione presente. Invaginazione
assente. [non valutato]
AZM
Carattere E. woodruffi E. parawoodruffi [sp. in esame]
Curvatura regione distale
Leggermente incurvata.
Fortemente incurvata
(7-shaped). Fortemente incurvata.
Estensione AZM Poco estese (13-15 μm). Molto estese (18-20 μm). Apparentemente poco
estese.
Curvatura regione
prossimale Poco incurvata. Molto incurvata. Molto incurvata. Posizione estremità prossimale Anteriore ai cirri trasversali. Prossima ai cirri trasversali. Prossima ai cirri trasversali.
Apparato nucleare
Carattere E. woodruffi E. parawoodruffi [sp. in esame]
Lunghezza braccia distali
Braccia di pari lunghezza.
Braccio sinistro lungo il doppio del destro.
Braccio sinistro lungo il doppio del destro.
Spessore braccia
distali Spesse. Sottili.
Apparentemente sottili.
Argiroma
Carattere E. woodruffi E. parawoodruffi [sp. in esame]
Argiroma Doppio-eurystomus Doppio-eurystomus [non valutato] Tabella I.6: Raffronto delle differenze morfologiche osservabili fra E. woodruffi, E. parawoodruffi e
la specie in esame (cellule del ceppo OS 52/1). Per le informazioni relative ad E. woodruffi cfr. Inamori, 2006; per quelle relative ad E. parawoodruffi cfr. Inamori, 2006 e Song and Bradbury 1997.
I dati relativi alla disposizione delle AZM e soprattutto quelli relativi alla conformazione macronucleare supportano l’identificazione della specie con E. parawoodruffi. È senza dubbio possibile escludere qualsiasi altra specie di Euplotes ad oggi descritta ad eccezione di
E. woodruffi, che possiede un macronucleo simile. Le differenze sin qui esposte sono
caratteri morfologici più sottili e di più difficile interpretazione rispetto alla forma del macronucleo, e probabilmente nessuno di essi è in grado di fornire di per sé una chiave diagnostica per discriminare fra le due specie. Tuttavia, sono stati riscontrati almeno sette punti in comune fra quanto osservato e la descrizione di E. parawoodruffi, di cui almeno uno (conformazione apparato nucleare) di facile individuazione, a fronte di due soli caratteri (spessore del corpo, estensione AZM), peraltro di dubbia interpretazione, apparentemente condivisi con E. woodruffi.
La caratterizzazione molecolare è stata di scarso aiuto per la discriminazione fra le due specie. Il marcatore molecolare utilizzato, la sequenza completa del gene codificante per il 18S rRNA, non sembra infatti differire significativamente fra di esse: la sequenza di OS 52/1 differisce infatti dello stesso numero di nucleotidi (due) sia da quella di E. woodruffi che da quella di E. parawoodruffi presenti in banca dati (sebbene l’appaiamento con la sequenza di E. woodruffi depositata sia più corto di due nucleotidi: cfr. Parte Seconda,
Risultati, presente capitolo). I due nucleotidi che differiscono non sono gli stessi nelle due
sequenze. Infine, la notevole percentuale di dissimilarità registrata fra la sequenza di OS 52/1 e quella del ceppo BHY di E. woodruffi (AF492707: Song 2002) è probabilmente imputabile alla cattiva qualità di quest’ultima piuttosto che alla variabilità interspecifica.
È possibile che tali specie si siano separate in tempi relativamente recenti, e che il gene in esame non abbia ancora accumulato un numero di mutazioni sufficiente per differenziarsi significativamente fra di esse. L’utilizzo di un marcatore molecolare più sensibile (geni mitocondriali e/o codificanti per proteine) consentirebbe probabilmente di distinguere fra le due specie.
I.3.2: Caratterizzazione del simbionte principale
La presenza di simbionti omikron-simili in E. parawoodruffi era attesa sulla base di quanto riportato in letteratura per la specie vicina E. woodruffi (Heckmann et al., 1983). Il batterio trovato all’interno di tale specie presenta una distribuzione all’interno della cellula ospite effettivamente paragonabile a quella descritta per Polynucleobacter spp. (es. Heckmann et
al., 1983; Heckmann et al., 1987). Alcune differenze sono state tuttavia notate a livello
dell’ultrastruttura; la caratterizzazione molecolare della sequenza del 16S rDNA ha in seguito mostrato l’esistenza di una divergenza significativa da Polynucleobacter spp.
I simbionti omikron descritti originariamente in E. aediculatus (Heckmann, 1975) e quelli
omikron-simili successivamente individuati in molte altre specie di Euplotes (Heckmann et al., 1983), sono tutti accomunati dal fatto di possedere, all’interno del citoplasma, un numero
variabile (in genere 3-9; in alcuni casi 12: Görtz, 2003) di strutture elettrondense, presentanti un nucleo centrale di aspetto cromatinico (ma la cui natura non è stata determinata: Görtz, 2003), collocate a distanze regolari tra loro e denominate “nucleoidi” (Heckmann, 1975). Tale elemento è, apparentemente, l’unico carattere esclusivamente condiviso da tutti i batteri
omikron-simili, che presentano invece numerose differenze per quanto riguarda dimensioni,
forma ed altre caratteristiche (Görtz, 2003). I simbionti di OS 52/1 presentano caratteristiche morfologiche (forma e dimensioni) paragonabili a quelle di Polynucleobacter spp.; tuttavia, l’osservazione al microscopio a trasmissione ha rivelato l’assenza di strutture assimilabili a nucleoidi. Qualcosa di analogo era stato segnalato nel 1983 (Heckmann et al.), quando furono individuati batteri privi di nucleoidi all’interno di due ceppi monoclonali di E.
aediculatus (nei ceppi in questione non era presente P. necessarius). Tali batteri apparivano
inoltre differire per forma, dimensioni e numero di simbionti per cellula ospite rispetto a P.
necessarius (Heckmann et al., 1983). Tali caratteri sono tuttavia piuttosto variabili in
simbionti di ospiti differenti (Görtz, 2003); l’abbondanza all’interno dell’ospite varia inoltre a seconda della fase del ciclo cellulare e delle condizioni di coltura (Heckmann, 1983). Tuttavia, la mancanza di nucleoidi, unitamente alle differenze citate, appariva indizio sufficiente per “[essere] convinti che essi appartengano ad una classe di batteri completamente diversa […] da [quella dei] simbionti essenziali” (Heckmann et al., 1983). Sui ceppi infetti da questo secondo tipo di batterio furono ugualmente eseguiti gli esperimenti di aposimbiosi e reinfezione necessari per verificare l’essenzialità del simbionte per la sopravvivenza dell’ospite (descrizione riportata all’inizio del presente capitolo). Tali esperimenti dimostrarono che la simbiosi in esame era necessaria all’ospite così come lo è quella con batteri omikron-simili; inoltre, il simbionte era in grado di indurre nuovamente la divisione cellulare in cellule aposimbionti di E. aediculatus precedentemente infette da P.
necessarius, così come la reinfezione con P. necessarius di cellule aposimbionti dei ceppi in
presente nei due ceppi analizzati, gli autori conclusero che il secondo batterio vi si era sostituito funzionalmente (Heckmann et al., 1983). In conclusione, la presenza di batteri privi di nucleoidi in Euplotes sp. è già stata segnalata, ed in un caso ne è stata accertata l’essenzialità per l’ospite e l’avvenuta sostituzione funzionale ai battei omikron-simili. Durante il presente studio non sono stati eseguiti esperimenti di aposimbiosi su E.
parawoodruffi; è tuttavia possibile che, anche in questo caso, un simbionte privo di nucleoidi
abbia sostituito funzionalmente un batterio Polynucleobacter-simile, dal momento che questi non sono sicuramente presenti nel ceppo analizzato. La creazione di cellule aposimbionti di
E. parawoodruffi è necessaria per stabilire se l’ospite sia effettivamente incapace di dividersi
senza il simbionte. Analoghi esperimenti, eseguiti sulle specie di Euplotes con cirrotipo “9
type 1” (fra cui l’affine E. woodruffi: Heckmann et al., 1983; Fujishima and Heckmann,
1984), fanno tuttavia supporre che anche in questo caso tale simbiosi sia necessaria.
L’analisi filogenetica della sequenza del 16S rDNA ha confermato il basso grado di parentela del batterio osservato con i simbionti omikron-simili già sequenziati e, più in generale, con l’intero cluster “Polynucleobacter”. Tali sequenze si collocano nel cosiddetto “ParaPoly cluster”, in posizione basale rispetto al clade comprendente i genn.
Polynucleobacter e Ralstonia/Wautersia, che risultano filogeneticamente correlati fra loro.
Strettamente associata ad esse (bootstrap value 100% con 1000 pseudorepliche) si trova la sequenza di un simbionte presente nel ceppo FL(12)-VI di E. octocarinatus (un secondo ceppo, Fan10, possiede invece un simbionte omikron-simile filogeneticamente vicino a P.
necessarius: Vannini, unpubl.). La stabilità della posizione appare confermata dal fatto che
essa si ripete, senza variazioni apprezzabili, in tutti gli alberi calcolati, a prescindere dal criterio utilizzato (Neighbor-joining method, Parsimony criterion, Maximum-likelihood
criterion), dai filtri e dalle correzioni applicate. La distanza filogenetica che separa il clade
comprendente i simbionti di E. parawoodruffi e del ceppo di E. octocarinatus di cui sopra dal “Polynucleobacter cluster” e dal suo sister group (genn. Ralstonia/Wautersia) appare significativa. È possibile ipotizzare che tali batteri appartengano ad un genere diverso da
Polynucleobacter, compreso nella famiglia delle Burkholderiaceae, non ancora descritto. In
tale genere sarebbe da ascriversi anche il simbionte di E. octocarinatus la cui sequenza risulta associata a quello di E. parawoodruffi.
La conferma di tale risultato porterebbe a dover riconsiderare lo status della sonda oligonucleotidica Poly862, precedentemente definita come specifica per organismi appartenenti al genere Polynucleobacter o affini ad esso. Tale sonda non è teoricamente in grado di legarsi a batteri del genere Ralstonia e Wautersia, costituenti il sister group del “Polynucleobacter cluster”; tuttavia, riconosce specificamente i batteri simbionti di OS 52/1, che non appartengono a tale gruppo, bensì al “ParaPoly cluster”. La sonda riconosce pertanto organismi appartenenti a due diversi gruppi filetici, e probabilmente collocabili in differenti
generi: la sua definizione dovrebbe pertanto essere indicata come “specifica per batteri simbionti principali di Euplotes spp. appartenenti al ‘Polynucleobacter cluster’ od al ‘ParaPoly cluster’”.