2.Materiali e metodi
2.1 Linee murine utilizzate
-Per gli esperimenti di caratterizzazione delle popolazioni ematopoietiche sono stati utilizzati topi C57BL/6 (Harlan Lab).
-Per i saggi di ripopolazione competitiva a lungo termine sono stati usati come donatori strain congenici di topi C57BL/6-Ly5.1 (Charles River), C57BL/6(Ly5.2) wild-type e knock-out per ciclina E1 e topi a background misto C129/C57BL/6 wild-type e knock-out per ciclina E1 e transgenici per GFP; come recipienti sono stati usati topi C57BL/6 Ly5.1-Ly5.2 e un ceppo misto di C129/C57BL/6.
Tutti i topi sono stati mantenuti nello stabulario del Campus Ifom-IEO (Milano) e tutte le procedure sperimentali sono state condotte rispettando la direttiva EU 86/609 del Ministero italiano della Sanità.
2.2 Genotipizzazione
La genotipizzazione è stata condotta per PCR sul DNA estratto da biopsie caudali. Per amplificare la sequenza relativa al gene ciclina E1 sono stati usati i seguenti pirmers: -primer forward allele wild-type 5’-CGCATACTGAGACACAGACT (4052)
-primer forward allele knock-out 5’-GATCTCTCGTGGGATCATTG (4053) -primer reverse comune 5’-CGCCATGGTTATCCGGGAGATGG (4051)
Protocollo di estrazione del DNA
1) Lisi del tessuto caudale mediante risospensione in 400 μL di soluzione lisante formata da 100Mm Tris HCl pH 8.5, 0,5M EDTA, 10% SDS, 5M NaCl e Proteinasi K (20 mg/mL).
2) Incubazione per 12 ore 55°C in agitazione
3) Precipitazione in isopropanolo (400 μL), agitazione delicata e trasferimento del DNA precipitato in 400 μL di acqua.
4) Incubazione per 20’ a 37°C in agitazione 5) Incubazione per 5’ a 95°C
6) Dopo un graduale ritorno a temperatura ambiente, la soluzione contenete DNA puo’ essere usata per la genotipizzazione o conservata a -20°C
Protocollo di PCR
1) Risospensione di 3 μL di DNA in 17 μL di miscela di reazione. 11,8 μL di acqua, 2 μL di buffer 10X, 0,3 μL di deossinucleotidi, 0,1 μL di forward primer, 0,1 μL di reverse primer, 0,4 μL di dimetilsolfossido e 0,3 μL di TaqPolimerasi (Quiagen)
2)Programmazione del termocycler (T3000 Biometra): • Denaturazione 94°C per 5’
• 34 cicli trifasici (94°C per 30’’; 55°C per 30’’; 72 °C per 45’’) • Estensione finale 72°C per 5’
2.3 Immunodetezione di antigeni cellulari di superficie e analisi FACS
La citometria a flusso, nota come FACS o Fluorescence-Activated Cell Sorting, è un mezzo molto potente per analizzare in una determinata popolazione di cellule diversi parametri come morfologia, grandezza, contenuto di DNA per valutare la progressione del ciclo cellulare o l’apoptosi ed espressione di specifiche proteine grazie ad un precedente passaggio di staining con anticorpi legati a fluorocromi.
Tale approccio è stato usato per la caratterizzazione fenotipica delle diverse popolazioni ematopoietiche presenti nel midollo, purificabili grazie all’espressione di un set specifico e univoco di marcatori di superficie.
Dopo l’isolamento, le cellule mononucleate del midollo osseo (BMMNCs) subiscono uno staining con anticorpi monoclonali specifici per tali marcatori legati a fluorocromi che ne permettono la rilevazione.
Protocollo di colorazione
1) Dissezione dei due femori e relative tibie, pulizia delle singole ossa con garze sterili e ottenimento di una sospensione cellulare in PBS sterile mediante crashing;
2) Filtraggio della sospensione cellulare attraverso un filtro da 70μm
3) Isolamento della popolazione di cellule mononucleate del midollo (BMMNCs) grazie a centrifugazione su gradiente di densità con Ficoll-Histopaque -1083(Sigma Aldrich).In dettaglio, la sospensione cellulare è centrifugata per 45 minuti a 1500rpm a 4°C; la centrifuga è settata a minima velocità di accelerazione e decelerazione in modo da garantire l’accumulo delle cellule mononucleate in un anello centrale e la precipitazione delle altre tipologie cellulari, quali eritrociti, adipociti, cellule mesenchimali.
4) il protocollo di staining è per 5-6x106 BMMNCs: • Lavaggio con 1mL di PBS
• Incubazione di 15 minuti in ghiaccio in 50 μL di una soluzione di PBS contenente 5% BSA(Bovin serum albumine), 5% di rat serum e streptavidina (1μg/mL) .Questo processo è definito “blocking” e ha lo scopo di minimizzare le possibili interazioni aspecifiche
• Lavaggio le cellule con 1mL di PBS
• Incubazione di 45 minuti in ghiaccio e al buio in 50 μL di PBS1%BSA contenente gli anticorpi primari diluiti 1:100.
• Lavaggio le cellule con 1mL di PBS
• (Se necessaria) incubazione di 30minuti in ghiaccio e al buio in 50 μL di PBS1%BSA contenente l’ anticorpo secondario diluito 1:100.
• Fissaggio delle cellule mediante incubazione in una soluzione di PBS2%formaldeide per un periodo di 15 minuti in ghiaccio
• Le cellule vanno mantenute ad una temperatura di 4°C fino all’ analisi FACS • Le analisi sono state effettuate con FACSCanto II della Becton Dickinson
equipaggiato con tre laser (405nm 488nm e 633nm).Per l'acquisizione e l'analisi dei campioni è stato utilizzato il software BDFacsDiVa 6.0
Elenco degli anticorpi utilizzati
Anticorpi primari (eBioscience)
PE anti-mouse CD117 (c-Kit, cKit)clone 2B8 cod 12-1171 APC anti-mouse CD117 (c-Kit, cKit) clone 2B8 cod 17-1171
Phycoerythrin-Cy5.5 (PE-Cy5.5) anti-mouse CD117 (c-Kit, cKit) clone 2B8 cod 35-1171
FITC anti-mouse Ly-6A/E (Sca-1, Sca1)clone D7 11-5981 PE anti-mouse Ly-6A/E (Sca-1, Sca1) clone D7 12-5981
Biotin anti-mouse Ly-6A/E (Sca-1, Sca1) clone D7 13-5981
Phycoerythrin-Cy5.5 (PE-Cy5.5) anti-mouse Ly-6A/E (Sca-1, Sca1) clone D7 35-5981 Biotin anti-mouse CD34 clone RAM34 cod 13-0341
Fluorescein isothiocyanate (FITC) anti-mouse CD34 clone RAM34 cod 11-0341 PE anti-mouse CD135 (Flk-2, Flk2, Flt3, Flt-3, Ly-72)clone A2F10 cod 12-1351 PE anti-mouse CD4 (L3T4) clone GK1.5 cod 12-0041
FITC anti-mouse CD8 (alpha subunit, CD8a, Ly-2)clone 53-6.7 cod 11-0081 PE anti-mouse/human CD45R (B220) clone RA3-6B2 cod 12-0452
PE-Cy7 anti-mouse/human CD45R (B220)clone RA3-6B2 cod25-0452
Biotin anti-mouse TER-119 (TER119, Erythroid cells, Ly-76) clone TER-119 cod 13-5921
PE-Cy7 anti-mouse TER-119 (TER119, Erythroid cells, Ly-76) clone TER-119 cod 25-5921
PE anti-mouse Ly-6G (Gr-1, Gr1) clone RB6-8C5 cod 12-5931 Biotin anti-mouse Ly-6G (Gr-1, Gr1) clone RB6-8C5 cod 13-5931 PE-Cy7 anti-mouse Ly-6G (Gr-1, Gr1) clone RB6-8C5 cod 25-5931
Biotin anti-mouse CD11b (Integrin alpha M, Mac-1 alpha) clone M1/70 cod13-0112 PE-Cy7 anti-mouse CD11b (Integrin alpha M, Mac-1 alpha) clone M1/70 cod 25-0112
25-0031 PE-Cy7 Hamster anti-mouse CD3 (epsilon subunit; CD3e) 145-2C11 anti-mouse-IL7R coniugato con
12-1271 PE anti-mouse CD127 (Interleukin-7 Receptor alpha, 7 Receptor alpha, IL-7Ra, IL7Ra) A7R34
Per determinare le cellule negative per i marker di lineage (Lin- cells) è stato usato un cocktail di anticorpi anti-marcatori di lineage contenente anticorpi anti-CD3e, anti-CD8, anti-B220, anti-TER-119, anti-Gr1 e anti-Mac1 coniugati al medesimo fluorocromo (ficoeritrina-cianina7) e diluiti 1:200 in PBS1%BSA.
Anticorpi secondari (BD Pharmingen™)
Streptavidin-Fluorescein Isothiocyanate (SAv-FITC) FITC Streptavidin 0.5 mg cod 554060
Streptavidin-Allophycocyanin (SAv-APC) APC Streptavidin cod 349024
Streptavidin-Allophycocyanin-Cy7 (SAv-APC-Cy7)APC-Cy™7 Streptavidin 0.1 mg cod 554063
Streptavidin-Phycoerythrin (SAv-PE) PE Streptavidin 0.5 mg cod 554061 Streptavidin-Phycoerythrin-Cy5 (SAv-PE-Cy5) PE-Cy™5 Streptavidin 0.1 mg cod 554062
Streptavidin-Phycoerythrin-Cy7 (SAv-PE-Cy7) PE-Cy™7 Streptavidin 0.1 mg cod 557598
2.4 Saggio di ripopolazione competitiva a lungo termine (Long Term
Competitive Repopulation Assay)
Il saggio di ripopolazione competitiva è un saggio staminale funzionale che si basa sulla capacità unica delle cellule staminali ematopoietiche di ricostituire un intero sistema ematopoietico. Esso permette di stabilire in maniera semiquantitativa il potenziale di ripopolazione di un midollo oggetto di indagine, definito “test”, rispetto ad un altro midollo, definito “competitor”. Il potenziale di ripopolazione viene determinato
valutando a livello del sangue periferico il contributo relativo dei due midolli usati per ricostituire il sistema ematopoietico dei topi recipienti.
Tecnicamente, consiste nel trapiantare in un topo recipiente congenico letalmente irradiato (“host”) uno egual numero di BMMNCs isolate dal midollo “test” e dal midollo “competitor” e verificare dopo un certo periodo l’esito della ricostituzione mediante analisi del chimerismo del sangue periferico dell’ospite, che rifletterà la capacità ripopolativa del midollo test in rapporto a quella del midollo competitor.
Per discriminare i midolli usati per la ricostituzione si usano donatori differenti a livello di marcatori genetici o fenotipici. A questo scopo, è spesso sfruttato il polimorfismo allelico di un antigene cellulare di superficie, il locus Ly5 (CD45, common leukocyte antigen). Esistono infatti due diverse forme alleliche di tale proteina chiamate Ly5.1 e Ly5.2 perfettamente identiche a livello funzionale e facilmente distinguibili grazie ad anticorpi monoclonali specifici per i due epitopi.
In questo lavoro, sono stati utilizzati midolli “test” relativi a strain C57BL/6Ly5.2, midolli “competitor” isolati da strain C57BL/6Ly5.1 e il trapianto è stato effettuato in topi “host” C57BL/6Ly5.1-Ly5.2.
Un’altra modalità qui sfruttata è l’utilizzo di midolli test isolati da topi transgenici per GFP. La loro progenie è facilmente individuabile nel sangue periferico in quanto GFP positiva; le rimanenti cellule GFP negative sono il prodotto del midollo competitore o residuale dell’ospite. La positività per la GFP è rilevata mediante analisi FACS del sangue periferico.
Il saggio di ripopolazione seriale è un assay funzionale per misurare la capacità di self-renewal di un determinato midollo.
Consiste nell’usare un midollo test per ricostituire un topo letalmente irradiato e verificare la sopravvivenza dell’ospite, ossia la piena capacità delle cellule trapiantate di ricostituire l’intero sistema ematopoietico. Il numero di trapianti seriali che il midollo in
oggetto riuscirà a sostenere permettendo la sopravvivenza dell’ospite riflette la sua capacità di self-renewal.
Nel nostro caso, per analizzare l’efficienza di self-renewal del midollo test in rapporto al competitor è stato effettuato un passaggio del trapianto competitivo primario in seriale, ossia il midollo del topo trapiantato in maniera competitiva che ha già ripopolato il sistema ematopoietico dell’ospite primario è trapiantato in un nuovo ospite letalmente irradiato, definito ospite secondario. Dopo 3 mesi è stata verificata l’efficienza di ripopolamento sul sangue periferico in modo analogo al trapianto primario in competizione.
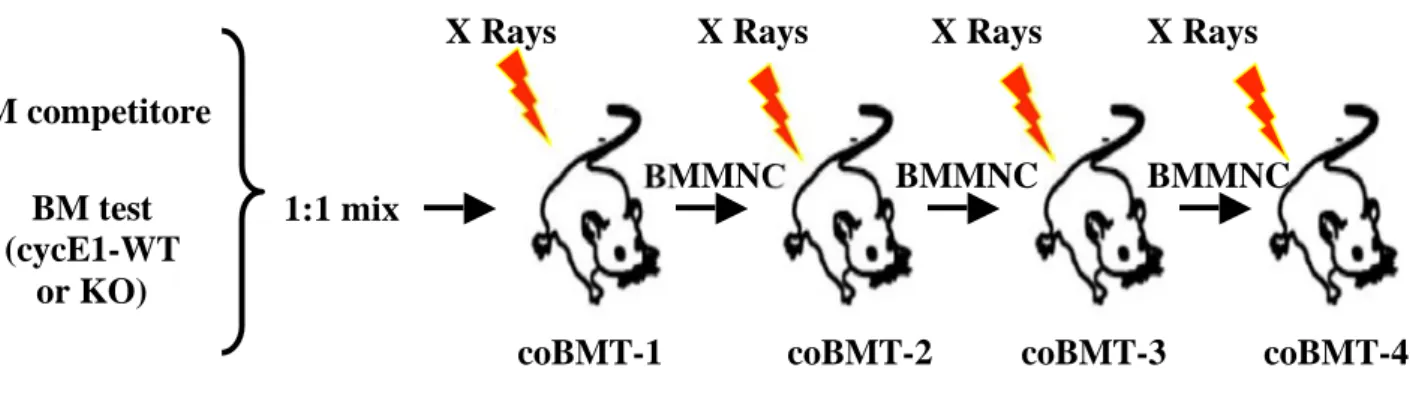
Sono stati effettuati in modo analogo un secondo ed un terzo passaggio. Il protocollo di ricostituzione seriale è identico a quello primario: ad ogni passaggio, vengono impiantate 2x106 BMMNCs (vedi Fig. 1.1.).
Figura 1.1: Saggio di ricostituzione competitiva a lungo termine: riassunto schematico dell’approccio
sperimentale adottato
Protocollo di ricostituzione primaria
1) Derivazione di BMMNCs in modo analogo ai punti 1) 2) 3) del protocollo di staining 2) Preparazione di una sospensione cellulare composta da 1x106 di BMMNC test e da 1x106 di BMMNC competitor da concentrare in un volume finale di 500 μL.
coBMT-4 coBMT-1 X Rays BM test (cycE1-WT or KO) BM competitore 1:1 mix BMMNC coBMT-2 coBMT-3 BMMNC BMMNC
5) Ricostituzione di un topo ospite letalmente irradiato mediante iniezione nella vena caudale. L’iniezione è preceduta da una fase di riscaldamento di 10 minuti in modo da provocare la dilatazione dei vasi. Il topo viene irradiato il giorno precedente alla ricostituzione con 9,5 Gy (dose di 1,7Gy al minuto) e, nel periodo successivo alla ricostituzione, viene trattato con antibiotici per tre settimane (neomicina solfato 1g/L di acqua).
7) Monitoraggio della ricostituzione a lungo termine mediante analisi del chimerismo sul sangue periferico. Il primo test è effettuato a 3 mesi dal trapianto, periodo necessario per la piena ricostituzione del compartimento ematopoietico e ripetuto al sesto mese.
Protocollo del test del sangue periferico
1) Riscaldamento dei topi per circa 10 minuti in modo da garantire la dilatazione dei vasi 2) Prelievo dalla vena caudale di circa 100 μL di sangue periferico e aggiunta di 20 μL di EDTA pH8 0,5 M come fattore anticoagulante.
3) Si effettuano 3 cicli di lisi eritrocitica usando, per ogni ciclo, 1 mL di buffer lisante freddo ( composto da 3,25g di NH4 Cl, 0,4g di KHCO3 e 0,0148 g di Na2EDTA in 300mL
di acqua distillata) per eliminare gli eritrociti
4) Staining per i marcatori di superficie Ly5.1-FITC e Ly5.2-PE.
• Incubazione di 15 minuti in ghiaccio in 50 μL di una soluzione di PBS5%BSA (blocking buffer)
• Lavaggio con 1mL di PBS
• Risospensione in un buffer di staining composto da 50 μL di PBS1%BSA contenente gli anticorpi primari anti Ly5.1 e anti Ly5.2 diluiti 1:100; incubazione di 45 minuti in ghiaccio
• Fissazione delle cellule mediante incubazione in una soluzione di formaldeide al 2% in PBS per un periodo di 15 minuti in ghiaccio
• Le cellule vanno mantenute ad una temperatura di 4°C fino all’analisi FACS
Gli anticorpi usati sono eBioscience
FITC anti-mouse CD45.1 (SJL, old Ly5.2, new Ly5.1) clone A20 codice 11-0453 PE anti-mouse CD45.2 (C57BL/6, Balb/c, old Ly5.1, new Ly5.2) clone104 codice12-0454
La percentuale del chimerismo è calcolata come segue:
% chimerism = (%test-derived cells) x 100/ (% test-derived cells + % competitor-derived cells)
2.5 Trattamento con 5-fluorouracile (5-FU)
Il 5-fluorouracile (5-FU) è un agente citotossico che causa una deleplezione mirata delle cellule in attiva proliferazione e, in questo lavoro, è stato utilizzato allo scopo di analizzare la risposta ripopolativa delle HSC allo stress.
Il sistema ematopoietico è stato analizzato 4 giorni dopo il trattamento mediante isolamento delle popolazioni staminali e caratterizzazione del loro ciclo cellulare con tecniche di FACS.
Protocollo per il trattamento con 5-FU:
1) Somministrazione mediante iniezione intraperitoneale di una soluzione sterile di 5FU (Fluorouracile TEVA 5g/mL) in PBS con una singola dose di 150mg/kg di massa corporea.
2) Derivazione di BMMNCs in modo analogo ai punti 1) 2) 3) del protocollo di staining 3) Staining degli antigeni di superficie per caratterizzare le singole popolazioni (vd protocollo)
4) Staining degli antigeni intracellulari e analisi del ciclo cellulare (vd paragrafo seguente)
2.6 Analisi del ciclo cellulare
Al fine di caratterizzare il ciclo cellulare e la proliferazione delle singole popolazioni staminali ematopoietiche in condizioni normali e in seguito a stress citotossico, è stata esaminata l’incorporazione della bromodeossiuridina
La 5-bromo2’-desossi-uridina o BrDU è un analogo della timidina e viene incorporata nel DNA neosintetizzato dalle cellule che entrano in fase S del ciclo cellulare. Essa può essere poi facilmente rilevata mediante staining con un anticorpo monoclonale anti BrDU e un’analisi al FACS, previa permeabilizzazione cellulare.
Le cellule positive per la BrDU sono cellule che stanno attivamente sintetizzando DNA, quindi sono ciclanti; per individuare precisamente la loro fase a livello di ciclo cellulare l’incorporazione di BrDU è associata ad una marcatura con Hoechst 33342, un fluorocromo utilizzato per stimare la quantità di DNA.
Protocollo di marcatura BrDU/ Hoechst 33342
E’ stato usato il protocollo di staining relativo al BrDU flow kit (BD Pharmingen) con un sistema di labelling in vivo:
1) Circa 14 ore prima del processamento, somministrazione mediante iniezione peritoneale di una singola dose di 1 gr di BrDU (100 μL di una soluzione sterile di BrDU 10 mg/mL in PBS- BD Pharmingen).
2) Derivazione di BMMNCs in modo analogo ai punti 1) 2) 3) del protocollo di staining 3) Staining degli antigeni di superficie per discriminare le singole popolazioni
4) Staining per la rivelazione della BrDU Il protocollo è per 10-15 x106cellule:
a) Fissazione e permeabilizzazione delle cellule con il BD Cytofix/Cytoperm Buffer • Risospendere le cellule in 100μL di BD Cytofix/Cytoperm Buffer
• Incubazione per 30 minuti in ghiaccio • Lavaggio con 1 mL di Perm/Wash buffer
b) Incubazione delle cellule in BD Cytoperm plus Buffer
• Risospendere le cellule in 100μL di BD Cytoperm plus Buffer • Incubazione per 10 minuti in ghiaccio
• Lavaggio con 1 mL di Perm/Wash buffer c) Ri-fissazione delle cellule
• Risospendere le cellule in 100μL di BD Cytofix/Cytoperm Buffer • Incubazione per 30 minuti in ghiaccio
• Lavaggio con 1 mL di Perm/Wash buffer
d) Trattamento delle cellule con DNAsi per esporre la BrDU incorporata
• Risospendere le cellule in 100μL di una soluzione di DNAsi 300μg/mL(soluzione stock 1mg/mL in PBS da diluire)
• Incubazione per 1 ora a 37°C
• Lavaggio con 1 mL di Perm/Wash buffer e) Staining BrDU
• Risospendere le cellule in 50μL di Perm/Wahs buffer contenente un anticorpo specifico anti-BrDU coniugato a FITC diluito 1:100
• Incubazione per 40 minuti a temperatura ambiente • Lavaggio con 1 mL di Perm/Wash buffer
f) Fissazione delle cellule mediante incubazione in 400 μL di una soluzione al 4% di formaldeide in PBS;10 minuti a temperatura ambiente.
g) Staining per la quantificazione del contenuto in DNA
• Risospendere le cellule in 1,4mL di una soluzione contenente Hoest diluito in PBS1%BSA (9μL/100mL)
• Incubazione per 30 minuti in ghiaccio • Lavaggio con 1 mL di Perm/Wash buffer • Risospensione in 800μL di PBS
Le cellule vanno mantenute al buio ed ad una temperatura di 4°C fino all’analisi FACS
2.7 Long-term culture initiating cell (LTC-IC)
E’ un saggio funzionale in vitro che si propone di fornire un’alternativa ai saggi di ripopolazione in vivo e di quantificare le cellule ematopoietiche primitive presenti in un midollo in base alla loro capacità di “ripopolare” un feeder layer di cellule stromali irradiate dando origine colonie e sostenerle a lungo termine.
Il feeder layer ha la funzione di mimare in qualche modo la nicchia ematopoietica e rilasciare i fattori necessari al mantenimento della staminalità. L’irradiamento è necessario per eliminare le possibili cellule ematopoietiche relative al donatore del midollo che possono falsare il numero di LTC-IC relative al midollo da testare.
Il saggio consiste nel piastrare un piccolo numero di cellule isolate dal midollo del topo su uno stromal layer confluente e precedentemente irradiato.
La coltura va mantenuta per circa 4 settimane: durante questo periodo si verificherà la morte di tutte le cellule terminalmente differenziate e lo sviluppo di colonie relative a tutte le cellule clonogeniche più o meno primitive presenti nella sospensione di cellule midollari.
Al termine della coltura tutte le cellule presenti nella piastra, aderenti e non, vengono staccate e nuovamente piastrate in metilcellulosa per effettuare un colony forming cell assay (CFC).
In questo modo, viene stabilita la capacità clonogenica e proliferativa delle cellule di midollo dopo ben un mese di coltura: le cellule ancora in grado di formare altre colonie e quindi di proliferare e differenziare a lungo termine sono stimate come cellule primitive. Il conteggio del numero di colonie presenti nel saggio finale, generalmente mieloidi, fornisce una misura relativamente precisa del numero di tali cellule primitive presenti nella sospensione inizialmente piastrata sul feeder layer.
Protocollo LTC-IC
1) Dissezione dei due femori e relative tibie, pulizia delle singole ossa con garze sterili e ottenimento di una sospensione cellulare mediante crashing;
2) Filtraggio della sospensione cellulare attraverso un filtro da 70μm 3) Centrifuga 10 minuti 1500 rpm a 4°C
4) Risospensione del pellett in Stromal Medium 5) Conta delle cellule con il colorante Tuerk
6) Piastratura di 50x106cellule su una piastra da 150cm di diametro
7) Coltura delle cellule per circa 15 giorni, cambiando mezzo ogni 48 ore 8) 7 giorni prima dell’ espansione delle Lin-, piastrare 800.000cellule/6wells
9) quando le cellule sono in confluenza, circa 2 giorni dopo la piastratura, cambiare il medium e conservarlo come medium condizionato a 4°C e irradiare con 15Gy
10) Il giorno dopo l’irradiamento, sostituire il medium con quello condizionato
11) Dopo altri 2/3 giorni si può procedere con la piastratura di circa 250.000 BMMNCs/6wells, ottenute come già specificato nel precedente protocollo.
Stromal medium (500mL) • IMDM+glutamax 375mL • Horse Serum 62,5mL
• Fetal calf serum nordamericano (FSC NA) 62,5mL • Penicillina/Streptomicina 100X
• 2-mercaptoetanolo 1000X • Idrocortisone 1380μL
12) Le cellule vengono lasciate in coltura sul feeder layer stromale per circa 4 settimane, cambiando opportunamente il mezzo
13) Al termine del periodo di coltura tutte le cellule presenti nella piastra, aderenti e non, vengono piastrate in colture di metilcellulosa (CFC)
14) Dopo un periodo di coltura di 12 giorni, si procede al conteggio e all’analisi di tutte le colonie: ogni colonia indica una cellula primitiva responsabile della sua origine e del suo mantenimento a lungo termine, quindi una LTC-IC.
2.8 Colony forming cell assay (CFC)
E’ un saggio staminale in vitro capace di determinare in modo quantitativo la capacità proliferativa e clonogenica di una popolazione in un preciso momento, sia che essa sia appena stata isolata dal midollo osseo che dopo un processo di coltura, come avviene nell’ LTC-IC.
Tale approccio permette di quantificare i progenitori ematopoietici multipotenti o già ristretti ad un unico lineage differenziativo e consiste nel piastrare un basso numero di cellule (25000 BMMNCs) in un medium semisolido di metilcellulosa arricchito con una combinazione di citochine specifica per il tipo di differenziamento che si vuole studiare. La metilcellulosa impedisce la mobilità cellulare e garantisce che la progenie di una cellula clonogenica rimanga nello stesso sito in cui è stata prodotta in modo che le colonie neoformate siano discrete; la specifica combinazione di citochine, invece, è responsabile della proliferazione e del differenziamento dei progenitori verso un certo lineage. Dopo un periodo di coltura di 10-15 giorni, è possibile contare il numero di colonie sviluppato ed eventualmente procedere con una classificazione della loro tipologia su base morfologica: il numero di colonie è linearmente proporzionale al numero di cellule clonogeniche, quindi progenitori o, nel caso di cellule derivanti da una coltura a lungo termine, cellule ancora più primitive.
Protocollo CFC
1) Risospensione di 25000 cellule in 250μL di RPMI
2) Aggiunta di 1250 μL di metilcellulosa arricchita con citochine
3) Miscelare bene in modo da distribuire omogeneamente le cellule e piastrare in piastre da 35mm di diametro.
La metilcellulosa supplementata è preparata nel seguente modo
a) differenziamento mieloide (per identificare i progenitori mieloidi GMP) • 8mL di metilcellulosa (Methocult SF M3226)
• 0,5 mL di RPMI (Lonza)
• 50 μL 100ng/mL di Stem cell factor o SCF (Peprotech cod 250-03 ) • 30ng/mL di Interleuchina 3 o IL3 (Peprotech cod 213-13)
• 10ng/mL di Interleuchina 6 o IL6 (Peprotech cod 216-16)
• 60ng/mL di Granulocyte Colony-Stimulating Factor o G-CSF (Peprotech cod 250-05) • 10 μL 60ng/mL di Granulocyte/Macrophage Colony-Stimulating o GM-CSF(Peprotech cod 315-03) c) differenziamento linfoide • 8mL di metilcellulosa (Methocult SF M3226) • 0,5 mL di RPMI (Lonza)
• 1,5 mL di fetal bovine serum o FBS (Stem Cell) • 20ng/mL di Flt3 ligand o FL (Peprotech cod 300-19) • 10ng/mL di interleuchina 7 o IL7 (Peprotech cod 217-17)
b) differenziamento eritroide
• 8mL di metilcellulosa supplementata con eritropoietina (Methocult SF M3334)
• 0,5 mL di RPMI (Lonza)
• 1,5 mL di fetal bovine serum o FBS (Stem Cell)
• 50 μL 100ng/mL di Stem cell factor o SCF (Peprotech cod 250-03 ) • 30ng/mL di Interleuchina 3 o IL3 (Peprotech cod 213-13)
4) Incubare a 37°C, 5% di CO2 ,95% di umidità per 12 giorni