4. RISULTATI
Durante il mio lavoro di ricerca ho applicato tecniche di biologia molecolare per verificare l’ipotesi che gli effetti a lungo termine indotti dal CFC, sia a livello comportamentale che elettrofisiologico, siano dovuti ad una modulazione di specifici geni a livello delle regioni medio-temporali del cervello di ratto.
4.1 Risultati dello screening primario di geni candidati a
essere differenzialmente espressi in seguito a CFC
Precedentemente al mio internato di tesi, sono state costruite e clonate due librerie sottrattive di cDNA utilizzando la tecnica dell’SSH: la libreria forward, contenente i trascritti dei geni modulati positivamente dal CFC, e la libreria reverse, costituita, invece, dai trascritti dei geni modulati negativamente da questo tipo di condizionamento. La tecnica della SSH è un metodo molto efficiente di analisi dei patterns di espressione genica che permette di ottenere librerie sottrattive di cDNA arricchite di trascritti differenzialmente espressi. Tuttavia le librerie così costruite possono ancora contenere dei cloni comuni che possono rappresentare dei falsi positivi. Tali cloni sono eliminati tramite lo
Screening primario e i cloni rimanenti rappresentano le sequenze dei
geni candidati a essere differenzialmente espressi dopo il CFC. In questa tesi sono riportati i dati relativi allo screening primario di cloni sia della libreria forward che reverse.
Nella prima parte dello screening, gli inserti sono stati amplificati tramite PCR da colonia, utilizzando primers complementari alle regioni presenti ai due lati del sito di clonaggio. I trascritti amplificati sono stati analizzati su un gel di agarosio al 2%. I cloni sono stati fatti correre con un marker di peso molecolare e sono stati selezionati solo i cloni che presentano un amplificato unico. L’eterogeneità dei pesi molecolari dei prodotti di PCR suggerisce che la maggior parte dei cloni non è ridondante e quindi che i cloni sono altamente rappresentativi (Fig.11).
Fig.11: Colony PCR di alcuni dei cloni ottenuti. Lane 1: Marker IX (New England BioLabs);
Lanes 2-21: prodotti di PCR da colonia di alcuni dei cloni ottenuti.
Per ciascun clone, 1 l prodotto di colony PCR è stato trasferito, dopo esser stato denaturato, sulle due membrane. Le membrane sono state ibridate utilizzando come sonde i cDNA delle due librerie sottrattive marcate non radioattivamente (Paragrafo 3.9).
Nella Fig.12 a pag. 65, sono mostrati i risultati ottenuti dall’ibridazione di alcuni cloni della libreria forward con la sonda forward (1) e la sonda
reverse (2).
Fig.12: Segnali di ibridazione di cloni forward con la sonda della libreria forward (1) e
reverse (2).
In base ai segnali di ibridazione ottenuti (interpretati secondo il protocollo descritto nel PCR-Select Differential Screening Kit User
Manual, BD Biosciences), sono stati selezionati da entrambe le librerie
quei cloni che mostrano un segnale positivo con una sonda ma non con l’altra, o che, comunque, mostrano un segnale differente nell’ibridazione con le due sonde (alcuni cloni selezionati sono indicati dalle frecce nella Fig.12). Le sequenze dei cloni scelti, tramite lo screening primario, rappresentano i geni candidati ad essere differenzialmente espressi in seguito a CFC nelle regioni medio-temporali nel cervello di ratto.
4.2 Analisi delle sequenze
In seguito allo screening primario sono stati individuati 58 cloni candidati ad essere differenzialmente espressi per la libreria forward e 30 cloni per la libreria reverse. In seguito all’analisi bioinformatica delle
loro sequenze è stato possibile suddividerli in categorie funzionali distinte quali: metabolismo mitocondriale, turn-over proteico, trasduzione del segnale, formazione e rilascio di vescicole, regolazione della trascrizione, funzioni varie e funzione sconosciuta. Nella Fig.13, la percentuale riportata per ognuna delle categorie funzionali rappresenta il numero dei cloni che hanno mostrato similarità con sequenze geniche depositate in banca dati la cui funzione è indicata dalla categoria stessa.
Fig.13: Suddivisione funzionale dei geni individuati con lo screening primario.
In questa tesi sperimentale sono discussi i cloni “5ND3”, “5VD6”, “5VF8” e “7NB6” le cui sequenze appartengono alle categorie funzionali “Trasduzione del segnale” e “Formazione e rilascio di vescicole”.
Turn-over proteico 17% Metabolismo mitocondriale 31% Apoptosi 3% Trasduzione del segnale 20% Funzioni varie 14% Formazione e rilascio di vescicole 8% Regolazione trascrizione 4% Funzione sconosciuta 3%
4.2.1 Sequenza del clone 5ND3
La sequenza ottenuta tramite sequenziamento automatico per il clone 5ND3 è la seguente:
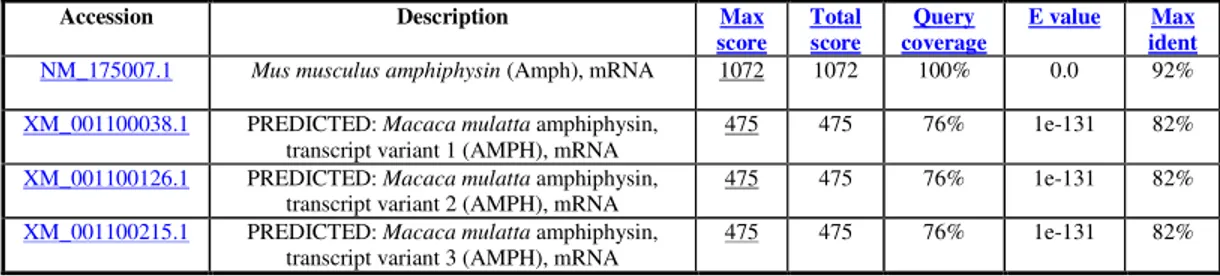
ed è stata confrontata con le sequenze depositate in banca dati (GenBank), utilizzando programmi come: BLASTN e FASTA. Nella tabella 2 sono riportati i risultati ottenuti con il BLASTN.
Tabella 2: Risultati ottenuti con BLASTN per la sequenza del clone 5ND3.
La sequenza che mostra più alta similarità è una porzione della regione 3’-UTR della sequenza NM175007.1 nella regione compresa fra i
5’…ACTTGATTCCCCTCCCACCAAATCTAAAGTAGCTTTCTCCAGACCAGAC TTTGACTTCTTTGTATCAAGTGTGTGGTGTTGAGTGATTGCCCTGCAGATGTG ATGAGTTATCCTGTAGTAAGACATTATCTTACTCCCTTGCCCGATGTGAGTG AGCACGTGCTCTCTTTTCCTTTCAAGTTTACTGCGATTAAAACTGAACAGCT GCAAACGTTCTTAACAGTCTTTTCCAATCACCTCTATATACATCTATACGGA CACACAGTCGCACAAGCTAGTTCCCATGCCTGCTGGACCTATGTGTATGCAG ATGGCTACATATACATAGCTGATTCCTTCCCTTTCCACTGTAGCTCTTCGCCT TCGTCATTGATACACGCATTGCTCTTATTCGTGGACATTACAGCTGCAGTTTT GCCTTGAGGCAAAACCACTCATCCCATTGCCAAAACAGTTAGTTGACAGTG TCTGTGTGTGTTGTGTGCAGTTTTCAGGATGATATAACAGATACAGTGTCTA CGCTGCTGCCCCTTTGGAGAGAGCAGGGCCTGGTTGTATAGCTGCCCAGCTG CTCAGTCCAGTAAATCCATGGACCAGCAACTATGCAGTCATCATTTCTGCCT TTATATGATTGGCTGGATGAGGGGGCTAAAGAACCTATTTACAAAAGCTCC TATATACTTCCACATTCTATTTATGCAACTCATCTTCACAGATCATTCTAAAA TGAACGAGGTGATGTATGCTGT…3’
Accession Description Max
score Total score coverageQuery E value identMax
NM_175007.1 Mus musculus amphiphysin (Amph), mRNA 1072 1072 100% 0.0 92%
XM_001100038.1 PREDICTED: Macaca mulatta amphiphysin,
transcript variant 1 (AMPH), mRNA 475 475 76% 1e-131 82%
XM_001100126.1 PREDICTED: Macaca mulatta amphiphysin,
transcript variant 2 (AMPH), mRNA 475 475 76% 1e-131 82%
XM_001100215.1 PREDICTED: Macaca mulatta amphiphysin,
nucleotidi 2378 e 3124. Tale sequenza codifica per l’mRNA dell’ Amfifisina, come mostrato dal seguente allineamento:
ref|NM_175007.1| Mus musculus amphiphysin (Amph), mRNA
Score = 1072 bits (580), Expect = 0.0
Identities = 702/758 (92%), Gaps = 20/758 (2%) Strand=Plus/Plus 5ND3 ACTTGATTCCCCTCCCACCAAATCTAAAGTAGCTTTCTCCAGACCAGACTTTGACTTCTT 60 ||||||||| ||||||||||| ||||||||||| |||||||||||||||||||||||||| Sbjct 2378 ACTTGATTCTCCTCCCACCAAGTCTAAAGTAGCCTTCTCCAGACCAGACTTTGACTTCTT 2437 5ND3 TGTATCAAGTGTGTGGTGTTGAGTGATTGCCCTGCAGATGTGATGAGTTATCCTGTAGTA 120 || |||||||||||||||| ||||| ||||||||||||||||||||||||||||||||| Sbjct 2438 TGCATCAAGTGTGTGGTGTCGAGTGGTTGCCCTGCAGATGTGATGAGTTATCCTGTAGTG 2497 5ND3 AGACATTATCTTACTCCCTTGCCCG-ATGTGAGTGAGCACGTGCTCTCTTTTCCTTTCAA 179 |||||| |||||||||||||||||| || | |||||| | ||||||||| |||||||| Sbjct 2498 AGACATCATCTTACTCCCTTGCCCGAAT-T--GTGAGCTCCTGCTCTCTTGCCCTTTCAA 2554 5ND3 GTTTACTGCGATTAAAACTGAACAGCTGCAAACGTTCTTAACAGTCTTTTCCAATCACCT 239 |||||||||| ||||||||||||||||||||||||||||||| ||||| ||||||||||| Sbjct 2555 GTTTACTGCGTTTAAAACTGAACAGCTGCAAACGTTCTTAACGGTCTTCTCCAATCACCT 2614 5ND3 CTATATACATC-TATACGGACACACAGTCGCACAAGCTAGTTCCCATGCCTGCTGGACCT 298 ||||||||| | | |||||||||||||||||||||||||| ||||||||||||||||||| Sbjct 2615 CTATATACA-CATGTACGGACACACAGTCGCACAAGCTAG-TCCCATGCCTGCTGGACCT 2672 5ND3 ATGTGTATGCAGATGGCTACATATACATAGCTGATTCCTTCCCTTTCCACTGTAGCTCTT 358 ||||||||||||||||||||||||||||||||||| |||||||||||||||||||||||| Sbjct 2673 ATGTGTATGCAGATGGCTACATATACATAGCTGATCCCTTCCCTTTCCACTGTAGCTCTT 2732 5ND3 CGCCT-T---CGTCATTGATACACGCATTGCTCTTATTCGT-GGACATTACAGCTGCAGT 413 | ||| | ||||||||||||||||||||||||||| || || ||||||||||||||| Sbjct 2733 CTCCTCTAAGTGTCATTGATACACGCATTGCTCTTATTTGTCGG-CATTACAGCTGCAGT 2791 5ND3 TTTGCCTTGAGGCAAAACCACTCATCCCATTGCCAAAACAGTTAGTTGACAgtgtctgtg 473 ||||||||| |||||||||| |||||||||||||||||| ||||||||||||||||| Sbjct 2792 TTTGCCTTGGGGCAAAACCATTCATCCCATTGCCAAAAC----AGTTGACAGTGTCTGTG 2847 5ND3 tgtgttgtgtgCAGTTTTCAGGATGATATAACAGATA-CAGTGTCTACGCTGCTGCCCCT 532 ||||||||||||||||||||||||||||||||||||| | | ||| |||||||||||||| Sbjct 2848 TGTGTTGTGTGCAGTTTTCAGGATGATATAACAGATAGC-GCGTCCACGCTGCTGCCCCT 2906 5ND3 TTGGAGAGAGCAGGGCCTGGTTGTATAGCTGCCCAGCTGCTCAGTCCAG-TAAATCCATG 591 ||||| |||||||||||||||| | |||||||||||||||||||||||| ||||| |||| Sbjct 2907 TTGGAAAGAGCAGGGCCTGGTTATGTAGCTGCCCAGCTGCTCAGTCCAGATAAATACATG 2966 5ND3 GACCAGCAACTATGCAGTCATCATTTCTGCCTTTATATGATTGGCTGGATGAGGGGGCTA 651 ||| ||||||||||||||||||||| ||||||||| |||||||||||| |||||||| | Sbjct 2967 GACTAGCAACTATGCAGTCATCATTCCTGCCTTTACGTGATTGGCTGGACGAGGGGGCAA 3026 5ND3 AAGAACCTATTTACAAAAGCTCCTATATACTTCCACATTCTATTTATGCAACTCATCTTC 711 ||||||||||||| |||||||||||||||||||||| ||||||||||||||||||||||| Sbjct 3027 AAGAACCTATTTATAAAAGCTCCTATATACTTCCACGTTCTATTTATGCAACTCATCTTC 3086 5ND3 ACAGATCATTCTAAAATGAACGAGGTGATGTATGCTGT 749 |||||||||||||||||||||||||||||||||||||| Sbjct 3087 ACAGATCATTCTAAAATGAACGAGGTGATGTATGCTGT 3124
La sequenza del clone 5ND3 mostra similarità di sequenza con le sequenze degli mRNA di varie isoforme della proteina amfifisina nel topo, nel macaco e nell’Uomo. L’identità di sequenza con l’amfifisina di ratto non è rilevata. È stato quindi ripetuta l’analisi con il BLASTN scegliendo, anzichè il database Reference RNA Sequences, quello
Reference Genomic Sequences del genoma del ratto. Di seguito è
riportato l’allineamento ottenuto:
ref|NW_047492.2|Rn17_WGA2014_4 Rattus norvegicus chromosome 17 genomic contig, reference assembly; Features flanking this part of subject sequence:
24085 bp at 5' side: CDS 253 bp at 3' side: amphiphysin 1 5ND3 ACTTGATTCCCCTCCCACCAAATCTAAAGTAGCTTTCTCCAGACCAGACTTTGACTTCTT 60 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900649 ACTTGATTCCCCTCCCACCAAATCTAAAGTAGCTTTCTCCAGACCAGACTTTGACTTCTT 6900590 5ND3 TGTATCAAGTGTGTGGTGTTGAGTGATTGCCCTGCAGATGTGATGAGTTATCCTGTAGTA 120 || ||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900589 TGCATCAAGTGTGTGGTGTTGAGTGATTGCCCTGCAGATGTGATGAGTTATCCTGTAGTA 6900530 5ND3 AGACATTATCTTACTCCCTTGCCCGATGTGAGTGAGCACGTGCTCTCTTTTCCTTTCAAG 180 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900529 AGACATTATCTTACTCCCTTGCCCGATGTGAGTGAGCACGTGCTCTCTTTTCCTTTCAAG 6900470 5ND3 TTTACTGCGATTAAAACTGAACAGCTGCAAACGTTCTTAACAGTCTTTTCCAATCACCTC 240 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900469 TTTACTGCGATTAAAACTGAACAGCTGCAAACGTTCTTAACAGTCTTTTCCAATCACCTC 6900410 5ND3 TATATACATCTATACGGACACACAGTCGCACAAGCTAGTTCCCATGCCTGCTGGACCTAT 300 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900409 TATATACATCTATACGGACACACAGTCGCACAAGCTAGTTCCCATGCCTGCTGGACCTAT 6900350 5ND3 GTGTATGCAGATGGCTACATATACATAGCTGATTCCTTCCCTTTCCACTGTAGCTCTTCG 360 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900349 GTGTATGCAGATGGCTACATATACATAGCTGATTCCTTCCCTTTCCACTGTAGCTCTTCG 6900290 5ND3 CCTTCGTCATTGATACACGCATTGCTCTTATTCGTGGACATTACAGCTGCAGTTTTGCCT 420 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900289 CCTTCGTCATTGATACACGCATTGCTCTTATTCGTGGACATTACAGCTGCAGTTTTGCCT 6900230 5ND3 TGAGGCAAAACCACTCATCCCATTGCCAAAACAGTTAGTTGACAgtgtctgtgtgtgttg 480 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900229 TGAGGCAAAACCACTCATCCCATTGCCAAAACAGTTAGTTGACAGTGTCTGTGTGTGTTG 6900170 5ND3 tgtgCAGTTTTCAGGATGATATAACAGATACAGTGTCTACGCTGCTGCCCCTTTGGAGAG 540 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900169 TGTGCAGTTTTCAGGATGATATAACAGATACAGTGTCTACGCTGCTGCCCCTTTGGAGAG 6900110
Questa seconda analisi bio-informatica della sequenza del clone 5ND3 dimostra che c’è una discrepanza tra la sequenza dell’mRNA e il modello del gene. La discrepanza può essere causata dall’allineamento delle sequenze o dalla sequenza della proteina tradotta. L’allineamento può presentare, infatti, dei gaps o presentare similarità con due o più regioni genomiche. Per quanto riguarda invece la proteina possono essere rilevate differenze tra la sequenza aminoacidica ottenuta da una sequenza di mRNA identificata e la sequenza ottenuta a partire dalla sequenza dell’intero gene oppure dalla terminazione prematura della regione codificante nella sequenza genomica.
4.2.2 Sequenza del clone 5VD6
La sequenza ottenuta tramite sequenziamento automatico per il clone 5VD6 è la seguente: 5’…TCTACAGGAACAGACCATAATACCAGCTGTCAGACAGCGTAAGGAACGGAGAG GTGGCATCTCCTGTGATCGGAGAGAAGATGGTTTGAGCCGGAGCAGAGCCATGGGC ACGTCTTCAGAAAAGGCCTGCTAGTTGGACCCACCTTGTGAGCACTGATCTGTGTG GATGATGTCCAAGTTACCACATGAGACTGAACCAGATCAGAAGCTCCTGCAAAAAG 5ND3 AGCAGGGCCTGGTTGTATAGCTGCCCAGCTGCTCAGTCCAGTAAATCCATGGACCAGCAA 600 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 6900109 AGCAGGGCCTGGTTGTATAGCTGCCCAGCTGCTCAGTCCAGTAAATCCATGGACCAGCAA 6900050 5ND3 CTATGCAGTCATCATTTCTGCCTTTATATGATTGGCTGGATGAGGGGGCTAAAGAACCTA 660 |||||||||||||||||||||||||||||||||||||| ||||||||||||||||||||| Sbjct 6900049 CTATGCAGTCATCATTTCTGCCTTTATATGATTGGCTGTATGAGGGGGCTAAAGAACCTA 6899990 5ND3 TTTACAAAAGCTCCTATATACTTCCACATTCTATTTATGCAACTCATCTTCACAGATCAT 720 ||||||||||||||||||||||| |||||||||||||||||||||||||||||||||||| Sbjct 6899989 TTTACAAAAGCTCCTATATACTTTCACATTCTATTTATGCAACTCATCTTCACAGATCAT 6899930 5ND3 TCTAAAATGAACGAGGTGATGTATGCTGT 749 ||||||||||||||||||||||||||||| Sbjct 6899929 TCTAAAATGAACGAGGTGATGTATGCTGT 6899901
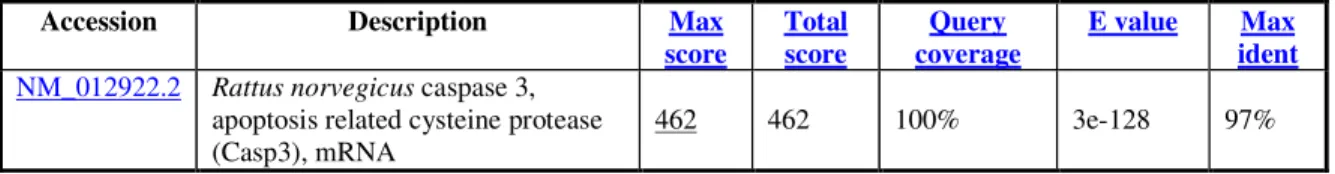
ed è stata confrontata con le sequenze depositate in banca dati (GenBank), utilizzando programmi come: BLASTN e FASTA. Nella tabella 3 sono riportati i risultati ottenuti con il BLASTN.
Tabella 3: Risultati ottenuti con BLASTN per la sequenza del clone 5VD6.
La sequenza che mostra similarità più alta è una porzione della regione 3’-UTR della sequenza NM012922.2, nella regione compresa fra i nucleotidi 1723 e 1994. Tale sequenza codifica per l’mRNA della Caspasi 3, come mostrato dal seguente allineamento:
ref|NM_012922.2| Rattus norvegicus caspase 3, apoptosis related cysteine protease (Casp3), mRNA 5VD6 TCTACAGGAACAGACCATAATACCAGCTGTCAG--ACAGCGTAAGGAACGGAGAGGTGGC 58 ||||||||||||||||||||||||||||||||| | ||||||||||| ||||||||||| Sbjct 1723 TCTACAGGAACAGACCATAATACCAGCTGTCAGTCAGAGCGTAAGGAAAGGAGAGGTGGC 1782 5VD6 ATCTCCTGTGATCGGAGAGAAGATGGTTTGAGCCGGAGCAGAGCCATGGGCACGTCTTCA 118 |||||||||||| ||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 1783 ATCTCCTGTGATTGGAGAGAAGATGGTTTGAGCCGGAGCAGAGCCATGGGCACGTCTTCA 1842 5VD6 GAAAAGGCCTGCTAGTTGGACCCACCTTGTGAGCACTGATCTGTGTGGATGATGTCCAAG 178 |||||||||||||||||||||||||||||||||||||| ||||||||||||||||||||| Sbjct 1843 GAAAAGGCCTGCTAGTTGGACCCACCTTGTGAGCACTGGTCTGTGTGGATGATGTCCAAG 1902 5VD6 TTACCACATGAGACTGAACCAGATCAGAAGCTCCTGCAAAAAGGCTTTCCCAGAGTGCTG 238 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 1903 TTACCACATGAGACTGAACCAGATCAGAAGCTCCTGCAAAAAGGCTTTCCCAGAGTGCTG 1962 5VD6 AGCGTCTCAAGAAAGTTCACTGGACTCCATGT 270 ||| |||||||||||||||||||||||||||| Sbjct 1963 AGCATCTCAAGAAAGTTCACTGGACTCCATGT 1994
Accession Description Max
score Total score coverage Query E value ident Max
NM_012922.2 Rattus norvegicus caspase 3, apoptosis related cysteine protease
4.2.3 Sequenza del clone 5VF8
La sequenza ottenuta tramite sequenziamento automatico per il clone 5VF8 è la seguente:
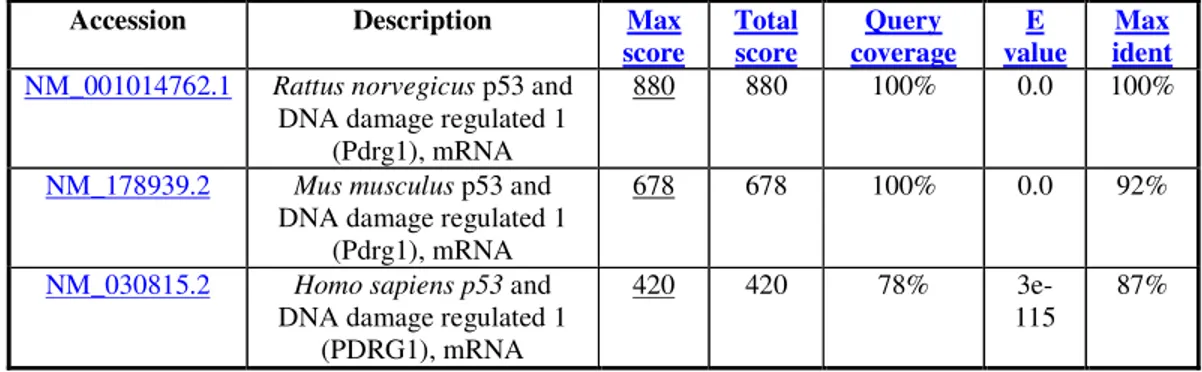
ed è stata confrontata con le sequenze depositate in banca dati (GenBank), utilizzando programmi come: BLASTN e FASTA. Nella tabella 4 sono riportati i risultati ottenuti con il BLASTN.
Accession Description Max
score Total score coverageQuery valueE identMax
NM_001014762.1 Rattus norvegicus p53 and
DNA damage regulated 1 (Pdrg1), mRNA
880 880 100% 0.0 100%
NM_178939.2 Mus musculus p53 and
DNA damage regulated 1 (Pdrg1), mRNA
678 678 100% 0.0 92%
NM_030815.2 Homo sapiens p53 and
DNA damage regulated 1 (PDRG1), mRNA
420 420 78%
3e-115 87%
Tabella 4: Risultati ottenuti con BLASTN per la sequenza del clone 5VF8.
La sequenza che mostra similarità più alta è con gran parte della CDS e una piccola parte del 3’-UTR della sequenza NM001014762.1, nella regione compresa fra i nucleotidi 68 e 543. Tale sequenza codifica nella
5’…ACCTGGTCGAAGTGGAGGAGCTGGCAGAGGCAGTGCTCTCAGACAAG CGGCAGATTGTAGACCTGGACACCAAGAGGAATCAGAACCGAGAGGGCC TGAGGGCCCTACAGAAAGACCCGAGTGTCTCTGAAGACGTGATGGTTTGC TTTGGGAACATGTTCATCAAAATGCCTCACCTTAAGACGAAGGAAATGAT CCAGAAAGATCAGGAGCACCTGGATAAAGAAATAGAAAGGCTGCGGAGC CAACTGAAAGTGAAAGTTAACCGCCTCCTAGAAGCCCAAGGAAAACCGGA GCTAAAGGGTTTTAACCTGAACCCCCTCAACCAGGATGAGCTTAAAGCCC TCCAGGTCATCTTGAAAGGATGAGACTCACAGCAGGATGGGGACCTGTGA CTAGGACCCTCCCCAGCCTTGAGACCTGCAAGGACAGGATTGTCTGGTCA AGGGGACAGGCATAAGGAATGTGGCTTCTGT…3’
regione codificante dell’mRNA della proteina P53, come mostrato dal seguente allineamento:
Qui di seguito è mostrata la sequenza amminoacidica tradotta a partire dalla sequenza del clone tramite il programma ExPASy-Translate Tool:
…T Met V S P E A D R V L R Y L V E V E E L A E A V L S D K R Q I V D L D T K R N Q N R E G L R A L Q K D P S V S E D V Met V C F G N Met F I K Met P H L K T K E Met I Q K D Q E H L D K E I E R L R S Q L K V K V N R L L E A Q G K P E L K G F N L N P L N Q D E L K A L Q V I L K G…
ref|NM_001014762.1| Rattus norvegicus p53 and DNA damage regulated 1 (Pdrg1), mRNA 5VF8 ACCTGGTCGAAGTGGAGGAGCTGGCAGAGGCAGTGCTCTCAGACAAGCGGCAGATTGTAG 60 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 68 ACCTGGTCGAAGTGGAGGAGCTGGCAGAGGCAGTGCTCTCAGACAAGCGGCAGATTGTAG 127 5VF8 ACCTGGACACCAAGAGGAATCAGAACCGAGAGGGCCTGAGGGCCCTACAGAAAGACCCGA 120 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 128 ACCTGGACACCAAGAGGAATCAGAACCGAGAGGGCCTGAGGGCCCTACAGAAAGACCCGA 187 5VF8 GTGTCTCTGAAGACGTGATGGTTTGCTTTGGGAACATGTTCATCAAAATGCCTCACCTTA 180 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 188 GTGTCTCTGAAGACGTGATGGTTTGCTTTGGGAACATGTTCATCAAAATGCCTCACCTTA 247 5VF8 AGACGAAGGAAATGATCCAGAAAGATCAGGAGCACCTGGATAAAGAAATAGAAAGGCTGC 240 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 248 AGACGAAGGAAATGATCCAGAAAGATCAGGAGCACCTGGATAAAGAAATAGAAAGGCTGC 307 5VF8 GGAGCCAACTGAAAGTGAAAGTTAACCGCCTCCTAGAAGCCCAAGGAAAACCGGAGCTAA 300 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 308 GGAGCCAACTGAAAGTGAAAGTTAACCGCCTCCTAGAAGCCCAAGGAAAACCGGAGCTAA 367 5VF8 AGGGTTTTAACCTGAACCCCCTCAACCAGGATGAGCTTAAAGCCCTCCAGGTCATCTTGA 360 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 368 AGGGTTTTAACCTGAACCCCCTCAACCAGGATGAGCTTAAAGCCCTCCAGGTCATCTTGA 427 5VF8 AAGGATGAGACTCACAGCAGGATGGGGACCTGTGACTAGGACCCTCCCCAGCCTTGAGAC 420 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 428 AAGGATGAGACTCACAGCAGGATGGGGACCTGTGACTAGGACCCTCCCCAGCCTTGAGAC 487 5VF8 CTGCAAGGACAGGATTGTCTGGTCAAGGGGACAGGCATAAGGAATGTGGCTTCTGT 476 |||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 488 CTGCAAGGACAGGATTGTCTGGTCAAGGGGACAGGCATAAGGAATGTGGCTTCTGT 543
4.2.4 Sequenza del clone 7NB6
La sequenza ottenuta tramite sequenziamento automatico per il clone 7NB6 è la seguente:
ed è stata confrontata con le sequenze depositate in banca dati (GenBank), utilizzando programmi come: BLASTN e FASTA. Nella tabella 5 sono riportati i risultati ottenuti con il BLASTN.
Accession Description Max
score Total score coverageQuery valueE identMax
NM_017166.1 Rattus norvegicus stathmin
1 (Stmn1), mRNA 617 617 100% 175 8e- 99%
NM_019641.3 Mus musculus stathmin 1
(Stmn1), mRNA 538 538 100% 151 6e- 95%
Tabella 5: Risultati ottenuti con BLASTN per la sequenza del clone 7NB6.
La sequenza che mostra similarità più alta è una porzione della regione 3’-UTR della sequenza NM017166.1, nella regione compresa fra i nucleotidi 674 e 1017. Tale sequenza codifica per l’mRNA della Statmina 1, come mostrato dal seguente allineamento:
5’…ACTGGCCAGTGTCATTTACTTTTTCCCTCCTGACAAATATTCTAGAA GCTGATGTAGGACCGTATAGGTAGATCCAGACCGTGAGATGTTTTAGG GGCTCAAGGGGAGAAACTGAAAGTGTTTTGCTCTTTTTTAAAGTGTTGG TCTTTCTAACGTAGCTATTTTTCTCGTTGCATCTTTTCCTCTCGGGCACA CTCGGTGTGCTGGGTTAATGGCTAGTGCTGTATTGACTGTGGAAGACGT TCGTGAAGAGTATGTAGTGGCTTCTTCCAACCCATTAGATGCTGAGTAT CTGTTCACTTTGCGATCCCAATTCTGTCCCAATCTCACCAGATGCTACT GT…3’
4.3 Risultati dell’analisi di espressione tramite
RT-REAL-TIME PCR
Per analizzare l’espressione dei geni con cui i cloni 5ND3, 5VD6, 5VF8 e 7NB6 hanno mostrato omologia di sequenza è stata utilizzata la tecnica della RT-PCR real-time. I livelli di espressione dei geni di interesse rispetto al gene housekeeping G3PDH sono stati calcolati con il metodo del 2-∆∆CT (paragrafi 3.13 e 3.14). In particolare è emerso che il CFC modula negativamente l’espressione dell’mRNA dell’amfifisina e positivamente gli mRNA di caspasi 3, p53 e statmina. Nella Fig.14(A) a pag.76 è mostrato che, dopo il CFC, nelle regioni medio-temporali del cervello di ratto l’espressione del gene dell’amfifisina diminuisce della metà rispetto al campione CFC. Nella Fig.14(B) a pag.77 è mostrato che, dopo il CFC, nelle regioni medio-temporali del cervello di ratto
ref|NM_017166.1| Rattus norvegicus stathmin 1 (Stmn1), mRNA
7NB6 ACTGGCCAGTGTCATTTACTTTTTCCCT-CCTGACAAATATTCTAGAAGCTGATGTAGG- 58 |||||||||||||||||||||||| ||| |||||||||||||||||||||||||||||| Sbjct 674 ACTGGCCAGTGTCATTTACTTTTT-CCTCCCTGACAAATATTCTAGAAGCTGATGTAGGA 732 7NB6 ACCGTATAGGTAGATCCAGACCGTGAGATGTTTTAGGGGCTCAAGGGGAGAAACTGAAAG 118 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 733 ACCGTATAGGTAGATCCAGACCGTGAGATGTTTTAGGGGCTCAAGGGGAGAAACTGAAAG 792 7NB6 TGTTTTGCTCTTTTTTAAAGTGTTGGTCTTTCTAACGTAGCTATTTTTCTCGTTGCATCT 178 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 793 TGTTTTGCTCTTTTTTAAAGTGTTGGTCTTTCTAACGTAGCTATTTTTCTCGTTGCATCT 852 7NB6 TTTCCTCTCGGGCACACTCGGTGTGCTGGGTTAATGGCTAGTGCTGTATTGACTGTGGAA 238 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 853 TTTCCTCTCGGGCACACTCGGTGTGCTGGGTTAATGGCTAGTGCTGTATTGACTGTGGAA 912 7NB6 GACGTTCGTGAAGAGTATGTAGTGGCTTCTTCCAACCCATTAGATGCTGAGTATCTGTTC 298 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 913 GACGTTCGTGAAGAGTATGTAGTGGCTTCTTCCAACCCATTAGATGCTGAGTATCTGTTC 972 7NB6 ACTTTGCGATCCCAATTCTGTCCCAATCTCACCAGATGCTACTGT 343 ||||||||||||||||||||||||||||||||||||||||||||| Sbjct 973 ACTTTGCGATCCCAATTCTGTCCCAATCTCACCAGATGCTACTGT 1017
campione naïve. Nella Fig.14(C) a pag.77 è mostrato che, dopo il CFC, nelle regioni medio-temporali del cervello di ratto l’espressione del gene p53 aumenta più del doppio rispetto al campione naïve. Nella Fig.14(D) a pag 78 è mostrato che, dopo il CFC, nelle regioni medio-temporali del cervello di ratto l’espressione del gene statmina 1 aumenta di due volte rispetto al campione naïve. I dati ottenuti per entrambi i geni sono statisticamente significativi (unpaired t test, p < di 0,05 n= 9).
Fig.14 (A): Livelli di espressione del mRNA dell’amfifisina calcolato con il metodo 2-∆∆CT.
(*)L’analisi statistica è stata eseguita tramite unpaired t test (p < 0.05, n = 9). I dati sono mostrati come le medie ± S.D (naïve 1± 0.177; CFC 0.456± 0.134).
naive CFC 0.00 0.25 0.50 0.75 1.00 1.25
*
A m ph ip hy si n / G 3P D H m R N A(B): Livelli di espressione del mRNA della caspasi 3 calcolato con il metodo 2-∆∆CT.
(*)L’analisi statistica è stata eseguita tramite unpaired t test (p < 0.05, n = 9). I dati sono mostrati come le medie ± S.D (naïve 1± 0.189; CFC 3.165± 0.228).
(C): Livelli di espressione del mRNA della p53 calcolato con il metodo 2-∆∆CT. (*)L’analisi
statistica è stata eseguita tramite unpaired t test (p < 0.05, n = 9). I dati sono mostrati come le medie ± S.D (naïve 1± 0.163; CFC 2.23± 0.227) CFC 0.0 0.5 1.0 1.5 2.0 2.5 3.0 3.5 C as pa se 3 / G 3P D H m R N A naïve
*
CFC 0.0 0.5 1.0 1.5 2.0 2.5 P5 3/ G 3P D H m R N A naïve*
(D): Livelli di espressione del mRNA della statmina calcolato con il metodo 2-∆∆CT.
(*)L’analisi statistica è stata eseguita tramite unpaired t test (p < 0.05, n = 9). I dati sono mostrati come le medie ± S.D (naïve 1± 0.215; CFC 2.020± 0.239).