3.1
I
SOLAMENTO DEL CDNA
DEL GENELEAFY
COTYLEDON1-LIKE
INHELIANTHUS ANNUUS.
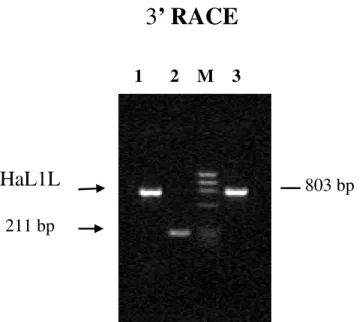
Il cDNA del gene LEAFY COTYLEDON1-LIKE (L1L) è stato isolato dalla specie Helianthus annuus per mezzo delle tecniche 3’ e 5’RACE. Inizialmente sono stati usati oligonucleotidi degenerati in grado di amplificare una porzione del cDNA specifica per il gene L1L a partire da RNA estratto da embrioni immaturi di 5-7 giorni circa dalla presunta data di fecondazione del fiore del disco (5-7 GDF; giorni dopo la fecondazione). Gli oligonucleotidi (Tabella 1) sono stati scelti su un allineamento, ottenuto con il programma ClustalW, di sequenze aminoacidiche, depositate in banca-dati, relative alle proteine LEAFY COTYLEDON1 (LEC1) e LEAFY COTYLEDON1-LIKE (L1L) di Arabidopsis thaliana e della proteina omologa a L1L in Phaseolus coccineus (PcL1L). Con l’oligonucleotide (LEC2) specifico per L1L, nella regione 5’, è stata condotta la reazione di 3’ RACE. L’elettroforesi su gel d’Agarosio 2% dei prodotti d’amplificazione ha evidenziato un frammento di cDNA della dimensione attesa di circa 803 bp (Figura. 7).
3’
RACE
Figura 7: Elettroforesi su gel di Agarosio al 2% colorato con Bromuro di Etidio. Corsia 1 e 3 cDNA per HaL1L, amplificato con la coppia di oligonucleotdi LEC2-P9 nella reazione di 3’ RACE. Corsia 2, amplificazione con coppia di oligonucleotidi VL1-VL2 collocati all’interno dell’amplificato LEC2-P9. Corsia M, “Marker”di pesi molecolari.
Il frammento amplificato, clonato e quindi sequenziato contiene sia una porzione codificante di circa 628 bp sia la regione 3’ UTR con la coda di poli(A) di circa 175 bp. L’utilizzo del software Blast-N, che individua sequenze omologhe nelle banche-dati, ha indicato che il frammento sequenziato appartiene al gene L1L e codifica per una porzione della proteina attesa. Esso presenta, infatti, un alto grado di similarità con la sequenza dei geni LEAFY COTYLEDON1-LIKE di Arabidopsis thaliana e Phaseolus coccineus. Sulla sequenza nucleotidica, ottenuta dalla 3’ RACE, per Helianthus annus sono stati disegnati oligonucleotidi specifici utilizzati nella successiva 5’ RACE (Tabella 1).
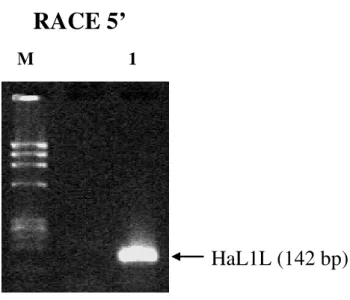
Il prodotto d’amplificazione, ottenuto con l’oligonucleotide CHI 5-1 ed evidenzato mediante corsa elettroforetica su gel d’Agarosio 2% (Figura. 8 ), è un frammento di cDNA della dimensione attesa di circa 142 bp.
HaL1L
1 2 M 3
803 bp 211 bp
Figura 8: Elettroforesi su gel di Agarosio al 2% colorato con Bromuro di Etidio. Corsia 1, cDNA per HaL1L, amplificato nella reazione di 5’ RACE. Corsia M, “Marker” di pesi molecolari.
Il sequenziamento, che è seguito al clonaggio, ha reso possibile la ricostruzione di tutta la sequenza di cDNA del gene L1L nella specie Helianthus annus (HaL1L) che copre tutta la parte codificante del messaggero, estendendosi da una zona a monte del codone di inizio trascrizione (ATG) alla coda di poli-A. I due amplificati ottenuti dalla 5’ RACE e dalla 3’ RACE presentano una sovrapposizione tra loro di circa 72 bp. Quindi tutto il cDNA è costituito da 820 bp e la porzione codificante contiene 645 bp. Infine, sulla sequenza nucleotidica del cDNA sono stati individuati due oligonucleotidi: CHI-TcDNA (situato al 5’, precisamente 21 nucleotidi a monte del codone di inizio traduzione) e CHI-SR (situato al 3’, esattamente 118 nucleotidi a valle del codone di stop). Con questi oligonucleotidi è stato possibile ottenere un amplificato che, clonato, ha confermato la sequenza ricostruita.
RACE 5’
M 1
selezionato e la sequenza contenuta, lunga 859 bp, è stata depositata in banca-dati. (Numero di accesso: AJ863116)
Sequenza cDNA
CTAGAGAGAGACAATTCCCTCCCTTAGAGAGAGAAACATATGGAACGTGGAGGAGGTTTC CATGGCTACCACAGGCTCCCCATCCACCCTACATCTGGGATGCAACAGCCGGATATGAAG CAGAAGCAACCAGACACAACCAACACCACATTTACAGAAGACAATGAGTGCATTGTTAGA GAGCAAGACCGCTTTATGCCGATAGCAAACGTGATCAGGATCATGCGAAAGATCTTGCCA CCTCACGCGAAGATCTCTGACGACGCAAAAGAAACGATCCAAGAATGTGTTTCCGAGTAC ATTAGTTTTGTGACAGGTGAAGCGAATGACCGTTGCCAACGCGAGCAGAGAAAGACTATC ACAGCTGAAGATGTGTTGTGGGCTATGAGCAAACTCGGGTTTGATGACTATATCGAACCG TTGACTGTGTATCTTCATCGCTACAGGGAGTTTGATGGGGGTGAGCGTGGGTCGATAAGG GGTGAGCCGCTTGTCAAGAGGGCTGCGGCTACTGCTGACCCTGGTCCGTTTGGTATGGGT CCGTTTGTGCCTGGTTTTCACATGGGTCATCATAATGGGTTCTTTGGTCCTGCTAGCATT GCTGGTTTCTTTAAGGACCCGTCGAGTGCGGCTGGCCAGTCTGGGCCTGCTGGTTTCGAG CCGTATGCTCAGTGTAAAGACTAACTGGTTTCGATTTTCGAGCCGTGTGTTGGTTGGTTT GTTTTTTTTTCTTGTTTGTTTGTCTTCTTGGTCTTGGAGCAACAAATTACATTGGGTCAA ACTTTGGCAAGGCATGTAAAAACTGACACTCTCCATCTACTGGTAGTAATCTGCTTTTCT TCCAAAAAAAAAAAAAAAA
Figura 9: Sequenza del cDNA del gene HaL1L di H. annuus. In arancio sono evidenziate le regioni 5’ e 3’ UTR mentre i codoni di inizio e di stop sono identificati dal colore rosso.
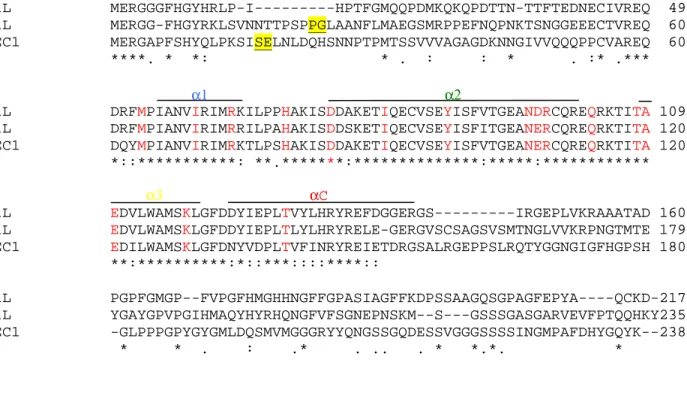
La figura 9 riporta la sequenza dell’intero cDNA ottenuto. Esso è composto di 645 nucleotidi che rappresentano il segmento codificante un peptide, mentre 39 nucleoditi costituiscono la regione 5’ non tradotta (UTR), e 159 la regione 3’ UTR, alla quale fa seguito una coda di 16 Adenine. L’allineamento, ottenuto con il programma ClustalW, della sequenza aminoacidica della proteina dedotta HaL1L con quelle di LEC1 e L1L di Arabidopsis thaliana, ha evidenziato la mancanza di una frammento aminoacidico compreso tra gli aminoacidi 13-25 in L1L e 14-25 in LEC1 di A. thaliana (Figura 10). La regione codificante questa porzione, sul DNA di A. thaliana, è interrotta da introni.
HaL1L MERGGGFHGYHRLP-I---HPTFGMQQPDMKQKQPDTTN-TTFTEDNECIVREQ 49 AtL1L MERGG-FHGYRKLSVNNTTPSPPGLAANFLMAEGSMRPPEFNQPNKTSNGGEEECTVREQ 60 AtLEC1 MERGAPFSHYQLPKSISELNLDQHSNNPTPMTSSVVVAGAGDKNNGIVVQQQPPCVAREQ 60 ****. * *: * . : : * . :* .*** α1 α2
HaL1L DRFMPIANVIRIMRKILPPHAKISDDAKETIQECVSEYISFVTGEANDRCQREQRKTITA 109 AtL1L DRFMPIANVIRIMRRILPAHAKISDDSKETIQECVSEYISFITGEANERCQREQRKTITA 120 AtLEC1 DQYMPIANVIRIMRKTLPSHAKISDDAKETIQECVSEYISFVTGEANERCQREQRKTITA 120 *::***********: **.*******:**************:*****:************
α3 αC
HaL1L EDVLWAMSKLGFDDYIEPLTVYLHRYREFDGGERGS---IRGEPLVKRAAATAD 160 AtL1L EDVLWAMSKLGFDDYIEPLTLYLHRYRELE-GERGVSCSAGSVSMTNGLVVKRPNGTMTE 179 AtLEC1 EDILWAMSKLGFDNYVDPLTVFINRYREIETDRGSALRGEPPSLRQTYGGNGIGFHGPSH 180 **:**********:*::***::::****::
HaL1L PGPFGMGP--FVPGFHMGHHNGFFGPASIAGFFKDPSSAAGQSGPAGFEPYA----QCKD-217 AtL1L YGAYGPVPGIHMAQYHYRHQNGFVFSGNEPNSKM--S---GSSSGASGARVEVFPTQQHKY235 AtLEC1 -GLPPPGPYGYGMLDQSMVMGGGRYYQNGSSGQDESSVGGGSSSSINGMPAFDHYGQYK--238 * * . : .* . .. . * *.*. *
Figura 10. Allineamento della sequenza aminoacidica della proteina LEAFY
COTYLEDON1-LIKE di Helianthus annuus
con le sequenze aminoacidiche delle proteine LEAFY COTYLEDON1 e LEAFY
COTYLEDON1-LIKEdi Arabidopsis thaliana.
In rosso sono evidenziati gli aminoacidi che contraddistinguono, nell’ambito delle proteine HAP3, le proteine di tipo LEC1. In giallo sono evidenziati i punti di inserzione degli introni presenti nelle sequenze AtLEC1 e AtLlL. Le linee individuano gli aminoacidi facenti parte delle α-eliche nel motivo di ripegamento istonico.
Numeri di accesso delle sequenze in banca dati. AtL1L: AY138461, AtLEC1: AF036684; HaL1L AJ863116
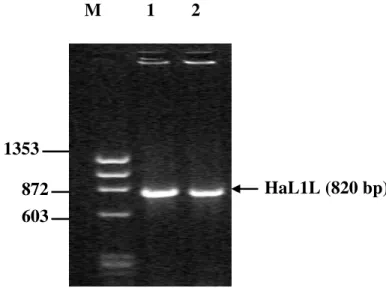
Le informazioni ricavate dal sequenziamento del cDNA sono state utilizzate per la scelta di oligonucleotidi specifici, CHI-TcDNA e CHI-SR, con i quali è stata amplificata la sequenza del DNA genomico corrispondente all’intero cDNA. L’amplificato ottenuto dal DNA genomico è della stessa lunghezza dell’amplificato derivato dal cDNA (Figura 11 ).
Figura 11. Elettroforesi su gel di Agarosio al 2% colorato con Bromuro di Etidio. Amplificazione con la coppia di oligonucleotidi CHI-TcDNA e CHI-SR: corsia 1, amplificato di DNA genomico; corsia 2, amplificato di cDNA per HaL1L; corsia M, “Marker” di pesi molecolari.
L’amplificato da DNA, clonato e sequenziato, mostra inoltre la medesima sequenza del cDNA, e, come evidenziato chiaramente dall’allineamento della figura 12, manca di sequenze introniche.
M 1 2 HaL1L (820 bp) 1353 872 603
DNA CTAGAGAGAGACAATTCCCTCCCTTAGAGAGAGAAACATATGGAACGTGGAGGAGGTTTC 60 cDNA CTAGAGAGAGACAATTCCCTCCCTTAGAGAGAGAAACATATGGAACGTGGAGGAGGTTTC 60 ************************************************************ DNA CATGGCTACCACAGGCTCCCCATCCACCCTACATCTGGGATGCAACAGCCGGATATGAAG 120 cDNA CATGGCTACCACAGGCTCCCCATCCACCCTACATCTGGGATGCAACAGCCGGATATGAAG 120 ************************************************************ DNA CAGAAGCAACCAGACACAACCAACACCACATTTACAGAAGACAATGAGTGCATTGTTAGA 180 cDNA CAGAAGCAACCAGACACAACCAACACCACATTTACAGAAGACAATGAGTGCATTGTTAGA 180 ************************************************************ DNA GAGCAAGACCGCTTTATGCCGATAGCAAACGTGATCAGGATCATGCGAAAGATCTTGCCA 240 cDNA GAGCAAGACCGCTTTATGCCGATAGCAAACGTGATCAGGATCATGCGAAAGATCTTGCCA 240 ************************************************************ DNA CCTCACGCGAAGATCTCTGACGACGCAAAAGAAACGATCCAAGAATGTGTTTCCGAGTAC 300 cDNA CCTCACGCGAAGATCTCTGACGACGCAAAAGAAACGATCCAAGAATGTGTTTCCGAGTAC 300 ************************************************************ DNA ATTAGTTTTGTGACAGGTGAAGCGAATGACCGTTGCCAACGCGAGCAGAGAAAGACTATC 360 cDNA ATTAGTTTTGTGACAGGTGAAGCGAATGACCGTTGCCAACGCGAGCAGAGAAAGACTATC 360 ************************************************************ DNA ACAGCTGAAGATGTGTTGTGGGCTATGAGCAAACTCGGGTTTGATGACTATATCGAACCG 420 cDNA ACAGCTGAAGATGTGTTGTGGGCTATGAGCAAACTCGGGTTTGATGACTATATCGAACCG 420 ************************************************************ DNA TTGACTGTGTATCTTCATCGCTACAGGGAGTTTGATGGGGGTGAGCGTGGGTCGATAAGG 480 cDNA TTGACTGTGTATCTTCATCGCTACAGGGAGTTTGATGGGGGTGAGCGTGGGTCGATAAGG 480 ************************************************************ DNA GGTGAGCCGCTTGTCAAGAGGGCTGCGGCTACTGCTGACCCTGGTCCGTTTGGTATGGGT 540 cDNA GGTGAGCCGCTTGTCAAGAGGGCTGCGGCTACTGCTGACCCTGGTCCGTTTGGTATGGGT 540 ************************************************************ DNA CCGTTTGTGCCTGGTTTTCACATGGGTCATCATAATGGGTTCTTTGGTCCTGCTAGCATT 600 cDNA CCGTTTGTGCCTGGTTTTCACATGGGTCATCATAATGGGTTCTTTGGTCCTGCTAGCATT 600 ************************************************************ DNA GCTGGTTTCTTTAAGGACCCGTCGAGTGCGGCTGGCCAGTCTGGGCCTGCTGGTTTCGAG 660 cDNA GCTGGTTTCTTTAAGGACCCGTCGAGTGCGGCTGGCCAGTCTGGGCCTGCTGGTTTCGAG 660 ************************************************************ DNA CCGTATGCTCAGTGTAAAGACTAACTGGTTTCGATTTTCGAGCCGTGTGTTGGTTGGTTT 720 cDNA CCGTATGCTCAGTGTAAAGACTAACTGGTTTCGATTTTCGAGCCGTGTGTTGGTTGGTTT 720 ************************************************************ DNA GTTTTTTTTTCTTGTTTGTTTGTCTTCTTGGTCTTGGAGCAACAAATTACATTGGGTCAA 780 cDNA GTTTTTTTTTCTTGTTTGTTTGTCTTCTTGGTCTTGGAGCAACAAATTACATTGGGTCAA 780 ************************************************************ DNA ACTTTGGCAAGGCATGTAAAAACTGACACTCTCCATCTAC--- 820 cDNA ACTTTGGCAAGGCATGTAAAAACTGACACTCTCCATCTACTGGTAGTAATCTGCTTTTCT 840 ****************************************
3.2
A
NALISI DELLA SEQUENZA AMINOACIDICAIl peptide dedotto dalla sequenza nucleotidica con il programma Translate (http://swissmodel.expasy.org/) mostra una sequenza di 214 residui aminoacidici (Figura 13). La sequenza aminoacidica è stata allineata, con il programma ClustalW, alle sequenze aminoacidiche delle proteine L1L e LEC1 di Arabidopsis thaliana. Dall’allineamento è emerso che la proteina dedotta, HaL1L, presenta una regione altamente conservata condivisa con entrambe le sequenze di A. thaliana, ma il numero di aminoacidi identici è maggiore rispetto all’L1L (56%) piuttosto che all’LEC1 (45%). La regione dove la conservazione di sequenza è massima corrisponde al dominio B, caratteristico delle proteine HAP3, all’interno del quale si riconosce la sequenza consenso identificativa delle proteine Hap3: C-V-S-E-x-I-S-F- [LIVM]-T-[SG]-E-A-[SC]-[DE]-[KRQ]-C (Figura 10 e 13). Inoltre, nella sequenza HaL1L sono individuabili gli aminoacidi che distinguono le proteine HAP3 di “tipo LEC1” dalle HAP3 di “tipo non-LEC1”. La conservazione aminoacidica identificativa riguarda una metionina in posizione 53, un’isoleucina in posizione 59, l’arginina in posizione 63, l’istidina 69, isoleucina 80, tirosina 87, asparagina 96, arginina 98, glutamina 103, treonina 108, alanina 109, glutamato 110, lisina 118 e treonina 129, non è invece mantenuta la conservazione dell’acido glutammico in posizione 97 delle proteine LEC1 e L1L di A. thaliana che, nell’HaL1L è sostituito da un aspartato.
Sequenza proteica dedotta HaL1L:
MERGGGFHGYHRLPIHPTFGMQQPDMKQKQPDTTNTTFTEDNECIVREQD 50
RFMPIANVIRIMRKILPPHAKISDDAKETIQECVSEYISFVTGEANDRCQ 100
REQRKTITAEDVLWAMSKLGFDDYIEPLTVYLHRYREFDGGERGSIRGEP 150
LVKRAAATADPGPFGMGPFVPGFHMGHHNGFFGPASIAGFFKDPSSAAGQ 200
SGPAGFEPYAQCKD 214
Figura 13. In grigio: dominio B. In blu:”histone fold motif”
Sottolineata: sequenza consenso identificativa delle proteine Hap3: C-V-S-E-x-I-S-F- [LIVM]-T-[SG]-E-A-[SC]-[DE]-[KRQ]-C in rosso: dominio di legame con le TBP (TATA “Binding Protein”)
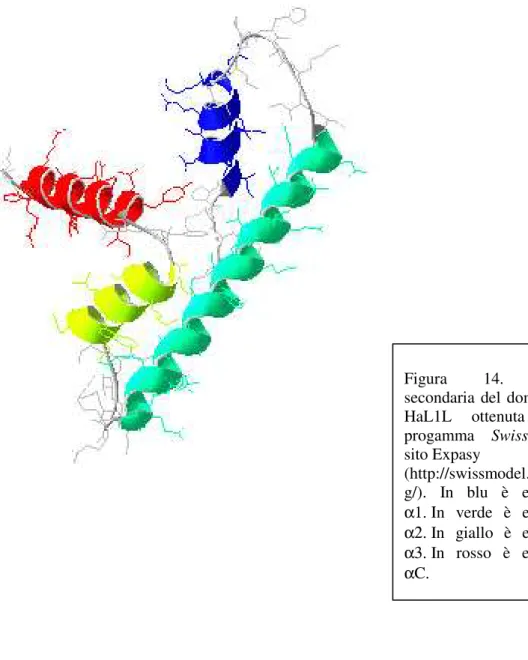
Nella regione conservata è presente anche un motivo strutturale di tipo istonico (“histone fold motif”, HFM). La struttura secondaria della proteina HaL1L, ottenuta con il progamma Swissmodel (http://swissmodel.expasy.org/), ha confermato la presenza in questo motivo strutturale (Figura 14) di quattro eliche: α-eliche, α1, α2 α3 e αC, separate da corte regioni dette anse L1, L2 e L3.
Altre caratteristiche delle proteine che contengono l’HFM sono evidenziabili nell’HaL1L: alanine in posizioni critiche nelle seconde metà delle due eliche, α2 e α3; residui basici in α1; l’alternanza di aminoacidi carichi e idrofobici in α2 e residui basici e una treonina nell’ansa L2 (Figura. 10 ). Infine va osservato che nel dominio HFM si ritrova la sequenza aminoacidica 117 SKLGFDDYIEPL 128, che rappresenta un dominio di legame con TBP (TATA “Binding Protein”) secondo Bellorini (1997).
Figura 14. Struttura secondaria del dominio B di HaL1L ottenuta con il progamma Swissmodel al sito Expasy (http://swissmodel.expasy.or g/). In blu è evidenziata α1. Ιn verde è evidenziata α2. In giallo è evidenziata α3. In rosso è evidenziata αC.
3.4
S
TUDIO DELL’
ESPRESSIONE DEL GENEHaL1L
L’espressione del gene HaL1L è stata valutata con due diverse tecniche: RT-PCR relativa ed ibridazione in situ. La RT-PCR Relativa, con la quale è stato stimato il livello di espressione in diversi tipi di materiale vegetale, consiste nella co-amplificazione, nella stessa reazione di PCR, del cDNA di interesse e di un controllo interno (cDNA di un gene costitutivamente espresso), utilizzando due coppie di oligonucleotidi specifici, con Tm (temperature di appaiamento) analoghe. Questa tecnica è idonea ad evidenziare la presenza di trascritti rari o poco abbondanti come quelli dei fattori di trascrizione. I frammenti da amplificare, del cDNA d’interesse e del controllo interno, devono essere compatibili tra loro, ovvero non devono formarsi prodotti ibridi dovuti al legame tra i due amplificati, e nella co-amplificazione non devono essere prodotte bande aggiuntive rispetto a quelle attese. Affinchè i dati ottenuti dalla RT-PCR relativa siano significativi, la reazione di PCR deve essere terminata quando i prodotti, sia del controllo interno che del cDNA di interesse, risultino evidenziabili e siano ancora all’interno della fase esponenziale. Per i nostri campioni, il numero dei cicli scelto è 26 e in alcuni casi 30. Il gene costitutivo utilizzato è quello per la β−actina, e la coppia di oligonucleotidi ACT3 e ACT5 (Tabella 1) permette l’amplificazione di un frammento di cDNA lungo 243 bp, distinguibile, ma non troppo diverso, dal frammento, di 175 bp, ottenuto con gli oligonucleotidi CHI-REL e CHI-TcDNA, per i cDNA di HaL1L (Figura 6). Le quantità relative per ogni prodotto di PCR sono state quantificate mediante la scansione diretta del gel di Agarosio 2% con il densitometro UVP Image Store 5000 (Ultra Violet Product Ltd, Cambridge, England)
cellulari o tissutali. Il segnale d’espressione, dipende dal legame tra l’RNA messagero del gene d’interesse e la sonda marcata. Quest’ultima è costituita da una specifica sequenza RNA complementare al trascritto da studiare in situ, sonda antisenso, marcata con metodo non radioattivo. L’eventuale presenza di falsi segnali positivi è valutata, in ciascun esperimento di ISH, con l’uso di una sonda molecolare senso caratterizzata dalla sequenza identica al trascritto del gene da studiare.
3.4.1ESPRESSIONE DEL GENE HAL1L IN EMBRIONI ZIGOTICI DI HELIANTHUS ANNUUS
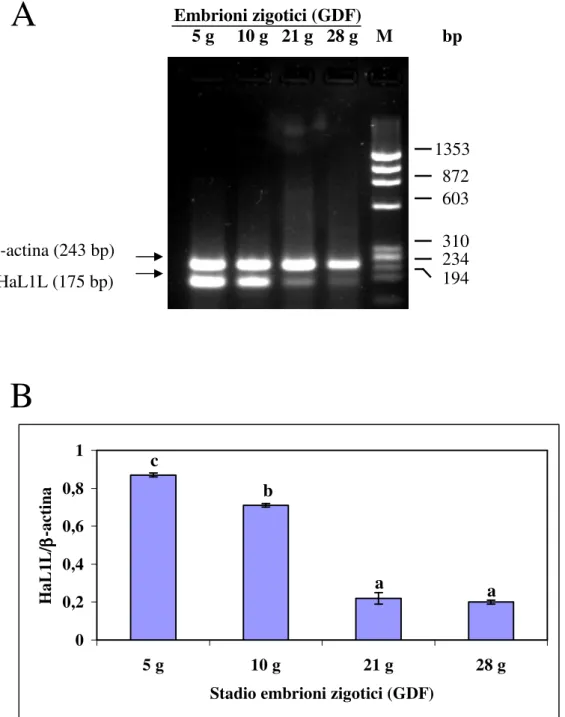
Inizialmente, l’espressione del gene HaL1L è stata valutata, mediante RT-PCR Relativa, in embrioni zigotici poiché, in letteratura, il ruolo del gene L1L, isolato per la prima in Arabidopsis thaliana, è stato associato all’embriogenesi zigotica. L’analisi è stata condotta in embrioni zigotici di Helianthus annuus a quattro diversi stadi di sviluppo: 5, 10, 21 e 28 giorni dalla fecondazione (GDF). I risultati ottenuti, riportati in figura 15, evidenziano che l’espressione del gene è presente, a 26 cicli di PCR, in tutti gli stadi di sviluppo considerati, sebbene a livelli diversi.
Embrioni zigotici (GDF) 5 g 10 g 21 g 28 g M bp β-actina (243 bp) HaL1L (175 bp) 0 0,2 0,4 0,6 0,8 1 5 g 10 g 21 g 28 g Stadio embrioni zigotici (GDF)
H aL 1L /ββββ -ac ti n a 1353 872 603 310 234 194 a a b c
A
B
Figura 15. Livello di espressione relativa di HaL1L stimato mediante RT-PCR. L’amplificazione dei trascritti per HaL1L e β-actina è condotta usando 5 µg di RNA totale estratto da embrioni zigotici a stadi diversi di sviluppo (giorni dalla fecondazione: GDF). Il livello di espressione del trascritto per
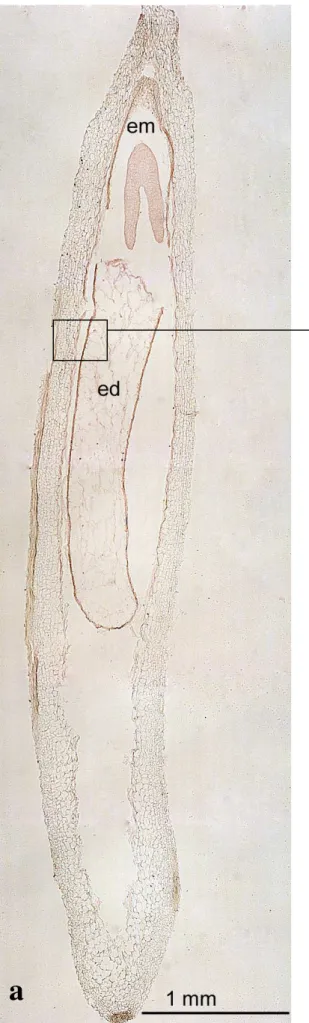
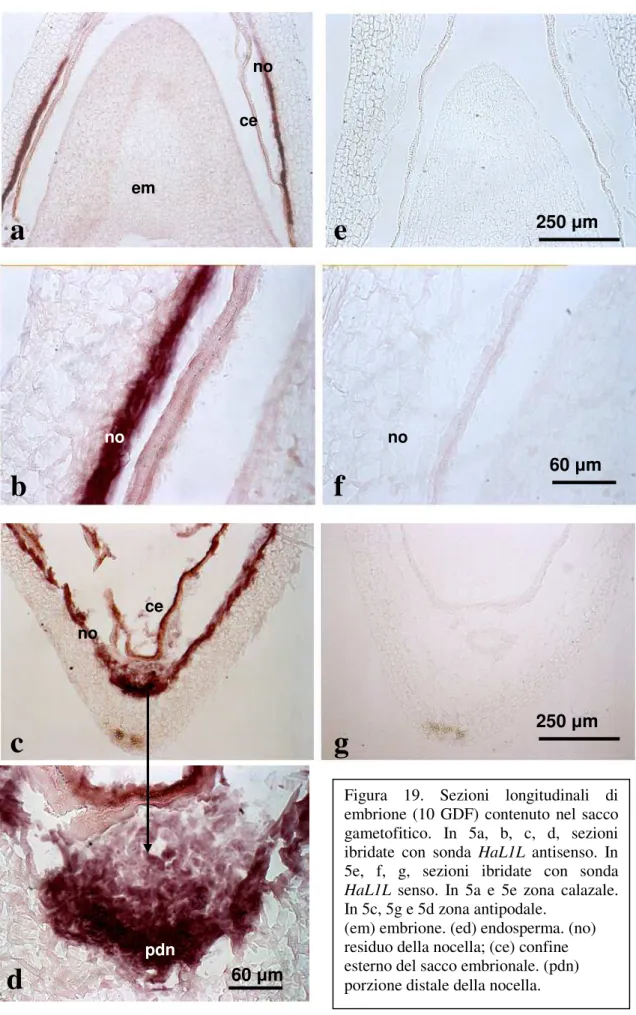
L’analisi statistica ha evidenziato che il livello di espressione, molto elevato negli embrioni di 5 e 10 giorni, decresce in maniera consistente negli stadi di sviluppo embrionale più avanzati presi in considerazione (Figura 15 B). Il livello significativamente più elevato di espressione del gene HaL1L è stato rilevato negli embrioni zigoti allo stadio di sviluppo di 5 giorni dalla fecondazione, mentre non si riscontra una differenza significativa tra gli embrioni di 21 e 28 giorni. Mediante esperimenti d’ibridazione in situ, su sezioni longitudinali di semi di Helianthus annus di 5 giorni (Figura 16 e 17) e 10 giorni (Figura 18 e 19) dalla fecondazione inclusi in paraffina, l’espressione del gene HaL1L è visualizzata sostanzialmente a livello dell’embrione, dell’endosperma e dei tessuti materni. L’esame a maggiore ingrandimento della sezione longitudinale del seme di circa 5 giorni, dalla data di fecondazione del fiore del disco, evidenzia il segnale di ibridazione a livello di specifici domini cellulari come il residuo della nocella, il confine esterno del sacco embrionale e le cellule dell’endosperma (Figura 16 e 17). Un’analoga localizzazione cellulare del segnale, indicativo dell’espressione di HaL1L, è stata individuata anche nelle sezioni longitudinali del seme dopo circa 10 giorni dalla data di fecondazione (Figura 18). Tuttavia, in quest’ultimo campione, è rilevabile un marcato segnale d’espressione nella zona della finestra, parte calazale della nocella, che rimane attiva con la funzione di veicolare nutrimenti dallo sporofito al gametofito (Figura 19 c, d) (Forino et al., 2000).
b
a
c
d
500µm em em no no ce ed ed ce ce no em ed60µm
ce
ed
b
Figura 17. a: Sezione longitudinale di un seme con embrione di circa 5 giorni dalla data di fecondazione del fiore del disco (5 GDF) ibridata con sonda HaL1L antisenso. (em) embrione zigotico allo stadio di torpedine. (ed) endosperma.
Nel riquadro laterale b, particolare a maggiore ingrandimento per valutare il segnale di ibridazione a livello dei vari domini cellulari: (no) residuo della nocella; (ce) confine esterno del sacco embrionale; (ed) endosperma.
no
a
b
ts
ce
ed
em
a
b
c
g
f
e
pdn no no 250 µm 60 µm 250 µm em ce no ce noFigura 19. Sezioni longitudinali di embrione (10 GDF) contenuto nel sacco gametofitico. In 5a, b, c, d, sezioni ibridate con sonda HaL1L antisenso. In 5e, f, g, sezioni ibridate con sonda
HaL1L senso. In 5a e 5e zona calazale. In 5c, 5g e 5d zona antipodale.
(em) embrione. (ed) endosperma. (no) residuo della nocella; (ce) confine esterno del sacco embrionale. (pdn) porzione distale della nocella.
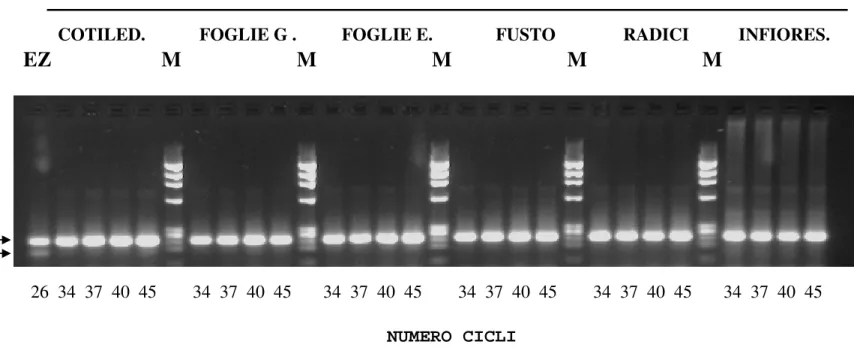
Dopo aver accertato in che modo il gene si esprime a livello dell’embrione zigotico e del seme, era necessario esaminare la sua espressione negli stadi post-embrionali di sviluppo. In particolare, l’RNA, utilizzato per condurre l’RT-PCR, è stato estratto dai seguenti organi di H. annuus: foglie cotiledonari di 10 giorni, foglie giovani (2 cm di lunghezza), foglie espanse (15 cm di lunghezza), fusto di piante allo stadio di bottone fiorale, radici di piante allo stadio di bottone fiorale e infiorescenze immature (capolini a 15 giorni dall’antesi). Sulla base di quanto accertato negli embrioni zigotici è stato deciso di condurre una prima serie di RT-PCR relativa utilizzando 26 cicli di amplificazione, ma con tali condizioni non si è evidenziata l’espressione del gene nei vari tipi di organi analizzati (dato non mostrato). Aumentando a 45 il numero dei cicli (Figura 20), è stata accertata la trascrizione del gene HaL1L nei cotiledoni, nelle foglie espanse e nelle infiorescenze immature. Tuttavia, come appare evidente sul gel di Agarosio colorato con bromuro di etidio (Figura 20), la banda relativa al trascritto per HaL1L è risultata sempre molto flebile rispetto a quella per la β-actina. Per questi campioni non è stato possibile misurare il livello d’espressione, e quindi effettuare l’analisi statistica, anche perché la banda pertinente alla β-actina non è più all’interno della fase esponenziale. Visto il basso livello di espressione accertato con l’RT-PCR, si è ritenuto poco opportuno effettuare, sui medesimi organi vegetali, gli esperimenti di ibridazione in situ. I risultati ottenuti lasciano, infatti, ipotizzare che i trascritti siano presenti in questi organi in quantità inferiori ai limiti di risoluzione permessi dalla tecnica ISH.
ORGANI
EZ M M M M M
COTILED. FOGLIE G . FOGLIE E. FUSTO RADICI INFIORES.
NUMERO CICLI
26 34 37 40 45 34 37 40 45 34 37 40 45 34 37 40 45 34 37 40 45 34 37 40 45
β-actina HaL1L
Figura 20. Amplificazione dei trascritti per HaL1L e β-actina, mediante RT-PCR, condotta usando 5 µg di RNA totale estratto da embrioni zigotici al decimo giorno dalla fecondazione (giorni dalla fecondazione: GDF) e dai seguenti tipi di organi: cotiledoni, foglie giovani, foglie espanse, fusto, radici ed infiorescenze immature. I trascritti per HaL1L e β-actina sono separati in gel di agarosio (2%) colorato con etidio bromuro. Le amplificazioni sono effettuate con diversi numeri di cicli in un intervallo
Il coinvolgimento dell’espressione del gene HaL1L nei fenomeni di morfogenesi avventizia è stato inizialmente valutato con la tecnica della RT-PCR in embrioni somatici prelevati dal clone EMB-2 dell’ibrido interspecifico H. annuus x H. tuberosus cresciuto in vitro. Come controllo è stato utilizzato l’RNA estratto da embrioni zigotici di H. annuus al decimo giorno dalla fecondazione (EZ). La separazione dei prodotti di amplificazione in gel di Agarosio (Figura 21 A) ha evidenziato una banda elettroforetica più intensa nel campione EZ. In particolare, l’analisi statistica dimostra che negli embrioni zigotici il livello di espressione di HaL1L è significativamente più alto (Figura 21 B).
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 EZ (10 g) ES H aL 1L /ββββ -ac ti n a
M EZ ES
(10 g)
ββββ-actina (243 bp) HaL1L (175 bp)bp
1018 517/506 220/201 298b
a
A
B
Embrioni SomaticiFigura 21. Livello di espressione relativa di HaL1L stimato mediante RT-PCR. L’amplificazione dei trascritti per HaL1L e β-actina è condotta usando 5 µg di RNA totale estratto da embrioni zigotici (ES) al decimo giorno dalla fecondazione ed da embrioni somatici (ES). Il livello di espressione del trascritto per HaL1L , normalizzato rispetto al trascritto per la β-actina (B) è stimato misurando la fluorescenza dei prodotti di PCR separati in gel di agarosio (2%) colorato con etidio bromuro (A). I valori in B seguiti dalla stessa lettera non sono significativamente differenti (P= 0,05) in accordo al test di Tukey.
Mediante RT-PCR relativa è stato valutato, se ed in quale misura, il trascritto del gene HaL1L si accumula nei tessuti fogliari con fenomeni di epifillia che caratterizzano il clone morfogenetico EMB-2 dell’ibrido interspecifico H. annuus x H. tuberosus. A tale scopo è stato estratto l’RNA totale dal seguente materiale: embrioni zigotici al decimo giorno dalla fecondazione di H. annuus (EZ), foglie dell’ibrido interspecifico H. annuus x H. tuberosus (clone A-2) non epifillico cresciuto in vitro, foglie dell’ibrido interspecifico H. annuus x H. tuberosus (clone EMB-2, cresciuto in vitro) che non manifestano segni evidenti di epifillia (EMB-2 NE), foglie dell’ibrido interspecifico H. annuus x H. tuberosus (clone EMB-2, cresciuto in vitro) che manifestano segni evidenti di epifillia (EMB-2 EP) (Figura 22 EMB-2 EP), embrioni somatici prelevati dal clone epifillico EMB-2 cresciuto in vitro (ES) (Figura 22 ES) e da germogli avventizi prelevati dal clone epifillico EMB-2 (GA) (Figura 22 GA). La reazione d’amplificazione, condotta per 30 cicli, ha prodotto, per tutti i campioni analizzati, amplificati che sono stati visualizzati su gel di Agarosio al 2% colorato con bromuro di etidio (Figura 22 A). L’espressione più elevata del gene HaL1L è manifestata dagli embrioni zigotici al decimo giorno dalla fecondazione. L’analisi statistica (Figura 22 B) ha confermato che esiste una significativa differenza tra il livello d’espressione degli embrioni zigotici (EZ) e degli embrioni somatici (ES). Nell’ambito delle foglie del clone epifillico EMB-2, la quantità di trascritto significativamente maggiore è stata accertata nelle foglie epifilliche (EMB-2 EP), mentre non sono emerse differenze tra EMB-2 NE e i germogli avventizi (GA). Infine, il livello di espressione nei due ultimi campioni non è risultato significativamente diverso da quello evidenziato nelle
M EZ A-2 EMB-2 EMB-2 ES GA (10 g) NE EP HaL1L (175) bp) ββββ-actina (243 bp) bp 603 310 234 194 118 GA ES EMB-2 EP 0 0,2 0,4 0,6 0,8 1
EZ (10 g) A-2 EMB-2 NE EMB-2 EP ES GA
H a L 1 L /ββββ -a ct in a
B
a a a b c dA
All’analisi con RT-PCR relativa sono seguiti esperimenti di ibridazione in situ. Sezioni trasversali di foglie di tipo EP cresciute in vivo del clone epifillico EMB-2 e del controllo A-2, sono state ibridate con la sonda antisenso (Figura 24; nella Figura 23 le stesse sezione sono colorate con ematossilina). Le strutture ectopiche, presenti sulla pagina adassiale delle foglie EP, sono di tipo organogenetico poichè costituite da germogli avventizi. A livello dell’intera foglia, e nei particolari ingranditi delle proliferazioni organogenetiche ectopiche (indicate dalle frecce), non si evidenzia la presenza di un segnale positivo. Nelle figure 24 c e 24 e, a livello dello strato epidermico delle strutture ectopiche, si nota una colorazione più intensa non imputabile tuttavia a ibridazione specifica, poiché riscontrabile anche nell’analogo materiale ibridato con la sonda senso (Figure 25). Un’ulteriore conferma dell’interpretazione appena formulata è fornita dall’esame dei risultati ottenuti con il materiale di controllo A-2. Nelle sezioni trasversali di foglie cresciute in vivo del clone non morfogenetico A-2 (Figura 26), l’ibridazione in situ con la sonda antisenso ha generato, infatti, lo stesso tipo di colorazione ottenuto con la sonda senso. In particolare, a livello del parenchima clorofilliano a palizzata il segnale ottenuto mostra una sostanziale
Figura 22. Livello di espressione relativa di HaL1L stimato mediante RT-PCR. L’amplificazione dei trascritti per HaL1L e β-actina è condotta usando 5 µg di RNA totale estratto dal seguente materiale: embrioni zigotici a 10 giorni dalla fecondazione (EZ) foglie dell’ibrido interspecifico A-2, foglie del clone EMB-2 che non manifestano epifillia (EMB-2 NE), foglie del clone EMB-2 che manifestano epifillia (EMB-2 EP), embrioni somatici isolati (ES), germogli avventizi (GA). Il livello di espressione del trascritto per HaL1L , normalizzato rispetto al trascritto per la β-actina (B) è stimato misurando la fluorescenza dei prodotti di PCR separati in gel di agarosio (2%) colorato con etidio bromuro (A). I valori in B seguiti dalla stessa lettera non sono significativamente differenti (P= 0,05) in accordo al test di Tukey.
Figura 23. Sezioni trasversali di foglie colorate con ematossilina cresciute in vivo dell’ibrido interspecifico H. annuus x H. tuberosus A2 (Fig.7a e 7b) e del clone variante con epifillia EMB-2 (Fig. 7c e 7d). In Fig.7e, 7f, 7g, 7h particolari ingranditi di foglie EMB-2 con proliferazioni ectopiche (indicate dalle frecce) sulla pagina adassiale.
a
b
c
d
e
f
g
h
60 µµµµm 500 µµµµmsonda HaL1L antisenso. In Fig.8c, 8d, 8e, 8f particolari ingranditi di proliferazioni ectopiche (indicate dalle frecce) sulla pagina adassiale da individui diversi.
500 µm
a
b
Figura 25. Sezioni trasversali di foglie cresciute in vivo del clone epifillico EMB-2 (Fig. 25c e 25d) ibridate con la sonda HaL1L senso. In Fig.9c, 9d, 9e, 9f particolari ingranditi di proliferazioni ectopiche sulla pagina adassiale da individui diversi.
500 µm 60 µm
a
c
b
f
e
d
Figura 26. Sezioni trasversali di foglie cresciute in vivo dell’ibrido interspecifico H. annuus x H.
tuberosus A2. In 26a e 26b sezioni ibridate con la sonda HaL1L antisenso In 26c e 26d sezioni ibridate con la sonda HaL1L senso. (la) lamina fogliare. (ne) nervatura.