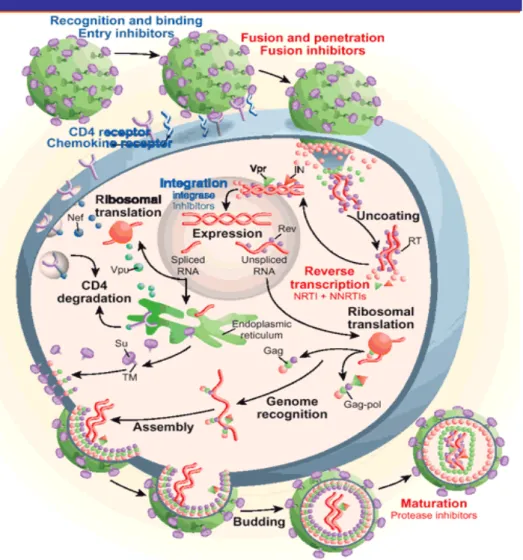
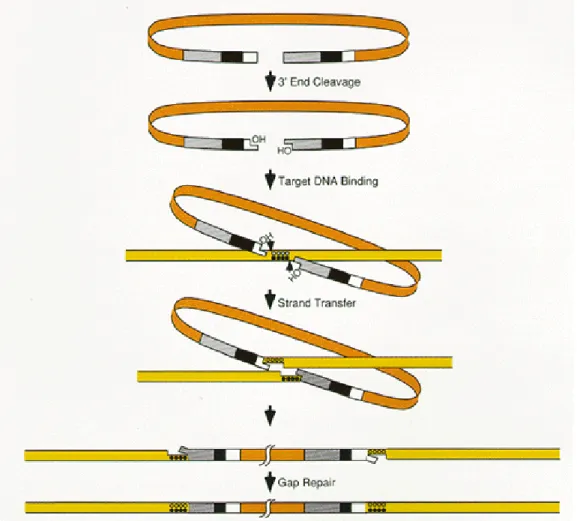
Figura 1.3 Meccanismo di integrazione: l’integrasi catalizza una reazione endonucleasica a carico del dinucleotide GT posto alle estremità 3’ del DNA virale. I gruppi –OH rimasti liberi servono da sito di attacco del DNA virale in quello cellulare. Una volta completata l’integrazione, la sequenza di DNA fiancheggiante il provirus rimasta a singolo filamento viene riparata grazie all’azione di enzimi cellulari.
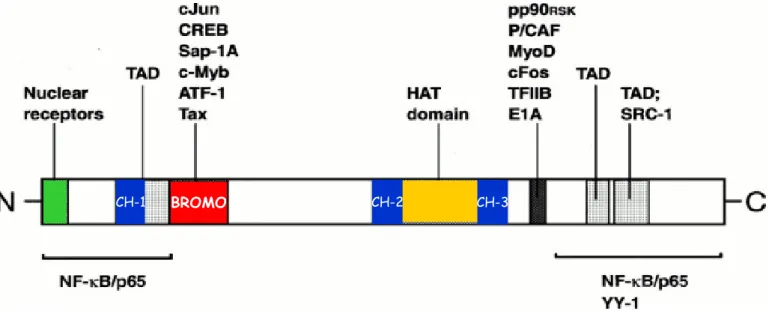
Figura 1.4 Struttura di p300/CBP
BROMO
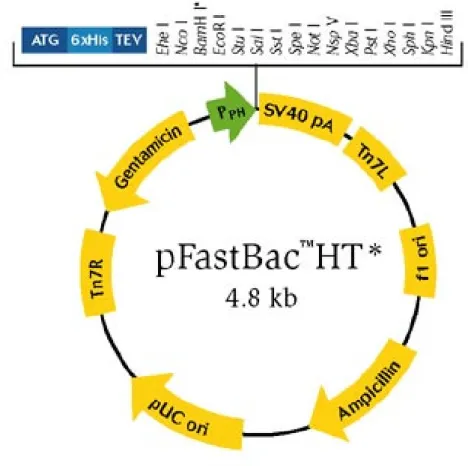
Figura 3.1 Struttura del plasmide pFastBac: la regione compresa tra i siti Tn7L e Tn7R è quella in grado di trasporre. All’interno della regione sono indicati i siti corrispondenti alla sequenza genica che conferisce resistenza alla gentamicina, il promotore della poliedrina (PPH) e il sito di clonaggio.
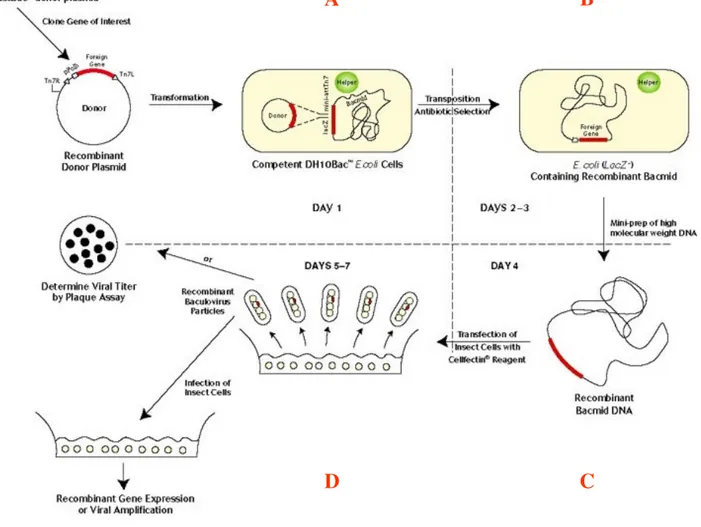
Figura 3.2 Schema rappresentativo dei principali passaggi sperimentali seguiti per la produzione di proteine ricombinanti con il Baculovirus Expression Vector System
A B
C D
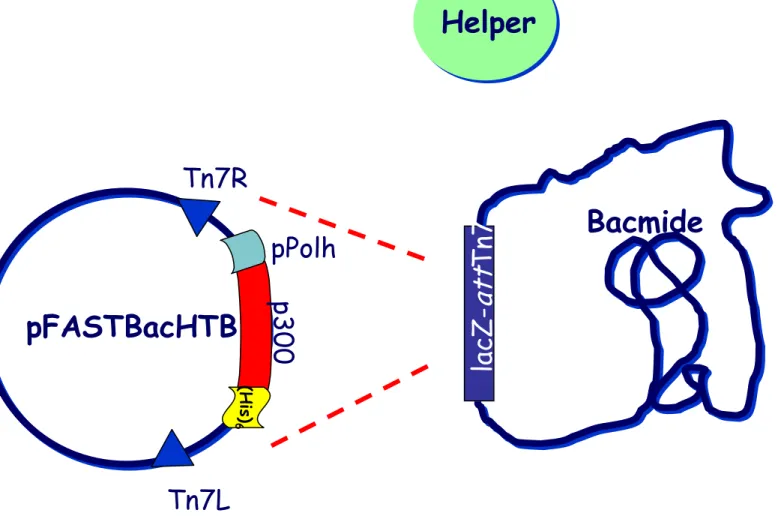
Figura 3.3 Reazione di trasposizione: La regione del plasmide pFASTBac compresa tra i siti Tn7L e Tn7R traspone nel sito mini-attTn7 del bacmide distruggendo così il gene LacZ. La trasposizione, che viene mediata da una trasposasi prodotta dal plasmide T helper genera un bacmide ricombinante contenente la sequenza di p300 al posto di quella della poliedrina.
Helper
p3
0
0
Tn7R
Tn7L
pPolh
pFASTBacHTB
Bacmide
la
cZ
-at
t
T
n7
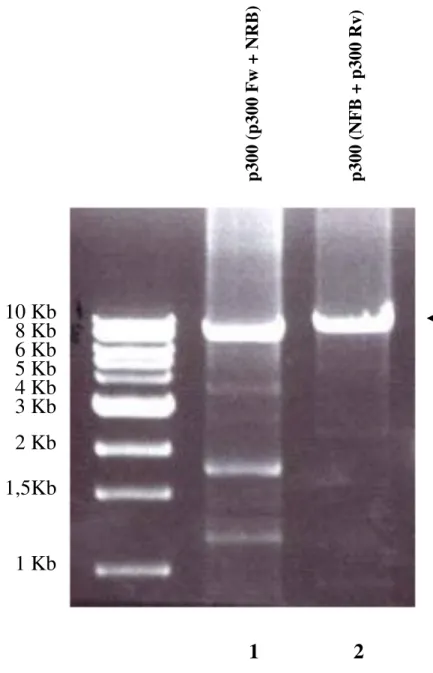
(H is ) 6Figura 3.4 Analisi tramite PCR del bacmide ricombinante: In corsia 1 è presente il prodotto della reazione di PCR eseguita utilizzando il primer NFB e il reverse
complementare a p300; nella corsia 2 è presente il prodotto ottenuto da una reazione di PCR eseguita utilizzando il primer forward complementare a p300 e NRB.
p 3 0 0 ( p 3 0 0 F w + N R B ) p 3 0 0 ( N F B + p 3 0 0 R v ) 1 2 p300 10 Kb 8 Kb 6 Kb 5 Kb 4 Kb 3 Kb 2 Kb 1,5Kb 1 Kb
Figura 3.5 Espressione di p300 ricombinante in cellule Sf9 infettate con diverse concentrazioni di preparato virale: le cellule infettate con diluizioni virali diverse sono state analizzate mediante Western Blotting utilizzando un anticorpo che riconosce p300. In corsia 1 è presente il controllo costituito da cellule non infettate, nelle corsie 2, 3, 4, 5 e 6 cellule infettate con diluizioni 1:10, 1:5, 1:3, 1:2 e 1:1.
N O N IN F E T T A T E D IL U IZ IO N E 1 :1 0 D IL U IZ IO N E 1 :5 D IL U IZ IO N E 1 :3 D IL U IZ IO N E 1 :2 D IL U IZ IO N E 1 :1 p300 1 2 3 4 5 6 200 kDa 116 kDa 97 kDa 45 kDa 66 kDa
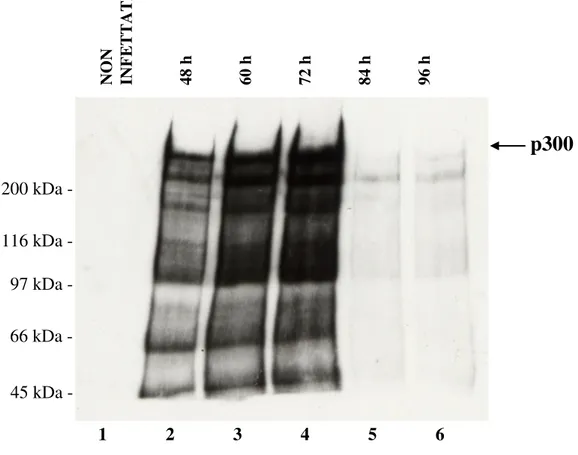
-Figura 3.6 Espressione di p300 ricombinante in cellule Sf9 raccolte a tempi diversi dall’infezione: le cellule di insetto sono state infettate con diluizione virale 1:1, raccolte 48 h (corsia 2), 60 h (corsia 3), 72 h (corsia 4), 84 h (corsia 5) e 96 h (corsia 6) dopo l’infezione e analizzate mediante Western Blotting utilizzando un anticorpo che riconosce p300.
Corsia 1: Western Blotting su cellule non infettate.
N O N IN F E T T A T E 4 8 h 6 0 h 7 2 h 8 4 h 9 6 h p300 1 2 3 4 5 6 200 kDa 116 kDa 97 kDa 45 kDa 66 kDa
-Figura 3.7 Cromatogramma di eluizione: la retta di colore verde indica il passaggio della soluzione di eluizione, la curva in rosso indica il picco di assorbimento a 280 nm.
2 3 4 5 6 7 8 9 10 11 12 Waste
Figura 3.8 Analisi dei prodotti di purificazione di p300 tramite FPLC su gel di poliacrilamide: il lisato (corsia 1 e 10), il lisato privo dei componenti
insolubili cellulari (“sopranatante”, corsia 2), il “flow through” (corsia 3) e i prodotti dell’eluizione di p300 (corsie 4-15) vengono analizzati tramite SDS-PAGE e colorati con blu di coomassie. L IS A T O S O P R A N A T A N T E F L O W T H R O U G H T E L U IZ IO N E 2 E L U IZ IO N E 3 E L U IZ IO N E 4 E L U IZ IO N E 5 E L U IZ IO N E 6 E L U IZ IO N E 7 200 116 97 kDa-p300 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 L IS A T O E L U IZ IO N E 8 E L U IZ IO N E 9 E L U IZ IO N E 1 0 E L U IZ IO N E 1 1 E L U IZ IO N E 1 2 200 116 97 kDa-p300
Figura 3.9 Analisi tramite Western Blotting dei prodotti di purificazione: il lisato
(corsia 1), il sopranatante (corsia 2), il pellet (corsia 3) il “flow through” (corsia 4), i lavaggi (corsie 5-8), le eluizioni (corsie 9 e 10), la resina lavata con EDTA (corsia 11) e la frazione proteica rimasta legata alla resina (corsia 12) vengono analizzati con un anticorpo che riconosce p300. L IS A T O S O P R A N A T A N T E P E L L E T F L O W T H R O U G H T W A S H 1 W A S H 2 W A S H 3 W A S H 4 1 2 3 4 5 6 7 8 21 45 116 31 66 97 200 kDa-p300 E L U IZ IO N E 1 E L U IZ IO N E 2 E D T A R E S IN A 9 10 11 12 p300 21 45 116 31 66 97 200
kDa-Figura 3.10 Saggio di acetilazione con p300 sugli istoni: gli istoni sono stati incubati con p300 e AcetilCoA14C in assenza (corsia 1) e in presenza (corsia 2) di LysCoA, separati
tramite SDS-PAGE e colorati con blu di coomassie (Figura 3.10a). Il livello di acetilazione è stato rilevato esponendo il gel al Cyclone (Figura 3.10b).
p 3 0 0 + I S T O N I p 3 0 0 + I S T O N I + L y sC o A 45 kDa 6 kDa 21 kDa 31 kDa 66 kDa 97 kDa 116 kDa 200 kDa Istoni 1 2 p 3 0 0 + I S T O N I p 3 0 0 + I S T O N I + L y sC o A 1 2
Figura 3.11 Saggio di acetilazione di p300 sull’integrasi: p300 da sola (corsia 1), con BSA (corsia 2), l’integrasi wild-type (corsia 3) e l’integrasi mutata (corsia 4) sono state incubate con AcetilCoA14C, separate tramite elettroforesi SDS-PAGE e colorate con blu di coomassie
(Figura 3.11a). L’acetilazione viene rilevata esponendo il gel al Cyclone. (Figura 3.11b)
116 kDa p 3 0 0 p 3 0 0 + B S A p 3 0 0 + I N w t p 3 0 0 + I N m u t 31 kDa 45 kDa 66 kDa 97 kDa 200 kDa p300 IN 1 2 3 4 IN p 3 0 0 p 3 0 0 + B S A p 3 0 0 + I N w t p 3 0 0 + I N m u t 1 2 3 4
Figura 3.12 Saggio di “in vitro pulldown” tra 35S integrasi e p300: l’integrasi marcata radioattivamente è stata incubata con la resina (Talon) in presenza (corsia 1) o in assenza (corsia 2) di p300. I campioni sono stati poi separati tramite elettroforesi SDS-PAGE e colorati con blu di coomassie. (Figura 3.12a). Il gel è stato poi esposto al Cyclone per rilevare la radioattività presente. (Figura 3.12b).
IN 1 2 97 21 31 45 66 116 200 kDa-1 2 p 3 0 0 + I N IN p3 0 0 + I N IN
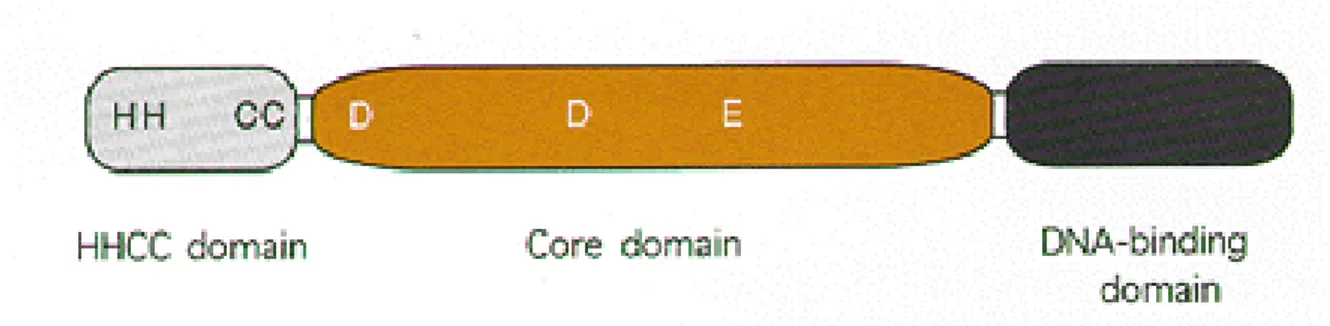
Figura 3.13 Saggio di “in vitro pulldown” tra p300 e i deleti dell’integrasi:
rappresentazione schematica dell’integrasi in cui vengono evideniati i dominii proteici. Nella parte sottostante sono rappresentati i frammenti di integrasi deleti a partire dal C-terminale utilizzati nei saagi di legame in vitro (Figura 3.13a). L’integrasi full-length (corsia 2) e i frammenti di integrasi (corsie 3-5) marcati con 35S sono stati incubati con p300
e successivamnete con la resina Talon in grado di legare le code di istidina di p300. In corsia 1 l’integrasi full-length è stat incubata con la resina in assenza di p300. I prodotti di legame sono poi stati analizzati tramite elettroforesi SDS-PAGE e colorati con coomassie
(Figura 3.13b). Il gel è stato poi esposto al Cyclone. (Figura 3.13c).
75 kDa-IN ( 1 -2 8 8 ) p 3 0 0 + I N ( 1 -2 8 8 ) 20 25 37 50 100 kDa-250 kDa- p300 p 3 0 0 + I N ( 1 -2 7 2 ) p 3 0 0 + I N ( 1 -2 6 3 ) p 3 0 0 + I N ( 1 -2 4 3 ) 1 2 3 4 5 IN ( 1 -2 8 8 ) p 3 0 0 + I N ( 1 -2 8 8 ) IN p 3 0 0 + I N ( 1 -2 7 2 ) p 3 0 0 + I N ( 1 -2 6 3 ) p 3 0 0 + I N ( 1 -2 4 3 ) 1 2 3 4 5 (1-263) (1-243) C-term Core N-term IN (1-288) (1-272) 1 51 212 288 Figura 3.13a Figura 3.13c Figura 3.13b
Figura 3.14 Saggio di attività enzimatica (strand transfer) dell’integrasi in seguito ad acetilazione con p300: l’attività di strand transfer dell’integrasi viene misurata dopo incubazione con p300 in assenza (corsie 2, 4 e 6) e in presenza (corsie 3, 5 e 7 di
AcetilCoA. Corsia 1: substrato senza integrasi e AcetilCoA. Corsia 8: substrato incubato solo con l’integrasi.
+ + + IN AcCoA p300 + IN 1 2 3 4 5 6 7 8 S P
Figura 3.15 Saggio di attività enzimatica (strand transfer) dell’integrasi w.t. e mutata dopo incubazione con p300: l’attività di strand transfer dell’integrasi wild-type (corsie 1 e 2) e dell’integrasi mutata nelle lisine 264, 266 e 273 (corsie 3 e 4) è stata misurata dopo incubazione con p300 in assenza (corsie 1 e 4) e in presenza (corsie 2 e 3) di AcetilCoA. Corsia 5: substrato (S) incubato senza integrasi, p300 e AcetilCoA.
+ + p300 1 2 3 4 5 AcCoA S IN wt IN mut P