65
4. RISULTATI
Caratteristiche epidemiologiche della casistica
L’analisi dei dati riguardanti la casistica, oggetto di studio, è stata eseguita mediante l’uso di un foglio di lavoro fornito dal software statistico StatSoft
STATISTICA 8.0, che è stato arricchito da variabili riguardanti lo stato mutazionale dei geni in esame.
La casistica è costituita da 60 pazienti, 35 uomini e 25 donne, con un’età media al momento del primo intervento chirurgico di 62 anni [età minore 31, età maggiore 84]. Nel 70% dei casi la sede del tumore primitivo è a livello del colon, seguita dal 21,7% dal sigma-retto, 3,3% dal tenue; in due casi nella diagnosi non risultava indicata la localizzazione del tumore primitivo. La presenza di metastasi è stata riscontrata nell’84% dei casi a livello epatico, il 10% presentava una metastasi a livello polmonare, il 3% a livello ovarico, mentre un caso presentava metastasi atriale e in un altro era indicata un’infiltrazione vaginale; tale distribuzione conferma i dati epidemiologici presenti in letteratura (81).
Per ciò che riguarda il tipo di materiale di partenza da cui è stato estratto il DNA, si è trattato nel 95% dei casi di vetrini istologici e nel 5% dei casi di biopsie. La percentuale di cellule neoplastiche era compresa tra il 50 ed il 90%, 8 casi presentavano una percentuale sotto il 50% (40% e 30%) e solo in un caso la percentuale era del 5%. Ad ogni modo abbiamo ritenuto opportuno procedere con l’estrazione del DNA anche in quei casi la cui percentuale di cellule neoplastiche si mostrava al di sotto dei valori indicati nei protocolli per la determinazione dello stato mutazionale.
66
Estrazione e quantificazione del DNA
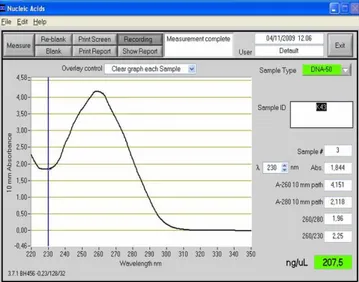
L’analisi spettrofotometrica del DNA estratto dai prelievi istologici di tumore primitivo e metastasi, ha dato risultati compresi tra 3,55 ng/µl e 572 ng/µl. Solo in un caso la quantità di DNA è risultata essere inferiore a 5 ng/µl, quantità minima indicata sul protocollo di amplificazione del DNA; ciò nonostante, abbiamo ritenuto opportuno proseguire nella successiva reazione di amplificazione.
Il 73% dei campioni ha mostrato un rapporto A260/A280 compreso tra 1,8 e 2, tale intervallo è indice di buona qualità del DNA estratto, in termini di purezza. Nel 24% dei casi il rapporto A260/A280 è risultato compreso tra 2,01 e 2,21, mentre il 3%, nello specifico due casi, hanno mostrato un rapporto A260/A280 di 3,16 e 6,17.
Di seguito, viene riportata un’immagine esemplificativa delle analisi spettrofotometriche (Figura 1):
67
Real-Time PCR
La resa della reazione di amplificazione dei codoni 12 e 13 del gene KRas e dell’esone 15 del gene BRAF è stata verificata mediante analisi end-point. In ogni seduta di PCR sono stati utilizzati 2 controlli: un controllo negativo (o bianco della reazione) ed un controllo positivo (DNA sintetico). La reazione di amplificazione di questi controlli è risultata idonea: il controllo negativo non ha mostrato tracce di amplificazione, il controllo positivo si è regolarmente amplificato.
Figura 8. Curve di amplificazione di 24 campioni; la freccia in blu indica il controllo positivo di reazione, la freccia in rosso indica il controllo negativo o bianco della reazione.
Sono stati ammessi all’analisi successiva di pirosequenziamento tutti i campioni che, dopo l’analisi end-point dei risultati, hanno mostrato un segnale di fluorescenza maggiore di 75. Diversi campioni sono stati comunque ammessi al pirosequenziamento nonostante il segnale fosse
68
compreso tra 25 e 75, utilizzando per l’analisi successiva, così come suggerisce il protocollo, una quantità maggiore di PCR. Al contrario, alcuni campioni che mostravano un segnale di fluorescenza inferiore a 25 sono stati giudicati non idonei per il pirosequenziamento.
Figura 9. Analisi end-point della reazione di amplificazione in cui la negative threshold e la positive threshold delineano l’intervallo compreso tra 25 e 75. I 24 campioni amplificati mostrano un segnale di fluorescenza uguale a 100; in posizione 25 si trova il controllo positivo di reazione, in posizione 26 il controllo negativo.
In dettaglio:
a. dalla reazione di amplificazione effettuata sul DNA estratto dai 60 tumori primitivi sono risultati:
69
per i codoni 12 e 13 del gene KRas 59 casi idonei (di cui 11 idonei con un livello di fluorescenza compreso tra 25 e 75), 1 non idoneo.
per il codone 61 del gene KRas 58 casi idonei (di cui 7 idonei con un livello di fluorescenza compreso tra 25 e 75), 2 non idonei.
per l’esone 15 del gene BRAF 59 casi idonei (di cui 5 idonei con un livello di fluorescenza compreso tra 25 e 75), 1 non idoneo.
b. dalla reazione di amplificazione effettuata sul DNA estratto dalle metastasi sono risultati
per i codoni 12 e 13 del gene KRas 16 casi idonei (di cui 2 idonei con un livello di fluorescenza compreso tra 25 e 75), 1 non idoneo.
per il codone 61 del gene KRas 29 casi idonei (di cui 6 idonei con un livello di fluorescenza compreso tra 25 e 75), 3 non idonei.
per l’esone 15 del gene BRAF 22 casi idonei (di cui 8 idonei con un livello di fluorescenza compreso tra 25 e 75), 1 non idoneo.
70
Pirosequenziamento
La determinazione dello stato mutazionale dei codoni 12, 13 e 61 del gene
KRas e dell’esone 15 del gene BRAF è stata eseguita mediante metodica di
pirosequenziamento.
Dalle analisi che ho eseguito sui tumori primitivi è emerso che: su 59 casi analizzati per KRas codone 12 e 13:
27 pazienti risultavano wild-type
32 pazienti presentavano una mutazione nel codone 12 e 13 del gene KRas. La caratterizzazione molecolare delle mutazioni è riportata in Tabella 1; di seguito, la figura 9 si riferisce ai pirogrammi relativi al profilo wild-type e ad alcune delle mutazioni osservate.
Stato mutazionale N° pazienti c.34 G>T (p.G12C) 2 c.34 G>A (p.G12S) 3 c.35 G>A (p.G12D) 10 c.35 G>C (p.G12A) 1 c.35 G>T (p.G12V) 12 c.38 G>A (p.G13D) 4 TOTALE 32
71
Figura 10. Nel riquadro A è riportato il programma di un campione wild-type, il riquadro B mostra il programma di un campione che presenta la mutazione c.34 G>T (p. G12C); nei riquadri C e D sono riportati i programmi di due campioni che mostrano rispettivamente le mutazioni c.34 G>A (p. G12S) e c.35 G>A (p. G12D). Le frecce rosse indicano il segnale relativo alla presenza dell’allele mutato.
su 58 casi analizzati per KRas codone 61: 57 pazienti risultavano wild-type
1 presentava una mutazione nel codone 61 del gene KRas in particolar modo, la mutazione Q61R.
su 59 casi analizzati per BRAF esone 15: 51 pazienti risultavano wild-type
72
Figura 11. In altro è riportato il programma di un campione wild-type, il pirogramma in basso indica la presenza della mutazione c.1799 T>A (p.V600E). La freccia rossa indica il segnale relativo alla presenza dell’allele mutato.
Le analisi di pirosequenziamento effettuate sulle metastasi hanno dato i seguenti risultati:
su 16 pazienti analizzati per il codone 12 e 13 di KRas: 9 pazienti risultavano wild-type
7 presentavano una mutazione nel codone 12 e 13 del gene KRAS . In particolare:
www w
73
I 29 pazienti analizzati per il codone 61 del gene KRAS sono risultati tutti wildtype.
Su 22 pazienti analizzati per l’esone 15 del gene BRAF: 19 pazienti risultavano wild-type
3 presentavano la mutazione V600E nell’esone 15 del gene BRAF. Stato mutazionale N° pazienti
c.34 G>A (p.G12S) 1 c.35 G>T (p.G12V) 2 c.35 G>A (p.G12D) 3 c.38 G>A (p.G13D) 1
74
Valutazione dei risultati
Complessivamente, integrando i risultati ottenuti in questo studio e quelli di cui ho potuto usufruire poiché ottenuti a scopo diagnostico, è stato possibile valutare i codoni 12 e 13 del gene KRas di 57 pazienti metastatici. In due casi non è stato possibile fare alcuna valutazioni poiché il primitivo, in un caso, e la metastasi, nell’altro, sono risultati non idonei alla reazione di amplificazione.
Dalla correlazione dei risultati ottenuti sui prelievi di tessuto tumorale primitivo e metastatico, è emersa una concordanza sullo stato mutazionale dei codoni 12 e 13 del gene KRas del 95%. In 54 casi lo stato mutazionale dei suddetti codoni si è rivelato omogeneo tra il tumore primitivo e la metastasi corrispondente, in 3 casi, invece, è stata riscontrata eterogeneità tra tessuto tumorale primitivo e metastasi.
Grafico 1. Correlazione tra lo stato mutazionale del codone 12 e 13 del gene KRas tra tumore primitivo e metastasi (WT: wild-type; M: mutato.)
75 In particolar modo:
in 26 casi sia il tumore primitivo che quello metastatico sono risultati wild-type;
10 casi hanno mostrato su entrambi i prelievi la mutazione c.35 G>A (p.G12D);
8 casi hanno mostrato su entrambi i prelievi la mutazione c.35 G>T (p.G12V);
4 casi hanno mostrato su entrambi i prelievi la mutazione c.38 G>A (p.G13D);
3 casi hanno mostrato su entrambi i prelievi la mutazione c.34 G>A (p.G12S);
2 casi ha mostrato su entrambi i prelievi la mutazione c.34 G>T (p.G12C). 1 caso ha mostrato su entrambi i prelievi la mutazione c.35 G>C (p.G12A);
In 3 casi è stata riscontrata una discordanza dello stato mutazionale dei codoni esaminati; in dettaglio, sul tumore primitivo è stata ritrovata la mutazione c.35 G>T (p.G12V) che non è stata riscontrata nella metastasi che ha dato esito wild-type.
Degno di nota è il caso di un campione, non inserito nella nostra casistica ai fini della valutazione, che ha mostrato la mutazione c.35 G>T (p.G12V) nel tumore primitivo e un risultato indeterminato sul tumore metastatico.
76
Grafico 2. Caratterizzazione molecolare delle mutazioni del codone 12 e 13 del gene KRAS a confronto tra tumore primitivo e metastasi.
Per il codone 61 del gene KRas ho potuto valutare i dati in 56 casi sui 60 arruolati nello studio. È stata riscontrata una concordanza dello stato mutazionale tra tumore primitivo e tumore metastatico del 98%. L’unico caso a risultare discordante è quello in cui lo stato mutazionale del tumore primitivo mostra una mutazione, precisamente la Q61R, mentre lo stato mutazionale del tumore metastatico è risultato essere wild-type.
77
Grafico 3. Correlazione dello lo stato mutazionale del codone 61 del gene KRAS tra tumore primitivo e metastasi (WT: wild-type; M: mutato.)
L’analisi dei dati sullo stato mutazionale del codone 600 del gene BRAF condotti su prelievi di tessuto tumorale primitivo e sulla metastasi si è resa possibile complessivamente per 57 dei 60 casi arruolati. I risultati hanno messo in evidenza una concordanza del 96%. In particolar modo:
- 49 casi hanno mostrato un profilo wildt-ype sia nel tumore primitivo che sulla metastasi,
- in 6 casi è stata riscontrata la mutazione V600E sul tumore primitivo e sulla metastasi.
- 2 casi, invece, hanno mostrato una discordanza dello stato mutazionale; in particolare, in entrambi i casi il tumore primitivo risultava mutato V600E mentre il tumore metastatico non presentava tale mutazione.
78
Grafico 4. Correlazione dello stato mutazionale dell’esone 15 del gene BRAF tra tumore primitivo e metastasi (WT: wild-type.)
79
5. DISCUSSIONE
Nei paesi occidentali il carcinoma del colon-retto (Colorectal Cancer, CRC) è uno dei tumori più diffusi e rappresenta la terza causa di morte dovuta a neoplasie maligne, dopo il tumore della mammella nella donna e quello del polmone nell'uomo. Al momento della diagnosi si registra, nella maggior parte dei casi, la presenza di disseminazione a distanza della neoplasia, con sede epatica e in minor parte polmonare, ovarica e atriale. Negli ultimi anni, per il trattamento dei CRC in stadio avanzato, sono state messe a punto terapie farmacologiche mirate, che si basano sulla somministrazione di anticorpi monoclonali (mAb) rivolti verso il recettore per il fattore di crescita epiteliale (EGFR), quali il Cetuximab e il Panitumumab (82, 83); tali terapie, come osservato in diversi studi, risultano efficienti soltanto nei pazienti wild-type per il gene KRas. Tuttavia la presenza di mutazioni a carico del gene
KRas spiega solamente il 30-40% circa dei casi di mancata risposta al
trattamento, pertanto si è rivelato prioritario identificare ulteriori determinanti genetici di resistenza alle terapie rivolte contro l’EGFR nei carcinomi del colon-retto. Evidenze scientifiche hanno indicato che anche l’attivazione oncogenica di BRAF influenzi la risposta agli anticorpi monoclonali anti-EGFR. La corretta determinazione dello stato mutazionale dei suddetti geni è, quindi, essenziale nell’impostazione del trattamento
terapeutico appropriato per ciascun paziente. Tuttavia, l’eterogeneità dello stato mutazionale dei geni KRas e BRAF tra il
tumore primitivo e la sua metastasi, potrebbe complicare l’esatta determinazione del profilo genetico dei pazienti.
80
Nello studio da me condotto è stata presa in esame la distribuzione dello stato mutazionale dei suddetti geni tra il CRC primitivo e la relativa metastasi con lo scopo di stabilire se l’analisi mutazionale condotta sul tumore primitivo o sulla metastasi sia sufficiente a definire lo stato mutazionale del paziente. A tale fine, l’analisi mutazionale dei codoni 12, 13 e 61 del gene KRas e del codone 600 del gene BRAF è stata condotta su 60 pazienti affetti da CRC, selezionati da una casistica di 556 pazienti, per i quali era disponibile sia un prelievo di tessuto tumorale primitivo che un prelievo di tessuto metastatico.
L’analisi mutazionale eseguita sui tumori primitivi per i codoni 12 e 13 dell’esone 2 del gene KRas ha mostrato una frequenza di mutazione del 54%, per il codone 61 dell’esone 3 del gene KRas è stata riscontrata una frequenza di mutazione dell’1% e del 13% per il codone 600 del gene BRAF . Tali risultati sono in linea con quelli ottenuti in altri studi (20, 21, 79).
Dalla correlazione dei dati sullo stato mutazionale dei codoni 12 e 13 dell’esone 2 del gene KRas è emersa una concordanza del 95% (54/57 casi) tra il tessuto tumorale primitivo e la relativa metastasi. Tutti i casi che hanno mostrato eterogeneità dello stato mutazionale (5%, 3/57) presentavano la mutazione G12V nel tumore primitivo mentre le metastasi sono risultate wild-type. Il codone 61 del gene KRas ha mostrato una concordanza dello stato mutazionale tra tumore primitivo e metastasi del 98% (55/56 casi), per questo codone l’unico caso risultato discordante ha presentato una mutazione (Q61R) nel tumore primitivo e uno stato wild-type nella metastasi. Per quanto riguarda il codone 600 del gene BRAF è stata osservata una concordanza tra il tumore primitivo e la metastasi nel 96% dei casi (55/57 casi), in entrambi i casi in cui è stata riscontrata eterogeneità il tumore primitivo mostrava la mutazione V600E, al contrario la metastasi risultava wild-type. Questo studio ha confermato che le mutazioni a carico dei due geni
81
presi in esame sono mutualmente esclusive. Tale risultato è stato osservato anche in altri studi (31, 34)., in cui è stato riportato che meno dell’1% dei tumori mutati per BRAF risultano essere mutati anche per KRas.
Complessivamente, dalla valutazione dei risultati è stata riscontrata una concordanza dello stato mutazionale tra tumore primitivo e metastasi dell’89,3%, e tutti i casi discordanti (10,7% dei casi) hanno mostrato una mutazione nel tumore primitivo non rilevata nella metastasi.
Da un punto di vista biologico, la scomparsa della mutazione nella metastasi potrebbe essere spiegata, ad esempio, attraverso l’ipotesi che le cellule tumorali potrebbero aver lasciato la sede primitiva di neoplasia prima dell’acquisizione della mutazione (84); tale ipotesi però non trova accordo con la sequenza di eventi molecolari descritta da Fearon e Vogelstein, secondo cui le mutazioni a carico di KRas avvengono negli stadi precoci. L’assenza della mutazione nella metastasi potrebbe essere spiegata anche attraverso l’ipotesi che differenti cloni cellulari con numerose alterazioni genetiche coesistano nello stesso tumore (85). Oppure, la scomparsa della mutazione potrebbe essere dovuta all’aumento dell’instabilità cromosomica (in termini di perdita e guadagno di parti di cromosoma) che si osserva durante la progressione tumorale dei CRC (86).
Dal punto di vista clinico, l’eterogeneità emersa in questo studio suggerisce che la determinazione dello stato mutazionale dei geni KRas e BRAF eseguita sul tumore primitivo o sulla metastasi potrebbe essere insufficiente a definire il profilo genetico del paziente con carcinoma del colon retto in avanzato stato di malattia. Dalle osservazioni emerse dal nostro studio, infatti, la sola genotipizzazione del tumore primitivo, che in tutti i casi eterogenei risultava mutato, avrebbe determinato l’esclusione del paziente dal trattamento con mAb anti-EGFR, dal quale invece, visto lo stato wild-type della metastasi, avrebbe potuto trarne beneficio.
82
In conclusione, questo studio, confermando l’eterogeneità dello stato mutazionale tra il tumore primitivo e la metastasi corrispondente, suggerisce, dal punto di vista biologico la necessità di approfondire ulteriormente gli aspetti molecolari della progressione tumorale, e dal punto di vista clinico diversi spunti di riflessione e di discussione volti alla coerente impostazione del trattamento terapeutico con mAb anti-EGFR da parte dell’oncologo. Ad oggi, infatti, non esistono delle linee guida che indicano di completare la genotipizzazione della metastasi laddove sia noto lo stato mutazionale del tumore primitivo, i nostri risultati propongono la necessità di determinare anche lo stato mutazionale della metastasi in modo da avere un profilo genetico il più completo ed esaustivo possibile del paziente.