CAPITOLO 1: INTRODUZIONE
1.1 Classificazione
La famiglia Retroviridae viene suddivisa in tre sottofamiglie:
• Gli oncovirus, che comprendono principalmente virus in grado di indurre trasformazione cellulare in vitro e in vivo.
• I lentivirus, che instaurano in genere infezioni croniche con lungo periodo di incubazione.
• Gli spumavirus, che comprendono virus che infettano varie specie animali e non ancora associati a specifiche patologie.
Il virus dell’immunodeficienza felina (FIV) è un retrovirus appartenente alla sottofamiglia dei lentivirus, il cui ospite naturale è il gatto, nel quale induce una sindrome da immunodeficienza simile all’AIDS nell’uomo; questa proprietà ed altre similitudini tra HIV e FIV rendono quest’ultimo un eccellente modello animale per la ricerca di farmaci antivirali e vaccini per l’AIDS (Burkhard and Dean, 2003).
in seguito all’infezione, i lentivirus, come gli altri retrovirus, sono in grado di integrare il loro genoma in quello dell’ospite, ma a differenza di tutti i membri delle altre sottofamiglie hanno la capacità di infettare e integrarsi anche in cellule che non sono in attiva replicazione (Bukrisky et al.,1993). Questa proprietà rende i lentivirus un ottimo mezzo di veicolazione di geni eterologhi anche in cellule in fase quiescente, di gran lunga predominante in un organismo vivente, e per questo motivo sono oggetto di intensa ricerca per la produzione di vettori per scopi vaccinali o terapia genica.
1.2 Morfologia
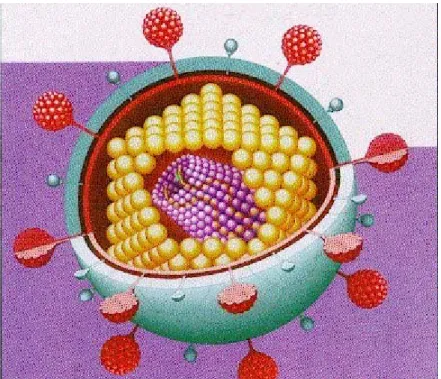
Il virione maturo di FIV (Fig 1.1) è costituito da una particella sferico-elissoidale con diametro di 100-125 nm, in cui si distinguono una regione esterna detta envelope e una zona centrale elettrondensa (core) contentente il genoma.
L’envelope è costituito da un doppio strato fosfolipidico di derivazione cellulare, sul quale si ancorano due proteine virus-specifiche: la glicoproteina di superficie (SU) e la proteina tansmembrana (TM). La SU, che costituisce l’antirecettore virale, è una glicoproteina altamente glicosilata di peso molecolare di 95 KDa. La TM, di 40 KDa, ancora la SU attraverso legami non covalenti ed è coinvolta nella fusione del virus con la cellula bersaglio.
Figura 1.1: Particella virionica matura di FIV (da 1.cba.unige.it/virologia/HIVD1.jpg, modificata)
La proteina di matrice (MA), associata internamente all’envelope, garantisce la corretta incorporazione delle proteine di superficie nel virione maturo.
Il core centrale comprende il capside a simmetria icosaedrica, formato dall’associazione di diverse unità di proteina capsidica (CA).
Quest’ultimo racchiude il genoma virale di RNA a polarità positiva associato alla proteina nucleocapsidica (NC) e diverse proteine enzimatiche quali: trascrittasi inversa (RT), integrasi (IN), proteasi (PR) e dUTPasi (DU).
1.3 Organizzazione genomica
Come tutti i virus appartenenti alla famiglia Retroviridae, il genoma di FIV è costituito da due copie di RNA a singolo filamento con polarità positiva che presentano un cap metilato al 5’ e una coda di poly-A al 3’.
Lungo il genoma si possono riconoscere tre grandi cornici di lettura (ORF) denominate gag, pol ed env, che codificano per le principali proteine strutturali e funzionali del virione, ed altre ORF più piccole che esprimono proteine regolatorie (Talbott et al., 1989; Tomonaga and Mikami, 1996) (Fig 1.2).
Una volta entrato nel citoplasma, l’RNA virale è retrotrascritto a DNA bicatenario e integrato come provirus nel genoma della cellula ospite.
1.3.1 LONG TERMINAL REPEAT
Il genoma provirale è fiancheggiato da due LTR generate dalla duplicazione delle sequenze terminali dell’RNA virale durante la retrotrascrizione. Ciascun LTR è costituita da una regione U3 (unique 3’), R (repeat), U5 (unique 5’). Nella regione U3 sono presenti sequenze cis-agenti necessarie per l’inizio della trascrizione del genoma provirale, come la TATA box e siti di legame per fattori di trascrizione quali AP-1, AP-4, ATE, C/EPB, NF1, e NF-kB. Mutazioni o delezioni di questi siti dimostrano che queste regioni giocano un ruolo fondamentale nell’attività basale di promotore dell’LTR (Thompson et
al., 1994; Bigornia et al., 2001).
ga
g
pol
vif
or
f-A
rev
en
v
RRE
LT RFi
gur
a
1.
2:
Gen
oma
p
rov
ir
al
e di
F
IV
LT Rga
g
pol
vif
or
f-A
rev
en
v
RRE
LT RFi
gur
a
1.
2:
Gen
oma
p
rov
ir
al
e di
F
IV
LT R 4La regione R è essenziale per il processo di retrotrascrizione e replicazione virale.
Nella regione U5 si trovano sequenze che facilitano l’inizio della retrotrascrizione e parte del segnale di incapsidamento necessario al
packaging dell’RNA genomico nel virione maturo.
1.3.2 Geni strutturali
Gag
Il gene gag codifica per una poliproteina di 50 KDa che viene processata nelle tre principali proteine del core. La porzione N-terminale codifica per la matrice (MA p15), quella centrale per la proteina capsidica (CA p25)e quella C-terminale per la nucelocapsidica (NC p8).
La MA è legata alla superficie interna dell’envelope, come una proteina periferica di membrana e conferisce rigidità alla struttura. La parte aminoterminale è soggetta alla miristilazione, una modificazione post-traduzionale che consiste nel legame di molecole di acido miristico alla proteina; questa modificazione garantisce un legame più stretto della MA con la membrana.
La CA è la più conservata tra le proteine di Gag, costituisce l’involucro proteico del core e determina la forma icosaedrica del capside.
La NC è strettamente associata all’RNA virale ed è implicata in diverse fasi del ciclo replicativo virale. Questa proteina, infatti, interviene nel processo di dimerizzazione delle due molecole di RNA virale, facilita l’annealing del tRNA al primer binding site (PBS) al 5’ dell’RNA virale, media il trasferimento del filamento a polarità negativa di cDNA dall’estremità 5’ all’estremità 3’ dell’RNA virale durante la retrotrascrizione, incrementa la processività della RT e gioca un ruolo fondamentale nell’assemblaggio delle particelle virali (Moscardini et al., 2002)
Pol
Il gene pol di FIV è trascritto inizialmente come RNA messaggero gag-pol. Successivamente attraverso uno spostamento della cornice di lettura da parte dei ribosomi, non è riconosciuto il codone di stop presente su gag e la traduzione continua fino alla fine di pol. Il precursore poliproteico è quindi scisso dalla proteasi virale e produce gli enzimi virali PR, RT, DU e IN.
La PR è una piccola endoproteasi di 13KDa, adibita al processamento di Gag e Gag-Pol nei rispettivi prodotti maturi. Il sito attivo è presente al centro della proteina e costituito da due molecole di acido aspartico che catalizzano la scissione dei precursori proteici.
La RT è un eterodimero costituito da due subunità di 51KDa e 66KDa che, nella porzione C-terminale di quest’ultima, presenta un frammento con attività di Rnasi-H. La RT, caratteristica di tutti i retrovirus, è responsabile della retrotrascrizione dell’RNA virale in DNA bicatenario.
La DU promuove l’idrolisi di dUTP in dUMP in modo da prevenire un’errata incorporazione di deossiuridina durante la sintesi di DNA ed è quindi importante per l’infettività virale (Payne et al., 2001).
La IN è un multimero che presenta un motivo Zinc finger nella regione N-terminale implicata nel legame con il DNA e uno nella regione coinvolta nell’integrazione del DNA retrotrascritto nel genoma della cellula ospite.
Env
Il gene env codifica per un unico precursore proteico di 130 KDa che, in seguito al taglio proteolitico, dà origine alle due glicoproteine di superficie SU (gp95) e TM (gp36), coinvolte nelle prime fasi dell’infezione virale.
La SU, infatti, si lega a recettore e corecettore cellulare e rappresenta il principale determinante del tropismo di FIV. La TM promuove la fusione tra
envelope e membrana cellulare. La SU, inoltre, contiene epitopi che
inducono la formazione di anticorpi neutralizzanti e di altri effettori della risposta immune dell’ospite.
Il gene env è quello che presenta il più alto tasso di variabilità. Le zone soggette a mutazione non sono però localizzate casualmente, ma in nove regioni ben definite denominate regioni ipervariabili (V). Due di esse si trovano nella regione all’N-terminale (V1 e V2), quattro nella proteina SU (V3-V6) e tre nel dominio codificante la TM (V7-V9). La presenza di regioni ipervariabili, in particolare a livello della SU, sembra avere un ruolo importante nell’elusione del sistema immunitario, che non è in grado di adattarsi rapidamente alle mutazioni degli epitopi antigenici virali (Bendinelli
et al., 2001).
1.3.3. Geni regolatori
Vif
Il gene vif di FIV è simile per lunghezza e localizzazione a quello dei lentivirus dei primati, come HIV, con i quali però non condivide nessuna omologia genetica.
La regione che codifica Vif ha inizio subito dopo il codone di stop del gene
pol.
Vif è un determinante essenziale per l’infettività del virione sia in vitro che
in vivo (Tomonaga et al., 1992; Lockridge et al., 1999) ed è principalmente
localizzato nel citoplasma di cellule infettate.
Studi effettuati negli ultimi anni, tuttavia, ne hanno rilevato la presenza anche nel nucleo (Charterji et al., 2000). Queste osservazioni indicano che forse Vif svolge il suo ruolo principale proprio a livello del nucleo conferendo, in qualche modo, infettività alla progenie virale.
ORF-A
FIV ORF-A è un piccolo gene che codifica per una proteina di circa 9KDa necessaria per l’efficiente replicazione virale in alcuni istotipi cellulari sia in
vitro che in vivo (Pistello et al., 2002; Waters et al., 1996).
Recenti studi hanno evidenziato che questa proteina è importante nelle fasi finali del ciclo replicativo di FIV e in particolare nella formazione del virione
maturo (Gemeniano et al., 2003). La scoperta della localizzazione nucleare di ORF-A e della capacità di indurre arresto del ciclo cellulare in G2
suggeriscono che la funzione di questa proteina possa essere analoga a quella di Vpr in HIV (Gemeniano et al., 2004).
Rev e Rev responsive element
Il gene rev è presente in tutti i lentivirus e il suo prodotto è un fattore chiave nella replicazione.
Rev è codificata da due esoni: il primo è localizzato all’estremità 5’ dell’ORF di env, mentre il secondo si trova dopo la fine di env e si estende fino all’U3 dell’LTR (Phillips et al., 1992).
Rev agisce a livello post-trascrizionale promuovendo l’export nucleare dell’mRNA unspliced o singly spliced. Questo tipo di messaggeri, infatti, presentano delle sequenze inibitorie cis-agenti (INS), analoghe a quelle trovate in HIV (Graf et al., 2000), che impediscono il passaggio attraverso la membrana nucleare.
Rev svolge la sua attività legandosi, attraverso un dominio basico, ad un Rev responsive element (RRE), localizzato all’estremità 3’ del gene env e presente in tutti gli mRNA unspliced e single spliced. L’RRE esibisce una particolare struttura secondaria che favorisce il legame con Rev (Vaishnav et al., 1991). Quest’ultimo è in grado di svolgere la sua funzione di shuttle grazie alla presenza di due sequenze aminoacidiche specifiche: un nuclear
localisation signal (NLS) e un nuclear export signal (NES) che ne facilitano il
passaggio attraverso la membrana nucleare (Henderson and Percipalle, 1997).
In virtù di tali proprietà, Rev rappresenta una delle proteine dalle quali dipende lo stato di latenza o di infezione produttiva della cellula. Infatti, ad alte concentrazioni di Rev, mRNA unspliced o single spliced, codificante per le proteine strutturali del virione, è trasportato nel citoplasma e tradotto determinando così la produzione di progenie virali. Quando invece la concentrazione di Rev è troppo bassa questo non avviene (Felber et al.,
1989). Rev rappresenta quindi l’interruttore che innesca l’espressione genica nel ciclo replicativo dei lentivirus.
1.4 Ciclo Replicativo
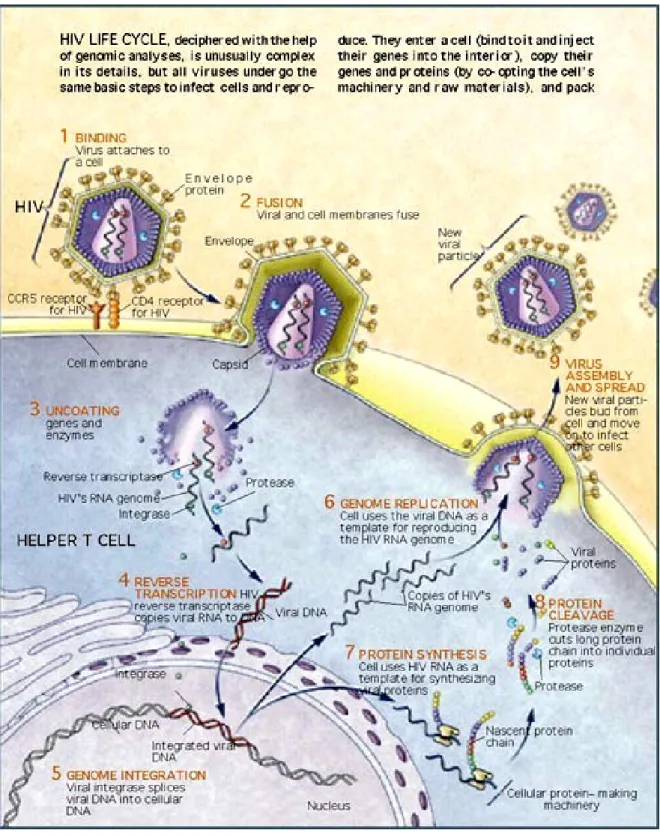
Il ciclo replicativo di FIV è simile a quello degli altri lentivirus (Fig 1.3) e comporta la fusione dell’envelope con la membrana plasmatica della cellula bersaglio, la liberazione al suo interno dell’acido nucleico, l’integrazione nel genoma dell’ospite e la formazione di nuova progenie virale.
La prima fase consiste nell’attacco da parte della proteina SU al recettore CD134 e corecettore CXCR4 presenti sulla membrana cellulare (Shimojima
et al., 2004). I cambiamenti conformazionali subiti dalla glicoproteina di
superficie in seguito a tale legame consentono l’esposizione dei domini della proteina TM che media poi la fusione tra la membrana virale e quella della cellula ospite garantendo il rilascio del capside nel citoplasma. In questo compartimento il capside si disgrega liberando l’RNA virale che, per azione della RT, viene retrotrascritto in una molecola completa di DNA a doppio filamento. Il DNA a doppia elica entra nel nucleo attraverso un meccanismo di trasporto attivo del complesso di pre-integrazione e, grazie all’azione dell’integrasi virale, si inserisce nel genoma della cellula ospite.
Questo enzima, infatti, è in grado di riconoscere le LTR e tagliarne le quattro basi terminali generando estremità 3’ spaiate.
L’IN taglia anche il genoma cellulare, nel quale produce estremità fosforilate al 5’ che si legano covalentemente ai gruppi ossidrilici 3’ del DNA virale. Una volta integratosi, il provirus è trascritto dalla RNA polimerasi II cellulare a partire dalla regione R, producendo numerose copie di RNA che nel citoplasma verranno in parte tradotte in proteine virali e in parte incorporate nel virus progenie.
L’ultima fase consiste nell’assemblaggio del virione per il quale sono necessarie le proteine MA, CA, NC. Quest’ultima interagisce con il genoma, permettendo la sua introduzione nel virione nascente, la CA interagisce con
Figura 1.3: Ciclo replicativo di FIV (da
hiv.buffalo.edu/assets/images/hiv_virus_in_action, modificata)
altre CA di altre poliproteine, la MA interagisce con la membrana cellulare, nella quale permette l’incorporazione delle proteine SU e TM. Il segnale di incapsidamento (ψ), necessario affinché due copie di RNA virale siano inserite nel capside, è localizzato nella regione compresa tra l’UTR e l’inizio del gene gag (Browning et al., 2003). Durante l’assemblaggio vengono anche incorporati i precursori Pol che verranno in seguito processati nei quattro enzimi RT, IN, DU e PR.
Il ciclo si conclude con il processo di gemmazione, durante il quale il virus acquisisce l’envelope e viene rilasciato dalla cellula come virione maturo.
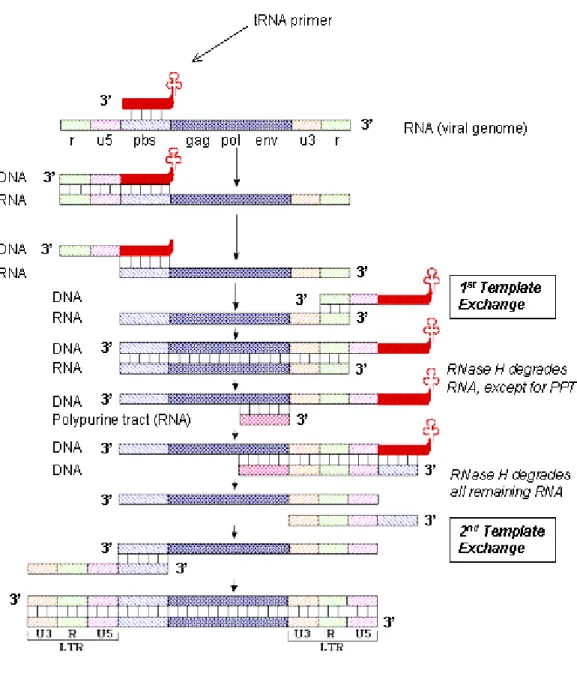
1.4.1 La retrotrascrizione
La retrotrascrizione (Fig 1.4) è uno dei punti chiave della replicazione dei retrovirus poiché la trasformazione dell’RNA virale in un frammento di DNA a doppio filamento ne garantisce l’integrazione nel genoma della cellula bersaglio.
Questo processo è possibile grazie all’attività della RT, una DNA polimerasi RNA dipendente, che, come le altre polimerasi, necessita di un innesco. Il primer è costituito da un tRNA-lys cellulare che si appaia con un sito localizzato a circa 200 basi di distanza dall’estremità 5’ dell’RNA virale denominato primer binding site (PBS). La RT parte da questo punto e retrotrascrive le regioni U5 e R arrestandosi al 5’ della molecola di RNA. A questo punto avviene il primo salto in cui la RT, il tRNA e il breve frammento di DNA sintetizzato si portano all’estremità 3’ del genoma virale. Questo passaggio è reso possibile dalla presenza di due regioni R identiche alle due estremità e dall’attività RNasi-H della RT che degrada la regione R al 5’ dell’RNA stampo consentendo il rilascio del DNA neosintetizzato e il suo legame alla regione R al 3’. Da qui riparte la sintesi del filamento negativo di DNA che procede fino al 5’ dello stampo di RNA.
Il risultato finale del salto e dell’allungamento della catena è l’addizione di un segmento U3 all’estremità 5’ e la formazione della sequenza U3-R-U5 che da origine agli LTR.
Figura 1.4: Meccanismo di retrotrascrizione (da
science.uwaterloo.ca./corse_notes/biology/biol442/revtrans.htlm,
modificata)
Terminata la sintesi del filamento negativo, la RT avvia quella del frammento positivo partendo da un frammento di RNA, presente a livello della regione U3 e noto come polypurine tract (PPT), residuo della degradazione operata dalla RNasi-H. Questo processo prevede un secondo salto e la conseguente generazione di una seconda LTR all’altra estremità della molecola.
1.5 Vettori virali
Caratteristica peculiare dei virus è quella di trasportare e liberare il proprio genoma all’interno delle cellule ed utilizzare l’apparato biosintetico cellulare per esprimere i propri geni.
Il concetto sul quale si basano la sintesi e lo sviluppo di un vettore virale è quello di sfruttare le capacità proprie dei virus per veicolare geni eterologhi in opportune cellule bersaglio a scopo terapeutico o vaccinale.
Nel primo caso l’isolamento e la caratterizzazione funzionale dei geni umani ha favorito lo sviluppo di nuovi approcci terapeutici per la cura di patologie genetiche basati sulla sostituzione del gene mutato con una copia funzionale veicolata nelle cellule deputate alla correzione del difetto genetico.
In ambito vaccinale riuscire ad indirizzare in cellule specifiche (cellule presentanti l’antigene) l’espressione di un transgene codificante per un immunogeno potrebbe risultare un approccio efficace nell’induzione di una robusta risposta immunitaria contro patogeni in grado di eludere facilmente il sistema immunitario.
Nel tentativo di perfezionare efficienza e efficacia di gene delivery sono state elaborate diverse tecniche che utilizzano approcci fisici e comprendono l’incapsulamento del DNA nei liposomi (Gao and Huang, 1995) o l’inoculo di plasmidi d’espressione eucariotica sia per inoculazione diretta sia con gene
gun technology (Prince 1998). Tuttavia l’utilizzo dei vettori virali si è rivelato
nettamente più efficiente e rappresenta, ad oggi, la strategia più utilizzata. A questo scopo sono stati realizzati diversi vettori virali basati su adenovirus, herpes simplex virus (HSV), virus adeno-associati (AAV) e
retrovirus. Per aumentare il livello di sicurezza questi virus sono stati opportunamente ingegnerizzati in modo tale da poter continuare ad esprimere il gene d’interesse nelle cellule bersaglio pur perdendo le proprietà infettive.
1.5.1 Vettori adenovirali
Gli adenovirus sono virus a DNA a doppio filamento di circa 30-35 Kb responsabili di diverse patologie umane, principalmente a carico del sistema respiratorio.
I vettori di prima generazione prodotti da virus adenovirali presentavano la sola sostituzione del gene E1, importante per l’espressione genica e la replicazione, con un gene eterologo. Il gene E1 veniva poi fornito in trans da cellule specifiche in cui il vettore era prodotto (Graham and Prevec, 1995).
Una seconda generazione di vettori adenovirali è stata realizzata grazie all’eliminazione di una regione più vasta del genoma virale che comprendeva i geni E2 ed E4 e all’uso di appropriate linee cellulari per la produzione del virus (Wang and Finer, 1996), tuttavia, in questo caso, la durata dell’espressione genica era notevolmente ridotta.
Ulteriori ricerche hanno portato alla sintesi di vettori di terza generazione (gutted vectors) contenenti come sequenze di derivazione virale soltanto le
viral terminal repeat e la sequenza di incapsidamento (Parks et al., 1996).
Questi vettori possono essere prodotti in una linea cellulare che fornisce E1 e in presenza di un virus helper, difettivo di questo gene, che esprime, in trans, tutte le altre proteine virali. La difficoltà incontrata in questo approccio consiste nella purificazione del vettore di trasferimento da quello
helper; non si rilevano inoltre aumenti significativi nell’espressione genica.
Inoltre, uno dei principali problemi per l’impiego di vettori adenovirali è l’alta risposta cellulo-mediata e umorale che si scatena contro le proteine dell’involucro adenovirale quando iniettato in vivo (Robbins e Ghivizzani, 1998) e che, se da un lato impedisce l’espressione del gene eterologo,
dall’altro può determinare anche notevoli problemi di sicurezza, in quanto può portare all’induzione di shock anafilattico nell’ospite.
A causa delle attuali limitazioni, i vettori adenovirali sono inappropriati per trattare sia disordini genetici, in cui è necessaria un’espressione genica a lungo termine, sia per la vaccinazione in cui gran parte della risposta immunitaria indotta è rivolta verso epitopi virali.
1.5.2 Vettori HSV
HSV è un virus a DNA a doppio filamento di circa 150 Kb codificante per più di 70 proteine virali.
I vettori basati su questo virus hanno il vantaggio di avere un’elevata capacità di clonaggio, di infettare cellule non in attiva replicazione e di stabilire latenza in alcuni tipi cellulari.
L’interesse in questo genere di vettori nasce anche dalla possibilità di rimanere latenti nelle cellule neuronali, proprietà che li rende un potenziale mezzo per il trattamento di patologie neurologiche quali morbo di Parkinson e Alzheimer (Fink et al., 2003; Maguir-Zeis et al., 2001).
Per rendere HSV replicazione difettivo è necessario eliminare l’espressione delle proteine che agiscono nelle prime fasi dell’infezione e che attivano l’espressione genica di altri promotori di HSV (Marconi et al., 1996; Wu et
al., 1996).
Uno dei problemi connessi all’impiego di questi vettori è la tossicità rilevata in diversi istotipi cellulari. Una possibile soluzione consiste nella delezione di numerosi prodotti dei geni a risposta immediata o nell’uso di ampliconi; in questo approccio, vengono co-trasfettati un plasmide contenente l’origine di replicazione e la sequenza di packaging di HSV con un cosmide contenente il genoma di HSV. Il virus risultante contiene solo le sequenze del plasmide e la tossicità viene eliminata (Heister et al., 2004).
Come gli adenovirus, anche i vettori HSV perdono rapidamente la capacità di esprimere il transgene. Per questo motivo e per le intrinseche proprietà patogene non ancora risolte, il loro impiego è, a tutt’oggi, piuttosto limitato.
1.5.3. Vettori AAV
I virus adeno-associati (AAV) fanno parte della famiglia Parvoviridae, sono costituiti da DNA a singolo filamento e necessitano di un virus helper come un adenovirus o HSV per replicarsi.
AAV codifica per due proteine denominate rep e cap, che, nella costruzione di un vettore AAV, possono essere delete e fornite in trans per la produzione della particella virale (Rolling e Samulski, 1995).
Un vettore di questo genere può essere trasfettato in cellule con un adenovirus mancante di E1 che svolge la funzione di virus helper.
AAV wild-type può infettare cellule umane quiescenti e integrarsi stabilmente in un locus specifico del cromosoma 19; quest’ultima proprietà è fondamentale per garantire un’espressione a lungo termine dei geni eterologhi. Inoltre, non sembrano esistere patologie associate all’infezione da AAV e il sito di integrazione non codifica per alcun gene importante.
Tuttavia il virus ricombinante, con un gene terapeutico al posto dei geni capsidici e della replicasi virale, perde la capacità di integrarsi stabilmente e in alcuni tipi cellulari resta episomiale. Un ulteriore svantaggio è che il genoma virale può ospitare geni eterologhi di lunghezza non maggiore a 5.2 Kb.
Per tali motivi, se da un lato AAV sembra essere un eccellente meccanismo di veicolazione di geni eterologhi, dall’altro la sua applicazione pratica resta limitata.
1.5.4 Vettori retrovirali
I retrovirus sono gli unici virus noti a possedere genoma diploide, formato da due copie uguali di una molecola di RNA monocatenario, ciascuna di circa 9 Kb, unite fra loro alla estremità 5’ da un appaiamento delle basi.
Numerosi vettori retrovirali sono stati realizzati a partire dal virus della leucemia murina (MLV) (Miller et al., 1995) e sono stati i primi ad essere
impiegati in un trial clinico per correggere la deficienza di adenosina deaminasi, causata da un disordine genetico (Blaese et al. 1995).
Nella costruzione di un vettore di trasferimento tutti gli elementi in cis, necessari per l’espressione genica sono mantenuti; i geni gag, pol ed env possono invece essere rimossi e forniti in trans attraverso l’uso di più plasmidi o con l’utilizzo di cellule helper ingegnerizzate (packaging cells) in grado di esprimere le proteine virali necessarie.
I vettori retrovirali hanno il vantaggio di poter trasportare fino a 10 Kb di DNA eterologo e di garantire un’espressione a lungo termine dei geni eterologhi grazie alla loro capacità di integrarsi nel genoma della cellula ospite.
Tuttavia, la maggior parte dei retrovirus non è in grado di attraversare la membrana nucleare e l’infezione è limitata alle cellule che vanno incontro a mitosi perché durante questa fase la membrana si disgrega; per questo motivo, i vettori retrovirali possono essere utilizzati per trasdurre solo cellule in attiva replicazione.
Il loro impiego, quindi, è limitato ad alcuni casi specifici, come la terapia anticancro (in cui le cellule mutate si replicano molto velocemente), ma non possono essere utilizzati per tutte quelle patologie che coinvolgono cellule quiescenti.
1.5.5 Vettori lentivirali
I lentivirus hanno molte caratteristiche in comune con i retrovirus; hanno, però, evoluto un meccanismo di trasporto attivo al nucleo che permette loro il passaggio attraverso la membrana nucleare. I vettori lentivirali presentano quindi un vantaggio in più rispetto a quelli retrovirali, poiché sono in grado di replicare anche in cellule quiescenti.
Uno dei primi modelli di vettori lentivirali è stato sviluppato a partire da HIV-1 (Naldini et al., 1996). La maggior parte dei vettori HIV si basa su un sistema split-component in cui le sequenze cis-agenti, necessarie per il trasferimento del genoma virale alle cellule bersaglio, sono separate da
quelle codificanti per proteine virali trans-agenti. In un sistema di questo tipo vengono solitamente realizzati tre costrutti a DNA: uno di packaging per l’espressione di proteine strutturali ed enzimi necessari per il ciclo vitale del virus e forniti da gag e pol, un costrutto che esprime l’envelope virale e un terzo vettore in cui è clonato il transgene e che mantiene le sequenze
cis-agenti necessarie per retrotrascrizione, integrazione e incapsidamento.
Quest’ultimo, infatti, è l’unico costrutto a mantenere la sequenza ψ e ad essere incapsidato nella particella virionica. I tre plasmidi vengono inoculati in cellule specifiche e, una volta raggiunto il nucleo, vengono trascritti e tradotti. Il risultato finale è la formazione di un virus ingegnerizzato contenente una sequenza di RNA codificante per il gene eterologo al posto del genoma virale. Le particelle virali così prodotte vengono utilizzate per trasdurre cellule target. In seguito all’interazione tra antirecettore virale e recettore cellulare, l’RNA viene rilasciato nella cellula, dove, dopo retrotrascrizione e ingresso nel nucleo, si integra nel genoma ospite ed esprime il transgene in maniera costitutiva (Fig 1.5).
La tendenza nella produzione di un vettore basato su questo sistema consiste nel separare il più possibile le componenti del genoma virale e ridurre al minimo la produzione di proteine non necessarie, per rendere altamente improbabile la possibilità di ricombinazione e formazione di una particella virale infettante. Seguendo questa linea, è iniziata la ricerca per la produzione di vettori di terza generazione self-inactivating (SIN). Il principio su cui si basa un vettore di questo genere è che, una volta che è avvenuto il
gene delivery, il virus perde alcuni elementi cis-agenti necessari per un altro
ciclo di replicazione.
Per la realizzazione di un vettore SIN, oltre alla delezione di parte dell’LTR in 5’, si va ad agire sulla regione U3 al 3’ LTR nella quale vengono eliminati la TATA box e i siti di legame per i fattori di trascrizione Sp1 e NF-kB (Miyoshi
et al., 1998).
Poichè durante la retrotrascrizione l’U3 deleto all’estremità 3’ fa da templato per generare l’LTR al 5’, il DNA provirale così sintetizzato ha entrambe le LTR inattive. In questo stato, l’unica regione del genoma provirale ad essere trascritta è quella della cassetta di espressione perché sotto controllo del
Transgene Gag Pol Env Costrutto vettore Costrutto di packaging Costrutto env Trasfezione Trasduzione Proteina d’interesse Transgene Gag Pol Env Costrutto vettore Costrutto di packaging Costrutto env Trasfezione Trasduzione Proteina d’interesse
Figura 1.5: Meccanismo d’azione di un vettore lentivirale basato sul
sistema split-component
promotore interno. L’espressione del gene eterologo è, quindi, ugualmente garantita e l’efficacia dei vettori SIN nel trasdurre cellule quiescenti è paragonabile a quella dei vettori di seconda generazione (Zufferey et al., 1998). In un vettore di questo genere l’assenza di una regione U3 completa, unita alla presenza al 5’ di un LTR ibrida (CMV-LTR), riduce notevolmente la regione di omologia e, di conseguenza, la possibilità di formazione di una particella virionica wild-type.
L’impiego di un vettore SIN presenta un ulteriore vantaggio in quanto l’inserimento casuale del provirus nel genoma della cellula ospite può indurre mutagenesi inserzionale per attivazione di proto-oncogeni cellulari che, in seguito all’inserzione, si vengono a trovare a valle dell’LTR (Trono, 2003). La delezione dell’U3 riduce notevolmente questo rischio poiché rende l’LTR completamente inattiva.
Tutti i diversi tipi di vettori HIV realizzati sono in grado di trasdurre diversi tipi cellulari e funzionano molto bene in modelli animali (Kordower et al., 2000; Consiglio et al., 2001; Baekelandt et al., 2002); tuttavia, poichè i vettori HIV derivano da un agente infettivo che produce una patologia letale nell’uomo, l’impiego in trial clinici, ad oggi, non è consentito.
Una valida alternativa è rappresentata dall’utilizzo di vettori basati su lentivirus che non danno patologia nell’uomo, come SIV, o, ad ulteriore garanzia di sicurezza, che infettano i non primati come FIV.
1.5.6 Vettori FIV
Non ci sono evidenze che FIV sia patogeno per l’uomo; infatti, numerose persone esposte al virus, a causa di morsi o ferite provocate da gatti infetti, non hanno mai manifestato sieroconversione (Willet et al., 2003).
Questa restrizione è dovuta ad un insieme di fattori che comprendono la bassa attività dell’LTR, la funzionalità subottimale del sistema Rev-RRE e il ridotto tropismo dell’envelope in cellule umane.
Poiché FIV è un lentivirus molto simile ad HIV, un vettore FIV-derivato unisce ai vantaggi funzionali di HIV, l’assenza di infettività allo stato
wild-type nell’uomo. Sebbene queste considerazioni suggeriscano un’elevata 20
sicurezza nell’impiego di vettori FIV, allo stesso tempo pongono il problema della necessità di adattare il modello FIV a cellule umane.
Il primo passo nella produzione dei vettori FIV è stato, quindi, quello di sostituire la regione U3 del 5’ LTR con un forte promotore ubiquitario costitutivamente espresso, quale quello del citomegalovirus umano (CMV) (Poeschla et al., 1998).
Questa singola modificazione ha consentito la produzione di numerose particelle vettore anche in cellule umane e ha portato alla realizzazione di vettori FIV di seconda generazione che, come quelli HIV, si basano sull’utilizzo di un sistema split component costituito da tre costrutti: un vettore di trasferimento, uno di packaging e un costrutto codificante per l’env. Nel vettore di trasferimento viene sostiuito l’U3 dell’LTR con un promotore più forte ed è mantenuta la sequenza d’incapsidamento (ψ) che è compresa tra la 5’ UTR e le prime 100 paia basi di gag (Browning et al., 2003), mentre vengono delete le ORF di gag e pol fornite dal packaging. L’espressione del transgene è, solitamente, sotto il controllo di un promotore interno che, nella maggior parte dei casi, è il CMV o il promotore della fosfoglicerato chinasi murina (PGK).
Nel vettore di packaging le LTR al 5’ e al 3’ sono sostituite rispettivamente con il CMV e con una regione di poliadenilazione, viene inoltre deleta la sequenza ψ per impedirne l’inserimento nel virione maturo.
Analogamente al vettore di packaging, anche nel costrutto codificante per l’envelope manca la sequenza ψ. E’ quest’ultimo costrutto a determinare il tropismo del vettore che può essere quindi pseudotipizzato con l’utilizzo di
envelope eterologhi al fine di facilitarne l’applicazione a diversi tipi cellulari.
Per studi in vitro, si utilizzano principalmente l’envelope anfotropica di MLV o quella codificante per la proteina G del virus della stomatite vescicolare (VSV-G).
Il recettore dell’envelope anfotropica è una proteina di trasporto di fosfato che si lega con l’antirecettore in maniera altamente specifica (Kozak et al., 1995); la proteina G del VSV, invece, interagisce con un fosfolipide di membrana ubiquitario permettendo l’impiego dei vettori su un gran numero di istotipi cellulari (Friedmann e Yee, 1995).
Inoltre, a causa della maggior stabilità e della grande resistenza a stress meccanici del VSV-G è possibile concentrare le particelle utilizzando l’ultrancentrifugazione aumentando, così, il titolo del vettore.
Per quello che riguarda le applicazioni in vivo, il vettore può essere pseudotipizzato con envelope di virus che attaccano un tessuto specifico, garantendo l’ingresso del DNA eterologo solo nelle cellule che lo necessitano o permettendo di andare ad agire anche sui tessuti più resistenti all’infezione.
Al fine di eliminare tutte le regioni codificanti per fattori non necessari alla produzione e alla funzionalità del sistema, nella realizzazione dei vettori FIV di seconda generazione, sono state analizzate le proteine Vif, Orf-A e Rev per determinare il loro ruolo nei costrutti vettore e di packaging. Dallo studio è emerso che non c’è differenza significativa nell’efficienza di trasduzione dei costrutti in cui sono presenti Vif e Orf-A da quelli in cui queste due proteine sono delete (Johnston et al., 1999).
Il complesso Rev/RRE appare invece strettamente necessario per ottenere alti titoli di vettore, poiché garantisce l’export nucleare degli mRNA
unspliced e single spliced che daranno origine alle principali proteine
virioniche (Gasmi et al., 1999). In particolare è stato osservato che l’inserimento dell’RRE prima della cassetta interna di espressione del transgene contribuisce ad aumentare ulteriormente il titolo (Johnston et al., 1999).
In tutti i costrutti che costituiscono il sistema split component, quindi, è necessaria la presenza di un sistema di export nucleare quale Rev/RRE o il
costitutive transport element (CTE), una sequenza altamente conservata del
virus Mason-Pfizer della scimmia, che facilita il trasporto nucleo-citoplasma attraverso un meccanismo Rev indipendente (Curran et al., 2000); mentre le regioni vif e ORF-A possono essere delete.
1.6 Applicazioni dei vettori lentivirali
Per l’applicazione dei vettori lentivirali a scopo di terapia genica o vaccinazione si distingue una tecnica ex vivo e una tecnica in vivo. Nel primo caso cellule precedentemente raccolte dal paziente e mantenute in coltura sono trasdotte con il vettore terapeutico e reinoculate nel paziente. Il metodo diretto, cioè in vivo, prevede che le particelle di vettore, pseudotipizzate con un’envelope appropriata, siano prodotte dalla trasfezione di cellule in coltura e inoculate direttamente nel paziente. La scelta tra l’una e l’altra tecnica è dettata dall’accessibilità del distretto corporeo interessato.
I lentivirus, a differenza dei retrovirus, sono in grado di infettare anche cellule quiescenti quali staminali, muscolari, ematopoietiche, epatociti, cellule dendritiche, linfociti T e neuroni; le principali applicazioni in vivo ed
ex vivo dei vettori lentivirali riguardano, quindi, questo tipo di cellule.
Le cellule staminali ematopoietiche, in particolare, sono un target molto interessante per terapia genica, a causa del loro potenziale di differenziamento in cellule del sangue. Questa caratteristica le rende particolarmente adatte per il trattamento di numerose emoglobinopatie, prime fra tutti la β-talassemia e l’anemia a cellule falciformi, in cui il trasferimento di un singolo gene dovrebbe, teoricamente, eliminare il difetto genetico. In esperimenti condotti su topi affetti da β-talassemia intermedia è stata rilevata la sintesi a lungo termine di emoglobina chimerica, a seguito dell’impianto nel midollo osseo di cellule trasdotte con un vettore lentivirale codificante per il gene della β-globina umana (May et al. 2002).
L’espressione a lungo termine di geni terapeutici è molto importante anche a livello del fegato per la cura di patologie epatiche e sistemiche che comprendono disturbi metabolici, emofilia e ipercolesterolemia; poiché gli epatociti sono quiescenti, i vettori lentivirali sono quelli più adatti per la veicolazione dei geni eterologhi in queste cellule.
Vettori FIV si sono rilevati altamente efficienti sia nella trasduzione di cellule epatiche in vitro, specialmente utilizzando promotori tessuto-specifici, sia
nell’espressione a lungo termine del transgene in vivo (Condiotti et al., 2004).
Come i vettori adenovirali, anche quelli lentivirali sono in grado di trasdurre cellule retiniche e possono essere applicabili come terapia per la cura di disturbi oculari. Questi vettori presentano marcate differenze correlate alle loro capacità biologiche, tuttavia entrambi inducono espressione a lungo termine del gene eterologo nell’epitelio pigmentato retinale (RPE) a seguito di iniezione subretinale nell’occhio della cavia (Loewen et al. 2004).
Studi condotti in parallelo con vettori adenovirali e FIV hanno dimostrato che questi ultimi risultano più promettenti, dal momento che sono in grado di trasdurre una regione più ampia di RPE e causano una minor distruzione dell’architettura retinica che, nel caso dei vettori adenovirali, è da attribuire all’alta risposta immunitaria scatenata nell’ospite dagli adenovirus (Reichel
et al., 2001).
I vettori FIV sono in grado di trasdurre anche cellule immunitarie del cervello e neuroni (Kyrkanides et al., 2003) e possono rappresentare un buon strumento per la cura di alcune patologie a carico del sistema nervoso centrale (SNC). Vettori FIV-derivati sono in grado di veicolare geni eterologhi anche nelle cellule della materia bianca (Alisky et al., 2003) che gioca un ruolo essenziale nello svolgimento delle funzioni cerebrali e nell’interazione tra le diverse regioni cerebrali (Filey, 1998).
Poichè diverse patologie neurodegenerative, quali la leucodistrofia metacromica e la sclerosi multipla, agiscono a livello della materia bianca e alcuni tumori cerebrali la utilizzano per diffondere nelle diverse aree del cervello (Geer et al 1997), vettori FIV potrebbero rivelarsi un ottimo strumento per sviluppare un nuovo approccio terapeutico per la cura di queste malattie.
Vettori lentivirali sono stati applicati anche nell’ambito della cura della fibrosi cistica, causata da una mutazione del gene codificante per un canale di trasporto del Cl-, importante affinché i polmoni svolgano la loro normale
funzione. La sostituzione di questo gene con la copia funzionale, quindi, può essere un punto cruciale nella cura di questa patologia.
I primi vettori codificanti per il cystic fibrosis transmembrane conductance
regulator (CFTR) ad essere stati testati sono quelli adenovirali, perché il loro
naturale sito di infezione è l’epitelio respiratorio, ma vettori di questo genere, oltre ad indurre una forte risposta immunitaria nell’ospite, determinavano una graduale perdita nell’espressione genica (Wilson et al., 1994).
Risultati più soddisfacenti sono stati ottenuti utilizzando vettori lentivirali che rappresentano un efficace mezzo di trasduzione per cellule epiteliali respiratorie quiescenti. Infatti vettori FIV codificanti per il gene CFTR, testati
in vitro su cellule di epitelio respiratorio umano, hanno determinato una
correzione nel trasporto del Cl- persistente per tutta la durata della coltura
cellulare. Quando questo approccio è stato applicato in vivo, lo stesso vettore FIV ha trasdotto dall’1 al 14% di cellule dell’epitelio respiratorio di coniglio (Wang et al., 2002).
Finora la limitazione principale nell’impiego in vivo è stata la bassa efficienza di trasduzione dei vettori applicati alla superficie apicale dell’epitelio respiratorio.
Tuttavia, studi più recenti hanno suggerito che il recettore α per il folato (FRα), espresso abbondantemente nell’epitelio respiratorio umano polarizzato, è coinvolto nell’ingresso dei filovirus nella cellula bersaglio e, di conseguenza, vettori FIV pseudotipizzati con envelope dei virus di Marburg o dell’Ebola hanno dimostrato una buona efficienza di trasduzione di colture cellulari derivanti dall’epitelio respiratorio umano (Sinn et al., 2003).
Un’altra patologia in cui è auspicabile l’utilizzo di un vettore lentivirale a scopo terapeutico è l’artrite reumatoide, una malattia sistemica autoimmune che colpisce la membrana sinoviale delle articolazioni portando ad una progressiva distruzione della cartilagine. E’ stato dimostrato che l’espressione di geni codificanti per l’interleuchina 1 e di inibitori del tumor
necrosis factor α (TNF-α) è in grado di invertire la progressione della
malattia nel coniglio (Otani et al., 1996). Poiché i sinoviociti si dividono molto lentamente in coltura, i vettori lentivirali si rivelano particolarmente adatti alla loro trasduzione; infatti, vettori FIV applicati in vitro hanno trasdotto il 60% di queste cellule (Lin et al., 2004). In un esperimento
condotto in parallelo in vivo, sinoviociti umani trasdotti con TNF-α e iniettati nella giuntura del ginocchio di topi con immunodeficienza severa combinata (SCID) hanno riprodotto una situazione patologica simile all’artrite reumatoide, inducendo erosione della cartilagine dell’osso in una settimana. I vettori FIV sembrano essere quindi, non solo un ottimo mezzo terapeutico, ma anche uno strumento efficace per creare modelli animali adatti allo studio di determinate patologie umane.
Un possibile e importante impiego dei vettori lentivirali riguarda la veicolazione di geni eterologhi in ambito vaccinale. In questo ambito particolare attenzione è rivolta alla veicolazione del gene eterologo (immunogeno) alle cellule dendritiche (DC), le cellule presentanti l’antigene più efficienti (APC) (Akbari et al., 1999). Le DC catturano e trasferiscono le informazioni ricevute dall’esterno al sistema immunitario dell’ospite e sono importanti per l’attivazione della risposta immune primaria, per l’induzione di una tolleranza immunologia e per la regolazione del tipo di risposta cellulo-mediata da attivare. Infatti le DC esprimono recettori per la porzione costante (Fc) delle immunoglobuline G (IgG) che mediano l’internalizzazione dei complessi antigene-IgG e promuovono l’efficiente presentazione dell’antigene da parte del complesso maggiore di istocompatibilità (MHC) di classe I e II.
Nell’ambito della terapia antitumorale la possibilità di produrre un vaccino efficace dipende dall’abilità di indurre una risposta cellulo-mediata specifica verso il tumore e notevoli passi avanti in questo campo sono stati fatti dall’ identificazione di antigeni tumorali specifici (Bertolaccini and Oliviero, 2001).
I linfociti citotossici (CTL) sono la componente critica per la risposta immune ai tumori perchè, insieme alle cellule natural killer, si infiltrano nel tessuto tumorale e sono in grado di controllarne la crescita; l’induzione di un alta risposta CTL è, quindi, un punto chiave per la corretta strategia di vaccinazione.
Le DC sono potenti attivatori della risposta CTL e sono possibili induttori di immunità antitumorale quando stimolate con peptici antigenici o proteine; per questo motivo molti lavori si sono concentrati sull’ottimizzazione di
strategie che vanno ad agire su queste cellule e, in particolare, sui vettori virali che sono in grado di trasdurre le DC (Brossart et al. 1997). L’impiego di questi ultimi in vivo è stato finora limitato dalla piccola frazione captata dalle DC quando il vettore è inoculato nell’animale, tuttavia da recenti studi è stato dimostrato che se il vettore esprime una proteina di fusione Ag-Fc, la cattura e la presentazione dell’Ag da parte delle DC è notevolmente aumentata (You et al., 2001).
Tra le diverse classi di vettori virali quelli di terza generazione derivati dai lentivirus si sono rilevati tra i più efficienti, infatti, dopo essere stati introdotti come strumento per la trasduzione ex vivo di DC (Esslinger et al., 2002), si sono dimostrati anche molto efficienti nel trasdurle in vivo e nell’indurre una risposta CTL molto forte (Esslinger et al., 2003). In questo caso l’efficienza di un vettore lentivirale è dovuta alla sua capacità di trasdurre le DC immature e quiescenti, che si rivela cruciale per la sua efficacia come vaccino. Questo alto livello di efficienza è da attribuire anche alla capacità dei vettori lentivirali di sfuggire all’azione del sistema immunitario. Infatti, uno dei principali problemi nell’impiego di altri vettori virali è che spesso questi derivano da agenti patogeni già utilizzati per la produzione di vaccini, basati su virus attenuati, verso i quali è presente un’immunità pre-esistente che interferisce con la funzionalità del vettore. In questo senso può essere auspicabile l’impiego di vettori basati su un virus che non infetta l’uomo, come FIV, verso i quali non esiste immunità acquisita precedentemente.