UNIVERSITÀ DI PISA
FACOLTÀ DI MEDICINA VETERINARIA SCUOLA DI SPECIALIZZAZIONE
“SANITÀ ANIMALE, ALLEVAMENTO E PRODUZIONI ZOOTECNICHE ”
“HEPATITIS E VIRUS (HEV): INDAGINI
SIEROLOGICHE, BIOMOLECOLARI E PATOLOGICHE
SULLA POPOLAZIONE DI CINGHIALI (Sus scrofa)
DELLA PROVINCIA DI PISA”
RELATORE
Prof. Alessandro Poli
CANDIDATO
Dott. Roberto Nardini
CORRELATORE Dott. Maurizio Mazzei
«dedicato a HGW XX/7, con gratitudine»
“Blessed be the man who expects nothing, for he shall never be disappointed” (Alexander Pope)
INDICE
INTRODUZIONE...1
PARTE I: HEPATITIS E VIRUS: EZIOLOGIA, EPIDEMIOLOGIA E RISCHIO ZOONOSICO CAPITOLO 1: EZIOLOGIA...3
1.1 CARATTERISTICHE GENERALI DEL VIRUS...3
1.2 STRUTTURA GENOMICA...4
1.3 INQUADRAMENTO TASSONOMICO...6
1.4 GENOTIPI VIRALI...…...7
CAPITOLO 2: EPIDEMIOLOGIA...9
2.1 EPIDEMIOLOGIA NELL’UOMO...9
2.1.1. Caratteristiche dell’infezione nei paesi in via di sviluppo...10
2.1.2. Caratteristiche dell’infezione nei paesi industrializzati...12
2.2 EPIDEMIOLOGIA NELLE ALTRE SPECIE ANIMALI RECETTIVE ALL’INFEZIONE...14
2.2.1. HEV nel suino...14
2.2.2. HEV nel Cinghiale...16
2.2.3. Avian HEV...17
2.3 DISTRIBUZIONE GEOGRAFICA DEI DIVERSI GENOTIPI DI HEV...18
CAPITOLO 3: PATOGENESI E SINTOMATOLOGIA...20
3.1 LA MALATTIA NEL SUINO...21
3.1.1. Patogenesi...…...21
CAPITOLO 4: HEV COME AGENTE DI ZOONOSI...28
4.1 POSSIBILITÀ DI INFEZIONE INTERSPECIFICA...28
4.2 OMOLOGIA TRA CEPPI UMANI E ANIMALI CIRCOLANTI SULLO STESSO TERRITORIO...29
4.3 HEV COME AGENTE DI ZOONOSI A TRASMISSIONE ALIMENTARE...32
PARTE II: INDAGINI SIEROLOGICHE, BIOMOLECOLARI E PATOLOGICHE SULLA POPOLAZIONE DI CINGHIALI (Sus scrofa) DELLA PROVINCIA DI PISA CAPITOLO 5: MATERIALI E METODI...36
5.1 CALCOLO DELLA NUMEROSITÀ CAMPIONARIA...36
5.2 ANIMALI...…...37
5.3 METODICHE DI DIAGNOSI DIRETTA..…...39
5.3.1 NESTED-PCR su pool di campioni fecali...39
5.3.1.1. Estrazione del RNA virale...39
5.3.1.1.1. Estrazione RNA...…..40
5.3.1.1.2. Retrotrascrizione...…..40
5.3.1.2 Prima PCR...40
5.3.1.3 Nested-PCR...….41
5.3.2 Nested-PCR su campioni singoli...42
5.4 METODICHE DI DIAGNOSI INDIRETTA...42
5.4.1. ELISA su siero...42
5.4.2. Analisi istologica su campioni di fegato.…...42
5.5 ANALISI STATISTICA...43
CAPITOLO 6: RISULTATI...44
6.1 METODICHE DI DIAGNOSI DIRETTA...44
6.1.2 Nested-PCR su pool di pool di campioni singoli....45
6.2 METODICHE DI DIAGNOSI INDIRETTA...46
6.2.1 ELISA su siero...46
6.2.2 Analisi istologica su campioni di fegato...48
CAPITOLO 7: DISCUSSIONE E CONCLUSIONI...50
BIBLIOGRAFIA...51
FONTI ICONOGRAFICHE...62
INTRODUZIONE
L'epatite E è una malattia infettiva ad eziologia virale che nell’uomo presenta sintomi clinici di epatite acuta e tassi di letalità prossimi al 2%, che possono superare il 20% nelle donne in gravidanza. L’agente responsabile è il virus dell’epatite E (Hepatitis E Virus, HEV).
Nell'uomo la malattia costituisce un importante problema di sanità pubblica nei paesi in via di sviluppo dove i focolai, spesso epidemici e prevalentemente dovuti alla contaminazione delle acque, possono presentare un numero elevato di casi.
Nei paesi industrializzati l'andamento è stato invece inizialmente sporadico; i primi casi sono stati riscontrati in persone di ritorno da paesi con infezione endemica; con il passare del tempo invece l'infezione ha assunto anche in molti paesi europei un carattere di endemicità. La trasmissione in questi paesi non si trasmette con il ciclo orofecale, ma avviene principalmente attraverso il contatto con animali infetti o loro prodotti.
L'epatite è stata riscontrata in numerose specie animali, ma il suino è considerato la principale specie reservoir, potenzialmente fonte di trasmissione per l'uomo sia per contatto diretto o indiretto, sia per via alimentare.
Anche il cinghiale è stato oggetto di studi di prevalenza rivelando la presenza diffusa del virus in molti paesi del mondo; focalizzando l'attenzione sul possibile ruolo dei suini selvatici nella trasmissione dell'infezione, in particolare in categorie professionali (operatori forestali, cacciatori, ristoratori) che vengono a contatto con gli animali vivi o loro prodotto.
Lo scopo di questa tesi di specializzazione è di descrivere i risultati di uno studio di prevalenza di HEV effettuato su un campione della popolazione di cinghiali della provincia di Pisa, effettuato utilizzando tecniche sierologiche, biomolecolari ed istologiche.
PARTE I:
HEPATITIS E VIRUS: EZIOLOGIA,
EPIDEMIOLOGIA E RISCHIO
CAPITOLO 1: EZIOLOGIA DELL’EPATITE E 1.1 CARATTERISTICHE GENERALI DEL VIRUS
Il virus dell’epatite E è un virus della famiglia Hepeviridae, genere
Hepevirus, di 27-34 nm, a simmetria icosaedrica e sprovvisto di
envelope (Figura 1). Il genoma è costituito da un RNA a singolo filamento positivo, con una coda poly(A) all’estremità 3’ . I virioni sono costituiti da una proteina maggiore del capside di dimensioni di 72kDa, e da una piccola proteina (1.5 kDa) la cui funzione rimane tuttora sconosciuta .
Figura 1: Immagine di HEV al microscopio elettronico
Il virus dell’Epatite E è in grado di tollerare variazioni di pH da alcalino ad acido, perché sopravvive all’acidità gastrica, ma non sembra tollerare elevate concentrazioni di sali. Per quanto riguarda la resistenza del virus al calore, temperature di 50-60°C si sono rilevate in grado di inattivarlo fino al 95%.
1.2 STRUTTURA GENOMICA
HEV è un virus a RNA a singolo filamento e ad orientamento positivo, di dimensioni di circa 7,3 kb . La regione codificante è preceduta da una corta regione non codificante (UTR, untranslated
region) di circa 25 nucleotidi, ed è seguita da una seconda UTR più
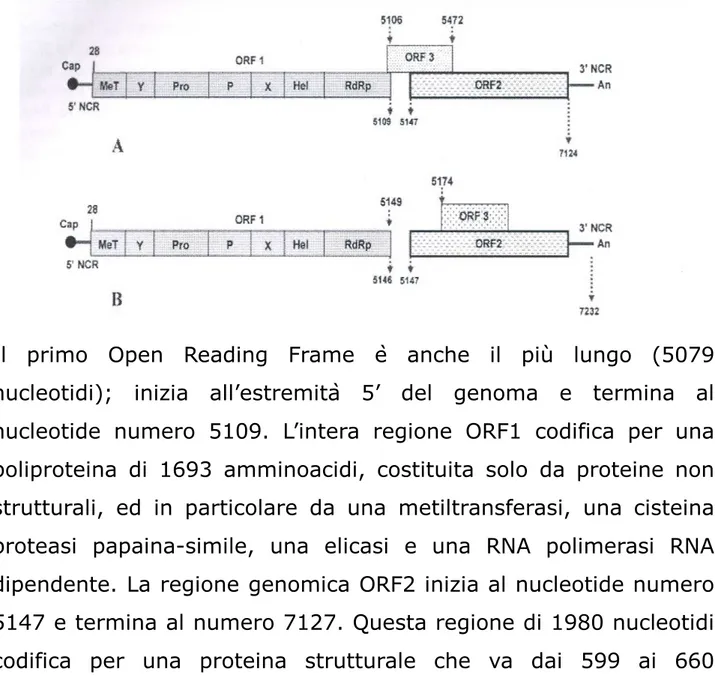
lunga (65-74 nucleotidi) e da una estremità poly(A) (1). La regione codificante è costituita da tre Open Reading Frames (ORF), chiamati ORF1, ORF2 e ORF3 (Figura 2).
Figura 2: Rappresentazione schematica del genoma di HEV
Il primo Open Reading Frame è anche il più lungo (5079 nucleotidi); inizia all’estremità 5’ del genoma e termina al nucleotide numero 5109. L’intera regione ORF1 codifica per una poliproteina di 1693 amminoacidi, costituita solo da proteine non strutturali, ed in particolare da una metiltransferasi, una cisteina proteasi papaina-simile, una elicasi e una RNA polimerasi RNA dipendente. La regione genomica ORF2 inizia al nucleotide numero 5147 e termina al numero 7127. Questa regione di 1980 nucleotidi codifica per una proteina strutturale che va dai 599 ai 660
amminoacidi (1). All’estremità 5’ della regione ORF2 si trova una regione di circa 300-450 nucleotidi che è la più conservata tra i diversi ceppi identificati di HEV, e che è stata recentemente utilizzate come riferimento per una classificazione in sottotipi dei diversi genotipi di HEV.
La proteina sintetizzata dalla regione ORF2 (pORF2) è una glicoproteina strutturale del peso di 88 KDa, caratterizzata dalla presenza di amminoglicani e da una regione ricca di arginina e lisina all’estremità N-terminale che costituisce il sito di ancoraggio al reticolo endoplasmatico. Inizialmente sintetizzata come precursore, viene successivamente processata tramite clivaggio in una proteina matura e glicosilata in tre siti (fenomeno frequente nelle proteine dell’envelope ma raro per una proteina capsidica). Studi condotti in vitro hanno dimostrato che la proteina è espressa sia a livello intracellulare sia sulla superficie cellulare . La proteina codificata dalla regione ORF2 è la proteina strutturale del capside, ed è responsabile della risposta anticorpale . Nonostante esistano notevoli differenze genomiche trai vari ceppi di HEV, a tutt’oggi viene riconosciuto un singolo sierotipo (1).
La regione ORF3 è di lunghezza di 369-372 basi, e si sovrappone alla regione ORF2 per 331 nucleotidi. Codifica per una piccola fosfoproteina immunogenica di lunghezza massima di 123 amminoacidi, di funzione non chiara, ma che sembra interagire con la proteina codificata dalla proteina ORF2 (1). La fosfoproteina presenta due domini idrofobici all’interno della sua molecola (alfa eliche transmembrana) con i quali sembra in grado, infatti, di legarsi al citoscheletro delle cellule epatiche. Inoltre tale proteina
crea un complesso con la proteina capsidica pORF2 fungendo da sito di ancoraggio per l’assemblamento del virione .
1.3 INQUADRAMENTO TASSONOMICO
Dopo la prima identificazione del virus tramite microscopia elettronica avvenuta nel 1983, fu suggerito di inserire il virus nella famiglia Picornaviridae, come il virus dell’epatite A di tipo 2 .
A seguito del clonaggio e sequenziamento completo del primo ceppo di HEV (Burma strain), per l’organizzazione genomica e le caratteristiche morfologiche, HEV fu invece inserito provvisoriamente in un genere separato della famiglia Caliciviridae . Questa classificazione era stata adottata a seguito dell’analisi della regione genomica codificante la proteina capsidica, che portava alla suddivisione della famiglia Caliciviridae in 5 principali cluster: Calicivirus umani Norwalk-like e Sapporo-like, virus dell’epatite E, esantema vescicolare da calicivirus swine-like e lapine (2). In seguito, prendendo in considerazione le più conservate regioni genomiche codificanti per l’elicasi e la polimerasi, sono stati inclusi con relativa sicurezza nella famiglia Caliciviridae solo quattro gruppi, escludendone il virus dell’epatite E (2).
In sequito, questa decisione è stata validata dall’inserimento nello studio comparativo, delle altre due famiglie virali inizialmente prese in considerazione per la filogenesi di HEV, le Togaviridae e le
Picornaviridae. Lo studio filogenetico della regione polimerasica ha
portato a concludere che la distanza esistente tra le famiglie
Caliciviridae, Picornaviridae, Togaviridae e HEV è la stessa (2) . Ad
oggi il virus è l'unico del genere Hepesvirus, della famiglia Hepeviridae, famiglia non inclusa ancora in nessun ordine. Nella
stessa famiglia del HEV è compreso il virus dell'epatite E aviare, seppur ancora non incluso in un genere definito.
1.4 GENOTIPI VIRALI
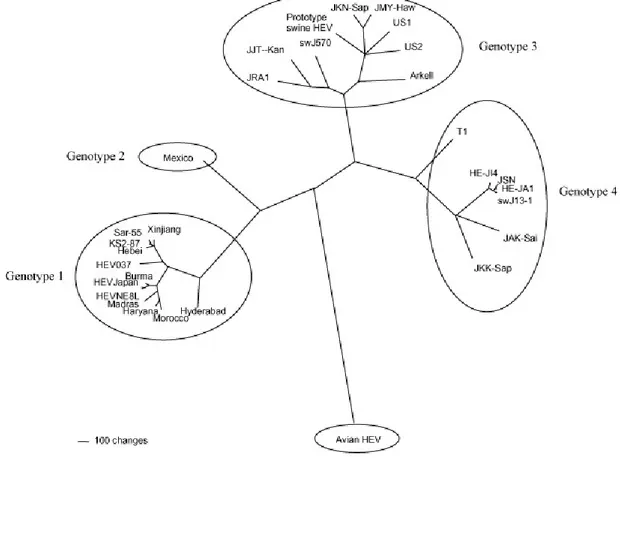
Anche se finora è stato identificato un solo sierotipo (3), una notevole diversità genomica viene osservata trai diversi ceppi di HEV ad oggi identificati (1). Diversi schemi per la classificazione del virus sono stati proposti (4), ma finora quello più accettato prevede la classificazione dei diversi isolati di HEV in 4 maggiori genotipi. Mentre i ceppi del genotipo 1 e 2 sono stati isolati solo dall’uomo (tranne che nel caso di un ceppo suino cambogiano identificato da Caron et al. 2006 (5), appartenente al genotipo 1), quelli del genotipo 3 e 4 sono stati identificati sia nell’uomo che in diverse specie animali, tra cui il suino, il cinghiale, il cervo e la mangusta. Il genotipo 1 fu identificato e sequenziato per la prima volta nel 1991, a partire da un campione proveniente dal Myanmar (formalmente Burma). Questo ceppo di riferimento presenta più dell’88,2% di identità nucleotidica con ceppi isolati in altri paesi dell’Asia e dell’Africa, che vengono per questo fatti rientrare nel genotipo 1 (1).
Nel 1992 è stato sequenziato un isolato virale proveniente da un focolaio verificatosi in Messico nel 1986, che si presentava nettamente distinto dal Burma strain, e per questo è stato classificato in un secondo genotipo.
Nel 1997, è stato riportato un caso di epatite E negli Stati Uniti, da un paziente che non aveva un’anamnesi di viaggi all’estero; questo ceppo considerato autoctono è stato sequenziato, ed è andato a costituire il prototipo di un terzo genotipo. Successivamente è stato
dimostrato che virus appartenenti a questo genotipo sono praticamente diffusi in tutto il mondo.
Il genotipo 4 è stato identificato nel 1999 in Cina, e differiva dai ceppi appartenenti al genotipo 1 circolanti sullo stesso territorio. Isolati appartenenti a questo genotipo sono stati identificati anche in Giappone e a Taiwan.
Recentemente è stata ipotizzata un’ulteriore suddivisione dei 4 genotipi in sottotipi, basandosi sulle sequenze dei 300-450 nucleotidi terminali della regione genomica dell’ORF2. Secondo questo studio, i genotipi 1,2,3 e 4 si possono suddividere rispettivamente in 5, in 2 , 10 e 7 sottotipi (Figura 3).
CAPITOLO 2: EPIDEMIOLOGIA 2.1 EPIDEMIOLOGIA NELL’UOMO
Si possono riconoscere due distinti pattern epidemiologici di infezione da HEV nell’uomo: endemico con focolai epidemici, che si manifesta prevalentemente nelle aree tropicali e subtropicali; sporadico, proprio invece dei Paesi industrializzati.
Nei Paesi in via di sviluppo l’infezione viene considerata endemica e si manifesta con focolai epidemici di vasta portata generalmente conseguenti alla contaminazione delle fonti d’acqua.
Nei Paesi industrializzati, invece, dove gli standard igienico-sanitari sono più elevati, l’infezione si manifesta sporadicamente, generalmente correlata a viaggi nelle aree endemiche, ma anche autoctona, probabilmente di natura zoonotico.
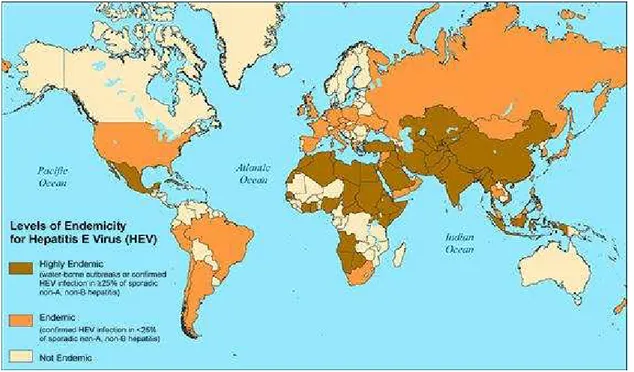
Nella maggior parte delle aree endemiche la sieroprevalenza è di circa il 5% nei bambini sotto 10 anni e tende ad aumentare con valori che raggiungono il 10-40% negli adulti sopra i 25 anni. Diversi studi sieroepidemiologici hanno inoltre rilevato una notevole prevalenza anticorpale anti-HEV (5-20%) nella popolazione sana di molti Paesi industrializzati, facendo pensare ad un’elevata diffusione dell’infezione, seppur in genere a livello subclinico. In Figura 4 è mostrata la diffusione di HEV a livello mondiale.
Figura 4: Livello di endemicità di HEV nei paesi del mondo
2.1.1 Caratteristiche dell’infezione nei Paesi in via di sviluppo
Il virus dell’epatite E è ampiamente diffuso nei paesi tropicali e sub-tropicali in via di sviluppo: gran parte dell’Asia, Nord Africa, Medio Oriente ed America centro-meridionale, dove l’infezione si manifesta in modo epidemico . Il tasso di letalità correlato all’infezione da HEV si aggira nei focolai epidemici intorno allo 0,2-4%, anche se raggiunge picchi del 20% in donne in gravidanza, specialmente nel primo trimestre.
La principale via di trasmissione dell’infezione è quella orofecale, in particolare legata alla contaminazione delle acque, in particolare in concomitanza di forti piogge e inondazioni. Altra via di trasmissione considerata è quella verticale: è stato dimostrato che il 50% delle donne che si infettano durante il terzo trimestre di gravidanza trasmettono l’infezione al feto.
Se l’età possa rappresentare un fattore di rischio per l’infezione è un aspetto ancora da chiarire dato che sull’andamento demografico dell’infezione da HEV in letteratura tutt’oggi esistono dati incompleti e contrastanti (6;7). Le epidemie colpiscono fasce molto ampie di popolazione e possono essere prolungate nel tempo con tassi di attacco che vanno dall’1% al 15%, e con i giovani adulti più colpiti dei bambini (8). Essendo l’epatite E un’infezione con trasmissione oro-fecale, ci si aspetterebbe di osservare pattern di infezione simili ad altri virus con le stesse caratteristiche, come il virus dell’epatite A (HAV) (6). Invece, in Paesi come l’India in cui entrambi i virus sono endemici, il virus dell’epatite A infetta maggiormente i bambini intorno ai 5 anni di età, mentre la maggior parte dei bambini e almeno la metà degli adulti sono sieronegativi per anticorpi anti-HEV. Le ragioni per cui le sieroprevalenze distribuite per classi d’età sono molto variabili, anche in Paesi con le stesse condizioni igienico sanitarie, rimangono ancora da chiarire (6).
Altri fattori di rischio sembrano essere anche le condizioni e gli stili di vita: studi condotti in Malesia hanno evidenziato una sostanziale differenza nella presenza di anticorpi anti-HEV in donatori di sangue sani provenienti da un contesto urbano (2,5%) e in persone provenienti da zone rurali (45-50%).
2.1.2 Caratteristiche dell’infezione nei Paesi industrializzati
In USA, Giappone ed Europa sono segnalati sempre più spesso casi sporadici di malattia in soggetti che non risultano aver viaggiato all’estero in zone a rischio. I ceppi isolati in tali episodi sono risultati geneticamente differenti rispetto a quelli isolati nei Paesi in via di sviluppo, facendo supporre che questi casi di malattia siano ascrivibili a virus endemici sul territorio e non ad infezioni contratte durante il soggiorno in Paesi a rischio. Numerosi studi sieroepidemiologici hanno inoltre rilevato una notevole prevalenza anticorpale anti-HEV (5-20%) nella popolazione sana di molti Paesi industrializzati, facendo ipotizzare un’elevata diffusione dell’infezione, seppur in genere a livello subclinico.
Casi autoctoni di epatite E sono stati segnalati nei seguenti Paesi industrializzati: UK, Olanda, Spagna, Germania, Francia, Italia, Austria, Grecia, Giappone, USA, Canada. La maggior parte dei casi riportati coinvolgono persone di ritorno da viaggi nelle cosiddette aree iper-endemiche, e la maggior parte degli studi disponibili in letteratura descrivono casi isolati e in piccolo numero.
In Italia, il virus dell’epatite E sembra essere responsabile di circa il 10% delle epatiti virali non-A, non-B, non-C (9). La maggior parte dei casi di malattia sono stati registrati in viaggiatori provenienti da aree in via di sviluppo considerate tradizionalmente endemiche. Tuttavia, nel 1999, una nuova variante di HEV è stata identificata nelle feci di un paziente che non aveva viaggiato né era venuto a contatto con individui di ritorno da tali zone. Questo stipite si è rivelato geneticamente diverso dai virus isolati in altri Paesi, mostrando una relativa analogia nucleotidica solo con i ceppi
americani del genotipo 3 ed è stato quindi considerato originario del territorio italiano.
Dal punto di vista sierologico, la presenza di anticorpi anti-HEV è stata rilevata in diverse regioni nel nostro paese , con prevalenze che oscillano tra l’1% ed il 5%. I valori di sieropositività più elevati sono stati riscontrati tra gli emodializzati, tossicodipendenti e persone positive ad altri marcatori di epatiti virali post-transfusionali. Inoltre è stato evidenziato un gradiente di sieropositività che va da Nord a Sud (9). La maggiore prevalenza nel Sud del Paese è probabilmente ascrivibile alla maggiore vicinanza di paesi in cui la malattia è considerata da molti anni endemica e all’elevato flusso migratorio da tali zone. Alcuni Autori ritengono che anche la pratica assai diffusa al Sud di consumare molluschi crudi possa essere un fattore di rischio aggiuntivo.
Per giustificare le frazioni di positività sierologiche e virologiche cosiddette autoctone, sono state avanzate diverse ipotesi, tutte correlate alla presenza di un serbatoio animale presente sul territorio . Le modalità di acquisizione dell’infezione comprendono la via alimentare , il contatto con animali infetti , l’esposizione ai reflui di allevamenti suinicoli infetti tramite la rete fognaria. Eccezione alla sporadicità dell’infezione sono i focolai di origine alimentare che sono stati riportati in Giappone. In questi casi infatti l’infezione non si è manifestata in maniera isolata, ma in forma di outbreaks su piccola scala, collegati all’assunzione dello stesso alimento contaminato (7).
Possibili fattori di rischio correlati all’acquisizione della malattia nei paesi industrializzati sono quindi tutte le condizioni associate al
contatto con animali serbatoio come l’esposizione professionale a suini infetti, sia in allevamento che al macello, il contatto domestico con suini tenuti come pet (10), l’assunzione di carne e organi infetti crudi o poco cotti.
2.2 EPIDEMIOLOGIA NELLE ALTRE SPECIE ANIMALI RECETTIVE ALL’INFEZIONE
Molte specie animali si sono rivelate recettive all’infezione, o per dimostrazione diretta della presenza del virus, o per la messa in evidenza di anticorpi anti-HEV nel siero (11).
La presenza di anticorpi anti-HEV è stata documentata in topi, polli, cani, vacche, pecore, capre, scimmie, manguste, cavie, suini, cinghiali, cervi, gatti, cavalli, uccelli, bufali. RNA di HEV è stato documentato in un numero ridotto di specie, rispetto a quelle in cui è stata riscontrata la presenza di anticorpi. Il primo ceppo virale animale è stato identificato nel suino -swHEV - , ma successivamente HEV è stato identificato in altre specie ed in particolare in cervi, cinghiali, manguste, cavalli. Recentemente, è stata dimostrata la presenza di HEV RNA in un elevato numero di specie selvatiche ospitate in una riserva zoo-like in Cina. Tra questi rientrano anche orsi e leopardi. HEV è stato identificato anche negli uccelli (12), ma i ceppi aviari differiscono molto da quelli dei mammiferi, quindi l’avian HEV è stato classificato separatamente.
2.2.1 HEV nel suino
Il primo ceppo di HEV suino è stato identificato nel 1997 negli Stati Uniti . Il virus si è rivelato simile ma distinto dai ceppi circolanti nell’uomo nello stesso territorio: il gene ORF2 aveva il 79-80% di identità a livello nucleotidico e il 90-92% a livello aminoacidico con
dei ceppi umani identificati nella stessa regione . Dal momento della sua scoperta, SwineHEV è stato identificato in suini provenienti da tutto il mondo , e si è sempre rivelato molto simile ai ceppi circolanti nell’uomo, tranne che in India, dove i ceppi circolanti nelle due specie sembrano essere distinti . Attualmente esistono numerosi studi che descrivono l’epidemiologia del virus dell’epatite E in suini allevati in diverse regioni del mondo, e di seguito alcuni di questi verranno descritti nel dettaglio (Tabella 1).
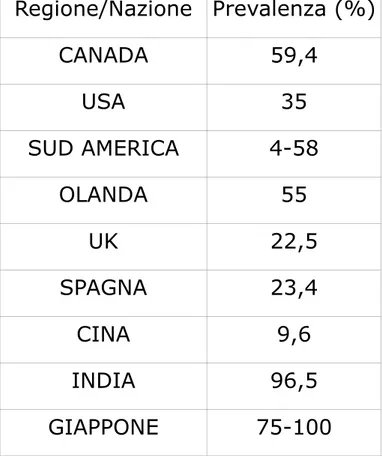
Tabella 1: Prevalenza di HEV in alcune nazioni o regioni, del mondo
Regione/Nazione Prevalenza (%) CANADA 59,4 USA 35 SUD AMERICA 4-58 OLANDA 55 UK 22,5 SPAGNA 23,4 CINA 9,6 INDIA 96,5 GIAPPONE 75-100
Gli studi condotti in Spagna sono di particolare interesse per le evidenze emerse a riguardo della prevalenza dell’infezione nelle diverse classi di età. Nel primo sono stati presi in considerazione i diversi stadi di produzione dei suini in allevamento, per identificare l’eventuale presenza di un’età a maggiore rischio di infezione. La
prevalenza complessiva dell’infezione era del 23,4%, e la fase più a rischio è risultata essere quella del primo mese di ingrasso (60% di positivi sui testati) e del magronaggio (41,7% di positività). Dato interessante è stato il rinvenimento di positività anche relativamente elevate (21,9%) nelle scrofette; si tratta infatti della prima testimonianza dell’infezione da HEV nei riproduttori . Nel secondo studio è stata presa in considerazione la sieroprevalenza in 41 allevamenti dislocati sul territorio spagnolo. Quaranta degli allevamenti testati erano positivi per la presenza di IgG anti-HEV (97,6%), e la classe di età con una più elevata positività anticorpale era quella delle scrofe anziane (60,8%), seguita da quella dei suinetti di 3-6 settimane (36,2%) .
2.2.2 HEV nel cinghiale
Riguardo alle popolazioni di in un recente studio condotto in Spagna è stata dimostrata una sieropositività del 42,7% e una prevalenza del virus del 19,6% in campioni di cinghiale provenienti da aree diverse. Interessante è anche il fatto che è stata riscontrata una prevalenza significativamente maggiore dell’RNA virale in campioni provenienti da animali in età giovanile (41,2%) piuttosto che in quelli di animali sub-adulti (10,5%) (13) .
Anche in Germania è stato condotto uno studio su campioni di siero di cinghiale raccolti negli anni 1995-1996. Il virus è stato rinvenuto nel 5,3% dei campioni; questa ridotta prevalenza dell’infezione rispetto allo studio spagnolo potrebbe essere dovuta alla prolungata conservazione dei campioni tedeschi prima di essere testati (14).
provenienti da un territorio limitato della regione Emilia-Romgna si è rilevata una prevalenza del 25%.
Studi recenti condotti nel Lazio, in Lombardia ed Emilia romagna riportano prevalenza sierologiche che vanno dal 12,8 al 72,5% e virologiche dal 6,3 al 7,3 %.
2.2.3 Avian HEV
Il primo ceppo di epatite E aviare fu identificato nel 2001 negli Stati Uniti (12) da polli con la sindrome di epatomegalia splenomegalia (HS syndrome). Questa malattia del pollame era una patologia emergente in Nord America, ma fino al 2001 l’agente eziologico non era stato individuato (24). Nel 2001 fu identificato un virus da campioni di bile di polli affetti da HS che, sequenziato, ha rivelato avere struttura genomica e sequenza nucleotidica simile a quella dell’HEV umano. I genoma dell’avian HEV è più corto di quello dei mammiferi di circa 600 paia di basi, avendo una lunghezza complessiva di 6,6 Kb (12). Anche se l’identità nucleotidica trai ceppi di epatite E aviari e umani è solo del 50% circa, parti dei domini funzionali della regione ORF1 della elicasi e della metiltransferasi sono relativamente conservati, supportando il fatto che anche l’HEV aviario sia un membro della famiglia Hepeviridae (2). In un recente studio sono stati testati campioni di diversi animali, sia con primers disegnati per la ricerca di ceppi aviari di HEV, che con primers per individuare i ceppi dei mammiferi. Nessun campione è risultato positivo per la ricerca di ceppi aviari, ma due campioni provenienti da uccelli si sono rivelati positivi per la ricerca di ceppi dei mammiferi . I ceppi identificati avevano il 98% di omologia nucletidica con quelli isolati dai mammiferi dello stesso
sito; gli Autori hanno pertanto ipotizzato una possibile trasmissione di HEV dai mammiferi agli uccelli, che sarebbero quindi recettivi all’infezione da HEV dei mammiferi .
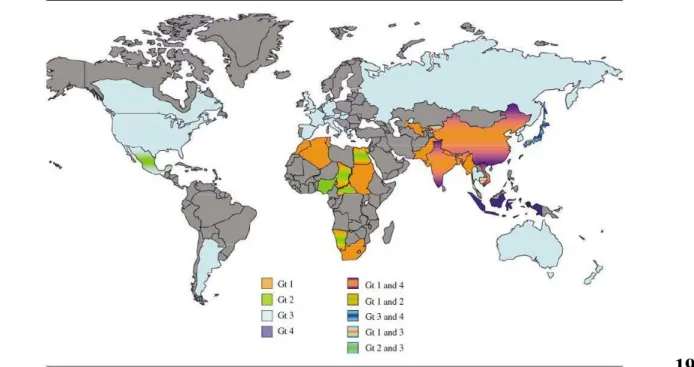
2.3 DISTRIBUZIONE GEOGRAFICA DEI DIVERSI GENOTIPI DI HEV
Il genotipo 1 può essere suddiviso in 5 sottotipi, che presentano diversi pattern di distribuzione geografica (15). Negli ultimi 20 anni i virus appartenenti a questo genotipo sono stati responsabili di focolai di malattia in India, e negli ultimi 15 anni la loro circolazione è stata dimostrata anche in Nepal (16,17) .
Il sottotipo 1a è stato isolato in epidemie nel Burma (18), ma anche in Pakistan e Giappone (19,20).
Il sottotipo 1b è prevalentemente riscontrato in Cina, dove è stato responsabile di focolai epidemici durante gli anni 1986-1988 (21) . Il sottotipo 1c è stato probabilmente introdotto dalla Cina in India e in Pakistan, dove si manifesta con focolai che precedono la stagione delle piogge (17) .
I sottotipi 1d e 1e sono stati identificati solo in Africa, il primo solo nel nord del Paese, mentre il secondo in tutto il continente (19) . Il genotipo 2 è stato identificato solo in poche aree, e per la prima volta in Messico (22).
Il genotipo 3a è stato identificato principalmente negli USA, in Giappone, ma anche in Corea e in Olanda. Sembra che ci siano due gruppi all’interno di questo sottotipo, uno indigeno degli Stati Uniti e l’altro del Giappone, e che da qui questi si siano poi diffusi nelle altre regioni (15).
tra loro: Argentina, Austria, Italia e Nuova Zelanda .
Diversamente dal genotipo 3, pur presentando lo stesso grado di variabilità, il genotipo 4 è stato rinvenuto solo in paesi asiatici (15). Le sequenze provenienti dalla Cina sono state classificate in 3 sottotipi, mentre le sequenze provenienti da Giappone ed India sono state raccolte ciascuna in un diverso sottotipo (15).
Particolare è il caso del Giappone: sul suo territorio circolano ceppi appartenenti al genotipo 3 e 4, e sono entrambi responsabili di casi di trasmissione all’uomo (15). A questo proposito esiste uno studio in bibliografia, in cui viene dimostrato che i ceppi sia del genotipo 3 che del 4 circolanti in Giappone formano dei clusters isolati e distinti rispetto a quelli circolanti in altri Paesi. In particolare, utilizzando la tecnica del molecoular tracing, viene avanzata l’ipotesi che il progenitore di questi ceppi sia stato introdotto nel Paese agli inizi del 1900, quando numerosi tipi di suini di razza Yorkshire vennero introdotti dallo UK in Giappone (23).In Figura 5 è mostrata la distribuzione mondiale dei genotipi di HEV.
CAPITOLO 3: PATOGENESI E SINTOMATOLOGIA
Per quanto riguarda la strategia di replicazione del virus, il modello che è stato proposto si basa su quello di altri virus a RNA a singolo filamento positivo ben caratterizzati (25). Le cellule in cui avviene la replicazione virale sono prevalentemente gli epatociti, ma anche altri siti di replicazione extraepatica sono stati identificati, come il piccolo intestino, la milza, i linfonodi e i monociti . Una volta penetrato in una cellula permissiva, l’ORF1 viene tradotto nella poliproteina, e grazie all’enzima RNA polimerasi RNA dipendente, probabilmente sfruttando gli enzimi della cellula ospite, l’RNA viene trascritto in un filamento ad orientamento negativo . Questo filamento negativo rappresenta la forma replicativa del virus e serve come stampo per la sintesi di altri filamenti di RNA positivo. Come avviene per gli alphavirus, in questo RNA intermedio si trova una sequenza che agisce come promoter subgenomico per la trascrizione di una proteina strutturale (o più di una) . Queste a loro volta vanno a circondare il genoma virale per la costituzione dei nuovi virioni infettanti. Nonostante queste conoscenze, le esatte modalità di questa strategia replicativa devono essere ulteriormente chiarite. I filamenti di RNA negativo si ritrovano solo negli epatociti e negli altri siti primari di replicazione, mentre nella bile e nel siero si ritrovano solo filamenti positivi di RNA. Recentemente (26) è stato dimostrato che il virus si localizza primariamente nel reticolo endoplasmatico della cellula ospite, che probabilmente è anche il sito primario di replicazione.
3.1 LA MALATTIA NEL SUINO 3.2.1 Patogenesi
Analogamente a quanto accade nell’uomo, si ritiene che la via principale di trasmissione sia quella oro-fecale; non ci sono evidenze di trasmissione verticale del virus (27;28). Sperimentalmente, è stato dimostrato la trasmissione dell’infezione da animali inoculati ad animali non infettati ma tenuti a contatto con quelli inoculati, confermando la contagiosità del virus (27; 28;29). L’infezione sperimentale per via oro-fecale è possibile, ma è difficile da riprodurre e richiede in genere esposizioni ripetute al virus; per questo motivo viene generalmente utilizzata la via endovenosa (27;29).
In analogia a quanto riportato per l’uomo, non è ancora chiaro in che modo HEV, una volta penetrato nel suino, raggiunga il fegato né quali siano i siti iniziali di replicazione. In animali infettati sperimentalmente per via endovenosa è possibile rilevare, tramite polymerase chain reaction (PCR) o ibridazione in-situ, RNA virale in numerosi tessuti extraepatici, anche in assenza di viremia, fino a 20-27 giorni post-infezione (p.i.). La forma replicativa del virus, a filamento negativo, oltre che nel fegato si ritrova soprattutto nel tratto intestinale e nei linfonodi (30;31)
Recentemente HEV è stato ricercato in organi e tessuti di suini affetti da diverse condizioni patologiche, ed è stato dimostrato che la bile è il campione più frequentemente positivo (76,9% degli animali esaminati erano positivi), seguita dai linfonodi mesenterici, fegato, feci e siero. La maggiore presenza del virus nella bile e nei linfonodi mesenterici sarebbe giustificata secondo gli Autori dal
fatto che questi organi rappresenterebbero i siti principali di replicazione virale (32).
La viremia ha una durata di circa 2 settimane mentre nelle feci è possibile ritrovare il virus fino a 3-4 settimane. La sieroconversione avviene 2-3 settimane p.i. (27;28;31;33). I tessuti in cui HEV replicherebbe prima e più a lungo (da 3 a 27 giorni p.i.) sono fegato, intestino tenue, colon e linfonodi (31;33). Queste osservazioni ed il fatto che in corso d’infezione sperimentale l’RNA virale sia riscontrabile nelle feci prima che nella bile ed in quantità circa 10 volte maggiore rispetto a quest’ultima, hanno fatto ipotizzare che, una volta penetrato per via oro-fecale e prima di indurre viremia, il virus replichi nell’intestino (27;28). Le indagini virologiche effettuate sul siero e/o sulle feci di animali in allevamento hanno dimostrato che RNA di HEV è rilevabile principalmente in animali di 2-5 mesi d’età, mentre in genere animali sotto i 2 mesi e sopra i 6-8 mesi di vita sono negativi (30;34;35;36). In seguito a queste rilevazioni e poiché si ritiene che l’immunità materna duri circa 2 mesi, si ipotizza che l’infezione naturale avvenga a circa 2-3 mesi d’età (29;33).
All’infezione segue la viremia, che dura circa 1-2 settimane e quindi l’escrezione del virus attraverso le feci per circa 3-4 settimane (3-5 mesi d’età), con successiva sieroconversione ed eliminazione del virus ad opera del sistema immunitario (27;28,33). L’infezione sarebbe quindi di breve durata e si autoestinguerebbe nel giro di poche settimane (27,33). Va tuttavia chiarito che in recenti lavori (37) il virus è stato rinvenuto anche in animali vicini all’età di macellazione (circa 6 mesi in Spagna, dove lo studio è stato
condotto), e nei riproduttori (animali con età superiore agli 8 mesi). Negli USA (38) inoltre il virus è stato rinvenuto nel fegato di animali macellati, quindi al di sopra dei 3-5 mesi di età indicati nei primi studi di infezione sperimentale. Se si tratti di un’infezione prolungata nel tempo o acquisita tardivamente o di reinfezione è ancora da chiarire, anche perché fino ad adesso è stata accertata la presenza di un solo sierotipo di HEV, ma questi dati potrebbero mettere in discussione tale acquisizione . Si potrebbe infatti trattare di infezioni acquisite più tardi nel corso della vita dell’animale, causate da ceppi verso cui la precedente immunità non era proteggente (37). Dal punto di vista sierologico, l’immunità passiva materna decresce a partire da 3-4 settimane e si estingue a 8-9 settimane di vita. E’ raro tuttavia trovare animali sieropositivi sotto 1-2 mesi d’età, questo probabilmente a causa della sensibilità non adeguata dei test sierologici (39). Dopo l’infezione gli animali sviluppano anticorpi nei confronti del virus montando prima una risposta anticorpale in cui prevalgono le IgM, seguite dopo una settimana circa da un innalzamento delle IgG. A questo punto le IgM decrescono rapidamente nel giro di 1-2 settimane mentre le IgG incrementano costantemente per diverse settimane. La sieroconversione avviene in seguito alla fase viremica intorno ai 3-4 mesi di vita (picco anticorpale a 4 mesi) e gli animali restano positivi fino a 5-6 mesi di vita, quando le IgG cominciano lentamente a decrescere (31;39;40).
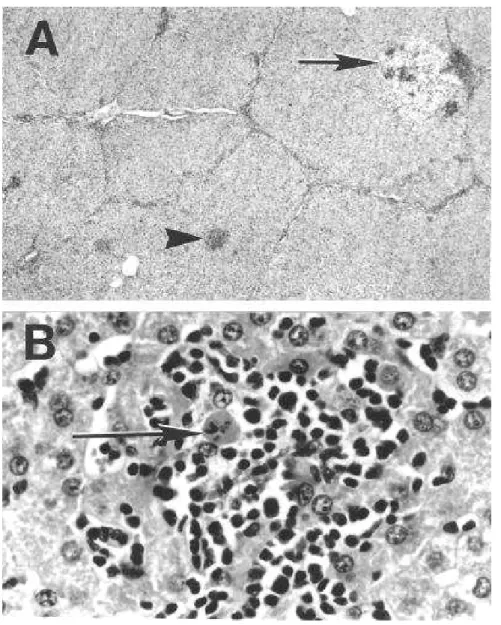
Le lesioni anatomopatologiche nel suino sono rilevabili nel fegato solo a livello istologico: si caratterizzano per la presenza di infiltrati linfoplasmocitari multifocali sinusoidali e periportali e di aree focali
a distribuzione irregolare di vacuolizzazione e necrosi epatocellulare (39) (Figura 6).
Recentemente sono stati condotti due studi sulle lesioni epatiche rilevabili in suini affetti da infezione da HEV (32). Nel primo studio il 57,7 % dei suini risultati positivi per HEV presentavano lesioni, esclusivamente microscopiche, di epatite. I suini risultati negativi per la ricerca del virus HEV presentavano lesioni di epatite solo nel 16,3 % dei casi (tale differenza è risultata statisticamente significativa). Le lesioni epatiche erano caratterizzate da una infiltrazione di entità variabile di mononucleati, infiltrati infiammatori principalmente localizzati nello spazio periportale oppure distribuiti variamente nel parenchima epatico (32). Nel secondo studio l’RNA di HEV è stato rinvenuto nel 55% dei fegati con lesioni epatiche, sempre a livello microscopico, caratterizzate da infiltrazioni focali di linfociti, plasmacellule e macrofagi nei sinusoidi epatici, e i qualche caso da infiltrazioni a livello portale.
Figura 6: Lesioni istopatologiche in fegati di suino (da Meng et al., 1997) A: Epatite linfoplasmocitaria multifocale (x10)
3.2.2 Caratteristiche cliniche
Il virus non sembra essere particolarmente patogeno per il suino domestico, né nei cinghiali. Lo studio della malattia in animali naturalmente infetti o inoculati sperimentalmente ha dimostrato che HEV provoca di regola infezioni subcliniche con segni di epatite rilevabile solo a livello istologico (28; 31; 33; 41). Dal punto di vista macroscopico, in animali infettati sperimentalmente è stato talvolta rilevato un ingrossamento dei linfonodi mesenterici ed epatici (33). Da rilevare che ceppi umani inoculati sperimentalmente nel suino sembrano, in genere, provocare infezioni più severe dal punto di vista delle alterazioni istologiche rispetto a quelle ottenute con i ceppi suini. Alcuni Autori (28; 31), hanno ipotizzato che il virus nel suino possa avere un comportamento analogo a quello dell’epatite A nell’uomo il quale è patogeno solo nei soggetti adulti e non nei giovani. Secondo questa ipotesi, nella maggior parte dei casi la malattia non si manifesterebbe in modo clinicamente evidente in quanto la maggioranza dei suini adulti sarebbe protetta dall’infezione per aver incontrato il virus in uno stadio giovanile di vita, sviluppando così un’immunità attiva proteggente (28). In uno studio recente, tuttavia, 12 scrofe sieronegative sono state inoculate per via endovenosa con un ceppo di HEV suino e durante il periodo della sperimentazione, non sono stati rilevati segni clinici di malattia né nelle madri né nei relativi feti. Una leggera epatite linfoistiocitaria è stata osservata in 4 delle 12 scrofe, mentre non è stato registrato alcun effetto sulla vitalità, taglia, peso alla nascita e incremento ponderale giornaliero della prole (42). Alcuni Autori ritengono che
HEV, di per sé scarsamente patogeno, possa però agire in sinergia con altri agenti virali come ad esempio il circovirus suino tipo 2 (PCV2), determinando così malattia (32;43). Indagini a tale proposito sono state condotte in due studi: nel primo (32), non è stato possibile identificare nessuna correlazione statistica tra la presenza del virus HEV e lesioni anatomopatologiche riconducibili ad infezione da PCV2, mentre nel secondo è stato possibile identificare una correlazione statistica. Se i due virus possano agire in sinergia o essere l’uno predisponente la presenza dell’altro resta comunque ancora da chiarire (43).
CAPITOLO 4: HEV COME AGENTE DI ZOONOSI
Dopo il riscontro di positività virologiche e sirologiche per HEV in alcune specie animali ed in particolare nei suidi, l’ipotesi che alcune specie animali possano rappresentare un serbatoio di infezione per l’uomo si è fatta sempre più fondata
Inoltre l’esistenza di un serbatoio animale di HEV consente di giustificare i casi cosiddetti autoctoni di infezione (15). A sostegno di questa ipotesi, esistono numerose prove del potenziale zoonosico del virus HEV, che verranno di seguito schematizzate.
4.1 POSSIBILITA’ DI INFEZIONE INTERSPECIFICA
Il primo tentativo di infezione interspecifica con ceppi di HEV è da fare risalire al 1990 (44) quando in Russia un suino fu infettato con un ceppo asiatico di HEV isolato da un paziente affetto dalla forma clinica della malattia. Gli Autori però non sequenziarono il ceppo identificato nel suino dopo l’infezione, rendendo impossibile stabilire se quello rinvenuto fosse il ceppo utilizzato per l’inoculo o se l’animale fosse già precedentemente infetto con un ceppo suino (44). Nel 1998 (27) alcuni Autori avevano tentato di infettare suini SPF con due ceppi di HEV di origine umana, uno appartenente al genotipo 1 (Sar-55) e uno al genotipo 2 (Mex-14). Anche in questo caso l’infezione non ebbe successo, probabilmente per la lontananza genetica di questi ceppi da quelli che normalmente infettano il suino (solo 75% di omologia nucleotidica).
Quando l’infezione è stata condotta invece con un ceppo appartenente al genotipo 3, e quindi più simile a quelli naturalmente circolanti nella popolazione suina (US-2), l’infezione ha avuto successo ed è stata confermata dalla messa in evidenza
nell’animale di una sequenza nucleotidica identica a quella del ceppo di inoculo (28). Per provare la trasmissibilità di ceppi suini all’uomo, preliminarmente è stata effettuata l’inoculazione di ceppi umani a primati non umani . E’ stato così dimostrato che le scimmie potevano essere infettate con ceppi umani appartenenti al genotipo 1, 2, 3 (45). Successivamente è stata dimostrata anche la possibilità di trasmissione di ceppi suini ai primati non umani. In particolare, un ceppo di probabile origine suina rinvenuto nella rete fognaria in prossimità di un macello è stato in grado di infettare primati non umani (46).
Tramite queste sperimentazioni è stato quindi possibile dimostrare che HEV, ed in particolare i ceppi appartenenti a genotipo 3, possono essere trasmessi dall’uomo al suino e viceversa, dando così la prima conferma all’ipotesi che gli animali, e specialmente il suino, giochino un ruolo importante nella trasmissione nell’infezione da HEV, soprattutto nei casi cosiddetti autoctoni di infezione.
4.2 OMOLOGIA TRA CEPPI UMANI E ANIMALI CIRCOLANTI SULLO STESSO TERRITORIO
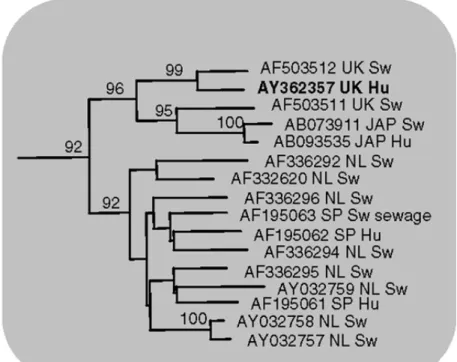
Ceppi di HEV identificati nell’uomo tendono a presentare elevate omologie nucleotidiche se circolanti nello stesso territorio, cioè tendono a formare dei clusters geografici (1) (Figura 7). I ceppi appartenenti ai genotipi 1 e 2 hanno tra loro minori differenze nucleotidiche rispetto a quelli appartenenti al genotipo 3 e 4 e questo sembra essere dovuto alla contemporanea circolazione di questi ultimi, sia negli animali che nell’uomo. Mentre i primi due genotipi (1 e 2) hanno una trasmissione di tipo oro fecale, per gli altri due genotipi (3 e 4), le vie di trasmissione si differenziano,
essendo implicate diverse specie animali; sembra quindi che, a simili pattern di trasmissione, corrispondono gli stessi gradi di divergenza genetica (15).
A dimostrazione della trasmissibilità di ceppi animali all’uomo stanno le numerose evidenze che ceppi animali e umani circolanti sullo stesso territorio manifestano gradi di omologia nucleotidica molto elevati, soprattutto se comparati con quelli circolanti su altri territori (47; 48). Sin dalla scoperta del primo ceppo animale di HEV negli USA, si era notato come questo presentasse elevate omologie nucleotidiche con i ceppi umani statunitensi (39). Successivamente questa osservazione è stata confermata a Taiwan (49) e in Giappone (1). Nel 2001 poi (50), i primi ceppi suini europei sono stati identificati in Olanda, e si sono dimostrati geneticamente vicini a ceppi di europei di HEV di origine umana. Nel 2002 è stato condotto uno studio su larga scala su suini provenienti da diverse regioni geografiche degli Stati Uniti (47), che ha evidenziato come i ceppi suini presentassero tra loro un’identità nucleotidica dell’88-100% e, se comparati con il prototipo umano di HEV, del 89-98%. Nel 2004 nel Regno Unito (34) sequenze di HEV identificate in suini, comparate con un ceppo di HEV identificato da un caso umano autoctono di epatite E in UK, si sono dimostrate molto vicine nell’albero filogenetico.
In Spagna, nel 2006 (32), in un’indagine condotta in suini presentanti diverse patologie, sono stati identificati ceppi con un identità nucleotidica variabile tra 85,7% e 100% tra loro, e che avevano un’identità dell’85,1-97,6% con due ceppi spagnoli di origine umana.
Anche in Italia (51;52), ceppi suini hanno rivelato una stretta omologia nucleotidica con ceppi identificati nell’uomo.
Tutti questi dati presenti in bibliografia sostengono l’ipotesi che vi sia una trasmissione interspecifica di ceppi di virus dell’epatite E appartenenti al genotipo 3 e 4 dagli animali, in particolare dal suino, all’uomo, e che quindi sussistano delle vie di trasmissione di tipo zoonotico.
4.3 HEV COME AGENTE DI ZOONOSI A TRASMISSIONE ALIMENTARE
Indagini effettuate in Giappone hanno rivelato che alla base di una parte considerevole dei casi di infezione umana da HEV che si sono verificati, vi è verosimilmente la trasmissione alimentare (15;53). In particolare, alcuni pazienti hanno manifestato una sintomatologia acuta, successivamente attribuita ad epatite E, dopo aver consumato carne cruda di cervo Sika; successivamente le indagini biomolecolari condotte sulla carne sospettata hanno rilevato, in quest’ultima, la presenza di sequenze virali aventi elevate percentuali di omologia con quelle rinvenute nelle persone che l’avevano mangiata (53;54). In uno di questi episodi di trasmissione alimentare, un ceppo di HEV geneticamente identico a quello identificato nei casi umani è stato ritrovato nella carne di cervo Sika implicata nel focolaio umano ed ancora conservata nel congelatore dei pazienti (53). In aggiunta a ciò, uno studio successivo ha messo in evidenza, sempre in Giappone, che consumatori di carne di selvaggina cacciata cruda presentavano valori di sieroprevalenza più elevate rispetto a gruppi di controllo (7). Episodi di malattia si sono verificati anche dopo il consumo di carne di cinghiale alla griglia o bollita (55) e, più frequentemente, di fegato crudo di cinghiale (56). Anche in queste occasioni, le sequenze virali identificate nel siero dei pazienti infetti avevano presentato elevate percentuali di omologia con quelle presenti nella carne che avevano mangiato prima dell’inizio dei sintomi (55).
Anche il consumo di fegato di suino è stato implicato nella trasmissione dell’infezione dopo che, una serie di casi di epatite E,
si erano verificati in persone che avevano mangiato del fegato di maiale grigliato non completamente cotto. In seguito a questi episodi sono state effettuate delle indagini anche in alcune macellerie giapponesi e l’1,9% dei fegati in vendita analizzati è risultato positivo alla ricerca del virus (57). In un recente studio condotto negli USA l’11% dei fegati esposti per la vendita in un supermercato era positivo per la presenza di HEV. Omogenati di questi fegati sono stati successivamente somministrati a suini SPF, che si sono infettati con i ceppi identificati negli organi, a dimostrazione del fatto che HEV non solo era presente, ma anche infettante. Sottoposti a diverse tipologie di cottura, è stato inoltre dimostrato che fegati riscaldati a 56°C per 1 ora contenevano ancora materiale infettante. A temperature di 191°C (temperatura interna 71°C) per 5 ' o in acqua bollente per 5 ' il virus sembra invece inattivarsi, dal momento che i suini inoculati con tale materiale non si sono infettati (38).
Recentemente, anche in Olanda è stato riportato un caso di possibile trasmissione alimentare di HEV collegato all’assunzione di carne di suino poco cotta, anche se risultati definitivi non sono stati prodotti dagli Autori (58;59).
Nel sud-est dell’Ungheria sono stati condotti studi che hanno messo in evidenza una maggiore incidenza di casi di HEV in persone con l’abitudine di ingerire salsicce di suino preparate in casa, ma non è stata riportata l’evidenziazione diretta del virus nei prodotti carnei in questione (60;61).
Anche in Texas si è tentato di collegare un caso di epatite autoctona umana al consumo di carne poco cotta, ma indagini condotte sulla
carne avanzata hanno prodotto risultati negativi (62).
Nel suino è stato dimostrato che HEV replica soprattutto nel fegato e nel tratto intestinale (63); l’indagine svolta su fegati di maiale venduti in alcune macellerie di Hokkaido ha rilevato, in questi, la presenza di elevate cariche virali (56). Il muscolo invece non sembra essere una sede elettiva della replicazione virale, anche se è comunque possibile che possa pervenire in queste sedi durante la viremia (7). Per quanto riguarda le specie selvatiche, non ci sono ancora notizie certe di quale sia la dinamica replicativa del virus, anche se l’elevata carica virale rilevata nel muscolo delle specie testate (105 copie per grammo), fa pensare ad una possibile replicazione del virus anche a questo livello (53).
Sembra che anche l’ingestione di molluschi filtratori crudi rappresenti un fattore di rischio per l’acquisizione dell’infezione, come dimostrato delle più elevate sieroprevalenze riscontrate in persone con questa abitudine alimentare (64), o che semplicemente vivono in zone costiere (65).
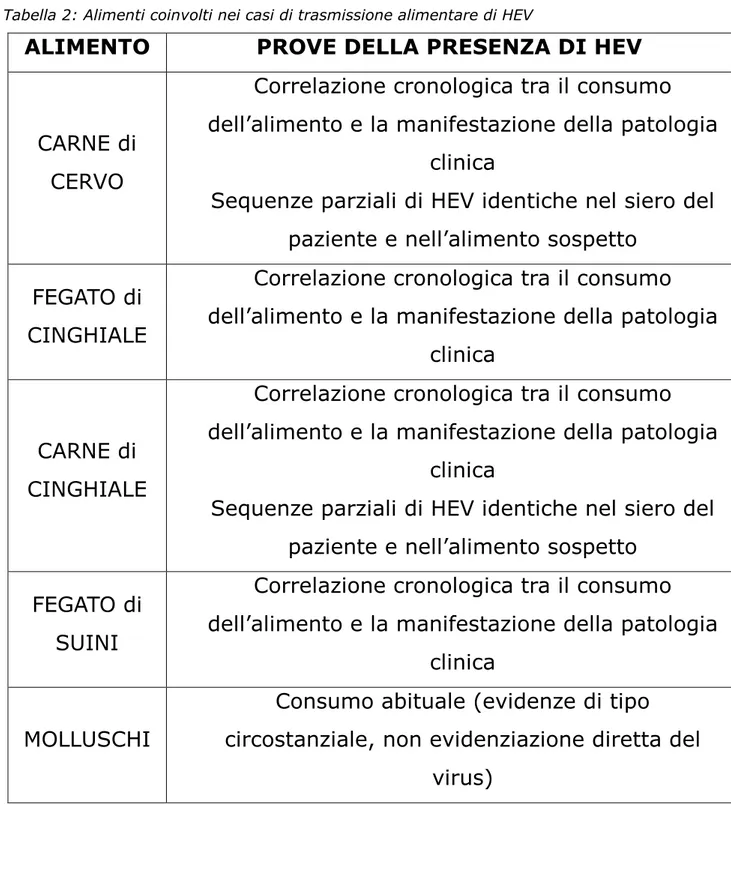
In Tabella 2 vengono schematizzati gli alimenti fino ad oggi coinvolti in casi di trasmissione alimentare di HEV, e i riscontri virologici ottenuti da ciascun tipo di alimento.
Tabella 2: Alimenti coinvolti nei casi di trasmissione alimentare di HEV
ALIMENTO PROVE DELLA PRESENZA DI HEV
CARNE di CERVO
Correlazione cronologica tra il consumo dell’alimento e la manifestazione della patologia
clinica
Sequenze parziali di HEV identiche nel siero del paziente e nell’alimento sospetto
FEGATO di CINGHIALE
Correlazione cronologica tra il consumo dell’alimento e la manifestazione della patologia
clinica
CARNE di CINGHIALE
Correlazione cronologica tra il consumo dell’alimento e la manifestazione della patologia
clinica
Sequenze parziali di HEV identiche nel siero del paziente e nell’alimento sospetto
FEGATO di SUINI
Correlazione cronologica tra il consumo dell’alimento e la manifestazione della patologia
clinica
MOLLUSCHI
Consumo abituale (evidenze di tipo
circostanziale, non evidenziazione diretta del virus)
PARTE II:
INDAGINI SIEROLOGICHE,
BIOMOLECOLARI E PATOLOGICHE
SULLA POPOLAZIONE DI
CINGHIALI
(Sus scrofa) DELLA PROVINCIA
DI PISA
CAPITOLO 5: MATERIALI E METODI 5.1 CALCOLO DELLA NUMEROSITÀ CAMPIONARIA
Per il calcolo della numerosità campionaria sono stati raccolti dalla bibliografia i dati di prevalenza di HEV nelle popolazioni di cinghiali, sia in Italia che in altre nazioni. Sulla base di questi dati, e considerando anche le disponibilità dei cacciatori che collaboravano alla raccolta dei sieri, si è deciso di stabilire una sieroprevalenza attesa del 10% (0,1)- seppur inferiore alle prevalenze riscontrate in altre regioni-, un livello di confidenza del 95% ed una precisione del 10%.
La consistenza della popolazione di cinghiali nella provincia di Pisa, derivata da dati indiretti cinegenetici, è stata stimata in circa 6000 unità, secondo il piano annuale di gestione della provincia di Pisa. Nel grafico 1 sono mostrati i capi abbattuti nella provincia di Pisa nel periodo 2005-2011. (66)
Per il calcolo della numerosità campionaria si è utilizzata la formula seguente:
n=
Il numero di soggetti ritenuti necessari allo studio è risultato pari a 34. Essendo il campionamento effettuato su dei cluster di popolazione si è moltiplicato per un fattore di design effect pari a due (67;68). Il campione totale è risultato pari a 68 campioni. Essendo il campione inferiore al 5% della popolazione stimata non si è ritenuto necessario applicare la correzione per popolazioni finite.
5.2 ANIMALI
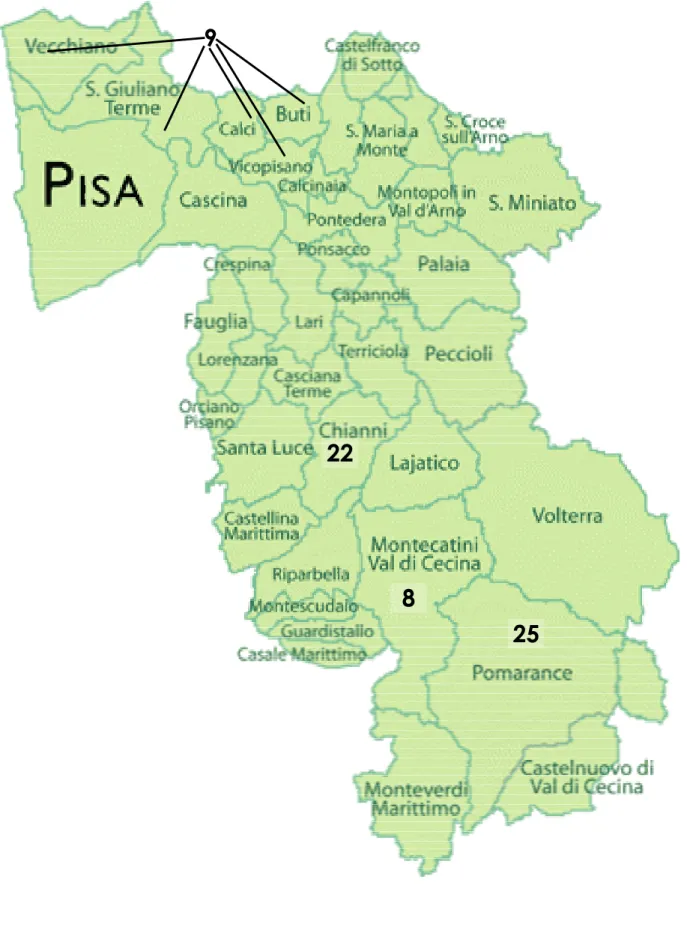
I campioni raccolti derivano da un campionamento effettuato durante la stagione venatoria 2011-2012 in diverse zone della provincia di Pisa. Nella Figura 8 è mostrata la cartina politica della provincia con indicati i comuni e il relativo numero di campioni. Ogni soggetto è stato classificato in base al sesso ed alla classe di età. É stato deciso di utilizzare tre classi di età: giovane, subadulto e adulto. Ad ogni zona di prelievo è stato assegnato un codice alfanumerico con cui contrassegnare i soggetti. Sono stati prelevati un totale di 64 soggetti, 31 femmine e 26 maschi, 26 adulti e 26 subadulti. Dei 64 soggetti, 2 sono privi del campione di feci e sangue e 12 non sono stati classificati secondo la classe di età e sesso.Da ogni soggetto sono stati raccolti: un campione di feci, un campione di sangue intero, e due campioni di fegato, uno da conservare a –20°C per la biologia molecolare ed una porzione
d2
Figura 8 Cartina politica della Provincia Pisana con i comuni e i relativi campioni prelevati
25
8
22
9
rappresentativa del fegato di circa 3x3cm, fissata in formalina
tamponata al 10% (pH 7,4) per le successive indagini istopatologiche.
5.3 METODICHE DI DIAGNOSI DIRETTA 5.3.1 Nested-PCR su pool di campioni fecali 5.3.1.1 Estrazione del RNA virale
I campioni fecali sono stati suddivisi in 19 pool per luogo di prelievo e classe di età. In tabella 3 sono riportati i soggetti componenti i pool con luogo di prelievo classe di età e sesso.
Tabella 3:Riepilogo dei soggetti componenti i pool: identificazione, zona di prelievo, classe di età e sesso.
Ogni pool è stato costituito con circa 3 grammi di feci, composti da parti uguali di ogni campione, pesate con una bilancia di precisione e raccolte in una provetta da 50 ml. Ad ogni provetta è stato aggiunto il 10% P/V di PBS 1X e delle sfere di vetro per facilitare
Pool Soggetto Zona di prelievo Classe di età Sesso Pool Soggetto Zona di prelievo Classe di età Sesso
L1 Pomarance S F P1 Comuni dei Monti Pisani S M L2 Pomarance S F P2 Comuni dei Monti Pisani A M L3 Pomarance S M P3 Comuni dei Monti Pisani
L4 Pomarance A F P4 Comuni dei Monti Pisani SR2 Pomarance A F P5 Comuni dei Monti Pisani SR3 Pomarance A F P6 Comuni dei Monti Pisani SR5 Pomarance A F P7 Comuni dei Monti Pisani SR6 Pomarance A F P8 Comuni dei Monti Pisani SR7 Pomarance A F P9 Comuni dei Monti Pisani
SR8 Pomarance A F S27 Chianni A F SR9 Pomarance A F S28 Chianni A F SR22 Pomarance A F S30 Chianni A F SR1 Pomarance A M S31 Chianni A F SR21 Pomarance A M S32 Chianni A F SR25 Pomarance A M S13 Chianni A M SR4 Pomarance S F S14 Chianni A M SR12 Pomarance S F S25 Chianni A M SR13 Pomarance S F S26 Chianni A M SR14 Pomarance S F S29 Chianni A M SR23 Pomarance S F S12 Chianni G M SR24 Pomarance S F S15 Chianni G M SR26 Pomarance S F S24 Chianni S F SR27 Pomarance S F S11 Chianni S M SR10 Pomarance S M S16 Chianni S M SR11 Pomarance S M S17 Chianni S M M1 Montecatini Val di Cecina M S18 Chianni S M M2 Montecatini Val di Cecina F S19 Chianni S M M3 Montecatini Val di Cecina F S20 Chianni S M M4 Montecatini Val di Cecina F S21 Chianni S M M5 Montecatini Val di Cecina F S22 Chianni S M S1 Montecatini Val di Cecina A M S23 Chianni S M S2 Montecatini Val di Cecina A F
S3 Montecatini Val di Cecina A F
6 16 17 7 18 8 19 9 4 14 5 15 1 10 11 2 12 3 13
l'omogeneneizzazione. Le provette sono state quindi agitate su un Vortex e centrifugate per 20 ' a 3000 rpm a 4°C. Il surnatante è stato quindi raccolto in provette da 2 ml e centrifugato per 10 ' a 14000 rpm. Il surnatante è stato quindi trasferito su altre provette da 2 ml e stoccato a -20°C.
5.3.1.1.1 Estrazione RNA
Per l'estrazione del RNA è stato utilizzato il kit QIAGEN ® QIAamp ® Viral RNA utilizzando lo spin protocol e effettuando l'eluizione con due passaggi di buffer AVE, in modo da incrementare fino al 10% la raccolta di RNA, come consigliato dal manuale di uso del kit. Questo kit sfrutta le proprietà selettive chelanti di una membrana di gel silicato su cui il materiale è fatto correre grazie ad una centrifuga. Due aliquote per ogni pool sono state trattate separatamente fino al momento del passaggio sulla membrana, dove sono state riunite per assicurare una maggiore resa di RNA.
5.3.1.1.2 Retrotrascrizione
Per la retrotrascrizione del RNA in cDNA, è stato utilizzato il kit QIAGEN ® QuantiTect ® Reverse transcription. La retrotrascrizione comprende una prima fase di eliminazione di DNA genomico, appartenente a cellule presenti nel campione e eventualmente estratto insieme al RNA, ed una seconda fase di retrotrascrizione vera e propria.
5.3.1.2 Prima PCR
È stata effettuata una prima PCR utilizzando il kit QIAGEN ® HotStartTaq DNA Polymerase con i primer esterni 3156 e 3157 secondo il protocollo proposto da Meng (47). In tabella 4 e 5 sono mostrate le sequenze dei primers e la loro posizione nel genoma di
HEV; e le caratteristiche dei cicli di amplificazione. I prodotti della Nested PCR sono stati fatti correre su gel al 2% di agarosio utilizzando il marker MassRuler™ Low Range DNA Ladder, ready-to-use fermentas.
Tabella 4: Sequenze dei primers utilizzati per la prima PCR e loro posizione nel genoma di HEV
Primer Sequenza Posizione nel genoma
3156-EF Senso AAYTATGCMCAGTACCGGGTTG 5687-5708
3157-ER Antisenso
CCCTTATCCTGCTGAGCATTCTC 6395-6417
Tabella 5: Valori di temperatura, tempo e numero di cicli
Temperatura (°C) Tempo (minuti) Numero di cicli
Denaturazione 95 15 1 Denaturazione 94 0,5 Annealing 60 0,5 Estensione 72 1 40 Estensione finale 72 10 1 4 ∞ ∞ 5.3.1.2 Nested-PCR
La nested-PCR è stata condotta utilizzando lo stesso kit della precedente ma impiegando dei primer interni, 3158 e 3159 (37, 47). In tabella 6 e 7 sono mostrate le sequenze dei primers, la loro posizione nel genoma di HEV; e le caratteristiche dei cicli di amplificazione. Il prodotto atteso della nested è un peptide di 347 bp.
Tabella 6: Sequenze dei primers utilizzati per la Nested-PCR e loro posizione nel genoma di HEV
Primer Sequenza Posizione nel genoma
3158-EF Sense GTYATGYTYYGCATACATGGCT 5972-5993
Tabella 7: Valori di temperatura, tempo e numero di cicli della nested PCR
Temperatura (°C) Tempo (minuti) Numero di cicli
Denaturazione 95 15 1 Denaturazione 94 0,5 Annealing 55 0,5 Estensione 72 1 40 Estensione finale 72 10 1 4 ∞ ∞
5.3.2 Nested-PCR su campioni singoli
I campioni componenti i pool risultati positivi alla prima Nested-PCR, sono stati analizzati singolarmente secondo il protocollo sopradescritto. Per i soggetti per i quali il quantitativo di feci è risultato insufficiente per una seconda analisi, sono stati utilizzati i campioni di fegato. L’estrazione dell’RNA è stata preceduta da un trattamento con il QIAGEN Tyssue-Liser ®.
5.4 METODICHE DI DIAGNOSI INDIRETTA 5.4.1 ELISA su siero
I campioni di sieri di tutti i soggetti sono stati analizzati con un ELISA indiretta utilizzando il kit HEV Ab della AXIOM Diagnostic ®. Il kit è formato da strip di polistirene preadsorbite con l'antigene HEV corrispondente alla proteina strutturale codificata dal ORF-2 del virus nativo. I campioni sono stati classificati come positivi, negativi o dubbi, secondo i criteri di interpretazione fornite dalla ditta.
5.4.2 Analisi istologica su campioni di fegato
positivi in PCR sono stati condotti esami istologici. Porzioni rappresentative dei fegati campionati sono state fissate in formalina tamponata pH 7,4 ed incluse in paraffina. Sezioni seriali di 4 µm sono state colorate con Ematossilina-Eosina per le visioni generali.
5.5 ANALISI STATISTICA
I dati raccolti sono stati elaborati per stimare la prevalenza sierologica e virologica del HEV con i relativi intervalli di confidenza al 95%. Il test esatto di Fisher è stato utilizzato per verificare se vi fossero differenze significative all’interno di sesso e classe di età per quanto riguarda i risultati sierologici e virologici, utilizzando il sito http://www.quantitativeskills.com/sisa/statistics/fisher.htm. Inoltre è stato calcolato il valore predittivo positivo dei quadri patologici riscontrati rispetto alla positività.
CAPITOLO 6: RISULTATI 6.1 METODICHE DI DIAGNOSI DIRETTA 6.1.1 Nested-PCR su pool di campioni fecali
Dei 19 pool di feci 4 sono risultati positivi alla Nested-PCR. In figura 9 è mostrata una foto del gel elettroforetico.
Figura 9: Immagine del gel elettroforetico dei prodotti della Nested-PCR di pool di campioni fecali
Due dei pool provenivano dal comune di Pomarance ed erano formati tutti da soggetti subadulti, 2 maschi e 8 femmine; gli altri due pool erano provenienti da Chianni, uno formato da tre femmine adulte e l'altro da due giovani maschi.
In tabella 8 sono mostrati i dati relativi ai soggetti dei pool positivi.
Tabella 8 Dati relativi ai soggetti componenti i pool risulati positivi alla Nested-PCR
6.1.2 Nested-PCR su pool di pool di campioni singoli
Dei 12 soggetti dei 4 pool risultati positivi alla Nested-PCR 6 sono risultati positivi alle Nested-PCR. Quattro sono subadulti, 3 maschi e 1 femmina e due femmine adulte.
In figura 10 è mostrata una foto del gel elettroforetico.
Figura 10: Immagine del gel elettroforetico dei prodotti della Nested-PCR di campioni fecali di singoli soggetti
Marker
Pool Soggetto Zona di prelievo Età Sesso
POOL 5 SR4 S F SR12 S F SR13 S F SR14 S F POOL 7 SR27 S F SR10 S M SR11 S M POOL 13 S27 Chianni A F S28 Chianni A F S30 Chianni A F POOL 16 S12 Chianni G M S15 Chianni G M Serrazzano Serrazzano Serrazzano Serrazzano Serrazzano Serrazzano Serrazzano
6.2 METODICHE DI DIAGNOSI INDIRETTA 6.2.1 ELISA su siero
Dei 60 campioni esaminati 32 sono risultati positivi, due dubbi e 26 negativi. In tabella 9 sono riassunti i risultati delle tre prove effettuate.
Tabella 9: Riepilogo risultati delle prove effettuate
Zona di prelievo Età Sesso PCR POOL PCR SINGOLO ELISA AXIOM
L1 Pomarance S F Negativo Positivo
L2 Pomarance S F Negativo Campione assente
L3 Pomarance S M Negativo Campione assente
L4 Pomarance A F Negativo Positivo
SR2 Pomarance A F Negativo Positivo
SR3 Pomarance A F Negativo Positivo
SR5 Pomarance A F Negativo Positivo
SR6 Pomarance A F Negativo Positivo
SR7 Pomarance A F Negativo Positivo
SR8 Pomarance A F Negativo Positivo
SR9 Pomarance A F Negativo Negativo
SR22 Pomarance A F Negativo Positivo
SR1 Pomarance A M Negativo Positivo
SR21 Pomarance A M Negativo Dubbio
SR25 Pomarance A M Negativo Positivo
SR4 Pomarance S F Positivo Negativo Negativo
SR12 Pomarance S F Positivo Negativo Positivo
SR13 Pomarance S F Positivo Positivo Positivo
SR14 Pomarance S F Positivo Negativo Negativo
SR23 Pomarance S F Negativo Negativo
SR24 Pomarance S F Negativo Positivo
SR26 Pomarance S F Negativo Negativo
SR27 Pomarance S F Positivo Negativo Negativo
SR10 Pomarance S M Positivo Positivo Negativo
SR11 Pomarance S M Positivo Negativo Positivo
M1 Montecatini Val di Cecina M Negativo Positivo
M2 Montecatini Val di Cecina F Negativo Positivo
M3 Montecatini Val di Cecina F Negativo Positivo
M4 Montecatini Val di Cecina F Negativo Negativo
M5 Montecatini Val di Cecina F Negativo Negativo
S1 Montecatini Val di Cecina A M Negativo Positivo
S2 Montecatini Val di Cecina A F Negativo Negativo
S3 Montecatini Val di Cecina A F Negativo Positivo
P1 Comuni dei Monti Pisani S M Negativo Negativo
P2 Comuni dei Monti Pisani A M Negativo Positivo
P3 Comuni dei Monti Pisani Negativo Negativo
P4 Comuni dei Monti Pisani Negativo Campione assente
P5 Comuni dei Monti Pisani Negativo Negativo
P6 Comuni dei Monti Pisani Negativo Negativo
P7 Comuni dei Monti Pisani Negativo Negativo
P8 Comuni dei Monti Pisani Negativo Dubbio
P9 Comuni dei Monti Pisani Negativo Positivo
S27 Chianni A F Positivo Negativo Positivo
S28 Chianni A F Positivo Positivo Positivo
S30 Chianni A F Positivo Positivo Positivo
S31 Chianni A F Negativo Positivo
S32 Chianni A F Negativo Positivo
S13 Chianni A M Negativo Positivo
S14 Chianni A M Negativo Positivo
S25 Chianni A M Negativo Negativo
S26 Chianni A M Negativo Negativo
S29 Chianni A M Negativo Positivo
S12 Chianni G M Positivo Positivo Negativo
S15 Chianni G M Positivo Positivo Positivo
S24 Chianni S F Negativo Negativo
S11 Chianni S M Negativo Campione assente
S16 Chianni S M Negativo Negativo
S17 Chianni S M Negativo Negativo
S18 Chianni S M Negativo Negativo
S19 Chianni S M Negativo Negativo
S20 Chianni S M Negativo Negativo
S21 Chianni S M Negativo Positivo
S22 Chianni S M Negativo Negativo
S23 Chianni S M Negativo Negativo
POOL 17 POOL18 POOL 19 POOL 13 POOL 14 POOL 15 POOL 16 POOL 9 POOL 10 POOL 11 POOL 12 POOL 5 POOL 6 POOL 7 POOL 8 POOL 1 POOL2 POOL3 POOL 4