A
Al
lm
m
a
a
M
M
at
a
te
er
r
S
St
tu
ud
di
io
or
ru
um
m
–
–
U
U
ni
n
iv
v
er
e
rs
si
it
tà
à
d
di
i
B
Bo
ol
lo
og
gn
na
a
DOTTORATO DI RICERCA
in
Colture erbacee, Genetica agraria e Sistemi Agroterritoriali
Ciclo XXI
AGR/07 GENETICA AGRARIA
IDENTIFICAZIONE DI MUTANTI DI INTERESSE
AGRONOMICO IN ORZO MEDIANTE APPROCCI
DI GENETICA DIRETTA E INVERSA
Presentata da: Dott. Riccardo Bovina
Coordinatore Dottorato Relatore Prof Giovanni Dinelli Prof.ssa Maria Corinna Saguineti
INDICE DEGLI ARGOMENTI
I INTRODUZIONE
11.
MUTAGENESI……….
21.1Materiale vegetale per il trattamento mutageno………. 5
1.2Fattoti che influenzano la mutagenesi……….. 6
1.3La mutagenesi fisica………... 7
1.4Mutagenesi chimica………... 8
1.4.1 L’azide sodica………. 9
1.5Ruolo della mutagenesi nel miglioramento genetico………... 13
2.
LA GENETICA INVERSA………
162.1Strategie di genetica inversa………. 18
2.2Mutagenesi mirata in genetica inversa……… 18
2.2.1 Gene targeting………. 19
2.2.2 Silenziamento genico……….. 19
2.2.3 Nucleasi “zinc-finger”……… 20
2.3Mutagenesi casuale in genetica inversa……… 21
2.3.1 Mutagenesi inserzionale……….. 21
2.3.2 Mutagenesi fisica: Deletagene……… 24
2.4Mutagenesi chimica in genetica inversa: il TILLING……… 25
2.4.1 Produzione di una popolazione mutagenizzata………... 28
2.4.1.1Scelta della specie……… 28
2.4.1.2Scelta del mutageno chimico………... 29
2.4.1.3Trattamento mutageno………. 31
2.4.2 Scelta del gene bersaglio……… 33
2.4.3 Analisi molecolare TILLING………. 34
2.4.3.1Pooling e formazione dell’eteroduplex……… 34
2.4.3.3Visualizzazione dei frammenti……… 37
2.4.3.4Sequenziamento e analisi delle mutazioni………... 39
2.5Dall’analisi molecolare al fenotipo………... 40
2.6Ecotilling………. 42
2.7Esempi di applicazione della strategia TILLING………... 44
2.7.1 Risorse TILLING in orzo……… 46
2.8 Utilizzo del TILLING per il miglioramento genetico………. 47
3.
L’ORZO………...
493.1Domesticazione della specie……….. 49
3.2Botanica e tassonomia………... 50
3.3Miglioramento genetico……… 50
3.4Impiego ed importanza economica……….. 52
3.5Caratteristiche morfologiche……… 53
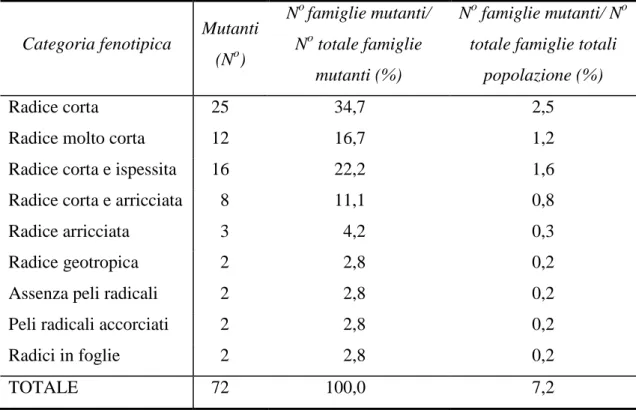
3.6Importanza ed analisi dell’apparato radicale in orzo……… 58
3.6.1 Mutanti della radice e loro utilizzo……… 58
3.6.2 Identificazione di Quantitative Trait Locus (QTL) e utilizzo in programmi di selezione assistita……… 61
3.7Esigenze ambientali e sostenibilità………. 61
II OBIETTIVI
64III MATERIALI E METODI
67 1. La risorsa TILLMore………... 682. Allevamento ed analisi fenotipica delle famiglie M3……….. 69
3. Campionamento ed estrazione del DNA ……… 70
4. Preparazione del DNA tramite diluizione e pooling ………. 71
5. Aspetti molecolari della procedura TILLING………... 71
5.1 Selezione dei geni e progettazione dei primer……… 71
5.3 Digestione e purificazione……….. 76
5.4 Separazione elettroforetica………. 77
5.5 Sequenziamento……….. 78
6. Analisi delle famiglie segreganti M4 con marcatori CAPS………... 79
7. Analisi bioinformatica delle proteine mutanti………... 82
8. Ricerca ed osservazione dei mutanti radicali ………... 83
9. Analisi dei mutanti allo stereo microscopio………... 84
IV RISULTATI E DISCUSSIONE
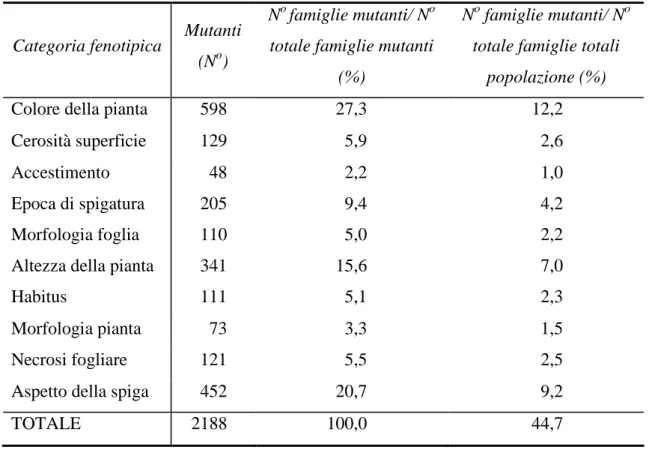
86 1. Frequenza e descrizione dei mutanti M3 ………... 872. Frequenza e descrizione dei mutanti radicali……… 97
3. Identificazione di mutanti per geni coinvolti nello sviluppo e architettura radicale ………... 100
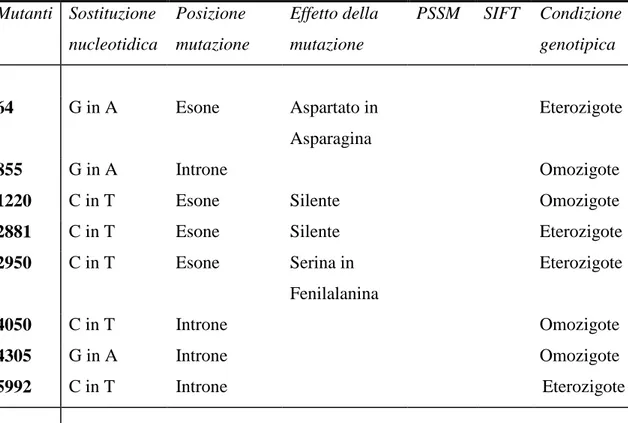
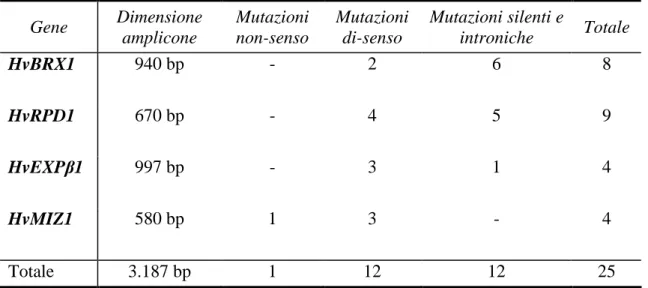
4. Analisi delle mutazioni identificate ……… 106
4.1 Analisi della direzione delle mutazioni……….. 106
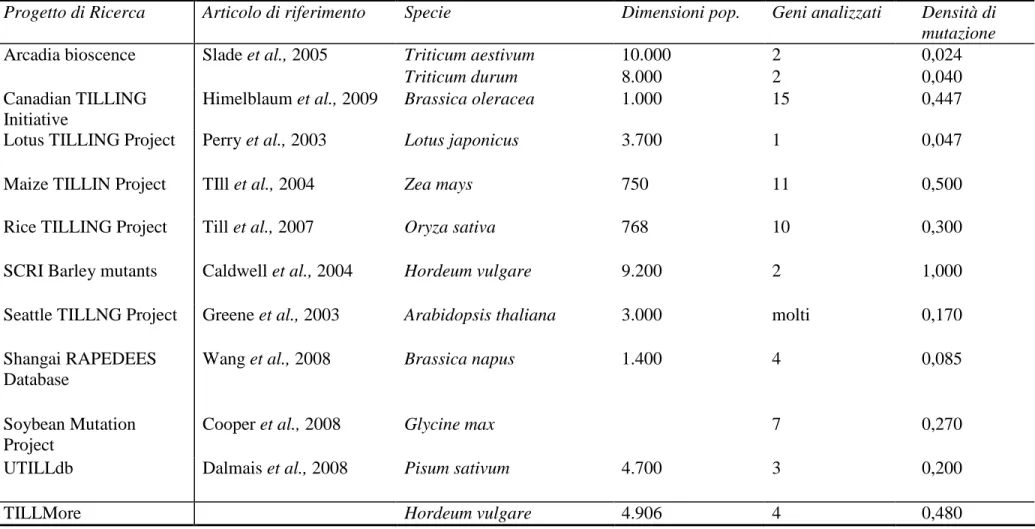
4.2 Analisi della densità di mutazione………. 107
5. Analisi CAPS delle famiglie mutanti ………. 110
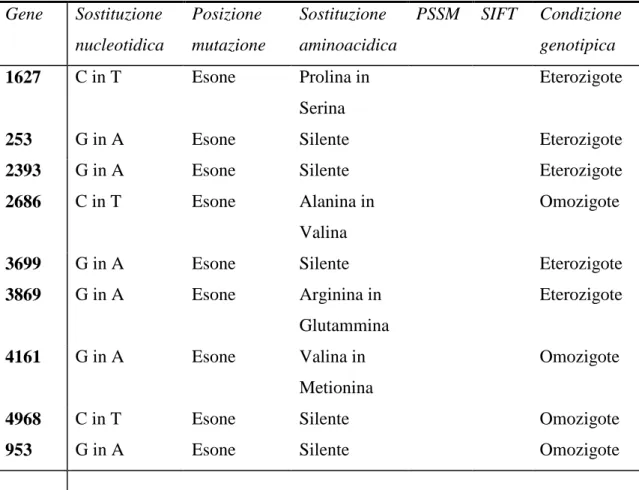
6. Predizione dell’effetto delle mutazioni del gene HvEXPβ1 mediante analisi bioinformatica……….. 111
7. Analisi fenotipica dei mutanti radicali………... 114
V CONCLUSIONI
116VI BIBLIOGRAFIA
121TABELLE
1451. LA MUTAGENESI
La mutagenesi può essere definita come l’insieme di tecniche che consentono di indurre mutazioni in un determinato organismo riproducendo così in modo artificiale ciò che avviene da sempre in natura; le mutazioni spontanee sono infatti un processo biologico fondamentale per la creazione della variabilità genetica presente nelle popolazioni naturali e, insieme alla ricombinazione genica, sono alla base dei fenomeni evolutivi.
Le mutazioni consistono in una alterazione del materiale genetico ereditario a diversi livelli:
• mutazioni genomiche: comportano alterazioni del numero cromosomico e sono distinte in aneuploidia o euploidia;
• mutazioni cromosomiche: comportano alterazioni della struttura dei cromosomi e sono distinte in delezioni, duplicazioni, inversioni e traslocazioni;
• mutazioni geniche: interessano una o poche paia di basi in un singolo locus genico (si parla perciò di mutazioni puntiformi) e sono distinte in inserzioni o delezioni di basi e sostituzioni; in quest’ultimo caso si distinguono transizioni (sostituzione di purina con purina o pirimidina con pirimidina) e transversioni (purina sostituita da pirimidina e viceversa).
Per quanto riguarda gli effetti dei vari tipi di mutazioni, quelle genomiche e cromosomiche possono essere deleterie e, nella maggior parte dei casi, comportano alterazioni molto più evidenti rispetto a quelle geniche; pertanto sono queste ultime le più utili ai fini di studi di genetica o per applicazioni nel miglioramento genetico.
La sostituzione di una base, quando interessa regioni geniche codificanti (esoni), comporta il cambiamento di una tripletta che codifica per un aminoacido della proteina con diversi effetti sulla funzionalità della proteina stessa; si distinguono infatti mutazioni silenti, in cui, per la degenerazione del codice genetico, il codone alterato codifica per lo stesso aminoacido; mutazioni neutre se si ha cambiamento di un
aminoacido tale però da non alterare la funzione proteica; mutazioni di-senso (missense mutation) in cui il codone modificato specifica un aminoacido diverso che altera in modo più o meno evidente, senza tuttavia sopprimerla, la funzionalità della proteina a seconda del sito mutato, e si possono originare quindi diverse varianti fenotipiche; mutazioni non-senso (nonsense mutation) quando si forma un codone di-stop che causa la terminazione prematura della sintesi proteica con perdita della sequenza a valle del sito mutato e produzione di una proteina troncata in genere non funzionale. Inoltre mutazioni a livello dei siti di splicing nelle giunzioni tra introni ed esoni possono causare la perdita di tali siti o la produzione di nuovi, con conseguente produzione di proteine troncate.
Le inserzioni o delezioni di una singola coppia di basi sono dette anche mutazioni “frameshift” perché comportano uno slittamento del modulo di lettura delle triplette causando un’alterazione della sequenza aminoacidica a valle del sito mutato con perdita, in genere, della funzionalità della proteina codificata.
Talvolta si possono verificare anche retromutazioni che comportano la reversione della mutazione originale (detta anche “forward mutation”) che inattiva il gene selvatico, ripristinando la funzione della proteina; le retromutazioni richiedono però condizioni più specifiche e si verificano quindi con frequenze inferiori di circa 10 volte rispetto alle “forward mutation”.
In generale quindi le mutazioni possono provocare diversi effetti (letali, favorevoli o sfavorevoli) e, negli ultimi due casi, possono pertanto essere trasmesse alla progenie fissandosi nel corso delle successive generazioni oppure essere eliminate per effetto della selezione naturale.
Nell’ambito delle specie vegetali coltivate è importante considerare che anche l’azione selettiva operata dall’uomo in migliaia di anni di domesticazione ha avuto un ruolo fondamentale, favorendo il mantenimento degli alleli per caratteristiche agronomiche superiori tra tutti quelli generatisi in seguito a mutazioni spontanee nella popolazione.
funzione, e quindi recessive (mentre quelle con acquisto di funzione sono per lo più dominanti); restano così mascherate a livello fenotipico dall’allele dominante quando presenti in eterozigosi, o possono essere eliminate dalla selezione se presenti in omozigosi.
Alla luce di tutte queste considerazioni, si sono sviluppati gli studi di mutagenesi sperimentale, che sfruttano l’azione di agenti mutageni fisici o chimici per ottenere mutazioni indotte con una frequenza più alta rispetto a quelle spontanee. Lo sviluppo delle tecniche di biologia molecolare ha reso disponibile anche un'altra classe di mutageni, quelli “biologici”, come i trasposoni o il T-DNA di Agrobacterium
tumefaciens, che permettono l’inserzione di DNA esogeno nei geni modificandone la
sequenza (Alonso et al., 2003); si parla in questo caso di mutagenesi inserzionale, che sarà approfondita nel capitolo sulla genetica inversa.
Tra i primi lavori di mutagenesi si possono citare quelli di Muller su Drosophila
melanogaster (Muller, 1927) e di Stadler su cereali quali orzo e mais (Stadler, 1928a;
Stadler, 1928b), che fornirono una dimostrazione della capacità di un agente fisico come i raggi X di incrementare la frequenza di mutazione in modo significativo.
In seguito a queste scoperte e sino agli anni ’40 si assiste così allo sviluppo della radiogenetica, cioè studi di genetica in cui si sfrutta l’azione mutagena di radiazioni penetranti quali quelle ionizzanti (raggi X, raggi gamma); i materiali d’elezione erano appunto Drosophila melanogaster e cereali (Ward, 1934; Eriksson, 1963), e proprio in orzo si ottennero i primi risultati applicativi grazie all’opera dello svedese Gustafsson (1963).
Al periodo della seconda guerra mondiale risale invece la scoperta da parte di Auerbach di un primo potente mutageno chimico, l’iprite, e si avviano studi più approfonditi grazie anche allo sviluppo della genetica dei microrganismi, sfruttando i ridotti tempi di moltiplicazione e la possibilità di lavorare con un numero molto elevato di individui in spazi ridotti, rispetto agli organismi superiori.
Notevoli progressi nell’ambito della mutagenesi si sono poi ottenuti utilizzando specie vegetali come Arabidopsis thaliana, una piccola crucifera, con ciclo vitale
breve, e Tradescantia, una specie erbacea perenne, entrambe provviste di pochi cromosomi e quindi utili per studi di citogenetica.
Sempre maggiore importanza ha poi acquisito il trattamento su seme, che per le sue peculiari caratteristiche si è rivelato particolarmente idoneo a questo tipo di esperimenti, e molte specie di cereali (soprattutto autogame come frumento, orzo, riso, ma anche allogame come il mais) hanno assunto un ruolo di primo piano negli studi di mutagenesi artificiale applicati alla genetica (Bianchi e Marocco, 1989).
1.1 MATERIALE VEGETALE PER IL TRATTAMENTO MUTAGENO
Nelle piante a riproduzione gamica i trattamenti mutageni si effettuano principalmente su seme o su polline.
L’utilizzo del polline offre alcuni vantaggi quali la possibilità di lavorare con grandi quantità di materiale in spazi ridotti e di utilizzare anche le radiazioni ultraviolette (U.V.); inoltre, fecondando una pianta portaseme demasculata coi granuli pollinici mutagenizzati, si possono ottenere piante interamente eterozigoti per la mutazione. Un limite è che il trattamento va effettuato in loco, o non a grandi distanza, in quanto il trasporto e la conservazione del polline risulta in genere abbastanza complesso. Il seme al contrario può essere facilmente inviato a centri appositi per la mutagenesi e conservato per un certo periodo prima di essere impiegato, inoltre è una struttura più resistente del polline e subisce quindi meno danni fisiologici in seguito a certi trattamenti. Tuttavia vi sono anche alcuni problemi: mutazioni che interessano l’endosperma non sono utili in quanto non conservate nelle generazioni successive; inoltre, essendo l’embrione una struttura pluricellulare, le mutazioni possono interessare solo alcune cellule, e in cellule diverse si possono verificare mutazioni diverse. Dal seme M1 (ottenuto in seguito a trattamento del cosiddetto seme M0) si
originano pertanto piante M1 con una certa frequenza di chimerismo e portanti le
mutazioni allo stato eterozigote. Queste mutazioni possono poi segregare ed essere fissate in omozigosi nella generazione M2, ottenuta per autofecondazione delle piante
della linea germinale. Le mutazioni a livello delle cellule somatiche, invece, non sono trasmissibili alla progenie e quindi sono perse nelle generazioni successive alla M1. Dal
numero delle cellule progenitrici, definito da Li e Redei “genetically effective cell number” (GECN), dipendono i rapporti di segregazione che indicano il numero di mutanti omozigoti attesi in M2 (Koornneef, 2002). Indicazioni quali il GECN e la
strategia riproduttiva (autogamia o allogamia) della specie da sottoporre a mutagenesi, influiscono sul numero di piante M1 da allevare e su quello di M2 da saggiare per
ottenere un certo numero di mutanti. In genere per i cereali è consigliabile raccogliere separatamente tutte le spighe delle piante M1 e allevare le M2 in fila per spiga oppure
raccogliere e seminare un seme per ogni spiga M1, un metodo quest’ultimo per
aumentare l’efficienza di analisi contenendo l’ampiezza della popolazione M2; è
importante inoltre proseguire la selezione dei mutanti anche nelle famiglie M3.
Nel caso di specie a propagazione vegetativa si trattano parti di pianta (marze o talee), provviste di gemme contenenti gli apici meristematici, in grado di dividersi attivamente originando i vari organi della pianta. Le gemme sono strutture multicellulari e, come nel caso del trattamento ai semi, si ha quindi la formazione di chimere di vario tipo; per ottenere un mutante non chimerico si possono prelevare cellule solo dal settore gemmario mutato e, ove possibile, rigenerare la pianta tramite coltura.
1.2 FATTORI CHE INFLUENZANO LA MUTAGENESI
Sulla base di vari studi, come quelli di Cadelcott e Konzak negli anni ’60 su semi di orzo, si sono evidenziati i vari fattori da cui dipende il risultato del trattamento mutageno. Tra questi fondamentale è sicuramente la specie vegetale su cui si opera, considerandone la sensibilità a diversi agenti, la modalità riproduttiva, il livello di ploidia; molto importante è anche lo stato metabolico e la fase del ciclo cellulare in cui si trova il materiale da sottoporre a mutagenesi, nonché le caratteristiche del trattamento applicato, come tipo e dose di agente mutageno (in generale è preferibile
non usare la dose massima al fine di limitare le mutazioni indesiderate o letali e la sterilità delle piante M1). Anche altri parametri come presenza di ossigeno,
temperatura, pressione e pH possono essere variati per aumentare l’efficienza a seconda dell’agente impiegato (Koornneef, 2002); inoltre per i semi, e in misura minore per il polline, bisogna tener presente che prolungando l’intervallo di tempo tra il trattamento e la germinazione si può avere un aumento dell’effetto mutageno detto “effetto storage” (Bianchi e Marocco, 1989).
1.3 MUTAGENESI FISICA
Per realizzare questo tipo di mutagenesi si utilizzano agenti fisici riconducibili a due tipologie principali:
- radiazioni ionizzanti che, nell’attraversare la materia vivente, causano la formazione di ioni e radicali liberi a livello di composti cellulari in seguito a collisioni casuali tra atomi con cessione di energia. Questi processi di ionizzazione sono probabilmente la causa principale degli effetti biologici delle radiazioni che, oltre a un danno diretto al DNA, determinano anche danni indiretti a vari componenti cellulari. Si osservano in genere rotture di vari legami nei filamenti del DNA e formazione di nuovi legami anomali, eventi che possono determinare mutazioni geniche ma anche molte aberrazioni cromosomiche, con conseguente sterilità di vario grado. Tra gli agenti di questo tipo troviamo i raggi X, molto usati e meno pericolosi rispetto ai raggi gamma (emessi ad esempio dal cobalto radioattivo) o ai neutroni veloci ottenuti in reattori nucleari. Essendo le radiazioni ionizzanti dotate di alto potere penetrante possono essere usate per trattare semi o parti vegetative della pianta, oltre che polline.
- radiazioni non ionizzanti, come i raggi U.V. che provocano eccitazione degli elettroni delle basi puriniche e pirimidiniche da cui sono assorbiti, ma non ionizzazioni. Ciò determina varie alterazioni del DNA come la formazione di dimeri di timina, con problemi nella replicazione, e conseguenti mutazioni geniche di vario tipo (sostituzioni, delezioni o inserzioni). I raggi U.V. hanno scarso potere penetrante e il loro uso è
1.4 MUTAGENESI CHIMICA
Questo tipo di mutagenesi sfrutta l’azione di vari composti chimici molto efficaci nell’indurre mutazioni benché alcuni provochino contemporaneamente un’elevata letalità, riducendo l’efficienza del trattamento. Proprio per questo è fondamentale valutare bene, tramite saggi appositi, la dose ottimale di mutageno da impiegare per ottenere il maggior numero di mutazioni utili tenendo però basso il numero di quelle letali o dei mutanti sterili.
Rispetto a quella fisica, la mutagenesi chimica presenta alcuni vantaggi: l’ampio spettro di molecole mutagene permette di effettuare studi più approfonditi; è possibile ottenere mutazioni più specifiche (ad esempio transizioni o transversioni), scegliendo un certo composto chimico, rispetto a quelle prodotte dalle radiazioni. Inoltre gli agenti chimici risultano più utili ai fini del miglioramento genetico in quanto inducono principalmente mutazioni geniche a differenza dei mutageni fisici che causano anche aberrazioni cromosomiche spesso deleterie.
In generale si distinguono tre classi principali di mutageni chimici in base al loro meccanismo d’azione:
• Analoghi di basi: composti aventi struttura simile a quella delle basi puriniche o pirimidiniche e che possono essere pertanto incorporati nel DNA durante la replicazione causando errori d’appaiamento che danno origine a sostituzioni di basi. Tra questi composti troviamo il 5-bromouracile e la 2-aminopurina.
• Agenti intercalanti: composti che si legano alla doppia elica del DNA distorcendola e provocando durante la replicazione l’aggiunta o la perdita di una o poche coppie di basi, cioè inserzioni o delezioni, che comportano uno slittamento del modulo di lettura delle triplette (“frameshift mutation”). Gli intercalanti, come le acridine o il bromuro d’etidio, causano pertanto effetti più rilevanti rispetto agli altri mutageni chimici che inducono invece sostituzioni di basi.
• Agenti alchilanti: composti che reagiscono con gli acidi nucleici causando modificazioni tramite alchilazione dei nucleotidi, con conseguenti sostituzioni o, più raramente, delezioni di basi. L’evento indotto più di frequente è la formazione di 7-alchilguanina che si appaia non con la citosina ma con la timina e, in seguito a replicazione del DNA, si ha una transizione (la coppia G-C è sostituita da quella A-T); si può verificare anche la depurinazione della guanina che causa invece delezioni. In generale agiscono come alchilanti i composti nitrosi, i solfati e sulfonati, gli epossidi, le etilenimine, la formaldeide e l’iprite. Più in particolare si possono citare nell’ambito di questa classe alcune delle sostanze più usate per la mutagenesi delle piante coltivate: l’etilmetansulfonato (EMS), molto usato in Arabidopsis thaliana, il dietilsolfato (dES), l’etilenimina (EI), la nitrosoguanidina (NG), l’etilnitrosourea (ENU) e la metilnitrosourea (MNU).
I composti di questa classe sono molto efficienti come mutageni, in quanto prontamente disponibili e molto penetranti in soluzione acquosa o sospesi in olio, ma presentano, come tutti i mutageni chimici, rischi per la salute umana essendo potenti cancerogeni e volatili; richiedono pertanto l’uso di opportune precauzioni da parte degli operatori.
Un discorso a parte riguarda gli agenti in grado di causare raddoppiamento del numero cromosomico cellulare, tra cui il più importante è la colchicina, che agisce bloccando la formazione del fuso mitotico e impedendo quindi la disgiunzione dei cromatidi.
1.4.1L’AZIDE SODICA
L’azide sodica (NaN3) è un composto di largo impiego, già da tempo utilizzato nei
laboratori chimici per l’ampio spettro di reazioni a cui può dar luogo, oltre a numerose applicazioni in varie industrie (ad esempio come generatore d’azoto per gli airbag) o in agricoltura per le sue proprietà erbicide, nematocide e fungicide (limitate però dalla alta
volatilità e bassa persistenza nel suolo). In ambito scientifico la sua importanza risiede soprattutto nell’essere un potente mutageno chimico, pur non essendo un agente alchilante, che si è rivelato tra i più efficaci in orzo e, in diversa misura anche in altre specie vegetali quali Triticum monococcum, Glycine max, Oryza sativa, Vicia faba (ma non Arabidopsis thaliana), oltre che in lievito, in Salmonella typhimurium e in
Escherichia Coli (Kleinhofs et al., 1978).
Le azidi inorganiche, di cui fa parte la NaN3 sono caratterizzate da legami ionici
mentre le azidi organiche hanno legami covalenti. Studi sulle azidi inorganiche si sono sviluppate a partire dalla scoperta dell’acido idrazoico nel 1890, evidenziando le proprietà tossiche di tali composti, prima fra tutte la capacità d’inibire la respirazione (Kleinhofs et al., 1978). Il notevole potenziale mutageno dell’azide è stato poi scoperto indirettamente grazie a studi di Spence (1965) sui meccanismi di riparazione delle rotture cromosomiche indotte in semi di orzo irradiati, una parte dei quali trattati con azide e originanti una popolazione di piante usate come controllo negativo (in quanto l’effetto inibitorio sulla respirazione sembra rallentare il processo di riparazione). Tra queste ultime si osservò però un numero significativo di mutanti clorofilla-deficienti (circa il 6%) indotti dall’azide stessa e non dalle radiazioni (Afsar Awan et al., 1980); questa frequenza di mutazione aumenta fino al 20% usando soluzioni a pH inferiore al pKa dell’azide (quindi pH < 4,8; pH ottimale = 3): in tal modo la specie predominante è la molecola non carica dell’acido idrazoico (HN3) che penetra più facilmente
attraverso le membrane cellulari rispetto allo ione N3- (Kleinhofs et al.,1978).
Un ulteriore notevole aumento della frequenza di mutazione si può ottenere sottoponendo i semi ad imbibizione alla temperatura di 0 °C e pre-germinazione a 20 °C in acqua addizionata di ossigeno per alcune ore (a seconda della specie). Questo pretrattamento è efficace anche per altri mutageni chimici che, nel caso di mutagenesi su seme, per poter reagire col DNA delle cellule meristematiche, devono diffondere nell’embrione attraverso la membrana di rivestimento (testa) del seme. Questo tessuto di rivestimento presenta diversa permeabilità ai diversi composti a seconda della specie vegetale considerata e ciò spiega in parte le differenti risposte ai trattamenti. In
generale, comunque, l’imbibizione, idratando i tessuti, serve proprio a facilitare la penetrazione dei composti chimici e a proteggere i sistemi cellulari da danni fisiologici (Konzak, 1987). Questo pretrattamento ha anche un effetto di stimolazione dei processi metabolici e della sintesi di ATP, influendo così sull’attività mitotica e le altre fasi del ciclo cellulare, e ciò è importante poiché l’azione mutagena dell’azide dipende dallo stato del DNA nelle cellule embrionali. Vari esperimenti hanno infatti evidenziato come la frequenza di mutanti clorofilliani sia incrementata quando la fase di pre-germinazione a 20°C è protratta per 8-16 ore e, poiché dopo tale periodo di tempo le cellule dell’apice del germoglio sono nella fase S del ciclo cellulare, si è ipotizzato che l’azide sia un mutageno attivo proprio in questa fase in cui avviene la replicazione del materiale genetico (Sander e Nilan, 1974). Tuttavia, tempi di pre-germinazione troppo prolungati causano un incremento della letalità del trattamento rendendo necessario un ulteriore aumento del numero di semi da trattare.
Alcuni studi hanno evidenziato che l’acido gibberellico, aggiunto dopo il trattamento, incrementa in modo indiretto l’effetto dell’azide, in quanto attiva enzimi promuoventi una maggiore crescita della pianta e quindi la sua sopravvivenza, anche in presenza di mutazioni. Altre sostanze, tra cui il cianuro di potassio, hanno invece un’influenza negativa sull’efficacia dell’azide, riducendone l’attività sui semi trattati (Prina e Favret, 1983).
A livello fenotipico, in seguito a trattamenti con azide sodica, si sono ottenute alcune categorie di mutanti utili di tipo morfologico come i semi-dwarf, fisiologico quali linee a spigatura precoce, e biochimico tra cui mutanti che accumulano prolina (Bright et al., 1981) e quelli privi di proantocianidina o antocianina (Olsen et al., 1993). Per quanto riguarda gli effetti dell’azide sodica, i molti esperimenti condotti in orzo hanno evidenziato come questa molecola induca in popolazioni M2 un’elevata frequenza
(superiore al 50%) di mutazioni clorofilliane (Nilan et al., 1973) e un alto livello di sterilità delle piante M1 (Prina et al., 1983). Si è osservata inoltre sterilità parziale nelle
generazioni M2 e fino a generazioni anche avanzate di selezione; questo aspetto limita
NaN3. Si sono condotti perciò degli studi per caratterizzare le linee mutanti aventi
sterilità parziale in modo da comprenderne i meccanismi e manipolarle per tentare di ridurre il fenomeno.
Questi lavori (Crispi et al., 1987) hanno portato alla conclusione che la sterilità parziale non dipende da alterazioni della struttura o del numero di cromosomi e neppure dalla presenza di geni letali per l’embrione o per l’endosperma, bensì da interazioni tra l’ambiente e mutazioni geniche minori e recessive, letali quando presenti in omozigosi, e che devono essere quindi allo stato eterozigote nelle linee mutanti inizialmente selezionate; tale sterilità è infatti eliminabile incrociando il mutante col parentale non trattato.
L’azide può indurre anche anormalità meiotiche pur non causando aberrazioni cromosomiche; uno dei pochi casi descritti è infatti una traslocazione reciproca che coinvolge i cromosomi 6 e 7 di orzo (Lehman et al., 1977). Queste anomalie alla meiosi sono probabilmente correlate all’alta sterilità delle piante M1 e possono
renderne conto, almeno in parte, anche in assenza di mutazioni cromosomiche visibili (Prina et al., 1983).
L’azione dell’azide sodica si esplica a livello del DNA tramite l’induzione di sostituzioni di basi, che causano spesso mutazioni di-senso; nel caso del gene Ant18 (coinvolto nella sintesi di antocianina) di orzo si sono osservate transizioni in direzione G/C – A/T, tre volte più frequenti rispetto a quelle A/T – G/C, e, in numero inferiore, anche transversioni del tipo A/T – T/A (Olsen et al., 1993).
La peculiarità dell’azide consiste nell’essere un raro esempio di promutageno; esso viene, infatti, metabolizzato e convertito, in alcune piante (Hordeum vulgare) o batteri (Salmonella), in un composto stabile e dotato di attività mutagena vera e propria, l’azidoalanina (Owais et al., 1983) tramite condensazione della NaN3 con
O-acetilserina catalizzata dall’enzima O-O-acetilserina sulfidrilasi. L’azidoalanina potrebbe interagire direttamente col DNA oppure essere convertita in un altro composto (ad esempio l’azidopiruvato); ma in entrambi i casi l’interazione col DNA avviene a livello
del gruppo azide (Olsen et al., 1993). Da studi in vitro sull’effetto dell’azidoalanina in embrioni di orzo, il meccanismo d’azione sembra essere l’induzione di siti sensibili alle endonucleasi e alla proteinasi K, con rotture a singolo filamento, e l’interazione con proteine ed enzimi coinvolti nel metabolismo del DNA (Veleminsky et al., 1987a; Veleminsky et al., 1987b).
1.5 RUOLO DELLA MUTAGENESI NEL MIGLIORAMENTO GENETICO
La mutagenesi sperimentale è stata largamente utilizzata in passato per studi di genetica classica, soprattutto sulla natura del gene e sulla trasmissibilità dei caratteri, oltre a quelli sulle relazioni sistematiche tra le specie entro un certo genere e sulla struttura genetica delle popolazioni (importanti a questo proposito gli studi sul genere
Triticum di McKey e Konzak negli anni ‘60).
Più di recente inoltre si sono abbinate le potenzialità della mutagenesi con un approccio di tipo molecolare per approfondire le indagini di genomica funzionale. La mutagenesi infatti, inducendo l’inattivazione di diversi loci genici, permette di creare ampie popolazioni di mutanti da usare per analisi di genetica diretta o inversa. Se da un lato il ricorso a mutanti cosiddetti “biologici”, quali i trasposoni o il T-DNA, è stato molto utile per il progresso della genomica funzionale in specie vegetali modello, nel caso dei cereali la mancanza di sistemi di trasformazione semplici e l’ampiezza notevole del genoma ha limitato l’utilità di questi sistemi, rendendo preferibile l’uso dei mutageni tradizionali chimici o fisici (Caldwell et al., 2004).
Tuttavia una delle sue applicazioni più importanti ha riguardato, soprattutto nei decenni passati, il miglioramento genetico tradizionale delle piante. Quest’ultimo si basa sulla selezione di nuove cultivar con caratteristiche superiori e ha pertanto come presupposto la presenza di variabilità genetica entro la popolazione. Una volta esaurita la variabilità presente nel germoplasma naturale si può ricorrere alla mutagenesi per creare nuova variabilità ed effettuare quindi il processo di selezione dei genotipi desiderati.
Una prima strategia usata già negli anni ’50-’60, detta “direct mutation breeding”, consisteva nell’induzione e selezione di un mutante che può costituire una nuova cultivar, in cui si è realizzato il miglioramento di un carattere monogenico (a eredità mendeliana semplice) mantenendo inalterati gli altri caratteri (MacKey, 1965).
In questo modo è stato possibile ridurre il tempo di sviluppo di varietà migliorate aumentando quindi i benefici ottenibili da esse (Micke e Donini, 1982) se si considera che uno dei fattori limitanti il miglioramento genetico consiste proprio nei lunghi tempi necessari per le fasi ripetute di incrocio e selezione prima di arrivare al rilascio di nuove cultivar.
A questo proposito si possono citare gli studi di Yamagata (1981) i quali hanno evidenziato che, nei cereali diploidi come l’orzo, il periodo di tempo per realizzare una cultivar mutante con questo metodo varia da 4 a 7 anni, circa la metà di quello normalmente richiesto col breeding senza mutagenesi.
Successivamente si è sviluppata un’altra strategia, detta “mutation cross-breeding”, che consiste nell’incrocio tra un genotipo mutante e uno coltivato (seguito da varie generazioni di reincrocio) per introdurre il carattere utile in quest’ ultimo, oppure nell’incrocio tra due mutanti combinando così i caratteri voluti nello stesso background genetico. Questo metodo permette lo sviluppo indiretto di nuove varietà; ma in questo caso, però, non si ha il vantaggio della riduzione dei tempi perché bisogna considerare anche le fasi di incrocio delle linee mutanti e di selezione dei ricombinanti utili (Yamagata, 1981).
In generale, l’approccio del “mutation breeding” può essere perciò visto come un metodo per l’ottenimento di cultivar migliorate complementare agli altri a disposizione dei breeder, come l’incrocio intraspecifico e interspecifico o tecniche basate sulle colture in vitro (induzione di variazione somaclonale, fusione di protoplasti, ottenimento di aploidi raddoppiati), senza considerare l’ingegneria genetica.
La scelta di un metodo dipende dalla specie vegetale considerata, dalla tecnologia di cui si dispone e da vari altri fattori; la mutagenesi resta, probabilmente, tra gli approcci tradizionali uno di quelli con maggior potenzialità nella creazione di nuova variabilità a
livello genetico, e che permette quindi di produrre le maggiori modificazioni a livello fisiologico e morfologico.
Tanti sono infatti i risultati ottenuti ad oggi con questa tecnica; tra i più importanti per fini applicativi si citano: resistenza a patogeni e parassiti o a stress abiotici, modificazioni morfologiche degli organi vegetativi e riproduttivi, maschiosterilità, variazione dell’epoca di fioritura, miglioramento qualitativo. Per citare qualche esempio si possono ricordare i frumenti a taglia ridotta e aumentata produttività, resistenti alla ruggine dei cereali, o varianti ad alto contenuto proteico nella cariosside (Corpuz et al., 1983); per l’orzo, i mutanti ad alto contenuto proteico, ad esempio con livelli più elevati dell’amminoacido lisina (Eggum, 1978) o quelli privi di antocianine e proantocianidine nel tegumento del seme, usati nel breeding di cultivar da malto per evitare la stabilizzazione chimica della birra contro l’opacità da polifenoli (Olsen et al., 1993).
In conclusione è importante sottolineare come il successo di un programma di miglioramento genetico basato sul “mutation breeding” dipenda, in buona parte, da un’accurata scelta del materiale iniziale e dalla pianificazione della strategia più idonea in base agli obiettivi prefissati, valutando anche le possibili interazioni tra geni, come gli effetti epistatici, o l’associazione tra una mutazione utile e una dannosa. Inoltre bisogna tener presente la necessità di pianificare un trattamento su ampia scala e di saggiare molte piante della generazione M2 per aumentare la probabilità di ottenere
almeno un mutante utile per il carattere d’interesse; ciò è necessario in quanto: non è possibile usare dosi troppo alte di mutageno, la mutagenesi chimica o fisica non è sito-specifica e vi è elevato chimerismo delle piante M1.
Infine, dalle precedenti considerazioni si evince come queste strategie siano applicabili solo al miglioramento di caratteri qualitativi (a controllo monogenico) o a caratteri quantitativi (a controllo poligenico) in cui sia noto un “major gene”, perché risulterebbe molto difficile ottenere contemporaneamente mutazioni utili in tutti i loci che controllano un carattere quantitativo.
2. LA GENETICA INVERSA
Con il termine “genomica funzionale” vengono indicate una serie di differenti metodologie che hanno lo scopo di desumere la funzione dei geni su scala genomica. L’utilizzo di queste tecniche è stato notevolmente facilitato dallo sviluppo di nuove tecnologie sempre più raffinate, efficaci ed applicabili a quasi tutti gli organismi. L’utilizzo di strumenti bioinformatici per l’allineamento di sequenze omologhe tra organismi differenti e lo studio dei pattern di espressione genica su larga scala sono solo due tra gli innumerevoli esempi di strategie di genomica funzionale utilizzati attualmente. Nonostante queste tecnologie siano estremamente importanti per dedurre informazioni generali su una nuova sequenza genica (la somiglianza con altre sequenze a funzione nota o il livello di espressione) non possono essere adottate per definire l’esatta funzione del gene e la sua espressione a livello fenotipico (Henikoff e Comai, 2003).
In passato veniva utilizzato, per stabilire la funzione di nuove sequenze geniche, un approccio di analisi denominato di “genetica diretta”, che permette di risalire dal fenotipo (es. un’alterazione morfologica) alla sequenza relativa che lo determina. Oggi sono disponibili, grazie a più efficienti tecniche di sequenziamento, una crescente quantità di sequenze, delle quali non si conosce la funzione associata. Infatti, fino ad oggi sono state pubblicate, e sono a completa disposizione della comunità scientifica, le sequenze dei genomi di Arabidopsis thaliana (The Arabidopsis Initiative, 2000), Oryza
sativa (Goff et al., 2002), Populus trichocarpa (Tuskan et al., 2006), Vitis vinifera
(Jaillon et al., 2007) e Sorghum bicolor (Paterson et al., 2009). Di conseguenza, sono state sviluppate tecniche denominate di “genetica inversa” che, partendo dalle sequenze conosciute, tentano di risalire alla funzione delle sequenze stesse. In generale le procedure iniziano con l’acquisizione della sequenza codificante di un gene potenzialmente di interesse e si tenta poi di indurre, tramite differenti strategie di mutagenesi, alterazioni al fine di modificarne la funzionalità. Una successiva analisi fenotipica del mutante permetterà di attribuire una funzione al gene bersaglio. Il flusso
di informazioni delle due strategie di analisi genetica può essere riassunto come indicato nella figura di seguito riportata:
Disporre di una popolazione di varianti (naturali od indotte artificialmente) è il primo passaggio sia per analisi di genetica diretta che inversa.
• Genetica diretta. si inizia con la selezione di un processo biologico od un aspetto morfo-fisiologico di interesse a cui segue il vaglio della popolazione per l’individuazione di fenotipi alterati. L’ultima fase è l’identificazione e il clonaggio del gene/i responsabile della mutazione/variazione fenotipica.
• Genetica inversa. si parte con la selezione di uno o più geni a sequenza nota ma a funzione ignota. Segue uno screening molecolare sequenza-specifico per individuare varianti nella porzione genomica selezionata. L’analisi fenotipica delle varianti individuate consentirà di associare alla variazione di sequenza una variazione morfo-fisiologica.
Genetica diretta
Genetica inversa
SEQUENZA
…
G
T
A
C
G
T
AAA
T
…
…
G
T
A
C
A
T
AAA
T
…
Selezione di un processo biologico di interesse Screening della popolazione per individuare fenotipi alterati Mappaggio e clonaggio del gene responsabiledel fenotipo Selezione di un gene di interesse Screening molecolare della popolazione sequenza-specifico Analisi del fenotipo dei
mutanti identificati
2.1 STRATEGIE DI GENETICA INVERSA
Diverse sono le metodologie proposte per studi di genetica inversa; per semplicità possono essere raggruppate in:
1) Strategie nelle quali il bersaglio della mutagenesi è uno specifico locus di interesse; in questo caso parleremo di tecniche basate su mutagenesi mirata. Purtroppo queste tecniche non sono ancora state affinate per il loro utilizzo in organismi eucariotici multicellulari. Tutte queste tecniche si basano sulla trasformazione genetica che non è facilmente applicabile a tutte le specie. In particolare, utilizzando tecniche di ricombinazione omologa, si sono ottenuti successi solo in poche specie modello quali
Mus musculus, Drosophila melanogaster e in campo vegetale nella briofita Physcomytrella Patens (Shaefer et al., 2000) in Arabidopsis thaliana (Beetham et al.,
1999; Kempin et al., 1997) e recentemente in Oryza sativa (Iida e Terada., 2004).
2) Strategie nelle quali è prevista la realizzazione di una popolazione mutagenizzata in maniera aspecifica, seguita da un’analisi effettuata su specifici geni; queste saranno indicate come tecniche basate su mutagenesi casuale. A questo scopo esistono tre strategie principali utilizzate per indurre mutazioni casuali all’interno di un genoma: la mutagenesi inserzionale, basata sull’inserzione di T-DNA e di trasposoni, la mutagenesi fisica e la mutagenesi chimica.
2.2 MUTAGENESI MIRATA IN GENETICA INVERSA
Per quanto concerne le strategie di “mutagenesi mirata” in campo vegetale, di primaria importanza sono i risultati ottenuti in Arabidopsis thaliana e Oryza sativa, le uniche specie per cui la trasformazione genetica non presenta più limitazioni tecnologiche.
2.2.1GENE TARGETING
E’ la strategia di mutagenesi mirata in assoluto più precisa; si realizza inducendo la ricombinazione omologa tra la sequenza del gene bersaglio del genoma ospite ed una sequenza di DNA estranea introdotta per trasformazione genetica opportunamente modificata per essere non funzionale. Se il processo di ricombinazione omologa avviene correttamente, uno specifico gene funzionale è sostituito con una copia quasi identica ma non funzionale (Mengiste e Paszkowski, 1999). Questa tecnologia consente di modificare una specifica sequenza senza determinare cambiamenti in nessun’altra regione del genoma. Sebbene i primi tentativi abbiano dato risultati apprezzabili (Beetham et al., 1999; Kepim et al., 1997), la tecnica è ancora praticamente inutilizzata su larga scala in campo vegetale a causa della bassa frequenza di ricombinazione omologa (Adams et al., 2002). Recenti avanzamenti tecnologici ed una miglior comprensione del meccanismo molecolare alla base della ricombinazione omologa, potrebbero rendere in futuro più semplice il suo utilizzo (Terada et al., 2002; Hanin e Paskowski, 2003). Ad esempio, e’ stato recentemente dimostrato che la sovraespressione in Arabidopsis thaliana del gene di lievito RAD54, coinvolto nel rimodellamento della cromatina, determina un incremento sostanziale dei trasformati per ricombinazione omologa (Shaked et al., 2005). Ancora, la messa a punto di un efficiente protocollo di selezione dei trasformati basato su un sistema a “doppia selezione positiva-negativa”, ha permesso di ottenere una quantià piuttosto elevata di trasformati per ricombinazione omologa per il gene della alcool deidrogenasi in Oryza
sativa (Terada et al., 2007).
2.2.2SILENZIAMENTO GENICO
Il fenomeno è stato descritto per la prima volta in petunia da due differenti gruppi di ricerca (van der Krol et al., 1990; Napoli et al., 1990); obiettivo di entrambi i ricercatorti era quello di ottenere fiori con colore più intenso introducendo nel genoma il gene della calcone sintetasi. Al contrario osservarono una riduzione di espressione sia
del gene endogeno che del gene estraneo introdotto. La tecnologia, oggi comunemente denominata “silenziamento genico”, viene utilizzata per attenuare l’espressione di specifici geni introducendo nel genoma ospite sequenze con elevata omologia. Dal punto di vista molecolare, l’espressione di elevati livelli di uno specifico dsRNA (RNA a doppio filamento) comporta la diffusione di un segnale di silenziamento genico di uno o pochi geni endogeni in maniera sequenza-specifica. Il silenziamento genico sembra essersi evoluto quasi senza modifiche in tutti gli organismi eucariotici superiori come difesa contro virus e trasposoni attivi (Robertson et al., 2004). Essendo l’unica strategia di mutagenesi che agisce a livello post-trascrizionale presenta alcuni peculiari vantaggi:
1) E’ possibile ridurre l’espressione di più geni contemporaneamente (es. membri di una famiglia genica) inducendo la sovra-epressione di una sequenza comune a tutti. La tecnica è stata utilizzata per silenziare i geni della Glutatione trasferasi di Arabidopsis
thaliana (Sappl et al., 2008)
2) Il silenziamento genico è molto interessante anche per lo studio di geni essenziali per la vitalità della pianta poiché determina in genere una semplice attenuazione dell’espressione genica (Waterhouse e Helliwell, 2003).
Purtroppo, la variabilità degli effetti in specie e tessuti differenti complica enormemente l’interpretazione e la riproducibilità dei risultati ottenuti utilizzando questa strategia. Inoltre, l’espressione del transgene può variare tra piante differenti della stessa specie e tra generazioni successive.
2.2.3NUCLEASI ZINC-FINGER
Recentemente è stata descritta una nuova metodologia che sfrutta proteine di fusione tra un dominio di riconoscimento specifico del DNA a “zinc-finger” ed un dominio nucleasico in grado di generare tagli a doppio filamento (Lloyd et al., 2005). Il domino a “zinc-finger” riconosce sequenze specifiche del DNA mentre il dominio nucleasico genera tagli introducendo mutazioni stabili. E’ stato dimostrato che l’espressione di una nucleasi “zinc-finger” sequenza specifica in Arabidopsis thaliana è in grado generare
mutazioni stabili (inserzioni e delezioni) in planta (Lloyd et al., 2005). L’ampia scelta di domini a “zinc-finger”, ciascuno con differente specificità, potrebbe permettere l’uso della tecnica per indurre mutazioni in quasi tutti i geni di un organismo.
2.3 MUTAGENESI CASUALE IN GENETICA INVERSA
Una ampia scelta di mutageni fisici, chimici o biologici possono essere utilizzati per indurre alterazioni utili per studi di genetica inversa. Ciascun mutageno è caratterizzato da una diversa efficienza (numero di eventi di mutazione per genoma) e conseguenza molecolare (varia da sostituzioni a singolo nucleotide fino a inserzioni e riarrangiamento di intere porzioni cromosomiche; Feldmann et al., 1994). La natura del danno a livello molecolare è strettamente correlato con l’effetto che la mutazione avrà sul fenotipo del mutante: delezioni, inserzioni e riarrangiamneti indurranno con elevata probabilità mutazioni con perdita di funzione, mentre mutazioni puntiformi avranno diversi effetti sulla funzionalità del gene (attività ridotta, attività eliminata o alterata).
2.3.1MUTAGENESI INSERZIONALE
La tecnica della mutagenesi inserzionale consiste nell’introdurre, all’interno del genoma di una specie, nuovi tratti di DNA a sequenza nota. Questi possono determinare, se si inseriscono all’interno di una sequenza genica, la perdita della sua funzione (mutazioni “knock-out”). Sottoponendo al trattamento di mutagenesi un elevato numero di individui è teoricamente possibile ottenere inserzioni in tutte le sequenze geniche della specie sottoposta al trattamento. Le due principali strategie utilizzate a questo fine sono il “T-tagging” e il “Transposon-tagging”.
T-TAGGING
La tecnica del “T-tagging” consiste nel produrre una ampia popolazione di individui mutagenizzati a seguito di trasformazione con il batterio Agrobacterium tumefaciens. Questo batterio possiede la capacità intrinseca di trasformare le piante attraverso la
trasmissione di un segmento di DNA (comunemente chiamato T-DNA) che penetra all'interno delle cellule vegetali e si integra in maniera casuale nel loro genoma. La tecnica del “T-tagging” è stata utilizzata con successo per studi di genomica funzionale in Arabidopsis thaliana e Oryza sativa (Jeon et al., 2000), specie che presentano un genoma di dimensioni relativamente ridotte e per le quali è disponibile un efficiente ed economico sistema di trasformazione (Sallaud et al., 2003). Il numero di eventi di trasposizione, e quindi di inserzione del T-DNA, deve essere molto alto (dell’ordine delle decine di migliaia) al fine di garantire una discreta probabilità che un qualsiasi locus genico sia stato interessato in almeno un caso (Kolesnik et al., 2004; Sallaud et
al., 2004). Per contro, si possono verificare eventi di inserzione complessi
(riarrangiamenti cromosomici o inserzioni a copia multipla) che rendono più complicata l’analisi genetica dei trasformati.
TRASPOSON-TAGGING
Gli elementi trasponibili, o trasposoni, sono particolari sequenze di DNA dotate di mobilità capaci, cioè, di trasferirsi da una regione all’altra del genoma (McClintock, 1953). Tale capacità viene sfruttata per generare dei mutanti da inserzione; su queste basi si fonda la tecnica del “Transposon-tagging”. Una categoria di elementi trasponibili, ampiamente utilizzati in programmi di “tagging” nei vegetali, sono stati individuati inizialmente nel genoma di Zea mays. Questi trasposoni sono raggruppabili in famiglie in cui si distinguono elementi autonomi, dotati della capacità intrinseca di escindersi e trasporsi, e elementi non autonomi o difettivi. Questi ultimi sono incapaci di trasporsi autonomamente e necessitano della presenza di un elemento autonomo, della stessa famiglia, che fornisca “in-trans” le funzioni necessarie alla trasposizione. Tra le famiglie di elementi trasponibili maggiormente utilizzate per programmi di “Transposon-tagging” dei vegetali troviamo la Ac/Ds, la Spm/dSpm. Gli elementi trasponibili Ac e Spm sono i membri autonomi delle due famiglie mentre Ds e dSpm sono, invece, quelli non autonomi. Queste famiglie possono essere trasferite nel genoma di altre specie, dove la trasposizione dei singoli elementi determina la produzione di mutanti da inserzione. Se l’evento di inserzione avviene all’interno di
una sequenza genica, può essere alterata l’espressione del gene e quindi può verificarsi un effetto rilevabile fenotipicamente (Maes et al., 1999). La famiglia Ac/Ds è stata ampiamente utilizzata per programmi di “Transposon-tagging” in sistemi eterologhi:
Arabidopsis thaliana (Parinov et al., 1999), Oryza sativa (Izawa et al., 1997) e Hordeum vulgare (Koprek et al., 2000, Cooper et al., 2004), otre che ampiamente in Zea mays (Bruntell, 2002). La famglia Spm/dSpm è invece stata utilizzata quasi
esclusivamente in Arabidopsis thaliana (Speulman et al., 1999; Tissier et al., 1999). Un’altra classe di elementi trasponibili utilizzati in programmi di “Transposon-tagging” sono i retrotrasposoni. Questi elementi sono presenti in tutte le specie vegetali ed in quantità molto elevate in piante con genoma ampio e complesso. La principale differenza rispetto ai comuni trasposoni risiede nel particolare meccanismo di trasposizione: il retrotrasposone viene trascritto in una forma intermedia a RNA e, successivamente, una trascrittasi inversa, codificata dall’elemento, retrotrascrive la sequenza in una forma definitiva a DNA che viene integrata in un nuovo sito del genoma. La tecnica del “Transposon-tagging” mediante retrotrasposoni è stata inizialmente utilizzata su Drosophila melanogaster (Bingham et al., 1981) e lievito (Smith et al., 1996). Nonostante l’abbondanza di questi elementi nelle piante, solo una piccola porzione di retrotrasposoni sembra essere attiva. In relazione alla loro frequenza di trasposizione Tnt1, Tto1 di Nicotiana tabacum e Tos17 di Oryza sativa sembrano essere i soli adatti al tagging nei vegetali (Kumar e Bennetzen, 1999). In particolare il retrotrasposone Tos17 è stato già utilizzato in programmi di genetica inversa di Oryza sativa (Hirochika, 1999).
ANALISI MOLECOLARE DEI MUTANTI INSERZIONALI
Una popolazione mutagenizzata con elementi inserzionali può essere sottoposta ad analisi, al fine di rintracciare la pianta che porta l’elemento di inserzione all’interno del gene di interesse, tramite due principali approcci molecolari.
Un primo metodo, già applicato in Drosophila melanogaster, Caenorhabditis
specifico per un gene target, di cui è nota la sequenza, ed un primer specifico per la sequenza di inserzione. Avviene così l’amplificazione solo quando l’inserto è situato all’interno o in prossimità della sequenza di interesse. Grazie all’elevata sensibilità e specificità della tecnica PCR, questo tipo di analisi può essere condotta su pool molto ampi di individui e permette una considerevole riduzione del numero di campioni da sottoporre ad analisi. Ad un eventuale risultato positivo all’interno del pool, seguirà la ricerca del singolo campione portante l’inserzione (Speulman et al., 1999). Un secondo metodo prevede il sequenziamento sistematico per un gran numero di piante delle regioni di DNA fiancheggianti l’inserto. Per Arabidopsis thaliana e Oryza sativa, specie nelle quali è già implementata tale strategia, sono stati creati database “Tag & Sequence” (TES) contenenti le informazioni di sequenza che possono poi essere confrontate in maniera automatica e veloce con sequenze di interesse per i singoli ricercatori. Tale risorsa riduce tutte le fasi di analisi e ricerca di uno specifico mutante ad una semplice ricerca in database. TES per Arabidopsis thaliana sono state create sia utilizzando il T-DNA che elementi Ds (Ortega et al., 2002) e in Oryza sativa utilizzando solo il T-DNA (An et al., 2005; Ryu et al., 2004).
2.3.2MUTAGENESI FISICA:DELETAGENE
La “Deletagene” è una strategia di genetica inversa che si avvale di un approccio molecolare basato su PCR per identificare, in un pool di campioni, mutazioni indotte da radiazioni ionizzanti ad alta energia o neutroni veloci (Li et al., 2001). L’amplificazione PCR viene realizzata con una coppia di primer fiancheggiante un gene di interesse ed è seguita dalla visualizzazione dei frammenti mediante elettroforesi. La visualizzazione di un amplificato di dimensioni ridotte rispetto all’atteso indica la presenza di una delezione indotta dal trattamento mutageno. La tecnica presenta alcuni vantaggi peculiari derivati dall’utilizzo della mutagenesi fisica:
1) è facilmente applicabile in specie per cui non sono disponibili protocolli efficienti di trasformazione.
2) è possibile, con un solo evento di mutagenesi, eliminare un gruppo di geni in stretta associazione (es geni ripetuti in tandem; Li et al, 2002).
Nonostante i considerevoli vantaggi, la tecnica non è mai stata utilizzata per analisi di genetica inversa su larga scala; uno dei pochi risultati applicativi è stata l’individuazione di mutazioni in alcuni geni codificanti per fattori di trascrizione di
Arabidopsis thaliana. Una successiva analisi fenotipica ha dimostrato il loro
coinvolgimento nella resistenza sistemica acquisita (Zhang et al., 2003). E’ stata sviluppata una popolazione “Deletagene” anche in Oryza sativa al fine di identificare mutazioni in geni coinvolti nella resistenza a patogeni (Manosalva et al., 2003); l’analisi molecolare ha consentito di identificare mutazioni in due differenti geni della Fenilalanina Ammonio Liasi (PAL) enzima chiave nella biosintesi dei composti fenolici. La mancata diffusione della tecnica è forse dovuta alla simultanea comparsa di nuove e più efficienti strategie di genetica inversa basate su mutagenesi chimica.
2.4 MUTAGENESI CHIMICA IN GENETICA INVERSA: IL TILLING
Recentemente è stata sviluppata una nuova strategia di genetica inversa denominata TILLING (Targeting Induced Local Lesion IN Genomes), che combina mutagenesi chimica e “screening” molecolare basato sulla reazione PCR su un ampia popolazione di individui, al fine di identificare nuove varianti alleliche prodotte dal trattamento mutageno (McCallum et al., 2000). Mutageni chimici opportuni, quali l’EMS (Etil metan sulfonato) o l’azide sodica, inducono principalmente mutazioni geniche puntiformi e permettono quindi, a differenza della mutagenesi inserzionale, di ottenere una serie di varianti alleliche per locus risultanti in una alterazione o una perdita di funzione (knockout mutation; Henikoff e Comai, 2003). Inoltre, l’elevata densità con cui sono indotte queste mutazioni rendono la tecnica applicabile anche a sequenze geniche di dimensioni ridotte. Un altro aspetto vantaggioso della mutagenesi chimica è la sua applicabilità alla maggior parte delle specie in quanto non richiede efficienti protocolli di trasformazione e rigenerazione. Per queste caratteristiche il TILLING
rappresenta una strategia tra le più promettenti per studi di genomica funzionale nelle piante.
Per quanto concerne l’analisi molecolare, la prima fase di un saggio TILLING consiste in una amplificazione PCR della sequenza bersaglio effettuata su una miscela di DNA genomici estratti da diverse piante mutagenizzate. Una successiva fase di denaturazione e rinaturazione consente poi la formazione di molecole eteroduplex tra ampliconi provenienti da campioni differenti. Nel caso sia presente all’interno della miscela (pool) una variante indotta dal trattamento mutageno, si ha la formazione di appaiamenti imperfetti a singolo nucleotide. Segue una digestione enzimatica con una particolare nucleasi in grado di riconoscere come siti di restrizione gli appaiamenti imperfetti a singolo a nucleotide con la formazione di frammenti a basso peso molecolare. La visualizzazione dei frammenti ottenuti avviene tramite una elettroforesi su gel denaturante o, in alternativa, con elettroforesi capillare. In presenza di un risultato positivo il DNA genomico di ogni singolo componente del pool viene miscelato con una uguale quantità di DNA genomico del “wild-type” e sottoposto alle varie fasi di analisi molecolare precedentemente descritte. In questo modo è possibile identificare il singolo campione portante la mutazione. La presenza della mutazione puntiforme viene infine verificata tramite sequenziamento. Una descrizione più dettagliata degli aspetti tecnici della strategia TILLING sarà riportata in seguito.
Prima di procedere con l’analisi delle differenti fasi della strategia TILLING, risulta opportuno mettere a confronto nella seguente tabella le principali strategie di genetica inversa utilizzate in campo vegetale. Per ciascuna tecnica sono stati riportati i vantaggi e gli svantaggi che la caratterizzano e la contraddistinguono dalle altre strategie.
TECNICA
VANTAGGI
SVANTAGGI
MUTAGENESI MIRATA
Ricombinazione Omologa
• Consente di alterare un gene specifico
• Induce mutazioni stabili
• Bassissima efficienza in specie vegetali
trasformazione genetica Silenziamento
genico
• Effetti differenti sulla funzionalità della proteina (è possibile studiare geni essenziali)
• E’ possibile restringere l’analisi a specifici tessuti o stadi di sviluppo
• Studi di famiglie geniche
• E’ impossibile prevedere il livello di silenziamento • Mutazioni instabili • Strategia basata su trasformazione genetica Nucleasi zinc-finger
• Consente di alterare un gene specifico
• Induce mutazioni stabili
• Strategia basata su trasformazione genetica
• Bassa efficienza
• Utilizzata solo a livello sperimentale MUTAGENESI CASUALE Mutagenesi inserzionale • Alta efficienza
• Possono essere indotte mutazioni a perdita di funzione od acquisto di funzione
• Induce mutazioni stabili
• Mutagenesi casuale
• Non utilizzabile per lo studio di geni essenziali
• Strategia basata su trasformazione genetica DELETAGENE • Consente l’analisi di più geni in
stretta associazione
• Non è richiesta trasformazione genetica (applicabile a tutti gli organismi)
• Mutazioni stabili
• Mutagenesi casuale
• Induce solo mutazioni con perdita di funzione
TILLING • Alta efficienza
• Induce mutazioni con un ampio spettro di effetti sul fenotipo (è possibile lo studio di geni essenziali)
• Non richiede trasformazione genetica (applicabile a tutti gli organismi)
• Mutazioni stabili
2.4.1PRODUZIONE DI UNA POPOLAZIONE MUTAGENIZZATA
La realizzazione di una adeguata popolazione di mutanti è un aspetto di primaria importanza per l’ottenimento di una buona risorsa TILLING. In effetti, le scelte attuate in sede di mutagenesi chimica avranno ripercussioni su tutte le fasi successive e sulla qualità della risorsa. In questa prima fase, è indubbiamente fondamentale il conseguimento di una elevata frequenza di mutazione pur mantenendo bassi il livello di letalità ed il livello di sterilità.
Di primaria importanza nella strategia TILLING sono quindi la scelta della specie da sottoporre a trattamento e del mutageno da utilizzare.
2.4.1.1SCELTA DELLA SPECIE
Le specie ideali per l’analisi TILLING hanno un genoma diploide, piccolo, povero in sequenze ripetute e ricco di sequenze codificanti; in generale, quindi, un genoma molto semplice. In tali specie le mutazioni puntiformi interessano con maggiore probabilità sequenze geniche ed hanno con maggiore frequenza una ripercussione sul fenotipo (Caldwell et al., 2004). Tuttavia, la grande maggioranza dei genomi degli organismi eucariotici, in particolare dei vegetali, sono caratterizzati da un elevato grado di complessità; presentano geni ripetuti in tandem, famiglie geniche, elevata quantità di sequenze ripetute e geni interrotti da lunghe sequenze non codificanti. Inoltre, l’evoluzione di molte specie vegetali è avvenuta in seguito a parziale o totale duplicazione del genoma (Bowers et al., 2005; Paterson et al., 2006). Ad esempio, il genoma di frumento, una delle specie maggiormente coltivate, è interamente duplicato (Feuillet e Keller, 2002). Per le analisi TILLING sarebbe preferibile impiegare delle specie simili dal punto di vista evolutivo ma con un genoma più semplice. Tali specie sono considerate dei “modelli” per determinate classi filogenetiche (ad esempio ricordiamo l’orzo per le Triticae).
L’analisi molecolare è altresì complicata dall’elevato grado di eterozigosi che contraddistingue quasi tutti i genomi degli organismi superiori; risulta difficile
distinguere tra mutazioni indotte e polimorfismi preesistenti. L’eterozigosi è un aspetto che complica l’analisi molecolari ma non è da considerare un problema insormontabile. In effetti, buoni risultati sono stati ottenuti anche in specie altamente eterozigoti, come ad esempio Drosophila melanogaster (Winkler et al., 2004) e Danio rerio (Draper et
al., 2004; Wienholds et al., 2003). In questi casi è buona regola attuare il trattamento di
mutagenesi su pochi individui molto simili tra loro (o preferibilmente solo uno) al fine di ridurre il più possibile la variabilità genetica iniziale (Comai e Henikoff, 2006).
2.4.1.2SCELTA DEL MUTAGENO CHIMICO
Numerosissime informazioni, circa le caratteristiche dei principali agenti mutageni e dei protocolli per il loro utilizzo, sono oggi a disposizione della comunità scientifica grazie all’ampio utilizzo della mutagenesi sia per il miglioramento genetico delle specie agrarie sia per analisi di genetica diretta. Alcuni dei principali agenti mutageni (chimici e fisici) utilizzati nel settore vegetale, i loro effetti prevalenti ed il loro impiego in esperimenti di genetica inversa sono elencati nella tabella di seguito riportata:
AGENTE MUTAGENO
CATEGORIA EFFETTO UTILIZZO
Raggi UV Mutageno fisico Dimeri di pririmidine ed errori nella replicazione
Non utilizzato Raggi-X,
Raggi gamma
Mutageno fisico Ionizzazione di molecole organiche con formazione di radicali che causano rotture nel DNA e alterazione nelle purine e pirimidine
Deletagene
Neutroni veloci Mutageno fisico Danni consistenti al DNA Deletagene
2-amminopurina5-bromouracile
Mutageno chimico Appaiamento errato della basi azotate: induce transizioni da A/T a G/C
Non utilizzato Bromuro di etidio,
Proflavina, Arancio
Mutageno chimico Agrnti intercalanti: causano delezioni, inserzioni e spostamento del modulo di
acridina lettura EtilMetanSulfonato
(EMS)
Mutageno chimico Modificazione della Guanina: induce transizioni G/C in A/T
TILLING N-Etil-N-Nitrosurea
(ENU)
Mutageno chimico Modificazioni delle Timina: induce tran versioni A/T in T/A
TILLING Acido Nitroso Mutageno chimico Modificazioni della Citosina: indice
transizioni A/T in G/C
Non utilizzato Idrossilammina Mutageno chimico Modificazioni della Citosina: induce
transizioni G/C in A/T
Non utilizzato Azide sodica Mutageno chimico Meccanismo di azione non noto TILLING
La frequenza di mutazione indotta in una specie di interesse è il primo aspetto che deve essere considerato per la scelta di un mutageno chimico (Henikoff e Comai, 2003). Come regola generale, un mutageno è da considerarsi efficace se induce un numero elevato di mutazioni per genoma. Per questa ragione, la sostanza maggiormente utilizzata per analisi TILLING in campo vegetale è stato l’Etil-Metan-Sulfonato (EMS), un mutageno chimico che produce un numero molto elevato di transizioni nel genoma delle piante. Inoltre, l’EMS introduce pochissime alterazioni cromosomiche ed ha un effetto molto limitato sulla fertilità e la vitalità delle piante (Henikoff e Comai, 2003). L’EMS induce un’ampia gamma di alterazioni: prevalentemente mutazioni puntiformi e, in percentuale molto inferiore, piccole inserzioni e delezioni (Greene et al., 2003; Till et al., 2003). Dal un punto di vista biochimico agisce alchilando residui di guanina e determina un appaiamento errato: la guanina alchilata riconosce come nucleotide complementare una timina così da indurre transizioni in direzione G/C-A/T. Un esperimento di genetica inversa condotto su 192 geni di Arabidopsis thaliana ha messo in evidenza che il 99% delle mutazioni indotte da EMS sono transizioni G/C-A/T (Greene et al., 2003). L’EMS ha dato ottimi risultati in varie specie vegetali: Arabidopsis thaliana (McCallun et al., 2000), Hordeum
vulgare (Calwell et al., 2004), Lotus japonicus (Perry et al., 2003), Triticum spp.
(Slade et al., 2005), Zea mays (Till et al., 2004) e Oryza sativa (Till et al., 2007). Solo di recente è stato impiegata anche l’azide sodica (NaN3) per analisi TILLING, sia da