Studio termodinamico e cinetico dell'interazione del 4',6-diamidino-2-phenylindole (DAPI) con il DNA
Testo completo
(2) Indice Capitolo 1 .............................................................................................................................. 3 Introduzione ....................................................................................................................... 3 1.1. Il DNA ..................................................................................................................... 4. 1.1.1. La struttura primaria ........................................................................................ 5. 1.1.2. La struttura secondaria ..................................................................................... 6. 1.2. Interazioni DNA-colorante...................................................................................... 8. 1.2.1. Complesso esterno ........................................................................................... 8. 1.2.2. Groove binding ................................................................................................ 8. 1.2.3. Intercalazione ................................................................................................. 10. Capitolo 2 ............................................................................................................................ 13 Materiali e metodi ............................................................................................................ 13 2.1 Materiali ..................................................................................................................... 13 2.2 Metodi ........................................................................................................................ 14 2.2.1 Trattamento del CT-DNA.................................................................................... 14 2.2.2 Misure di pH ........................................................................................................ 14 2.2.3 Misure spettrofotometriche ................................................................................. 14 2.2.4 Misure spettrofluorimetriche ............................................................................... 16 2.2.5 Titolazioni............................................................................................................ 18 2.2.6 Misure di viscosità ............................................................................................... 19 2.2.7 Tecnica T-jump ................................................................................................... 19 Capitolo 3 ............................................................................................................................ 23 Risultati ............................................................................................................................ 23 3.1 Studio termodinamico ................................................................................................ 23 3.1.1 Titolazioni spettrofotometriche ........................................................................... 24.
(3) 3.1.2 Titolazioni spettrofluorimetriche ......................................................................... 29 3.1.3 Dipendenza dell’equilibrio dalla temperatura ..................................................... 36 3.1.4 Sintesi e discussione dei dati termodinamici ....................................................... 39 3.2 Misure di viscosità ..................................................................................................... 41 3.3 Misure di quenching di fluorescenza ........................................................................ 43 3.4 Studio cinetico............................................................................................................ 46 3.4.1 Effetto cinetico veloce ......................................................................................... 46 3.4.2 Effetto cinetico lento............................................................................................ 48 3.4.3 Sintesi e discussione dei dati cinetici................................................................... 52 Capitolo 4 ............................................................................................................................ 55 Conclusioni ...................................................................................................................... 55 Appendice I .......................................................................................................................... 59 Appendice II ......................................................................................................................... 61 Appendice III ....................................................................................................................... 65 Bibliografia .......................................................................................................................... 71. 2.
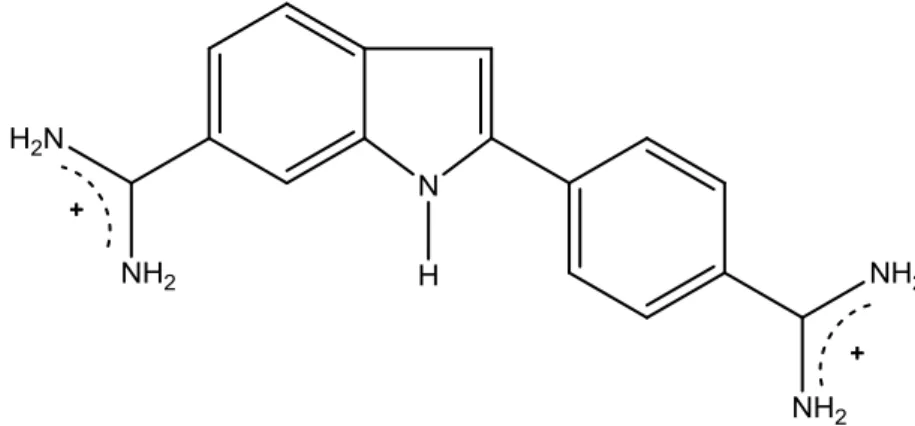
(4) Capitolo 1. Introduzione In questo lavoro di tesi viene presentato uno studio termodinamico e cinetico dell’interazione del colorante fluorescente 4’,6-diamidino-2-fenilindolo (DAPI), la cui formula di struttura è mostrata in Figura 1.1, con DNA naturale di tipo Calf Thymus.. Figura 1.1 Formula di struttura del 4’,6-diamidino-2-fenilindolo (DAPI).. Il DAPI, originariamente sintetizzato come analogo del Berenil (Dann et al., 1971), un farmaco antiparassitario per la cura della tripanosomiasi, ha trovato successivamente largo impiego in biochimica e in citochimica come agente di staining, ovvero per la colorazione di biosubstrati (per questa ragione d’ora in avanti sarà indicato anche come colorante). Quando il DAPI si lega al DNA, prediligendo le sequenze AT (Kapuściński, 1995), la sua resa quantica aumenta fortemente ed è per questo motivo che viene ampiamente utilizzato per colorare il nucleo delle cellule. Il meccanismo dell’interazione tra il DAPI e il DNA non è però ancora del tutto chiaro. Sebbene il legame alla minor groove del DNA sia il più accreditato (Schipper & Nordén, 1981; Zimmer & Wähner, 1986; Kubista et al., 1987; Jeppesen & Nielsen, 1989), esistono evidenze della presenza di un secondo modo di binding, più debole, che fanno supporre la.
(5) Capitolo 1. possibile presenza di fenomeni di tipo intercalativo (Kania et al., 1976; Kapuściński & Skoczylas, 1978; Kapuściński & Szer, 1979; Chandra & Mildner, 1979) o di formazione di un complesso esterno (Stepień et al., 1979). Il tipo di legame che queste molecole stabiliscono con il DNA riveste una notevole importanza. Interazioni di tipo “groove”, ovvero nella scanalatura, non modificano significativamente la struttura del polinucleotide, mentre al contrario, fenomeni di tipo intercalativo alterano profondamente il DNA. Per questa ragione, gli intercalanti sono considerati molecole potenzialmente cancerogene (Jeffrey, 1985; Ferguson & Denny, 1991; Stephens et al., 2000). Tuttavia, grazie alla loro capacità di inibire la trascrizione e la replicazione del DNA, tali molecole hanno recentemente trovato impiego come chemioterapici per la loro proprietà di inibire la crescita delle cellule neoplastiche (Braña et al., 2001). Lo scopo di questo lavoro è quindi quello di ottenere nuovi parametri termodinamici e cinetici al variare delle concentrazioni dei reagenti, del loro rapporto, della temperatura e della natura del solvente, al fine di introdurre nuovi elementi utili alla valutazione del modo di binding del DAPI con il DNA.. 1.1 Il DNA Gli acidi nucleici sono molecole complesse che sovrintendono alla trasmissione dei caratteri ereditari, e in particolare il DNA, presente nel nucleo delle cellule eucariote, è il depositario del corredo genetico. L’integrità del DNA, in termini di sequenza e di conformazione, è essenziale ai fini dell’espressione genica. Data la complessità degli acidi nucleici, per comprenderne la forma occorre anzitutto identificarne le strutture primaria, secondaria e terziaria. La struttura primaria di un polinucleotide è costituita dalla sequenza dei componenti costituenti il filamento, quella secondaria è data dall’avvolgimento dei filamenti che danno origine ad una doppia elica, infine, la struttura terziaria, è costituita dai ripiegamenti successivi della doppia elica.. 4.
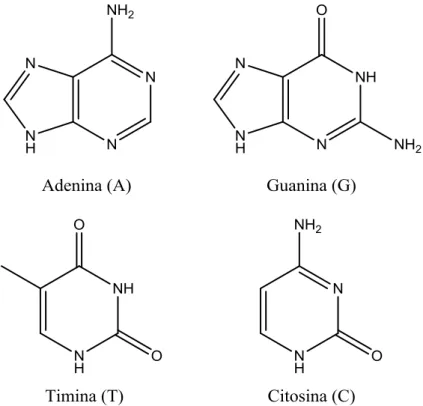
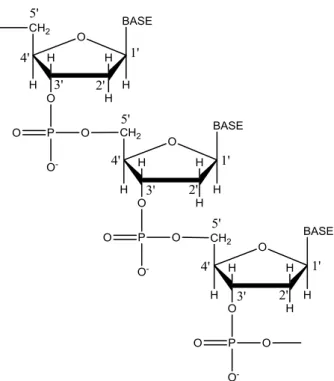
(6) Introduzione. 1.1.1 La struttura primaria La struttura monomerica del DNA, chiamata nucleotide, è costituita da tre unità fondamentali: un pentoso (2-deossi-d-ribosio, un monosaccaride a 5 atomi di carbonio), un gruppo fosforico ed un sistema eteroaromatico a scelta tra un gruppo di 4 basi (2 di tipo purinico, Adenina e Guanina, e 2 di tipo pirimidinico, Timina e Citosina) la cui struttura è riportata in Figura 1.2.. Figura 1.2 Struttura delle basi azotate costituenti il DNA.. Le basi azotate si legano all’atomo di carbonio in posizione 1’ del deossiribosio mediante l’atomo di azoto in posizione 1’ di una pirimidina oppure mediante l’atomo di azoto in posizione 9’ di una purina; si costituisce così un singolo nucleotide. Ciascun nucleotide della catena si lega al successivo tramite un gruppo fosforico che esterifica l’ossidrile in posizione 3’ di un pentoso e quello in posizione 5’ del pentoso successivo (Figura 1.3). I nucleotidi si uniscono formando un legame fosfodiesterico dando luogo a filamenti costituiti da uno scheletro in cui si alternano residui fosforici e di deossiribosio.. 5.
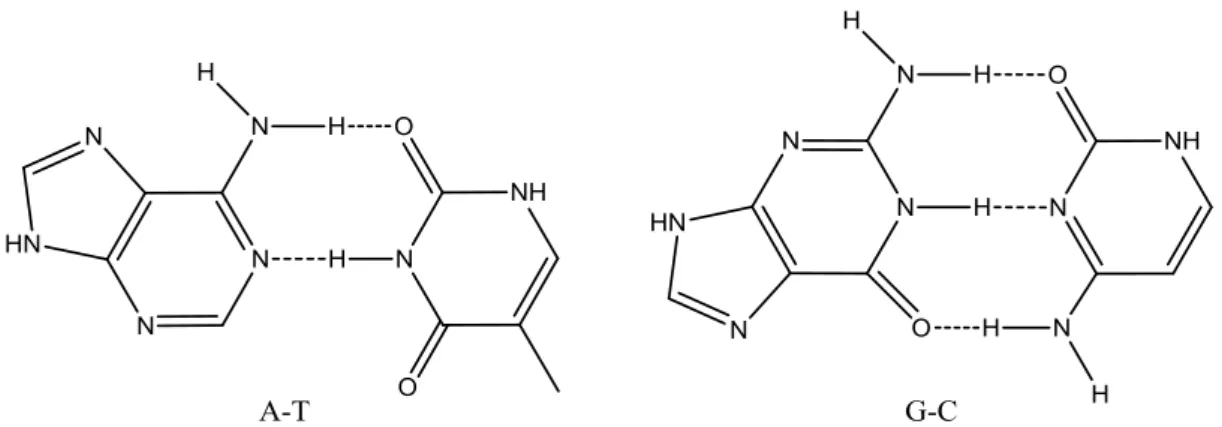
(7) Capitolo 1. Figura 1.3 Connessione di nucleotidi in un filamento singolo di DNA.. 1.1.2 La struttura secondaria Il primo modello accurato della struttura secondaria del DNA è stato presentato nel 1953 da James Watson e Francis Crick (Watson & Crick, 1953). Essi proposero un modello secondo il quale il DNA è formato da due filamenti di polinucleotidi orientati in direzione opposta l’uno rispetto all’altro e disposti secondo una doppia elica destrogira (forma B del DNA); le basi azotate sono rivolte verso l’interno dell’elica stessa mentre le unità fosfato verso l’esterno. I due filamenti sono tenuti assieme dai legami a idrogeno presenti tra le basi azotate e dalle interazioni non covalenti tra i piani delle basi (stacking interactions). Una caratteristica fondamentale, sulla quale si basa la stabilità di questa struttura, è la specificità dei legami tra le basi azotate. Il modello proposto da Watson e Crick prevede un principio di complementarità delle basi azotate secondo il quale l’Adenina si accoppia sempre con la Timina mentre la Citosina si accoppia sempre con la Guanina (Figura 1.4).. 6.
(8) Introduzione. Figura 1.4 Strutture delle coppie di basi proposte da Watson e Crick.. Una rappresentazione tridimensionale della struttura del DNA è rappresentata in Figura 1.5.. solco minore (minor groove) 6Å. passo dell’elica (10 paia di basi) = 34 Å. solco maggiore (major groove) 12 Å. Figura 1.5 Struttura tridimensionale della doppia elica.. Le basi planari sono disposte perpendicolarmente all’asse dell’elica, le basi adiacenti presentano una separazione di 3.4 Å e, poiché la struttura dell’elica si ripete ogni 34 Å, 7.
(9) Capitolo 1. sono presenti 10 residui per ogni giro. Infine, ciascuna base ruota di 36° rispetto a quella precedente, cosicché 10 basi completano un giro dell’elica (10×36°=360°).. 1.2 Interazioni DNA-colorante Un colorante può interagire non covalentemente con il DNA in tre diversi modi: complesso esterno, groove binding o intercalazione.. 1.2.1 Complesso esterno I gruppi fosfato, disposti esternamente alla doppia elica del DNA, conferiscono ad esso una carica negativa. La maggior parte dei coloranti, invece, essendo di tipo cationico, presenta una carica positiva. Il complesso esterno è quindi stabilizzato da interazioni di tipo elettrostatico e, in eccesso di colorante, anche da interazioni non covalenti tra molecole di colorante adiacenti, note come “interazioni di stacking”. La costante di formazione del complesso esterno mostra una notevole dipendenza dalla forza ionica del mezzo, infatti quando quest’ultima aumenta, parte delle cariche negative del DNA sono neutralizzate dai cationi salini (effetto schermo). Conseguentemente l’interazione elettrostatica DNA/colorante diventa più debole e la costante di binding decresce. E’ stato osservato che il contributo dell’interazione elettrostatica alla stabilità del complesso DAPI/DNA è in molti casi importante (Misra & Honig, 1995; Medhi et al., 1999).. 1.2.2 Groove binding Nel caso del groove binding, la molecola di colorante si dispone all’esterno della doppia elica occupando uno dei due solchi della stessa. Le molecole di piccole dimensioni tendono a posizionarsi nel solco minore (Neidle, 2001) mentre molecole molto grandi, come le proteine, si legano preferenzialmente al solco maggiore (Hannon, 2007). Esistono diversi tipi di molecole che possono legarsi ai solchi del DNA, caratterizzate tuttavia da alcune proprietà comuni. Si tratta, infatti, solitamente di strutture rigide, 8.
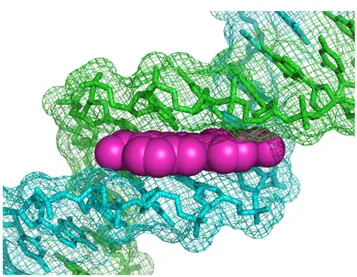
(10) Introduzione. costituite da anelli aromatici complanari e caricate positivamente (anche se non mancano esempi di molecole neutre). L’interazione della molecola al solco minore avviene mediante forti interazioni elettrostatiche con i gruppi fosfato, interazioni di tipo van der Waals e ponti a idrogeno. La Figura 1.6 mostra la disposizione di un “groove binder” all’interno del solco minore della doppia elica del DNA.. Figura 1.6 Interazione di un groove binder al solco minore della doppia elica del DNA.. La maggior parte dei groove binder mostra una preferenza per il solco minore delle regioni di DNA ricche di sequenze AT (Reddy et al., 2001), questa preferenza può essere spiegata tenendo conto delle seguenti caratteristiche: . il solco minore formato dalle sequenze AT è più stretto di quello formato dalla sequenze GC, ciò favorisce le interazioni di tipo van der Waals.. . la superficie delle regioni AT del solco minore presenta un potenziale elettrostatico negativo, ciò favorisce l’interazione con i leganti cationici, come sono la maggior parte dei groove binder. Nelle regioni GC il potenziale è invece altamente positivo (Neidle, 2001).. . il solco minore nelle regioni AT è liscio mentre, nelle regioni GC, il gruppo amminico della Guanina, orientato verso il solco stesso, può impedire stericamente l’interazione con il colorante (Kopka et al., 1993).. 9.
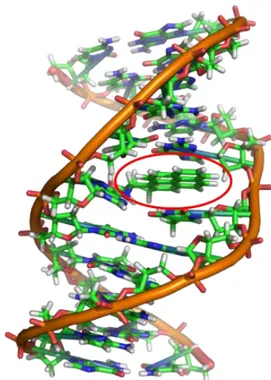
(11) Capitolo 1. Una molecola che si lega al solco occupa normalmente tra le 3 e le 5 paia di basi (AbuDaya et al., 1995) e provoca modifiche strutturali minori rispetto all’intercalazione. L’adattamento della molecola alla forma del solco, e viceversa, aumenta le interazioni dando luogo a complessi molto stabili.. 1.2.3 Intercalazione Il modello dell’intercalazione fu inizialmente proposto da Lerman nel 1961 (Lerman, 1961). Il colorante si inserisce tra due paia di basi consecutive della doppia elica (Figura 1.7) stabilizzandosi grazie alle interazioni di stacking tra la molecola stessa e le basi del DNA. Possono stabilirsi anche legami a idrogeno, interazioni idrofobiche ed elettrostatiche.. Figura 1.7 Intercalazione di una molecola di benzopirene (cerchio rosso) tra due paia di basi del DNA.. Come conseguenza dell’intercalazione, si producono importanti deformazioni della struttura a doppia elica (Figura 1.8). La reazione di intercalazione è accompagnata da un aumento della distanza di separazione tra le paia di basi.. 10.
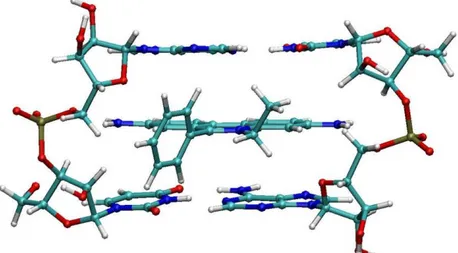
(12) Introduzione. Questa fenomeno implica lo svolgimento (unwinding) della doppia elica, la cui entità dipende dalla struttura dell’intercalante. Per quanto detto nel paragrafo 1.1.2, la rotazione. Figura 1.8 Deformazione della doppia elica in seguito ad una reazione di intercalazione. tra le paia di basi è di 36°. La reazione di intercalazione del bromuro di etidio con il DNA (Figura 1.9), ad esempio, riduce l’angolo a 10°, producendo un unwinding di 26°. La proflavina, un’altra molecola intercalante, produce invece un unwinding di 17°. Anche se l’unwinding avviene solamente al sito di legame, la sua influenza si propaga lungo tutta la struttura a doppia elica. Conseguentemente, l’intercalazione produce effetti a lungo raggio tanto allo struttura quanto alla stabilità del DNA stesso.. Figura 1.9 Complesso di intercalazione tra il bromuro di etidio e due paia di basi del DNA.. 11.
(13) Capitolo 1. Le molecole intercalanti hanno generalmente struttura planare costituita da anelli aromatici, ai quali posso essere legati vari gruppi laterali, e sono di solito caricate positivamente. Questo facilita l’avvicinamento all’elica del DNA che presenta una superficie di carica negativa. Gli intercalanti presentano una specificità limitata a causa della loro difficoltà nel riconoscere più di una coppia di basi, sono per questo leganti molto meno selettivi dei groove binder. Molecole di questo tipo presentano proprietà antitumorali per la loro capacità di interrompere i processi di replicazione, trascrizione e riparazione del DNA. Tuttavia, il loro potere antitumorale non dipende soltanto dalla possibilità di formare complessi di intercalazione ma anche dalla loro citotossicità, cioè dalla capacità di inibire l’enzima Topoisomerasi II mediante la formazione di complessi ternari stabili DNAintercalante-Topo (Martinez & Chacon-Garcia, 2005).. 12.
(14)
Figura
Documenti correlati
Per il primo sottospazio abbiamo un sistema
¨ La progettazione fisica di un db si propone non solo di fornire delle strutture dati appropriate, ma anche di garantire delle buone performance del database system. ¨
¨ Se ogni record ha esattamente la stessa dimensione (in byte), il file è detto essere costituito da record a lunghezza fissa.. ¨ Se record diversi hanno dimensioni diverse, il file
Il campo di indicizzazione è un campo chiave, detto chiave secondaria, contenente un valore distinto per ogni record nel file dati.. Il campo di indicizzazione è un campo non
¤ Isolation: Una transazione non deve rendere visibili i suoi aggiornamenti ad altre transazioni finché non è committed (responsabilità del sistema per il controllo
¨ Al più una transazione può mantenere il lock su un elemento X; vale a dire che due transazioni non possono accedere allo stesso elemento concorrentemente....
Una transazione non raggiunge il punto di commit finché tutte le sue operazioni di aggiornamento non sono registrate nel log e il log è scritto su disco.. ¨ L’algoritmo è NO-UNDO
¤ Un singolo sito è designato come sito primario il quale fa da coordinatore per la gestione delle transazioni. Communications neteork