INTRODUZIONE
1- Il sistema nervoso centrale (SNC)
Lo sviluppo del sistema nervoso centrale (SNC) è un processo complesso che consiste nella specificazione di molteplici tipi neurali distinti e nella formazione di circuiti funzionali che controllano le attività vitali e comportamentali dell’organismo adulto. Questa struttura estremamente sofisticata origina da una popolazione omogenea di cellule che, precocemente nell’embrione, costituiscono un epitelio pseudostratificato, chiamato neuroectoderma, dal quale si forma la piastra neurale. La parte anteriore della piastra neurale corrisponde alla regione presuntiva del cervello mentre quella più posteriore alla regione presuntiva del midollo spinale. Durante la neurulazione, la piastra neurale inizia a piegarsi ventralmente lungo la linea mediana e a chiudersi dorsalmente per dare origine al tubo neurale, il quale risulterà poi isolato dal restante ectoderma superficiale, che invece formerà l'epidermide. Ancor prima che la parte posteriore del tubo neurale si sia chiusa completamente, nella parte più rostrale si assiste alla comparsa di tre vescicole che rappresentano i primordi di prosencefalo, mesencefalo e rombencefalo. Dal rombencefalo origineranno una serie di strutture nervose con svariate funzioni: dalla parte più anteriore il cervelletto, responsabile della coordinazione motoria, della postura e dell’equilibrio, e, dalla parte più caudale, il ponte e il midollo allungato, sede dei nuclei motori e della componente sensoriale dei nervi cranici V-XII, dei neuroni del sistema reticolare e degli interneuroni deputati alla regolazione di molte attività del sistema nervoso autonomo, come la frequenza cardiaca e il ritmo respiratorio. Inoltre, dalla porzione più dorsale del tubo neurale derivata dalla regione compresa tra la piastra neurale e l’ectoderma a destino epidermico, avranno origine le cellule della cresta neurale cranica. Queste costituiscono una popolazione eterogenea di cellule non ancora differenziate in grado di migrare a livello degli archi faringei e dare origine alla componente sensoriale dei nervi cranici ed alla maggior parte delle cartilagini e delle ossa della faccia e del collo (si vedano per un compendio Köntges e Lumsden, 1996; Le Lievre e Le Douarin, 1975; Baker e Bronner-Fraser, 1997).
2- Induzione neurale e regionalizzazione antero-posteriore
Il differenziamento delle cellule del tessuto nervoso, così come per gli altri tessuti, è la conseguenza di un complesso programma che dirige l’espressione di geni specifici in singole cellule. Il destino differenziativo di una cellula dipende in parte dai segnali a cui questa è esposta, diretta conseguenza della sua localizzazione all’interno dell’embrione, e in parte dall’insieme dei geni espressi dalla cellula stessa (recettori, molecole di trasduzione, fattori di trascrizione) come conseguenza degli eventi induttivi a cui è stata precedentemente sottoposta.I primi esperimenti riguardanti l’induzione neurale risalgono al 1924, quando Hans Speman e Hilde Mangold proposero che negli anfibi il differenziamento della piastra neurale dall’ectoderma dipendesse dai segnali prodotti da un gruppo specializzato di cellule più tardi chiamate organizzatore di Speman (Speman e Mangold, 1924). Il processo di regionalizzazione antero-posteriore della piastra neurale è intimamente connesso con il processo di induzione neurale stesso. Secondo il modello a due segnali proposto da Nieuwkoop, l’induzione neurale comporterebbe anche l’attribuzione di una specificità di posizione lungo l’asse antero-posteriore (Nieuwkoop et al., 1952). Nei vertebrati il sistema nervoso è indotto dalle cellule che daranno origine al mesoderma e che durante la gastrulazione involvono al di sotto della piastra neurale presuntiva. Sono state individuate molecole secrete (follistatin, nogging e chordin) e geni che codificano per fattori di trascrizione (goosecoid, HNF-3β e LIM-1) che hanno la capacità di conferire carattere neurale di tipo anteriore alle cellule ectodermiche. Questi geni sono stati identificati negli anfibi, ma la loro esistenza è stata dimostrata anche in altri vertebrati, rendendoli così ottimi candidati come segnali di attivazione. Gli attivatori neurali agiscono, inoltre, come antagonisti funzionali di fattori appartenenti alla famiglia dei Bone Morphogenetic Proteins (BMPs), i quali invece impediscono all'ectoderma di intraprendere un destino differenziativo neurale.
L’effetto anteriorizzante degli induttori neurali sarebbe contrastato più tardivamente dall’azione posteriorizzante di molecole trasformanti rappresentate da proteine secrete appartenenti sia alla famiglia dei geni Wnt (McGrew et al., 1997; McGrew et al., 1995) che alla famiglia dei geni FGF (McGrew et al., 1997). Anche l'acido retinoico (RA), prodotto principalmente nell'organizzatore, ha la capacità di posteriorizzare il tubo neurale (Hogan et al., 1992; Kraft et al., 1994). Studi effettuati su Xenopus hanno mostrato che il trattamento di embrioni con RA provoca la riduzione delle strutture cerebrali anteriori a favore dell’espansione delle strutture posteriori (Durston et al., 1989). Nel topo un eccesso di RA provoca trasformazioni posteriori
nella parte caudale del cervello e nello scheletro assiale (Kessel e Gruss, 1991; Marschall et al., 1992).
L’azione integrata e coordinata di questi segnali suddivide progressivamente il tubo neurale lungo il suo asse rostro-caudale conferendo alle cellule caratteristiche molecolari specifiche mediante l’espressione differenziale di geni come Emx2, espresso nel prosencefalo, Otx2, espresso nel pro- e mesencefalo, Krox20 e kreisler, espressi nel rombencefalo.
3- La segmentazione del rombencefalo
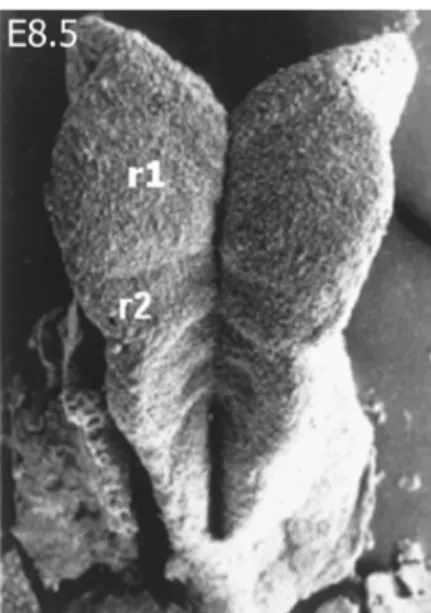
Durante l’embriogenesi molti tessuti sono suddivisi in unità morfologiche ripetute, denominate segmenti, che differenziano in una serie di strutture omologhe simili, benché distinte. Queste strutture ripetute sono fondamentali per determinare l’organizzazione del piano corporeo di molti animali e, da un punto di vista evolutivo, questa suddivisione modulare costituisce la base per la diversificazione e la specializzazione delle diverse parti del corpo, come risulta particolarmente evidente negli artropodi. Nei vertebrati una chiara segmentazione si osserva nel mesoderma che dà origine ai somiti, ma anche a livello del faringe, con la formazione degli archi branchiali, e nel sistema nervoso centrale a livello del rombencefalo. Infatti, durante lo sviluppo precoce, il rombencefalo dei vertebrati è suddiviso transitoriamente in una serie di compartimenti ripetuti, denominati rombomeri (r), che nel topo sono morfologicamente distinguibili tra 8.5 e 10.5 dpc (days post coitum) (Fig 1).
Fig 1. Sviluppo del rombencefalo.
Microfotografia al microscopio elettronico a scansione della regione dorsale di un embrione a 8.5 giorni di gestazione. Si riconosce la suddivisione del rombencefalo in compartimenti.
Questi segmenti anticipano il differenziamento di una serie di strutture, come per esempio i nervi cranici, che lungo l’asse antero-posteriore (A/P) si sviluppano in accordo con questa architettura segmentale. La suddivisione in rombomeri ha quindi un profondo significato funzionale in quanto ciascun rombomero possiede un’identità unica che ne determina il successivo sviluppo. La prova dell’organizzazione metamerica del rombencefalo deriva da studi condotti nel pollo sulla formazione dei nervi cranici con cui è stato dimostrato che i motoneuroni dei nervi trigemino (V), facciale (VII) e glossofaringeo (IX) sono infatti generati rispettivamente a livello di r2, r4, e r6 (Lumsden e Keynes, 1989; Simon e Lumsden, 1993). Inoltre, colorazioni retrograde dei corpi cellulari hanno permesso di mostrare un’organizzazione basata sulla cooperazione di due rombomeri successivi: ciascun nervo è costituito da assoni originati da motoneuroni localizzati in due rombomeri adiacenti (Fortin et al., 1995). La compartimentalizzazione del rombencefalo, oltre a sostenere l’organizzazione neurale, partecipa alla specificazione segmentale delle cellule della cresta neurale che migrano negli archi branchiali contribuendo alla componente sensoriale dei nervi cranici e in parte alle strutture scheletriche della faccia e del collo.
L’osservazione di una precisa organizzazione cellulare del rombencefalo lungo l’asse A/P presuppone che ogni rombomero possa stabilire e mantenere una specifica identità segmentale. Negli ultimi anni sono stati identificati geni che presentano un profilo di espressione ristretto alle regioni rombomeriche, con limiti di espressione che coincidono con il confine di rombomeri adiacenti, come ad esempio Krox-20, kreisler e Gbx2. L’analisi di topi mutanti per questi geni, ottenuti mediante ricombinazione omologa nelle cellule staminali embrionali (ES), ha permesso di indagarne il ruolo durante il processo di segmentazione.
Il gene Krox20 mostra un’espressione segmentale, precedente la formazione morfologica dei rombomeri, ristretta a r3 e r5 (Wilkinson et al., 1989). Topi knockout per il gene Krox20 non presentano il r3 e r5 (Schneider-Maunoury et al., 1997; Schneider-Maunoury et al., 1993; Swiatek e Gridley, 1993). Kreisler è espresso nei territori presuntivi di r5 e r6, anch’esso sin da prima che la segmentazione sia visibile anatomicamente (Cordes e Barsh, 1994) e i topi mutanti per questo gene mostrano una parziale delezione del territorio di r5 e r6 e una perdita della segmentazione morfologica nella regione di r4 e r7 (Frohman et al., 1993; McKay et al., 1994; Moens et al., 1998). Il gene Gbx2 è espresso molto precocemente nel SNC, a partire da 7.5 dpc, e ha un limite anteriore di espressione nella regione tra il mesencefalo e il rombencefalo, denominata istmo (Wassarman et al., 1997). L’inattivazione di questo gene porta ad una riduzione e ad una specificazione anormale della regione
rombencefalica anteriore (r1-r3) e dell’istmo. Grazie a queste caratteristiche peculiari Krox-20, kreisler e Gbx2 sono stati considerati i possibili candidati per il controllo della segmentazione e dell’identità posizionale delle cellule nel rombencefalo.
In realtà, anche il gene Hoxa1, appartenente alla famiglia dei geni Hox (che verrà trattata in dettaglio più avanti) è espresso precocemente sino al confine tra r3 e r4 e la sua espressione diminuisce prima della segmentazione morfologica dei rombomeri (Murphy et al.,1991). Il mutante Hoxa1, mostra forti alterazioni a carico di r4 e r5, che risultano ridotti, e inoltre mostra la perdita del confine con r6 (Carpenter et al., 1993; Mark et al., 1993).
Come conseguenza della segmentazione si ha la formazione dei confini tra rombomeri adiacenti che rappresentano confini di “lineage”, vale a dire che le cellule originate all’interno di un dato rombomero sono confinate al suo interno e non possono migrare nel rombomero adiacente (Fraser et al., 1990). Le basi cellulari per la formazione di questi compartimenti furono suggerite da esperimenti di trapianto nel pollo che indicarono che le cellule di r3 e r5 potevano mescolarsi le une con le altre, ma formavano dei confini molto netti se giustapposte alle cellule di r2, r4 o r6. Questi dati rivelano un’alternanza nelle proprietà cellulari dei rombomeri, come l’adesione cellulare, che potrebbe spiegare perché le cellule di un rombomero si aggreghino preferenzialmente tra di loro piuttosto che con le cellule di compartimenti adiacenti (Guthrie e Lumsden, 1991; Guthrie et al., 1993). Effettivamente è stata osservata una periodicità alternata nell’espressione di molecole coinvolte nell’adesione cellulare che presentano un dominio di espressione ristretto ai rombomeri. Per esempio tra il gruppo dei recettori con attività tirosina-chinasica (RTK) Eph, e dei loro ligandi, ephrins (Xu e Wilkinson, 1997), il gene EphA4 è fortemente espresso in r3 e r5 e debolmente in r2 (Gilardi-Hebenstreit et al., 1992; Nieto et al., 1992). In Xenopus e Danio rerio, o zebrafish, è possibile studiare il ruolo di uno specifico gene mediante aumento di espressione (esperimenti di “gain-of-function”) o silenziamento (esperimenti di “loss-of-function”) del gene stesso. L’iniezione, in embrioni di Xenopus e zebrafish, di mRNA codificante per la forma dominante negativa del recettore EphA4 comporta la migrazione anomala di cellule appartenenti ai rombomeri 3 e 5 nei rombomeri 2, 4 e 6 (Xu et al.,1995).
Sulla base di questi dati è stato proposto un modello per il controllo genetico della segmentazione del rombencefalo (Schneider-Maunoury et al., 1998): il gradiente morfogenetico responsabile della regionalizzazione antero-posteriore precoce del neuroectoderma che compare durante la gastrulazione, induce l’attivazione trascrizionale di una prima serie di geni tra cui buoni candidati sono Gbx2, Krox20,
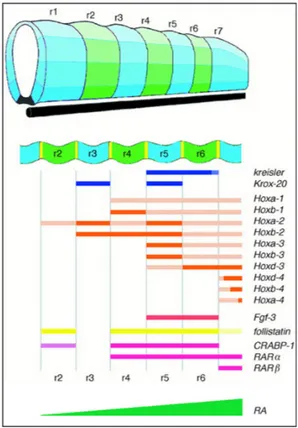
Hoxa1 e kreisler. La loro espressione suddivide il rombencefalo in ampi territori che successivamente, grazie all’espressione di geni che agiscono a valle, mediando la formazione dei confini rombomerici, vengono suddivisi in distinti rombomeri. Una volta che è avvenuta la segmentazione e che i confini dei vari rombomeri sono stati definiti si ha l’espressione di una famiglia di geni responsabili della specificazione dell’identità dei rombomeri, i geni Hox. I geni Hox sono fattori di trascrizione che mostrano un dominio di espressione segmentale nel rombencefalo e conferiscono un valore posizionale lungo l’asse A/P alle cellule di ciascun rombomero integrando le informazioni che derivano dai gradienti morfogenetici precoci, dall’espressione dei geni della segmentazione e da altri geni Hox eventualmente espressi nello stesso segmento (Fig 2).
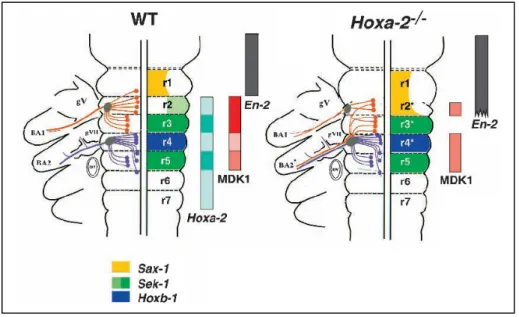
Fig 2. Molecole coinvolte nella segmentazione del rombencefalo.
Nel diagramma è rappresentata l’espressione genica dei fattori coinvolti nella segmentazione e s p e c i f i c a z i o n e d e l l ’ i d e n t i t à segmentale del rombencefalo.
4- I geni Hox
4.1- I geni “homeobox”
I geni della famiglia “homeobox” codificano per un ampio gruppo di fattori di trascrizione contenenti una regione in grado di legare il DNA lunga circa 60 aminoacidi chiamata omeodominio. L’omeodominio presenta un caratteristico motivo di
elica-ansa-elica (“helix-turn-helix”) comune a molte proteine che legano il DNA. Numerosi geni contenenti omeodominio sono espressi durante lo sviluppo embrionale spesso regolandone i processi chiave. Questa sequenza è stata identificata per la prima volta in un particolare gruppo di geni di Drosophila in grado di controllare lo sviluppo del piano corporeo e denominati geni omeotici. Il termine omeotico deriva dall’osservazione che questi geni, se mutati, provocano alterazioni note come trasformazioni omeotiche, in cui una struttura caratteristica di un dato segmento è sostituita da quella normalmente derivata da un segmento di un diverso livello assiale.
4.1.1- I geni omeotici di Drosophila
Drosophila melanogaster, grazie ad una grande quantità di informazioni ottenute da studi di genetica classica, rappresenta un sistema modello ideale per lo studio dei meccanismi che regolano i processi dello sviluppo. Questo modello, nonostante sia un invertebrato, ha avuto un ruolo cruciale per comprendere le basi genetiche dello sviluppo dei vertebrati: un esempio classico è proprio quello che riguarda i geni Hox.
Durante lo sviluppo larvale, il piano corporeo di Drosophila, viene suddiviso in una serie di segmenti che, con il procedere dello sviluppo, acquisiscono identità distinte. Il prodotto dei geni materni, localizzati nell’uovo secondo un preciso ordine spaziale nel corso dell’oogenesi, determinano i principali assi corporei del futuro individuo (si veda per un compendio Patel, 1994) e stabiliscono le informazioni di posizione che attivano una serie gerarchica di geni zigotici, i geni “gap”, i geni “pair-rule” ed i geni “segment polarity” che, a loro volta, determinano la formazione dei segmenti, stabilendone numero e polarità. Successivamente, i geni appartenenti alla famiglia dei geni selettori omeotici (geni HOM), espressi in diverse combinazioni lungo l’asse A/P, stabiliscono l’identità dei segmenti. Mutazioni a carico di questi geni risultano in trasformazioni omeotiche in cui si assiste alla trasformazione della struttura normalmente originata da uno specifico segmento nella struttura derivata da un segmento che si trova in un’altra posizione lungo l’asse A/P. Per esempio, la mutazione del gene Antennapedia provoca la trasformazione delle antenne in un paio di zampe, in quanto un segmento della testa acquisisce l’identità di un segmento più posteriore, mantenendo tuttavia inalterato il numero complessivo dei segmenti (Schneuwely e Gehring, 1985).
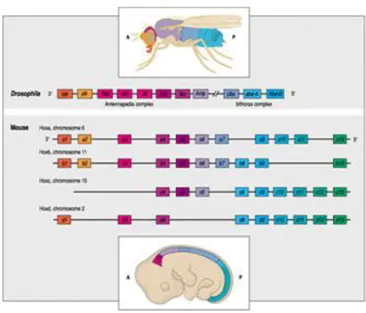
I geni HOM sono raggruppati in due complessi: il complesso Antennapedia (ANT-C) e il complesso Bithorax (BX-C). Il complesso ANT-C comprende cinque geni omeotici: labial (lab) proboscipedia (pb), Deformed (dfb), sex combs reduced (scr) e Antennapedia (Antp) (si veda per un compendio Kaufman et al., 1990), mentre il complesso BX-C ne comprende tre Ultrabithorax (Ubx), abdominal A (abdA) e Abdominal B (AbdB) (Karch et al., 1985; Regulski et al., 1985; Sanchez-Herrero et al., 1985). In seguito all’osservazione della similarità tra i geni omeotici di Drosophila ed altre specie animali si è dato un altro nome ai due complessi, chiamandoli entrambi HOM-C (“HOMeotic Complex”).
4.1.2- I geni omeotici dei vertebrati: i geni Hox
4.1.2.a - Origine e caratteristiche degli Hox
Nei vertebrati tetrapodi i geni omeotici (Hox) sono raggruppati in quattro complessi (HoxA, HoxB, HoxC e HoxD) localizzati su cromosomi distinti. La struttura genomica e l’organizzazione simile a quella dei complessi HOM-C di Drosophila e le profonde omologie nella sequenza amminoacidica delle proteine, hanno suggerito che questa famiglia di geni derivi per amplificazione e duplicazione da un gene ancestrale comune che negli artropodi avrebbe dato origine ai due complessi ANT-C e BX-C. Nei vertebrati, poi, la duplicazione dell’intero complesso ancestrale avrebbe portato alla formazione di quattro complessi indipendenti (Kappen et al., 1989). Questa ipotesi evolutiva è sostenuta dall’identificazione nell’anfiosso Brachiostoma floridae, u n cefalocordato primitivo, di un solo complesso Hox (Garcia-Fernandez e Holland, 1994). Come conseguenza della duplicazione del complesso ancestrale nei vertebrati è possibile individuare geni equivalenti, detti paraloghi, che presentano un elevato livello di omologia. Nel topo sono presenti 39 geni Hox appartenenti a 13 diversi gruppi paraloghi. Non tutti i paraloghi sono rappresentati in ogni complesso in quanto alcuni geni infatti, si sarebbero persi durante l’evoluzione (si veda per un compendio Krumlauf, 1992) (Fig 3).
Fig 3. Evoluzione dei geni Hox. Rappresentazione
schematica del complesso
HOM-C di Drosophila e dei
complessi Hox nei Vertebrati in cui sono evidenziati i gruppi paraloghi.
Una caratteristica peculiare che accomuna i geni Hox è la correlazione tra la loro posizione sul cromosoma ed il corrispondente dominio spaziale e temporale con cui questi sono espressi lungo l’asse A/P dell’embrione. Questa caratteristica viene definita colinearità spaziale e temporale: i geni localizzati più al 3’ sul cromosoma mostrano infatti un’espressione più precoce ed un limite anteriore di espressione più rostrale rispetto ai quelli localizzati più vicino all’estremità 5’ (Lewis, 1978; Dolle et al., 1991; Hunt et al., 1991; Izpisua-Belmonte et al., 1991).
La profonda omologia e le caratteristiche di espressione dei geni Hox sono comuni in molte specie animali e riflettono il grado di conservazione della funzione di queste proteine anche in specie filogeneticamente distanti (Slack et al., 1993). Questi geni condividono infatti un ruolo fondamentale nel controllo della morfogenesi precoce e in particolare nella specificazione dell’identità regionale lungo l’asse A/P embrionale in tutto il regno animale.
4.1.2.b - Espressione dei geni Hox
Il dominio spazio-temporale di espressione dei geni Hox, per l’importante ruolo da essi svolto, è finemente regolato durante lo sviluppo embrionale Nei mammiferi i geni Hox dei quattro gruppi paraloghi presentano domini di espressione che nel rombencefalo mostrano un dominio anteriore di espressione in corrispondenza del confine tra due rombomeri adiacenti, con una periodicità di due segmenti (Gaunt et al., 1989; Gaunt, 1988; Hunt et al., 1991; Murphy e Hill, 1991; Prince e Lumsden, 1994;
Wilkinson et al., 1989). Ad esempio il confine anteriore di espressione di Hoxb2 corrisponde al confine tra r2 ed r3, e quello di Hoxb3 coincide con quello tra r4 ed r5. I domini di espressione dei geni Hox vengono stabiliti precocemente durante lo sviluppo del tubo neurale, prima che la formazione dei rombomeri risulti anatomicamente evidente. Tuttavia, l’espressione di alcuni geni Hox persiste fino a stadi avanzati dello sviluppo embrionale, aprendo l’ipotesi di un loro ruolo nel controllo del differenziamento di popolazioni neuronali di origine rombencefalica. I geni Hox sono espressi anche nelle cellule della cresta neurale cranica (NCC) generate nella regione dorsale del rombencefalo e che successivamente migrano negli archi branchiali del corrispondente livello assiale. Da questa popolazione di cellule hanno origine lo scheletro e le cartilagini della faccia e del collo e la componente sensoriale dei nervi cranici (Hunt et al., 1991; Prince e Lumsden, 1994; Wilkinson et al., 1989). Sembra tuttavia che l’espressione dei geni Hox sia regolata in maniera indipendente nelle NCC e nel tubo neurale (Prince e Lumsden, 1994). Per esempio Hoxa2 è espresso sino al confine tra r1 ed r2, ma non nelle creste neurali che originano da r2 e migrano nel primo arco branchiale, che è popolato da NCC che non esprimono geni Hox. Inoltre Hoxa1, normalmente espresso in r4, non è presente nelle NCC che da questo rombomero migrano nel secondo arco faringeo.
Il controllo coordinato dei domini di espressione dei geni Hox definisce uno specifico “codice Hox” all’interno di ciascun segmento, ovvero una combinazione di distinti geni Hox nei diversi rombomeri e nelle NCC che popolano specifici archi branchiali. Questo codice costituisce un sistema di coordinate spaziali che permette alle cellule di acquisire specifici valori posizionali lungo l’asse A/P e di attuare uno specifico programma differenziativo (Hunt e Krumlauf, 1991; Hunt et al., 1991).
4.1.2.c - Fattori regolatori dell’espressione segmentale dei geni Hox
L’espressione modulare dei geni Hox è finemente regolata per fornire una corretta identità segmentale dei rombomeri. Non sono tuttavia del tutto chiariti i meccanismi che portano all’attivazione dei geni Hox lungo l’asse neurale.
Krox20 e kreisler sono fattori di trascrizione della famiglia “zinc-finger”, e sono buoni geni candidati per questa funzione, in quanto per entrambi è stata dimostrata la funzione di attivazione diretta per alcuni geni Hox. Krox20 regola l’espressione di Hoxa2 e Hoxb2 in r3 e in r5 (Nonchev et al., 1996; Schneider-Maunoury et al., 1993),
mentre Kreisler regola l’espressione di Hoxa3 e Hoxb3 rispettivamente in r5/r6 e in r5 (Manzanares et al., 1997, 1999).
Anche l’acido retinoico (RA) è un buon candidato come regolatore dell’espressione dei geni Hox. Gli effetti dell’RA sono mediati da recettori nucleari (RARs) che legano direttamente sequenze specifiche di DNA localizzate nel promotore di numerosi geni Hox, chiamate elementi di risposta all’acido retinoico (RAREs). I geni Hox presentano, oltre alla colinearità spaziale e temporale descritta in precedenza, anche una colinearità nella sensibilità all’RA: i geni localizzati al 3’ sul cromosoma sono più sensibili all’RA (Simeone et al., 1990). Il trattamento di embrioni di topo con acido retinoico porta ad un’alterazione a carico dell’espressione dei geni Hox e si osserva nelle regioni più anteriori espressione di geni H o x solitamente espressi più posteriormente (Marshall et al.,1992).
Ci sono infine numerose evidenze a favore di un’autoregolazione dei geni Hox. Per esempio, l’elemento responsabile del mantenimento dell’espressione di Hoxb1 in r4 contiene siti per le proteine omeotiche del gruppo paralogo 1 (Popperl et al., 1995): un elemento “enhancer” presente sul promotore di Hoxb1 è in grado di legare Hoxb1 stesso, grazie alla cooperazione di membri della famiglia Exd/Pbx e di attivare un “feedback” positivo (Popperl et al., 1995). Inoltre l’analisi dei doppi mutanti Hoxa1/Hoxb1 ha mostrato che l’induzione di Hoxb1 in r4 è controllata dalla proteina Hoxa1 oltre che dall’acido retinoico. Quindi il promotore di Hoxb1 è controllato sia da un’autoregolazione sia da una regolazione crociata da parte di un altro gene della stessa famiglia (Gavalas et al., 1998; Studer et al., 1998). Queste evidenze sperimentali mostrano quindi che l’autoregolazione e la regolazione incrociata dei geni Hox rappresentano importanti meccanismi per la corretta attivazione e mantenimento della loro espressione in specifiche strutture (Gould et al., 1997; Maconochie et al., 1997).
4.2- Studio dei geni Hox nel topo: knockout e knockin
La possibilità di poter introdurre mutazioni in regioni specifiche del genoma di un organismo costituisce una straordinaria opportunità per studiare la funzione genica. Questo approccio sperimentale è stato applicato con successo allo studio dei meccanismi che regolano i processi dello sviluppo embrionale ed in particolare per comprendere il ruolo di quei geni che ne controllano i processi differenziativi, come i geni Hox.
Le cellule staminali embrionali (ES), isolate per la prima volta da Martin Evans all’inizio degli anni ’80, sono cellule totipotenti in grado di dare origine a tutti i tipi cellulari presenti in un organismo, comprese le cellule della linea germinale, i gameti. Le cellule ES di topo possono essere coltivate e manipolate in vitro senza alterarne le capacità differenziative. Questa possibilità ha costituito la prerogativa fondamentale per la generazione di topi in cui uno o più geni possono essere selettivamente inattivati. In seguito all’introduzione in cellule ES di un vettore a DNA opportunamente ingegnerizzato ed in grado di promuovere eventi di ricombinazione omologa, è possibile modificare la regione codificante dispecifici geni inattivandone la funzione (knockout genico). Le cellule così modificate (ricombinanti o eterozigoti per la mutazione introdotta) possono essere inserite all’interno di un embrione pre-impianto di topo allo stadio di blastocisti dove queste possono integrarsi con la massa cellulare interna dell’embrione ospite da cui può nascere un topo chimerico. Nel caso in cui le cellule ES ricombinanti contribuiscano alla formazione della linea germinale sarà possibile trasmettere alla progenie il carattere modificato in vitro ed ottenere un topo eterozigote che, dopo opportuni incroci, permetterà di ottenere topi knockout omozigoti per la mutazione introdotta.
Impiegando lo stesso approccio sperimentale possono essere generati animali in cui la sequenza codificante di uno specifico gene viene deleta (knockout) e, allo stesso tempo,sostituita con la sequenza codificante di un altro gene, knockin genico. La regione codificante introdotta può essere quella di un gene endogeno, oppure quella di un gene reporter come la proteina verde fluorescente (EGFP), la β-galattosidasi o la fosfatasi alcalina). In questo modo possono essere ottenuti topi in cui la regione codificante del gene introdotto risulta essere sotto il controllo trascrizionale del gene inattivato. Una delle principali applicazioni di questa tecnica permette, grazie alla presenza del gene reporter, di seguire, sia in animali eterozigoti (frequentemente identici al selvatico) sia nel contesto di animali mutanti, il destino differenziativo delle cellule in cui il gene inattivato è normalmente espresso.
4.3- Inattivazione genica tempo e tessuto specifica
Negli ultimi anni sono state messe a punto tecniche per inattivare la funzione di un gene di interesse in maniera tempo e tessuto specifica. Con questo approccio è stato possibile studiare il ruolo tardivo di geni la cui espressione persiste in diverse fasi di sviluppo ma la cui inattivazione (knockout convenzionale) portava ad una letalità
embrionale precoce. In maniera simile, è stato possibile studiare in popolazioni cellulari ristrette il ruolo di geni che mostrano ampi domini di espressione. Questa strategia si basa sull’impiego di un enzima, la ricombinasi Cre, in grado di promuovere eventi di ricombinazione somatica sito-specifica nel genoma di topo per eliminare in maniera controllata la regione contenente il gene di interesse. Il più comune di questi sistemi di ricombinazione è chiamato Cre/loxP.
4.3.1- ll Sistema inducibile Cre/loxP
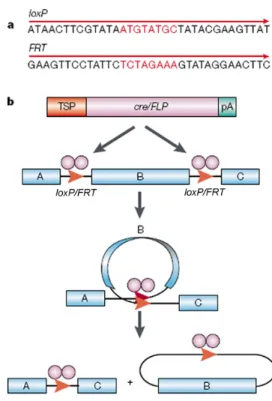
Il sistema Cre/loxP si basa su due elementi fondamentali: la ricombinasi Cre isolata dal fago P1 ed una particolare sequenza di DNA definita sito loxP. La ricombinasi Cre è un enzima che catalizza un evento di ricombinazione conservativa del DNA in seguito al riconoscimento di due siti loxP. I siti loxP sono sequenze di 34 nucleotidi, con estremità palindromiche e con una regione centrale di 8 nucleotidi che conferisce la direzionalità al sito. La posizione sul filamento di DNA ed il verso dei siti loxP determinano il tipo di ricombinazione promosso dalla ricombinasi Cre. Nel caso in cui due siti loxP siano posti in cis, ovvero sullo stesso filamento di DNA, ed orientati nello stesso verso, la ricombinasi Cre è in grado di catalizzare l’excisione del frammento compreso tra i due siti (definito frammento “floxed”) che viene successivamente degradato (Fig 4) La generazione di alleli in cui due siti loxP sono posti ai lati della sequenza codificante del gene di interesse permette di eliminare il gene stesso nelle cellule in cui è attiva la ricombinasi Cre. Normalmente la presenza dei siti loxP ai lati della regione codificante del gene di interesse (allele condizionale, “floxed”) non provoca alterazioni a carico dell’espressione del gene stesso rendendo così gli animali omozigoti per l’allele condizionale indistinguibili dagli animali selvatici. La combinazione di animali omozigoti per un allele condizionale con linee di topi transgenici in cui la ricombinasi Cre è posta sotto il controllo di un promotore tempo e tessuto specifico, permette di ottenere animali in cui il gene in analisi è deleto esclusivamente nelle cellule in cui la ricombinasi Cre è attiva.
Fig 4. Ricombinazione somatica in vivo: sistema Cre/loxP o Flp/FRT.
a) Sequenza nucleotidica dei siti loxP e FRT. b) Rappresentazione schematica del processo di ricombinazione per l’excisione di frammenti di DNA fiancheggiati dalle sequenze loxP o
FRT catalizzato dalle ricombinasi Cre o
Flip.
Esiste un altro sistema inducibile analogo al sistema Cre/loxP che può essere utilizzato per promuovere eventi di ricombinazione somatica sito specifica nel topo. Questo sistema prevede l’utilizzo della ricombinasi F l p, isolata dal lievito Saccharomyces cerevisiae, e di una sequenza specifica di DNA nota come sito FRT.
4.4- Analisi funzionale dei geni Hox
Nel topo l’alterazione mirata del codice Hox, mediante esperimenti di perdita di funzione (ricombinazione omologa nelle cellule embrionali staminali ES (vedi paragrafo 4.2) e mediante esperimenti di espressione ectopica di geni Hox, ha permesso di chiarire il ruolo funzionale dei membri di questa famiglia nella specificazione del rombencefalo, e delle NCC. Negli ultimi anni, con il perfezionamento delle tecniche di inattivazione genica, è aumentato in maniera considerevole il numero di dati derivante dallo studio di alleli nulli per geni Hox ottenuti nel topo (si veda per un compendio Krumlauf, 1994). L’analisi del fenotipo ottenuto dalla generazione di queste linee ha permesso di mostrare la presenza di difetti a carico del sistema nervoso centrale, dei derivati delle NCC, dello scheletro assiale, e degli arti. In alcuni casi i fenotipi osservati, sia negli esperimenti di guadagno che di perdita di funzione, sono riconducibili alle trasformazioni omeotiche osservate in Drosophila. Per esempio la perdita di funzione per H o x c 8, Hoxb4, H o x a 2, H o x d 3 e H o x d 1 3 mostra una
trasformazione omeotica anteriore di alcune strutture derivate dalle creste neurali e dello scheletro assiale (Le Mouellic et al., 1992; Condie e Capecchi, 1993; Dollé et al., 1993; Gendron-Maguire et al., 1993; Ramirez-Solis et al., 1993; Rijli et al., 1993), mentre si ha una trasformazione posteriore nella perdita di funzione per Hoxa5 e Hoxa11 (Jeannottee et al., 1993; Small e Potter, 1993). Mutazioni per perdita di funzione di Hoxa1 e Hoxa3 non mostrano invece trasformazioni omeotiche vere e proprie ma diverse alterazioni a livello dei derivati delle creste neurali craniche (Chisaka e Capecchi, 1991; Lufkin et al., 1991; Chisaka et al., 1992). Infine esperimenti di guadagno di funzione per i geni Hoxc6 e Hoxc8 risultano in trasformazioni omeotiche anteriori (Jegalian e DeRobertis, 1992; Pollok et al., 1992). Viceversa, nel caso dei geni Hoxa7 e Hoxd4, si assiste a trasformazioni omeotiche posteriori (Balling et al., 1989; Kessel et al., 1990; Lufkin et al., 1992).
Oltre ad alterazioni riconducibili a trasformazioni omeotiche classiche nel topo si osservano anche fenotipi che risultano essere meno severi di quanto atteso sulla base di un modello di espressione di tipo segmentale. Ciò può essere spiegato dall’effetto compensatorio di geni paraloghi (ridondanza genica) e dalla dominanza funzionale dei geni posteriori su quelli più anteriori espressi nello stesso dominio. Tuttavia, non è possibile formulare una regola universale in grado di prevedere il fenotipo che risulta da alterazioni del codice Hox: molte strutture, anziché cambiare identità vengono alterate o trasformate solo parzialmente. Questo potrebbe essere spiegato dall’azione combinata di più geni per conferire la corretta specificazione di una data struttura, per cui l’alterazione di uno solo di questi non è sufficiente a promuovere una trasformazione omeotica completa.
4.5- I geni Hox e la specificazione dell’identità segmentale
Il confronto tra i domini di espressione dei geni Hox evidenzia una stretta corrispondenza con regioni ben definite lungo tutto l’asse A/P dell’embrione suggerendo che questi geni siano coinvolti nella specificazione dell’identità segmentale durante lo sviluppo. La possibilità di generare animali mutanti per i geni Hox (si veda paragrafo 4.2) ha fornito uno straordinario sistema per comprendere le basi molecolari ed il ruolo funzionale dei componenti di questa numerosa famiglia genica nel processo di specificazione dell’identità segmentale durante lo sviluppo embrionale.
Nel mesoderma dei somiti, l’espressione modulare dei geni Hox, ha un ruolo nel determinare il numero e il tipo di vertebra che da essi origineranno in accordo con il
livello assiale. La perdita di funzione del gene Hoxc8, espresso normalmente nella regione toracica, porta alla trasformazione omeotica della prima vertebra lombare in una vertebra toracica (Le Mouellic et al., 1992). Un altro esempio è dato dal knockout di Hoxd11 che risulta nella trasformazione delle vertebre sacrali in quelle lombari (Favier et al., 1995). Infine l’eliminazione del gene Hoxb4, espresso normalmente nel mesoderma che darà origine alla seconda vertebra cervicale (epistrofeo) ma non a livello del somite da cui originano le cellule della prima vertebra cervicale (atlante), risulta nella trasformazione omeotica dell’epistrofeo in un secondo atlante (si veda per un compendio Duboule 1995).
Nel sistema nervoso centrale il rombencefalo, per la sua caratteristica organizzazione in segmenti e per la presenza di uno specifico codice Hox a livello dei singoli rombomeri e delle NCC, costituisce un modello particolarmente utile per lo studio dei meccanismi che regolano la specificazione del “pattern” A/P. Embrioni di topo recanti mutazioni per Hoxa1 (Carpenter et al., 1993; Dolle et al., 1993; Mark et al., 1993), Hoxb1 (Goddard et al., 1996; Studer et al., 1996), Hoxb2 (Barrow e Capecchi, 1996) e Hoxa2 (Gavalas et al., 1997) hanno messo in evidenza alterazioni a carico dello sviluppo del rombencefalo già a partire da stadi molto precoci dell’embriogenesi. Quest’ultimo rappresenta un caso particolarmente interessante che verrà trattato in dettaglio nel paragrafo successivo. Il knockout del gene Hoxa1 mostra alterazioni nella segmentazione del rombencefalo ed i territori dei rombomeri 4 e 5 sono drammaticamente ridotti andando a fondersi caudalmente con r6 per dare origine ad un territorio in cui non è più possibile individuare la normale segmentazione rombomerica (Dolle et al., 1993; Mark et al., 1993). Queste alterazioni sono all’origine di un anomalo sviluppo dei motoneuroni dei nervi abducente e facciale (VI e VII nervo cranico) ed una grave alterazione a carico della formazione dell’orecchio interno il cui normale sviluppo procede in seguito a segnali induttivi emanati da r4-6 (Carpenter et al., 1993; Mark et al., 1993; Pasqualetti et al., 2001). I topi mutanti per Hoxb1 non mostrano anomalie nel processo di segmentazione quanto piuttosto presentano alterazioni a carico della specificazione dell’identità rombomerica in particolare del rombomero 4. In embrioni mutanti Hoxb1-/- infatti analisi molecolari indicano che l’espressione di marcatori specifici per la regione presuntiva di questo rombomero, come il recettore tirosin-chinasico EphA2, non è alterata. Tuttavia è stato possibile mettere in evidenza l’espressione ectopica di marcatori di r2 a livello di r4 e questo suggerisce che nei mutanti Hoxb1 si abbia una parziale trasformazione omeotica di r4 in r2 (Studer et al., 1996). In accordo con questa ipotesi la componente motoria del nervo facciale (VII) di r4 migra in modo errato e riflette i movimenti tipici dei neuroni del
trigemino (V) specifici di r2 (Goddard et al., 1996; Studer et al., 1996). Hoxb1 risulta quindi importante nella specificazione dell’identità di r4 e per il suo mantenimento. L’analisi dei doppi mutanti Hoxa1/Hoxb1 (Gavalas et al., 1998; Studer et al., 1998) ha mostrato che la presenza di un meccanismo di autoregolazione di Hoxb1 sul suo stesso promotore e che per il mantenimento della sua trascrizione Hoxa1 ha un ruolo chiave Questo tipo di regolazione è necessaria affinché le cellule di r4 acquisiscano un destino che si mantenga fino a stadi tardivi dello sviluppo (Popperl et al., 1995; Studer et al., 1998).
I topi mutanti per Hoxb2 mostrano alterazioni a carico della componente motoria del VII nervo cranico (facciale) (Barrow e Capecchi, 1996). Mediante esperimenti di marcatura retrograda con DiI è stato infatti possibile mettere in evidenza una forte riduzione della componente motoria di questo nervo (Davenne et al.,1999). Il fenotipo osservato in assenza di Hoxb2 è da imputarsi alla perdita di espressione di Hoxb1 in r4 dimostrando che Hoxb2 è necessario per il mantenimento della sua espressione (Barrow e Capecchi, 1996). Inoltre, anche se nel mutante Hoxb2 non sono state osservate alterazioni a carico del processo di segmentazione del rombencefalo l’analisi del fenotipo nel doppio mutante Hoxa2/Hoxb2 ha messo in evidenza che questi due geni cooperano nel determinare i confini rombomerici di r3-r5.
4.6- Il gene Hoxa2
In genere l’interpretazione del fenotipo in animali mutanti per specifici geni Hox è ostacolata dalla presenza di altri geni appartenenti a questa famiglia che presentano “pattern” di espressione sovrapposti a quello del gene studiato. Hoxa2 è l’unico gene Hox espresso in r2 e, assieme a Hoxb2, nelle NCC del secondo arco branchiale che originano da r4 (Krumlauf, 1993; Prince e Lumsden, 1994; Mark et al., 1995). Per questo motivo con Hoxa2 si ha l’opportunità di studiare il ruolo di un gene Hox nel processo di specificazione dell’identità rombomerica e delle cellule della cresta neurale cranica in quanto la sua eliminazione porta alla generazione di territori che non esprimono altri geni della stessa famiglia.
4.6.1- Hoxa2 è il gene selettore dell’identità morfogenetica delle NCC del secondo arco branchiale
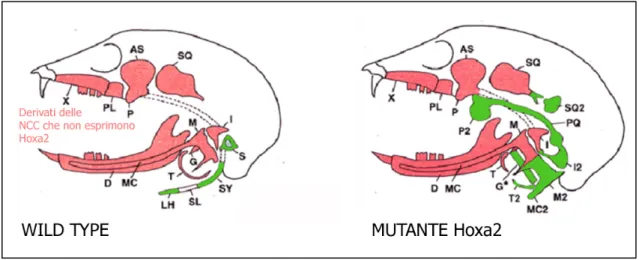
La componente scheletogenica delle cellule della cresta del secondo arco branchiale dà normalmente origine ad alcuni elementi scheletrici dell’orecchio medio come la staffa ed il processo stiloideo. Nel topo mutante per Hoxa2 queste strutture risultano assenti e si assiste ad una trasformazione omeotica a specchio dei derivati scheletrici delle NCC di r4, esprimenti Hoxa2, nei derivati scheletrici che normalmente si sviluppano nel primo arco branchiale e che originano dalle NCC di r1 e r2 (Gendron-Maguire et al., 1993; Rijli et al., 1993) (Fig 5). I risultati ottenuti dall’analisi degli embrioni mutanti per Hoxa2 hanno permesso di stabilire che, in maniera analoga ai geni selettori omeotici di Drosophila, questo gene funziona come gene selettore per il programma differenziativo del secondo arco branchiale (Rijli et al., 1993).
Fig 5. Rappresentazione schematica delle alterazioni riscontrate nello scheletro craniofacciale del topo mutante Hoxa2-/-. Sono mostrati gli elementi scheletrici derivati
dal primo arco branchiale (in rosa) e dal secondo arco branchiale (in verde) in un topo “wild type” e in un topo mutante per Hoxa2. Nel mutante si osserva una trasformazione omeotica a specchio degli elementi scheletrici derivati dal secondo arco branchiale, in elementi caratteristici del primo arco branchiale.
Esperimenti in Xenopus in cui Hoxa2 è stato attivato in maniera ectopica a livello del primo arco branchiale, hanno dimostrato come questo gene sia sufficiente a indurre una trasformazione omeotica degli elementi scheletrici del primo arco verso quelli caratteristici del secondo (Pasqualetti et al., 2000). Questo fenotipo, opposto a quello ottenuto nei topi knockout, mostra che nei vertebrati Hoxa2 svolge un ruolo nel
conferire alle NCC un’identità tipica del secondo arco branchiale, indicando una conservazione funzionale di Hoxa2 nell’evoluzione (Pasqualetti et al., 2000).
4.6.2- Hoxa2 stabilisce l’identità segmentale di r2 e r3
Nel mutante Hoxa2, a differenza di quanto osservato con Hoxa1, non si assiste ad alterazioni a carico della normale segmentazione del rombencefalo in quanto l’espressione dei marcatori Krox20, H o x b 2 , Hoxb1 e Hoxa3 non risulta alterata (Gendron-Maguire et al., 1993; Rijli et al., 1993). Gavalas e collaboratori hanno tuttavia dimostrato che in embrioni a stadio 10.5 dpc mutanti per Hoxa2 sono riscontrabili alterazioni molecolari a carico dei rombomeri 2 e 3 (Gavalas et al, 1997). Utilizzando marcatori della regione di r1, come En2 e Sax1, e r2 come EphA4, è stata dimostrata, nel mutante Hoxa2, una trasformazione dell’identità delle cellule di r2 in quella tipica di r1. Inoltre, come suggerito dalla perdita di espressione del marcatore molecolare PLZF, si osserva la perdita del confine tra i rombomeri 1 e 2 e, parzialmente, di quello tra r2 e r3 (Gavalas et al., 1997). L’analisi dettagliata di topi mutanti Hoxa2 ha rivelato un importante ruolo di questo gene nello sviluppo e nell’organizzazione di gruppi neuronali derivanti da r2 ed r3 (Barrow e Capecchi, 1996; Davenne et al., 1999; Gavalas et al., 1997). La traiettoria assonale dei motoneuroni trigeminali risulta alterata come messo in evidenza mediante esperimenti di marcatura retrograda in cui sono stati utilizzati DiI e DiO. I motoneuroni del nervo trigemino (V), originano normalmente in r2 e r3 e innervano il primo arco branchiale con i loro assoni che fuoriescono dal rombencefalo in corrispondenza di r2; in maniera analoga, i motoneuroni del nervo facciale (VII), originano in r4 e, attraverso questo stesso rombomero, innervano il secondo arco branchiale. Nel mutante Hoxa2 gli assoni di una sottopopolazione di motoneuroni trigeminali fuoriescono in corrispondenza di r4, innervando il secondo arco branchiale (Gavalas et al., 1997) (Fig 6). Le traiettorie degli assoni dei motoneuroni sono guidate da segnali attrattivi in corrispondenza delle regioni in cui i nervi cranici fuoriescono dal rombencefalo, dove le cellule della cresta neurale formano una struttura denominata “exit point”. E’ stato suggerito che Hoxa2 possa avere un ruolo nel conferire identità alle cellule delle creste neurali che migrano dal rombomero 4 per formare l’ “exit point” del VII nervo cranico, in maniera analoga a quanto osservato per le NCC scheletogeniche del secondo arco branchiale. In assenza di Hoxa2 il punto di uscita delle fibre motorie del nervo facciale presenta una parziale trasformazione della sua identità (r4) in quella tipica dell’ “exit point” del nervo trigemino (r2) attraendo quindi
parte degli assoni dei motoneuroni trigeminali che escono normalmente in corrispondenza del rombomero 2.
Fig 6. Riassunto schematico delle alterazioni riscontrate nel rombencefalo del topo mutante Hoxa2-/- a 10,5 dpc. Nel mutante
alcuni assoni del nervo trigemino fuoriescono in maniera anomala in corrispondenza del rombomero 4 da cui normalmente fuoriescono solo gli assoni del nervo facciale.
L’inattivazione di Hoxa2 provoca l’alterazione dei derivati di r3, sia da un punto di vista morfologico che molecolare, anche se in misura minore di quanto avviene per r2, presumibilmente a causa del fatto che in r3 Hoxa2 è co-espresso con il suo paralogo Hoxb2 (Krumlauf, 1993; Gavalas et al., 1997; Davenne et al., 1999). Studi su topi doppi mutanti per Hoxa2 e Hoxb2 hanno infatti messo in luce un’attività sinergica di questi due geni per la formazione di r3 e per la specificazione del pattern dorso ventrale della porzione rostrale del rombencefalo. (Davenne et al., 1999).
Dalla parte più anteriore del rombencefalo (rombomero 1), in cui non è presente espressione di geni Hox, originano i precursori delle cellule dello strato granulare del cervelletto (Marin e Puelles, 1995). Questi precursori migrano anteriormente in maniera tangenziale andando a disporsi nella zona superficiale dell’abbozzo del cervelletto per formare l’EGL (“External Granular Layer”, strato granulare esterno). Nel mutante Hoxa2, si osserva un ispessimento dello strato granulare esterno del cervelletto. L’interpretazione di questo fenotipo è stata attribuita alla perdita del confine tra r1 e r2 ed alla parziale trasformazione dell’identità di r2 in r1. Ciò avrebbe come conseguenza
una anomala migrazione delle cellule di r2 che, in assenza di Hoxa2, contribuiscono a formare lo strato granulare esterno (Galavas et al.,1997). Nel mutante Hoxa2 si osserva inoltre una forte riduzione della porzione più anteriore del nucleo cocleare, un nucleo simmetrico pari responsabile dell’integrazione delle informazioni uditive, che ha un’origine multisegmentale in quanto deriva da precursori che sono generati nei rombomeri da 2 a 5 (Farago et al., 2006). Non è comunque chiaro se in assenza di Hoxa2 si verifichi una perdita dei neuroni del nucleo stesso in seguito all’alterazione dell’identità dei suoi precursori in r2 e r3 oppure se essi siano reclutati da altre strutture anteriori a causa della perdita del codice Hox (Gavalas et al., 1997).
4.6.3- Strumenti genetici per lo studio del ruolo di Hoxa2
I dati ottenuti fino ad oggi relativi allo studio del ruolo di Hoxa2 sono stati ottenuti dall’analisi di una linea knockout convenzionale. Recentemente, sono stati generati nuovi alleli per questo gene con lo scopo di studiare più in dettaglio alcuni aspetti del ruolo funzionale di Hoxa2. In particolare, sono state generate, e sono disponibili nel laboratorio dell’Unità di Biologia Cellulare e dello Sviluppo, tre linee di topo. La prima linea (Hoxa2EGFP) è rappresentata da un allele knockin in cui il gene
reporter EGFP è stato sostituito alla regione codificante di Hoxa2 (Pasqualetti et al., 2002). Una seconda linea knockin (Hoxa2/τLacZ) è data da un allele in cui la regione codificante di Hoxa2 è stata sostituita con quella del gene reporter β-galattosidasi coniugato con la proteina τ. Una terza linea (Hoxa2flox) consiste invece in un allele condizionale dove due siti loxP sono stati posti ai lati della regione codificante del gene Hoxa2 (Ren et al., 2002; Santagati et al., 2005).
4.6.3.a- Linea knockin Hoxa2EGFP
La generazione di una linea knockin in cui la proteina verde fluorescente (EGFP) sostituisce uno specifico locus genico costituisce uno strumento genetico molto utile per marcare e confrontare in vivo le cellule che normalmente esprimono il gene di interesse sia in un contesto eterozigote, che non mostri anomalie rispetto al fenotipo selvatico, sia mutante per il gene stesso. Questo approccio offre quindi la possibilità di visualizzare direttamente eventuali fenotipi anormali.
Poiché negli animali eterozigoti per la mutazione Hoxa2 non sono riscontrabili differenze fenotipiche rispetto ad animali selvatici, recentemente è stato generato un allele knockin Hoxa2/EGFP (Hoxa2EGFP(loxP-neo-loxP)). Una peculiarità di questo allele è
dato dalla possibilità di promuovere l’espressione del gene reporter EGFP in maniera condizionale nelle cellule che normalmente esprimono Hoxa2, in seguito ad un evento di ricombinazione somatica mediata dalla ricombinasi Cre (Pasqualetti et al., 2002). In questa linea la regione codificante della EGFP, che è stata sostituita alla regione codificante di Hoxa2, risulta sotto il controllo trascrizionale dello stesso Hoxa2. In questa maniera la presenza di fluorescenza riflette l’espressione di Hoxa2. In questo allele knockin tuttavia, per la presenza di una cassetta di DNA fiancheggiata da siti loxP a valle della sequenza codificante del gene reporter con la capacità di silenziare il promotore di Hoxa2, non si ha espressione di EGFP in quanto L’incrocio della linea Hoxa2EGFP(loxP-neo-loxP) con una linea transgenica che esprime la ricombinasi Cre, provoca, nelle cellule in cui Cre è attiva, con l’excisione della cassetta di DNA, la derepressione trascrizionale della EGFP nelle cellule in cui Hoxa2 è normalmente espresso (Pasqualetti et al., 2002). In particolare, a seconda del promotore che controlla l’espressione di Cre, è possibile visualizzare distinte sottopopolazioni cellulari all’interno del dominio di espressione di Hoxa2. L’incrocio con la linea r2/Cre (Pasqualetti et al., 2002), in cui la ricombinasi Cre è attiva nel rombomero 2, promuove l’espressione di EGFP solo nelle cellule di r2 che esprimono normalmente Hoxa2. L’incrocio con una linea transgenica in cui la ricombinasi Cre è espressa in maniera ubiquitaria (CMV/Cre), porta alla generazione di una nuova linea Hoxa2EGFP in cui la
trascrizione della EGFP è attivata in tutto il dominio di Hoxa2 (Pasqualetti et al., 2002). Con questa linea è stato possibile mettere in evidenza che l’espressione di EGFP persiste negli embrioni Hoxa2EGFP fino a stadi tardivi di sviluppo e che in alcuni
distretti rimane visibile fino ad alcuni giorni dopo la nascita (Fig 7). La persistenza della fluorescenza suggerisce che il gene Hoxa2, la cui funzione è stata principalmente indagata in eventi precoci dello sviluppo embrionale, possa essere espresso e quindi avere un ruolo anche a stadi successivi, addirittura post-natali. Tuttavia la persistenza della proteina del gene reporter potrebbe essere dovuta alla sua elevata stabilità. Infatti la fluorescenza potrebbe essere ancora visibile anche se il promotore di Hoxa2, che ne regola l’espressione, a questi stadi non fosse più attivo. Inoltre la possibilità di marcare in maniera selettiva nel sistema nervoso centrale i neuroni che esprimono Hoxa2, ha permesso di individuare, nel contesto di animali mutanti per questo gene, alterazioni a carico di distinte strutture del rombecefalo. In particolare è stato possibile mettere in evidenza alterazioni a carico del nucleo pontino, che presenta un elevato livello di
espressione del gene reporter ed il cui sviluppo prosegue fino a stadi post-natali. Questa linea rappresenta quindi un ottimo strumento genetico che ha permesso di formulare l’ipotesi di un potenziale ruolo tardivo di Hoxa2 nello sviluppo del rombencefalo.
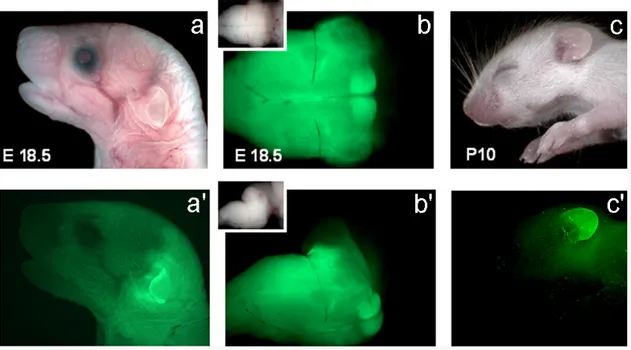
Fig 7. Espressione della EGFP nella linea knockin Hoxa2EGFP. In a) è mostrata la
fotografia in campo chiaro di un topo eterozigote a 18.5 dpc. In a’) la stessa immagine in fluorescenza mostra l’espressione della proteina verde fluorescente a livello dell’orecchio. (b,b’) Espressione di EGFP nel dominio di Hoxa2 nell’encefalo in visione ventrale e laterale; si può osservare un elevato livello di espressione in corrispondenza del nucleo pontino. (c,c’) A 10 giorni dopo la nascita l’espressione di EGFP persiste negli embrioni Hoxa2EGFP a livello dell’orecchio esterno.
4.6.3.b- Linea knockin Hoxa2/τLacZ
L’allele knockin Hoxa2/τLacZ presentala regione codificante di Hoxa2 sostituita da quella del gene reporter LacZ coniugato con la proteina tau (τ), una proteina del citoscheletro assonale, che ne promuove il trasporto lungo i processi assonali. Per la modalità con cui è stato preparato il costrutto di “targeting” impiegato nella generazione dell’allele knockin, il gene reporter τLacZ è espresso in una sottopopolazione dei neuroni che normalmente esprimono Hoxa2, come per esempio i neuroni del nucleo pontino. Questa linea si è rivelata quindi molto utile per visualizzare, durante lo sviluppo del rombencefalo, la formazione del nucleo pontino e delle sue proiezioni assonali sia nel contesto di animali eterozigoti che mutanti Hoxa2-/-.
4.6.3.c- Linea mutante condizionale Hoxa2 (Hoxa2flox)
La mutagenesi condizionale risulta particolarmente utile per lo studio di geni che, come Hoxa2, presentano ampi domini di espressione in quanto offre la possibilità di analizzarne il ruolo in distinte popolazioni cellulari. Recentemente è stato generato un allele condizionale per Hoxa2 (Hoxa2flox) in cui è stato inserito, a monte e a valle della regione codificante del gene, un sito loxP (Ren et al., 2002).Gli animali omozigoti per l’allele “floxed” (Hoxa2flox-/-) non presentano alterazioni fenotipiche riconducibili
all’introduzione dei siti loxP nel locus di Hoxa2. La combinazione della linea condizionale Hoxa2flox con linee transgeniche Cre che esprimono la ricombinasi in
maniera tessuto specifca permette di analizzare la funzione di Hoxa2 in maniera dettagliata. Infatti solamente nelle cellule in cui si esprime la ricombinasi Cre, viene promossa l’excisione della regione codificante di Hoxa2, abrogandone la funzione. Per esempio l’impiego della linea transgenica Wnt1/Cre (Danielian et al., 1998), in combinazione con l’allele condizionale, permette di eliminare Hoxa2 selettivamente nei precursori neurali che derivano dall’epitelio germinativo da cui hanno origine i neuroni del nucleo pontino (“rhombic lip”) mantenendo tuttavia la normale funzione di Hoxa2 nelle restanti strutture del rombencefalo. Questo strumento genetico rappresenta quindi un ottimo sistema per caratterizzare la funzione di Hoxa2 nei meccanismi che regolano lo sviluppo dei neuroni che danno origine al nucleo pontino.
5- Il sistema precerebellare
Nei vertebrati il cervelletto svolge un ruolo di primaria importanza nella coordinazione del movimento, nel controllo dell’equilibrio, del movimento degli occhi, dell’apprendimento motorio, e, verosimilmente, sembra anche essere implicato in funzioni cognitive (Mauk et al., 2000; Gao et al., 1996; Schmahmann, 1997). Le informazioni dirette al cervelletto che provengono dalla corteccia cerebrale e dal sistema somatosensoriale passano attraverso una serie di nuclei che nel loro insieme costituiscono il sistema precerebellare. Una volta elaborate dalla corteccia cerebellare, principalmente costituita dalle cellule granulari e del Purkinje, queste informazioni tornano al sistema nervoso centrale attraverso i tre nuclei profondi del cervelletto, dentato, interposito e del fastigio. Il sistema precerebellare è formato da cinque nuclei localizzati nel tronco dell’encefalo: il nucleo pontino (PN) ed il nucleo reticolo tegmentale (RTN), localizzati nella regione ventrale del ponte, il nucleo reticolare
laterale (LRN), il nucleo cuneato esterno (ECN) ed il nucleo dell’oliva inferiore (ION), localizzati nel midollo allungato (si veda per un compendio Altman e Bayer, 1997). Dal nucleo dell’oliva inferiore hanno origine le fibre rampicanti, che prendono contatto con le cellule del Purkinje nello strato molecolare del cervelletto. Gli altri quattro nuclei precerebellari complessivamente danno origine alle fibre muscoidi che proiettano alle cellule dei granuli (Altman e Bayer, 1987; Brodal e Bjaalie, 1992; Sotelo e Chedotal, 1997).
5.1- Origine del sistema precerebellare: il “rhombic lip”
His (1891) per la prima volta ipotizzò che il margine più dorsale del rombencefalo embrionale, denominato “rhombic lip”, fosse la regione del tubo neurale da cui originano i neuroni del sistema precerebellare e dello strato granulare esterno del cervelletto (EGL) da cui deriva lo strato delle cellule granulari. Successivamente, evidenze indirette a favore di questa ipotesi sono state ottenute mediante esperimenti di ablazione e marcature con perossidasi (Harkmark, 1954; Bourrat e Sotelo, 1990). Il “rhombic lip” è un epitelio germinativo specializzato del rombencefalo che durante l’embriogenesi origina molteplici tipi neurali che contribuiscono alla formazione di distinte strutture del sistema nervoso centrale. E’ noto che la parte più rostrale di questa struttura, dà origine non solo ai precursori delle cellule granulari del cervelletto (Alder et al., 1996; Hallonet et al., 1990; Zhang e Goldman, 1996), ma anche ad alcuni nuclei cerebellari profondi (nucleo dentato, nucleo del fastigio e nucleo interposito) (Wang et al., 2005; Machold et al. ,2005; Fink et al., 2006). Recentemente, mediante esperimenti di “fate mapping” nel topo, è stato dimostrato da diversi autori che dalla porzione del “rhombic lip” compresa tra i rombomeri 2 e 5 deriva il complesso dei nuclei cocleari responsabile dell’elaborazione delle informazioni uditive provenienti dalla coclea (Wang et al., 2005; Farago et al., 2006). Con lo stesso approccio sperimentale è stato dimostrato che il “rhombic lip” dei rombomeri più posteriori (r6-r8) contribuisce alla formazione del nucleo del ponte, del reticolo tegmentale e degli altri nuclei del sistema precerebellare localizzati nella porzione più caudale del rombencefalo (Farago et al., 2006) (Fig 8).
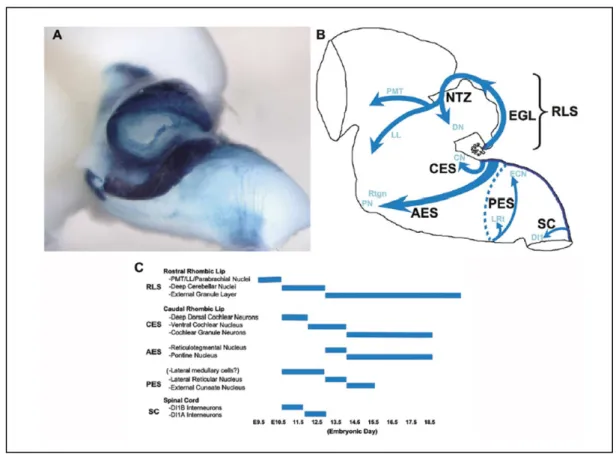
Fig 8. Schema riassuntivo delle vie di migrazione dei derivati neuronali del “rhombic lip”. A) Preparazione “whole mount” in visione laterale del rombencefalo di
un embrione di topo a 14dpc. In blu sono evidenziati i derivati neuronali del “rhombic lip” in migrazione che esprimono il gene Math1. B) Rappresentazione schematica delle strie migratorie dei neuroni derivati dal “rhombic lip”. C) Strutture derivate dal “rhombic lip” e progressione temporale del loro sviluppo (Modificata da Wang et al. 2005).
5.2- La migrazione dei precursori precerebellari
I precursori dei nuclei precerebellari, dal “rhombic lip”, migrano verso la loro destinazione finale nella porzione ventrale dl rombencefalo circumnavigando la regione ventro-laterale del midollo allungato ordinati in strie cellulari compatte (si veda per un compendio Sotelo, 2004). Sono state distinte tre diverse vie migratorie: la via extramurale anteriore (aes) intrapresa dai precursori del nucleo pontino e reticolo-tegmentale che migrano rostralmente passando immediatamente al di sotto della superficie piale fino a raggiungere il ponte ventrale sul lato ipsilaterale in corrispondenza del territorio derivato dai rombomeri 4 e 5 (Bourrat e Sotelo, 1988 e 1990; Marín e Puelles, 1995); la via extramurale posteriore (pes) che, attraverso una migrazione subpiale diretta al midollo allungato controlaterale, porta alla formazione dei
nuclei cuneato esterno e reticolo-laterale (Altman e Bayer, 1987; Bourrat e Sotelo, 1988, 1990); infine le cellule che danno origine al nucleo dell’oliva inferiore seguono una via di migrazione intramurale attraversando in profondità il parenchima per localizzarsi nella porzione ipsilaterale ventrale del midollo allungato (Bourrat e Sotelo, 1988, 1990).
I precursori dei nuclei precerebellari hanno origine a diversi stadi di sviluppo e, dopo aver raggiunto la loro destinazione finale nella porzione ventrale del rombencefalo, iniziano ad emettere le proiezioni assonali al cervelletto. Nel topo, i neuroni dell’oliva inferiore sono generati tra 10.5 e 11.5 dpc, e raggiungono la loro destinazione finale a 13.5 dpc (de Diego et al., 2002). Contemporaneamente si ha anche la formazione del nucleo reticolato laterale e del nucleo cuneato esterno (de Diego et al., 2002). Al contrario, i neuroni del nucleo pontino sono generati in una finestra temporale più ampia compresa tra il dodicesimo ed il sedicesimo giorno di gestazione raggiungendo il ponte ventrale tra 17.5 e 18.5 dpc. Poco prima della nascita, si ha la comparsa dei primi processi assonali dei neuroni del nucleo pontino che formeranno le fibre muscoidi, un processo che si protrae fino a diversi giorni dopo la nascita (Taber-Pierce, 1966) (Fig 9).
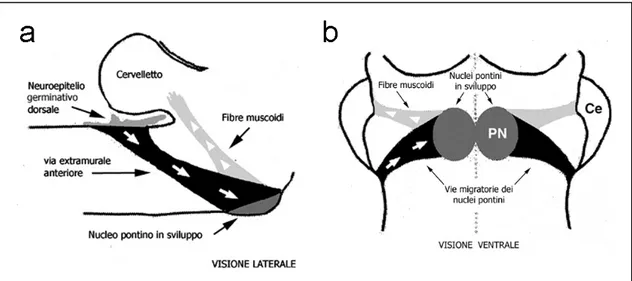
Fig 9. Sviluppo del nucleo pontino. Rappresentazione schematica della regione del
ponte in visione laterale (a) e ventrale (b). In nero è raffigurata la via di migrazione dorso-ventrale dei precursori del nucleo pontino; in grigio le proiezioni assonali che formano le fibre muscoidi. PN, nucleo pontino; Ce, cervelletto.
5.3- Alterazione della migrazione dei precursori pontini nel mutante Hoxa2
La disponibilità delle linee knockin descritte precedentemente ha permesso di analizzare alcuni aspetti dello sviluppo del rombencefalo sia in animali eterozigoti che nei mutanti Hoxa2-/-. La diretta visualizzazione della EGFP nella linea Hoxa2EGFP e
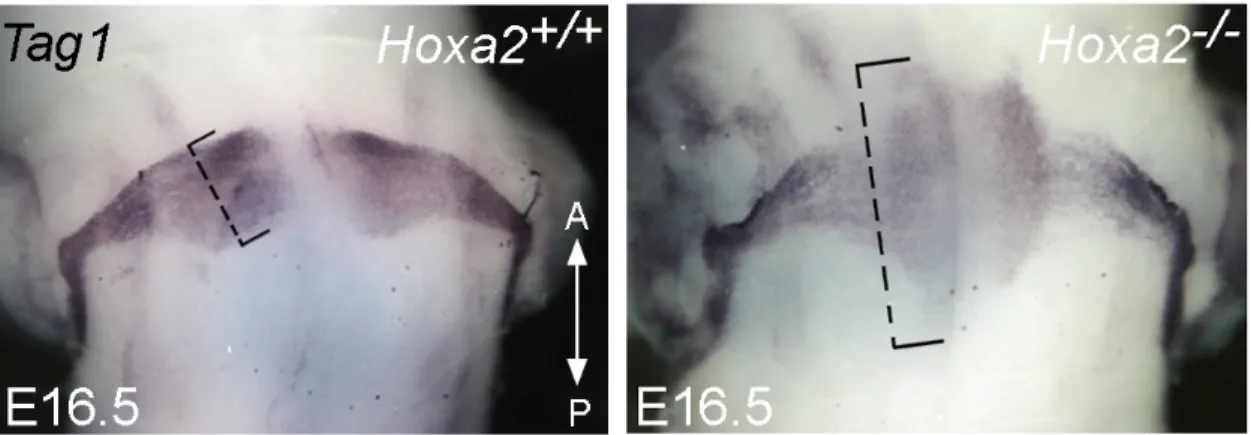
colorazioni Xgal effettuate su encefali di animali Hoxa2/τLacZ hanno messo in evidenza una malformazione a carico della formazione del nucleo pontino negli animali mutanti, Hoxa2-/-. Sin dagli stadi in cui i suoi precursori iniziano la migrazione verso la regione ventrale del ponte si può osservare una disorganizzazione consistente a livello del fronte del fascio migratorio. Come conseguenza i neuroni che daranno origine al nucleo del ponte si dispongono su un territorio molto più esteso in senso rostro-caudale rispetto a quanto si osservi negli embrioni eterozigoti. Questa osservazione è stata confermata effettuando esperimenti di ibridazione in situ con un marcatore molecolare specifico dei neuroni del nucleo pontino Tag1 che codifica per un membro della superfamiglia delle immunoglobuline (Fig 10).
Fig 10. Alterazione della migrazione dei precursori pontini nel mutante Hoxa2.
Ibridazione in situ con il marcatore Tag1 su preparati “whole mount” di embrioni a stadio E16.5 selvatico e mutante in visione ventrale. Rispetto a quanto si osserva nell’ embrione selvatico il nucleo pontino nel mutante Hoxa2 risulta marcatamente più esteso in senso rostro-caudale ed il fronte migratorio dei precursori pontini è disorganizzato.
L’evidenza di una alterazione a carico dei meccanismi che regolano la migrazione dei neuroni del nucleo pontino in assenza di Hoxa2 ha permesso di formulare l’ipotesi di un ruolo per questo gene omeotico nel controllo degli eventi di modellamento del sistema nervoso centrale come la migrazione neuronale, andando così oltre la classica concezione che attribuisce ai membri di questa famiglia un ruolo precoce durante lo sviluppo.
6- Controllo della formazione dei processi assonali
Successivamente agli eventi che controllano il differenziamento e la migrazione dei neuroni, la formazione di una complessa circuiteria neuronale è il processo finale che sottintende al corretto funzionamento del sistema nervoso centrale. La prerogativa essenziale per la formazione della corretta rete neuronale è data dalla capacità di ciascun neurone di indirizzare il proprio processo assonale verso precise regioni target del cervello, talvolta localizzate a grande distanza. Per far questo è necessario un meccanismo di controllo altamente elaborato in quanto l’assone in accrescimento deve essere in grado di proiettare nella giusta direzione, attraversare territori in cui sono presenti popolazioni eterogenee di neuroni e arrestare la propria crescita una volta raggiunto il corretto target. Ramon y Cajal osservò il cono di crescita assonale per la prima volta più di un secolo fa e propose che questa struttura avesse la funzione di ricevere i segnali necessari per la proiezione dell’assone verso il giusto bersaglio (Ramon y Cajal, 1893). Successivamente venne proposta l’ipotesi chemotattica secondo cui il cono di crescita risponde a gradienti di molecole segnale attraverso specifici recettori espressi sulla membrana plasmatica, permettendo all’assone di rispondere a segnali attrattiv i o repulsivi (Sperry, 1963). Oggi sappiamo non solo che queste ipotesi sono corrette, ma è stato anche possibile identificare alcune delle molecole segnale e dei recettori che controllano il processo di accrescimento assonale (“axon guidance”).
Tra i candidati che regolano la proiezione assonale ci sono molecole che hanno una funzione permissiva, consentendo cioè all’assone di muoversi attraverso la matrice extracellulare (MEC). Questo gruppo di molecole comprende la famiglia delle integrine e quella delle caderine. Le prime sono in grado di mediare le interazioni con i componenti della MEC, mentre le seconde sono proteine transmembrana capaci di mediare i contatti cellula-cellula. Una funzione analoga è svolta da alcuni componenti della super famiglia delle immunoglobuline, tra cui la meglio caratterizzata è la famiglia delle NCAM (“neural cell adhesion molecule”).
Negli ultimi anni sono state identificate una serie di molecole con un ruolo istruttivo, che possono cioè esercitare in maniera attiva un effetto attrattivo o repulsivo sull’assone in crescita. Le Netrine (Ntn) sono fattori secreti in grado di generare gradienti che furono identificati nei vertebrati per il loro ruolo nell’attrazione degli assoni commisurali (Tessier-Lavigne et al., 1988; Placzek et al.,1990). Successivamente sono stati identificati i recettori per queste molecole (UNC-40, DCC) che ne mediano l’effetto di attrazione sul cono di crescita assonale (Keino-Masu et al.,1996).