4. RISULTATI
4.1 Amplificazione della regione SSU-ITS-LSU dei funghi AM con i
primers NS31/LSU-Glom1 e AML1/ LSU-Glom1
Allo scopo di amplificare una porzione del DNA ribosomale che
comprendesse contemporaneamente la regione SSU (parziale), la
regione ITS (completa) e la regione LSU (parziale) sono stati utilizzati i
primers NS31 e LSUGlom1. Un frammento di ~2200 bp è stato
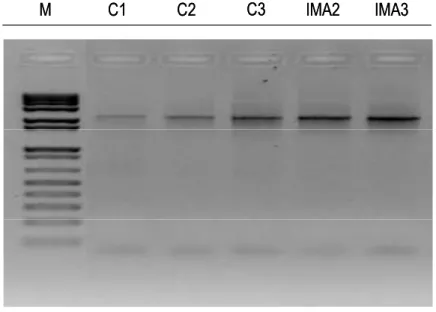
ottenuto in sette dei nove campioni prelevati ad un mese
dall’emergenza delle piante. Un esempio di tale amplificazione è
mostrato in Fig.10.
C1
C2
C3
IMA2
IMA3
M
C1
C2
C3
IMA2
IMA3
M
Figura 10. Prodotto di amplificazione del frammento di ~2200 bp dell’rDNA ottenuto con la coppia di primers NS31-LSUGlom1. Da sinistra il marker (M) 1kb plus DNA ladder (Invitrogen), poi i campioni C1, C2, C3, IMA2 e IMA3.
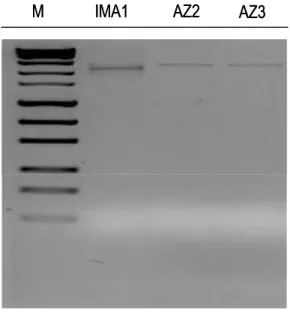
Poiché dagli estratti dei campioni di radice di due piante inoculate
con il fungo AZ225C (campioni AZ2 e AZ3) non si otteneva un
amplificato sufficiente per il clonaggio, tali campioni sono stati
sottoposti ad amplificazione con i primers AML1/LSUGlom1, ottenendo
un frammento di ~2400 bp (Fig.11).
IMA1
AZ2
AZ3
M
IMA1
AZ2
AZ3
M
Figura 11. Prodotti di amplificazione dell’rDNA nucleare. Da sinistra il marker (M) 1 kb plus DNA ladder (Invitrogen), poi i campioni IMA1, AZ2 e AZ3. L’amplificato del campione IMA1, di ~2200 bp, è stato ottenuto con la coppia di primers NS31-LSUGlom1, mentre gli amplificati di AZ2 e AZ3, di ~2400 bp, sono stati ottenuti con la coppia di primers AML1-LSUGlom1.
4.2 Ricchezza e variabilità fungina AM tramite RFLPs a due mesi
dalla germinazione delle piante di M. sativa
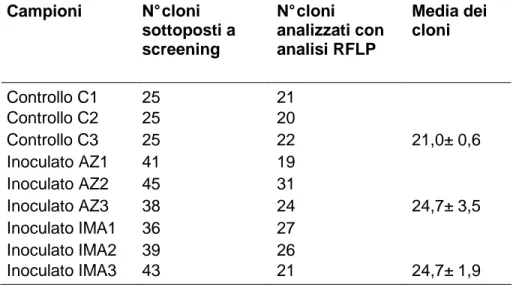
Nove librerie clonali sono state ottenute dall’analisi di 317 cloni,
derivanti dal DNA dalle radici delle piante di M. sativa sottoposte ai tre
trattamenti: i) piante di controllo non inoculate (C1, C2, C3); ii) piante
inoculate con il fungo AZ225C (AZ1, AZ2, AZ3); iii) piante inoculate
con il fungo IMA1 (IMA1, IMA2, IMA3). In totale sono state sottoposte
a screening con i primers SP6/T7 317 colonie, di cui 211 (66,5%)
hanno prodotto una banda della grandezza attesa (~550 bp) dopo
amplificazione nested con i primers NS31/AM1. Tale porzione
dell’SSU è stata sottoposta ad analisi mediante RFLPs. Sono stati
analizzati in media (± errore standard) 21,0 ± 0,6 cloni nel controllo,
24,7 ± 3,5 cloni nelle piante inoculate con AZ225C e 24,7 ± 1,9 cloni
nelle piante inoculate con IMA1 (Tab. 3).
Ogni prodotto di PCR di ciascun clone per campione di radice è
stato digerito con i due enzimi di restrizione HinfI e Hsp92II. Per ogni
clone è stata ottenuta una coppia di patterns, sulla base della quale è
stata effettuata l’analisi. Teoricamente l’insieme dei patterns ottenuti
con i due enzimi suddetti è in grado di caratterizzare una singola
specie di fungo AM.
Tabella 3. Numero di cloni sottoposti a screening e analizzati tramite RFLPs della porzione di SSU amplificata dai primers NS31/AM1. I valori rappresentano le medie ± ES di tre repliche per ciascun trattamento fungino AM.
Campioni
N° cloni
sottoposti a
screening
N° cloni
analizzati con
analisi RFLP
Media dei
cloni
Controllo C1
25
21
Controllo C2
25
20
Controllo C3
25
22
21,0± 0,6
Inoculato AZ1
41
19
Inoculato AZ2
45
31
Inoculato AZ3
38
24
24,7± 3,5
Inoculato IMA1
36
27
Inoculato IMA2
39
26
Inoculato IMA3
43
21
24,7± 1,9
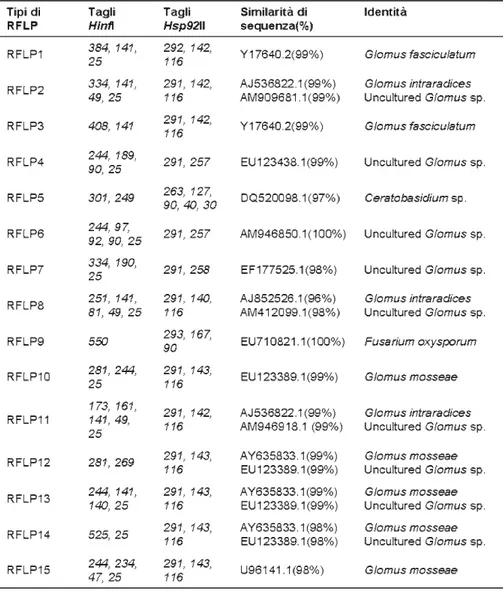
Dall’analisi delle sequenze dell’SSU parziale ottenute dalle spore
degli isolati IMA1 e AZ225C già presenti in banca dati e ottenute da
Pellegrino (2007), tagliate in silico, è stato possibile identificare un
profilo specifico, caratteristico di ognuno dei due funghi inoculati,
costituito dall’RFLP10 sia per l’inoculo AZ225C che per l’inoculo IMA1
(Tab. 4).
Tabella 4. Profili RFLPs ottenuti dai campioni radicali di M. sativa inoculati con G. mosseae AZ225C e IMA1, e non inoculati (controllo).
Sono state identificate 15 coppie di patterns diverse. La ricchezza
e diversità dei profili di RFLPs variava tra le tre differenti tesi.
Le tre piante appartenenti alla tesi di controllo hanno prodotto 9
patterns di RFLPs (Fig. 12 e Fig. 15); quelle appartenenti alla tesi
inoculo con il fungo AZ225C hanno prodotto 3 patterns di RFLPs (Fig.
13 e Fig. 15); infine quelle appartenenti alla tesi inoculo con il fungo
IMA1 hanno prodotto 6 patterns di RFLPs (Fig. 14 e Fig. 15).
Il numero totale di RFLPs (SR) è stato usato come stima della
diversità della comunità fungina AM entro le radici. Dall’analisi è
possibile stimare che in media il numero delle specie AM che
colonizzava le radici delle diverse tesi era di 4.0 nelle piante controllo,
3.3 nelle piante inoculate con IMA1 e 1.6 in quelle inoculate con
AZ225C (Tab. 5).
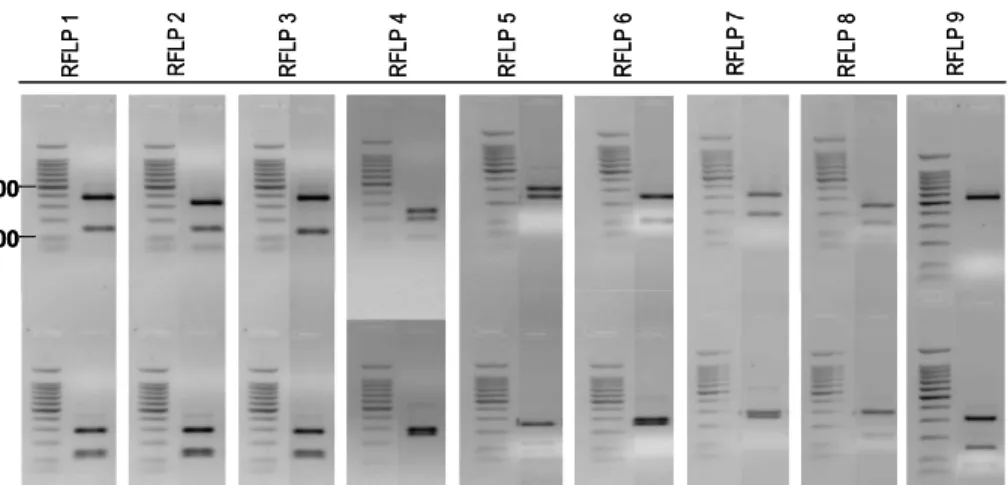
R F L P 1 R F L P 3 R F L P 2 R F L P 4 R F L P 5 R F L P 6 R F L P 7 R F L P 8 R F L P 9 500 100 R F L P 1 R F L P 3 R F L P 2 R F L P 4 R F L P 5 R F L P 6 R F L P 7 R F L P 8 R F L P 9 500 100Figura 12. Profili di restrizione ottenuti nella tesi controllo. Ogni profilo è riportato a destra del marker 100 bp DNA ladder (Promega), ed è costituito dal prodotto di digestione con HinfI (lanes in alto), e da quello con Hsp92II (lanes in basso).
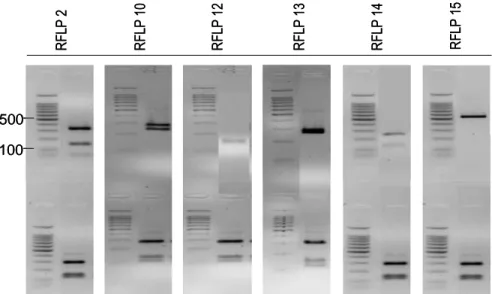
R F L P 1 0 R F L P 1 6 R F L P 1 5 500 100 R F L P 1 0 R F L P 1 6 R F L P 1 5 R F L P 1 0 R F L P 1 6 R F L P 1 5 500 100
Figura 13. Profili di restrizione ottenuti nella tesi AZ225C. Ogni profilo è riportato a destra del marker 100 bp DNA ladder (Promega), ed è costituito dal prodotto di digestione con HinfI (lanes in alto), e da quello con Hsp92II (lanes in basso). R F L P 2 R F L P 1 0 R F L P 1 2 R F L P 1 3 R F L P 1 4 R F L P 1 5 500 100 R F L P 2 R F L P 1 0 R F L P 1 2 R F L P 1 3 R F L P 1 4 R F L P 1 5 500 100
Figura 14. Profili di restrizione ottenuti nella tesi IMA1. Ogni profilo è riportato a destra del marker 100 bp DNA ladder (Promega), ed è costituito dal prodotto di digestione con HinfI (lanes in alto), e da quello con Hsp92II
Il numero totale di RFLP (SR) era significativamente influenzato dal
trattamento fungino (P < 0.02) (Tab. 5). La media del numero totale di
RFLPs variava significativamente tra i trattamenti inoculati con
AZ225C e i controlli, mentre le piante inoculate con IMA1
presentavano valori intermedi (Tab. 5).
Figura 15. Tipi di profili RFLPs di funghi micorrizici arbuscolari e loro frequenza nelle radici di Medicago sativa non inoculate_controllo (a) e inoculate con
Glomus mosseae AZ225C (b) e IMA1
(c). I valori sono medie di tre repliche per ogni trattamento.
Tabella 5. Ricchezza delle specie (SR) e indice di diversità , indice di Shannon (Ho) nelle radici di M. sativa inoculate con Glomus mosseae
AZ225C e IMA1. Anova ad una via per testare l’ipotesi nulla di non effetto del trattamento fungino sulla ricchezza delle specie e sull’indice di Shannon.
Ricchezza
delle specie
SR
Indice
di Shannon
Ho
Controllo
4.00 ± 0.58 b
0.93 ± 0.10 b
AZ225 C
1.67 ± 0.33 a
0,10 ± 0.05 a
IMA1
3.33 ± 0.33 ab
0.73 ± 0.16 b
I valori sono medie (n=3) ± l’ES. . I valori in ciascuna colonna non seguiti dalle stesse lettere sono significativamente diversi (SR, P = 0.021; Ho, P = 0.005).
Nelle piante controllo l’RFLP1, l’RFLP2 e l’RFLP4 erano quelli
maggiormente presenti, presentando valori medi di il 40.3, 18.6 e 24.9
(Tab. 6). Nelle piante inoculate con il fungo AZ225C l’RFLP10
rappresentava valori medi di 97.5, mentre in quelle inoculate con il
fungo IMA1, l’RFLP10 e l’RFLP2 presentavano valori medi di 39.1 e
50.4 rispettivamente (Tab. 6). E’ da notare che l’RFLP10 corrisponde
al profilo che identifica i due funghi inoculati. Il test di Levene di
omogeneità della varianza sulla % di presenza degli RFLPs, ha messo
in evidenza valori statisticamente significativi, per cui si è resa
necessaria un’analisi non parametrica. Da questa analisi a coppie sulle
percentuali di ciascun RFLP è stato evidenziato che l’unico RFLP che
variava significativamente tra tesi Controllo vs tesi AZ225C e tra tesi
Controllo vs tesi IMA1 era l’RFLP10 (P = 0.037, Controllo vs AZ225C;
P = 0.037, controllo vs IMA1), mentre nel confronto tesi AZ225 vs tesi
IMA1 sia l’RFLP2 che l’RFLP10 risultavano significativamente diversi
(P = 0.037 e P = 0.050, rispettivamente). L’RFLP2 risultava essere
significativamente maggiore nelle tesi inoculate con IMA1 e l’RFLP10
nelle tesi inoculate con AZ225C.
Tabella 6. Percentuali degli RFLPs delle comunità fungine AM presenti nelle radici di M. sativa inoculate con G. mosseae AZ225C e IMA1 e non inoculate (controllo). I valori sono medie (n=3) ± l’ES.
Tipi di RFLPs
Controllo AZ225C IMA1
RFLP1 40,3 ± 20,2 0 RFLP2 18,6 ± 9,3 0 50.4 ± 19,9 RFLP3 1,6 ± 1,6 0 0 RFLP4 24,9 ± 22,3 0 0 RFLP5 1,7 ± 1,7 0 0 RFLP6 8,3 ± 8,3 0 0 RFLP7 1,5 ± 1,5 0 0 RFLP8 1,5 ± 1,5 0 0 RFLP9 1,5 ± 1,5 0 0 RFLP10 0 97,5 ± 1,3 39,1 ± 22,7 RFLP11 0 0 1,2 ± 1,2 RFLP12 0 0 1,3 ± 1,3 RFLP13 0 0 6,3 ± 6,3 RFLP14 0 1,4 ± 1,4 1,6 ± 1,6 RFLP15 0 1,1 ± 1,1 0
La diversità fungina AM (H
o) era influenzata in modo
statisticamente significativo dal trattamento fungino (P = 0.005). La
media della diversità di funghi AM espressa mediante tale indice
mostrava differenze statisticamente significative tra i diversi
trattamenti. Le tesi IMA1 presentavano valori medi di H
0pari a 0.10,
statisticamente diversi rispetto a AZ225C (0.73) e Controllo (0.93), che
mostravano valori simili tra loro.
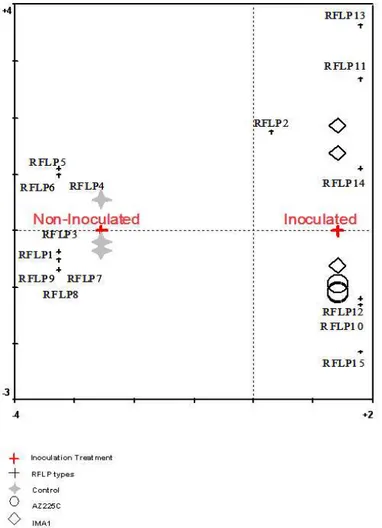
In aggiunta le comunità fungine AM sono state comparate con la
CCA usando i dati di abbondanza relativa degli RFLPs. La CCA
mostrava come le comunità fungine AM che colonizzavano le radici di
M. sativa sottoposta al trattamento inoculazione erano diverse, e
evidenziava come alcuni RFLPs erano caratteristici del trattamento
IMA1 e AZ225C (RFLP 2, RFLP10 e RFLP12) e del controllo (RFLP1,
RFLP3, RFLP4, RFLP5, RFLP 6, RFLP7, RFLP8 e RFLP9) (Fig. 16).
La varianza spiegata dal fattore inoculazione sulla prima asse era pari
al 31.1% ed il fattore inoculazione correlava con tale asse per il 100%.
Il Montecarlo test in seguito a 999 permutazioni era statisticamente
significativo (P = 0.013) per il fattore inoculazione.
Figura 16. Biplot della CCA dei tipi di RFLPs e del fattore ambientale inoculazione entro le radici di M. sativa inoculate con Glomus mosseae IMA1 e AZ225C, e entro le radici di piante di controllo (n = 9). La prima asse spiega il 78% della varianza spiegata da tutti gli assi canonici e le differenze sono significative (P = 0,013).
4.3 Ricchezza e variabilità fungina AM tramite analisi delle
sequenze SSU a due mesi dalla germinazione delle piante di M.
sativa
Sulla base dei profili di restrizione ottenuti sono stati sequenziati, da 1
fino a 10 cloni per ogni profilo. I cloni sono stati sequenziati sia in 5’
(sequenze SSU), sia in 3’ (sequenze ITS2-LSU). Dopo il
sequenziamento della porzione relativa all’SSU dei cloni derivanti dalle
nove librerie i 15 profili identificati si sono ridotti a 5 tipi di sequenze
(Tab. 7). Nove profili di RFLPs derivavano da un solo clone (RFLP3,
RFLP5, RFLP7, RFLP8, RFLP9, RFLP11, RFLP12, RFLP14,
RFLP15); due di questi (RFLP5 e RFLP9), dopo essere stati sottoposti
alla procedura BLAST, risultavano sequenze di funghi non micorrizici
(Ceratobasidium sp. e Fusarium oxysporum), sei (RFLP3, RFLP8,
RFLP11, RFLP12, RFLP14, RFLP15), dopo essere stati analizzati sia
con procedura BLAST che filogeneticamente, mostravano sequenze
identiche ai profili con numero di cloni maggiore, e cioè l’RFLP12,
l’RFLP14 e l’RFLP15 avevano sequenze simili a quelle dell’RFLP10,
l’RFLP8 e l’RFLP11 avevano sequenze simili all’RFLP2, e l’RFLP3
aveva sequenze simili all’RFLP1.
Dei 96 plasmidi sequenziati abbiamo ottenuto 75 sequenze di SSU
parziale di Glomeromycota (98%-100% di omologia) utilizzabili per
l’analisi filogenetica NJ. Le 75 sequenze ottenute, insieme ad altre 69
derivanti da GenBank, sono state utilizzate per effettuare l’analisi
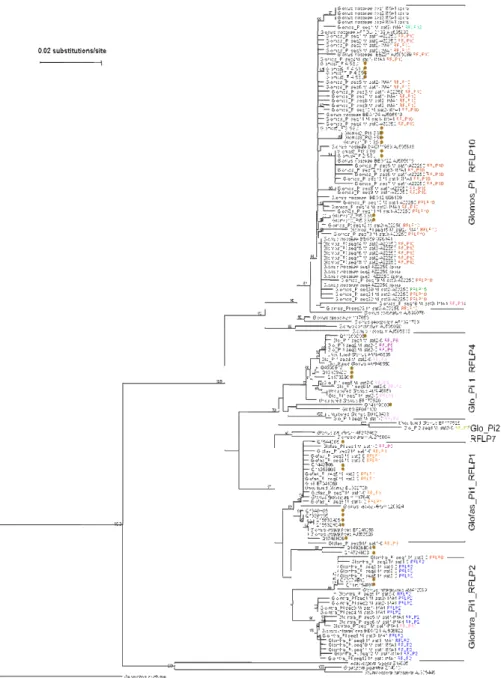
filogenetica NJ sulla porzione (Fig. 17).
L’analisi NJ ha permesso di identificare cinque gruppi di sequenze,
nominate come segue Glofas_Pi RFLP1; Glointra_Pi RFLP2; Glo_Pi1
RFLP4; Glo_Pi2 RFLP7; Glomos_Pi RFLP10 relazionando specie e/o
filotipo al profilo RFLP di appartenza (Tab. 7). I 5 tipi di sequenze
appartenevano tutti alla famiglia delle Glomeraceae, e non sono state
Figura 17. Analisi filogenetica NJ dei Glomeromycota presenti nelle radici di piante di
M. sativa inoculate con G. mosseae AZ225C e IMA1 e piante non inoculate (controlli). Le sequenze in grassetto sono state ottenute in questo lavoro di tesi. L’analisi è stata ottenuta dall’allineamento di 144 sequenze di un frammento di SSU (~ 550 bp). Geosiphon pyriforme è stato usato come outgroup. I valori di bootstrap (1000 repliche) sono mostrati per valori sopra il 40%. Le sequenze contrassegnate dal simbolo marrone sono state isolate da radici di piante trappola utilizzate per intrappolare i funghi indigeni presenti nel sito sperimentale.
ritrovate sequenze appartenenti alle famiglie Acaulosporaceae,
Gigasporaceae, Diversisporaceae. Inoltre non è stata ritrovata alcuna
sequenza appartenente alle famiglie delle Archaeosporaceae e
Paraglomeraceae, come atteso dall’uso, durante lo screening dei cloni,
del primer AM1, che non amplifica questi taxa (Redecker, 2001).
Nelle radici delle piante di controllo sono state ritrovate sequenze
simili a Glomus fasciculatum (99% di omologia), a G. intraradices
(96%-99% di omologia), e due tipi di sequenze simili a quelle del
genere Glomus. (98%-100% di omologia). Le sequenze simili a G.
fasciculatum denominate Glofas_Pi RFLP1 rappresentavano il gruppo
più abbondante (42,8%), le sequenza simili a G. intraradices,
Glointra_Pi RFLP2, erano presenti nelle radici con il 20,6%, e quelle
simili a Glomus sp., Glo_Pi1 RFLP4 e Glo_Pi2 RFLP7, erano presenti
con il 34,9% e 1,6%, rispettivamente (Tab. 7; Fig. 18).
Nelle radici delle piante inoculate con G. mosseae AZ225C è stato
trovato il 100% di sequenze simili a G. mosseae (99%-100% di
omologia) denominate Glomos_Pi RFLP10 (Tab. 7; Fig. 18).
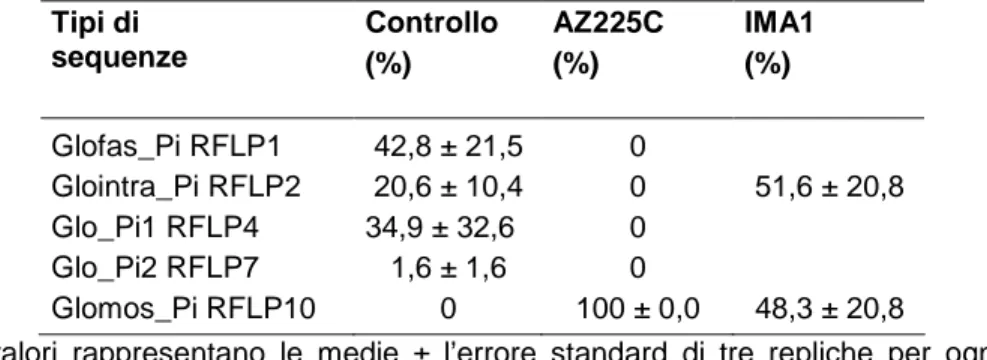
Tabella 7. Percentuali dei tipi di sequenze di funghi AM trovati nelle radici di M. sativa inoculate con Glomus mosseae AZ225C e IMA1, e non inoculate
(controllo). Tipi di sequenze Controllo (%) AZ225C (%) IMA1 (%) Glofas_Pi RFLP1 42,8 ± 21,5 0 Glointra_Pi RFLP2 20,6 ± 10,4 0 51,6 ± 20,8 Glo_Pi1 RFLP4 34,9 ± 32,6 0 Glo_Pi2 RFLP7 1,6 ± 1,6 0 Glomos_Pi RFLP10 0 100 ± 0,0 48,3 ± 20,8
I valori rappresentano le medie ± l’errore standard di tre repliche per ogni trattamento.
Nelle radici delle piante inoculate con G. mosseae IMA1 il 48,3% di
sequenze (Glomos_Pi RFLP10) erano simili a G. mosseae
(98%-100% di omologia) e il 51,6% (Glointra_Pi RFLP2) erano simili a G.
intraradices (99%-100% di omologia) (Tab. 7; Fig. 18).
I cinque tipi di sequenze ritrovate rientravano in clusters, dove
erano presenti anche sequenze simili isolate dalle radici di piante
Figura 18. Tipi di sequenze di funghi micorrizici arbuscolari e loro frequenza nelle radici di Medicago sativa non inoculate_controllo (a) e inoculate conGlomus mosseae AZ225C (b) e IMA1
(c). I valori sono medie di tre repliche per ogni trattamento.
trappola fatte crescere sul suolo prima dell’inoculo dei due isolati di G.
mosseae (Fig. 17).
Le percentuali di presenza dei tipi di sequenze AM ritrovati nelle
radici di M. sativa inoculate e non inoculate con i due isolati di G.
mosseae sono state analizzate tramite il test di Levene per valutare
l’omogeneità delle varianze. I valori statisticamente significativi del test
hanno reso necessaria un’analisi non parametrica. Dall’analisi a
coppie Mann-Whitney U è stato evidenziato che Glomos_Pi RFLP10
era l’unico tipo di sequenza che variava in modo statisticamente
significativo tra le piante di controllo vs le piante inoculate con G.
mosseae AZ225C (P = 0.025) e tra controllo vs G. mosseae IMA1 (P =
0.037). Nel confronto tra AZ225 vs IMA1 sia Glointra_Pi RFLP2 che
Glomos_Pi RFLP10 risultavano diversi in modo statisticamente
significativo (P = 0.037 e P = 0.037, rispettivamente). Nello specifico
Glointra_Pi RFLP2 risultava essere maggiore nelle tesi inoculate con
G. mosseae IMA1 e Glomos_Pi RFLP10 nelle tesi inoculate con G.
mosseae AZ225C.
In aggiunta le comunità fungine AM sono state comparate con la
RDA usando i dati di abbondanza relativa dei tipi di sequenze ottenute.
L’RDA ha mostrato come le comunità fungine AM che colonizzavano
le radici di M. sativa inoculate con i due isolati fungini di G. mosseae
erano diverse rispetto a quelle presenti nelle radici delle piante di
controllo, ed evidenziava come alcuni tipi di sequenza erano
caratteristici del trattamento IMA1 e AZ225C (Glomos_Pi RFLP10) e
del controllo (Glo_Pi1 RFLP4) (Fig. 19). La varianza spiegata dal
fattore inoculazione sulla prima asse era pari al 35,2% ed il fattore
inoculazione correlava con tale asse per il 100%. Il Montecarlo test in
seguito a 999 permutazioni mostrava una significatività di P = 0.08 per
il fattore inoculazione.
Figura 19. Biplot della RDA dei tipi di sequenza e del fattore ambientale inoculazione entro le radici di M. sativa inoculate con Glomus mosseae IMA1 e AZ225C, ed entro le radici di piante di controllo (n = 9). La prima asse spiega il 35,2% della varianza spiegata da tutti gli assi canonici e le differenze non sono significative (P = 0,08).
In media la SR, e cioè il numero di tipi di sequenze di funghi AM
che colonizzava le radici delle piante di controllo era di 2,3 ± 0,67, di 1
± 0,00 nelle piante inoculate con l’isolato AZ225C e di 2 ± 0,00 in
quelle inoculate con l’isolato IMA1. L’ANOVA ad una via non ha messo
in evidenza differenze statisticamente significative tra i diversi
trattamenti (P=0,11). La diversità fungina espressa dall’ H
0risultava di
0,5 ± 0,24 nelle radici delle piante di controllo, di 0,24 ± 0,04 nelle
radici inoculate con l’isolato IMA1, mentre di 0,00 ± 0,00 nelle radici
delle piante inoculate con l’isolato AZ225C. Anche per questo
parametro
non
sono
state
messe
in
evidenza
differenze
statisticamente significative tra i trattamenti (P=0,13).
4.4 Analisi della porzione ITS2 delle sequenze di G. mosseae
presenti nelle radici di M. sativa
Tutte le sequenze che mostravano nella porzione parziale di SSU
omologia con il fungo G. mosseae sono state selezionate per eseguire
un’analisi BLAST e un’analisi filogenetica NJ della porzione ITS2 dei
medesimi cloni. L’analisi BLAST è stata effettuata con le sequenze di
DNA da spora di AZ225C (AJ919273, AJ919273, G. mosseae AZ225C
seq1, G. mosseae AZ225C seq2, G. mosseae AZ225C seq3, G.
mosseae AZ225C seq4 e IMA1 (AM422114, AM422115). Le sequenze
ritrovate nelle radici delle piante inoculate con l’isolato AZ225C
mostravano in media un’omologia di 98% con le sequenze di AZ225C
derivate da spora presenti sia in GenBank sia sequenziate da
Pellegrino (2007), mentre l’omologia tra le sequenze di G. mosseae
presenti nelle radici inoculate con l’isolato IMA1 e le sequenze di
GenBank di tale isolato o sequenziate da Pellegrino (2007) era in
media 95%.
L’analisi NJ ha messo in evidenza che tutte le sequenze ITS2 di G.
mosseae ritrovate nelle piante inoculate con il fungo AZ225C
rientravano in un unico cluster insieme alle sequenze di tale fungo
isolate da spora (87% di valore di bootstrap) (Fig. 20). Anche tutte le
sequenze ITS2 di G. mosseae ritrovate nelle radici delle piante
inoculate con il fungo IMA1 rientravano in un unico cluster
comprendente le sequenze ITS2 isolate da spore (32% di valore di
bootstrap) (Fig. 20). Questi gruppi di sequenze divergevano dalle
sequenze di G. mosseae presenti nel suolo prima dell’inoculo (valore
di bootstrap 100%).
Figura 20. Analisi filogenetica NJ dei Glomeromycota focalizzata alla specie Glomus mosseae, e in particolare agli isolati AZ225C e IMA1 inoculati nel sito
sperimentale e agli isolati indigeni. Le sequenze in grassetto sono state ottenute da radici di Medicago sativa inoculate con G. mosseae (IMA1 e AZ225C) dopo due mesi dalla germinazione. Tale analisi è stata ottenuta dall’allineamento di 65 sequenze dell’ITS2 (~ 200 bp). Glomus geosporum è stato usato come outgroup. I valori di bootstrap (1000 repliche) sono mostrati per valori sopra il 20%.