4.
R
ISULTATI
4.1 Isolamento e clonaggio di
DjPHB2
Lo scopo iniziale di questo lavoro di Tesi è stato quello di isolare la sequenza completa del gene proibitina di planaria, a partire dalla sequenza di un clone EST già disponibile in banca dati (numero di accesso Gene Bank: Gi32904098), attraverso la metodica della 3’-5’ RACE. Gli amplificati ottenuti sono stati, quindi, clonati e mandati a sequenziare.
5’AAGCAGTGGTATCAACGCAGAGTACGCGGGGGCATTTTTTGAAAAAAAAATTCAACACGCCATTGCTTCATGGA TTTAAAAAATGTAGCTAATTCTTTTATGGGTAAAGGTAGTCCCAAAGGGTTGGGTGCGATGTTAATAGGAGGTGGT CTTATATTTGGATTAACTAAGGCATTTTATACAGTTGATGGTGGTCAAAGAAGTATTATTTTTAGTCGATTTGGTG GGATTAAAGAAAACATTTATGCTGAAGGACTTCATTTTAGAATTCCTGGTATACAATATCCCATCATTTTCGATGT GAGATCTCGTCCAAGAATAATTTCTTCTCCAACTGGAAGCAAAGATTTACAAATGGTGAATATTTCTCTGAGAGTA CTTAGTAGACCAGATATATTAAAAATTCCTGACATATACAGAAATCTCGGTGAAGATTACGACGAAAGAGTTCTGC CTTCAATATCAAATGAAGTTTTAAAAGCCGTTGTCGCTAAATTTAATGCCGGACAATTAATTACACAAAGAGAACA AGTTTCTCTGCTTATTCGGAAACTATTAATTGAAAGAGCTAAAGATTTTAACATAATCGTTGATGATGTTTCAATT ACTGATTTATCGTTTTCTAAACAATACGGCGAAGCAGTCGAAAGAAAACAGATTTCTCAACAAGAAGCTCAGCGAG CCCAATTTACTGTAATGCGAGCAAAACAAGAAAGACAACAGAAAATAGTCAATGCCGAAGGAGAAGCGCAAGCTGC AATATTGATCGGTGATGCTTTGTCAGCGAATTCAGGATATTTGAAATTGAGAAAAATAAAAGCATCAGAGAAAATC GCAAGAACGCTGTCAACAGCTCAAAACAGAGCTTATTTAAATGCGAATACATTGATGATGAACATCAATGAAAAAG AATTCAACGATTCAGTCGACAGAGTAGTGAAAAAGAAGTAAGATCGTAAAGTTGGGAAAGTTTTTGTAGTGTTGTT CGAATAAAATAAAGTTTAGAATTTTCAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAGAAAAAAAAAAAAAAAAA3’
Fig. 4.1: sequenza nucleotidica ricavata dal sequenziamento degli amplificati derivanti dalla 3’-5’RACE. In grassetto: ATG codone di inizio (Met) e TAA codone di terminazione
L’analisi della sequenza ottenuta ha rivelato che la lunghezza del clone completo, chiamato DjPHB2, possiede una lunghezza di 1.012 pb - a cui va aggiunta la coda di poli(A) - (Fig. 4.1). Il clone contiene una 5’ non tradotta (UTR) di 69 pb, una 3’ UTR di 61 pb e una open reading frame (ORF) di 882 pb, che codifica per una proteina putativa di 293 amminoacidi (Fig. 4.2).
M D L K N V A N S F M G K G S P K G L G A M L I G G G L I F G L T K A F Y T V D G G Q R S I I F S R F G G I K E N I Y A E G L H F R I P G I Q Y P I I F D V R S R P R I I S S P T G S K D L Q M V N I S L R V L S R P D I L K I P D I Y R N L G E D Y D E R V L P S I S N E V L K A V V A K F N A G Q L I T Q R E Q V S L L I R K L L I E R A K D F N I I V D D V S I T D L S F S K Q Y G E A V E R K Q I S Q Q E A Q R A Q F T V M R A K Q E R Q Q K I V N A E G E A Q A A I L I G D A L S A N S G Y L K L R K I K A S E K I A R T L S T A Q N R A Y L N A N T L M M N I N E K E F N D S V D R V V K K K
Fig. 4.2: sequenza della proteina putativa codificata da DjPHB2, costituita da 293 amminoacidi
Utilizzando il programma tblastx, disponibile in rete, è stata messa in evidenza una forte omologia di sequenza a livello amminoacidico (>80%) con proteine PHB2 di altri organismi. Tra le sequenze presentanti un maggiore grado di omologia con DjPHB2, vi sono quelle isolate in Xenopus laevis, Mus
musculus e Danio rerio.
D. japonica vs X. laevis
Identities = 177/291 (60%), Positives = 235/291 (80%), Gaps = 7/291 (2%)
D. japonica vs M. musculus
Identities = 177/298 (59%), Positives = 242/298 (81%), Gaps = 7/298 (2%)
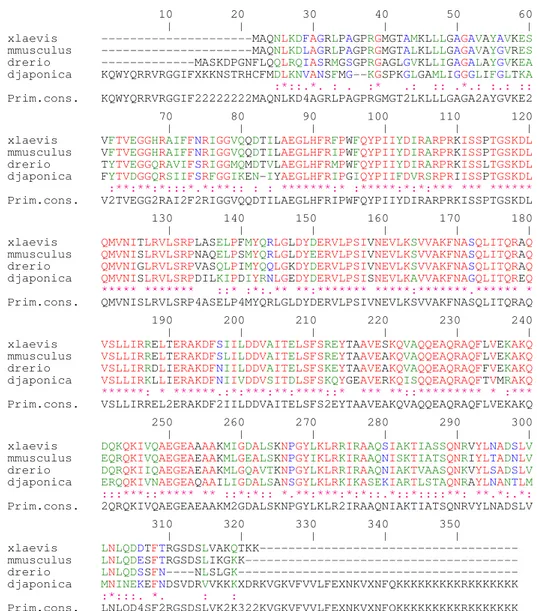
L’allineamento di queste proteine (Fig. 4.3), con il programma CLUSTAL
W, ha evidenziato la presenza di regioni amminoacidiche estremamente
conservate durante l’evoluzione tra i membri di questa famiglia proteica.
10 20 30 40 50 60 | | | | | | xlaevis ---MAQNLKDFAGRLPAGPRGMGTAMKLLLGAGAVAYAVKES
mmusculus ---MAQNLKDLAGRLPAGPRGMGTALKLLLGAGAVAYGVRES
drerio ---MASKDPGNFLQQLRQIASRMGSGPRGAGLGVKLLIGAGALAYGVKEA
djaponica KQWYQRRVRGGIFXKKNSTRHCFMDLKNVANSFMG--KGSPKGLGAMLIGGGLIFGLTKA
:*::.*. : . :* .: :: .*.: :.: :: Prim.cons. KQWYQRRVRGGIF22222222MAQNLKD4AGRLPAGPRGMGT2LKLLLGAGA2AYGVKE2 70 80 90 100 110 120 | | | | | | xlaevis VFTVEGGHRAIFFNRIGGVQQDTILAEGLHFRFPWFQYPIIYDIRARPRKISSPTGSKDL mmusculus VFTVEGGHRAIFFNRIGGVQQDTILAEGLHFRIPWFQYPIIYDIRARPRKISSPTGSKDL drerio TYTVEGGQRAVIFSRIGGMQMDTVLAEGLHFRMPWFQYPIIYDIRARPRKISSLTGSKDL djaponica FYTVDGGQRSIIFSRFGGIKEN-IYAEGLHFRIPGIQYPIIFDVRSRPRIISSPTGSKDL :**:**:*:::*.*:**:: : : *******:* :*****:*:*:*** *** ****** Prim.cons. V2TVEGG2RAI2F2RIGGVQQDTILAEGLHFRIPWFQYPIIYDIRARPRKISSPTGSKDL 130 140 150 160 170 180 | | | | | | xlaevis QMVNITLRVLSRPLASELPFMYQRLGLDYDERVLPSIVNEVLKSVVAKFNASQLITQRAQ mmusculus QMVNISLRVLSRPNAQELPSMYQRLGLDYEERVLPSIVNEVLKSVVAKFNASQLITQRAQ drerio QMVNIGLRVLSRPVASQLPIMYQQLGKDYDERVLPSIVNEVLKSVVAKFNASQLITQRAQ djaponica QMVNISLRVLSRPDILKIPDIYRNLGEDYDERVLPSISNEVLKAVVAKFNAGQLITQREQ ***** ******* ::* :*:.** **:******* *****:*******.****** * Prim.cons. QMVNISLRVLSRP4ASELP4MYQRLGLDYDERVLPSIVNEVLKSVVAKFNASQLITQRAQ 190 200 210 220 230 240 | | | | | | xlaevis VSLLIRRELTERAKDFSIILDDVAITELSFSREYTAAVESKQVAQQEAQRAQFLVEKAKQ mmusculus VSLLIRRELTERAKDFSLILDDVAITELSFSREYTAAVEAKQVAQQEAQRAQFLVEKAKQ drerio VSLLIRRDLIERAKDFNIILDDVAITELSFSKEYTAAVEAKQVAQQEAQRAQFFVEKAKQ djaponica VSLLIRKLLIERAKDFNIIVDDVSITDLSFSKQYGEAVERKQISQQEAQRAQFTVMRAKQ ******: * ******.:*:***:**:****::* *** **::********* * :*** Prim.cons. VSLLIRREL2ERAKDF2IILDDVAITELSFS2EYTAAVEAKQVAQQEAQRAQFLVEKAKQ 250 260 270 280 290 300 | | | | | | xlaevis DQKQKIVQAEGEAAAAKMIGDALSKNPGYLKLRRIRAAQSIAKTIASSQNRVYLNADSLV
mmusculus EQRQKIVQAEGEAEAAKMLGEALSKNPGYIKLRKIRAAQNISKTIATSQNRIYLTADNLV
drerio DQRQKIIQAEGEAEAAKMLGQAVTKNPGYLKLRRIRAAQNIAKTVAASQNKVYLSADSLV
djaponica ERQQKIVNAEGEAQAAILIGDALSANSGYLKLRKIKASEKIARTLSTAQNRAYLNANTLM
:::***::***** ** ::*:*:: *.**:***:*:*::.*::*::::**: **.*:.*: Prim.cons. 2QRQKIVQAEGEAEAAKM2GDALSKNPGYLKLR2IRAAQNIAKTIATSQNRVYLNADSLV 310 320 330 340 350 | | | | | xlaevis LNLQDDTFTRGSDSLVAKQTKK--- mmusculus LNLQDESFTRGSDSLIKGKK--- drerio LNLQDSSFN----NLSLGK--- djaponica MNINEKEFNDSVDRVVKKKXDRKVGKVFVVLFEXNKVXNFQKKKKKKKKKRKKKKKKK :*:::. *. : : Prim.cons. LNLQD4SF2RGSDSLVK2K322KVGKVFVVLFEXNKVXNFQKKKKKKKKKRKKKKKKK
Fig. 4.3: multiallineamento delle sequenze amminoacidiche della proteina PHB2 di Xenopus laevis, Mus musculus e Danio rerio contro la sequenza amminoacidica putativa di DjPHB2. (*) amminoacidi uguali - in rosso; (:) amminoacidi fortemente similari - in verde; (.) amminoacidi debolmente similari - in blu
L’analisi effettuata ha rivelato, infatti, la presenza anche in DjPHB2 di una regione centrale altamente conservata corrispondente al dominio SPFH, appartenente alla Band 7 Family, di cui fanno parte, oltre alle proibitine, stomatine, proteine citoplasmatiche antiproliferative e proteine batteriche HflC. La presenza di tale dominio in DjPHB2 è stata confermata anche mediante l’utilizzo del programma Pfam (amminoacidi 37-216; evidenziati in grassetto in Fig. 1.3).
La presenza del dominio SPFH è un ulteriore dato che permette di classificare il gene DjPHB2 come nuovo membro della famiglia delle proibitine.
Il gene DjPHB2 è stato anche confrontato con il clone PHB (numero di accesso Gene Bank: AY967616) recentemente isolato in un’altra specie di planaria, Schmidtea mediterranea. L’analisi, effettuata con il programma
blast2, ha rivelato un basso livello di omologia amminoacidica tra le due
sequenze (45% di identità e 70% di positività), facendo supporre che non si tratti della stessa proteina.
Un’analisi più approfondita del genoma di planaria ha infatti confermato l’esistenza anche in questo organismo di due omologhi del gene proibitina:
DjPHB2, omologo ai geni PHB2, e il clone AY967616, omologo ai geni PHB1.
4.2 Studi di espressione
Una volta terminato lo studio sulla sequenza, sono stati affrontati studi di espressione genica. Dati precedenti, ottenuti dall’utilizzo della metodica della PCR real time (Rossi et al., 2006), indicavano che DjPHB2 fosse espresso a livello dei neoblasti. Allo scopo di analizzare in maniera più approfondita l’espressione di DjPHB2, nel corso di questo lavoro di Tesi sono stati condotti esperimenti di ibridazione in situ whole mount sia in animali intatti che in rigenerazione.
4.2.1 Analisi dell’espressione di DjPHB2 in animali intatti
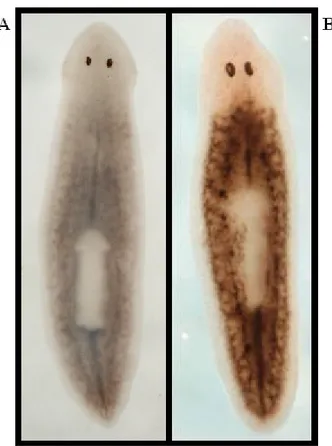
L’espressione di DjPHB2 è stata analizzata in animali intatti mediante esperimenti di ibridazione in situ whole mount. I dati ottenuti indicano che il gene è espresso in cellule accumulate lungo un cordone centrale dorsale, in
clusters laterali dorsali e in cellule sparse all’interno del parenchima.
La distribuzione delle cellule che esprimono DjPHB2 è paragonabile alla localizzazione delle cellule esprimenti il gene DjMCM2, noto marcatore molecolare di neoblasti (Fig. 4.4).
Fig. 4.4: analisi dell’espressione del gene DjPHB2 mediante esperimenti di ibridazioni in situ whole mount in animali intatti: DjPHB2 (A); DjMCM2 (B)
4.2.2 Analisi dell’espressione di DjPHB2 in animali rigeneranti
Allo scopo di analizzare l’espressione del gene durante la rigenerazione, gli animali sono stati amputati sopra e sotto il faringe, sono stati fissati a vari tempi dal taglio e quindi processati per esperimenti di ibridazione in situ whole
mount.
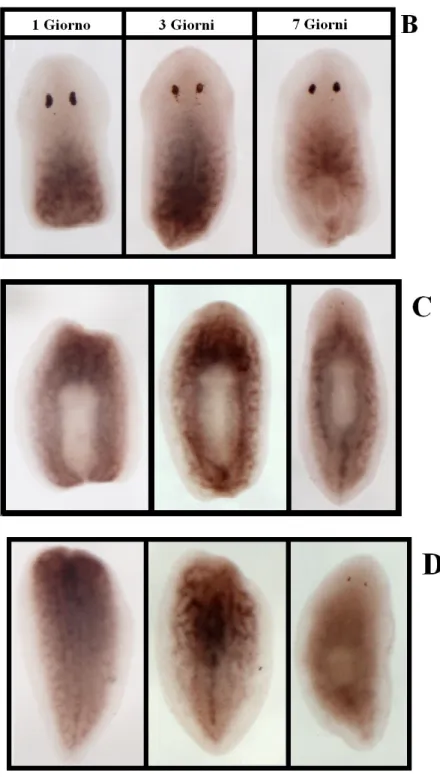
L’analisi dell’espressione ai vari tempi dal taglio ha rivelato quanto segue: ad 1 giorno dal taglio, l’espressione di DjPHB2 è evidenziabile al di sotto dell’epitelio; a 3 giorni dal taglio, DjPHB2 è espresso nelle cellule del blastema rigenerativo; successivamente DjPHB2 è espresso nella regione che sta rigenerando (Fig. 4.5).
A
Fig. 4.5 (A): schema di taglio per analisi dell’espressione del gene DjPHB2 in animali in rigenerazione, mediante esperimenti di ibridazioni in situ whole mount
Fig. 4.5: analisi dell’espressione del gene DjPHB2 mediante esperimenti di ibridazioni in situ whole mount in animali in rigenerazione (1, 3 e 7 giorni dal taglio): espressione di DjPHB2 in rigenerazione posteriore (B), bidirezionale (C) e anteriore (D)
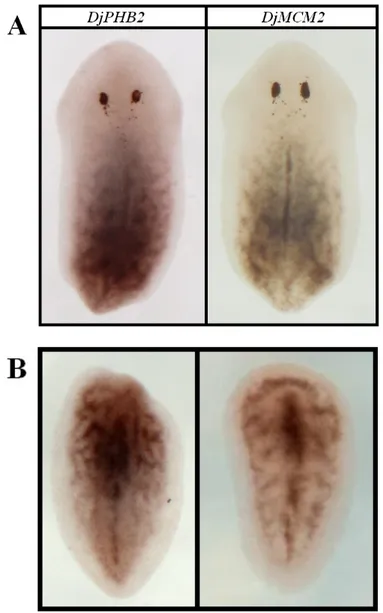
Confrontando l’espressione di DjPHB2 con quella di DjMCM2 durante la rigenerazione, appare evidente la diversa localizzazione di questi due marcatori di neoblasti: DjPHB2 è chiaramente espresso a livello del blastema rigenerativo, a differenza di DjMCM2, la cui espressione è limitata alla zona del post-blastema (Fig. 4.6).
Fig. 4.6: analisi dell’espressione del gene DjPHB2 mediante esperimenti di ibridazioni in situ whole mount in animali in rigenerazione (3 giorni dal taglio): espressione DjPHB2 e DjMCM2 in rigeneranti posteriori (A) e in rigeneranti anteriori (B)
4.3 Studi di funzione
Gli studi funzionali sono stati condotti mediante l’impiego della tecnica dell’RNAi con iniezioni successive di molecole di RNAi a doppia elica (dsDjPHB2) o di acqua per i controlli (per protocollo, vedi materiali e metodi). Lo studio è stato effettuato sia in animali intatti che in animali in rigenerazione.
4.3.1 Planarie intatte
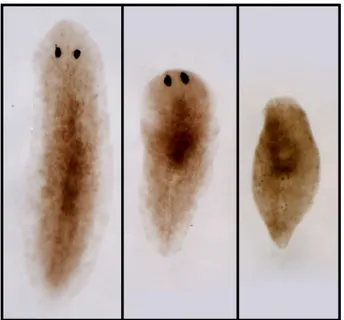
Analisi del fenotipo: a 25-30 giorni dalla prima iniezione, gli animali mostrano una diminuzione della motilità accompagnata da una progressiva degenerazione della testa con perdita iniziale delle auricole e, quindi, della parte della testa anteriore agli occhi, fino alla perdita completa della testa (Fig.
4.7). Per le planarie iniettate la morte incorre in media tra i 50 e i 60 giorni
Fig. 4.7: planarie intatte osservate al microscopio ottico: da sinistra verso destra - controllo; planaria iniettata con dsDjPHB2 che mostra una riduzione della porzione cefalica; planaria iniettata con dsDjPHB2 che mostra la degenerazione completa della testa
Analisi della distribuzione dei neoblasti: dato che il fenotipo ottenuto (degenerazione della testa) è tipico di planarie prive di neoblasti, lo scopo successivo del lavoro è stato quello di analizzare la distribuzione di queste cellule in animali in cui DjPHB2 era stato silenziato. A tale fine è stata analizzata l’espressione del marcatore molecolare di neoblasti DjMCM2.
I risultati ottenuti mostrano che il pattern di espressione del gene DjMCM2 è ridotto in animali in cui il gene DjPHB2 era stato silenziato, rispetto ai controlli.
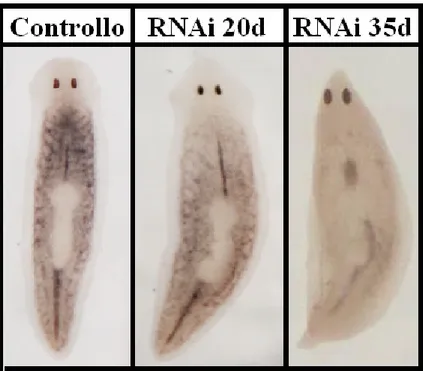
In specifico, si evidenzia un’iniziale diminuzione dei neoblasti sparsi nel parenchima, particolarmente di quelli localizzati nella zona anteriore al faringe. A 35 giorni dalla prima iniezione DjMCM2 è esclusivamente espresso lungo la linea mediana del corpo (Fig. 4.8).
Fig. 4.8: analisi dell’espressione del gene DjMCM2 mediante esperimenti di ibridazioni in situ whole mount in animali trattati con dsDjPHB2: 20 e 35 giorni dalla prima iniezione; a sinistra planaria di controllo a 35 giorni dalla prima iniezione con acqua
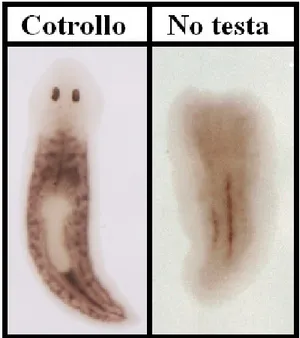
È possibile osservare la presenza del segnale lungo la linea mediana anche in planarie dal fenotipo estremo, senza testa (Fig. 4.9).
Fig. 4.9: analisi dell’espressione del gene DjMCM2 mediante esperimenti di ibridazioni in situ whole mount in animali di controllo e trattati con dsDjPHB2 45 giorni dopo la prima iniezione: a sinistra planaria di controllo iniettata con acqua; a destra planaria iniettata con dsDjPHB2 senza testa
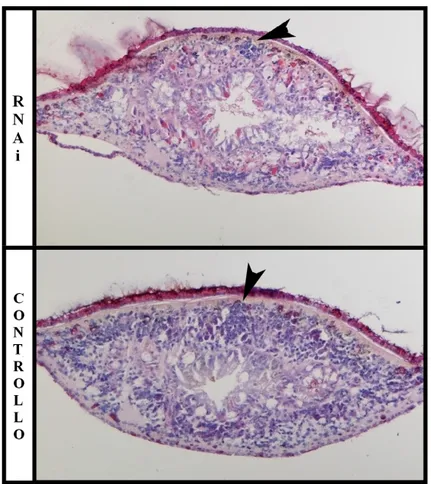
La presenza di neoblasti lungo la linea mediana del corpo e la marcata riduzione dei neoblasti sparsi nel parenchima a 35 giorni, è confermata anche dall’analisi istologica condotta su sezioni trasversali colorate in ematossilina ed eosina (Fig. 4.10).
Fig. 4.10: preparato istologico, colorato con ematossilina ed eosina, di sezioni trasversali a livello dell’intestino; le frecce indicano i “clusters” di neoblasti localizzati lungo la linea mediana dorsale del corpo di animali trattati con dsDjPHB2 e di controllo a 35 giorni dalla prima iniezione
Analisi della distribuzione di cellule in mitosi e loro quantificazione: al fine di analizzare la distribuzione delle cellule proliferanti sono stati effettuati esperimenti di immunolocalizzazione su sezioni trasversali con anticorpi anti-istone H3 fosforilato, specifico per cellule in mitosi, ossia per i neoblasti, essendo queste cellule le uniche proliferanti in planaria.
A 20 giorni dalla prima iniezione di dsDjPHB2, è possibile osservare una riduzione delle cellule in mitosi rispetto ai controlli. In accordo con quanto evidenziato dai dati precedenti di ibridazione in situ whole mount, i neoblasti rimanenti sono localizzati preferenzialmente lungo la linea mediana dorsale del corpo ed è osservabile una riduzione dei neoblasti sparsi all’interno del parenchima (Fig. 4.11).
A 35 giorni la quantità di cellule in mitosi è drasticamente ridotta: le poche cellule proliferanti che permangono sono situate esclusivamente lungo la linea mediana dorsale del corpo. (Fig. 4.12)
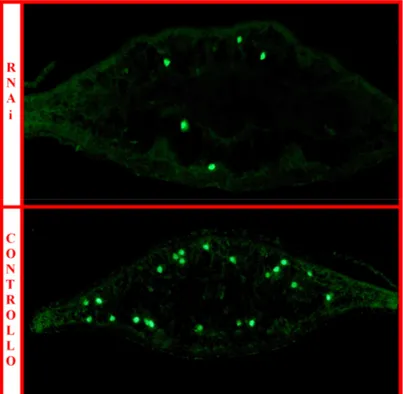
Fig. 4.12: immunolocalizzazione con anticorpi anti-istone H3 in planarie intatte di controllo e trattate con dsDjPHB2 a 35 giorni dalla prima iniezione. Le cellule in mitosi appaiono di color verde.
Allo scopo di quantificare il numero di cellule capaci di entrare nella fase M del ciclo cellulare, animali intatti di controllo e trattati con dsDjPHB2 35 giorni dopo la prima iniezione, sono stati posti in colchicina, bloccando, così, la divisione delle loro cellule. L’analisi dell’indice mitotico ha dimostrato una riduzione del 90% delle cellule proliferanti in animali silenziati per DjPHB2 rispetto al controllo (Graf. 4.1). Questi dati sono in accordo con quelli ottenuti mediante esperimenti di immunolocalizzazione.
0 20 40 60 80 100 120 K PROIB % M I T O S I
Graf. 4.1: quantificazione del numero di cellule in mitosi. Il valore del 100% è stato attribuito al controllo. La riduzione delle cellule mitotiche, osservata in animali intatti a 35 giorni dalla prima iniezione, è del 90% rispetto al controllo
4.3.2 Planarie in prima rigenerazione
Analisi del fenotipo: il silenziamento genico di DjPHB2 non interferisce con le capacità rigenerative dell’animale: il blastema rigenerativo si forma e avvengono i normali processi morfogenetici. Tuttavia, è possibile osservare un ritardo nella formazione del blastema così come nel ripristino delle parti mancanti negli animali iniettati con dsDjPHB2 rispetto ai controlli. Gli animali iniettati muoiono dopo 30-35 giorni dalla prima iniezione.
Analisi della distribuzione dei neoblasti: sebbene planarie iniettate siano in grado di rigenerare, esperimenti di ibridazione in situ whole mount, utilizzando il marcatore molecolare DjMCM2, hanno permesso di evidenziare una riduzione dei neoblasti localizzati nei clusters laterali così come di quelli sparsi in animali in cui il gene DjPHB2 era stato silenziato rispetto ai controlli. Questa differenza tra trattati e controlli è evidente maggiormente a livello delle teste in rigenerazione (Fig. 4.13).
Fig. 4.13: analisi dell’espressione del gene DjMCM2 mediante esperimenti di ibridazioni in situ whole mount in animali trattati con dsDjPHB2 e di
Successivamente, la dinamica di riduzione dei neoblasti è simile a quella osservata nelle planarie intatte: le cellule che esprimono DjMCM2, infatti, si riducono progressivamente e si ritrovano, a tempi tardivi, preferenzialmente a livello della linea mediana dorsale del corpo degli animali (Fig. 4.14).
Fig. 4.14: analisi dell’espressione del gene DjMCM2 mediante esperimenti di ibridazioni in situ whole mount in animali di controllo e trattati con dsDjPHB2 a 20 giorni dal taglio (prima rigenerazione): in alto rigenerazione posteriore; in basso rigenerazione anteriore
L’analisi dell’espressione del marcatore molecolare DjPiwi-1 rivela la presenza di cellule che esprimono questo gene lungo la linea mediana del corpo di planarie iniettate con dsDjPHB2 (Fig. 4.15).
Fig. 4.15: analisi dell’espressione del gene DjPiwi-1 mediante esperimenti di ibridazioni in situ whole mount in animali di controllo e trattati con dsDjPHB2 a 20 giorni dal taglio (in prima rigenerazione): in alto rigenerazione posteriore; in basso rigenerazione anteriore
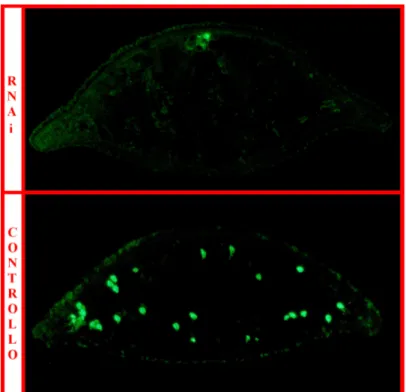
Analisi della distribuzione e della quantità di cellule in mitosi: mediante la tecnica di immunolocalizzazione con anticorpi anti-istone H3 fosforilato è stato osservato che la distribuzione delle cellule in mitosi a 20 giorni dal taglio è localizzata preferenzialmente lungo la linea mediana del corpo di animali trattati con dsDjPHB (Fig. 4.16). L’analisi dell’indice mitotico conferma la marcata riduzione (80%) dei neoblasti in animali trattati rispetto ai controlli.
Fig. 4.16: immunolocalizzazione mediante l’utilizzo di anticorpi anti-istone H3 in planarie di controllo e trattate con dsDjPHB2 a 20 giorni dal taglio (prima rigenerazione). Le cellule in mitosi appaiono di color verde
4.3.3 Planarie in seconda rigenerazione
Analisi del fenotipo: gli animali in seconda rigenerazione perdono le capacità rigenerative. In particolare, le teste non sono in grado di formare il blastema rigenerativo (95%) (Fig. 4.17 A); alcune (5%) formano inizialmente un piccolo blastema, che però regredisce in 1-2 giorni. La morte incorre dopo circa 14 giorni dal secondo taglio.
Le code, invece, riescono a formare il blastema rigenerativo anche se questo risulta essere più piccolo rispetto a quello presente nei controlli. Anche la testa rigenerata è di dimensioni ridotte rispetto a quella dei controlli ed appare arrotondata, senza le auricole, e presenta o un occhio solo, o due occhi, ma uno più piccolo ed uno più grande (Fig 4.17 B). Le planarie trattate muoiono entro 30 giorni dal taglio.
Fig. 4.17: immagini al microscopio ottico di planarie di controllo e trattate con dsDjPHB2 in seconda rigenerazione; rigenerazione posteriore a 7 giorni dal secondo taglio (A) e rigenerazione anteriore allo stesso tempo (B)
Analisi di microscopia elettronica a trasmissione (TEM): per comprendere il motivo per cui le teste non fossero in grado di formare un blastema rigenerativo, sono stati condotti esperimenti di microscopia elettronica a trasmissione. Le microfotografie mostrano la corretta chiusura della ferita, ma al di sotto dell’epitelio rimarginato, non è evidenziabile il tipico accumulo di neoblasti riscontrabile nei controlli a 3 giorni di rigenerazione dal secondo taglio, ma, piuttosto, l’accumulo di cellule in via di differenziamento (Fig.
Fig. 4.18: analisi di animali iniettati con dsDjPHB2 e di controllo a 3 giorni dal taglio mediante microscopia elettronica a trasmissione (TEM).
Nel controllo: la freccia rossa indica la zona in cui è localizzato l’epitelio rimarginato.
Nel trattato: la barra verde indica l’epitelio della ferita; il rettangolo rosso mostra cellule in via di differenziamento (RNAi). L’ingrandimento permette una visione più dettagliata dello stato di differenziamento delle cellule sottostanti l’epitelio rimarginato
Analisi della distribuzione dei neoblasti: esperimenti di ibridazione in situ
whole mount, utilizzando il marcatore molecolare DjMCM2, dimostrano che, in
planarie trattate con dsDjPHB2, incapaci di formare il blastema, a 7 giorni dal taglio (seconda rigenerazione), sono evidenziabili dei neoblasti localizzati preferenzialmente localizzati lungo la linea mediana dorsale del corpo (Fig.
4.19).
Fig. 4.19: analisi dell’espressione del gene DjMCM2 mediante esperimenti di ibridazioni in situ whole mount in animali di controllo e trattati con dsDjPHB2 a 7 giorni dal taglio (seconda rigenerazione). In figura sono mostrati animali in rigenerazione posteriore
L’analisi dell’espressione del marcatore molecolare DjPiwi-1, mediante esperimenti di ibridazione in situ whole mount, mostra la presenza, nella linea mediana del corpo degli animali trattati, di cellule positive anche per questo marcatore, a 7 giorni dal secondo taglio (Fig. 4.20).
Fig. 4.20: analisi dell’espressione del gene DjPiwi-1 mediante esperimenti di ibridazioni in situ whole mount in animali di controllo e trattati con dsDjPHB2 a 7 giorni dal taglio (seconda rigenerazione). In figura è mostrata la rigenerazione posteriore
La permanenza di neoblasti lungo la linea mediana di animali trattati con
dsDjPHB2 a 7 giorni dal taglio è confermata anche dall’analisi istologica
condotta su sezioni trasversali colorate in ematossilina ed eosina (Fig. 4.21).
Fig. 4.21: preparato istologico, colorato con ematossilina ed eosina, di sezioni trasversali a livello dell’intestino; le frecce indicano i clusters di neoblasti lungo la linea mediana dorsale del corpo dell’animale a 7 giorni dal taglio, sia in planarie in seconda rigenerazione trattate con dsDjPHB2, che nei controlli
Analisi della distribuzione e della quantità di cellule in mitosi: anche per gli animali in seconda rigenerazione sono stati condotti esperimenti di immunolocalizzazione su sezioni trasversali con anticorpi anti-istone H3 fosforilato per l’analisi della distribuzione delle cellule proliferanti (neoblasti).
A 7 giorni dal secondo taglio, in animali trattati con dsDjPHB2, le pochissime cellule proliferanti rimaste si trovano esclusivamente lungo la linea mediana dorsale del corpo (Fig. 4.22); in molte sezioni non è addirittura possibile riscontrare alcuna cellula positiva all’istone H3 fosforilato.
Fig. 4.22: immunolocalizzazione con anticorpi anti-istone H3 a 7 giorni dal taglio in planarie di controllo e trattate con dsDjPHB2 in seconda rigenerazione. Le cellule in mitosi appaiono di color verde
Per quantificare la riduzione delle cellule proliferanti, osservata negli esperimenti di immunolocalizzazione, è stato valutato l’indice mitotico a 3 giorni dal taglio in animali di controllo e trattati con dsDjPHB2, sia durante la rigenerazione posteriore che anteriore. Nelle teste, in cui il trattamento con l’RNAi determina effetti più marcati, già a 3 giorni dal taglio si ha, nel trattato, una riduzione del 95% delle cellule in mitosi rispetto al controllo, mentre nelle code la riduzione è del 70% (Graf. 4.2).
TESTE 3D II° RIGENERAZIONE 0 20 40 60 80 100 120 K PROIB % M I T O S I
CODE 3D II° RIGENERAZIONE
0 20 40 60 80 100 120 K PROIB % M I T O S I
⋅Graf. 4.2: quantificazione del numero di cellule in mitosi. L’indice è stato calcolato attribuendo al controllo il valore del 100%. La riduzione delle cellule mitotiche, osservata in animali in seconda rigenerazione a 3 giorni