M
ATERIALI E METODI
Pazienti
Circa 730 soggetti con diabete tipo 2 sono stati arruolati nello studio. Criteri di inclusione sono stati:
• l'età compresa tra 30 e 65 anni,
• l'età alla diagnosi del diabete superiore a 30 anni,
• il trattamento dietetico normoglicemizzante e/o con ipoglicemizzanti orali,
• il trattamento insulinico purchè iniziato dopo almeno due anni dal momento della diagnosi del diabete,
• la durata della malattia superiore o eguale a 2 anni.
Sono stati inclusi nello studio solo soggetti di origine caucasica; i pazienti non rappresentativi dell'etnia locale sono stati esclusi. Un semplice questionario è stato sottoposto a ciascun paziente reclutato nello studio. Così, in accordo all'EURODIAB Family Study ("EURODIAB IDDM Complication Study"), abbiamo ottenuto per ciascun paziente informazioni circa l'origine geografica delle due generazioni precedenti.
Un opportuno gruppo di controllo, con età sovrapponibile a quella dei pazienti diabetici, è stato accuratamente selezionato. Lo studio è stato approvato dal Comitato Etico dell'Università di Pisa. Ciascun soggetto ha concesso il consenso informato sia per studi clinici che per studi genetici.
La presenza di nefropatia diabetica è stata valutata sulla base dei livelli della creatinina plasmatica e dell'escrezione urinaria di albumina (AER). L’AER è stata misurata su almeno tre raccolte di urine delle 24 ore, ottenute in un periodo di 3-6 mesi precedente il reclutamento. I pazienti sono stati stratificati in soggetti senza danno renale, cioè normoalbuminurici (AER medio < 20 µg/min), soggetti con nefropatia incipiente, cioè con microalbuminuria persistente (AER compreso tra 20 e 200 µg/min, in almeno due delle tre raccolte urinarie eseguite) e individui con nefropatia conclamata, macroalbuminurici (AER > 200 µg/min; almeno in due raccolte su tre). I soggetti con creatinina plasmatica > 1.5 mg/dl, sono stati inclusi tra quelli con nefropatia conclamata.
Per la diagnosi di nefropatia conclamata erano richieste la coesistenza di retinopatia diabetica background o proliferante e/o l'assenza di evidenze cliniche e di laboratorio di cause di proteinuria diverse dalla nefropatia diabetica. Anche se la microalbuminuria e persino la nefropatia conclamata sono presenti con una certa frequenza già al momento della diagnosi del diabete (503), particolare attenzione è stata dedicata ai pazienti con proteinuria clinica persistente (nefropatia conclamata) comparsa entro 5 anni dalla diagnosi del diabete. Nei casi in cui lo stadio della nefropatia non poteva essere accuratamente confermato dai dati clinici di ciascun paziente, gli eventuali trattamenti con ACE-inibitori, antagonisti del recettore per l'angiotensina II e bloccanti dei canali del calcio non diidropiridinici, sono stati sospesi almeno 10 giorni prima della esecuzione delle raccolte urinarie. In questi pazienti, tre raccolte urinarie erano ottenute nell'arco delle successive due-tre settimane. Trattamenti con altri antiipertensivi erano consentiti. Nessuno degli altri trattamenti farmacologici (antiaggreganti piastrinici, ipolipemizzanti, etc) era sospeso in relazione alla inclusione nello studio.
L'escrezione urinaria di albumina è stata misurata con metodica immunoturbidimetrica (Tina-quant Albumin, Boehringer Mannheim) su aliquote conservate a -20°C. Il metodo ha sensibilità pari a 3.0 µg/mL e un coefficiente di variazione inter-assay inferiore al 8%, per un intervallo compreso tra 3 e 400 µg/mL.
La retinopatia, classificata come assente, background e proliferante, è stata valutata mediante oftalmoscopia diretta e foto del fondo oculare (2 campi, maculare e nasale, bilateralmente). La presenza di vasi neoformati, di proliferazioni fibrose, di emorragie vitreali e/o pre-retiniche e di esiti di fotocoagulazione laser definiscono lo stadio "proliferante" della retinopatia (134).
La diagnosi di ipertensione arteriosa è stata posta per valori di pressione sistolica > 140 mmHg e/o pressione diastolica > 90 mmHg in almeno tre diverse occasioni nei tre mesi precedenti lo studio o in presenza di terapia con anti-ipertensivi (128, 504).
La diagnosi di cardiopatia ischemica è stata posta in base alla storia personale di angina, infarto del miocardio o procedure di rivascolarizzazione coronarica e/o in base ad alterazioni elettrocardiografiche suggestive (Codice Minnesota) di cardiopatia ischemica.
L'emoglobina glicata (HbA1c) è stata misurata tramite HPLC (Diamat, Bio-Rad Labs., Richmond, CA). L'intervallo di normalità è compreso tra 4.1 e 6.1%.
L'indice di massa corporea (BMI) è stato calcolato come peso/altezza2
(Kg/m2
).
Con metodica standard, la reazione di Jaffè, si è provveduto alla misurazione della creatinina sierica.
Colesterolo totale, colesterolo HDL e trigliceridi sono stati misurati con metodica enzimatica (Boehringer-Mannheim). Il colesterolo LDL è stato calcolato con la formula di Friedewald.
Lo studio dei polimorfismi Leu55Met e Gln192Arg del gene che codifica per PON1 e del polimorfismo triallelico dell’ApoE sono stati effettuati utilizzando le seguenti procedure sperimentali:
•
•• estrazione di DNA da leucociti umani, ottenuti da sangue intero precedentemente congelato
•
•• amplificazione del frammento genico contenente la mutazione in oggetto mediante PCR (Polymerase Chain Reaction)
•
•• rilevazione del polimorfismo mediante analisi con enzima di restrizione (RFLP, Restriction Fragment Lenght Polymorphism).
Estrazione di DNA
Il DNA è stato estratto da leucociti ottenuti da campioni di sangue periferico, precedentemente prelevato in EDTA dai pazienti selezionati e conservato a -20°C. L’estrazione salina del DNA è stata condotta utilizzando un apposito protocollo per ottenere DNA di cellule eucariote da sangue congelato in accordo alla metodica descritta da Blin. e Stafford. (505). Tutti i reagenti utilizzati sono stati accuratamente sterilizzati prima dell’uso. Da ciascun campione di sangue portato a temperatura ambiente, vengono ottenute due aliquote di 5 mL ciascuna (estrazione in doppio). Le aliquote vengono poste in tubi falcon sterili e trattate con 45 mL di soluzione contenente Tris 20 mM, EDTA 5 mM, NaCl 150 mM e Triton X100. I campioni sono miscelati ed incubati in ghiaccio per 15 minuti, quindi centrifugati a 3000 rpm per 15 minuti alla temperatura di 4°C.
Eliminato il sovranatante, il pellet viene nuovamente sospeso nella soluzione TE/NaCl/Triton, agitato, incubato a 4°C per 15 minuti e centrifugato a 3000 rpm per 15 minuti. Il nuovo pellet così ottenuto viene sospeso in 2.5 mL di soluzione contenente Tris 20 mM, EDTA 5 mM, NaCl 150 mM, e trasferito in provette di vetro sterili.
Si aggiungono 150 µL di proteinasi K (10 mg/mL) e 100 µL di sodio dodecil solfato (SDS) al 10%, allo scopo di degradare le proteine residue. I campioni vengono quindi incubati per tutta la notte a 37°C; si procede ad aggiungere 1 mL di NaCl saturo e a coprire le provette con parafilm. Agitati per almeno 15 secondi, i campioni sono centrifugati a 2500 rpm per 15 minuti (4°C). Il sovranatante limpido, contenente il DNA, viene raccolto, evitando accuratamente di aspirare le proteine precipitate, e trasferito in provette di vetro sterili. I campioni vengono di nuovo agitati per almeno 15 secondi e successivamente centrifugati a 2500 rpm per 15 minuti, aggiungendo eventualmente, se il campione mostra evidenti impurità, 1 mL di NaCl saturo. Raccolto il sovranatante, viene aggiunta una quantità doppia in volume di etanolo assoluto; questa procedura permette di ottenere la precipitazione del DNA. Le provette, coperte con parafilm, vengono agitate delicatamente fino alla visualizzazione di una nubecola biancastra che tende a raccogliersi a gomitolo, il DNA. Il gomitolo viene raccolto sulla estremità di una pipetta pasteur uncinata e lasciato asciugare sotto una lampada o all’aria, in ambiente sterile. Le pipette pasteur, con il DNA essiccato vengono poste in eppendorf contenenti 100 µL di TE (Tris 10 mM, EDTA 1 mM) a pH 8.0 e lasciate a 37°C per almeno due ore. Disciolto il DNA nella soluzione, le pipette pasteur sono rimosse ed i campioni conservati a 4°C, per pochi mesi, oppure a -20°C, temperatura alla quale si mantengono inalterati per lunghi periodi di tempo. Benché la conservazione del DNA in una soluzione composta da Tris-EDTA (TE) risulti altamente efficiente, il TE può avere un effetto inibitorio sull’amplificazione genica (PCR). Pertanto, una aliquota del DNA conservato in TE, viene opportunamente trattata e disciolta in acqua bidistillata sterile; la procedura richiede l’utilizzo di NaCl ed etanolo freddo per precipitare il DNA conservato in TE. Dopo incubazione per l'intera durata della notte e dopo centrifugazione dei campioni si ottiene un pellet di DNA che viene quindi disciolto in acqua e conservato a 4°C. Prima di essere sottoposti ad amplificazione genica, i campioni di DNA vengono controllati su gel d’agarosio, utilizzando un marker di peso molecolare per DNA genomico, e tarati utilizzando uno spettrofotometro.
Taratura del DNA
Il DNA estratto da leucociti viene tarato utilizzando uno spettrofotometro che ci permette di valutare concentrazione e purezza dei campioni. Il rapporto tra l'assorbanza a 260 nm e l'assorbanza a 280 nm (A260/A280) è usato come misura qualitativa della purezza del DNA: un campione è considerato puro quando il rapporto A260/A280 è compreso tra 1.7 e 2.0. Se il rapporto è inferiore o superiore esistono contaminazioni rispettivamente di proteine o di RNA che possono interferire nelle successive analisi.
Con l'estrazione vengono ottenuti approssimativamente 500 ng/µL di DNA da ogni campione (5 mL) di sangue.
S
S
S
t
t
t
u
u
u
d
d
d
i
i
i
o
o
o
d
d
d
e
e
e
l
l
l
p
p
p
o
o
o
l
l
l
i
i
i
m
m
m
o
o
o
r
r
r
f
f
f
i
i
i
s
s
s
m
m
m
o
o
o
ε
ε2
ε
2
2
,
,
,
ε
ε3
ε
3
3
,
,
,
ε
ε4
ε
4
4
d
d
d
e
e
e
l
l
l
l
l
l
'
'
'
A
A
A
p
p
p
o
o
o
E
E
E
Il gene dell'ApoE è polimorfo. Gli alleli corrispondenti alle tre isoforme dell'ApoE sono denominati ε2, ε3, ε4 e le corrispondenti proteine sono dette E2, E3, E4. Dal polimorfismo genetico risultano sei fenotipi alternativi, che corrispondono alle tre situazioni di omozigosi (E2/E2, E3/E3, E4/E4) e alle tre possibili situazioni di eterozigosi (E2/E3, E2/E4, E3/E4).
PCR
La reazione di polimerizzazione è stata effettuata utilizzando le seguenti condizioni (506): 1 µg di DNA genomico, 30 pmoli di ciascun primer (F4: 5'-ACAGAATTCGCCCC-GGCCTGGTACAC-3', e F6: 5'-TAAGCTTGGCACGGCTGTCCAAGGA-3'), 1 mM dNTP, 10 mM Tris-HCl pH 8.3, 50 mM KCl, 1.5 mM MgCl2, 10% DMSO, 1.5 U di Taq-polimerasi in un volume totale di 30 µl.
La miscela di reazione è denaturata a 95°C per 5 minuti quindi sottoposta a 30 cicli di amplificazione ciascuno composto di un ciclo di denaturazione a 95°C per 60 secondi, uno di annealing a 60°C per 60 secondi, un ciclo di estensione a 70°C per 2 minuti, ed infine una estensione finale a 72°C per 7 minuti. I prodotti di PCR, pari a 244 bp, sono controllati su gel di agarosio al 1.4%.
RFLP
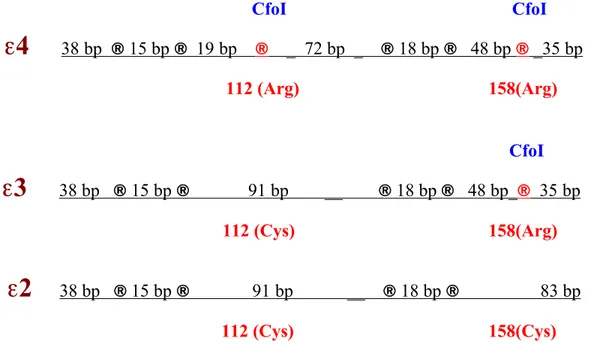
Ai prodotti amplificati con PCR viene aggiunto tampone specifico e 5 unità di enzima CfoI o HhaI (New England Biolabs), enzimi che effettuano digestione endonucleasica sulla stessa sequenza nucleotidica GCGC (figura 18). Successivamente, la miscela di reazione per la digestione delle sequenze dell'ApoE viene incubata a 37°C per 4 ore.
CfoI CfoI
ε4
38 bp ® 15 bp ® 19 bp ® _ 72 bp _ ® 18 bp ® 48 bp ® _35 bp 112 (Arg) 158(Arg) CfoIε3
38 bp ® 15 bp ® 91 bp __ ® 18 bp ® 48 bp_® 35 bp 112 (Cys) 158(Arg)ε2
38 bp ® 15 bp ® 91 bp __ ® 18 bp ® 83 bp 112 (Cys) 158(Cys)Figura 18 - Mappa di restrizione di CfoI. Siti di taglio dell’enzima sui frammenti di 244 bp corrispondenti ai
tre diversi alleli dell’Apo E.
Analisi elettroforetica su gel di poliacrilammide
Utilizzando un gel di poliacrilammide al 12%, i campioni amplificati e digeriti sono sottoposti a corsa elettroforetica effettuata per 2 ore a corrente costante (150 mA) utilizzando la cella D Gene System (Bio-Rad Labs., Richmond, CA).
I frammenti di restrizione sono poi visualizzati mediante colorazione all’argento del gel di poliacrilammide.
In figura 19 è disegnato uno schema dei possibili profili elettroforetici che si osservano dopo colorazione del gel ed i loro corrispondenti genotipi.
Scala in bp 2/2 3/3 4/4 2/3 2/4 3/4 --- 91 --- --- ---83 --- ---72 --- ---48 ---38 ---35 ---19 --- ---18 ---15
---Figura 19 - Profili elettroforetici dei 6 genotipi che si ottengono dopo restrizione enzimatica con CfoI dell’amplificato di 244 bp dell’ApoE.
La dimensione dei frammenti di restrizione ottenuti viene stimata mediante comparazione con le bande di peso noto di un marker (25 bp DNA Step Ladder, Promega).
La figura 20 mostra la sequenza (244 bp) che codifica l'isoforma E4 dopo amplificazione mediante PCR con i primer F4 e F6. La figura mostra i sei siti di taglio GCGC di CfoI nella sequenza dell'isoforma E4. Le sostituzioni nucleotidiche e aminoacidiche che distinguono le isoforme E2 e E3 sono allineate con la sequenza dell'isoforma E4. La sequenza dell'isoforma E3 codifica un residuo di cisteina alla posizione 112 (TGC) la quale elimina il sito di taglio nella sequenza E4; la sequenza dell'isoforma E3 ha 5 siti di taglio per CfoI. La sequenza E2 codifica cisteine in posizione 112 e 158 che eliminano i due siti di taglio relativi alla sequenza nucleotidica di E4, risultando in 4 siti di taglio.
In figura 21 è mostrato un esempio di gel di poliacrilammide dedicato alla analisi del polimorfismo triallelico dell'ApoE.
E4 DNA TAAGCTTG GCA CGG CTG TCC AAG GAG CTG CAG GCG GCG CAG GCC CGG CTG GGC
E4 proteina Ala Arg Leu Ser Lys Glu Leu Gln Ala Ala Gln Ala Arg Leu Gly
112
E2 T
E3 T
E4 GCG GAC ATG GAG GAC GTG CGCGGC CGC CTG GTG CAG TAC CGC GGC GAG GTG CAG GCC ATG
E4 Ala Asp Met Glu Asp Val Arg Gly Arg Leu Val Gln Tyr Arg Gly Glu Val Gln Ala Met
E3 Cys
E2 Cys
E4 CTC GGC CAG AGC ACC GAG GAG CTG CGG GTG CGC CTC GCC TCC CAC CTGCGC AAG CTG CGT
E4 Leu Gly Gln Ser Thr Glu Glu Leu Arg Val Arg Leu Ala Ser His Leu Arg Lys Leu Arg
158
E2 T
E3 C
E4 AAG CGG CTC CTC CGC GAT GCC GAT GAC CTG CAG AAGCGC CTG GCA GTG TAC CAG GCC GGG
E4 Lys Arg Leu Leu Arg Asp Ala Asp Asp Leu Gln Lys Arg Leu Ala Val Tyr Gln Ala Gly
E3 Arg
E2 Cys
E4 GCGAATTCTGT
Figura 20 - Sequenza nucleotidica e aminoacidica delle regioni amplificate che codificano le isoforme comuni
dell'ApoE. In rosso sono indicate le sequenze riconosciute da CfoI. In verde sono indicate le sequenze dei primer F6 e F4' (reverse complement di F4).
Figura 21 - Esempio di gel di poliacrilammide dedicato all'analisi del polimorfismo triallelico dell'ApoE.
marker
ε3/ε3 ε3/ε3 ε3/ε3 ε2/ε3 ε3/ε3 ε3/ε4 ε3/ε3 ε3/ε4
91 bp 72 bp
S
S
S
t
t
t
u
u
u
d
d
d
i
i
i
o
o
o
d
d
d
e
e
e
l
l
l
p
p
p
o
o
o
l
l
l
i
i
i
m
m
m
o
o
o
r
r
r
f
f
f
i
i
i
s
s
s
m
m
m
o
o
o
L
L
L
e
e
e
u
u
u
5
5
5
5
5
5
M
M
M
e
e
e
t
t
t
d
d
d
i
i
i
P
P
P
O
O
O
N
N
N
1
1
1
Il polimorfismo Leu55Met è una variante caratterizzata dalla sostituzione nell'esone 3 di una timina (T) con una citosina (C); questo comporta l'inserimento di una metionina (allele M) al posto di una leucina (allele L) alla posizione 55 della proteina matura.
PCR
La reazione di polimerizzazione è stata effettuata utilizzando le seguenti condizioni: 0.5 µg di D N A g e n o m i c o , 2 0 p m o l i d i c i a s c u n p r i m e r ( 4 5 1 ) ( s e n s o : 5 ' -GAAGAGTGATGTATAGCCCCAGTTT-3', e antisenso: 5'-TTTAATCCAGAGCTAA-TGAAAGCC-3'), Mastermix (Promega) contenente Taq polimerasi (1U) in un volume totale di 20 µL.
La miscela di reazione è denaturata a 95°C per 5 minuti, quindi sottoposta a 35 cicli di amplificazione ciascuno composto di un ciclo di denaturazione a 95°C per 60 secondi, uno di annealing a 61°C per 60 secondi ed un ciclo di estensione a 72°C per 60 secondi, seguito da una estensione finale a 72°C per 10 minuti.
I prodotti di PCR, pari a 170 bp, sono controllati su gel di agarosio al 1.4%. RFLP
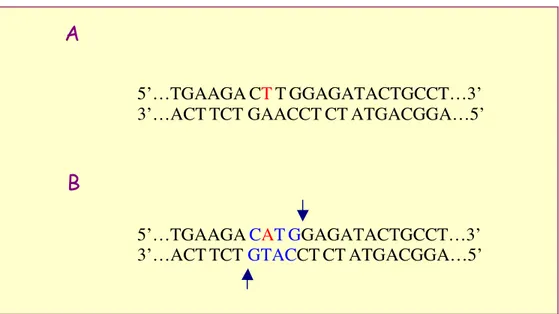
Ai prodotti amplificati con PCR viene aggiunto un tampone specifico e 2 U/µL di enzima NlaIII (New England Biolabs) (figura 22a). Successivamente la miscela di reazione per la digestione delle sequenze PON1 viene incubata a 37°C per tutta la notte.
Analisi elettroforetica su gel di poliacrilammide
Utilizzando un gel di poliacrilammide al 12%, i campioni amplificati e digeriti sono sottoposti a corsa elettroforetica effettuata per 2 ore a corrente costante (150 mA) utilizzando la cella D Gene System (Bio-Rad Labs., Richmond, CA).
I frammenti di restrizione sono poi visualizzati mediante colorazione all’argento del gel di poliacrilammide. La dimensione dei frammenti di restrizione ottenuti è stimata mediante confronto con le bande di peso noto di un marker (25 bp DNA Step Ladder, Promega). L'allele L non contiene il sito di taglio per NlaIII: su gel di poliacrilammide i soggetti omozigoti L/L presenteranno una sola banda corrispondente ad un frammento di 170 bp.
L'allele M, invece, presenta il sito di restrizione per NlaIII; i campioni amplificati di soggetti omozigoti M/M presenteranno, dopo digestione enzimatica, due frammenti pari a 126 bp e 44 bp. Infine, gli eterozigoti L/M presenteranno tre bande pari a 170 bp, 126 bp e 44 bp (figura 23).
Figura 22a - A Sequenza nucleotidica wild type della regione polimorfica 55; in rosso è indicato il nucleotide che
può variare. B Sequenza nucleotidica mutata della regione polimorfica 55; in rosso è indicato il nucleotide che può variare; in blu è indicata la sequenza di basi riconosciuta da NlaIII, le frecce indicano i siti di taglio dell’enzima.
Figura 22b: A Sequenza nucleotidica wild type della regione polimorfica 192; in rosso è indicato il nucleotide che
può variare. B Sequenza nucleotidica mutata della regione polimorfica 192; in rosso è indicato il nucleotide che può variare; in blu è indicata la sequenza di basi riconosciuta da AlwI, le frecce indicano i siti di taglio dell’enzima.
A
B
5’…TTCTTGACCCCTACTTACGATCCTGG…3’
3’…AAGAACTGGGGATGAATGCTAGGACC…5’
5’…TTCTTGACCCCTACT TACAATCCT GG…3’
3’…AAGAACTGGGGATGAATGTTAGGACC…5’
A
B
5’…TGAAGACATGGAGATACTGCCT…3’ 3’…ACTTCT GTACCTCTATGACGGA…5’ 5’…TGAAGACTTGGAGATACTGCCT…3’ 3’…ACTTCT GAACCTCTATGACGGA…5’
Figura 23 - Esempio di gel di poliacrilammide dedicato alla analisi del polimorfismo Leu55Met di PON1.
S
S
S
t
t
t
u
u
u
d
d
d
i
i
i
o
o
o
d
d
d
e
e
e
l
l
l
p
p
p
o
o
o
l
l
l
i
i
i
m
m
m
o
o
o
r
r
r
f
f
f
i
i
i
s
s
s
m
m
m
o
o
o
G
G
G
l
l
l
n
n
n
1
1
1
9
9
9
2
2
2
A
A
A
r
r
r
g
g
g
d
d
d
i
i
i
P
P
P
O
O
O
N
N
N
1
1
1
Il polimorfismo Gln192Arg è caratterizzato dalla sostituzione dell'aminoacido glutamina (allele Q, isoenzima A) alla posizione 192 con una arginina (allele R, isoenzima B), dovuto alla sostituzione A → G nell'esone 6 del gene che codifica per PON1.
PCR
La reazione di polimerizzazione è stata effettuata utilizzando le seguenti condizioni: 0.5 µg di DNA genomico, 20 pmoli di ciascun primer (451) (senso: 5'-TATTGTTGCTGT-GGGACCTGAG-3', e antisenso: 5'-CACGCTAAACCCAAATACATCTC-3'), Mastermix (Promega) contenente Taq polimerasi (1U) in un volume totale di 20 µL. La miscela di reazione è denaturata a 95°C per 5 minuti, quindi sottoposta a 35 cicli di amplificazione ciascuno composto di un ciclo di denaturazione a 95°C per 60 secondi, uno di annealing a 61°C per 60 secondi ed un ciclo di estensione a 72°C per 60 secondi, seguito da una estensione finale a 72°C per 10 minuti. I prodotti di PCR, pari a 99 bp, sono controllati su gel di agarosio al 1.4%.
RFLP
Ai prodotti amplificati con PCR viene aggiunto un tampone specifico e 2 U/µL di enzima AlwI (New England Biolabs) (figura 22b). Successivamente la miscela di reazione per la digestione delle sequenze PON1 viene incubata a 37°C per tutta la notte.
Analisi elettroforetica su gel di poliacrilammide
Utilizzando un gel di poliacrilammide al 12%, i campioni amplificati e digeriti sono sottoposti a corsa elettroforetica effettuata per 2 ore a corrente costante (150 mA) utilizzando la cella D Gene System (Bio-Rad Labs., Richmond, CA).
I frammenti di restrizione sono poi visualizzati mediante colorazione all’argento del gel di poliacrilammide. La dimensione dei frammenti di restrizione ottenuti è stimata mediante confronto con le bande di peso noto di un marker (25 bp DNA Step Ladder, Promega).
L'allele R presenta un sito di restrizione che porta a due prodotti: un frammento di 68 bp ed uno di 31 bp, la presenza di tali frammenti sarà indicativa di omozigosi R/R; l'allele Q non ha siti di taglio per AlwI, quindi sul gel sarà presente un unico frammento di 99 bp nei soggetti omozigoti Q/Q; il genotipo Q/R presenterà, dopo digestione enzimatica, tre frammenti pari a 99 bp, 68 bp e 31 bp (figura 24).
M
MM QQQ///RRR QQQ///QQQ QQQ//Q/QQ QQQ///RRR QQQ///QQQ QQQ///QQQ RRR///RRRQQQ///QQQQQQ///RRR QQQ///QQQ QQQ///QQQ QQQ///RRR
Figura 24 - Esempio di gel di poliacrilammide dedicato alla analisi del polimorfismo Gln192Arg di PON1.
31 bp 68 bp 99 bp
A
A
A
n
n
n
a
a
a
l
l
l
i
i
i
s
s
s
i
i
i
d
d
d
e
e
e
l
l
l
l
l
l
a
a
a
a
a
a
t
t
t
t
t
t
i
i
i
v
v
v
i
i
i
t
t
t
à
à
à
d
d
d
i
i
i
P
P
P
O
O
O
N
N
N
1
1
1
Prima dell’analisi dell’attività di PON1, il siero dei controlli e dei diabetici è preincubato con 5 µmol/L di eserina per 10 minuti, a temperatura ambiente, per inibire l’attività sierica della butirilcolinesterasi, che è elevata nel diabete e potrebbe interferire con la determinazione dell’attività della paraoxonasi nel siero di individui con diabete. Esperimenti hanno dimostrato che queste condizioni inibiscono completamente la butirilcolinesterasi senza danneggiare l’attività di PON1 (469).
L’attività di PON1, in accordo alla metodica descritta da Mackness B e coll. (461), è misurata aggiungendo al siero 1 mL di tampone TRIS-HCl (100 mmol/L, pH: 8.0) contenente 2 mmol/L di CaCl2 e 5.5 mmol/L di paraoxon (0,0-dietihyl-0-p-nitrophenylphosphate; Sigma, Poole, UK). La
velocità di generazione del p-nitrofenolo è determinata a 25°C, con l’uso di uno spettrofotometro a registrazione continua ad una lunghezza d'onda di 405 nm.
Per controllare la variabilità intra-assay abbiamo dosato più volte lo stesso campione all'interno di uno stesso saggio (C.V.: 4.8%); per controllare la variabilità inter-assay abbiamo misurato più volte l'attività di uno stesso campione in dosaggi diversi (C.V.: 20% per valori bassi, C.V.: 11.3% per valori alti).
D
D
D
e
e
e
t
t
t
e
e
e
r
r
r
m
m
m
i
i
i
n
n
n
a
a
a
z
z
z
i
i
i
o
o
o
n
n
n
e
e
e
d
d
d
e
e
e
l
l
l
l
l
l
a
a
a
c
c
c
o
o
o
n
n
n
c
c
c
e
e
e
n
n
n
t
t
t
r
r
r
a
a
a
z
z
z
i
i
i
o
o
o
n
n
n
e
e
e
p
p
p
l
l
l
a
a
a
s
s
s
m
m
m
a
a
a
t
t
t
i
i
i
c
c
c
a
a
a
d
d
d
e
e
e
l
l
l
l
l
l
e
e
e
L
L
L
D
D
D
L
L
L
o
o
o
s
s
s
s
s
s
i
i
i
d
d
d
a
a
a
t
t
t
e
e
e
Negli stessi pazienti con diabete tipo 2, reclutati per lo studio dei polimorfismi, sono stati determinati i livelli plasmatici di LDL ossidate, un importante fattore di rischio indipendente di malattia coronarica (497). La misura della concentrazione delle LDL ossidate è stata effettuata utilizzando il kit “Oxidized LDL ELISA” (Mercodia AB, Uppsala, Sweden). I campioni di plasma utilizzati, sono stati ottenuti mediante prelievi in EDTA, centrifugati e conservati a –20°C.
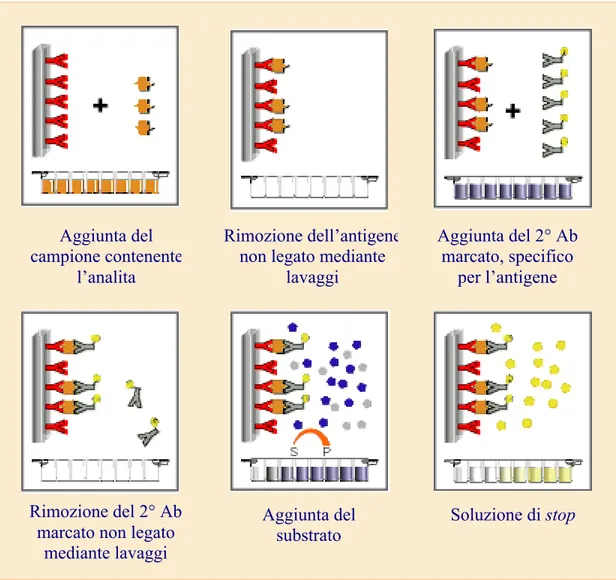
Il saggio immunoenzimatico utilizzato si basa sulla tecnica diretta a sandwich in cui due anticorpi monoclonali reagiscono con determinanti antigenici distinti sulla molecola apoB ossidata. Durante l’incubazione le LDL ossidate presenti nel campione reagiscono con anticorpi anti-LDL-ox legati al pozzetto di microtitolazione. Dopo il lavaggio, che rimuove le componenti di plasma non reattivo, un anticorpo anti-ApoB coniugato con perossidasi riconosce l’LDL-ox legata alla fase solida. Effettuata la seconda incubazione con l’anti-apoB coniugato ed un successivo lavaggio che rimuove l’anticorpo non legato, il complesso LDL-ox-coniugato viene rilevato mediante reazione con 3,3’,5,5’-Tetrametilbenzidina (TMB). L’aggiunta di acido permette lo sviluppo di una colorazione finale la cui intensità viene determinata mediante lettura spettrofotometrica ad una lunghezza d'onda di 450 nm (figura 25).
Figura 25 - Principio del metodo ELISA impiegato nel dosaggio delle LDL ossidate.
A causa dell’alta sensibilità del saggio, i campioni vengono diluiti mediante due passaggi successivi:
diluizione 1/81 → 25 µL di campione + 2000 µL di soluzione tampone
diluizione 1/6561 → 25 µL di diluizione del campione 1/81 + 2000 µL di soluzione tampone
La diluizione viene effettuata lo stesso giorno in cui si procede con il saggio. Il test viene eseguito in doppio per ciascun campione. In ogni saggio, quale controllo interno, le LDL-ox sono state dosate su quello stesso campione che in precedenza era stato utilizzato per definire la variabilità intra-assay (dosaggio multiplo nello stesso esperimento) e quella inter-assay (dosaggio di uno stesso campione in esperimenti successivi).
Rimozione dell’antigene non legato mediante
lavaggi
Soluzione di stop Aggiunta del
substrato Rimozione del 2° Ab
marcato non legato mediante lavaggi Aggiunta del campione contenente l’analita Aggiunta del 2° Ab marcato, specifico per l’antigene
Calcolo dei risultati
Dalle letture spettrofotometriche vengono mediati i valori ottenuti per ciascuna doppietta. I valori di assorbanza ottenuti per gli standards sono utilizzati per costruire una curva di taratura che mette in relazione le concentrazioni note degli standards e le relative assorbanze.
Le concentrazioni dei campioni e dei controlli, espresse in U/L, vengono calcolate utilizzando l’equazione della curva di taratura e moltiplicando il valore ottenuto per il fattore di diluizione (6.561).