1. Stato dell’arte dei biosensori di DNA
Lo scopo di questo lavoro è la realizzazione di dispositivi MEMS, quali microbilance risonanti in modalità torsionale proposti come biosensori a rivelazione gravimetrica. I sensori di massa infatti, tramite opportuni trattamenti biochimici, possono risultare sensibili ad un certo tipo di molecola. Un’interessante applicazione è quella per la rivelazione del DNA. Pertanto, poiché questi dispositivi appartengono ad una particolare categoria di biosensori, ovvero biosensori di DNA a rivelazione gravimetrica, vale la pena di presentare una panoramica sui biosensori in generale.
I biosensori stanno assumendo negli ultimi anni un sempre crescente interesse grazie alle loro numerose applicazioni: dal settore ambientale per il monitoraggio dell’inquinamento, all’agro-alimentare per la valutazione della qualità dei cibi o per la ricerca sugli OGM, al settore industriale per il monitoraggio di molti processi, a quello bio-medicale per la diagnostica di vario genere e il controllo di molti parametri.
Caratteristica fondamentale di un biosensore è l’interazione tra un elemento sensibile di origine biologica (es. un enzima o un anticorpo) e un trasduttore [1].
L’obiettivo di realizzare un biosensore “ideale” caratterizzato da facilità d’uso, costi contenuti, specificità, tempi di risposta ridotti ha reso e rende necessari elevati investimenti a supporto della ricerca scientifica.
Una recente review [1] riporta che nel mondo sono presenti all’incirca duecento aziende che si occupano di biosensori. La maggior parte di esse produce dispositivi basati su tecnologie consolidate mentre solo poche ne
stanno sviluppando di nuove. La commercializzazione della tecnologia biosensoristica è infatti rimasta indietro rispetto ai risultati ottenuti nei laboratori di ricerca per ragioni di costi, problemi di stabilità e sensibilità. Nel settore medicale, elemento di traino nel mercato, la maggior parte dei dispositivi in commercio si basa su meccanismi di trasduzione elettrochimici e ottici. I biosensori per l’analisi quantitativa del glucosio presente nel sangue coprono l’85% del mercato, visto il problema dilagante dell’obesità e l’allarmante aumento del diabete nei paesi industrializzati. Tra i dispositivi in commercio si annoverano: Nova’s StatStripTM Glucose Monitor della Nova biomedical, il Glucometer® Elite Diabetes Care System della Bayer AG (Diagnostic division).
Un posto rilevante è occupato anche dalla Neogen Corporation che commercializza una gamma di prodotti dedicati alla sicurezza del cibo e degli animali basati su procedimenti di analisi biochimiche tradizionali (test ELISA).
Altri dispostivi in commercio riguardano il monitoraggio di interazioni biologiche in tempo reale come la tecnologia BIAcore (Biacore international AB) che fornisce un sistema non invasivo e label-free basato sul principio di SPR (Surface Plasmon Resonance) una tecnica di trasduzione ottica.
Negli ultimi quindici anni è stata rivolta grande attenzione ai biosensori di affinità che sfruttano la capacità selettiva di alcune molecole di legare con determinate specie chimiche [1]. Tra questi una posizione emergente nell’ambito della ricerca è occupata dai biosensori di DNA perché consentono la diagnosi di malattie genetiche e la rivelazione di agenti patogeni batterici e virali. A differenza di enzimi o anticorpi, il DNA forma strati per il riconoscimento biologico facilmente sintetizzabili e altamente stabili inoltre i sensori di DNA non presentano problemi di rigenerazione perché l’ibridazione del DNA è reversibile con la temperatura tramite procedimenti di denaturazione [2].
I sensori di DNA sono basati sulla capacità di catene singole di DNA (ssDNA: single stranded DNA) di legarsi (ibridizzarsi) solo con catene singole
che hanno una sequenza complementare di DNA. Per la rivelazione di una determinata sequenza di DNA occorre effettuare la funzionalizzazione del dispositivo, ovvero la preparazione della superficie del sensore, tramite vari trattamenti, per consentire l’ancoraggio di catene singole di DNA recettrici (probes). I metodi di funzionalizzazione, differenti a seconda della superficie del sensore, necessitano della preventiva pulizia della superficie sensibile per la rimozione di contaminazioni. Nel caso di superfici in vetro o ossido di silicio la funzionalizzazione avviene generalmente tramite silanizzazione e prevede le fasi seguenti:
• esposizione ad una soluzione di attivazione (acidi o basi organiche) che permette di legare alla superficie gruppi idrossilici (OH);
• silanizzazione, tramite esposizione della superficie ad una soluzione contenenti silani, che permette di legare alla superficie gruppi amminici (NH2) che consentono l’immobilizzazione delle molecole probe tramite legame covalente.
È anche diffusa la procedura di funzionalizzazione che avviene tramite modifica delle molecole probes con un gruppo tiolo, in grado di legarsi con un metallo deposto sull’area sensibile del dispositivo.
Terminata la fase di funzionalizzazione, il sensore viene esposto ad una soluzione contenente l’analita ovvero sequenze di catene singole di DNA da rivelare (target). L’ibridazione avviene solo se nel DNA da analizzare è presente la sequenza complementare a quella precedentemente immobilizzata. Pertanto il meccanismo di riconoscimento tramite l’interazione probe-target è altamente specifico.
Per la rivelazione può essere necessario effettuare la marcatura (“labeling”) delle sequenze target. Questa consiste nel legare elementi reattivi (es. elementi fluorescenti) ai ssDNA. Si possono quindi distinguere sensori che presentano necessità di etichettatura (sensori “labeled”), che rende complesse e prolungate le fasi di preparazione del sensore, con conseguente aumento di
costi e tempo, da quelli più vantaggiosi che non la richiedono (sensori
“label-free”).
I dispositivi attualmente presenti sul mercato si basano su tecniche di rivelazione a fluorescenza (es. Gene chip® microarray della Affymetrix). Sono in grado di svolgere in parallelo un gran numero di test biochimici con elevata affidabilità ma presentano lo svantaggio di strumenti di lettura ottici costosi e la preventiva manipolazione del sensore per il “labeling”. Inoltre la non portabilità degli strumenti di misura rende difficoltose le misure in situ.
Per rendere la diagnostica medicale più pratica, efficiente e poco costosa la ricerca nel campo della microelettronica si sta focalizzando sullo sviluppo di dispositivi MEMS (Micro-Electro-Mechanical System) CMOS-compatibili. Tale tecnologia permette, con processi tecnologici standard, d’integrare sullo stesso substrato di silicio sia parti micromeccaniche, in grado di interagire con l’ambiente circostante, sia l’elettronica per il condizionamento del segnali. Grazie alla scalabilità delle dimensioni, è possibile integrare tali microsistemi in array. Pertanto tali dispositivi offrono i seguenti vantaggi: sono in grado di effettuare la multianalisi ovvero la rivelazione contemporanea di più specie biologiche, sono di pratico utilizzo grazie ad una rivelazione di tipo elettrico e poco costosi grazie alla compatibilità con processi tecnologici standard.
Il presente capitolo ha l’obiettivo di descrivere alcune soluzioni di biosensori di DNA proposti in letteratura che differiscono tra loro per processo di fabbricazione, meccanismo di sensing e metodo di lettura.
I sensori di DNA possono essere classificati in base al meccanismo di sensing sfruttato e cioè quale parametro fisico viene variato in seguito al fenomeno di ibridazione del DNA. Verranno pertanto passate in rassegna le seguenti categorie di biosensori:
• biosensori basati su tecniche convenzionali (DNA microarray a fluorescenza);
• biosensori ottici basati sul silicio poroso; • dispositivi a rivelazione di carica (ISFET); • biosensori di impedenza;
• biosensori gravimetrici.
Il dispositivo di cui tratterà il presente lavoro di tesi appartiene a quest’ultima categoria: è un microrisonatore meccanico (MEMS) la cui frequenza di risonanza varia in funzione della massa ad esso legata.
Nella descrizione dei vari dispositivi, aspetto degno di nota è la valutazione se questi sono immuni o meno dal problema dello “sticking”. Questo può presentarsi quando la struttura sospesa, realizzata in tecnologia MEMS, è in vicinanza (pochi μm) della parte fissa. In tal caso in presenza di liquido, infiltratosi sotto la struttura, si può verificare l’adesione permanente tra le due parti per effetto della tensione superficiale che esercita una forza attrattiva. Il dispositivo diventa a causa di ciò inutilizzabile.
Tale problema può presentarsi sia in fase di realizzazione (surface
micromachining) che di funzionalizzazione del dispositivo.
A riguardo delle prestazioni, data la diversità delle tipologie di dispositivi proposti, risulta difficile effettuarne un confronto in termini di sensibilità. Un metodo di raffronto più semplice ma altrettanto valido è la considerazione sulla necessità o meno di etichettatura delle molecole target di DNA. Tale procedura, in alcuni casi necessaria al meccanismo di rivelazione (es. rivelazione a fluorescenza), in altri utile ad aumentarne la sensibilità, prevede trattamenti aggiuntivi con conseguente spreco di costi e tempo e non consente una rivelazione in tempo reale [3].
1.1. DNA microarray
Un DNA microarray (DNA chip o gene chip) è costituito da un insieme di probe di DNA disposte a matrice attaccate ad un supporto di vetro, plastica o silicio [4]. Ogni probe è a sua volta costituita da un segmento di DNA a singola elica (ssDNA) relativa ad un gene. Tali array sono usati per la diagnosi di malattie genetiche, per la rivelazione contemporanea di molteplici agenti infettivi, analisi genetiche comparative, applicazioni in campo forense e farmaceutico. Attualmente la maggior parte dei dispositivi in commercio prevedono una rivelazione di tipo ottico essendo le molecole target marcate con un elemento fluorescente (sensori “labeled”) [5].
In letteratura è riportato il caso di un dispositivo integrato “usa e getta” per l’identificazione di agenti di bioterrorismo come l’antrace che provoca malattie infettive spesso mortali [6]. Esso è costituito da un chip basato su microarray di oligonucleotidi ad alta densità (CustomArray) commercializzato dalla Combimatrix, compatibile con la tecnologia CMOS. È costituito da un array di 12544 microelettrodi ciascuno con diametro di 44μm. In corrispondenza di ciascun microelettrodo è stata effettuata la sintesi degli oligonucleotidi di DNA tramite un metodo elettrochimico. In Fig. 1.1 è illustrata una fotografia al microscopio ottico del dispositivo della Combimatrix [7, 8] di prima generazione (CME9608I) costituito da un array di 1024 elettrodi ciascuno con diametro di 100μm.
Come evidenziato dalla Fig. 1.2, il chip è posto in contatto con una cartuccia microfluidica che automatizza i passi di manipolazione dei fluidi necessari per le fasi di labeling delle catene di DNA target con un elemento fluorescente e successiva ibridazione. L’intera operazione è molto laboriosa e delicata anche per la necessità di una preventiva amplificazione tramite PCR (Polymerase Chain Reaction) del DNA. Questa consiste in una serie di reazioni a catena che permettono di amplificare enormemente uno specifico frammento
Fig. 1.1 Immagine del DNA microarray della Combimatrix di prima generazione (CME9608I) [7,8].
Fig. 1.2 (A) Schema del dispositivo composto da una cartuccia microfluidica e dal DNA chip della Combimatrix (CustomArray slide). Nella cartuccia sono presenti cinque micropompe elettrochimiche (riga in alto), sei valvole di controllo “a becco d’anatra”, cinque camere di immagazzinamento (storage) per diverse soluzioni e reagenti, una camera per microarray e una camera di scarico (waste chamber). Ogni camera di immagazzinamento possiede due aperture: una per l’ingresso della soluzione e l’altra per la fuoriuscita d’aria durante le fase di caricamento. (B) Fotografia del dispositivo incapsulato alla cartuccia [6].
di DNA grazie all’enzima DNA polimerasi. La lettura ottica, dopo la rimozione del chip dalla cartuccia, avviene tramite uno scanner a fluorescenza. Il dispositivo ha permesso con successo l’identificazione dell’agente patogeno (antrace). I risultati ottenuti tramite il software della Combimatrix (Microarray Imager Software) sono infatti uguali a quelli derivanti dal trattamento manuale del chip.
1.2. Cantilever/membrane a flessione statica come
sensori di DNA
Le tecniche di microlavorazione del silicio sono largamente utilizzate per la realizzazione di dispositivi MEMS come cantilever (travi a mensola) e membrane. Tali dispositivi sfruttano lo stress indotto sulle strutture in seguito al fenomeno di ibridazione del DNA come meccanismo di sensing. Come illustrato nello schema seguente (Fig. 1.3), l’interazione chimica tra molecole probe e target si traduce nel piegamento della struttura [9].
Fig. 1.3 Principio di funzionamento del cantilever (flessione statica) [9].
Per quanto riguarda le tecniche di lettura la più comune è quella ottica ma un metodo sicuramente preferibile è quello piezoresistivo poiché fornisce un segnale di uscita elettrico. Il metodo capacitivo richiede l’integrazione della
struttura sospesa a distanza non superiore a qualche micron dall’elettrodo di riferimento posizionato sulla parte fissa. Questa distanza ridotta tra gli elettrodi può dar luogo a problemi di “sticking” in fase di ibridazione del DNA.
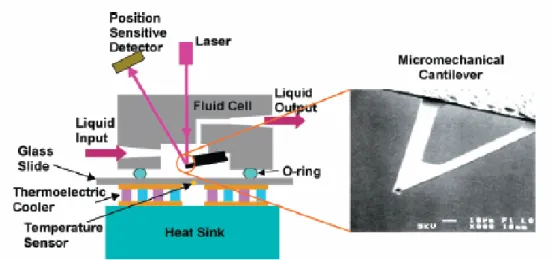
In un dispositivo di questo tipo è stata effettuata con successo la funzionalizzazione label-free di cantilever di un AFM (Atomic Force Microscopy) per rivelare la presenza di danni (mismatches) in un singolo nucleotide di DNA [10].
La rivelazione del polimorfismo di un singolo nucleotide (SNP: single Nucleotide Polymorphism) è un passo chiave nella ricerca per la diagnosi di alcune malattie (es. fibrosi cistica).
Per la funzionalizzazione la superficie dei cantilever è stata ricoperta tramite evaporazione in vuoto con uno strato di oro (20nm) che consente l’immobilizzazione dei ssDNA, una volta modificati con tiolo. La deflessione statica del cantilever in seguito al fenomeno di ibridazione è stata rivelata con un sistema ottico costituito da un laser puntato sulla parte superiore dell’estremo libero del cantilever e un fotodiodo che, raccogliendo il raggio riflesso, produce un segnale di uscita dipendente dalla posizione (1V= 100nm di deflessione) (Fig. 1.4).
Fig. 1.4 Rappresentazione schematica del set-up di misura per il cantilever del AFM (a sinistra). Immagine al SEM della geometria del cantilever (a destra) [10].
I risultati sperimentali, riportati nel grafico di Fig. 1.5, illustrano che elemento discriminante per la presenza di danni interni (internal mismatches) nelle sequenze target di DNA è la deflessione verso il basso dei cantilever che altrimenti si piegherebbero verso l’alto. L’ampiezza della deflessione risulta proporzionale al numero dei danni interni (1 o 2) ma il meccanismo di ciò è ancora poco chiaro.
Fig. 1.5 Andamento della deflessione del cantilever in funzione del tempo per sequenze target complementari, danneggiate e non complementari [10].
In [11] è stato proposto un biochip basato su microcantilever realizzati a partire da un processo CMOS con un meccanismo di sensing piezoresistivo che consente una lettura elettronica. I piezoresistori sono integrati sui cantilever. Sono realizzati sfuttando la variazione di resistenza del posilicio di gate di tale processo in seguito allo stress meccanico indotto dal fenomeno di ibridazione del DNA. Per massimizzare l’efficienza lo strato piezoresistivo è diviso in 4 zone (Fig. 1.6 b) a diversa concentrazione di drogante.
Come si nota dal disegno (Fig. 1.6 c) in parallelo ad ogni cantilever per il biosensing ne è integrato uno di riferimento per la cancellazione del rumore termico dovuto ai piezoresistori.
Ogni struttura è rilasciata tramite bulk micromachining con un attacco anisotropo in plasma ICP-RIE (Inductively Couplet Plasma Reactive Ion Etching).
Fig. 1.6 (a) Struttura del cantilver (con meccanismo di sensing piezoresistivo) a quattro strati. (b) Vista dall’alto del cantilever in cui si notano le quattro zone a diverso drogaggio: le zone1-3 ad elevato drogaggio per le connessioni elettriche e contatto ohmico, la zona 4 a basso drogaggio per un maggiore effetto piezoresistivo. (c) Schema della disposizione dei cantilever nel dispositivo. Sono presenti 4 regioni di sensing, ognuna delle quali dispone di tre coppie di microleve in parallelo e due regioni per le misure di riferimento. Immagine modificata presente in [11].
È stato deposto uno strato di cromo e oro per evaporazione, la cui geometria è stata definita con la tecnica di litografia lift-off, per consentire l’immobilizzazione delle singole eliche di DNA (ssDNA) modificate con gruppi tiolo. I risultati sperimentali hanno mostrato che il meccanismo di immobilizzazione è compatibile con il modello di Langmuir [12] (Fig. 1.7). La tensione misurata dopo immobilizzazione delle molecole probe può essere espressa tramite il modello di adsorbimento di Langmuir (Fig1.7) semplificato che si basa sulle seguenti ipotesi:
• l’adsorbimento riguarda il ricoprimento di un monostrato; • i siti di adsorbimento sono tutti equivalenti tra di loro.
) 1 ( kt e A Voff Vout = + − − (1)
in cui Voff indica la tensione di offset, K la costante di velocità di
adsorbimento/desorbimento, A una costante.
Fig. 1.7 Tensione misurata in funzione del tempo dopo immobilizzazione sulla superficie del cantilever (a tratto spesso) e curva che rappresenta il modello di Langmuir (a tratto sottile) [11].
Dopo aver esposto la superficie dei cantilever ad una soluzione contenente le molecole target di DNA, è stata rivelata in modalità label-free una diminuzione dell’ampiezza della tensione misurata con andamento analogo a quello di Fig. 1.7 a conferma della formazione del legame.
Com’è noto i parametri meccanici dei cantilever hanno un ruolo fondamentale nella determinazione della sensibilità. Per aumentarla è possibile ricoprirne la superficie con materiali colloidali o polimeri che presentano un valore del modulo di Young inferiore rispetto a quello dei materiali con cui vengono generalmente realizzati come silicio o nitruro di silicio [13].
A titolo di esempio si riporta un array di cantilever basati sul polimero SU-8 un fotoresist epossidico con elevata resistenza chimica [14].
Fig. 1.8 Principali passi di processo per la realizzazione del dispositivo ovvero del cantilever a flessione statica basato sul polimero SU-8. Immagine modificata presente in [14].
Il dispositivo è stato realizzato a partire da un substrato di silicio su cui è stato deposto un layer metallico sacrificale (cromo-oro-cromo). Dopo la deposizione per spinning di uno strato di resist SU-8, successivamente litografato per la definizione della geometria dei cantilever, è stato realizzato lo strato di immobilizzazione delle molecole di DNA tramite la litografia lift-off di un film metallico costituito da oro e titanio. Infine è stato deposto per spinning uno strato di SU-8 di 400 μm per la definizione del supporto della struttura che è stata rilasciata con surface micromachining tramite attacco wet dello strato metallico (Au/Cr/Au) deposto inizialmente. In Fig. 1.8 è illustrato quanto finora descritto.
Il vantaggio di tale dispositivo è che il rilascio della struttura avviene tramite una procedura di surface micromachining che consente di evitare il problema dello “sticking”.
Il sistema di lettura usato è stato quello ottico tradizionale usato nel AFM precedentemente descritto, in questo caso però l’illuminazione avviene sul retro del cantilever ovvero dove è presente lo strato di oro.
A causa di problemi di stabilità su tale dispositivo sono state finora effettuate solo prove di immobilizzazione di ssDNA che hanno permesso di riscontrare una maggiore sensibilità di tali cantilever rispetto a quelli basati su nitruro di silicio.
Recentemente è stato realizzato un dispositivo [15] che presenta un metodo di lettura vantaggioso in quanto misura semplicemente la variazione della corrente di drain in un MOS. Tramite le tecniche di microlavorazione del silicio, è stato realizzato un array di cantilever che presentano ognuno, in corrispondenza del punto di ancoraggio ovvero quello sottoposto a maggior stress, un MOSFET.
La deflessione dei cantilever in seguito all’interazione probe-target, induce stress nel canale del MOS con conseguente variazione della mobilità dei portatori che si traduce in una variazione della corrente di drain che può essere facilmente misurata. Nella figura seguente (Fig. 1.9), che schematizza quanto descritto, sono presenti due cantilever: uno in nitruro di silicio usato come riferimento e l’altro ricoperto da uno strato d’oro come dispositivo di sensing.
Fig. 1.9 Rappresentazione schematica del meccanismo di sensing dell’array di cantilever ciascuno dei quali presenta un MOSFET nel punto di ancoraggio [15].
A partire da un wafer di tipo SOI (silicon-on-insulator) che presenta uno strato sepolto di ossido (2,5μm) e uno strato epitassiale di silicio (1,5 μm), sono stati realizzati i MOSFET di tipo n in tecnologia CMOS standard. In seguito è stata definita la geometria dei microcantilever che sono stati poi rilasciati con l’attacco dello strato di ossido.
Gli esperimenti di funzionalizzazione label-free hanno dato esiti positivi. In Fig. 1.10 sono illustrati i risultati nel caso di interazione biotina-streptavidina. Per i cantilever di sensing (ricoperti da uno strato di oro) la deflessione in seguito al legame ha portato ad un decremento della corrente di
drain misurata, mentre nei cantilever di riferimento (nitruro) la corrente è
rimasta invariata.
Gli esperimenti sono stati condotti per diverse concentrazioni delle molecole target.
Fig. 1.10 Andamenti della corrente di drain di un MOS, integrato in corrispondenza dell’ancoraggio di un cantilener, in funzione della tensione [15].
Al momento non sono ancora stati eseguiti esperimenti su tale dispositivo con DNA.
Come accennato all’inizio del paragrafo una rivelazione di tipo capacitivo si può ottenere tramite membrane. Con le tecniche di microlavorazione si possono realizzare delle membrane sospese ciascuna delle quali forma con il substrato le due facce di un condensatore la cui capacità varia in seguito alla deformazione della membrana per effetto delle interazioni molecolari alla sua superficie.
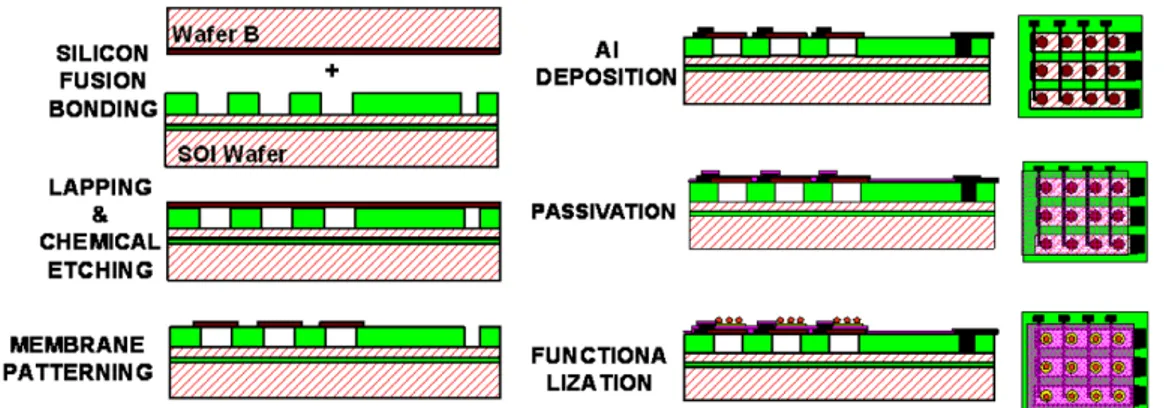
Recentemente è stato realizzato un dispositivo basato su una matrice di membrane capacitive finalizzato all’analisi di DNA [16].
Nella figura seguente (Fig. 1.11) sono descritti i principali step di fabbricazione di tale dispositivo.
Fig. 1.11 Principali passi del processo di fabbricazione dell’array di sensori a rivelazione capacitiva. [16].
Ciascuna membrana è stata realizzata con un processo dedicato che ha previsto la fusione (bonding) tra un wafer SOI che costituisce il substrato, e un wafer di silicio. Il wafer SOI è stato drogato con fosforo per la realizzazione degli elettrodi fissi del condensatore, mentre quello di silicio con uno strato epitassiale di SiGeB per la realizzazione della membrana dopo la litografia. Dopo la fusione, il wafer di silicio (wafer B) è stato lappato e attaccato completamente tramite wet etching fino a scoprire lo strato di SiGeB. In seguito è stata definita la geometria di ciascuna membrana tramite litografia. Infine è stata effettuata la deposizione e definizione delle metal di alluminio per i contatti.
Nota positiva di tali strutture è che sono immuni dal problema dello “sticking” in quanto le membrane insistono su una cavità completamente isolata dall’ambiente esterno, per cui non vi è la possibilità che, in presenza di umidità, le particelle d’acqua possano rimanere intrappolate negli spazi tra le membrane e il substrato.
Finora il dispositivo è stato testato solo come sensore chimico per cui nulla può ancora dirsi a riguardo della sua validità come sensore di DNA.
1.3. Biosensori basati su silicio poroso
Il silicio poroso ha suscitato grande interesse come potenziale piattaforma per le applicazioni biosensoristiche grazie al suo elevato rapporto superficie-volume, che consente una interazione efficace con gli analiti di interesse. È realizzato tramite etching anodico del silicio in soluzione di acido fluoridrico. Tale processo di anodizzazione consente di formare pori le cui caratteristiche sono controllate mediante i parametri di processo. Variando la densità di corrente [17] e il tempo di erogazione della stessa si possono ottenere strati a differente porosità e quindi con diverse proprietà ottiche a partite dalla superficie di silicio in contatto con la soluzione elettrochimica. Il meccanismo di sensing si basa sulla variazione dell’indice di rifrazione del materiale in seguito al riempimento dei pori con le molecole target che può esse rivelata per mezzo di tecniche interferometriche (film di Fabry-Perot) o risonanti (microcavità).
Di recente è stato realizzato un dispositivo costituito da una guida d’onda risonante basata su silicio poroso per la rivelazione label-free di DNA [18].
A partire da un substrato di silicio p+ sono stati realizzati, tramite attacco elettrochimico, due strati con pori di diversa densità: uno a bassa porosità (56%) sovrapposto ad uno a porosità più alta (84%) con spessore
rispettivamente di 310 nm e 1330 nm (Fig. 1.12). La porosità è definita come il rapporto tra il volume occupato dai pori (vuoto) e il volume totale del materiale (pieno).
L’accoppiamento del fascio laser all’interno della guida è stato ottenuto mediante un prisma. Se l’angolo di riflessione nel prisma supera l’angolo critico, si ha il fenomeno della riflessione interna totale che produce un campo nel gap di aria tra prisma e guida che permette l’innesco del modo fondamentale (TE0) in quest’ultima.
Fig. 1.12 Schema del dispositivo costituito da una guida d’onda risonante basata su silicio poroso [18].
Fig. 1.13 (a) Grafico della riflettanza in funzione dell’angolo di risonanza. Si nota la variazione di esso dopo ibridazione. (b) Grafico della riflettanza in funzione dell’angolo di risonanza. L’angolo di risonanza si mostra invariato dopo esposizione a sequenze non complementari di DNA [18].
La variazione dell’indice di rifrazione della guida, in seguito all’assorbimento di biomolecole da parte dei pori, da luogo alla variazione dell’angolo di innesco (angolo di risonanza in guida) e quindi della percentuale di luce riflessa uscente dal prisma (riflettanza) che è stata misurata tramite un fotodiodo posto all’uscita di esso.
In Fig. 1.13 sono mostrati i risultati sperimentali che evidenziano una variazione dell’angolo di risonanza solo in presenza di ibridazione del DNA.
Altra tecnica utilizzata per la rivelazione della variazione dell’indice di rifrazione è l’interferometria in luce bianca. La luce riflessa alla base e alla sommità di uno strato di silicio poroso produce uno spettro di interferenza. Nel dispositivo riportato in [19] in seguito all’interazione probe-target, dovuto al fenomeno di ibridazione del DNA, si induce la variazione dell’indice di rifrazione dello strato di silicio poroso e quindi della lunghezza d’onda dello spettro di interferenza. Questa è stata misurata tramite un CCD (Charge-Couplet-Device) (Fig. 1.14).
Fig. 1.14 Rappresentazione schematica del dispositivo basato su silicio poroso con tecnica di rivelazione a interferometria in luce bianca [19].
Sono stati realizzati pori con un diametro di 200 nm tale da fornire una elevata superficie per l’interazione molecolare.
I risultati sperimentali hanno evidenziato la validità del dispositivo nel rivelare la presenza di diverse specie biochimiche come proteine (streptavidina e anticorpi), piccole molecole organiche (biotina e digoxigenina) e sequenze di DNA senza la necessità di marcatori (label-free). Dalle figure seguenti si nota che solo in caso di esposizione con sequenze di DNA complementare (in una soluzione con 2•10-12 M di DNA) (Fig. 1.15 A) è stato misurato uno spostamento verso lunghezze d’onda minori dello spettro di interferenza. Nel caso di esposizione con sequenze non complementari non si osserva alcuno shift per soluzioni con concentrazioni inferiori a 10-9 M.
Fig. 1.15 Grafici dello spettro interferometrico di riflessione in funzione della lunghezza d’onda: (A) esposizione in presenza di DNA complementare (concentrazione di 2*10-12 M in soluzione), (B) esposizione con una soluzione contenente sequenze di DNA non complementari. Immagine modificata presente in [19].
Altro esempio da menzionare è quello di un biosensore di DNA basato su microcavità risonanti in silicio poroso [20]. È stato possibile restringere la banda di emissione di uno strato di silicio poroso (strato attivo) inserendolo tra due riflettori di Bragg distribuiti a loro volta realizzati alternando periodicamente strati di silicio a diversa porosità.
La struttura è stata realizzata tramite attacco elettrochimico a partire da un substrato di silicio ossidato termicamente.
Le immagini al SEM illustrano diversi aspetti della struttura (Fig. 1.16). I pori dello strato superficiale di silicio hanno un diametro di 20 nm abbastanza grandi da consentire l’immobilizzazione dei nucleotidi di DNA che hanno un diametro di circa 2 nm.
Il parametro di sensing è sempre l’indice di rifrazione la cui variazione, in questo caso, è stata rivelata misurando lo shift dei picchi dello spettro di luminescenza, dopo l’ibridazione, tramite spettroscopia infrarossa in trasformata di Fourier (FTIR). I risultati mostrati in Fig. 1.17 hanno evidenziato uno spostamento verso il rosso (7 nm) dello spettro dopo immobilizzazione delle molecole target di DNA in modalità label-free.
Fig. 1.16 Fotografie al SEM della struttura costituita da microcavità risonanti in silicio poroso. (A) sezione degli specchi multipli: in chiaro strato a bassa porosità (43%), in scuro strato ad alta porosità (62%). (B) visione dall’alto dello strato superficiale con pori di circa 20 nm di diametro. (C) sezione dello strato attivo con 68% di porosità e 60-75 nm di diametro [20].
Fig. 1.17 Andamento dello spettro di luminescenza in funzione della lunghezza d’onda: (a) in presenza di DNA complementare; (b) in presenza di DNA non complementare. Si nota che in questo caso il segnale rimane invariato [20].
1.4. Biosensori a rivelazione di carica
A questa categoria appartengono i dispositivi basati su transistori ad effetto di campo detti ISFET (Ione Sensitive Field Effect Transistor). Si tratta di dispositivi privati dell’elettrodo di gate nei quali l’isolante (ossido di silicio o nitruro) è direttamente esposto alla soluzione elettrochimica in cui viene immerso un elettrodo di riferimento [21].
In seguito all’interazione probe-target delle molecole di DNA si assiste ad una variazione della carica superficiale che ne induce una di segno opposto nella regione di carica spaziale del semiconduttore. Si ottiene in tal modo la variazione della popolazione di inversione nel canale e quindi della corrente di
drain che può essere misurata.
Questo meccanismo di trasduzione consente una rivelazione elettrica
A titolo di esempio si può citare un lavoro di ricerca [22] che propone come biosensore un ISFET basato su silicio amorfo idrogenato (a-Si:H), semiconduttore ampiamente usato per la realizzazione di celle solari, transistori a film sottile (TFT) e display piatti.
La realizzazione del dispositivo prevede un processo dedicato a partire da un substrato di vetro. Come illustrato (Fig. 1.18) i contatti di source e drain sono stati ottenuti tramite sputtering di uno doppio strato di alluminio e di titanio-tungsteno. La definizione della geometria è avvenuta tramite wet
etching per l’alluminio e RIE (Reactive Ion Etching) per lo strato di Ti/W. In
seguito sono stati deposti tramite PECVD (Plasma Enhanced Chemical Vapour Deposition) uno strato di silicio amorfo idrogenato di tipo n+ a 250°C e uno di SiNx a 350°C. Dopo definizione di essi è stato aggiunto uno strato ulteriore di passivazione di SiNx per isolare elettricamente il dispositivo dalla soluzione elettrochimica ed infine è stato definito tramite lift-off un layer di 75nm di SiO2 che costituisce l’elemento sensibile al riconoscimento biologico. L’elettrodo di Ag/AgCl immerso nella soluzione funge da gate.
Fig. 1.18 (a) Sezione del dispositivo ISFET basato su silicio amorfo idrogenato (a-Si:H). (b) Package del chip [22].
I risultati sperimentali (Fig. 1.19) mostrano le variazioni della curva di trasferimento Ids-Vg misurate dopo ogni fase di trattamento della superficie del
dispositivo: pulizia, funzionalizzazione tramite silanizzazione, pre-ibridazione con BSA (Bovine Serum Albumin) per evitare adsorbimenti non specifici ed ibridazione.
Fig. 1.19 Andamento della curva di trasferimento dopo ogni trattamento. L’inserto mostra un allargamento della curva [22].
Comunque, affinché i risultati ottenuti siano maggiormente affidabili è necessaria una miglior comprensione a riguardo del comportamento del dispositivo.
1.5. Biosensori impedenzimetrici
Si tratta di dispositivi in cui il meccanismo di sensing si basa sulla variazione di impedenza (resistenza e/o capacità) che può essere misurata elettricamente.
Tra questi si può menzionare un dispositivo [23] che non consente una rivelazione label-free poiché necessita della marcatura delle sequenze probe di DNA, dopo modifica con un gruppo tiolo (SH-), con nanoparticelle d’oro (AuNPs). Esso è costituito da un array di coppie di microelettrodi
convenzionali. Nel gap di separazione di ciascuna coppia, in seguito all’interazione probe-target, si viene a creare uno strato di nanoparticelle d’oro che induce una variazione della conduttività che è stata misurata come variazione di resistenza tramite un multimetro.
Il processo di fabbricazione ha previsto la definizione dei microelettrodi (60nm di Au su 5nm di Ti) con 20μm di gap con tecniche fotolitografiche standard a partire da un substrato di silicio su cui è stato cresciuto 1μm di ossido.
Per migliorare la sensibilità di risposta è stato necessario ricoprire la superficie con uno strato di argento, ottenuto tramite ripetute esposizioni ad una soluzione di AgNO3 e idrochinone.
Uno studio più recente [24] ha dato risultati soddisfacenti senza la necessità di questo passo aggiuntivo.
Dalla figura sottostante (Fig. 1.20), che riporta le principali sequenze di fabbricazione, si nota che la definizione del gap tra gli elettrodi è stata realizzata tramite un processo di lift-off.
Fig. 1.20 Principali passi di fabbricazione dei microelettrodi. Immagine modificata presente in [24].
Dopo diversi trattamenti di pulizia, il substrato è stato immerso in una soluzione di silani per la funzionalizzazione dell’ossido in corrispondenza del gap e trattato con una soluzione di nanoparticelle d’oro per la formazione del monostrato SAM (Self Assembled Monolayer). Le fasi successive sono illustrate in Fig. 1.21 e, dopo ibridazione delle molecole target, portano alla formazione di un multistrato di nanoparticelle.
Fig. 1.21 Passi di processo per l’immobilizzazione delle particelle d’oro e ibridazione del DNA [24].
I risultati sperimentali in Fig. 1.22 mostrano che solo in presenza di un multistrato, e quindi di molecole target, è stato misurato il passaggio di corrente.
Fig. 1.22 A sinistra le immagini al FE-SEM del gap tra i microelettrodi. Sulla destra i grafici corrispondenti della corrente in funzione della tensione applicata agli elettrodi. (a) Monostrato di AuNp. (b) Presenza di multistrato per l’ibridazione [24].
In letteratura sono riportati anche biosensori di impedenza che consentono una rivelazione label-free.
È stato realizzato un dispositivo basato su nano-eletttrodi interdigitati in titanio che è stato testato come immunosensore [25].
Il processo di fabbricazione è analogo a quello usato in un precedente lavoro in cui gli elettrodi sono stati realizzati in palladio (Fig. 1.23) [26].
A partire da un substrato di silicio è stato cresciuto termicamente uno strato di 1,2μm di ossido sul quale sono stati definiti gli elettrodi tramite litografia lift-off di Ti/Pd/Ti.
Lo schema in Fig. 1.24 illustra il principio di funzionamento del sensore che si basa sulla variazione delle linee del campo elettrico e quindi dell’impedenza misurata tra gli elettrodi in seguito alla formazione del legame.
Fig. 1.24 Schema del dispositivo basato su nano-elettrodi interdigitati : (a) prima della formazione del legame; (b) dopo la formazione del legame [25].
È stato utilizzato un sistema di misura ottico per rivelare l’avvenuto legame anticorpo-antigene di una proteina (PLAP: Placental Alcaline Phosphatase).
Il grafico seguente (Fig. 1.25) mostra la variazione dello spettro di assorbanza in seguito alla reazione enzimatica misurata tramite uno spettrometro UV-visibile.
Fig. 1.25 Grafici dell’assorbanza in funzione della lunghezza d’onda prima e dopo la formazione del legame antigene-anticorpo [25].
La maggior parte dei biosensori inpedenzimetrici riportati in letteratura sono realizzati su substrati di silicio o vetro mentre, solo recentemente sono stati presi in considerazione materiali polimerici che trovano ampia applicazione per la realizzazione di dispositivi lab-on-a-chip monouso.
A titolo di esempio si riporta la descrizione di un dispositivo [27] basato su un array di nano-elettrodi interdigitati (nIDA: nano interdigitated electrodes array) associato ad un sistema microfluidico (Fig. 1.26).
A riguardo della tecnologia di fabbricazione la Fig. 1.27 ne illustra i passi principali.
A partire da un substrato polimerico di COC (cyclic olefin copolymer) si è provveduto inizialmente alla deposizione e definizione di un film di cromo per rendere il substrato compatibile con i processi tecnologici. In seguito sono state
Fig. 1.26 Rappresentazione schematica del dispositivo basato su nano-elettrodi interdigitati associato ad un sistema microfluidico [27].
Fig. 1.27 Processo di fabbricazione dei nano-elettrodi interdigitati e del chip microfluidico [27].
effettuate in sequenza due litografie lift-off per la realizzazione degli nIDA e dei rispettivi pad. Successivamente esso è stato unito tramite bonding termico al chip microfluidico realizzato tramite una tecnica di stampo (plastic injection
molbing) del polimero su un disco di nickel.
Gli esperimenti di funzionalizzzazione sono stati condotti per la rivelazione label-free della presenza del legame antigene-anticorpo di immunoglobuline di topo anti-coniglio tramite misure di variazione di impedenza con EIS (Electrochemical Impedimetric Spectroscopy). Come si osserva dal grafico (Fig. 1.28) è stata misurata una variazione dello spettro solo per basse frequenze (20Hz-1KHz) in accordo con il modello usato.
Fig. 1.28 Andamento del modulo dell’impedenza in funzione della frequenza dopo la formazione del legame [27].
1.6. Dispositivi gravimetrici
Consistono in risonatori meccanici la cui frequenza di risonanza varia in funzione della variazione di massa conseguente al legame delle molecole da analizzare sulla loro superficie e pertanto sono anche detti microbilance.
Vale la pena menzionare i QCM (Quartz Crystal Microbalance) sottili dischi di diossido di silicio cristallino che, per il ben noto effetto piezoelettrico, sono in grado di entrare in risonanza meccanica se sottoposti ad un segnale elettrico ad una certa frequenza. Sono largamente utilizzati, grazie alle loro ottime prestazioni per l’analisi del DNA sebbene presentano gli svantaggi legati alla loro forma macroscopica [28].
A titolo di esempio si può menzionare un caso presente in letteratura in cui è stato utilizzato un quarzo AT-cut con fc=8 MHz per la rivelazione del batterio Escherichia coli O157:H7 responsabile della contaminazione dei cibi [29].
Di seguito (Fig. 1.29) sono mostrate le fasi di preparazione del dispositivo che prevedono: formazione del SAM (Self Assembled Monolayer) di tiolo, immobilizzazione delle nanoparticelle d’oro, immobilizzazione delle sequenze probe modificate con tiolo, ibridazione e amplificazione tramite PCR.
Fig. 1.29 Funzionalizzazione del QCM e procedura di rilevazione [29].
I risultati sperimentali mostramo uno shift di frequenza di circa 30Hz, apprezzabile in seguito al fenomeno di ibridazione.
I dispositivi gravimetrici su cui si sta focalizzando l’attenzione scientifica sono quelli realizzati con le tecnologie di microlavorazione del silicio poiché si tende alla realizzazione di oggetti integrati scalabili in dimensioni per consentire la multianalisi e rivelazione in tempo reale tramite un segnale di tipo elettrico. Tra questi una categoria particolarmente vantaggiosa è rappresentata da quelli compatibili con la tecnologia CMOS visto che richiedono pochi passi ulteriori di post-processing per la loro realizzazione.
Ad esempio i cantilever, già precedentemente menzionati nel funzionamento statico, si prestano anche ad un meccanismo di sensing dinamico.
In un lavoro di ricerca è stato realizzato un cantilever MEMS come microrisonatore meccanico per rilevare la presenza di un marker tumorale CEA (antigene carcino-embrionario) [30].
La realizzazione del dispositivo ha previsto la deposizione tramite sol-gel di un film piezoelettrico PZT (Titanato-Zirconato di piombo) usato per i meccanismi di attuazione e rivelazione.
Come illustrato nei principali step di processo in Fig. 1.30, prima e dopo la deposizione del PZT sono stati deposti due strati di Pt/Ti e di Pt rispettivamente a costituire gli elettrodi metallici. Il rilascio della struttura è avvenuto tramite bulk micromachining con attacco D-RIE (Deep-Reactive Ion Etching) del silicio.
Il dispositivo prevede un metodo di rivelazione ottico. La lettura della variazione della frequenza fondamentale di risonanza è stata effettuata tramite un vibrometro al laser doppler e lo schema di misura di Fig. 1.31.
I dati sperimentali hanno mostrato che, seppur presenti effetti trascurabili di legami non-specifici, il dispositivo è in grado di rivelare senza necessità di marcatori concentrazioni di 30pM, valore minimo per diagnosticare la presenza di un tumore, tramite uno scostamento di frequenza di 6.5KHz. Presenta però due svantaggi: processo di fabbricazione dedicato non compatibile con la
tecnologia CMOS che prevede inoltre l’integrazione di materiali inusuali (PZT) e strumento di lettura ingombrante e costoso.
Fig. 1.30 Procedura di fabbricazione del dispositivo costituito da un cantilever risonante. (sensing dinamico). In (e) si nota la deposizione e definizione di uno strato di materiale fotoresistivo (PR) come supporto per la realizzazione di un gap di aria realizzato per prevenire correnti di perdita tra i pad e l’elettrodo inferiore [30].
Fig. 1.31 Schema del sistema di misura ottico per la rivelazione della variazione della frequenza di risonanza del cantilever [30].
Un sensing di tipo gravimetrico può essere realizzato anche tramite membrane microlavorate che presentano uno strato di materiale piezoelettrico come sistema di rivelazione. Come esempio è riportato un dispositivo in cui la
membrana è stata liberata tramite tecniche standard di bulk micromachining [31]. Essa è costituita da uno strato di PZT racchiuso tra due elettrodi di Pt/Ti la cui geometria è stata definita in modo da ottenere sette sensori posizionati in forma esagonale. In corrispondenza di ciascuno di essi è stata definita sul retro della fetta una camera di reazione tramite uno strato di ossido in seguito coperto da un film di oro per la funzionalizzazione di ciascun sensore in modo da consentire la rivelazione contemporanea di più elementi biologici. Le immagini seguenti (Fig. 1.32) riportano quanto descritto.
Fig. 1.32 Struttura del dispositivo basato su membrane risonanti: (a) immagine al microscopio ottico della parte frontale; (b) immagine al SEM del retro. Immagine modificata presente in [31].
Il dispositivo è stato testato come immunosensore label-free per la rivelazione delle immunoglobuline di capra. In seguito alla formazione del legame anticorpo-antigene è stata misurata una diminuzione della frequenza di risonanza di circa 700Hz tramite un analizzatore di impedenza. Presenta però lo svantaggio di richiedere un processo dedicato di fabbricazione che ne aumenta i costi.
Fig. 1.33 Diagramma di fase dell’impedenza. Si nota come la frequenza di risonanza varia in seguito ad ogni passo di immobilizzazione [31].
Recentemente un gruppo di ricerca ha realizzato un sensore innovativo per la diagnosi di allergie tramite la rivelazione di immunoglobuline IgE [32].
A partire da un substrato di silicio è stata definita la geometria di uno strato di ZnO, materiale piezoelettrico deposto per RF-magnetron sputtering, racchiuso tra due strati metallici di Au/Cr litografati superiormente per fornire due trasduttori interdigitati di sensing e di drive rispettivamente (Fig. 1.34). Il rilascio della struttura (Si/SiO2/Si3N4/Cr/Au/ZnO) è avvenuto tramite bulk
micromachining. La funzionalizzazione, abbastanza complessa per i numerosi
passi di immobilizzazione, è stata effettuata sul retro del dispositivo (Fig. 1.35) dopo deposizione di uno strato di oro preceduto da un film di cromo per migliorarne l’adesione.
I risultati sperimentali, ottenuti tramite misure elettriche, hanno mostrato una variazione della frequenza di risonanza in seguito alla formazione del legame anticorpo-antigene di circa 9294Hz.
Fig. 1.34 Principali passi del processo di fabbricazione del dispositivo costituito da una membrana risonante. Immagine modificata in [32].
Fig. 1.35 (a) Configurazione finale del dispositivo basato su una membrana risonante. (b) Schema degli strati di immobilizzazione in successione: SAM di cistamina, glutaraldeide, anticorpi di IgE e antigeni. Immagine modificata presente in [32].
Come già accennato, il dispositivo oggetto di questo lavoro è un risonatore meccanico di tipo MEMS attuato magneticamente con meccanismo di sensing gravimetrico.
Al momento si intende fornire una descrizione a grandi linee riguardo la tecnologia e il funzionamento delle microbilance realizzate che verranno in seguito descritte approfonditamente.
Aspetto particolarmente vantaggioso è che le strutture sono ricavate a partire da un processo CMOS compatibile con pochi passi di post-processing. Questi hanno previsto la rimozione degli ossidi intermetallici ove non necessari e il rilascio della struttura tramite attacco del substrato di silicio con tecniche di
bulk micromachining.
Come si può notare nella figura seguente (Fig. 1.36) sono costituite da un piatto centrale di forma quadrata (o rettangolare) sospesa tramite due bracci ancorati al substrato che si comportano come molle torsionali. Tutta la struttura sospesa è costituita dagli strati dielettrici inter-metallici presenti nel processo e dalle metal di alluminio la cui geometria viene definita in modo da realizzare degli avvolgimenti di drive e di sensing per i meccanismi di attuazione e rivelazione magnetica rispettivamente.
Fig. 1.36 Struttura schematica del sensore costituito da una microbilancia risonante [33].
Il dispositivo si trova immerso in un campo di induzione magnetica esterno, generato tramite uno o più magneti permanenti, di intensità costante e direzione appartenente al piano della struttura. Facendo scorrere nella spira di drive (driving loop) una corrente sinusoidale con frequenza pari a quella di
risonanza meccanica della struttura, su di essa, essendo immersa nel campo magnetico, agisce la forza di Lorentz responsabile del momento torcente che provoca le oscillazioni della struttura. A causa del movimento viene indotta, secondo la legge di Faraday, una f.e.m. negli avvolgimenti di sensing (sensing loop) per effetto della variazione del flusso magnetico ad essi concatenato.
Pertanto altro aspetto vantaggioso di tale struttura risiede nel fatto che il segnale di uscita fornito è di tipo elettrico e quindi direttamente misurabile con un sistema elettronico. L’appesantimento della struttura, in seguito all’adsorbimento del DNA di cui si vuole rivelare la presenza, provoca la variazione del momento di inerzia e quindi della frequenza di risonanza del sensore che può essere agevolmente rivelata tramite la misura della tensione indotta ai capi delle spire di sensing.
![Fig. 1.1 Immagine del DNA microarray della Combimatrix di prima generazione (CME9608I) [7,8]](https://thumb-eu.123doks.com/thumbv2/123dokorg/7340751.91912/7.892.309.608.198.427/fig-immagine-dna-microarray-combimatrix-prima-generazione-cme.webp)
![Fig. 1.5 Andamento della deflessione del cantilever in funzione del tempo per sequenze target complementari, danneggiate e non complementari [10]](https://thumb-eu.123doks.com/thumbv2/123dokorg/7340751.91912/10.892.274.637.383.706/andamento-deflessione-cantilever-funzione-sequenze-complementari-danneggiate-complementari.webp)
![Fig. 1.7 Tensione misurata in funzione del tempo dopo immobilizzazione sulla superficie del cantilever (a tratto spesso) e curva che rappresenta il modello di Langmuir (a tratto sottile) [11]](https://thumb-eu.123doks.com/thumbv2/123dokorg/7340751.91912/12.892.293.616.370.629/tensione-misurata-funzione-immobilizzazione-superficie-cantilever-rappresenta-langmuir.webp)

![Fig. 1.9 Rappresentazione schematica del meccanismo di sensing dell’array di cantilever ciascuno dei quali presenta un MOSFET nel punto di ancoraggio [15]](https://thumb-eu.123doks.com/thumbv2/123dokorg/7340751.91912/14.892.194.722.697.983/rappresentazione-schematica-meccanismo-sensing-cantilever-ciascuno-presenta-ancoraggio.webp)
![Fig. 1.10 Andamenti della corrente di drain di un MOS, integrato in corrispondenza dell’ancoraggio di un cantilener, in funzione della tensione [15]](https://thumb-eu.123doks.com/thumbv2/123dokorg/7340751.91912/15.892.263.646.604.898/andamenti-corrente-integrato-corrispondenza-ancoraggio-cantilener-funzione-tensione.webp)
![Fig. 1.12 Schema del dispositivo costituito da una guida d’onda risonante basata su silicio poroso [18]](https://thumb-eu.123doks.com/thumbv2/123dokorg/7340751.91912/18.892.269.637.422.665/schema-dispositivo-costituito-guida-risonante-basata-silicio-poroso.webp)
![Fig. 1.14 Rappresentazione schematica del dispositivo basato su silicio poroso con tecnica di rivelazione a interferometria in luce bianca [19]](https://thumb-eu.123doks.com/thumbv2/123dokorg/7340751.91912/19.892.274.637.669.950/rappresentazione-schematica-dispositivo-basato-silicio-tecnica-rivelazione-interferometria.webp)