Capitolo 3
3.1 Introduzione di strumenti microfabbricati
per applicazioni biochimiche
Negli ultimi anni, tecnologie e scienze interdisciplinari sono state usate per creare una nuova generazione di strumenti integrati microfabbricati.
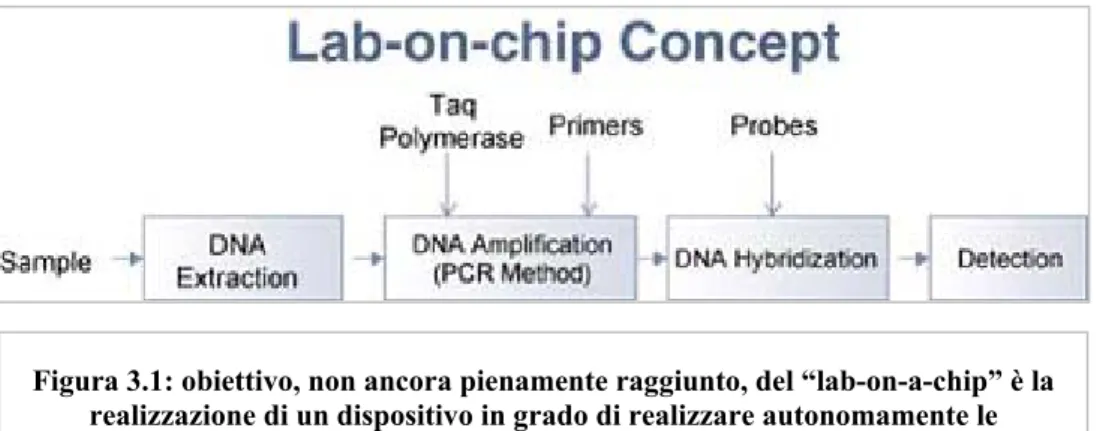
Questi nuovi strumenti vengono definiti “Lab-on-a-Chip” (o “Micro Total Analysis System, µTAS); essi consistono in apparecchiature che hanno come obiettivo l’integrazione su un singolo chip di più processi realizzati in laboratorio [1] (fig. 3.1).
Il loro sviluppo è stato possibile combinando sia tecnologie già esistenti che altre in evoluzione e includono la microlitografia, la microfabbricazione, la tecnologia MEMS (“Micro Electro Mechanical System”), la microfluidica e la nanotecnologia.
Come per l’industria elettronica, in cui i microchip in silicio sono stati il soggetto ideale per la miniaturizzazione, con la possibilità di aumentare la loro funzionalità e capacità, allo stesso modo nell’ingegneria chimica e
biochimica gli strumenti microfabbricati possono realizzare un insieme di funzioni come per esempio preparazione del campione, purificazione, separazioni…
La necessità di manipolare fluidi che si muovono in canali stretti (microfluidica) ha aperto nuove aree di ricerca, ha sviluppato nuovi metodi di fabbricazione per i sistemi fluidici, ha portato alla costruzione di complessi sistemi microfluidici e allo studio del moto di fluidi in canali di piccole dimensioni. Inoltre l’introduzione di tecniche fotolitografiche per la fabbricazione di microsistemi chimici e biochimici, ha incrementato esponenzialmente il numero di applicazioni in tale settore.
Particolarmente interessante è la tecnologia MEMS che applica sullo stesso wafer tecniche di lavorazione usate nella fabbricazione di circuiti integrati per costruire strumenti microscopici elettro-meccanici, come per esempio sensori [2]. La tecnologia MEMS permette di applicare la stessa economia dovuta all’integrazione su piccola scala della lavorazione dei wafer di silicio alla fabbricazione di strumenti meccanici. Per esempio i sensori prodotti usando le tecnologie convenzionali sono costruiti uno per volta, mentre usando la tecnologia MEMS, lo stesso sensore è realizzato in centinaia o migliaia di copie, con prestazioni costanti e basso costo unitario.
Una delle applicazioni della tecnologia MEMS con fluidi in movimento è stata la realizzazione di uno strumento in grado di realizzare
Figura 3.1: obiettivo, non ancora pienamente raggiunto, del “lab-on-a-chip” è la realizzazione di un dispositivo in grado di realizzare autonomamente le
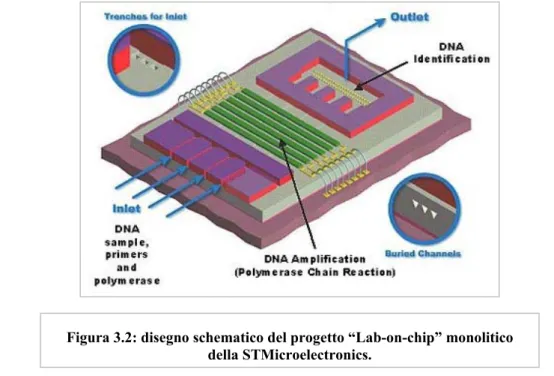
la “Polymerase Chain Reaction” (PCR) ottenendo uno strumento contenente canali in silicio per i reagenti e il campione, elementi riscaldanti per modificare le temperature durante il ciclo di amplificazione e sensori per il controllo della temperatura [3]. Le piccole dimensioni dei canali permettono l’uso di un minor quantitativo di costosi reagenti e la bassa capacità termica del silicio riduce il tempo necessario per la stabilizzazione delle temperature. Il risultato è che il tempo richiesto per realizzare l’amplificazione del campione con la PCR è ridotto da ore a minuti. L’inconveniente è che, non essendo possibile pulire lo strumento, esso è monouso. Inoltre sono in fase di studio progetti monolitici che consentono, oltre all’amplificazione, anche il riconoscimento delle sequenze di DNA
(fig. 3.2).
La miniaturizzazione consente quindi risultati molto superiori rispetto a quelli ottenuti con le tecniche tradizionali.
In conclusione, dalla miniaturizzazione ci si aspettano i seguenti vantaggi:
Figura 3.2: disegno schematico del progetto “Lab-on-chip” monolitico della STMicroelectronics.
• progettazione di sistemi compatti con diminuzione del loro peso e ingombro e quindi portabilità e possibilità di applicazione in luoghi diversi;
• parametri delle operazioni controllabili con precisione; • alto numero di dati elaborabili per unità di volume; • economia dei costi nella produzione di massa; • uso di una quantità minore di reagenti e campioni;
• possibilità di monitoraggio in tempo reale per una più rapida lettura dei dati raccolti.
Nei paragrafi che seguono verranno analizzate alcune metodologie di analisi di acidi nucleici realizzate su dispositivi microfabbricati attualmente in uso. Tali strumenti non realizzano ancora totalmente l’obiettivo che ci si è proposti della costruzione di un “Lab-on-a-Chip”, in quanto le varie operazioni di analisi vengono compiute su più di un dispositivo. Esse in ogni caso costituiscono metodologie di indagine molto potenti di genomi e hanno consentito, per esempio, il raggiungimento dell’importante traguardo del sequenziamento dell’intero genoma umano.
3.2 Realizzazione di Microarray per analisi di
acidi nucleici
Come visto da Southern, l’ibridazione fra acidi nucleici (uno dei quali è immobilizzato su una matrice) costituisce il principio di base per le indagini di sequenziamento degli acidi nucleici. La Southern blot quindi può essere vista come la prima forma di array per acidi nucleici [4].
Lo stesso principio di funzionamento (ibridazione di molecole complementari una delle quali opportunamente marcata per la sua individuazione) può essere usato per costruire DNA microarray, o chip genetici, che consentono esami dell’espressione genetica (per esempio l’analisi dell’ mRNA) permettendo innumerevoli applicazioni come la caratterizzazione di nuovi geni, l’identificazione di geni in percorsi biologici, l’analisi di variazioni genetiche, l’identificazione di obiettivi terapeutici… [5].
Classificandoli in base alla metodologia costruttiva, esistono tre tipi principali di microarray:
• microarray di cloni di DNA: microarray per uso specifico composti di oligonucleotidi oppure di cDNA (ovvero DNA complementare a singola catena ottenuto per clonazione da un campione di mRNA precedentemente isolato);
• microarray di oligonucleotidi prefabbricati: il posizionamento degli oligonucleotidi è fatto sfruttando l’attrazione elettrostatica esercitata su di loro da parte di microelettrodi;
• microarray di oligonucleotidi sintetizzati in situ: microarray ad alta densità contenenti oligonucleotidi sintetizzati usando tecniche fotolitografiche o di tipo“ink-jet”.
I microarray di cloni sono i più usati e possono analizzare RNA proveniente da due diversi campioni su un singolo chip; le limitazioni derivano dalla disponibilità di cloni e dalla qualità dei campioni di mRNA. Le altre due classi sono anche note come array microindirizzabili e permettono l’analisi dell’espressione di un gran numero di geni contemporaneamente ma possono analizzare un solo campione per chip, con costi considerevoli.
3.2.1
Microarray di cloni di DNA
Il principio chiave per la fabbricazione di microarray è che filamenti di DNA a singola catena legati fortemente a supporti rigidi non interagiscono fra loro ma si possono ibridare con RNA complementare [6]. La produzione di un array comincia quindi con la selezione delle sonde che devono essere posizionate sull’array. Tali sonde possono essere prelevate direttamente da librerie genomiche, da sorgenti istituzionali oppure da raccolte di cDNA totalmente o parzialmente sequenziato. Le sonde scelte sono quindi amplificate con il metodo della PCR e, dopo purificazione,
Figura 3.3: a sinistra si ha una visione globale del robot che depone in modo seriale differenti geni sul supporto rigido. A destra il dettaglio dello “spotter” nel momento della
posizionate con opportuno robot su un supporto rigido [7]. Il robot deposita un campione di ciascun gene precedentemente selezionato in posizioni prestabilite su un numero definito di matrici con una operazione seriale (fig. 3.3).
Esistono differenti tipi di iniettore (“spotter”) del robot: è possibile realizzare una “stampa” con uno strumento simile a una penna (fig. 3.4) o usare un tubo capillare al quale è applicata una piccola pressione costante che permette la deposizione del materiale
genetico. Sono anche usati iniettori di tipo inkjet del tutto simili a quelli usati dalle stampanti a inchiostro.
Il supporto dell’array, che inizialmente era costituito da membrane di nylon o nitrocellulosa, attualmente è quasi esclusivamente realizzato con vetrini da microscopio. L’utilizzo del vetro presenta i seguenti vantaggi [8]:
• i campioni di DNA possono essere legati covalentemente sulla sua superficie opportunamente trattata (con poly-L-lisina); • è un materiale duraturo che sopporta alte temperature;
• è un materiale non poroso e quindi il volume di ibridazione può essere minimizzato consentendo un miglior ancoraggio delle sonde e una minore diffusione del DNA depositato;
• come conseguenza della sua bassa fluorescenza intrinseca, esso non da contributi significativi al rumore di fondo durante la rivelazione.
In molti casi il DNA è fissato alla superficie della matrice con irradiazione ultravioletta. Il passo finale, che rende il microarray pronto all’uso, è quello di rendere una parte delle molecole di DNA depositate sul vetrino a singola catena, denaturandole mediante riscaldamento.
Figura 3.4: dettaglio di una punta per “spotting”
È possibile classificare i microarray, in base all’uso che ne viene fatto, in tre categorie [9, 10]:
1. cDNA microarray: per permettere l’analisi su larga scala di un gran quantitativo di mRNA come un indicatore dell’espressione genetica;
2. microarray SNP (“Single Nucleotide Polymorphism”) e array di mutazione: per rilevare polimorfismi o mutazioni in una popolazione usando array SNP o array progettati per rilevare mutazioni conosciute.
3. microarray CHG (“Comparative Hybridization Genomic”): per osservare perdite o guadagni genomici, o un cambiamento nel numero di copie di un gene particolare coinvolto in una malattia.
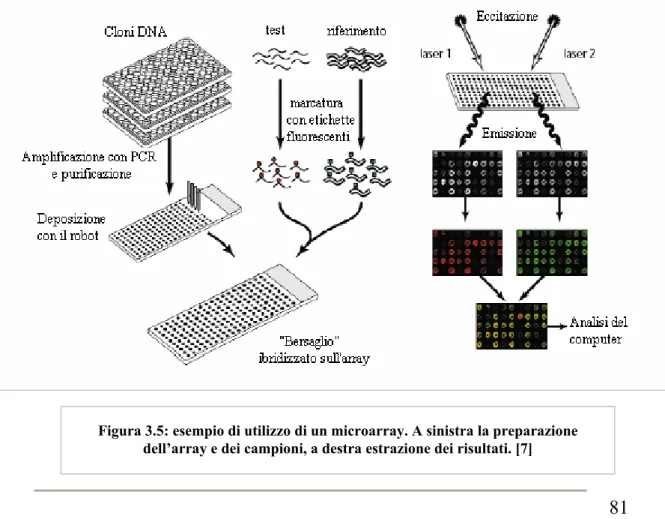
In figura 3.5 è illustrato un esempio del funzionamento del microarray nel caso di misurazione dell’espressione dell’mRNA dell’intero genoma.
Figura 3.5: esempio di utilizzo di un microarray. A sinistra la preparazione dell’array e dei campioni, a destra estrazione dei risultati. [7]
Innanzitutto viene realizzata la trascrizione inversa dell’mRNA estratto da un campione “test” che vogliamo analizzare e il cDNA derivato è marcato con una tintura fluorescente. È anche usato un campione di riferimento (per esempio un tessuto normale o un RNA biologicamente irrilevante) ed è etichettato con fluorocromo con tintura differente dalla precedente. L’analisi per fluorescenza richiede l’uso di questi due campioni bersaglio che, dopo essere stati mescolati, sono posizionati sul microarray e quindi lasciati ibridare con le sonde. Successivamente viene effettuato il confronto dell’espressione genica del test rispetto al riferimento usando sofisticati metodi di analisi dell’immagine. Il risultato immediato di un esperimento con microarray è un catalogo dell’attività trascrizionale per l’insieme dei geni rappresentati sulla griglia, che consente un chiarimento dell’insieme dei geni che sono responsabili di vari processi cellulari.
In questo momento i problemi maggiori si hanno nell’interpretazione dei risultati forniti dagli array, in quanto l’analisi statistica dei microarray è ancora a livello embrionale, soprattutto se paragonata all’avanzata tecnologia dei microarray stessi.
3.2.2 Microarray
di
oligonucleotidi prefabbricati
Negli array microindirizzabili sono usate altre tecniche per posizionare le sonde oligonucleotidiche. Nel caso di posizionamento di sonde sintetizzate fuori dal chip è possibile usare un metodo di attrazione elettrostatica oppure una tecnica di inkjet.
Nel primo caso è necessaria la costruzione di un array che contiene una elevata concentrazione di elettrodi molto piccoli (fig. 3.6) che stanno sotto uno strato di permeabilità molecolare (uno strato di idrogel contenente streptavidina) che serve a “intrappolare” il DNA (sia le sonde da
posizionare sull’array che, successivamente, il risultato dell’ibridazione delle sonde con il DNA bersaglio) [11, 12]. L’applicazione di una polarizzazione positiva a DC a un singolo microelettrodo, fa si che si abbia
un veloce trasporto e concentrazione di molecole di acidi nucleici, caricati negativamente, sopra la locazione selezionata. Gli acidi nucleici (DNA, RNA, polinucleotidi…) possono essere immobilizzati con un attacco diretto sullo strato permeabile che ricopre l’elettrodo, ma anche legandosi a un acido nucleico precedentemente indirizzato e già attaccato. La rapida concentrazione di acidi nucleici consente una notevole riduzione del tempo di ibridazione se comparato alle tecniche di ibridazione passiva; l’ibridazione avviene in secondi invece che in ore.
L’inversione del potenziale elettrico consente una rimozione veloce delle molecole che non si sono legate.
Ripetendo queste operazioni con specifiche sonde e attivando in maniera opportuna gli elettrodi, si ottiene l’assemblaggio della matrice di sonde desiderata. In questa operazione ha un ruolo cruciale lo strato permeabile agli ioni che è posizionato sopra i microelettrodi. Questo strato permette l’attacco degli acidi nucleici. Inoltre consente ad acidi nucleici a singola catena di essere “marcati elettronicamente” sopra questi microelettrodi e ibridarsi agli oligonucleotidi complementari ancorati.
La Nanogen usa tale metodologia per la fabbricazione del “NanoChip Electronic Microarray”, in cui elettrodi circolari di diametro di 80 µm sono
Figura 3.6: in alto dettaglio dell’array di elettrodi; sotto il
NanoChip Electronic Microarray della Nanogen.
posizionati su un chip multistrato (silicio-ossido di silicio-nitruro di silicio). In un chip di 0.7 cm2, l’array ha dimensioni di 2 mm2 e contiene 100 elettrodi (“DNA Test Sites”). L’array può essere posizionato in una apposita cartuccia (“NanoChip Cartridge”) (fig. 3.7).
La parte posteriore della cartuccia la predispone a essere collegata con la “NanoChip Molecular Biology Workstation” [13].
Tale sistema è composto di tre parti principali: un caricatore per inserire i campioni nella cartuccia, uno scanner per la rivelazione dei risultati e un computer che automatizza l’acquisizione dei dati e fornisce i risultati dell’analisi.
La particolarità del sistema è che è programmabile: l’utente può prepararsi da solo le sonde da inserire nei siti di rivelazione tramite indirizzamenti per righe, per colonne o anche per singola cella.
Figura 3.7: Nano Chip Electronic Microarray posizionato nella “NanoChip Cartridge”. In figura i due lati dalla cartuccia.
3.2.3 Microarray
di
oligonucleotidi sintetizzati in situ
La sintesi in situ di oligonucleotidi presenta un certo numero di vantaggi rispetto a quella precedentemente vista. I prodotti di questa sintesi hanno caratteristiche di omogeneità e alta qualità su tutte le celle che compongono l’array su cui vengono sintetizzati, con varie metodologie, oligonucleotidi diversi [14].
Gli array microindirizzabili (array DNA chip) sono una combinazione unica di tecnologie che permettono di fissare le sonde genetiche sulla superficie del microdispositivo. Si ha quindi la produzione di array con centinaia di migliaia di sonde differenti stipate con densità estremamente alta in posizioni note dell’array. Tale caratteristica permette ai ricercatori di ottenere una alta qualità di informazioni sul genoma umano usando piccoli volumi di campione.
Esistono differenti metodi di posizionamento delle sonde sulla superficie dell’array. Il metodo più conosciuto combina tecniche fotolitografiche usate nell’industria dei semiconduttori a tecniche di sintesi in fase solida, per ibridare direttamente su un wafer di quarzo le sonde oligonucleotidiche di lunghezza desiderata (di solito 25 nucleotidi). Questo tipo di tecnologia è stata derivata direttamente da studi fatti da Foder che ha usato tecniche di fotolitografia per la sintesi chimica in situ di materiale biochimico direttamente su silicio [15].
La fabbricazione del Gene Chip Affimetrix comincia da un wafer di quarzo di 5-inch quadrati [16]. Poiché il quarzo è un materiale idrossilato naturalmente, esso fornisce un eccellente substrato per l’attacco di elementi chimici. Su di esso vengono quindi posizionate molecole di collegamento sintetiche (molecole “linker”) modificate con gruppi di protezione rimovibili fotochimicamente che serviranno successivamente per
posizionare le sonde sull’array. La distanza fra queste molecole “linker” determina la densità di riempimento delle sonde.
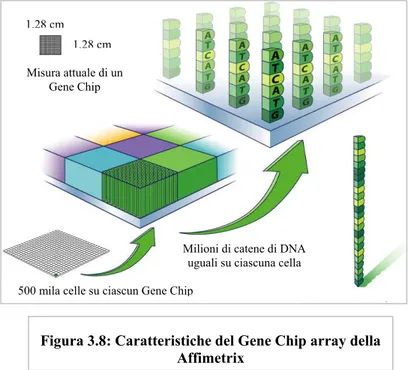
Con questo metodo è possibile costruire array con più di 500 mila locazioni (o celle) per le sonde contenute in 1.28 cm2. Ciascuna di queste locazioni contiene milioni di molecole identiche di DNA (diverse per ciascuna locazione) (fig. 3.8).
Le molecole “linker” attaccate alla matrice forniscono quindi una superficie di aggancio per le sonde; la superficie di aggancio deve essere spazialmente attivata dalla luce ultravioletta.
La sintesi delle sonde avviene in parallelo essendo conseguenza dell’aggiunta di un nucleotide A, C, T o G a più catene che crescono contemporaneamente.
Per definire quali catene oligonucleotidiche dovranno ricevere un oligonucleotide ad ogni passo, maschere fotolitografiche, con finestre di 18-20 µm2 ovvero di dimensioni pari a quella di una singola locazione, sono poste sopra il wafer su cui sono posizionate le molecole “linker”. Le
Milioni di catene di DNA uguali su ciascuna cella 500 mila celle su ciascun Gene Chip
1.28 cm 1.28 cm Misura attuale di un
Gene Chip
Figura 3.8: Caratteristiche del Gene Chip array della Affimetrix
finestre sono distribuite sulla maschera in base alla sequenza desiderata di ciascuna sonda (fig. 3.9).
Quando la luce ultravioletta colpisce la maschera nel primo passo di sintesi, i “linker” esposti vengono deprotetti e sono disponibili per l’accoppiamento con il nucleotide (fig. 3.10).
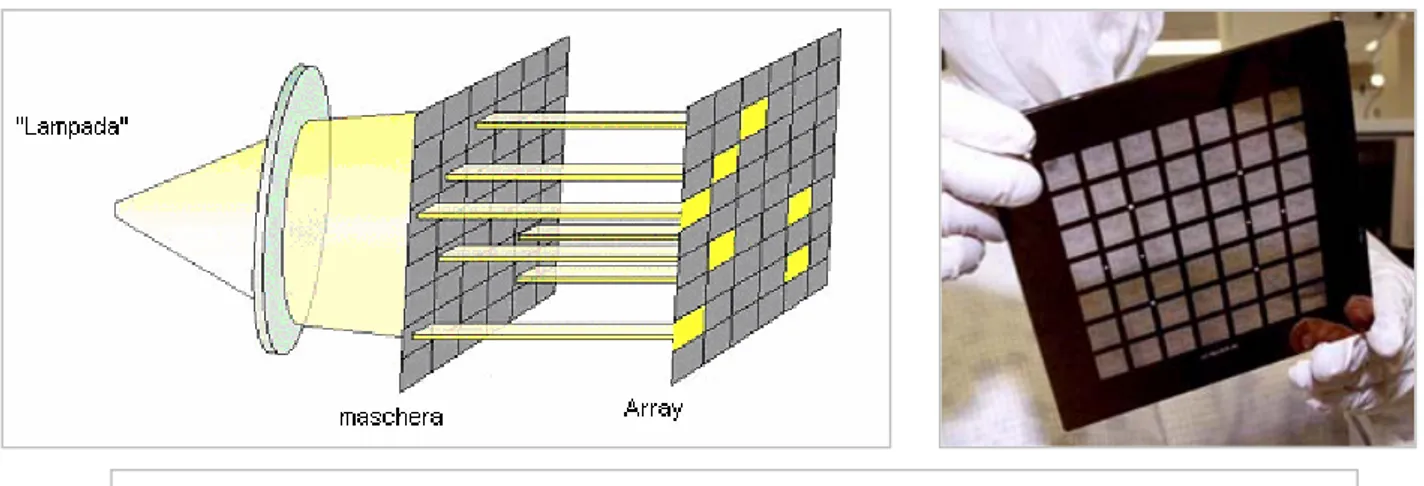
Figura 3.9: nella figura di sinistra si vede come la maschera possa far colpire dalla luce solo determinate locazioni. A destra una maschera.
Figura 3.10: ciclo di sintesi delle sonde sull’array. Nucleotidi differenti non possono essere inseriti in un unico passaggio: per sistemare ogni livello di una
La parte critica di questo passo è il processo di allineamento della maschera con il wafer prima di ciascun passo di sintesi. Per assicurare che questo passo sia accuratamente completato, le tracce di cromo presenti sul wafer e sulla maschera devono essere perfettamente allineate.
Una volta che le locazioni sono state attivate, una soluzione contenente un singolo tipo di desossinucleotidi è gettata sulla superficie del wafer e i nucleotidi si attaccano ai “linker” attivati con un accoppiamento chimico, dando inizio al processo di sintesi.
Il processo è altamente efficiente anche se talvolta l’aggancio fallisce. In tal caso le sonde con il nucleotide mancante vengono opportunamente “incappucciate” per bloccarne la crescita.
Nel passo successivo di sintesi, un’altra maschera è posizionata sopra il wafer per permettere un ulteriore ciclo di deprotezione e accoppiamento. Il processo è ripetuto fino a che le sonde non
raggiungono la lunghezza voluta (nell’esempio di figura 3.8, 25 nucleotidi).
Per esempio un insieme completo di 4n polidesossinucleotidi di lunghezza N (o un sottoinsieme) può essere sintetizzato in 4 x N cicli [17].
Esistono inoltre opportuni algoritmi che permettono di minimizzare il numero di maschere utilizzate coordinando la crescita delle sonde nelle diverse locazioni, individuando situazioni in cui più maschere possono essere utilizzate nello stesso tempo
[18]. Una volta completata la sintesi, i wafer sono tagliati; in funzione del numero di locazioni delle sonde per array, da un singolo wafer, è possibile produrre tra 49 e 400 array.
Figura 3.11: Gene Chip della Affimetrix pronto a essere
I risultanti singoli array sono quindi posizionati in apposite cartucce (fig. 3.11) in cui può circolare il flusso biologico da analizzare, opportunamente marcato.
Il campione di acido nucleico marcato con fluorescenza si lega sull’array con l’oligonucleotide complementare (fig. 3.12).
L’eccitazione laser, che entra dalla parte posteriore del supporto di quarzo (trasparente), è focalizzata all’interfaccia tra la superficie dell’array e la soluzione bersaglio ibridata; quando si ha l’eccitazione laser, solo dalle locazioni di array in cui è avvenuta l’ibridazione si ha l’emissione fluorescente (fig. 3.13).
L’emissione fluorescente è quindi raccolta tramite una lente e passa attraverso una serie di filtri ottici fino a un rivelatore sensibile. Con una semplice scansione, il laser irradia o traduce l’array (o entrambe le cose) e si ottiene una immagine bidimensionale dell’intensità di ibridazione.
Figura 3.12: esempio in cui frammenti di RNA marcati con fluorescenza si ibridano con le sonde posizionate sull’array.
Un altro metodo di ibridazione in situ di oligonucleotidi prevede la combinazione della tecnica inkjet usata dalle stampanti a inchiostro in commercio, con la chimica standard dei fosforamiditi, che consente la sintetizzazione di filamenti di DNA desiderati mediante l’aggiunta di monomeri attivati (deossiribonucleosidi 3´-fosforamiditi) a una catena legata a un supporto insolubile (vetro) [19].
Una normale stampante consiste in una serie di testine di stampa con più ugelli per ciascuna, attraverso cui l’inchiostro può fluire sulla carta. I vari ugelli sono controllati tramite computer.
La stampante viene usata in maniera del tutto simile se gli ugelli devono posizionare basi di acidi nucleici su vetro, invece di inchiostro su carta. Fondamentale è la scelta degli ugelli, che sono sottoposti a severi controlli di qualità, e del tipo di vetro che deve ottimizzare le condizioni di lavoro per lo scanner. Il vetro deve quindi essere di spessore uniforme, avere la superficie liscia, libera da aberrazioni, avvallamenti e presentare
Figura 3.13: durante l’analisi, dopo che è avvenuta l’ibridazione, l’array presenta zone ibridate in grado di emettere fluorescenza e zone “buie”,
minima fluorescenza propria. Inoltre, poiché naturalmente le basi non si legano al vetro, sulla sua superficie deve essere applicato un substrato attivatore che favorisce l’ancoraggio degli acidi nucleici.
Nel processo di stampa dei microarray di oligonucleotidi, le basi sono posizionate sull’array una alla volta secondo logica prestabilita per formare catene con sequenze desiderate (fig. 3.14). Con questo tipo di tecnologia è possibile produrre microarray di oligonucleotidi lunghi fino a 60 basi [20].
Con la tecnica ora descritta è anche possibile la deposisizione sul microarray di catene di cDNA o di oligonucleotidi presintetizzati. Ogni volta che un acido nucleico è posizionato, si può contollare visivamente in tempo reale se la procedura eseguita ha dato i risultati attesi.
Figura 3.14: processo di stampa degli array con la tecnica di inkjet. A) primo strato deposto di nucleotidi sulla superficie del microarray attivata. B) crescita degli oligonucleotidi. C-D) dettaglio del momento in cui una nuova base viene aggiunta alla