ALMA MATER STUDIORUM
UNIVERSITÀ DI BOLOGNA
DOTTORATO DI RICERCA IN
ONCOLOGIA E PATOLOGIA SPERIMENTALE
Progetto “Oncologia” - Indirizzo Biologia Molecolare
Ciclo XXIII
Settore scientifico-disciplinare di afferenza: MED/08
STUDIO DI MARCATORI IMMUNOISTOCHIMICI,
ANALISI FISH E RUOLO DEI MICRORNA NELLE
NEOPLASIE GLIALI DI BASSO E DI ALTO GRADO.
Presentata dal Dott. Gianluca Marucci
Coordinatore Dottorato
Relatore
Chiar.mo Prof.
Chiar.ma Prof.ssa
Sandro Grilli
Maria Pia Foschini
INDEX
INTRODUZIONE GENERALE ...7
Gliomi ...7
Classificazione istologica dei gliomi ...7
Marcatori di differenziazione oligodendrogliale ...8
I miRNA nei glioblastomi...8
Scopo della tesi ...9
SEZIONE 1. Profilo di espressione genica nei glioblastomi e valutazione immunoistochimica di IGFBP-2 e CDC20...11
1.1.1 Arrays nello studio dei gliomi ...11
1.1.2 Obiettivo dello studio ...11
1.2 Materiali e Metodi...13
1.2.1 Selezione dei casi ...13
1.2.2 Estrazione e marcatura dell’RNA...13
1.2.3 Analisi dei dati del microarray ...14
1.2.4 Validazione mediante RT-PCR ...15
1.2.5 Immunoistochimica ...16
1.3 Risultati ...19
1.3.1 Risultati di espressione genica...19
1.3.2 Risultati dell’analisi immunoistochimica...24
1.4 Discussione e conclusioni ...31
SEZIONE 2. Nogo-A: ruolo nella diagnosi di oligodendroglioma e nell’identificazione della codelezione 1p/19q...35
2.1.1 Olig-2...35
Index
2.1.3 Perdita cromosomica di 1p e 19q ...35
2.1.4 Obiettivo dello studio ...36
2.2 Materiali e Metodi...37
2.2.1 Selezione dei casi ...37
2.2.2 Analisi immunoistochimica...37
2.2.3 Analisi FISH per la ricerca della delezione di 1p e 19q ...39
2.2.4 Analisi dei dati...41
2.3 Risultati ...43
2.3.1 Risultati dell’analisi immunoistochimica ...43
2.3.2 Analisi statistica dei dati immunoistochimici...47
2.3.3 Risultati dell’analisi FISH ...48
2.4 Discussione e Conclusioni ...53
SEZIONE 3. miRNA nei glioblastomi...57
3.1.1 miRNA ...57
3.1.2 Modulazione della traduzione ...57
3.1.3 miRNA nelle neoplase umane ...58
3.1.4 Ruolo dei miRNA nell’oncogenesi e nella target-therapy ...59
3.1.5 Studi concernenti i miRNA nei glioblastomi ...59
3.1.6 Obiettivo dello studio ...61
3.2 Materiali e Metodi...63
3.2.1 Selezione dei casi e preparazione dei campioni ...63
3.2.2 Selezione dei miRNA...64
3.2.3 Disegno dei primers...65
3.2.4 Estrazione dei miRNA...65
3.2.5 qRT-PCR ...66
Index
INTRODUZIONE GENERALE
Gliomi
I gliomi costituiscono i tumori primitivi più comuni del Sistema Nervoso Centrale. Tra i glomi di alto grado, il glioblastoma (GL) mostra il decorso clinico più aggressivo ed una sopravvivenza media dopo la diagnosi di 14 mesi [1].
Nonostante i progressi delle indagini neuroradiologiche, delle tecniche chirurgiche e delle terapie post-chirurgiche (chemioterapia e radioterapia), l’aspettativa di vita dei pazienti con GL non ha subito significative modificazioni negli ultimi decenni.
D’altra parte, i tumori gliali con aspetti oligodendrogliali, anche quando presentano caratteristiche anaplastiche, generalmente mostrano una certa sensibilità a radio e chemioterapia ed una sopravvivenza migliore.
Classificazione istologica dei gliomi
Ancora oggi la classificazione istologica dei gliomi, secondo i criteri dettati dalla Organizzazione Mondiale della Sanità (World Health Organization, WHO) [2], rappresenta la variabile che maggiormente condiziona le decisioni terapeutiche e le previsioni prognostiche. Tuttavia la variabilità intra ed inter-osservatore comporta dei limiti nella riproducibilità diagnostica di queste neoplasie. Coons et al. hanno trovato una completa concordanza diagnostica tra quattro neuropatologi, esperti nella diagnosi dei glomi, nel 69% dei casi [3]. Allo stesso modo Aldape et al. [4] e Bruner et al. [5] hanno osservato una percentuale di discordanza diagnostica rispettivamente nel 23% e nel 42,8% dei casi.
Marcatori di differenziazione oligodendrogliale
In considerazione dei suddetti limiti nella diagnosi differenziale tra astrocitomi (A), oligodendrogliomi (OL) e oligoastrocitomi (OA), in passato numerosi studi sono stati realizzati con l'obiettivo di individuare marcatori immunoistochimici utili a mettere in
evidenza una differenziazione oligodendrogliale. A questo proposito sono state studiate varie molecole come la proteina S100, Leu7, CD57 e p25 [6,7], sebbene il loro reale valore
diagnostico rimanga fortemente controverso.
Recentemente è stato dimostrato che marcatori neuronali come il NeuN e la sinaptofisina sono espressi nei tumori oligodendrogliali, a prescindere dalla presenza di una
differenziazione neurocitica [2,8,9], ma le percentuali riportate per la sinaptofisina sono estremamente variabili, andando da una positività del 9,3% [10] ad una del 100% degli OL studiati [11].
Negli ultimi anni due molecole, Olig-2 e Nogo-A, sono state proposte come marcatori riproducibili di differenziazione oligodendrogliale [12, 13].
Inoltre gli OL mostrano frequentemente la contemporanea perdita cromosomica di 1p e 19q, una caratteristica che risulta predittiva di risposta alla chemioterapia. E’ inoltre un indice prognostico in quanto i casi di OL con codelezione hanno mostrato una sopravvivenza significativamente maggiore [14].
I miRNA nei glioblastomi
I miRNA (o microRNA) sono piccole molecole di RNA non codificanti in grado di regolare l’espressione genica agendo a livello post-trascrizionale.
cellulari [16, 17, 18, 19, 20, 21]. Nei GL in tre diversi studi indipendenti [21, 22, 23] sono stati identificati 17 miRNA overespressi e 33 miRNA ipoespressi.
Scopo della tesi.
Scopo della tesi è stato quello di sviluppare i seguenti progetti di ricerca:
1) studiare una serie di gliomi, sia di alto grado che di basso grado, a livello genico e proteico (immunoistochimico) per mettere in evidenza possibili marcatori di malignità;
2) verificare l'utilità di Nogo-A, Olig-2 e Sinaptofisina nel raggiungere una corretta diagnosi istologica di OL e valutare un possibile ruolo complementare nella identificazione mediante FISH della codelezione 1p19q;
3) delineare un profilo di espressione dei miRNA nei GL e paragonarlo con quello ottenuto da tessuto cerebrale non neoplastico.
SEZIONE 1. Profilo di espressione genica nei glioblastomi e valutazione
immunoistochimica di IGFBP-2 e CDC20
1.1.1 Arrays nello studio dei gliomi
Al fine di sviluppare una classificazione dei gliomi più oggettiva, studi recenti si sono concentrati sulle analisi genetico-molecolari [24], in particolare sull’uso di microarrays per identificare marcatori biomolecolari tumorali. Attualmente i pochi markers biomolecolari disponibili nei tumori cerebrali sono costituiti dalla delezione dei cromosomi 1p e 19q per gli oligodendrogliomi (OL) [25] e da C-MYC per il medulloblastoma [26]. Un singolo biomarcatore generalmente ha un valore predittivo limitato, soprattutto se altri geni o proteine giocano in ruolo nel determinare la prognosi. I DNA microarrays possono essere usati per identificare un gruppo di geni che, in associazione, contengono un numero maggiore di informazioni rispetto a un singolo biomarcatore. I geni emersi da tali microarrays vengono usualmente sottoposti a verifica mediante RT-PCR (reverse transcriptase polymerase chain reaction) e/o immunoistochimica.
1.1.2 Obiettivo dello studio
Una serie di 39 gliomi, suddivisi in 28 gliomi di alto grado e 11 gliomi di basso grado, è stata studiata a livello genico e proteico (immunoistochimico) per mettere in evidenza possibili marcatori genetici di malignità.
1.2 MATERIALI E METODI
1.2.1 Selezione dei casi
Sono stati selezionati random 100 casi di gliomi. Tutti i casi erano stati ricevuti nel nostro laboratorio non fissati. Un campione di tessuto neoplastico di ciascun caso era stato congelato con azoto liquido e conservato a −80°C nella banca dei tessuti congelati della Sezione di Anatomia Patologica del Dipartimento di Ematologia ed Oncologia
dell'Università di Bologna, presso l'Ospedale Bellaria (Bologna), tra il 1990 ed il 2002. La porzione di tessuto rimanente (speculare) era stata fissata in formalina, inclusa in paraffina e colorata con ematossilina/eosina per la diagnosi istologica di routine. Da ciascun paziente era stato ottenuto il consenso informato.
Soltanto i casi con un rapporto rRNA 28S/18S >1,5, misurato dal Bioanalyzer 2100 (Agilent, vedi sotto per i dettagli) e senza evidenza di degradazione ribosomiale, sono stati inclusi. Alcuni casi sono stati esclusi poiché il campione di tessuto prelevato era costituito unicamente da materiale necrotico; in altri casi nel campione congelato non era stato incluso tessuto neoplastico; in altri ancora, il tessuto non era sufficiente per poter ottenere una sezione al criostato con cui verificare la presenza di tessuto neoplastico. Pertanto dei 100 casi selezionati, solo 39 neoplasie gliali sono state arruolabili per questo studio: nello specifico si trattava di 28 GL e 11 gliomi di basso grado, ovvero 4 OL, 5 astrocitomi pilocitici (PA) e 2 astrocitomi grado II (FA). Tutti i tumori sono stati
rivalutati istologicamente e classificati in accordo alla Classificazione WHO del 2007 [2].
1.2.2 Estrazione e marcatura dell’RNA
I campioni sono stati trattati con RNAlater™ (Qiagen, Hilden, Germany). L’RNA è stato estratto (RNAeasy Protect mini kit, Qiagen, Hilden, Germany) e analizzato con il
Bioanalyzer 2100 (Agilent Technologies, Palo Alto, CA, USA). Sono stati usati
oligomicroarrays Agilent Human 1A contenenti sonde DNA 60-mer sintetizzate in situ. Su 19.061 spots erano presenti 17.086 sequenze trascritte uniche, corrispondenti a 15.989 geni umani unici.
La marcatura del cRNA, l’ibridizzazione all’array di 22K-geni e la determinazione dei rapporti di espressione sono stati effettuati come già descritto in precedenza [27]. Il cRNA è stato generato per mezzo di trascrizione in situ mediante RNA polimerasi T7 e marcato con Cy3-CTP (Perkin Elmer, Waltham, MA, USA) per l’RNA di riferimento oppure con Cy5-CTP (Perkin Elmer, Waltham, MA, USA) per l’RNA da testare. Come RNA di riferimento è stato utilizzato un pool di RNA totale dei glomi di basso grado arruolati per lo studio (quattro OL, cinque AP e due A).
Il campione marcato (0,75 μg) ed il cRNA di riferimento sono stati frammentati e
ibridizzati a 60°C per 17 ore. Dopo l’ibridizzazione, i vetrini sono stati lavati e analizzati con uno scanner laser confocale (Agilent Catalogue, Number G2565BA). L’intensità del segnale di fluorescenza nelle immagini scannerizzate è stata quantificata mediante apposito software e i dati di LogRatio utilizzati per le analisi statistiche.
1.2.3 Analisi dei dati del microarray
I dati sono stati analizzati con l’Agilent Feature Extraction Software, che esegue una localizzazione degli spots (find spot algorithm), elimina il cosiddetto rumore di fondo (cookie cutter algorithm) e riconosce gli spots con adeguato livello di saturazione (sono considerati tali quando più del 50% dei suoi pixels mostra un’intensità superiore a
Il metodo di classificazione cosiddetto “nearest shrunken centroid” è stato utilizzato per identificare i geni utili a classificare ed a meglio caratterizzare i glomi di alto grado rispetto a quelli di basso grado [30]. L’analisi è stata condotta utilizzando il PAMR package di Bioconductor. Una serie di geni espressi in maniera differente è stata evidenziata per mezzo del LIMMA package [31], usando un t statistico.
Il tempo di sopravvivenza è stato definito come il tempo intercorso tra la chirurgia e la morte per malattia. I livelli di espressione genica sono stati raggruppati in due categorie: alto livello e basso livello di espressione, assumendo 0 come valore di cut-point.
Un’analisi di sopravvivenza univariata è stata effettuata per ciascun gene emerso dal metodo “nearest shrunken centroid”, usando il log-rank test, e le curve di sopravvivenza sono state valutate con il metodo di Kaplan–Meier. Il modello di Cox (multivariate regression) è stato adottato considerando tutti i geni e l’età alla diagnosi come covariabili. Le analisi statistiche sono state eseguite utilizzando STATA (V.9).
1.2.4 Validazione mediante RT-PCR
La Real time RT-PCR è stata utilizzata in tutti i casi per confermare i risultati di
espressione genica dei microarray per 5 geni (insulin-like growth factor binding protein 2 (IGFBP-2), cell division cycle 20 homologue (CDC20), lymphocyte antigen 6 complex, locus H (LY6H), NADH dehydrogenase Fe-S protein 2 (NDUFS2) e transmembrane 4 super-family 2 (TM4SF2)). Sono stati studiati solo questi 5 geni in quanto erano quelli maggiormente discriminati tra i tumori di basso e di alto grado (vedi i “Risultati”). In breve, un’aliquota di 1 μg dello stesso RNA utilizzato per i microarray è stato retro-trascritto usando Super-Script™ First-Strand Synthesis System per RT-PCR (Invitrogen). La PCR è stata realizzata su un volume di reagenti di 25 μl contenente 10 pmol di ciascun
primer, 1 U di FastStart Taq DNA Polymerase™ (Roche), 1× GC rich solution (Roche), 4 mM MgCl2, 1× Rox (Invitrogen) e 1× GelStar in DMSO (Lonza).
La RT-PCR è stata eseguita mediante Applied Bio systems SDS-7000 thermal cycle (Applied Bio systems, CA, USA), seguendo 40 cicli di denaturazione a 95°C per 30 s, appaiamento dei primers a 58°C, estensione a 72°C per 30 s e acquisizione dei dati. Le sequenze dei primers sono elencate nella Tabella 1 e sono state estrapolate dall’RTPrimerDB database (http://medgen.ugent.be/rtprimerdb/) [32]. Quattro geni housekeeping sono stati scelti per la normalizzazione dei dati (ACTB, B2M, TBP e HPRT1). La quantificazione relativa è stata eseguita utilizzando qBase
(http://medgen.ugent.be/qbase/; Vandesompele et al.) [33]. Per l’analisi statistica è stato utilizzato il test di Wilcoxon, adottando la correzione di Bonferroni per i confronti multipli.
Tabella 1. Sequenze dei primers utilizzati per RT-PCR.
Nome del Gene Forward primer Reverse Primer
ACTB TTGCCGACAGGATGCAGAAGGA AGGTGGACAGCGAGGCCAGGAT
B2M ATGAGTATGCCTGCCGTGTGA GGCATCTTCAAACCTCCATG
TBP TGCACAGGAGCCAAGAGTGAA CACATCACAGCTCCCCACCA
HPRT TGACACTGGCAAAACAATGCCA GGTCCTTTTCACCAGCAAGCT
IGFBP2 GCCCTCTGGAGCACCTCTACT CATCTTGCACTGTTTGAGGTTGTAC CDC20 AGACCTGCCGTTACATTCCTTC GCCAGTACATTCCCAGAACTCC
LY6H GTGTGTGCCAGTGTCCGAATC AGTCACAGGAGGAGGCACACAT
NDUFS2 CAGGCCTATTCTCTAGCTGTGGA GCCATGATGTGGTTCAACAAAC TM4SF2 CTGCTGCATGAACGAAACTGAT CAGCGATGATTCCCATGTTAGT
1.2.5 Immunoistochimica
anaplastici (AO) e 1 oligoastrocitoma anaplastico(OAA)) sono stati selezionati random al fine di ampliare lo studio immunoistochimico.
I casi sono stati immunocolorati con gli anticorpi anti-GFAP (Dako, clone 6F2, diluizione 1:1200), anti-Ki 67 (Novocastra, clone Mib1, diluizione 1: 200), anti-IGFBP-2 (Cell Signalling Technology, policlonale, diluizione 1:25) ed anti-CDC20 (Santa Cruz Biotechnology, clone p55 CDC (E-7), diluizione 1:50). Per l’IGFBP-2 è stata effettuata una valutazione semiquantitativa della positività citoplasmatica, utilizzando il tessuto cerebrale normale come controllo positivo. Per il CDC-20 è stata realizzata una valutazione semiquantitativa della positività nucleare, utilizzando le cellule endoteliali come controllo positivo. L’assenza di anticorpo primario è stata utilizzata come controllo negativo. Il conteggio è stato effettuato su 10 aree neoplastiche a forte ingrandimento (ingrandimento X400) attribuendo i seguenti score:
Negativo: <5% di cellule con positività immunoistochimica; +: dal 5 al 10% di cellule positive;
++: > 11% di cellule positive.
Ai fini delle indagini statistiche sono stati considerati positivi i casi con uno score + e ++. Il Ki67 è stato valutato attribuendo una percentuale dopo aver valutato 10 campi a forte ingrandimento (X 400) per ciascun caso.
L’associazione tra grado del tumore ed espressione immunoistochimica di IGFBP-2 e CDC 20 è stata valutata usando il test di Fisher. L’analisi ANOVA è stata adottata per confrontare i livelli medi di espressione genica di IGFBP2 e CDC20 nei vari gruppi di score immunoistochimico (è stato utilizzato il test di Levene per valutare l’omogeneità di varianza). E’ stata valutata anche l’associazione di IGFBP-2 e CDC20 con la sopravvivenza. Le curve di sopravvivenza sono state elaborate usando il metodo standard di Kaplan-Meier ed il log-rank test.
1.3 RISULTATI
1.3.1 Risultati di espressione genica
I risultati di espressione genica sono stati ottenuti da 39 campioni (28 GL e 11 gliomi di basso grado) e sono disponibili sul sito web GEO (http://www.ncbi.nlm.nih.gov/geo/) con il numero di accesso GSE9885.
Tabella 2. Dati clinico-patologici ed immunoistochimici.
Caso n. Età Sex Sede (mesi)OS Ki67 X 10 HPFMitosi IGFBP2ICH CDC20ICH
GL1 68 F Fr 0,5 OD 10% 2 + -GL2 78 F Fr 5,2 8% 2 + + GL3 69 M Te 4,9 5% 1 + -GL4 41 M Pa 1,5 10% 1 + -GL5 43 M Fr 7,7 18% 3 ++ + GL6 71 F Pa 14,2 ND ND ND ND GL7 54 F Fr 14,5 15% 2 + + GL8 31 M Suprat 32,9 15% 4 ++ -GL9 31 M Te 18,6 25% 3 ++ + GL10 67 F Te 1,6 30% 12 ++ + GL12 73 M Suprat 1,9 10% 2 + + GL15 59 F Te 9,1 30% 5 + + GL16 36 F Te 18,8 15% 3 ++ + GL17 67 F Fr 5,8 8% 1 + + GL18 53 F Fr 26,3 20% 4 ++ ++ GL19 60 M Fr-Te 11,5 30% 7 + ++ GL20 58 M Fr 13,2 20% 4 +
-PA21 7 M Cereb Vivo dopo 122 2% 0 -
-PA22 10 F Cereb Vivo dopo 184 4% 1 -
-PA23 39 F Suprat Vivo dopo 105 0% 0 -
-FA24 29 M Fr Vivo dopo 175 ND ND ND ND
GL25 63 M Suprat 18,9 15% 2 ++ +
GL26 39 F Fr 10,1 20% 4 + ++
GL27 54 M Fr ND 5% 1 + +
PA28 26 M Te Vivo dopo 180 3% 1 +
-OL29 32 M Fr 105 1% 0 -
-OL30 60 F Suprat Vivo dopo 92 3% 0 -
-OL31 56 M Fr Vivo dopo 85 6% 0 -
-OL32 41 F Fr 132 5% 0 -
-PA33 3 F Cereb Vivo dopo 195 2% 1 -
-FA34 34 F Pa 134 2% 0 - -GL35 50 M Fr 14,2 8% 1 + ++ GL36 69 M Te 1,0 10% 4 - + GL37 67 F Suprat 19,8 20% 2 + ++ GL38 67 M Occ 0,4 OD 7% 1 + -GL42 57 M Te 10,2 6% 1 + -GL44 54 F Suprat 11,4 20% 2 + +
GL46 71 F Suprat Vivo dopo 42 6% 1 - +
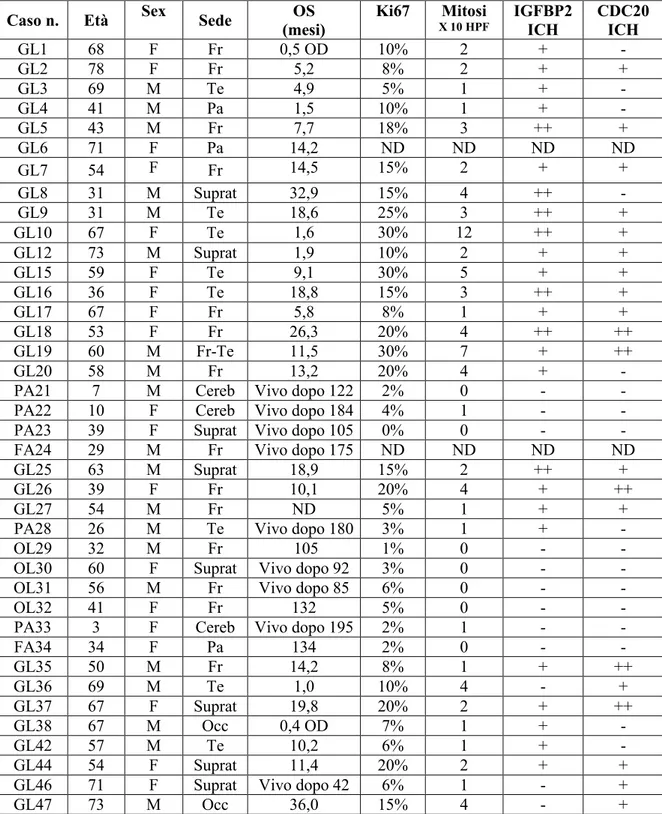
Il clustering gerarchico, basato sui dati di espressione genica e realizzato utilizzando il metodo di Ward (Figura 1),
Figura 1. Cluster dendrogram. gl= glioblastoma; pa= astrocitoma pilocitico; fa= astrocitoma fibrillare; ol= oligodendroglioma
ha separato due gruppi che mostrano una buona concordanza con l’istologia: 100% per gli OL (4 su 4 nel gruppo 1), 100% per PA (5 su 5 PA nel gruppo 1) e 50% per FA (1 su 2 nel gruppo 1). I GL sono stati inclusi dal cluster dendrogram nel gruppo 2 (25 su 28, 89,3%) insieme ad 1 FA.
Il tempo di sopravvivenza media per il gruppo 2 è stato di 10 mesi. Solo tre GL sono stati aggregati al gruppo 1 che includeva la maggior parte dei gliomi di basso grado. Tuttavia la sopravvivenza dei tre pazienti è stata rispettivamente di 2, 11 e 13 mesi, in linea con quella dei GL. Tutti gli altri casi del gruppo 1 sono vivi dopo 4 anni di follow-up.
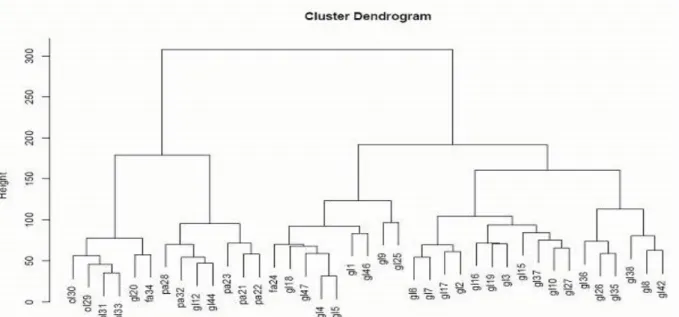
Il metodo “nearest shrunken centroid” ha identificato 9 geni (IGFBP-2, CDC20, LY6H, NDUFS2, TM4SF2, SNURF, GABARAPL1, AZGP1, BC016828) come quelli maggiormente significativi nel distinguere tra gliomi di basso e di alto grado. Tra di essi,
7 geni risultavano iperespressi nei gliomi di basso grado, ma ipoespressi nei GL; al contrario 2 geni (IGFBP-2 e CDC20) erano iperespressi nei GL ed ipoespressi nei gliomi di basso grado (Figura 2).
Figura 2. Espressione genica dei 9 geni selezionati dal metodo “nearest shrunken centroid”. In rosso i casi di GL, in verde i glomi di basso grado.
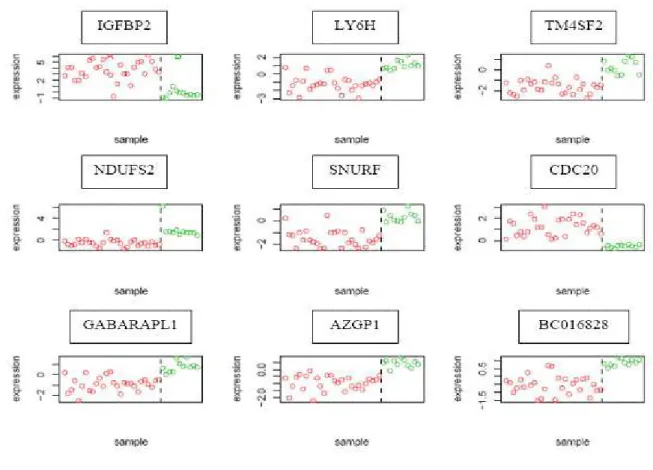
Tutti i 9 geni erano inclusi tra i 50 geni risultati maggiormente significativi negli arrays (Tabella 3).
Tabella 3. I 50 geni maggiormente significativi.
ProbeName GeneName logFC AveExpr t P.Value adj.P.Val B
1 A_23_P100653 ZBTB4 -1.031 11.444 -9.328 1.53406E-11 2.86251E-06 16.080 2 A_23_P169587 THC1942991 -1.113 7.533 -8.747 8.67318E-11 6.7725E-06 14.448 3 A_23_P71270 AZGP1 -1.388 8.699 -8.590 1.39631E-10 6.7725E-06 13.998 4 A_23_P117599 AHSA1 -1.312 8.259 -8.547 1.59044E-10 6.7725E-06 13.875
5 A_23_P67708 M65214 1.254 9.420 8.504 1.81473E-10 6.7725E-06 13.750
6 A_23_P216325 BC016828 -1.421 8.122 -8.296 3.42001E-10 7.90833E-06 13.150 7 A_23_P207125 NLGN2 -0.746 9.490 -8.295 3.43279E-10 7.90833E-06 13.147 8 A_23_P119943 IGFBP2 3.839 11.245 8.262 3.79834E-10 7.90833E-06 13.051 9 A_23_P60640 NM_017921 -1.030 8.070 -8.255 3.88939E-10 7.90833E-06 13.028 10 A_23_P155695 GAK -1.002 7.464 -8.227 4.23818E-10 7.90833E-06 12.947 11 A_23_P145297 NFKBIL1 -1.070 7.883 -8.151 5.34856E-10 8.20308E-06 12.726 12 A_23_P114144 NM_014008 -0.788 7.688 -8.145 5.44401E-10 8.20308E-06 12.710 13 A_23_P131899 SDCBP2 -0.950 8.064 -8.130 5.71498E-10 8.20308E-06 12.664 14 A_23_P120182 NDUFA10 -1.055 8.798 -7.945 1.01223E-09 1.2802E-05 12.121 15 A_23_P77066 SNURF -1.802 7.976 -7.940 1.02912E-09 1.2802E-05 12.106 16 A_23_P114185 TM4SF2 -1.931 8.810 -7.912 1.12048E-09 1.30674E-05 12.025 17 A_23_P156568 KLHDC3 -0.828 8.059 -7.800 1.5892E-09 1.73017E-05 11.693 18 A_23_P3574 NM_030819 -0.893 8.613 -7.785 1.669E-09 1.73017E-05 11.646 19 A_23_P162640 GABARAPL1 -1.708 9.221 -7.761 1.79763E-09 1.74373E-05 11.576 20 A_23_P1307 AKR1C1 -1.404 7.134 -7.748 1.86897E-09 1.74373E-05 11.539 21 A_23_P38497 SLC25A11 -0.739 7.773 -7.721 2.03425E-09 1.80755E-05 11.458 22 A_23_P149195 CDC20 1.803 8.811 7.695 2.20592E-09 1.83837E-05 11.381 23 A_23_P166609 DHX30 -1.086 8.637 -7.676 2.34274E-09 1.83837E-05 11.324 24 A_23_P72807 SHC3 -1.283 7.539 -7.673 2.3645E-09 1.83837E-05 11.315 25 A_23_P39024 NM_032332 -0.677 8.487 -7.602 2.95628E-09 2.13067E-05 11.103 26 A_23_P134914 LY6H -2.266 8.190 -7.599 2.9859E-09 2.13067E-05 11.093 27 A_23_P79251 EHD3 -1.050 7.326 -7.589 3.08301E-09 2.13067E-05 11.063 28 A_23_P112397 FBXW5 -1.379 8.549 -7.553 3.44924E-09 2.29864E-05 10.956 29 A_23_P76983 C14orf45 0.537 7.522 7.521 3.81187E-09 2.4527E-05 10.861 30 A_23_P210400 KCNQ2 -1.121 8.191 -7.447 4.81339E-09 2.99389E-05 10.639 31 A_23_P98037 NM_020123 -1.095 10.712 -7.434 5.0127E-09 3.01728E-05 10.600 32 A_23_P36647 NM_177441 -0.542 8.388 -7.371 6.12498E-09 3.57158E-05 10.410 33 A_23_P106778 SULT1A2 -0.798 8.062 -7.333 6.90225E-09 3.90285E-05 10.296 34 A_23_P149470 NDUFS2 -2.350 8.418 -7.318 7.23347E-09 3.93263E-05 10.251 35 A_23_P102517 PDE6D -0.989 8.365 -7.309 7.45277E-09 3.93263E-05 10.223 36 A_23_P256282 BX537520 -0.413 7.375 -7.303 7.58718E-09 3.93263E-05 10.206 37 A_23_P26640 BC033681 -1.269 7.077 -7.291 7.87552E-09 3.97176E-05 10.170 38 A_23_P211247 HRMT1L1 -0.965 8.239 -7.272 8.35588E-09 4.10312E-05 10.114 39 A_23_P158265 AF410455 0.687 10.525 7.257 8.7733E-09 4.19763E-05 10.067 40 A_23_P53211 PLEKHB1 -1.090 7.073 -7.213 1.00731E-08 4.60692E-05 9.936 41 A_23_P259166 NM_024863 -0.872 12.476 -7.212 1.01225E-08 4.60692E-05 9.931 42 A_23_P28233 AF037332 -0.968 7.269 -7.195 1.06802E-08 4.745E-05 9.880 43 A_23_P157545 NKX3-1 0.356 8.371 7.173 1.14421E-08 4.96526E-05 9.814 44 A_23_P144563 GRIA2 -1.047 7.889 -7.137 1.28315E-08 5.44164E-05 9.705 45 A_23_P70045 H2AFY 0.689 11.036 7.117 1.36699E-08 5.66838E-05 9.645 46 A_23_P133232 HARS -0.773 7.806 -7.090 1.49151E-08 6.05026E-05 9.562 47 A_23_P218412 SPAG7 -1.324 7.836 -7.061 1.63459E-08 6.48958E-05 9.475 48 A_23_P140876 ABCA3 -1.365 9.112 -7.041 1.74216E-08 6.77255E-05 9.414
49 A_23_P91902 SMC4L1 0.752 9.701 7.017 1.8814E-08 7.16457E-05 9.340
Legenda: log2-fold-change (logFC), average log2-expression across all the arrays and channels (AveExpr), moderated t-statistic (t), raw p-value (P.Value), p-value adjusted for multiple testing (adj.P.Value)
according to Benjamini and Hocberg’s method, log odds that the gene is differentially expressed (B). Un’adeguata quantità di mRNA, disponibile per 5 di questi 9 geni identificati con il metodo “nearest shrunken centroid”, è stata testata mediante RT-PCR. Quattro geni di riferimento sono stati utilizzati per la normalizzazione, come indicato da Vandesompele et al. [33]. Sono state messe in evidenza significative differenze di espressione tra i due gruppi (glomi di basso e di alto grado) in 3 dei 5 geni testati: IGFBP2 (p<0,0005), CDC20 (p<0,0005) e TM4SF2 (p<0,025) valutati con test di Wilcoxon.
1.3.2 Risultati dell’analisi immunoistochimica
I risultati immunoistochimici dei casi esaminati con analisi microarray sono riportati nella Tabella 2.
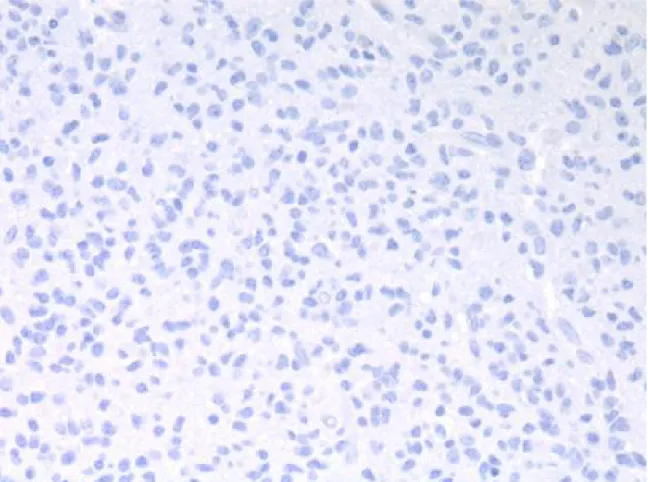
L'IGFBP-2 (considerando anche la serie di casi studiati solo con indagine di immunoistochimica) è risultato negativo in 19 gliomi di basso grado (figura 3). Figura 3. Oligodendroglioma, caso 30: le cellule sono risultate negative per IGFBP-2.
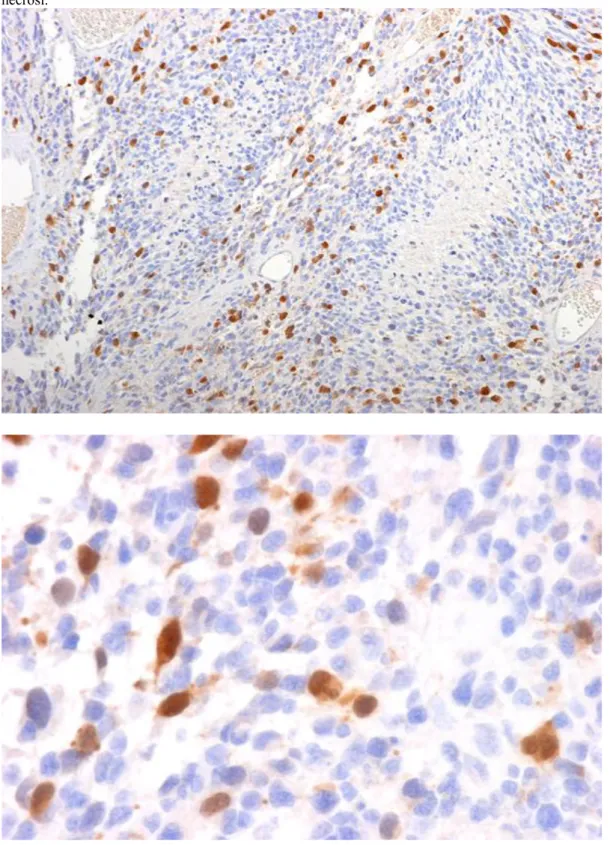
L'IGFBP2 era focalmente positivo (5% delle cellule) in 1 caso di PA (1/10=10%; 95%CI 0,3-44,5%) e in 1 AA su 10 gliomi grado III ( circa 20% di cellule positive). Al contrario è risultato positivo in 24 su 27 GL studiati (88,8%; 95%CI 69,7-95,1%); Fisher’s Exact Test: p=0,00002. L'immunopositività era citoplasmatica e nei GL appariva
significativamente più intensa nelle cellule adiacenti alle aree di necrosi a palizzata, mentre non sembrava correlata alla proliferazione microvascolare (Figure 4A e 4B). Figura 4A e B. Glioblastoma, caso 5: l'immunopositività per IGFBP-2 era citoplasmatica e maggiormente presente nelle cellule che palizzano attorno ai focolai di necrosi.
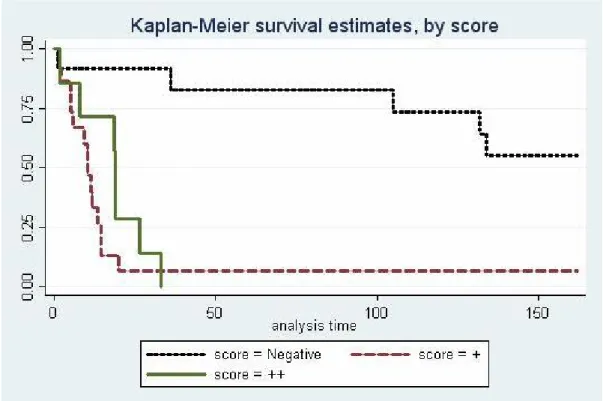
Per questo anticorpo, il log rank test risultava statisticamente significativo per gli score immunoistochimici (p<0,0001) (Figura 5).
Figura 5. Immunoespressione di IGFBP-2 correlata alla sopravvivenza.
La positività immunoistochimica di CDC-20 era nucleare e citoplasmatica. La
colorazione era negativa in tutti i 20 casi di glioma di basso grado (Fig. 6) con soltanto occasionali nuclei positivi in un A (FA34, meno del 5% di cellule positive).
Figura 6. Oligodendroglioma,caso 32: nessuna cellule era positiva per CDC-20.
Al contrario CDC-20 è risultato positivo in 3 AA su 10 gliomi grado III e in 20 su 27 GL (74,1%; 95%CI 57,5-90,6%); Fisher’s Exact Test: p<0,001 (Figure 7A e 7B).
Figura 7A e 7B. Glioblastoma, caso 19: A: Immunopositività nucleare per CDC-20. Le cellule positive sono prevalentemente localizzate attorno alle aree necrotiche. B: ma si osservano anche in aree prive di necrosi.
Per il CDC20, il log rank test è risultato statisticamente significativo per gli score immunoistochimici (p<0,0122).
Sono state evidenziate significative differenze nei livelli di espressione genica di IGFBP2 e CDC20 tra i corrispondenti gruppi immunoistochimici (p<0,001 per entrambi).
Inoltre come evidenziato nelle Tabelle 4A e 4B l'immunopositività di IGFBP2 e CDC20 nei GL è fortemente associata con l'indice di attività proliferativa (Ki67/Mib1).
Tabella 4A. Correlazione imunoistochimica tra IGFBP-2 e Ki67. GL: glioblastoma; (): tra parentesi il numero di casi.
GL (27) IGFBP-2 ++ (7) IGFBP-2 + (17) IGFBP-2 0 (3)
Ki67 medio 14,9% 19,7% 13,7% 11%
Tabella 4B. Correlazione imunoistochimica tra CDC20 e Ki67. GL: glioblastoma; (): tra parentesi il numero di casi.
GL (27) CDC20 ++ (5) CDC20 + (15) CDC20 0 (7)
Ki67 medio 14,9% 19,6% 15,3% 10,4%
Considerando i dati microarray relativi ai livelli di espressione, il log rank test è risultato statisticamente significativo per tutti i 9 geni selezionati (p<0,05). Inserendo gli stessi geni in un modello statistico di Cox, l'IGFBP-2 è emerso quale unico fattore indipendente predittivo di sopravvivenza.
1.4 DISCUSSIONE E CONCLUSIONI
Il presente studio di espressione genica dei gliomi umani ha messo in evidenza una condizione fortemente correlata (più del 95%) ai GL, rappresentata dalla iperespressione di Insulin-like growth factor binding protein 2 (IGFBP-2) e Cell division cycle 20 homolog (CDC 20). Questi dati sono stati convalidati da RT-qPCR ed
immunoistochimica.
L'IGFBP-2, membro di un'ampia famiglia di proteine, è normalmente espresso in cellule fetali ed è assente nella maggior parte dei tessuti adulti. Esso agisce non solo come vettore di Insulin-like growth factor (IGF), prolungandone l'emivita, ma anche come modulatore della disponibilità e dell'attività di IGF.
L'IGFBP-2 è iperespresso nei carcinomi dell'ovaio [34], del colon-retto [35], della prostata [36, 37] e del fegato [38] e nel neuroblastoma [39].
E' stato dimostrato che l'iperespressione di IGFBP-2 favorisce lo sviluppo e la
progressione del gliomi [40] e promuove la crescita invasiva dei GL [41]. Nei GL inoltre è stata osservata una correlazione positiva significativa tra l'espressione di IGFBP-2 e quella di MMP2 (metalloproteinase-2) [42] e VEGF (vascular-endothelial growth factor) [43]: ciò indica che l'espressione di IGFBP-2 è associata con pathways che promuovono l'invasività e l'angiogenesi.
Allo stesso modo dell'IGFBP-2, il CDC20 è stato trovato iperespresso nei gliomi di alto grado ed ipoespresso nei gliomi di basso grado ((p<0,0005), un risultato confermato anche dall'immunoistochimica. Questa molecola agisce come proteina di regolazione nel ciclo cellulare, in particolare è necessaria per due processi microtubulo-dipendenti, i movimenti che precedono l'anafase e la separazione dei cromosomi. Inoltre CDC20 è un fattore di regolazione di APC/C (anaphase promoting complex/cyclosome). I livelli di
CDC20 salgono nella mitosi e decrescono nella fase G1 iniziale. Il CDC20 è stato trovato iperespresso nei carcinomi dell'ovaio [44], dello stomaco [45], nei carcinoma squamoso del cavo orale [46] e nel carcinoma della mammella [47]. Recentemente è stato
dimostrato che il CDC20 è frequentemente up-regolato in molti tipi di neoplasia, inclusi i gliomi, e significativamente soppresso da p53 [48]. Dunque sembra che il CDC20
rappresenti un buon potenziale target terapeutico per un ampio spettro di neoplasie umane [48].
Nel presente studio sono state trovate differenze significative dei livelli di espressione genica di IGFBP-2 e CDC-20 tra i vari gruppi immunoistochimici (score 0, +, ++) (p<0,001 per entrambi).
L'IGFBP-2 ha immunocolorato 24 dei 27 GL e solo 1 dei 20 gliomi di basso grado. Allo stesso modo il CDC-20 era positivo in 20 dei 27 GL ed in 3 dei 10 gliomi grado III, mentre era negativo in tutti i 20 gliomi di basso grado.
Inoltre, come già osservato nel carcinoma gastrico [49], l'immunopositività di IGFBP2 e CDC20 nei GL è significativamente associata con l'indice di attività proliferativa (Ki67). Elmlinger et al [50] hanno dimostrato che le cellule gliomatose e le cellule
microcliali/macrofagiche IGFBP-2-positive si aggregano nelle immediate vicinanze delle aree di necrosi di gliomi umani e di GL di ratto e che l'iperespressione di IGFBP-2 correla positivamente con il grado del glioma.
La bassa percentuale di casi immunopositivi per IGFBP-2 e CDC-20 osservata tra i gliomi grado III può rappresentare un criterio diagnostico utile: può infatti essere un ausilio per ridurre la variabilità diagnostica inter-osservatore, particolarmente in piccole
Il TM4SF2 mostra un comportamento di espressione genica opposto rispetto a quello di IGFBP-2 e CDC20: è iperespresso nei gliomi di basso grado e ipoespresso in quelli di alto grado. Yokota et al. [51] hanno riportato risultati simili analizzando 11 GL (tutti ipoesprimevano il gene TM4SF2). Questo gene è associato a forme X-linked di ritardo mentale e malattie neuropsichiatriche come la corea di Huntington e la sindrome dell'X fragile [52]. La proteina codificata da TM4SF2, è un membro della “transmembrane 4 superfamily”, conosciuta anche con il nome di tetraspanina. La maggior parte di queste proteine sono proteine della superficie cellulare caratterizzate dalla presenza di 4 domini idrofobici. Queste proteine sono trasduttori di segnali che giocano un ruolo nella
regolazione dello sviluppo, della crescita e della mobilità cellulare.
In conclusione, i risultati osservati indicano che:
1- L'iperespressione genica di IGFBP-2 e CDC20 è fortemente correlata ai GL. Questi stessi geni in combinazione sono ipoespressi nei gliomi di basso grado, una differenza significativa che dimostra come la contemporanea iperespressione di IGFBP-2 e CDC20 è un segno di malignità nei gliomi.
2- L'espressione genica e proteica di IGFBP-2 e CDC-20 sono fortemente correlate, una relazione statisticamente significativa e probabilmente esclusiva dei gliomi maligni. 3- L'IGFBP-2 si è dimostrato il più importante fattore predittivo di sopravvivenza. 4- La positività immunoistochimica di IGFBP-2 e CDC-20 nelle cellule neoplastiche dei gliomi può essere utile per identificare i GL in piccole biopsie quando il tessuto è scarso. Dunque l'iperespressione di IGFBP-2 e CDC-20 potrebbe essere aggiunta agli abituali criteri istologici diagnostici come la necrosi e la proliferazione vascolare.
SEZIONE 2. Nogo-A: ruolo nella diagnosi di oligodendroglioma e nella
identificazione della codelezione 1p/19q
2.1.1 Olig-2
Olig-2 codifica per un fattore di trascrizione helix-loop-helix che gioca un ruolo critico nello sviluppo degli oligodendrociti [12].
I geni Olig-1 e Olig-2 sono stati descritti per la prima volta nel topo come fattori di trascrizione di tipo HLH che promuovono l’oligodendrogenesi e permangono espressi anche negli oligodendrociti maturi [53]. Topi knock-out per il gene Olig-2 mancano interamente del dominio dei precursori degli oligodendrociti nel tubo neurale.
2.1.2 Nogo-A
Nogo (Neurite outgrowth inhibitor) è una proteina che nell'uomo è codificata dal gene RTN4 ed è stata identificata come un inibitore della crescita neuritica specifico del sistema nervoso centrale. L'isoforma A del Nogo (Nogo-A) è un membro della famiglia dei reticuloni, fortemente espresso negli oligodendrociti e in misura minore nella mielina e nelle cellule neuronali [54, 55]. I reticuloni sono associati al reticolo endoplasmico e sono coinvolti nella secrezione neuroendocrina.
L'espressione di Nogo-A nei tumori gliali del sistema nervoso centrale è già stata descritta in circa l'80% degli OL and in poco più del 20% dei GL [13, 56].
2.1.3 Perdita cromosomica di 1p e 19q
Gli OL mostrano frequentemente la contemporanea perdita cromosomica di 1p e 19q (codelezione), una caratteristica che risulta predittiva di risposta alla chemioterapia. E’
inoltre un indice prognostico in quanto i casi di OL con codelezione hanno mostrato una sopravvivenza significativamente maggiore [14]. La codelezione è la conseguenza di una traslocazione non bilanciata tra il cromosoma 19 ed il cromosoma 1 [57, 58].
Attualmente l'identificazione della codelezione mediante analisi FISH (fluorescence in situ hybridization) rappresenta un dato estremamente importante nel trattamento dei pazienti con OL [59]. Tuttavia, soprattutto nei gliomi misti, i risultati della FISH possono essere influenzati dalla scelta delle aree neoplastiche sulle quali viene eseguito il test. Pertanto un marcatore immunoistochimico di differenziazione oligodendrogliale potrebbe essere utile sia per una corretta diagnosi istologica che per migliorare l'attendibilità dei risultati della FISH.
2.1.4 Obiettivo dello studio
L'obiettivo di questo studio è stato quello di verificare l'utilità di Nogo-A, Olig-2 e Sinaptofisina nel raggiungere una corretta diagnosi istologica di OL e di valutare un possibile ruolo complementare nella identificazione mediante FISH della codelezione 1p19q.
2.2 MATERIALI E METODI
2.2.1 Selezione dei casi
Centocinquantotto casi di neoplasie del sistema nervoso centrale, selezionati in maniera random dagli archivi della Sezione di Anatomia Patologica del Dipartimento di
Ematologia ed Oncologia dell'Università di Bologna, presso l'Ospedale Bellaria
(Bologna). I preparati istologici di tutti i casi sono stati rivisti e classificati seguendo la Classificazione WHO dei tumori del sistema nervoso centrale elaborata nel 2007 [2]. La serie di casi studiati includeva 24 OL, 23 oligodendrogliomi anaplastici (OL anaplastici), 2 OA, 2 oligoastrocitomi anaplastici (OA anaplastici), 30 GL, 2 A, 4 astrocitomi anaplastici (A anaplastici), 10 astrocitomi pilocitici (PA), 9 ependimomi, 12 ependimomi anaplastici, 10 neurocitomi centrali, 10 meningiomi, 10 papillomi dei plessi corioidei e 10 metastasi cerebrali di varie sedi primitive (1 melanoma, 3 carcinomi mammari, 1 carcinoma uroteliale, 1 adenocarcinoma gastrico, 2 carcinomi squamosi e 1 adenocarcinom del polmone, 1 carcinoma ovarico).
2.2.2 Analisi immunoistochimica
Sezioni standard di 4 μm, tagliate da inclusioni in paraffina selezionate, sono state
immunocolorate con 3 differenti anticorpi: 1) anti-Nogo-A (Neurite OutGrOwth inhibitor protein A) (Chemicon – Millipore Corporation, policlonale, diluizione 1: 500); 2) anti-Olig-2 (Oligodendrocyte transcription factor 2) (Chemicon – Millipore Corporation, policlonale, diluizione 1: 300); 3) anti-sinaptofisina (Neomarkers, policlonale, diluizione 1: 40).
Le reazioni immunoistochimiche sono state effettuate utilizzando l'UltraVision LP Large Volume Detection System HRP Polymer (Thermo Fisher Scientific, Fremont, CA, US) come descritto: la deparaffinazione e lo smascheramento antigenico sono stati ottenuti con W-CAP TEC BUFFER SOLUTION pH6 (Bio-Optica, Milano, Italia) a 95° per 25 minuti; l'inibizione delle perossidasi endogene è stata ottenuta in H2O2al 3%. Dopo aver
passato i vetrini in acqua distillata e in soluzione tampone (PBS-Tween 20, 1X, Bio-Optica, Milano), le sezioni sono state incubate in camera umida a temperatura ambiente per 5 minuti con soluzione Ultra V Block (Ultravision LP, LabVision Corporation). Successivamente le sezioni sono state lavate in soluzione tampone tre volte per 2 minuti, e incubate con l'anticorpo primario per 1 ora in camera umida a temperatura ambiente. Le sezioni sono state poi lavate in soluzione tampone e incubate con Primary Antibody Enhancer solution (Ultravision LP, LabVision Corporation) per 20 minuti in camera umida a temperatura ambiente. Dopo parecchi lavaggi in soluzione tampone, le sezioni sono state incubate per 30 minuti con HRP-polymer solution (Horse Radish Peroxides polymer, Ultravision LP, LabVision Corporation). Come sistema rivelatore è stata utilizzata la 3,3'-Diaminobenzidina (DAB) per 3 minuti.
La proteina Nogo-A è localizzata nel citoplasma; l'anticorpo mostra un pattern di immunoreattività finemente granulare e prevalentemente perinucleare. Olig-2 è una proteina nucleare, pertanto l'anticorpo colora i nuclei. In ciascun vetrino è stata aggiunta una sezione di un OL noto essere immunopositivo come controllo positivo. L'assenza dell'anticorpo primario è stata usata come controllo negativo. Il conteggio è stato
Score 1 (espressione debole): 5–24% di cellule positive; Score 2 (espressione moderata): 25–50% di cellule positive; Score 3 (espressione forte): > 50% di cellule positive.
Per propositi statistici sono stati considerati positivi i campioni che mostravano uno score 2 e 3.
2.2.3 Analisi FISH per la ricerca della delezione di 1p e 19q
L'analisi FISH era già stata effettuata prima della pianificazione di questo studio in 79 gliomi (OL, A, inclusi i GL, e gliomi misti). Otto casi erano stati esclusi in quanto il tessuto bioptico incluso in paraffina era scarso e dopo le colorazioni di routine, il tessuto neoplastico non era sufficiente per eseguire il test.
L'identificazione dello stato allelico di 1p/19q è stata ottenuta usando un'analisi FISH Dual-colour che è stata effettuata come descritto. Sezioni standard di 4 μm di tessuto fissato in formalina e incluso in paraffina sono state incubate a 56 °C per 2 ore,
deparaffinate mediante tre passaggi di lavaggio in Bioclear (Bio-Optica, Milano, Italia) di 15 minuti ciascuno, deidratate in due passaggi di 5 minuti in etanolo e lasciate asciugare all'aria a temperatura ambientale. Dopo incubazione in 2× SSC, pH 7,0 a 75 °C per 15 minuti, le sezioni sono state digerite con proteasi K (0,25 mg/mLin 2× SSC; pH 7.0) a 45°C per 15 minuti, sciacquate in 2× SSC(pH 7,0) a temperatura ambientale per 5 minuti e deidratate usando etanolo in una serie di concentrazioni crescenti (70%, 85%, 100%) per 2 minuti ciascuna. Le sonde Dual-Color Vysis LSI 1p36 / LSI 1q25 e LSI
19q13/19p13 (Vysis, Inc, Downers Grove, Illinois) sono state applicate sulle aree neoplastiche di ciascun vetrino, e l'area di ibridizzazione è stata coperta con un copri-oggetto e sigillata. I vetrini sono stati incubati a 75°C per 10 minuti al fine di consentire la
codenaturazione del DNA cromosomiale e di quello della sonda e successivamente a 37°C per 20 – 24 ore al fine di permettere la ibridizzazione. I lavaggi post-ibridizzazione sono stati effettuati in NP40 0,5%/ SSC 2X (pH 7,0 – 7,5) a 75°C per 2 minuti e in 2×SSC per 2 minuti a temperatura ambientale. Dopo che i campioni sono stati deidratati in etanolo come sopra, è stato applicato il DAPI (4 ′,6′-diamidino-2- phenylindole) diluito in Antifade 1µg/7ml (KREATECH Diagnostics, Amsterdam, Olanda) per colorare la cromatina. L'analisi FISH è stata effettuata usando un microscopio a fluorescenza Olympus BX61 (Olympus, Melville, New York, USA); in ciascun caso almeno 100 nuclei neoplastici sono stati contati, e il numero di copie di 1p36/1q25 e 19q13/19p25 (Fig. 1) è stato registrato per ogni nucleo.
Per designare un caso come deleto era necessario osservare un rapporto 1p36/1q25 < a 0,85 ed un rapporto 19q13/19p25 < a 0,90 [60].
In 31 casi, ovvero in quei gliomi risultati non deleti che all'esame immunoistochimico con anticorpo anti-Nogo-A mostravano una positività score 2 o 3, l'analisi FISH è stata
ripetuta, utilizzando le stesse sonde e gli stessi protocolli. In questo caso tuttavia le sonde Dual-Color Vysis LSI 1p36 / LSI 1q25 e LSI 19q13/19p13 sono state applicate su quelle aree neoplastiche che mostravano una maggiore immunopositività.
2.2.4 Analisi dei dati
Le differenze di espressione immunoistochimica sono state analizzate utilizzando il test del chi-quadrato. La correlazione della immunopositività del Nogo-A con lo stato allelico di 1p/19q è stata analizzata per mezzo del t test di Student. Un valore di p < 0,05 è stato adottato come livello di significatività.
2.3 RISULTATI
2.3.1 Risultati dell'analisi immunoistochimica
I risultati dell'analisi immunoistochimica maggiormente rilevanti sono riportati nella Tabella 1.
Tabella 1. Risultati dell'analisi immunoistochimica
Nogo-A Olig-2 Sinapto
Oligo Astro Misti Oligo Astro Misti Oligo Astro Misti Score 0 3 (6,4%) 19(52,8%) 0 1(2,1%) 4(11,1%) 0 12(25,6%) 28(77,8%) 1(25%) Score 1 8 (17%) 10(27,8%) 1(25%) 4(8,5%) 5(13,9%) 0 11(23,4%) 5(13,9%) 2(50%) Score 2 20 (42,6%) 4(11,1%) 2(50%) 19(40,5%) (33,3%)12 2(50%) 18(38,3%) 3(8,3%) 1(25%) Score 3 16 (34%) 3(8,3%) 1(25%) 23(48,9%) 15(41,7%) 2(50%) 6(12,7%) 0 0 Positivi 36/47 76,6% 7/3619,4% 3/475% 42/4789,4% 75%27/36 4/4100% 24/4751% 3/368,3% 1/425% Legenda: Sinapto: sinaptofisina; Oligo: oligodendrogliomi grado II e III; Astro: tumori astrocitari grado II, III, IV; Misti: gliomi misti.
Una immunopositività moderata o forte (scores 2 e 3) del Nogo-A è stata rilevata in 18/24 (75%) casi di OL (Fig. 2A), in 18/23 (78,2%) OL anaplastici (Fig. 2B) e in 2/2 (100%) OA. AL contrario le percentuali di casi positivi decrescono fortemente negli altri tipi di tumori; sono risultati positivi 1/2 (50%) OA anaplastici, 6/30 (20%) GL, 1/4 (25%) A anaplastici, 2/10 (20%) PA, 1/12 (8,3%) ependimoma anaplastico ed 1/10 (10%) neurocitoma centrale.
Figure 2A: un caso di OL con positività citoplasmatica perinucleare per Nogo-A.
Figura 2B: un caso di OL anaplastico con positività citoplasmatica diffusa per Nogo-A.
L'Olig-2 ha mostrato una positività moderata o forte (scores 2 e 3) in 22/24 (91,6%) OL, in 20/23 (86,9%) OL anaplastici (Fig. 3A), in 2/2 (100%) OA e in 2/2 (100%) OA anaplastici. Tuttavia esso è risultato positivo anche in 24/30 (80%) GL (Fig. 3B e C), in 2/4 (50%) A anaplastici, in 1/2 (50%) A, in 8/10 (80%) PA (Fig. 3D) e in 1/12 (8,3%) ependimoma anaplastico.
Figure 3B e C: due casi di GL intensamente positivi con Olig-2. Come si può osservare nella Figura 3B le cellule neoplastiche localizzate attorno ai focolai di necrosi risultano meno positive.
Figura 3D: un caso di PA immunocolorato con Olig-2.
La positività nucleare è risultata generalmente intensa ed un'ampia percentuale di casi sono stati giudicati score 3.
La Sinaptofisina ha mostrato una positività moderata o forte (scores 2 e 3) in 13/24 (54,1%) casi di OL, in 11/23 (47,8%) OL anaplastici, in 1/2 (50%) OA, in 3/30 (10%) GL, in 1/10 (10%) PA, in 10/10 (100%) neurocitomi centrali, in 1/12 (8,3%) ependimoma anaplastico e in 7/10 (70%) casi di papilloma dei plessi corioidei..
Le lesioni metastatiche (10 casi) erano completamente negative con tutti i 3 marcatori utilizzati.
2.3.2 Analisi statistica dei dati immunoistochimici
La sensibilità e specificità di Nogo-A nella diagnosi differenziale tra OL e tumori astrocitari sono risultate rispettivamente il 76,6% e l'80,6%.
Al contrario la specificità di Olig-2 per i tumori oligodendrogliali era solo il 25%, anche se la sensibilità era l’89,4%. Riguardo la Sinaptofisina, questo marcatore ha mostrato una specificità del 91,7%, ma la sensibilità negli oligodendrogliomi è risultata bassa (51%). Le differenze di espressione immunoistochimica di Nogo-A tra tumori oligodendrogliali e astrocitari è risultata statisticamente significativa (P = 0,001), pertanto il Nogo-A è stato scelto come marcatore di riferimento per selezionare le aree su cui ripetere l'analisi FISH.
2.3.3 Risultati dell'analisi FISH.
L'analisi FISH originale (eseguita in 79 casi) aveva messo in evidenza una perdita
combinata di 1p e 19q in 21 casi (26,6%), specificatamente in 12/23 (52,2%) OL, in 8/21 (38%) OL anaplastici e in 1/25 (4%) GL (Tabella 2A).
Tabella 2A. Risultati dell'analisi FISH eseguita prima della pianificazione dello studio Codelezione 1p/19q Delezione assente o solo parziale Score
Nogo-A 0 1 2 3 0 1 2 3
Casi 2/21
(9,5%) (14,3%)3/21 (28,6%)6/21 (47,6%)10/21 (24,2%)14/58 (22,4%)13/58 (31%)18/58 (22,4%)13/58
Dei 21 gliomi con codelezione di 1p/19q, 16 (76%) mostravano un'immunoespressione di Nogo-A moderata o forte (score 2 e 3), mentre 5 casi erano negativi: questa differenza non è risultata statisticamente significativa (P = 0,069) (Tabella 2B).
Dei 47 gliomi immunopositivi per Nogo-A, 16 casi erano codeleti, mentre 31 casi non mostravano la perdita contemporanea di 1p e 19q: dunque la positività del Nogo-A non correlava con lo stato allelico di 1p/19 (P = 0,997) (Tabella 2C).
Tabella 2C. Correlazione tra immunopositività di Nogo-A e codelezione. Casi immunopositivi (score 2-3)
Casi codeleti 16 Casi non codeleti 31 P = 0,997
Pertanto la positività immunoistochimica di Nogo-A non può essere considerato un fattore capace di predire lo stato allelico di 1p/19.
L'analisi FISH è stata ripetuta in 31 gliomi non deleti con una positività per Nogo-A moderata o forte (score 2 o 3). Questa volta l'indagine è stata effettuata nelle aree maggiormente immunopositive per Nogo-A (vedi Fig. 4A, 4B e 4C).
Figure 4A: un caso di OL anaplastico con immunopositività per Nogo-A moderata, ma con una distribuzione disomogenea.
Figura 4B. Maggiore ingrandimento dell'angolo superiore destro della Fig. 5A; questa area è stata giudicata non idonea per ripetere l'analisi FISH.
Figura 4C. Maggiore ingrandimento dell'angolo inferiore sinistro della Fig. 5A; questa area, maggiormente immunopositiva per Nogo-A, è stata selezionata per ripetere l'analisi FISH.
Adottando questa tecnica la codelezione di 1p/19q è stata evidenziata in 3 ulteriori OL (la percentuale di casi codeleti è salita da 52,2% a 65,2%), in altri 2 casi di OL anaplastici (da 38% a 47,6%), in 1 caso di OA, in 1 caso di OA anaplastico ed in 1 ulteriore caso di GL (da 4% a 8%). Questi 8 nuovi casi riconosciuti come codeleti avevano mostrato un'immunopositività per Nogo-A moderata in 7 casi e forte in 1 caso (vedi Tabella 3A).
Tabella 3A. Risultati dell'analisi FISH dopo aver ripetuto l'esame sulle aree maggiormente positive per Nogo-A
Codelezione 1p/19q Delezione assente o solo parziale Score
Nogo-A 0 1 2 3 0 1 2 3
Casi 2/29
(6,9%) (10,3%)3/29 (44,8%)13/29 (38%)11/29 (28%)14/50 (26%)13/50 (22%)11/50 (24%)12/50
Alla luce di queste modifiche, tra i 29 gliomi codeleti ben 24 (82,7%) mostravano una positività del Nogo-A moderata o forte (score 2 e 3), mentre 5 casi erano negativi: questa differenza è risultata statisticamente significativa (P = 0,001) (Tabella 3B).
Tabella 3B. Differenze nell'espressione immunoistochimica dei casi codeleti.
Casi immunopositivi (score 2-3) Casi immunonegativi (score 0-1)
Casi codeleti 24 5
P = 0,001
Tuttavia dei 47 gliomi immunopositivi per Nogo-A, 24 casi erano codeleti, mentre 23 non mostravano la codelezione: dunque la correlazione tra positività immunoistochimica di Nogo-A e stato allelico di 1p/19q è rimasta non significativa (P = 0,999) (Tab. 3C).
Tabella 3C. Correlazione tra immunopositività di Nogo-A e codelezione. Casi immunopositivi (score 2-3)
Casi codeleti 24 Casi non codeleti 23 P = 0,999
2.4. DISCUSSIONE E CONCLUSIONI
Sono stati fatti molti tentativi di identificare marcatori immunoistochimici utili nella diagnosi differenziale tra OL e A. In effetti un marcatore immunoistochimico di
differenziazione oligodendrogliale, se attendibile, sarebbe di aiuto non solo nella diagnosi istologica ma anche nelle indagini FISH.
I risultati di questo studio confermano che il Nogo-A colora le cellule neoplastiche oligodendrogliali con un'elevata sensibilità e specificità (rispettivamente 76,6% e 80,6%). L'anticorpo Olig-2 colora la maggioranza degli oligodendrogliomi, mostrando un’elevata sensibilità (89,4%), ma anche una larga fetta di tumori astrocitari, compresi i PA; pertanto la specificità è molto bassa (25%). Nonostante questo anticorpo inizialmente fosse
considerato un marcatore specifico di neoplasie oligodendrogliali [12], vari studi hanno già osservato che l'Olig-2 è universalmente espresso nei gliomi [13, 56, 61].
In effetti irisultati osservati sono in linea con i dati precedentemente pubblicati.
Kuhlmann et al [13] hanno osservato l’immunopositività del Nogo-A nel 71% dei tumori oligodendrogliali (76,6% nella nostra serie) e nel 24% dei GL (20% nella nostra serie). Jung et al [56] hanno dimostrato valori di sensibilità e specificità rispettivamente dell’86,9% e 57,1% (76,6% e 80,6% nel presente studio). Infine, come osservato in questo lavoro, già altri studi hanno osservato che l’OLIG2 è universalmente espresso nei gliomi diffusi [61].
La Sinaptofisina mostra una bassa sensibilità (51%) negli oligodendrogliomi; inoltre essa non permette di differenziare gli oligodendrogliomi dai neurocitomi.
E' noto che la delezione combinata di 1p/19q si associa ad un quadro istologico classico di oligodendroglioma e rappresenta un utile marcatore prognostico [60, 61]: in effetti sono state osservate significative differenze di sopravvivenza tra il gruppo dei casi “codeleti” (>123 mesi) ed il gruppo dei casi “non codeleti” (16 mesi) [62, 63]. Attualmente il trattamento standard di questi pazienti non può prescindere
dall'identificazione dello stato di 1p e 19q ed è estremamente importante riconoscere correttamente questo marcatore di chemosensibilità e di maggiore sopravvivenza. Nella nostra serie 47 dei 79 casi esaminati con analisi FISH risultavano positivi con Nogo-A (Score 2 e 3); di questi 47 casi positivi, 24 mostravano codelezione di 1p/19q, mentre gli altri 23 non avevano la codelezione. Dunque non si osserva una correlazione tra la positività di Nogo-A e lo stato allelico di 1p/19q (P = 0,997). Questi risultati
mettono in evidenza che l'analisi immunoistochimica con anticorpo anti-Nogo-A non può sostituire l'analisi FISH nel determinare lo stato allelico di 1p/19q. E' un fenomeno simile a quello osservato per lo stato di MGMT (O6-methylguanine-DNA methyltransferase) nei GL, dove la PCR metil-specifica continua ad essere preferita all'immunoistochimica [64, 65].
Tuttavia l'attendibilità dei risultati della FISH può essere condizionata dalle aree selezionate per eseguire l'analisi. Dunque nel presente lavoro, in considerazione dei risultati dell’immunoistochimica, l'analisi FISH è stata ripetuta sotto la guida del Nogo-A in quei gliomi “non deleti”, con uno score 2 o 3: l'esame è stato eseguito in quelle aree in cui il Nogo-A era risultato maggiormente positivo.
conseguenze che tale caratteristica biomolecolare implica nel trattamento di questi pazienti.
Inoltre nell'ambito dei casi codeleti (compresi i nuovi casi deleti individuati) il numero di casi immunopositivi è significativamente maggiore di quello dei casi negativi: 24 dei 29 casi codeleti sono risultati immunopositivi per Nogo-A (P = 0,001). Questi risultati suggeriscono che la FISH non può essere sostituita dall'immunoistochimica nell'analisi dello stato allelico di 1p/19q, ma se il test viene eseguito selezionando le aree
neoplastiche più immunopositive, è possibile evidenziare un numero maggiore di casi codeleti. Questa considerazione appare particolarmente significativa in quei casi con una immunopositività moderata e disomogenea (7 degli 8 casi riconosciuti come codeleti dopo FISH Nogo-A guidata, mostravano una immunopositività Score 2), in quanto a dispetto di un quadro morfologico oligodendrogliale omogeneo, l'immunoistochimica può mostrare una significativa variabilità da un'area all'altra.
In conclusione il presente studio
1) conferma che il Nogo-A è il marcatore istologico più utile e specifico nella diagnosi differenziale tra oligodendrogliomi e tumori astrocitari;
2) suggerisce che l’immunoistochimica con anticorpo anti-Nogo-A non può sostituire la FISH per determinare lo stato allelico di 1p/19q;
3) mette in evidenza che la sensibilità dell’esame FISH migliora quando questa analisi viene eseguita sulle aree neoplastiche maggiormente immunopositive per Nogo-A.
3. miRNA nei glioblastomi
3.1.1 miRNA
I miRNA (o microRNA) sono piccole molecole di RNA non codificanti in grado di regolare l’espressione genica agendo a livello post-trascrizionale.
La loro funzione è costituita dal silenziamento post-trascrizionale di RNA messaggeri (RNAm) target, attraverso l’inibizione della traduzione o la degradazione del RNAm bersaglio, modulando in tal modo un ampio spettro di processi fisiologici.
I miRNA regolano l’espressione genica utilizzando la rete di meccanismi cellulari coinvolti nel fenomeno dell’RNA interference, svolgendo funzioni endogene una volta attribuite esclusivamente agli small interfering RNA (sRNA).
I trascritti primari dei miRNA sono molecole lunghe anche alcune centinaia di basi, mentre i miRNA maturi, attivi, sono molecole a singolo filamento di 19-24 nucleotidi [66]. Ad oggi per il genoma umano sono stati già annotati più di 1000 miRNA nel database denominato miRBase (http://microrna.sanger.ac.uk/sequences/) e si stima che fino ad un terzo degli RNAm umani codificanti siano soggetti a questa rete di regolazione [67]. Un unico miRNA può modulare centinaia di targets differenti, mentre ciascun RNAm può essere regolato da uno o più miRNA [68, 69]
3.1.2 Modulazione della traduzione
Geni chiave, o essenziali, per una determinata funzione cellulare potrebbero essere fortemente trascritti ma tradotti in misura decisamente minore. Questo effetto sarebbe dovuto proprio alla modulazione negativa della traduzione ad opera dei miRNA.
In tal modo la cellula può disporre di un notevole numero di RNAm ma, allo stesso tempo, è in grado di controllare la produzione delle proteine. I miRNA possono garantire questa modulazione senza interferire con l’attività trascrizionale o con i processi di maturazione degli RNAm.
L’azione dei miRNA, se da un lato può inibire la traduzione di una determinata proteina, dall’altro può, nel contempo, portare all’aumento di espressione di un gene qualora l’azione del miRNA comporta l’inibizione di una proteina con azione regolatoria negativa.
3.1.3 miRNA nelle neoplasie umane
Diversi meccanismi possono condurre ad un’alterata espressione dei miRNA nelle neoplasie umane, sebbene la maggior parte degli studi abbiano identificato le aberrazioni cromosomiche come causa primaria.
L’espressione aberrante dei miRNA nei tumori umani è stata descritta per la prima volta nella leucemia linfatica cronica B (B-CLL), in cui la delezione, in omozigosi o
eterozigosi, a livello del locus 13q14 comporta la perdita di espressione di 15 e miR-16 [70]. Da allora un’espressione alterata dei miRNA è stata descritta in numerose
neoplasie, comprese quelle di colon [71], mammella [72], polmone [73], fegato [74] e linfomi [75].
Inoltre in diverse neoplasie è stata osservata un’elevata frequenza di alterazioni a livello di loci comprendenti regioni codificanti per miRNA.
Infine circa il 50% dei geni miRNA sono associati con isole CpG e quindi rappresentano possibili target per il sistema di metilazione del DNA [76].
3.1.4 Ruolo dei miRNA nell’oncogenesi e nella target-therapy
Il ruolo dei miRNA nello sviluppo delle neoplasie può essere spiegato con l’alterazione della regolazione dell’attività di oncogeni o oncosoppressori da parte di queste molecole. Ad esempio il target di miR-15a e miR-16-1 è stato identificato nella molecola Bcl-2 [79], l’archetipo dei geni antiapoptotici, la cui attivazione è considerata essere cruciale per i processi oncogenici di linfomi, leucemie e carcinomi del polmone. La perdita dei miRNA connessi con questo gene correla con l’overespressione di Bcl-2.
I miRNA costituiscono un possibile obiettivo per la target-therapy, funzionante su una rete di geni modulati da miRNA. In effetti il fenotipo fisiologico di una cellula viene modulato dall’attività di un limitato numero di miRNA.
Sebbene esistano già in letteratura studi che si sono proposti di tipizzare diverse neoplasie sulla base dell’espressione caratteristica di miRNA, delineando una vera e propria firma (signature) genetica, ancora poco si conosce sulla loro effettiva espressione nei tumori solidi.
Le informazioni ottenute possono rivelarsi utili per una migliore classificazione e tipizzazione dei tumori, permettendo inoltre una più accurata conoscenza dei processi neoplastici di trasformazione e oncogenesi.
3.1.5 Studi concernenti i miRNA nei glioblastomi
Nei GL in tre diversi studi indipendenti [21, 22, 23] sono stati identificati 17 miRNA overespressi e 33 miRNA ipoespressi (vedi Tabella 1).
In particolare miR-10b, miR-21, miR-124, miR-128-1, miR-137, miR-139, miR-218 e miR-323 sono stati trovati deregolati in almeno due studi. Livelli elevati di of miR-451 e miR-486 sono stati osservati nei GL primitivi [21].
Inoltre miR-21 si è rivelato uno dei miR più comunemente iperespresso nei GL, con una percentuale variabile tra il 44 ed il 100% dei casi che mostrano tale aberrazione [21, 23]. Tabella 1. miRNA alterati individuati nei GL in tre diversi studi indipendenti.
3.1.6 Obiettivo dello studio
Obiettivo di questo studio è stato quello di delineare un profilo di espressione dei miRNA nei GL e di paragonarlo con quello ottenuto da tessuto cerebrale non neoplastico. Inoltre è stato effettuato un confronto dei risultati ottenuti da tessuto congelato con quelli osservati su tessuto paraffinato. Lo studio non è stato ancora completato pertanto i risultati sono ancora parziali.
3.2 Materiali e Metodi
3.2.1 Selezione dei casi e preparazione dei campioni
Per questo studio sono stati arruolati 40 casi di GL operati presso l’Ospedale Bellaria di Bologna. Sono stati inoltre collezionati 10 campioni di tessuto cerebrale non neoplastico: in 5 casi tale materiale è stato ottenuto da pezzi chirurgici asportati per neoplasia, prelevando a distanza dal tumore tessuto macroscopicamente ed istologicamente indenne; nei restanti 5 casi il materiale è stato prelevato da tessuto corticale del lobo temporale asportato in pazienti operati per epilessia farmaco-resistente.
Tutti i casi sono stati ricevuti non fissati nel laboratorio della Sezione di Anatomia Patologica del Dipartimento di Ematologia ed Oncologia dell'Università di Bologna, presso l'Ospedale Bellaria (Bologna). Da ciascun paziente è stato ottenuto il consenso informato.
I campioni sono stati divisi in due porzioni: una parte è stata fissata in formalina, inclusa in paraffina e colorata con ematossilina/cosina (EE) per la diagnosi istologica di routine. Una parte è stata trattata per l’estrazione dei miRNA da tessuto non fissato. Su questo materiale è stata effettuata una sezione congelata, per valutare la significatività del campione, verificando la presenza di cellule neoplastiche; il campione è stato quindi incubato in RNA-later (Ambion, Austin, TX, USA) per un’ora a temperatura ambiente e infine congelato a -80°C.
Dal frammento fissato in formalina e incluso in paraffina (FFPE), utilizzato per le indagini di routine, sono state tagliate 4 sezioni consecutive da 20m ed una sezione da 4m colorata con EE. Mediante l’esame microscopico della sezione colorata con EE è stata selezionata la porzione di tessuto da esaminare, escludendo aree di necrosi o ricche
di infiltrato linfocitario. Quindi è stata effettuata la dissezione, tramite lama, della porzione selezionata.
3.2.2 Selezione dei miRNA
Sono stati presi in esame 19 miRNA (tabella 2), già segnalati in precedenti lavori su neoplasie cerebrali, scelti in base a funzione e possibili target. Oltre a questi 19 miRNA, sono stati selezionati 2 ulteriori miRNA (miR103 e miR191) e 2 sRNA (RNU49 e U54) che sono stati utilizzati come controlli di reazione (House-Keeping genes).
Tabella 2. Elenco dei miRNA e loro localizzazione cromosomica.
miRNA Localizzazione miR7 9q21.32 miR9 1q22 miR9* 1q22 miR10a 17q21.32 miR10b 2q31.1 miR15b 3q25.33 miR17 13q31.3 miR19a 13q31.3 miR20a 13q31.3 miR21 17q21.31 miR26a 3p22.2 miR27a 19p13.13 miR31 9p21.3 miR34a 1p36.22 miR92a 13q31.3 miR101 1p31.3 miR124 8p23.1 miR128 2q21.3 miR137 1p21.3 miR148a 7p15.2 miR182 7q32.2 miR184 15q25.1 miR221 Xp11.3
3.2.3 Disegno dei primers
Per poter selezionare specificatamente ogni miRNA sono stati disegnati 23 primers, ognuno complementare alla sequenza del miRNA da riconoscere. Per aumentare la specificità della reazione, esigenza dettata dal fatto che diversi miRNA possono differire tra loro anche per una singola base, a livello di tali nucleotidi “discriminanti” sono stati aggiunti degli LNA (Locked Nucleic Acid). Tali LNA sono dotati di un anello pirrolico che permette di aumentare la specificità del legame con la relativa base. L’efficienza dei primers è stata testata utilizzando diluizioni scalari di un pool di linee cellulari (MCF7 – carcinoma mammario; U87MG – GL; LNCaP – carcinoma prostatico).
3.2.4 Estrazione dei miRNA
Dal tessuto non fissato i miRNA sono stati estratti utilizzando il kit miRvana (Ambion, Austin, TX, USA). E’ stata adottata la procedura di arricchimento che permette di isolare la componente dei miRNA dall’RNA totale, secondo le indicazioni del protocollo. La quantità e la qualità dei miRNA così ottenuti è stata valutata tramite Bioanayzer 2100 (Agilent, Waldbronn, Germany) come mostrato in figura 1.
Figura 1. Profili ottenuti tramite Bioanalyzer 2100. A) Picco di fluorescenza e concentrazione corrispondente alla porzione arricchita in miRNA. B) Picchi di fluorescenza (subunità 18s e 28s) e concentrazione della frazione di RNA (privo di miRNA).
Per l’estrazione di miRNA da tessuto FFPE è stato utilizzato il kit RecoverAll (Ambion, Austin, TX, USA) e la quantità di RNA totale così ottenuto è stato valutato tramite fluorometro (Quant-iT RNA Assay kit Invitrogen, Oregon, USA).
3.2.5 qRT-PCR
I miRNA sono stati retrotrascritti utilizzando il kit NCODETM miRNA (Invitrogen,
Carlsbad, CA, USA), secondo le indicazioni del protocollo. Brevemente, viene effettuata una poliadenilazione dei miRNA, che servirà per fornire l’innesco al primer universale usato per la retrotrascrizione. Il cDNA così ottenuto viene amplificato utilizzando come primer forward il primer specifico per ogni singolo miRNA (disegnato come descritto in precedenza) e come primer reverse il primer universale. La reazione è stata effettuata utilizzando una Real-Time AB 7000.
3.2.6 Analisi Statistica
I risultati sono stati analizzati tramite i software per l’analisi di dati di Real-Time qBase (Biogazelle) e Data Assist (AB analytics). Le analisi statistiche sono state effettuate tramite il sotware GraphPad Prism.
3.3 RISULTATI
3.3.1 Confronto tra i risultati su campioni FFPE e quelli su campioni non fissati Abbiamo confrontato i profili d’espressione dei miRNA ottenuti dai tessuti non fissati con quelli ottenuti dai campioni FFPE. In tal modo abbiamo voluto verificare l’importanza della rappresentatività del tessuto esaminato nell’analisi dei miRNA. In effetti, nonostante venga effettuata una sezione congelata per valutare la composizione del tessuto in esame, quando si estrae da tessuto non fissato è più difficile determinare la percentuale di cellule neoplastiche rispetto al background di cellule non tumorali. La presenza di questi
“contaminanti” può portare a dei risultati che risentono dell’effetto del profilo di espressione dei miRNA presenti nelle cellule non neoplastiche.
Ne è prova il fatto che paragonando i livelli di espressione dei miRNA tra le due metodiche, i dati ottenuti non appaiono sovrapponibili (figure 2 e 3).
Figura 2. Istogramma raffigurante il fold change dell’espressione di ogni singolo miRNA nei
campioni FFPE e negli stessi campioni non fissati. Si può notare come vi siano notevoli differenze nei profili d’espressione, nonostante i pazienti in esame siano gli stessi.
Figura 3. Heat-map (Pearson’s correlation) dei singoli campioni FFPE (contrassegnati da una sigla con acronimo FFPE) e non fissati. I tessuti provenienti dagli stessi pazienti sono indicati con identici colori. E’ possibile osservare come gli stessi campioni clusterizzino differentemente in base al profilo d’espressione dei miRNA, a seconda che siano fissati o meno.
Tali discrepanze possono essere legate alla presenza di cellule “non neoplastiche” nel campione non fissato: si ottiene dunque un risultato non corrispondente al profilo del tumore stesso, ma altresì mescolato con il profilo d’espressione delle cellule non tumorali presenti nel campione, ad esempio i linfociti.
Alla luce di tali considerazioni per la determinazione del profilo di espressione dei miRNA nei GL ed il confronto dei risultati ottenuti con quelli di campioni non neoplastici, sono stati presi in considerazione i profili ottenuti da campioni FFPE. I risultati finora valutati si riferiscono a 15 GL e 5 casi non neoplastici.
3.3.2 miRNA nei glioblastomi
Nel confronto del profilo dei miRNA ottenuto dai GL con quello osservato in campioni non neoplastici abbiamo rilevato che alcuni miR sono sovraespressi (ad esempio miR10b, miR21 e miR27a) mentre altri downregolati (miR7, miR137) (vedi Tabella 3 e Figura 4).
Tabella 3. Fold-change dell’espressione di ogni miRNA nei GL rispetto al tessuto non neoplastico.
miR Fold Change
miR101 11,37 miR10a 25,31 miR10b 83,24 miR137 -6,12 miR17 11,67 miR182 20,32 miR20a 5,48 miR21 63,43 miR221 1,50 miR222 -1,07 miR26a 2,77 miR27a 24,22 miR31 1,56 miR330 2,34 miR34a 9,75 miR519d 1,83 miR7 -5,41 miR9 11,92 miR9* 13,06
Figura 4. Istogramma mostrante il Fold-change dell’espressione di ogni miRNA nei GL rispetto al tessuto non neoplastico
La clusterizzazione dei campioni mostra come i GL si raggruppino indipendentemente dai campioni normali (Figura 5).
Figura 5. In questa Heat-map (Pearson’s correlation) si può notare come i campioni di GL (contrassegnati da una sigla ROSSA) clusterizzino indipendentemente rispetto ai campioni non neoplastici (contrassegnati da una sigla BLU), in base al profilo di espressione dei miRNA.