C
C
a
a
p
p
i
i
t
t
o
o
l
l
o
o
I
I
I
I
I
I
Metodi di Analisi di Connettività
Funzionale ed Effettiva
III.1 Neuroimaging tramite tecnica fMRI
Attualmente esistono numerose e differenti tecniche non invasive di imaging cerebrale in grado di fornire mappe dell’attività funzionale del cervello umano in risposta all’esecuzione di determinati task.
Esse si basano su principi fisici diversi e indagano aspetti diversi dell’attività cerebrale, fornendo informazioni spesso complementari sull’attività neuronale: la fMRI (functional Magnetic Resonance Imaging) misura le risposte emodinamiche, la PET (Positron Emission Tomography) rileva l’attività metabolica mentre altre tecniche come MEG e EEG (magnetoencefalografia e elettroencefalografia) sfruttano la rilevazione dei segnali elettromagnetici prodotti dall’organismo.
Queste sono le tecniche più diffuse ed utilizzate nell’ambito del neuroimaging e tutte quante sono ottimi strumenti di indagine, tuttavia occorre sottolineare che oltre a cogliere aspetti diversi dell’attività neurale esse hanno anche delle
caratteristiche di risoluzione (sia spaziale che temporale) abbastanza differenti l’una dall’altra, perciò spesso capita che, in particolari situazioni di indagine, a seconda degli obiettivi perseguiti, l’una sia più efficace dell’altra o riesca meglio ad individuare le regioni cerebrali d’interesse.
In questo senso per le tecniche come la PET o la SPECT la risoluzione temporale è più bassa rispetto alle altre e si aggira intorno alle centinaia di secondi [9]. Ciò è dovuto al fatto che occorre trovare una via di mezzo tra il tempo di acquisizione e la quantità di radioisotopo somministrata al paziente, da cui dipende la risoluzione spaziale dell’indagine (nelle macchine più evolute può arrivare intorno ai 5 mm nominale [9] che si traduce in pratica in range di variazione superiore a 19 mm in piano e 8 mm in assiale). Questo è un po’ il limite dell’indagine PET: non è possibile aumentare a piacere la dose di radioattività assorbita dal paziente perché ne va della sua salute.
Le cose cambiano con le tecniche fMRI o EEG o MEG; infatti in questi casi non sono riscontrate controindicazioni per la salute del soggetto e quindi tali metodologie sono molto più utilizzate, rispetto alla PET, nell’ambito della ricerca scientifica. Nella fMRI si raggiungono risoluzioni spaziali abbastanza buone (1.5-3 mm) a scapito di quelle temporali che si aggirano sull’ordine della decina di secondi [9]. Dall’altra parte le indagini EEG e MEG hanno una altissima risoluzione temporale, sull’ordine del millisecondo, ma hanno una scarsa risoluzione spaziale (2-3 mm sulla superficie e più di 10 mm in direzione perpendicolare alla superficie) in dipendenza del numero di elettrodi applicati sulla superficie cranica [9]; tuttavia non si riesce con questa tecnica a distinguere la posizione di sorgenti profonde all’interno del volume poiché i segnali acquisiti sulla superficie sono la sommatoria dell’attività di regioni interne sovrapposte. E’ per questo che principalmente vengono impiegati per lo studio dei processi della corteccia esterna.
La prospettiva futura è quella di riuscire ad ottenere un tipo di indagine che permetta una elevata risoluzione sia spaziale che temporale. In quest’ottica sono stati avviati negli ultimi anni molti studi [9] che cercano di integrare queste due
tecniche di indagine con lo scopo di avere una buona risoluzione temporale attraverso la EEG ed una collocazione spaziale precisa grazie alla fMRI o PET.
III.1.1 Cenni di Risonanza Magnetica (MRI)
Il principio fisico che consente la formazione di immagini di risonanza magnetica è la Risonanza Magnetica Nucleare (NMR), che è stata studiata a partire dagli anni ’40 ed è stata impiegata per molti anni come strumento di analisi chimica.
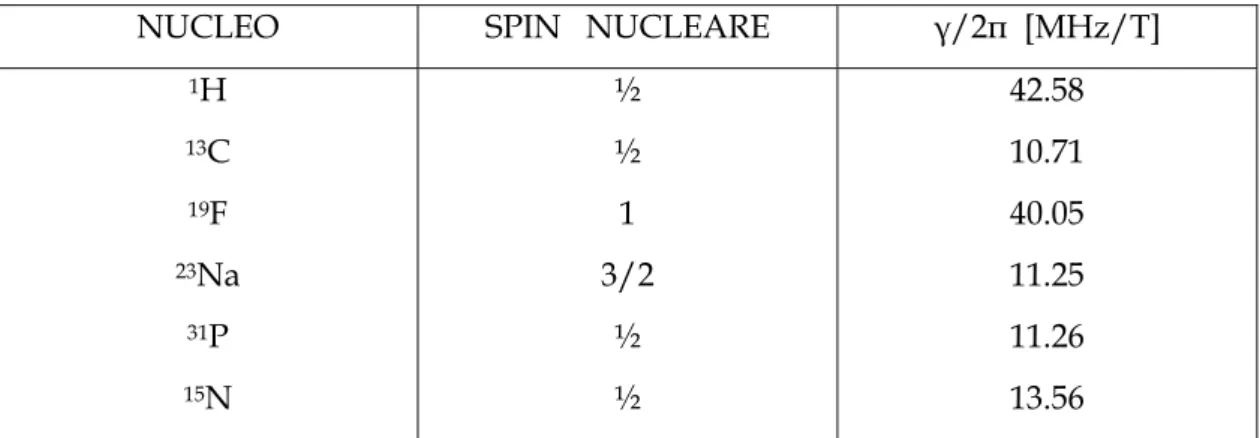
Si tratta di un fenomeno della meccanica quantistica per il quale il nucleo di un atomo contenente un numero dispari di protoni e/o neutroni presenta un momento magnetico proprio in seguito a quello che è chiamato uno spin nucleare. Questi isotopi risultano dunque suscettibili di NMR e tra di loro (1H, 13C, 19F, 23Na, 31P ed
altri ancora) l’ 1H è senza dubbio quello più importante nell’ utilizzo del NMR per la
formazione di bioimmagini che forniscano informazioni strutturali e morfometriche dei tessuti soffici viventi. Infatti il nostro organismo è per oltre i 2/3 in peso costituito di H2O, che è il componente principale di tutti i tessuti, fatta eccezione per
quelli mineralizzati (cioè ossei); questo, unito al segnale di risonanza magnetica nucleare particolarmente intenso al quale l’ 1H dà luogo, rende il MRI una tecnica
d’elezione per la valutazione anatomica ad alta risoluzione spaziale delle strutture biologiche molli.
Il tessuto biologico di cui si vuole ottenere una immagine, sostanzialmente basata sulla densità di protoni presenti, viene immerso in un campo di induzione magnetica B0 statico generato da un magnete superconduttore il quale deve essere
in grado di mantenere permanentemente questo campo, con accurate caratteristiche di omogeneità, anche per parecchi anni senza soluzione di continuità. Il modulo di B0 è dell’ordine di qualche Tesla (valori tipici per uso clinico sono di 1.5 T e 3 T),
cioè qualche migliaio di volte più intenso del campo magnetostatico terrestre ordinario; questo non ha finora mostrato effetti biologici rilevanti su coloro che vi sono esposti, ma pone serie controindicazioni all’applicazione su soggetti portatori
di protesi metalliche, pace-maker, neurostimolatori ed altri dispositivi con i quali il campo magnetico possa in qualunque modo interferire, così come all’introduzione di strumenti e materiali che non abbiano un comportamento il più possibile amagnetico.
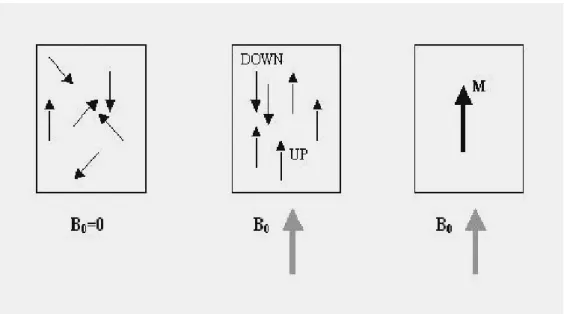
Gli atomi di 1H in condizioni di assenza di campi B0 intensi presentano momenti
magnetici aleatoriamente orientati in tutte le direzioni, generando globalmente una magnetizzazione netta M nulla. All’interno della regione interessata da B0 invece,
questi dipoli magnetici nucleari si allineano tutti a B0, avendo a disposizione due
orientamenti: quello parallelo (UP) e quello antiparallelo (DOWN) [Figura 3.1]. I versi di allineamento UP e DOWN corrispondono a due diversi livelli energetici per le particelle subnucleari: poiché il livello UP è a più bassa energia si osserva, a temperatura ambiente, una maggioranza (invero di poco prevalente) di spin nucleari orientati in modo parallelo a B0 ad ottenere un vettore magnetizzazione
netta risultante non nullo, come in figura 3.1.
Figura 3.1: Allineamento dei momenti magnetici di spin nucleare ad un campo magnetico esterno B0 e magnetizzazione risultante netta M
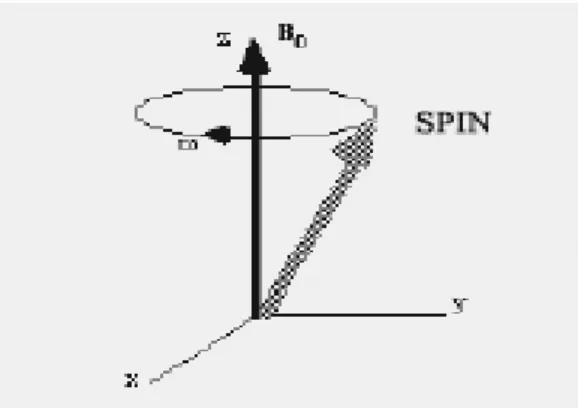
Il moto con il quale i dipoli magnetici intrinseci, e di conseguenza la magnetizzazione netta M che ne è la somma vettoriale, si allineano al campo statico esterno è particolare ed è detto di precessione intorno alla direzione di B0 (Figura 3.2).
Il moto di precessione di M è caratterizzato da una pulsazione angolare di risonanza ω, detta di Larmor, la quale soddisfa la seguente relazione:
0
B
ω γ
= ⋅
dove B0 è il modulo del campo statico applicato dall’esterno e
γ
, detto rapporto ocostante giromagnetica, è un fattore di proporzionalità specifico di ogni nuclide (in
Tabella 1.1 sono riportati i parametri NMR di alcuni nuclidi utilizzati in applicazioni mediche).
Figura 3.2: Precessione dello spin nucleare attorno ad un forte campo magnetico esterno
NUCLEO SPIN NUCLEARE γ/2π [MHz/T] 1H 13C 19F 23Na 31P 15N ½ ½ 1 3/2 ½ ½ 42.58 10.71 40.05 11.25 11.26 13.56
Se supponiamo di accendere un campo magnetico BL ortogonale al campo statico
esterno B0 e oscillante proprio alla frequenza di risonanza di Larmor, caratteristica
dei protoni che compongono in prevalenza i tessuti da mappare per un certo livello di B0, osserveremo un assorbimento di energia elettromagnetica da parte dei nuclei
di 1H, i cui momenti magnetici di spin sono nello stato a bassa energia UP allineati
al campo statico esterno e, conseguentemente, un loro passaggio alla posizione DOWN, cioè ad uno stato energetico eccitato in cui il loro orientamento è antiparallelo. Ciò è ottenuto con campi magnetici variabili a RF (questa è infatti la banda cui appartiene la frequenza di Larmor per i nuclidi di rilevanza biologica, ad esempio in un campo B0 di 1.5 T) generati con apposite bobine. In effetti il vettore
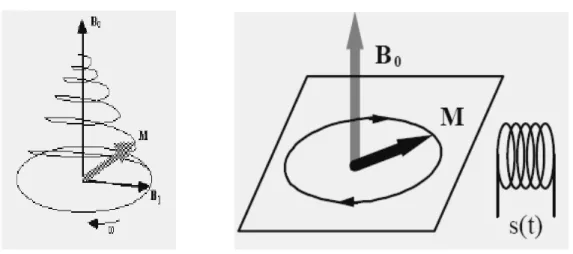
magnetizzazione netta M risponderà all’eccitazione esterna di durata Δt ruotando di un angolo
α
(detto flip angle) rispetto alla direzione di B0 con il solito moto diprecessione alla frequenza di Larmor specifica, allontanandosi dalla posizione di equilibrio (Figura 3.3).
Il flip angle vale
α
=γ
BLΔ t , e se la durata e l’intensità dell’impulso di eccitazione a RF sono sufficienti, può quindi essere di 90° (valore tipico) o di qualsiasi altra entità voluta. Una volta rimossa la sollecitazione BL gli spin magneticidei protoni tendono spontaneamente a riportarsi nello stato a bassa energia, cioè in direzione parallela a B0: ciò avviene con l’emissione da parte loro di energia
elettromagnetica alla stessa frequenza di risonanza, tramite un decadimento esponenziale detto FID (Free Induction Decay, Figura 3.4) che corrisponde ad un segnale rilevabile dalle stesse bobine utilizzate per la eccitazione precedente.
Figura 3.3: Precessione della magnetizzazione totale M durante un impulso di eccitazione a RF generato da una bobina
Figura 34: Segnale FID generato dal rilassamento spontaneo dei protoni in B0 dopo la rimozione della eccitazione col campo a RF
Questo processo di decadimento esponenziale può essere concettualmente suddiviso in due fasi:
• con una costante di tempo T2 detta di interazione spin-spin o trasversale,
decade il moto di precessione di M intorno al campo statico, ossia a causa del progressivo sfasamento degli spin che interagiscono fra loro nelle loro componenti orizzontali, la componente di M sul piano trasversale a B0
diminuisce fino ad annullarsi all’equilibrio; ciò è dovuto al progressivo sfasamento che si verifica fra i singoli momenti magnetici nucleari
• la magnetizzazione netta torna al suo valore di equilibrio, allineata col campo esterno B0, quindi la sua componente parallela a B0 cresce (da nulla
che era dopo una eccitazione tale che
α
=
9 0
°
) fino al valore precedentealla eccitazione col campo a RF, seguendo un andamento esponenziale con una costante di tempo T1 (detta spin-reticolo o longitudinale) che differisce da
T2 e dipende, come quest’ultima, dal tipo di tessuto sul quale è misurata
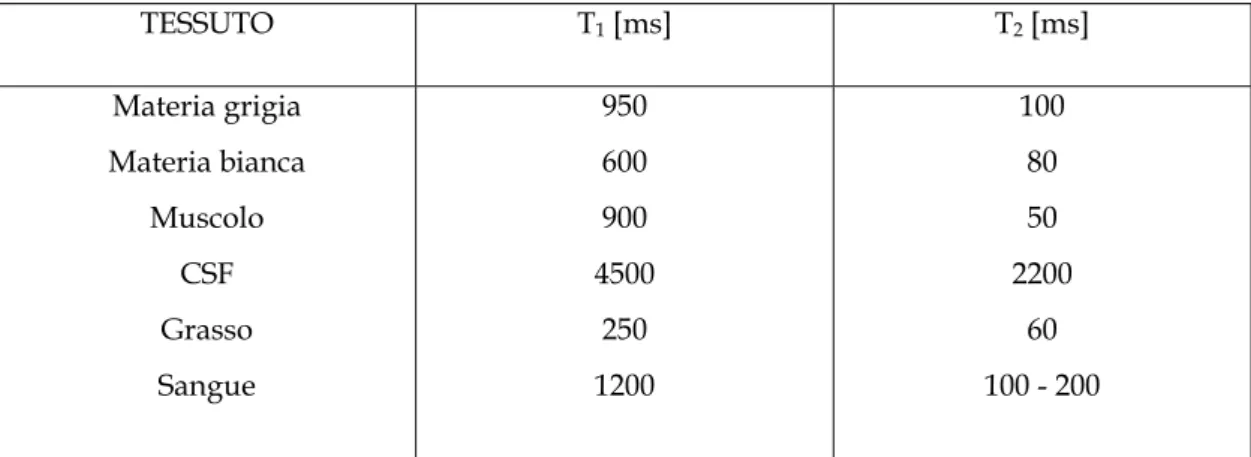
(Tabella 3.2).
Una ulteriore costante di tempo è quella indicata in letteratura come T2*, che tiene
conto sia delle interazioni spin-spin (come T2) che di eventuali disomogeneità del
campo statico, le quali determinerebbero velocità di perdita di fase tra gli spin nucleari disuniformi, a causa di valori diversi di B0, da tenere in conto nel calcolo
della frequenza di Larmor; si ha:
1/T
2*= 1/T
2+ 1/T
2discon 1/T
2dis=ΔB
0per unità di tempo
TESSUTO T1 [ms] T2 [ms] Materia grigia Materia bianca Muscolo CSF Grasso Sangue 950 600 900 4500 250 1200 100 80 50 2200 60 100 - 200
In generale l’MRI si basa sulla variazione da un tessuto all’altro di una di queste caratteristiche, o di una loro somma pesata, e della densità di specie chimiche MR-sensibili per ricavarne un segnale di contrasto utile alla formazione di una immagine.
Quindi le fasi della acquisizione dei segnali, utili alla successiva ricostruzione dell’immagine possono essere riepilogate così: accensione del campo esterno statico B0 , eccitazione con un campo a RF, acquisizione dei segnali MR di risposta con
bobine che fungono da ricevitori, impiegando procedure di eccitazione che consentano di mantenere informazione sulla posizione della sorgente del segnale. La localizzazione spaziale viene ottenuta grazie ad opportune sequenze di applicazione di gradienti spaziali di campo magnetico nelle tre direzioni principali. In sostanza si sfrutta il legame ottenuto fra la frequenza di Larmor e il campo totale in cui è immerso un certo tessuto:
Tabella 3.2 Diversi valori dei tempi dei rilassamento relativi a nuclidi 1H in tessuti biologici umani a 37 °C di temperatura e con un B0 di 1.5 T
Queste relazioni sono valide in una direzione x qualsiasi, f (x) è la frequenza di Larmor e G è una pendenza costante (G⋅x è il gradiente spaziale); in tal modo la frequenza di precessione di uno spin nucleare dipende dalla sua posizione x, ovvero esso genererà un segnale NMR con molte componenti frequenziali, una per ciascuna posizione. A partire dal legame fra f ed x possiamo risalire alla localizzazione del momento magnetico con quella frequenza dopo aver effettuato la trasformata di Fourier del segnale temporale captato dalla bobina.
III.1.2 Principi di fMRI
La Risonanza Magnetica Funzionale (fMRI) è una tecnica che si colloca nell’ambito del neuroimaging di classe MRI, ma il tipo di mappe che produce non è di carattere strutturale, bensì fornisce evidenze circa la ”attivazione” di determinate regioni del cervello in risposta a stimoli specifici.
Il contrasto è ottenuto, senza ricorrere a mezzi introdotti dall’esterno, spesso radioattivi e quindi con effetti collaterali, tramite il segnale BOLD (Blood Oxygen Level Dependent) che scaturisce da cambiamenti fisiologici dei livelli di ossigeno presenti nel sangue circolante nelle aree neuronali funzionalmente attive ad un certo istante.
Nonostante il diffuso ed oramai più che decennale impiego della fMRI, i meccanismi a livello dei neuroni determinanti il segnale MR che origina il contrasto BOLD, non sono ben assodati: in particolare ci sono più modelli alternativi in proposito di come siano collegate l’attività neuronale e l’ossigenazione del sangue. Ciò che invece è dimostrato da numerosi studi è il fatto che il segnale BOLD riflette
una attività dei neuroni [10,11], ed anche le ragioni per cui il livello di ossigeno nel sangue dà luogo ad un segnale di tipo MR sono ben stabilite. Illustriamo quest’ultimo aspetto.
Il cervello umano ha una massa inferiore al 2% di quella totale del corpo, ma consuma il 20% dell’intero fabbisogno di ossigeno di una persona; le ragioni di questo sono più di una ma la principale è che l’ossigeno è utilizzato per metabolizzare il glucosio, il quale fornisce l’energia necessaria alle attività cerebrali. Tuttavia il consumo di glucosio e la portata di sangue in condizione attivata sono tali che, il corrispondente aumento dell’utilizzo di ossigeno rispetto ad uno stato basale, lascia ugualmente nel sangue che irrora le zone attive un livello di ossigenazione aumentato: è questo che determina il segnale fMRI.
Difatti, data la bassa solubilità dell’ossigeno in ambiente acquoso, il meccanismo assolutamente dominante per trasportarlo nel sangue è quello di legarlo all’emoglobina (Hb), una proteina globulare contenente un atomo di ferro; questa si ossida (ossiemoglobina, HbO2) quando una molecola di ossigeno gli si lega, e si riduce
(desossiemoglobina, Hbr) quando la perde. Quello che interessa ai fini dell’fMRI è che l’emoglobina ridotta è paramagnetica, mentre l’emoglobina ossidata è diamagnetica: allora una accentuata presenza di Hbr distorce il campo magnetico statico B0
rendendolo disomogeneo e causando perciò una differenziazione delle frequenze di precessione dei nuclei, nonché una perdita di fase più rapida dei loro spin e quindi una diminuzione di ampiezza del segnale MR. Quest’ultimo a sua volta ha una certa costante di rilassamento T2*, la quale cambia con il livello di Hbr e quindi di O2
presenti nel sangue; ecco che è possibile costruire una immagine T2*-pesata, che è una
mappa funzionale del cervello, in base al suddetto legame tra ossigenazione ed attività dei neuroni; cioè quando essi si “accendono” richiamano sangue ossigenato, il cui flusso aumentato diminuisce il livello di Hbr in quelle aree, inducendo un aumento nel segnale fMRI. Tale grandezza però non ha un significato se espressa in termini assoluti (e di concrete unità di misura fisiche), perché non è una diretta misura della concentrazione di Hbr presente nel sangue, ma è appunto soltanto pesata da questa attraverso il T2*.
Parametro Effetto di una attivazione corticale
Flusso sanguineo ↑↑↑
Consumo di O2 ↑
Livello di O2 nel sangue ↑↑
Livello di Hbr ↓↓
Distorsione di B0 ↓↓
Dispersione di fase per M ↓↓
1/T2* effettivo ↓↓
Segnale T2* -pesato ↑↑
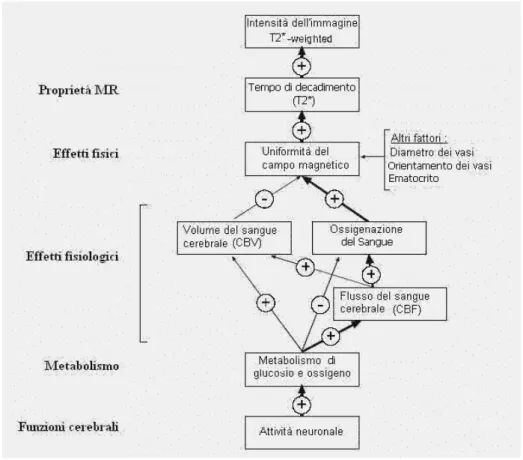
C’è da dire che gli effetti di uno stato corticale attivato sui diversi parametri fisici (di NMR) e fisiologici alla base del segnale fMRI BOLD sono numerosi e non tutti dello stesso segno (Tabella 3.3) e che il segnale BOLD trae origine da una loro complessa combinazione (schema in figura 3.5).
Tabella 3.3 Entità e segno dell’effetto di una attivazione corticale sui principali parametri fisici e fisiologici coinvolti nella fMRI
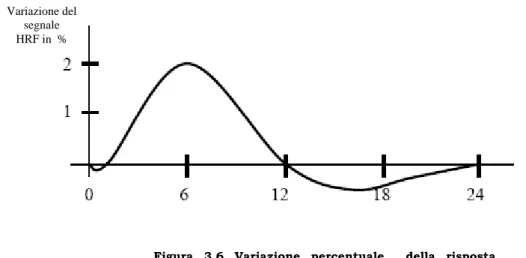
La Risonanza Magnetica Funzionale quindi misura una risposta emodinamica (HRF, figura 3.6) risultante dalla interazione di tutti i fattori schematizzati, ma che in ultima analisi è conseguenza della attivazione funzionale di un gruppo di neuroni. Le caratteristiche fondamentali della HRF sono le seguenti:
riflette indirettamente l’attività di un numeroso gruppo di neuroni
è marcatamente più lenta della risposta di un neurone o un loro gruppo ad uno stimolo
Figura 3.5: Schema delle interazioni fra i parametri che portano alla formazione di una immagine T2* -pesata, basata sul segnale BOLD. Il percorso in neretto è quello predominante nella maggior parte dei segnali fMRI
La prima questione (che sottintende un legame forte e sicuro fra risposta neurale ed emodinamica) ha come conseguenza che una risposta emodinamica ampia può essere dovuta sia alla forte attività di un substrato di neuroni relativamente piccoli, sia ad una debole attivazione di un loro gruppo molto nutrito. Inoltre è possibile correlare una variazione regionale della HRF con l’evidenza di esecuzione di una ben precisa funzione cerebrale solo ammettendo che i neuroni raggruppati in quello specifico sito siano preposti al medesimo compito fisiologico.
La seconda proprietà invece suggerisce una risoluzione temporale intrinsecamente modesta della fMRI (secondi contro decine o centinaia di millisecondi) rispetto alle tecniche di misura di segnali elettrofisiologici (EEG in primis), nonché la possibilità di mascheramento, a livello di misura BOLD, di neurodinamiche molto più rapide, invertendo perciò l’ordine cronologico reale fra gli eventi di attivazione di zone corticali distinte.
Sulla risoluzione spaziale della fMRI possiamo dire che essa è piuttosto buona (dell’ordine di 5 mm), ma occorre tenere presente che i grandi vasi sono sede di una riduzione del livello di Hbr che è più massiccia rispetto a quella cui si assiste nei piccoli vasi e quindi il massimo nel segnale BOLD può essere delocalizzato fino a
Variazione del segnale HRF in %
Figura 3.6 Variazione percentuale della risposta emodinamica nel tempo, in seguito ad un breve incremento della attività neuronale all’istante t = 0
qualche millimetro, rispetto al fuoco di attività neuronale; inoltre le elaborazioni sulle immagini BOLD attuate per ottenere le mappe funzionali finali riducono in genere ulteriormente la risoluzione spaziale originaria.
Con tutto ciò la questione critica forse più importante, specialmente in chiave dei progressi hardware che le macchine per fMRI potranno compiere, è quella legata alla ampiezza della attivazione, che è particolarmente piccola: dall’1% al 4% dello stato inattivo, in un campo statico tipico di 1.5 T: ossia il segnale fMRI è drammaticamente rumoroso.
Questo si affianca alle innumerevoli e difficilmente riducibili sorgenti di rumore riscontrabili nel neuroimaging di RM funzionale; infatti al rumore termico (in senso elettromagnetico), di quantizzazione (legato alla conversione A/D necessaria per gli algoritmi di ricostruzione delle immagini) e a quello intrinseco a tutta l’elettronica presente (amplificatori etc.), tipici di tutto il campo del MRI, nel caso dell’fMRI si aggiungono variazioni nel segnale ricevuto fortemente correlate col ciclo respiratorio e cardiaco del soggetto, lente derive temporali di origine non ancora stabilita (ma assenti se il soggetto nello scanner è un fantoccio inanimato) e movimenti della testa che, seppure dell’ordine di frazioni di un pixel (le cui dimensioni tipiche sono dell’ordine di 1-4 mm), possono causare false attivazioni anche superiori all’1% - 4% sopra citato per la variazione relativa della HRF.
Un ulteriore motivo di confondimento nei dati funzionali può essere dovuto ad attivazioni neuronali spontanee e non controllabili le quali rappresentano evidentemente un disturbo grave nella riconoscibilità di una certa attività come conseguente ad un determinato processo cognitivo, sensoriale o motorio. A questo scopo gli esperimenti che portano alla acquisizione di una mappa funzionale devono essere progettati con la massima cura e si fondano sul metodo sottrattivo; in sostanza l’immagine finale con le attivazioni è ottenuta come sottrazione fra un’ immagine fMRI, che potremmo definire di controllo, acquisita con il soggetto in uno stato in cui certamente (o per meglio dire in misura massimamente probabile) non sono attive le zone legate alla funzione studiata, e l’immagine ricostruita durante la condizione di attività del cervello esaminato: queste condizioni sono ottenute
stimolando ad hoc il paziente nello scanner (lo stimolo è spesso indicato come task) per sollecitarne la funzione cerebrale da individuare separandola nettamente da una
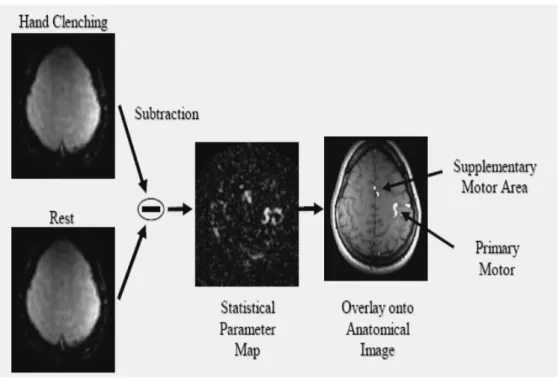
baseline. Il risultato del confronto mediato tra queste immagini (figura 3.7), o più
spesso una mappa statistica (SPM, Statistical Parameter Mapping) ottenuta ad esempio testando voxel per voxel l’ipotesi nulla di assenza di segnale (t-test, che equivale a confrontare voxel per voxel quello di immagini attivate con quello di immagini a riposo), determina la formazione di un overlay nel quale sono evidenziati soltanto quei voxel che differiscono dalla condizione di controllo e quindi rispondono al task predisposto, ossia sono coinvolti nella funzione studiata.
Si parla di overlay perché al fine di localizzare il più precisamente possibile le sedi anatomiche delle aree neurali attive, esso viene sovrapposto ad una immagine strutturale ad elevata risoluzione (l’underlay), acquisita preliminarmente alla funzionale con lo stesso tomografo MRI.
Figura 3.7 Esempio di metodo sottrattivo con formazione di una SPM nello studio della attivazione delle aree motorie in seguito ad “apertura” e “chiusura” di una mano
III.1.3 fMRI per l’olfatto
Gli studi funzionali sull’apparato olfattivo, in particolare relativamente alla corteccia olfattiva primaria e secondaria, si sono moltiplicati nell’ultimo decennio grazie all’avvento delle tecniche di neuroimaging funzionale in vivo: PET (Positron Emission Tomography) ed fMRI. Le due metodiche hanno prerogative diverse che le rendono più o meno adeguate alla ricerca nel campo dell’olfatto, ma quest’ultimo pone delle problematiche generali connaturate alle sue proprietà distintive ed anche particolari limitatamente alla risonanza magnetica funzionale [12].
I problemi intrinseci sono legati a numerosi fattori: la complessità della elaborazione degli stimoli olfattivi, il controllo automatico della respirazione che un individuo può attuare modulando la propria esposizione ad un odorante ed anche l’attenzione riposta nel rivelarne la presenza, il diverso percorso dei segnali nervosi a seconda che l’odorazione sia passiva o no oppure in base alla condizione se gli stimoli siano unimodali o bimodali o tali da coinvolgere circuiti non propriamente sensoriali, attivando il sistema limbico.
Una speciale attenzione merita inoltre il fatto che la risposta olfattiva e la sensibilità ad un certo odorante risentono, in caso di somministrazioni ripetute e/o continuate per lunghi periodi di tempo, dei fenomeni dell’adattamento e della abituazione [13]: il primo consiste in una diminuzione della risposta ad un segnale olfattorio che ha luogo a livello periferico e recettoriale, mentre il secondo riflette riduzioni che avvengono a livello di sistema olfattivo centrale; entrambi si prospettano come le cause, peraltro difficilmente separabili, della perdita di sensibilità per un certo odore che interviene dopo che un soggetto vi è stato lungamente esposto.
Tutti questi aspetti devono essere presi in considerazione con grande cura sia nell’interpretazione dei risultati che, soprattutto, nella progettazione dell’intero esperimento, con speciale riferimento al task di stimolazione, e interessano sia la PET che la fMRI. In più tutte le aree della corteccia olfattiva umana sono di dimensioni assai ridotte e perciò non agevolmente individuabili di per sé, se non con elevate risoluzioni.
Le questioni critiche che affliggono segnatamente l’analisi con risonanza magnetica funzionale dell’olfatto, sono essenzialmente riconducibili a due categorie: 1) i protocolli di stimolazione, 2) la collocazione anatomica delle zone coinvolte e la composizione delle strutture ad esse prossimali.
Il primo punto si riferisce in modo particolare ai requisiti che un apparato di somministrazione di uno stimolo olfattivo deve possedere per essere impiegabile in un esperimento di fMRI, soprattutto in relazione alla necessità che il sistema entri nella stanza della RM e addirittura nel magnete per raggiungere il naso del soggetto esaminato.
Circa la collocazione anatomica e la composizione delle zone possiamo dire che quelle che dovrebbero risultare attivate per lo più sono vicine a strutture disomogenee (osso, liquor, aria), collocate in prossimità di interfacce osso-aria ed osso-tessuto cerebrale, e che quindi vi potranno essere problemi in conseguenza di una marcata suscettibilità magnetica e un SNR ulteriormente abbassato, nonché incoerenza statistica fra le risposte emergenti dalla elaborazione dei dati.
Il MRI funzionale tuttavia possiede anche dei vantaggi nello studio dell’olfatto, perché rispetto alla PET non espone a radiazioni e quindi consente studi ripetuti di uno stesso soggetto (particolarmente importante in questo campo per la forte variabilità della risposta olfattiva e della anatomia cerebrale fra individui diversi) ed inoltre è caratterizzata da una risoluzione temporale migliore ed è pertanto indicata quando si vogliano prendere in considerazione gli andamenti temporali delle risposte.
III.2 Studi di Connettività Cerebrale su dati fMRI
L’utilizzo classico delle tecniche di neuroimaging è quello finalizzato all’individuazione delle funzioni specifiche delle regioni cerebrali ed il loro
isolamento (functional segregation). Solo più recentemente questo tipo di indagine è stato indirizzato verso studi di integrazione funzionale (functional integration) con lo scopo di ricostruire mappe funzionali che individuino le regioni coinvolte nello svolgimento di un compito specifico assegnato, nell’ottica di descrizione del cervello dal punto di vista della sua connettività.
In particolare la connettività funzionale cerca di capire quale sia la risposta complessiva del cervello, in seguito a determinati stimoli, ed individuare le aree correlate tra loro che contribuiscono allo svolgimento di quella risposta; invece dal punto di vista della connettività effettiva si cerca di capire come tali aree interagiscano tra di loro e come queste interazioni dipendano dai cambiamenti del contesto sperimentale.
Un punto di fondamentale importanza è la scelta degli strumenti di analisi più adatti a questo o quel genere di studio funzionale. La prima distinzione riguarda i metodi che vengono utilizzati nell’ambito della segregazione funzionale e quelli dedicati invece agli studi di integrazione funzionale ovvero di connettività (effettiva o funzionale) [14].
I primi sono quelli classici dell’analisi di dati di imaging funzionale; essi stimano come il singolo voxel sia correlato o meno con il paradigma sperimentale, infatti sono stati denominati voxel-based, senza tenere conto della eterogeneità della struttura della matrice di covarianza dei dati ricavata dall’esperimento; in altre parole tali metodi nel loro approccio all’analisi delle sequenze temporali non prendono assolutamente in considerazione le correlazioni che potrebbero intercorrere tra le varie regioni (ovvero tra i vari segnali) che sono appunto spiegate dalla matrice di covarianza dei dati.
Nella maggior parte dei casi questi sono approcci univariati che vanno a testare l’ipotesi nulla “assenza di segnale utile” basandosi su modelli cosiddetti sottrattivi, che confrontano due immagini relative a due momenti diversi dell’esperimento, ed implementando modelli di regressione multipla che sfruttano predittori legati al task di stimolo e alla risposta bold; solo a posteriori è possibile individuare la correlazione tra i vari voxel attivati.
In effetti essi isolano con molta efficacia le regioni attivate in seguito ad un determinato task ma non sono in grado di dire a priori che quelle regioni sono correlate tra loro perché facenti parte di un sistema di connettività; solo a posteriori e solo grazie al fatto che l’esperimento è stato progettato ad hoc per quel determinato task è possibile affermare che con molta probabilità le zone individuate compartecipano ad una risposta coerente del cervello.
In particolare il metodo più diffuso nell’analisi dei dati di neuroimaging è il GLM (general linear modelling); esso è sostanzialmente un modello di regressione che va a testare i contributi che diverse funzioni sorgente danno alla varianza del segnale in un singolo voxel. Filtrando il segnale task (che indica la sequenza con cui sono stati presentati gli stimoli) attraverso la risposta Bold si ottiene il cosiddetto segnale paradigma cioè la risposta ideale che dovremmo trovare in ogni regione attivata; questo segnale, insieme ad una base-line (che può essere costante, lineare o anche di ordine superiore), costituisce la sorgente coi cui si va a “regredire” i dati reali [15]. D’altra parte gli studi di integrazione funzionale adottano metodi di analisi molto differenti da quelli utilizzati nelle ricerche di segregazione; inoltre tali tecniche si differenziano ulteriormente a seconda che si appronti uno studio di connettività funzionale od effettiva.
Per quanto riguarda lo studio della connettività funzionale, si utilizzano
principalmente tecniche di analisi multivariata che sfruttano approcci descrittivi data-driven che vanno a caratterizzare le dinamiche psicofisiologiche delle diverse regioni cerebrali attraverso mappe funzionali cui sono associati i relativi modelli delle risposte temporali.
Attualmente i principali metodi utilizzati per gli studi di connettività funzionale sono [14,16-19]:
Principal Component Analysis (PCA)
Covariance Matrix & Singular Value Decomposition (SVD) Indipendent Component Analysis (ICA)
L’aspetto più interessante da sottolineare è che questi metodi lavorano direttamente o indirettamente sulla matrice di correlazione dei dati proiettando i segnali acquisiti in uno spazio le cui “dimensioni” hanno la caratteristica di essere incorrelate o addirittura indipendenti; le proiezioni dei dati in questo spazio costituiscono un set di componenti incorrelate (o indipendenti) la cui combinazione lineare (con coefficienti opportuni ricavati dalla decomposizione) permette di ricostruire i dati originari.
Quindi in fase di ricostruzione del segnale reale, a seconda di quale componente lo descrive maggiormente (in termini di varianza), è possibile stabilire quali regioni sono accomunate dalla presenza “forte” di quella particolare componente e quali invece sono spiegate maggiormente da una delle altre componenti; in questo senso regioni i cui segnali sono caratterizzati per lo più da componenti diverse possono essere considerate quanto meno incorrelate visto che lo sono i segnali ad esse associati.
Così si riesce a individuare quali regioni sono coinvolte, nel senso che sono correlate con il medesimo segnale, nella risposta a quel determinato task e quali invece sono assolutamente incorrelate con quell’evento, formando delle vere e proprie mappe funzionali.
In particolare occorre sottolineare che la PCA e la ICA sono metodi di trasformazione data-driven che non necessitano di alcun tipo di assunzione ed implementano una ricerca tipo BSS (Blind Source Separation). Dal punto di vista della connettività lavorano senza alcuna informazione circa le connessioni biologiche che intercorrono tra le varie regioni e quindi sono particolarmente indicate nei casi in cui non si conoscano le regioni implicate nello svolgimento di un determinato compito.
Per quanto riguarda invece l’analisi della connettività effettiva essa utilizza modelli statistici completamente diversi che lavorano su regioni selezionate e fanno assunzioni sulle connessioni anatomiche tra queste regioni in base a modelli di connettività strutturale [19]. Questo tipo di analisi può essere definita come hypothesis-driven piuttosto che data-driven ed è tanto più performante tanto più
sono precise le informazioni sulle aree funzionali rilevanti. I più importanti metodi di ricerca di connettività effettiva sono il SEM (structural equation modelling) e il DCM (dynamic causal modelling); il primo, fino ad oggi maggiormente impiegato dell’altro, soprattutto su dati fMRI, è stato sviluppato nel campo dell’econometria mentre il secondo è nato appositamente per l’analisi delle serie temporali da imaging funzionale.
Il SEM lavora utilizzando un set di regioni ed un set di connessioni direzionali stabilite, dove il significato di connessione tra A e B è inteso nel senso che A causa B. Le relazioni di causalità tra le varie regioni in questo caso non sono derivate dai dati ma sono ipotizzate a priori in base ad informazioni di connettività anatomica. Il sistema cervello è considerato statico e le connessioni tra le regioni sono viste come legami di causalità istantanea. Fissando i pesi da attribuire alla varie connessioni vengono automaticamente fissate le correlazioni che intercorrono tra i segnali delle varie regioni. Il principio su cui si basa la stima del modello di connessione (e quindi dei sui pesi) è proprio quello di settare tali coefficienti in modo tale da minimizzare la differenza tra la matrice di covarianza del modello e la matrice di covarianza dei dati sperimentali.
Nell’analisi DCM invece il cervello è considerato come un sistema deterministico e dinamico in cui gli stimoli esterni (ingressi) causano cambiamenti nell’attività neurale che a loro volta causano un cambiamento della risposta Bold rilevata dall’fMRI; in questo caso il termine “causa” acquista un significato ben diverso rispetto a quello che ha nel SEM, infatti qui per realizzare il modello dei dati si va a concatenare la risposta emodinamica con la risposta neurodinamica delle regioni cerebrali.
III.2.1 Metodi di analisi di Connettività Funzionale
Nello studio della connettività funzionale le metodologie di analisi più diffuse sono la PCA, la SVD e la ICA. Di seguito verranno presentati questi tre metodi ponendo
l’attenzione sui loro aspetti matematici e soprattutto sulle loro modalità di impiego in una prospettiva di indagine di integrazione funzionale, sottolineando i motivi che li rendono così efficaci in questo tipo di ricerca. Occorre precisare subito che questi strumenti statistici hanno aspetti in comune molto marcati e impieghi molto simili tra loro (a tal punto che la SVD e la PCA sono praticamente la stessa cosa) anche se differiscono sostanzialmente nel loro impianto teorico. Tuttavia vale la pena affrontare questa trattazione per puntualizzare i principi su cui si fondano ed evidenziare le loro potenzialità, cercando così di dare una panoramica delle più moderne metodiche di analisi statistica impiegate negli studi di connettività funzionale.
-
PCA : Principal Component Analysis
La PCA o trasformata di Karhunen-Loeve (K-L) nasce nell’ambito delle metodiche di analisi multivariata, cioè l’analisi di dati provenienti dall’osservazione di un certo numero di variabili su un gruppo di soggetti [25]. L’interpretazione di questi dati spesso non è immediata; soprattutto è difficile dire a priori quali e quante siano le variabili effettivamente necessarie e non ridondanti per una loro descrizione e successiva interpretazione. Pertanto il primo passo da compiere quando si eseguono analisi su più variabili è quello di verificare se tutte le variabili siano utili; i passi successivi riguardano l’effettiva descrizione dei dati ridotti e la loro classificazione. La sintesi interpretativa finale è molto più agevole su un numero di variabili ridotto e spesso un approccio di questo tipo permette anche di ridurre possibili dispersioni rispetto ad una complessità maggiore apparente.
La PCA è un metodo completo che può essere usato in tutte le fasi di riduzione, descrizione e classificazione di dati multivariati. Essa si basa su una trasformazione lineare delle variabili di partenza in altre variabili con proprietà particolari, tra cui quella di essere incorrelate tra loro ed ordinate in ordine decrescente a seconda della
quantità di informazione che ognuna di esse spiega. Infine non è necessario assumere alcuna ipotesi a priori sulla distribuzione dei dati e delle variabili.
Riassumendo gli obiettivi che si prefigge l’analisi PCA:
- Identificare nuove variabili significative; - Ridurre la dimensionalità del problema;
- Eliminare le variabili originarie che portano informazioni ridondanti; - Interpretare i dati dal punto di vista delle variabili o dei soggetti;
Intuitivamente, da un punto di vista geometrico una matrice Xnp rappresenta n punti dello spazio p vettoriale; con due sole variabili x1 e x2 è facile rappresentare graficamente l’insieme dei dati: ogni individuo ei è un punto sul piano x1 - x2 e il semplice esame visivo permette di studiare l’intensità del legame tra le due variabili e di ricercare gli individui che presentano caratteristiche diverse. Con un numero di variabili (p) superiore a tre non si riesce più a visualizzare graficamente la situazione.
L’operazione di scomposizione dei dati nelle loro componenti principali equivale a proiettare gli individui in un sottospazio q-dimensionale con q<p. La proiezione viene fatta in modo da minimizzare le distorsioni e conservare al meglio le distanze tra gli individui; il criterio adottato nella scelta del piano di destinazione è quello che rende massima la media dei quadrati delle distanze tra le proiezioni fi.
Dal punto di vista matematico supponiamo di avere a disposizione un set di dati organizzati per variabili in una matrice n x p , dove n è il numero delle osservazioni e p è il numero delle variabili:
x : vettore delle variabili [ ,1 2... ]
t p x= x x x
μ : vettore delle medie [ 1, 2... ]
t p
Σ : matrice di covarianza
Σ =
E x
[(
−
μ
) (
⋅ −
x
μ
) ]
tNella realtà le matrici μ e Σ non sono note ma si hanno a disposizione solamente le loro stime m e S ricavate sulla base delle osservazioni disponibili; in pratica ogni qualvolta si effettua una PCA si trova una stima di λi e ai , che sono rispettivamente
gli autovalori e gli autovettori di Σ.
Partendo dalle variabili originarie x1 , x2 , x3 …., xp la trasformazione K-L consente di ottenere nuove variabili y1 , y2 , y3 …., yp con le seguenti caratteristiche:
- sono fra loro incorrelate:
E y y
[ ,
i j]
=
0
cioèCov y y
( ,
i j)
=
0
- sono ordinate con varianza decrescente:
Var y
( )
1>
Var y
(
2)
>
...
>
Var y
(
p)
- la variabile
y
i è definita come combinazione lineare delle variabili originarie come: 1 1 2 2 ... t i i i pi p i y =a x⋅ +a ⋅ +x +a ⋅x = ⋅a x ; dove [ 1, 2 ... ] t i i i pi a = a a a è un vettore di costanti che soddisfa la seguente condizione di ortonormalità:2 1
1
p ii ia
==
∑
cioè: a atj⋅ =iIl problema di K-L trova soluzione nell’ambito dell’algebra lineare andando a scomporre la matrice di covarianza in una matrice di autovettori e di autovalori attraverso la scomposizione a valori singolari SVD:
1 se i = j 2 se i ≠ j
1
[
]
[
]
t t pS
U W V
U
V
λ
λ
⎡
⎤
⎢
⎥
⎢
⎥
=
⋅
⋅
=
⋅
⎢
⎥
⋅
⎢
⎥
⎢
⎥
⎣
⎦
i
i
U e V sono matrici che verificano la condizione di ortonormalità (U-1=Ut e quindi
UUt=I) mentre W è una matrice diagonale i cui valori sono gli autovalori della matrice di covarianza (definiti non negativi) e rappresentano le varianze delle componenti principali.
In sostanza si giunge a definire una matrice A (p x p) tale che
A
=
[ ,
a a
1 2...
a
p]
, dove gli ai indicano le colonne di A e rappresentano gli autovettori di S, tale che:t
Y
=
A X
⋅
.La matrice Y è la matrice contenente le componenti principali:
1 1 1 t t p p p
y
a
x
y
a
x
⎡
⎤
⎡
⎤
⎡
⎤
⎢
⎥
⎢
⋅
⎥
⋅
⎢
⋅
⎥
⎢
⎥
⎢
⎥
=
⋅
⎢
⎥
⎢
⎥
⎢
⋅
⎥
⋅
⎢
⋅
⎥
⎢
⎥
⎢
⎥
⎢
⎥
⎢
⎥
⎢
⎥
⎢
⎥
⎣
⎦
⎣
⎦
⎣
⎦
Si dimostra che la matrice di proiezione dei dati (A) corrisponde alla matrice (U) le cui colonne sono gli autovettori della matrice di covarianza S (calcolata come E[xxt]); mentre le colonne della matrice V sono gli autovettori della matrice di covarianza calcolata come E[xtx] cioè avendo scambiato nella matrice dei dati X le variabili con le osservazioni. Quindi:
Y U X
=
t⋅ = ⋅
X U
.La proprietà più importante di questa scomposizione è che la somma delle varianze originarie e delle componenti principali è la stessa:
1 1 1
v a r(
)
v a r(
)
v a r(
)
p p p i i i i i iy
λ
x
= = ==
=
∑
∑
∑
poichétr W
(
)
=
tr A
(
t⋅Σ ⋅
A
)
=
tr
(
Σ⋅ ⋅
A A
t)
=
tr
( )
Σ
.Grazie a questa proprietà si può valutare quale sia la porzione di varianza del segnale originario spiegata dalla i-esima componente principale:
1
(
)
i i p i i it r
λ
λ
λ
λ
==
∑
Infine occorre osservare che l’analisi PCA è legata strettamente alla scomposizione SVD effettuata direttamente sui dati; infatti, considerando un set di dati organizzati per righe (ogni riga è relativa ad un soggetto):
t x
X
= ⋅
U W V
⋅
e andando a calcolare la matrice di covarianza dei dati si ottiene:
cov(
)
t(
x t) (
x t t)
2 1 2 t t t x x p
w
S U W W U
U
U
w
⎡
⎤
⎢
⎥
= ⋅
⋅
⋅
= ⋅
⎢
⎥
⋅
⎢
⎥
⎣
⎦
i
Questo significa che gli autovalori della matrice di covarianza dei dati sono i quadrati dei valori singolari dei dati stessi ovvero che i valori singolari dei dati sono pari alla radice quadrata delle varianze delle componenti principali. Pertanto i due metodi, facendo le opportune considerazioni, sono del tutto analoghi e possono essere utilizzati indifferentemente portando agli stessi risultati.
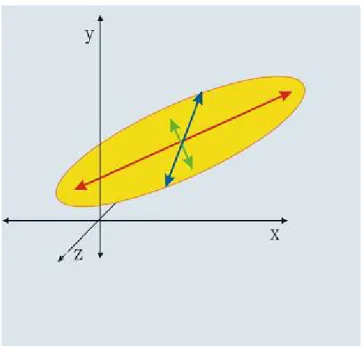
Come si deduce dalla descrizione matematica appena presentata, in un’analisi PCA gli strumenti che caratterizzano l’intero processo e che in fase di interpretazione descrivono i risultati sono sostanzialmente due: gli autovalori e gli autovettori della matrice di covarianza; i primi rappresentano le componenti principali ovvero la base di segnali incorrelati su cui vengono proiettati i dati, i secondi rappresentano la varianza degli autovalori e danno informazioni sulla importanza (in termini di informazione contenuta) delle singole PC. Per visualizzare meglio il funzionamento di questa scomposizione, immaginiamo di avere un problema “trivariato” (descritto cioè da un set di tre variabili) e di andare a rappresentare i dati in uno spazio tridimensionale di assi cartesiani (figura 3.8); si osserva come gli autovettori rappresentino l’unica base ortonormale su cui è possibile riproiettare il volume dei dati rappresentati dall’ellissoide.
Lo scopo classico della PCA ed in un certo senso anche quello più semplice è la riduzione dei dati nell’ambito dei problemi multivariati. L’obiettivo in questo caso è capire quali delle variabili in gioco possono essere eliminate dalle analisi successive dato il loro scarso contributo alla varianza totale del sistema, ovvero all’informazione totale contenuta nei dati.
La scelta dipende da diversi fattori tra i quali: il tipo di dati in esame, il tipo di informazioni che si vuole trarre da questi, l’accuratezza dei dati necessaria per le analisi successive e infine anche la dimensione della matrice dei dati. In questo senso la PCA è in grado di classificare le variabili in base al loro contributo alla descrizione dei dati; quindi permette di ridurre, in base alla percentuale di varianza che si vuol trattenere, la dimensionalità del problema sia dal punto di vista della selezione del numero di variabili significative che da quello dell’eliminazione del
Figura 3.8 : gli autovalori descrivono le direzioni della distribuzione dei dati; gli autovalori quantificano la varianza
rumore nei casi di analisi di segnali temporali (interpretati come realizzazioni di variabili aleatorie).
A partire dagli anni ’90, con l’affermazione delle tecniche di neuroimaging, la PCA ha trovato largo impiego in questo campo grazie alla sua capacità di riduzione della dimensionalità dei dati attraverso l’utilizzo della decorrelazione, che le permette di individuare le zone di attivazione e contemporaneamente evidenziarne le interazioni [14].
Infatti, come già accennato, essa estrae dai dati (in questo caso segnali campionati temporalmente che vengono interpretati come realizzazioni di variabili aleatorie cioè i singoli voxel) un set di componenti incorrelate tra loro di cui si conosce il contributo di informazione che ognuna fornisce ai dati stessi e riesce a individuare le regioni in cui è maggiormente presente l’informazione legata alle più significative componenti principali.
Quindi, nell’ipotesi che l’informazione prevalente nei dati e quindi rilevata dalle PCA, sia quella indotta dal task eseguito, è possibile estrarre le zone cerebrali maggiormente attivate in risposta allo stimolo.
Questo sembrerebbe un risultato di segregazione funzionale, in realtà il fatto che la PCA individui regioni il cui segnale è legato maggiormente ad una particolare componente principale implica che queste regioni siano in qualche modo compartecipi, dal punto di vista funzionale, alla stessa risposta cerebrale. Tali risultati sono interpretabili, a ragione, come un’evidenza della connettività funzionale tra quelle zone. In questo modo la PCA permette di estrarre dei modelli di correlazione spazio-temporale tra le varie regioni cerebrali che sono in grado di spiegare la maggior parte dell’informazione contenuta nei dati.
Attenzione al fatto che l’attivazione contemporanea di determinate regioni coinvolte nella riposta implica il loro coinvolgimento alla risposta complessiva del cervello ma non implica necessariamente una loro connettività effetiva, potrebbero essere attivazioni assolutamente indipendenti anche se correlate allo stesso stimolo.
Per convenzione, negli studi di imaging che si basano sull’analisi delle componenti principali, oltre agli autovalori (eigenvalue) e agli autovettori (eigenvectors o eigenvariets)
della matrice di correlazione dei dati, è utilizzato anche un altro parametro per la descrizione e la ricostruzione dei dati del problema, le cosiddette autoimmagini (eigenimages); queste sono delle mappe che contengono, per ogni voxel, il contributo (coefficiente di proporzionalità) che ogni componente principale dà a quel determinato segnale. Per chiarire quale sia il procedimento implementato in uno studio basato sulla PCA presentiamo un esempio (simulato) in cui si riassumono i passi successivi dell’analisi (figura 3.9):
1. Presentazione delle serie temporali di un immagine 1-D;
acquisizione fMRI, campione di 32 voxel.
2. Componenti Principali: rappresentano i segnali associati ad ogni
eigenimage.
3. Decomposizione dello spettro: da qui si evince il numero di PC
sufficienti a descrivere la maggior parte della varianza osservata
nei dati.
4. Eigenimages: rappresentano il contributo di ogni componente
principale alle serie temporali di ogni voxel.
5. Ricostruzione: utilizzando le prime tre componenti principali si
ricostruisce un immagine molto vicina a quella originale; significa
che è stata preservata la parte più significativa dell’informazione.
-
ICA : Indipendent Component Analysis
L’analisi delle componenti indipendenti è sostanzialmente un metodo di estrazione di singoli segnali da un insieme di segnali variamente combinati e mescolati. Il
Tempo (scansioni)
Voxel
Eigenimages Autovalori
Autovettori (PC)
Dati ricostruiti con le prime 3 PC Dati acquisiti
Figura 3.9 : esempio di analisi attraverso PCA di una sequenza fMRI (128 campioni) su una immagine 1-D di 32 voxel (1) (2) (4) (3) (5)
principio su cui si fonda è che realisticamente processi fisici diversi generano segnali indipendenti tra loro.
Il problema classico con il quale viene spiegato il funzionamento della ICA è quello del “cocktail party”: un party dove molte persone stanno parlando contemporaneamente ed un microfono che rileva un segnale audio dato dalla mescolanza di tutte le voci della festa. L’analisi delle componenti indipendenti riesce a rilevare le singole sorgenti (indipendenti tra loro), che sono proprio le singole voci delle persone presenti alla festa, senza avere a disposizione nessuna informazione sui segnali. In realtà nella pratica il numero di microfoni deve essere pari al numero di componenti indipendenti che si vuol stimare.
La ICA è un metodo di ricerca tipo BSS (Blind Source Separation) molto efficiente che ha come obiettivo la stima di un set di segnali statisticamente indipendenti che rappresentano la base da cui sono stati generati i dati originari; questa ricerca avviene andando a massimizzare l’entropia dei segnali estratti.
Dal punto di vista analitico [21] supponiamo di avere n osservazioni derivanti da mescolamenti lineari di n variabili aleatorie indipendenti
j 1 1 2 2
x
= ⋅ + ⋅ +
a s a s
j j...
+ ⋅
a s
jn n per j =1 :nDa notare che lavorare su variabili aleatorie è una generalizzazione del caso di segnali temporali che possono essere considerati in ogni istante come singole realizzazioni di tali variabili (per questo si omette la dipendenza temporale). Inoltre si assume, senza perdere di generalità, che le componenti si e i dati xi siano a valor medio nullo. Il problema viene solitamente posto in forma matriciale assumendo x vettore colonna formato dalle xJ , s vettore colonna formato dalle si e A matrice di mixing contenente gli aJi.
x=A s
⋅
La ICA va a stimare s ed A, che sono completamente sconosciute, facendo le ipotesi che le sorgenti siano statisticamente indipendenti e che le loro distribuzioni, seppur ignote, siano non-gaussiane. Si definisce la matrice di unmixing W come inversa di A che soddisfa la relazione
s W x
= ⋅
La matrice di mixing e il vettore delle sorgenti sono stimati a meno di una costante moltiplicativa che nel loro prodotto si elide permettendo una corretta ricostruzione dei dati iniziali; inoltre non è rispettato nessun ordine particolare, infatti è possibile ottenere permutazioni di A ed s senza che la stima sia falsata
1 1
x=k A P
⋅ ⋅
−⋅ ⋅ ⋅
P s k
−dove k è uno scalare e P una generica matrice di permutazione.
Considerando due variabili casuali y1 e y2 la definizione elementare di indipendenza è che la conoscenza del valore di y1 non ci dà alcuna informazione sul valore assunto da y2 e viceversa.
Da un punto di vista statistico questa definizione è espressa dalla condizione necessaria e sufficiente
1 2 1 1 2 2
(
,
)
(
)
(
)
p y y
=
p y p y
dove p(y1, y2) è la pdf congiunta delle due variabili mentre p1(y1) e p2(y2) sono le loro pdf marginali. La più importante proprietà che ne consegue è
{
1( ) ( )
1 2 2}
{
1( )
1} {
2( )
2}
E h y h y
=
E h y E h y
dove h1 e h2 sono due generiche funzioni assolutamente integrabili e naturalmente la definizione è estendibile ad un numero qualsiasi di variabili.
Il significato di tale proprietà è che, dato un insieme di n variabili aleatorie indipendenti, i momenti di ordine superiore al primo, calcolati sulla loro statistica n-esima, sono necessariamente nulli. Si potrebbe pensare di definire una scala di gradi di indipendenza a seconda del numero di momenti di ordine superiore che risultano uguali a zero; da questo punto di vista il caso di variabili incorrelate, che prevede covarianza nulla, può essere interpretato come una situazione di minima indipendenza tra le variabili.
Il criterio su cui si basa la ricerca fatta dalla ICA è proprio quello di massimizzare l’indipendenza delle sorgenti stimate attraverso il calcolo dei momenti di ordine superiore.
Stabilito il modello di dati cui fa riferimento la ICA (x = As), il problema che ora si presenta è quello di come poter stimare la matrice di mixing e quindi le componenti indipendenti che risolvono correttamente il problema.
Un primo metodo, piuttosto intuitivo e peraltro valido solo nel caso particolare di variabili uniformemente distribuite, è basato sulla valutazione geometrica delle direzioni delle colonne della matrice A. Consideriamo due sorgenti s1 e s2 con pdf
marginali uniformi e due variabili x1 e x2 derivanti dal loro mixing attraverso la matrice A; le due sorgenti sono assolutamente indipendenti in quanto il valore assunto da una non dà nessuna informazione su quello assunto dall’altra e ciò è riscontrabile anche dalla forma della loro pdf congiunta (figura 3.10a).
Attraverso la matrice di mescolamento le sorgenti vengono proiettate lungo le direzioni individuate dalle colonne della matrice A e si trasformano nelle variabili x1 e x2 , perdendo l’originaria indipendenza statistica; a riprova di questo si osservi come la pdf congiunta delle variabili mescolate sia deformata rispetto alla pdf congiunta delle sorgenti (fig. 3.10b).
In questa situazione le direzioni individuate dai vertici del parallelogramma corrispondono esattamente alle direzioni lungo le quali sono state proiettate le sorgenti, quindi se riuscissimo a valutare queste direzioni avremmo stimato le colonne della matrice di mixing risolvendo così l’intero problema.
In realtà il procedimento non è generalizzabile a variabili con pdf non uniformi e dal punto di vista computazionale è difficilmente realizzabile, tuttavia è utile per capire la dinamica delle trasformazioni statistiche che avvengono durante il mescolamento.
Figura 3.10 : a) funzione densità di probabilità congiunta delle sorgenti s1 ed s2 ; b) funzione densità di probabilità congiunta delle variabili x1 ed x2
Per esempio lo possiamo sfruttare per capire il motivo per cui la ICA non riesce a lavorare bene se le sorgenti indipendenti hanno distribuzione gaussiana.
Consideriamo due s1 e s2 indipendenti con pdf marginali gaussiane; la loro pdf congiunta risulta 2 2 1 2 ( ) 2 1 2
1
( ,
)
2
x xp x x
e
π
+ −=
Tale distribuzione ha simmetria circolare (fig. 3.11a), quindi, nel caso in cui si applichi una matrice di mescolamento ortogonale, le variabili x1 e x2 che ne derivano mantengono tale simmetria (fig. 3.11b) escludendo la possibilità di individuare graficamente le direzioni delle colonne della matrice A.
La spiegazione più rigorosa di questo fatto è che una qualsiasi trasformazione ortogonale applicata ad una distribuzione gaussiana nelle variabili (indipendenti) x1 e x2 ha esattamente la stessa distribuzione e mantiene l’indipendenza delle variabili. Quindi nel caso di sorgenti così distribuite la ICA può lavorare a meno di una trasformazione non ortogonale.
Figura 3.11: a) pdf congiunta delle sorgenti s1 e s2
(distribuite uniformemente); b) pdf congiunta delle variabili
Tutti i metodi di stima delle IC basano la loro ricerca sulla minimizzazione della gaussianità delle sorgenti stimate.
Questo tipo di approccio trova spiegazione nel Teorema del Limite Centrale il quale afferma che, date n variabili aleatorie iid, la loro somma è una variabile aleatoria con distribuzione che tende sempre più a quella “normale” al crescere di n.
Nel modello di dati previsto dalla ICA (x = As ; s = Wx ) i dati sono una combinazione lineare delle sorgenti (supposte iid) secondo la matrice A. Analogamente, quando andiamo ad effettuare la stima delle sorgenti, le interpretiamo come una particolare combinazione lineare dei dati secondo la matrice W.
Se consideriamo un generico vettore y = bT x, la stima che facciamo di s appartiene
sicuramente a questa famiglia di combinazioni lineari delle x, e ne rappresenta la componente a minima gaussianità; infatti:
T T T
i i i
y b x b As q s
=
=
=
=
∑
qs
dove bTA=qT ; quindi y in base al suddetto teorema è più gaussiana delle singole si e
massimamente non gaussiana quando uguaglia una delle si a meno di un fattore
moltiplicativo (questo in condizioni ideali). Il problema è che per noi q è incognito, quindi dobbiamo lavorare su b variandolo fintanto che la distribuzione della y risulti la meno gaussiana possibile.
Senza entrare ulteriormente nel dettaglio, ci basta sapere che per la valutazione della non gaussianità di una variabile aleatoria esistono varie tecniche che si basano sul calcolo di cumulanti di ordine superiore oppure sul concetto di Entropia; le tecniche utilizzate negli algoritmi che implementano la ICA, tra cui il più diffuso e completo è sicuramente il “fastICA”, sono la Kurtosis, la Neg-Entropia e la minimizzazione della mutua informazione.
Infine per completezza occorre sottolineare che nella pratica, per rendere la stima delle IC più semplice e meglio condizionata, è necessario effettuare dei processi preliminari sui dati: il centramento e lo sbiancamento.
Centramento : data la matrice dei dati (in ogni caso organizzata in modo da avere i segnali studiati sulle righe ), si toglie da ogni riga il suo valor medio in modo da riportare i segnali tutti centrati intorno allo zero. Anche le componenti indipendenti stimate risulteranno centrate cosicché, per riottenere i valori fisici, dovremo sommarci i corrispondenti valori medi (riordinati secondo la matrice di unmixing).
Sbiancamento : consiste nell’applicare alla matrice dei dati X una trasformazione lineare che li renda incorrelati in modo che la loro matrice di covarianza diventi la matrice identità; questa trasformazione è sempre possibile e vi sono vari metodi per effettuarla. Quello classico è basato sulla decomposizione a valori singolari (SVD) della matrice di covarianza dei dati C=E{xxT}=EDET, dove E è la matrice ortogonale degli autovettori di C e D è la matrice diagonale degli autovalori. I dati sbiancati si ottengono con la trasformazione:
1 2 T
x
=
E D
⋅
−⋅
E
⋅
x
Quindi lo sbiancamento trasforma la matrice di mixing A in una nuova matrice tale che:
1 2 T
L’utilità di questo processo sta nel sostanziale dimezzamento dei parametri da stimare, essendo à una matrice ortogonale, quindi caratterizzata da n(n-1)/2 gradi di libertà contro gli n² parametri di A.
Alternativamente è possibile utilizzare come trasformazione di whitening la PCA che ci dà l’ulteriore possibilità di scegliere gli autovalori significativi e quindi ridurre la dimensione dei dati prima di procedere con la ICA.
I campi della ricerca dove l’analisi delle componenti indipendenti ha trovato applicazione sono fondamentalmente due [21]: la creazione di modelli computazionali e l’analisi dei dati biomedici.
In questo ultimo settore, una delle prime applicazioni ha riguardato l’analisi dei segnali EEG dove la ICA è servita per estrarre i segnali associati alla risposta visiva. Assumendo che i segnali rilevati da ogni elettrodo fossero una combinazione di componenti temporali indipendenti, la ICA è stata impiegata per individuare tali sorgenti sfruttando la loro indipendenza temporale; in questo senso si parla di
temporal ICA.
Più di recente questa analisi è stata impiegata nello studio delle immagini funzionali ricavate da fMRI; qui la sua funzione è stata quella di individuare set di sorgenti indipendenti con l’obiettivo di isolare le regioni che contenessero le attivazioni legate al particolare task [18].
L’applicazione della ICA ai dati fMRI ha visto un cambiamento di prospettiva nel suo impiego; infatti con gli altri tipi di dato l’approccio è stato classicamente quello
temporale mentre nel caso di dati fMRI l’analisi si è indirizzata nel senso spaziale;
questo perché le dimensioni spaziali di questi dati sono molto più larghe della loro dimensione temporale (da 5000 a 25000 voxel contro qualche centinaia di campioni temporali) e quindi la mole di calcoli è molto minore rispetto ad un approccio temporale; al contrario succede per esempio nei dati da EEG in cui si hanno molti più campioni temporali rispetto al numero di punti spaziali acquisiti.
L’applicazione della sICA ai dati fMRI segue tipicamente due strade nell’interpretazione dei risultati: la prima consiste nel basare la selezione di una particolare componente indipendente ipotizzando un modello della risposta legata all’attivazione [17], l’altra invece esamina le mappe spaziali usando delle conoscenze pregresse sulle strutture cerebrali interessate in quel particolare task. Il secondo metodo è più inquadrato in un’ottica di segregazione funzionale, con lo scopo di stabilire le funzioni di singole aree selezionate, mentre il primo si sviluppa in un quadro di ricerca di connettività funzionale ovvero di individuazione spaziale di tutte le regioni legate ad un comune evento di stimolazione.
Dal punto di vista funzionale le regioni coinvolte nell’espletamento di una particolare funzione psicomotoria sono dislocate in vari punti del cervello e fanno
Figura 3.12 : rappresentazione schematica della scomposizione in componenti indipendenti dal punto di vista temporale e spaziale.
parte di uno schema di connettività appunto funzionale. Quindi le aree coinvolte nello svolgimento di una stessa risposta sicuramente saranno incorrelate dalle altre regioni non coinvolte i cui segnali saranno dovuti ad altri fenomeni indipendenti dal task eseguito.
Sulla base di questa affermazione si capisce come la ICA sia uno strumento di ricerca di modelli di connettività estremamente valido. Ogni componente indipendente prodotta dalla scomposizione consiste in una distribuzione spaziale di voxel (mappa indipendente) cui è anche associata una serie temporale; questo segnale rappresenta l’insieme dei pesi con cui va pesata la singola mappa indipendente nella ricostruzione, istante per istante, dei singoli fotogrammi dell’acquisizione originaria (figura 3.13).
In questo modo attraverso la scomposizione dei dati in mappe spazialmente indipendenti si possono isolare quelle regioni accomunate da una stessa componente di segnale, regioni che rappresentano un modello di connettività funzionale.
Figura 3.13 : Schema di decomposizione dei dati secondo sICA. Ogni mappa costituisce una componente indipendente e contiene, per ogni voxel, il peso sul segnale originario della componente di segnale associato alla singola mappa.
III.2.2 Metodi di analisi di Connettività Effettiva: DCM
Nell’ambito degli studi di connettività effettiva il SEM ed il DCM costituiscono le due tecniche di modellizzazione più diffuse ed utilizzate. Entrambe si prefiggono l’obiettivo di identificare le relazioni (come pesi delle connessioni ipotizzate) che intercorrono tra le varie regioni che entrano nel modello, tuttavia si discostano molto l’una dall’altra per quanto riguarda le ipotesi di lavoro assunte e l’analisi statistica implementata.
In questo lavoro di tesi è stato deciso di approfondire ed utilizzare come metodo di analisi il SEM a cui verrà dedicato ampio spazio nel capitolo successivo; questa scelta è derivata da un’approfondita ricerca in letteratura [19,23,24] di informazioni inerenti le due metodiche che ci ha permesso di valutare a priori le possibilità di utilizzare l’una o l’altra nel nostro caso specifico.
E’ quindi assolutamente importante presentare il modello DCM, nei suoi aspetti teorici e applicativi, cercando di sottolineare i motivi che hanno portato a rinunciare al suo impiego in questo studio.
DCM : Dynamic causal Modelling
Lo scopo di questa procedura di modellizzazione è dedurre le connessioni tra le varie aree cerebrali e quantificare il peso di queste interazioni, in funzione del contesto sperimentale (nel caso di fMRI il contesto è determinato dal modello della risposta emodinamica). Il DCM rappresenta un notevole passo in avanti rispetto ai già esistenti approcci alla connettività effettiva perché implementa un modello generativo delle risposte cerebrali più plausibile e dettagliato che tiene conto della loro natura non lineare e dinamica.
L’idea di base è costruire un modello molto realistico delle interconnessioni neurali, andando a capire come le attività sinaptiche o neurali vengano trasformate nei segnali emodinamici che si rilevano come risposte. Questo è possibile ipotizzando