Capitolo 1
1.1 I cromosomi e l’informazione ereditaria
Verso la metà del XIX secolo, Gregor Mendel iniziò alcuni studi su incroci di piante di pisello, che lo portarono a concludere che alcuni distinguibili tratti dei piselli erano ereditati dalle piante che avevano prodotto il seme (i “genitori”) in maniera statisticamente prevedibile. Solo molto più recentemente è stato scoperto che l’acido desossiribonucleico (DNA) contiene i codici biochimici per l’ereditarietà che Mendel aveva osservato [1].
Il DNA che è associato con una specifica caratteristica o con una funzione è noto come gene. L’intero insieme di informazioni rappresentate nel DNA è noto come genoma ed è stato oggetto di importanti studi interdisciplinari negli ultimi decenni.
Nell’uomo, il DNA è “impacchettato” in 23 coppie di cromosomi presenti nel nucleo di ogni cellula del corpo umano e ciascun genitore contribuisce con un cromosoma per ciascuna coppia. I cromosomi sono indicati con le lettere X e Y e i numeri da 1 a 22. Nelle cellule normali vi sono due coppie di cromosomi numerati, per un totale di quarantaquattro
cromosomi detti ordinari o autosomi, e altri due cromosomi, detti sessuali, che determinano appunto il sesso dell’individuo (cromosoma X e Y): nel caso di sesso femminile si hanno due cromosomi X, nel sesso maschile un cromosoma X e uno Y.
Le deviazioni del DNA dalla “normalità” sono note come mutazioni; esse possono essere ereditate, oppure derivare da particolari condizioni ambientali. Alcune mutazioni hanno un’impatto con la salute. Per esempio la presenza di un terzo cromosoma 21 causa la sindrome di Down.
1.2 Primi studi sul DNA
Il primo a individuare il DNA è stato Friedrich Miescher nel 1869 durante i suoi studi sul nucleo [2]. Purtroppo né lo stesso Miescher né i suoi contemporanei seppero apprezzare il possibile ruolo del DNA (allora chiamato “nucleina”) nella trasmissione dei caratteri ereditari.
Solamente nel 1944 tre biologi americani, Osvald Avery, Colin McLeod e Maclyn McCarty [3], fecero la scoperta che più di ogni altra contribuì a identificare il DNA (acido desossiribonucleico) come costituente dei geni e che ha aperto l’era moderna della ricerca scientifica.
Essi identificarono un “fattore trasformante” batterico descritto per la prima volta da Frederick Griffith nel 1928 [4]. Griffith, svolgendo esperimenti sui batteri pneumococco, scoprì che iniettando in un topo di laboratorio pneumococchi virulenti ma “inattivi” insieme a pneumococchi
Figura 1.2: doppia elica del DNA come fu presentata da Watson e Crick nell'articolo pubblicato su Nature nel 1953.
vivi ed innocui, il topo moriva. Cosa era successo? Il batterio morto aveva fornito al batterio inattivo una qualche sostanza che lo aveva reso attivo e mortale.
Tale sostanza (il “fattore trasformante”) poteva dunque causare un cambiamento ereditario della cellula batterica e aveva le caratteristiche di un gene, ovvero di un elemento ereditario che determina le proprietà innate dell’organismo.
Avery, McLeod e McCarty furono i primi a dimostrare che tale sostanza ereditaria era l’acido desossiribonucleico (DNA).
Dopo che la sostanza ereditaria fu identificata, il passo successivo fu scoprire la sua struttura chimico-fisica e come questa determinasse la sua azione nell’ereditarietà e nelle funzioni cellulari.
Tali informazioni furono ottenute da Watson e Crick che nel 1953 pubblicarono, sulla rivista “Nature”, il loro modello strutturale [5].
Fondamentali per la realizzazione di tale modello furono gli studi di Wilkins e Franklin che, separatamente, fecero esperimenti di diffrazione dei raggi X su DNA cristallizzato.
Franklin e Wilkins ottennero (fig. 1.1) chiare figure di diffrazione che confermarono la regolarità delle molecole di DNA e fecero pensare che
esse fossero elicoidali.
Watson e Crick proposero nel loro modello una struttura con due catene elicoidali ciascuna avvolta a spirale sullo stesso asse (fig. 1.2).
Tale struttura contiene l’intuizione geniale delle due catene antiparallele ed è stata confermata in quasi tutte le sue componenti negli studi successivi.
1.3 La struttura del DNA
Secondo il modello di Watson e Crick, le molecole di DNA sono formate da due lunghi filamenti avvolti a spirale a formare una doppia elica.
In termini chimici, ciascun filamento del DNA è un polimero di
nucleotidi, ovvero è formato da molti nucleotidi che si susseguono. Ciascun
nucleotide è formato dall’unione di un nucleoside con un gruppo fosfato. Il nucleoside è la combinazione di uno zucchero a 5 atomi di carbonio (nel caso del DNA il desossiribosio) e di una delle quattro basi azotate tipiche degli acidi nucleici (comunemente note come basi). È possibile classificare le basi del DNA in pirimidine e purine a seconda della struttura chimica monociclica o biciclica, rispettivamente, che presentano (fig. 1.3) [6].
Sono pirimidine la citosina (indicata con C) e la timina (T), mentre sono purine la guanina (G) e l’adenina (A).
I nucleotidi possono essere considerati molecole con direzionalità perché essi hanno gruppi fosfato (PO4) e gruppi idrossilici (OH) attaccati al
Normalmente il DNA consiste in due catene portanti (o “scheletri”), avvolte a spirale, composte da unità alternate di acido fosforico e desossiribosio legate fra loro da elementi trasversali di basi di purina e pirimidina (attaccate all’atomo di carbonio 1´ di ogni zucchero).
L’informazione genetica della molecola è codificata dalla sequenza delle basi (A, T, C e G) nel DNA.
In una data cellula l’ordine con cui le basi sono disposte lungo la catena non è affatto casuale: l’ordine delle basi contiene il “programma” che regola e distingue forme di vita diverse.
Affinché il programma abbia un significato biologico, la sequenza di A, T, C e G deve essere opportuna a seconda dell’informazione da codificare.
I due filamenti sono legati fra loro tramite legami a idrogeno che si formano fra le basi poste sulle due catene che si trovano una di fronte
Figura 1.4: possibili accoppiamenti fra le basi. [11]
all’altra. L’accoppiamento fra le basi (“base pairing”, indicato con bp1) sulle catene polinucleotidiche
è univoco: l’adenina si accoppia solo con la timina e la guanina solo con la citosina (fig. 1.4).
Inoltre la coppia T-A è tenuta insieme da due legami a idrogeno mentre la coppia C-G da tre.
Tali considerazioni furono derivate da Watson e Crick da analisi chimiche fatte da Chargaff negli anni ’50.
Egli dimostrò esistere un rapporto quasi unitario fra le basi di purina e quelle di pirimidina e in più mostrò che il numero di basi di T è uguale a quello di A e il numero di basi di C è uguale a quello di G.
Grande intuizione di Watson e Crick fu affermare che, essendo possibile formare solo specifiche coppie di basi, data la sequenza di basi su una delle due catene, la sequenza sull’altra catena è determinata automaticamente. Tale proprietà viene espressa dicendo che le basi sui due filamenti sono complementari e sta alla base di tutti gli studi fatti sul DNA.
Nella doppia elica i due filamenti complementari sono allineati in modo “antiparallelo”: le due sequenze di basi accoppiate sono legate a scheletri zucchero-fosfato disposti in senso opposto (una cresce aggiungendo nucleotidi in posizione 5´, l’altra in posizione 3´).
1
Quando si definiscono le dimensioni di un DNA, si utilizza il numero di coppie di basi. Originariamente per definire le dimensioni di un DNA si utilizzava il termine coppie di basi (bp,
base pairs) o chilo-coppie di basi (kbp, kilobase pairs). Oggi, per un DNA a doppia elica, si
preferisce abbreviare il termine kbp con quello di chilobasi (kb). Per il DNA a singolo filamento si preferisce definire la dimensione in termini di nucleotidi (nt).
Gli anelli di atomi di ogni coppia T-A e C-G giacciono orizzontalmente in un piano perpendicolare all’asse della doppia elica, distanziati di 0.34 nm dalla coppia di basi successiva mentre il diametro della molecola è 2.4 nm. La lunghezza complessiva della molecola di DNA può variare da qualche micron, nel caso dei virus (procarioti), fino al caso limite di alcuni centimetri nel caso di molecole animali o vegetali (eucarioti).
Il numero delle basi nel DNA è variabile in funzione del tipo di organismo. Tali basi sono sistemate in specifiche sequenze il cui insieme è detto genoma.
Nella tabella 1 sono indicate le dimensioni (in milioni di basi) di alcuni genomi [7].
La doppia elica può essere separata nei due filamenti che la compongono sottoponendola a un trattamento che rompa i legami a idrogeno; questo si può ottenere o in condizioni di pH estremo (ovvero pH < 3 o pH >10 ) o aumentando opportunamente la temperatura, cioè fornendo calore
alla doppia elica. La temperatura alla quale i filamenti si separano è nota come “temperatura di scioglimento” (Tm) ed è funzione della sequenza di
nucleosidi; per esempio se sono presenti tante coppie C-G, Tm è più alta
rispetto al caso in cui le coppie T-A siano predominanti, in quanto nel primo caso è necessario rompere tre legami a idrogeno che tengono unite le basi mentre nel secondo caso i legami da rompere sono due.
TABELLA 1 [7] Dimensioni di genomi (milioni di basi) Procarioti Mycoplasma genitalium Escherichia coli Bacillus megaterium 0.58 4.64 30.1 Eucarioti Funghi Saccharomyces cerevisiae Aspergillus nidulans 12.1 25.4 Protozoi Tetrahymena pyriformis 190 Invertebrati Drosophila melanogaster Locusta migratoria 180 5000 Vertebrati Fugu rubripes Homo Sapiens Mus musculus Triturus vulgaris Necturus maculosus 400 3200 3300 50000 160000
Indicativamente Tm è circa uguale a 90ºC.
La separazione dei filamenti è detta denaturazione.
La struttura di DNA che Watson e Crick hanno individuato, che fino ad ora è stata illustrata, è la “forma B”. Nella cellula il DNA è presente sempre nella forma B. In verità la doppia elica si sta rivelando poliformica: accanto alla forme tradizionali A e B, con elica destrorsa, si sono venute caratterizzando forme come la G (presente nei telomeri dei cromosomi eucariotici e così chiamata per l’abbondanza di residui G in un filamento); la H, tripla elica composta da due filamenti di pirimidine e uno di purine alternate fra loro; la Z con la sua elica sinistrorsa e l’andamento a zig-zag del legame fosfodiesterico [2].
1.4 La replicazione del DNA
A partire dal 1980, sistemi multiproteine in vitro hanno reso possibile una caratterizzazione dettagliata del meccanismo di replicazione e la soluzione del problema della sintesi di una nuova catena del DNA [8].
Il problema da risolvere era quello di capire come il DNA che forma un cromosoma potesse essere copiato per formare un nuovo cromosoma identico ogni volta che la cellula si riproduce.
Come precedentemente detto, il modello di Watson e Crick è basato su due filamenti di DNA appaiati che sono complementari nella loro sequenza nucleotidica.
Durante la sintesi di una nuova catena, una porzione di filamento preesistente si separa dal filamento complementare e funziona da stampo per la definizione dell’ordine in cui debbono essere inseriti i nuovi nucleotidi della catena in formazione (fig. 1.5). Il risultato finale sarà la nascita di due molecole figlie identiche sia fra loro che alla molecola madre da cui sono state generate. Tale processo è detto semiconservativo in quanto ogni nuova molecola è formata da un filamento completo della molecola originaria e da uno nuovo.
La replicazione semiconservativa comprende le seguenti fasi:
• rottura dei legami a idrogeno e svolgimento progressivo delle due catene polinucleotidiche (tale azione è svolta da enzimi); • le basi interagiscono con nucleotidi trifosfati (dATP, dTTP,
dGTP e dCTP) che hanno lo scopo di fornire i nucleosidi necessari alla costruzione delle nuove molecole;
• sintesi delle due nuove molecole di DNA .
Fondamentale per la sintesi è l’azione della molecola DNA polimerasi che permette l’aggiunta di nucleotidi, uno alla volta, alla terminazione del carbonio 3´ della nuova molecola, catalizzando quindi la crescita del filamento nella direzione chimica da 5´ a 3´ (copiando la catena parentale che va da 3´ a 5´).
È da notare che una delle due nuove catene (“ladding strand”) deve crescere nella direzione che va da 3´ a 5´ (in quanto copia del filamento che va da 5´ a 3´ ) e tale crescita non può essere catalizzata dalla sola DNA polimerasi.
Si è dimostrato che anche in questo secondo caso l’azione di crescita è promossa dalla DNA polimerasi che produce una lunga serie di piccoli frammenti (detti frammenti di Okasaki) tutti nella direzione che va da 5´ a 3´ che sono successivamente uniti dall’azione dell’enzima DNA ligasi per formare un unico filamento continuo di DNA.
Il procedere della replicazione fa si che le due catene polinucleotidiche diventino quattro e formino con le porzioni non ancora replicate una Y. La conformazione assunta prende il nome di forcella di replicazione e il punto di unione fra le porzioni della molecola già replicate e quelle da replicare è detto punto di crescita.
Nelle forcelle di replicazione formate nei sistemi artificiali, che hanno permesso lo studio di tale meccanismo, la velocità di replicazione è paragonabile a quella della cellula (da 500 a 1000 nucleotidi al secondo) e il modello di DNA è ricopiato con una notevole fedeltà.
1.5 La ricombinazione del DNA
Per ricombinazione si intende una varietà di processi che nella cellula portano a un riordinamento dell’informazione genetica.
La ricombinazione di DNA omologhi, richiede uno scambio di materiale genetico tra due doppie eliche di DNA, o segmenti della stessa molecola, che possiedono un’ampia regione di sequenze omologhe, ovvero le molecole che si ricombinano devono avere una notevole somiglianza nella sequenza di nucleotidi (ricombinazione omologa).
L’evento iniziale è una rottura della doppia elica. Essa è in alcuni casi provocata appositamente dalla cellula per iniziare una ricombinazione (per esempio nella ricombinazione programmata dei linfociti), ma può derivare anche da diverse cause esterne come radiazioni ionizzanti, agenti chimici o stress. In quest’ultimo caso, la cellula utilizza la ricombinazione per riparare un danno estremamente grave. Infatti, per una cellula in proliferazione, la presenza anche di una sola rottura della doppia elica non riparata risulterebbe letale.
Dopo che le catene sono state separate, si può procedere alla formazione di una unione eterodoppia (“eteroduplex”), ovvero una unione che incolla le due catene precedentemente separate: una catena proveniente da una elica deve formare coppie di basi con una catena proveniente da una seconda elica. In tale operazione interviene un enzima (Rec A nell’Escherichia Coli e Rad 51 nell’uomo) che catalizza la reazione di scambio dei filamenti di DNA2.
2 Per ulteriori approfondimenti sul meccanismo molecolare della ricombinazione e sul modello di
Da rimarcare che, oltre all’esistenza della ricombinazione omologa, esistono anche altre forme differenti di ricombinazione [9]:
• processo di ricombinazione sito-specifica, in cui è necessario solo un breve tratto di omologia, spesso inferiore a 15 bp, e gli enzimi coinvolti agiscono solo su queste sequenze;
• nella trasposizione, particolari sequenze di DNA, note come trasposoni, vengono inserite in una sequenza genomica, quasi indipendentemente dalla presenza di omologia di sequenza sul DNA nel quale avviene l’insersione. La trasposizione è un meccanismo attraverso il quale il materiale genetico può essere spostato da una posizione cromosomica a un’altra;
• la ricombinazione illegittima (rara), che avviene fra DNA non omologhi, indipendentemente da qualsiasi elemento di sequenza specifico.
1.6 Geni
e
proteine
Le proteine sono la parte essenziale di tutte le cellule, siano esse animali o vegetali, e il nostro corpo è fatto essenzialmente di proteine.
Nell’uomo sono presenti oltre 50000 differenti proteine e i loro compiti sono di primaria importanza.
Per esempio le proteine:
• modulano l'espressione dei geni e intervengono nella duplicazione, trascrizione e traduzione del DNA;
• regolano il metabolismo, come enzimi e come ormoni;
• trasportano svariate molecole attraverso i liquidi circolanti e attraverso le membrane cellulari;
• intervengono nella coagulazione del sangue; • proteggono l'organismo dalle infezioni (anticorpi); • danno luogo a strutture contrattili (muscoli e tendini);
• partecipano alla generazione e alla trasmissione degli impulsi nervosi;
• costituiscono la struttura dei tessuti di sostegno animali.
Le proteine sono delle macromolecole, cioè lunghe catene di atomi, che contengono carbonio, idrogeno, ossigeno, azoto e spesso zolfo, fosforo e metalli (come ferro e rame). Una proteina può essere costituita da una sola catena di atomi o da più di una catena, come nel caso dell’emoglobina [10]. Ciascuna di queste catene è detta catena polipeptidica e consiste in una successione di qualche decina o qualche centinaio di unità base dette
aminoacidi, tenute insieme da un legame chimico specifico, detto legame
peptidico (―NH―CO―), ottenuto per condensazione tra gruppi carbossilici (―COOH) e amminici (―NH) presenti su ciascun aminoacido.
Vi sono in tutto venti diversi tipi di aminoacidi (tabella 2) presenti nelle varie proteine e quindi si osserva che anche le catene proteiche si presentano come ripetizioni abbastanza monotone delle stesse lettere.
L’ordine degli aminoacidi di una proteina non è casuale, ma è una sequenza precisa e caratteristica di ciascuna proteina, definita dalla sequenza di basi degli acidi nucleici di un unico segmento in un filamento di DNA, ovvero da un gene.
Il ruolo del gene che codifica una determinata proteina è quello di specificare la natura e l’ordine degli aminoacidi che la compongono.
I geni variano in lunghezza fra loro e spesso sono composti da migliaia di basi; solo una parte di queste sequenze di basi (dette esoni) è in grado di codificare le proteine, la restante parte apparentemente non codificante (introni) è oggetto di studi ed è ipotizzabile avere un ruolo decisivo di controllo (per esempio gli introni sembrano regolare le interazioni fra geni diversi3) [11].
1.7 La sintesi delle proteine
Nelle cellule, la molecola di DNA non deve uscire dal nucleo per evitare traumi che potrebbero determinare un cambiamento nella sua sequenza e che quindi comprometterebbero la stabilità del genoma; inoltre
3 Lo studio di tali regioni di controllo rappresenta gran parte del lavoro sperimentale fatto dal
biologo molecolare al giorno d’oggi. Esse permettono l’espressione coordinata fra i vari geni e quindi in pratica l’organizzazione della cellula e della vita.
TABELLA 2 [10]
Nome aminoacido Simboli
Acido aspartico Asp (D) Acido glutammico Glu (E) Alanina Ala (A) Arginina Arg (R) Asparagina Asn (N) Cisteina Cys (C) Fenilalanina Phe (F) Glicina Gly (G) Glutammina Gln (Q) Isoleucina Ile (I)
Istidina His (H) Leucina Leu (L) Lisina Lys (K) Metionina Met (M) Prolina Pro (P) Serina Ser (S) Tirosina Tyr (Y) Treonina Thr (T) Triptofano Trp (W) Valina Val (V)
il DNA è impossibilitato a uscire dal nucleo a causa delle sue notevoli dimensioni.
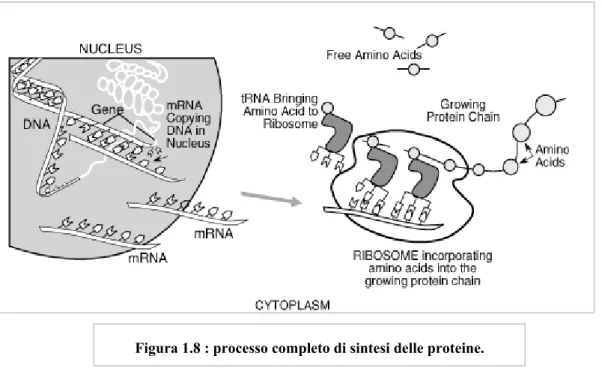
La fabbricazione delle proteine però avviene nel citoplasma in apposite strutture, i ribosomi, per cui l’informazione contenuta nel DNA deve essere portata al di fuori del nucleo.
La soluzione trovata dalla natura per superare tale difficoltà è stata quella di creare una struttura identica al DNA da un punto di vista funzionale ma di differente composizione chimica.
Tale copia è un altro acido nucleico chiamato acido ribonucleico (RNA).
Le catene di acido ribonucleico sono, come nel DNA, polimeri di nucleotidi con uno scheletro formato da zuccheri e fosfati a cui è attaccata una base.
Differenze fra le strutture RNA e DNA (fig. 1.6):
• nell’RNA lo zucchero è il ribosio (invece del desossiribosio presente nel DNA);
• le basi nucleotidiche che costituiscono l’RNA sono l’adenina, la guanina, la citosina (come nel DNA) e l’uracile (U), che sostituisce la timina presente nel DNA;
• le molecole di RNA sono normalmente a singolo filamento (struttura monomerica), anche se in alcuni casi sequenze complementari possono appaiarsi (per esempio nei virus), mentre il DNA è a doppio filamento.
Da un punto di vista funzionale, è possibile distinguere tre differenti tipi di RNA, ciascuno adibito a uno specifico ruolo e ugualmente indispensabile nella sintesi proteica: l’RNA messaggero (mRNA), l’RNA transfer (tRNA) e l’RNA ribosomiale (rRNA).
L’RNA messaggero viene prodotto nel nucleo cellulare sullo stampo di un gene. Tale processo, detto di trascrizione, è simile alla duplicazione e avviene dopo la separazione delle due eliche costituenti il DNA (azione svolta dall’enzima DNA polimerasi) e si realizza esclusivamente sul filamento che ha direzione che va da 5´ a 3´ (fig. 1.7).
I tratti di DNA esposti funzionano da stampo ordinando la sequenza dei nucleotidi in formazione nell’mRNA.
La regola di appaiamento risulta: • A se sul DNA è presente T; • G se è presente C (e viceversa); • uracile (U) se è presente A.
Sempre nel nucleo, l’mRNA “primitivo” appena costituitosi, viene sottoposto a un rimaneggiamento (detto splicing) che consiste nell’eliminazione degli introni. A questo punto l’mRNA “maturo” viene trasferito nel citoplasma e sistemato sui ribosomi dove viene letto (fig. 1.8).
Gli RNA transfer permettono la traduzione dell’informazione genetica nelle proteine in quanto sono in grado di trasportare sui ribosomi gli aminoacidi che occorrono per la sintesi. Infatti in base alle sequenze di nucleotidi dell’mRNA, esistono tRNA specifici che trasportano sull’mRNA gli aminoacidi corrispondenti (fig. 1.9).
L’RNA ribosomiale rappresenta la frazione di RNA sintetizzata nel nucleolo che forma lo scheletro dei ribosomi. L’rRNA è quindi il principale costitituente dei ribosomi e ha una ruolo sia strutturale che catalitico nella sintesi proteica.
1.8 Il codice genetico
L’informazione per la sintesi di una catena proteica è contenuta all’interno di un gene. Una data sequenza di basi del DNA (detta regione codificante del gene) custodisce il messaggio biologico che permette la realizzazione di sequenze di aminoacidi che costituiscono la proteina.
La conversione del messaggio genetico dal linguaggio dei nucleotidi a quello degli aminoacidi, prende il nome di traduzione e avviene secondo un codice stabilito detto codice genetico; esso è universale, cioè è valido per tutti gli organismi, dagli animali alle piante, ai batteri. Sono sufficienti tre nucleotidi del DNA per definire un aminoacido (secondo la corrispondenza imposta dal codice genetico); tale tripletta di nucleotidi è anche detta
codone nell’mRNA (fig. 1.9).
Ogni molecola di tRNA ha nella sua sequenza di basi un
anticodone, cioè una sequenza
complementare a un particolare codone. In questo modo l’mRNA può “chiamare” un dato aminoacido presentando un codone che viene “agganciato” dal codone complementare di una molecola di tRNA a cui è
attaccato l’aminoacido cercato.
Dal momento che esistono quattro tipi di basi (A, U, C e G) e che un aminoacido viene specificato dalla sequenza di tre basi, in totale esistono 43 possibili codoni, ovvero 64 possibili aminoacidi diversi.
Poiché però gli aminoacidi sono solo venti, si pone un problema di decifrazione del codice che ha impegnato numerosi studiosi (fra i quali lo stesso Crick che per primo formulò l’ipotesi che fossero necessarie tre basi per specificare un aminoacido) a cavallo degli anni ´50 e ´60 [12].
La comprensione del codice avvenne nel 1966 grazie alla collaborazione di numerosi ricercatori, fra i quali Niremberg, Ochoa e Mathaei, che individuarono quali codoni determinavano ciascuno dei venti aminoacidi (tabella 3). Nel corso dell’evoluzione, la natura ha risolto
l’eccesso di codoni rispetto al numero di aminoacidi con una
degenerazione del codice, ovvero un
codone codifica un solo aminoacido, ma lo stesso aminoacido può essere codificato da un certo numero di codoni diversi. In nessun caso comunque il codice risulta ambiguo. Il codice sarebbe ambiguo se lo stesso codone potesse codificare più di un
aminoacido, ma ciò non accade: ogni codone codifica sempre e solo un
aminoacido.
Per esempio, i sei codoni UUA, UUG, CUU, CUC, CUA e CUG indicano l'aminoacido leucina, i due codoni AGU e AGC corrispondono alla serina, mentre il solo codone UGG è attribuito al triptofano.
Esistono anche tre particolari codoni detti di STOP (dalla tabella essi sono: UAA, UAG e UGA), che sono necessari in quanto indicano la fine di una regione codificante (sono anche detti codoni di non senso). Se il codone di STOP non fosse presente al termine della regione codificante del gene, il messaggio genetico non potrebbe dirsi terminato e non svolgerebbe correttamente la sua funzione. Analogamente, ogni regione codificante ha un codone di inizio (AUG), che corrisponde all’aminoacido metionina.
1.9 Mutazioni
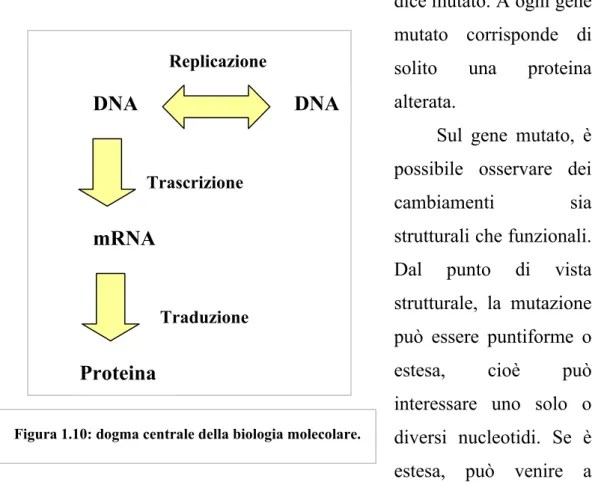
Abbiamo visto come si compie il ciclo fondamentale dell’informazione biologica, con il passaggio del messaggio genetico dal DNA all’RNA e da questo alla proteina. Tale flusso è anche detto dogma
centrale della biologia molecolare (fig. 1.10).
È possibile che un determinato nucleotide o più nucleotidi in un gene cambino natura? Se ciò accadesse, quali potrebbero essere le conseguenze nel nostro organismo?
L’alterazione di un nucleotide, o di alcuni nucleotidi, all’interno di un gene è un fenomeno possibile ed è detto mutazione e il gene in questione si dice mutato. A ogni gene mutato corrisponde di solito una proteina alterata.
Sul gene mutato, è possibile osservare dei
cambiamenti sia strutturali che funzionali.
Dal punto di vista strutturale, la mutazione può essere puntiforme o estesa, cioè può interessare uno solo o diversi nucleotidi. Se è estesa, può venire a mancare parte di un gene o addirittura un gene intero. In questi casi si parla di delezione parziale o totale e la proteina corrispondente può mancare del tutto o essere stravolta nella sua composizione e quindi nella sua funzione.
È anche possibile, viceversa, che una porzione del gene compaia ripetuta più di una volta: anche in questo caso il messaggio genetico viene sconvolto.
Consideriamo, come esempio, una mutazione puntiforme (fig. 1.11). Tale alterazione può consistere in una inserzione, in una delezione o in una sostituzione di un singolo nucleotide nella regione codificante di un gene. L’effetto di una inserzione o di una delezione è quello di una traslazione della sequenza delle basi di una posizione in avanti o indietro, rispettivamente (fig. 1.11.A e 1.11.B). Ciò comporta lo sconvolgimento
DNA DNA Replicazione mRNA Proteina Trascrizione Traduzione
della sequenza stessa e l’alterazione della lettura del messaggio genetico da quel punto in poi. Si osserva infatti che la successione delle triplette successive all’inserzione del nucleotide (C nell’esempio) o alla delezione del nucleotide (A) è differente da quella delle triplette nel caso normale.
La sostituzione di un nucleotide può causare tre effetti abbastanza diversi. Nel caso di mutazione sinonima la tripletta mutata codifica lo stesso aminoacido della tripletta iniziale. In tali casi ci si accorge dell’avvenuta mutazione solo se si analizza la sequenza nucleotidica. In figura 1.11.C si osserva che la modifica della tripletta GCA in GCG non crea problemi perché l’aminoacido codificato è sempre l’alanina.
Le mutazioni di senso sono quelle in cui la sostituzione di un nucleotide altera effettivamente la sequenza degli aminoacidi e quindi la
A T T G C A T C G A C C T T G A C A T T G C A C T C G A C C T T G A A T T G C T C G A C C T T G A C Inserzione di un nucleotide (C) Delezione di un nucleotide (A) A T T G C G T C G A C C T T G A C Sostituzione di un nucleotide (A con G)
A
B
C
Figura 1.11: esempio di mutazione puntiforme. [10]
Frazione di gene in condizioni normali
composizione della proteina corrispondente, causando spesso un effetto sulla salute del portatore.
Infine si hanno le mutazioni non senso o di terminazione in cui l’effetto della sostituzione è quello di trasformare una tripletta normale che codifica un aminoacido in una tripletta di STOP. La proteina risulterà quindi troncata in quel punto e potrebbe non svolgere le sue funzioni. Tale mutazione può essere deleteria.
Nella maggior parte dei casi, le mutazioni hanno un’origine spontanea, nascendo senza motivo e senza una causa specifica per un errore che ha luogo al momento della replicazione del DNA (fig. 1.10). Tale meccanismo infatti non è esente da errori anche se essi si presentano con una frequenza molto bassa (in media viene inserito un nucleotide sbagliato su 1 miliardo). In alcuni casi agenti ambientali (per esempio raggi ultravioletti e radiazioni) possono elevare il tasso di mutazione.
Da un punto di vista funzionale, si possono distinguere gli effetti delle varie mutazioni come:
• assenza di un qualsiasi effetto;
• riduzione più o meno grave di una funzione; • esagerazione di una funzione;
• creazione di una funzione totalmente nuova e diversa.
A esclusione del primo tipo di mutazioni che non producono alcun effetto, le altre producono una riduzione parziale o totale di una funzione. Per esempio, la proteina codificata da un gene mutato può non essere in grado di svolgere del tutto il proprio compito.
Si definisce allele ciascuna delle varie forme che può assumere un gene. In molti casi si può distinguere un allele normale e una serie di alleli mutati; in altri casi molti alleli possono essere considerati normali.
Non tutte le mutazioni hanno un effetto immediato sul nostro corpo: possediamo infatti due copie di ogni gene, una su ciascuna delle due copie
del cromosoma corrispondente. Se entrambe le copie dello stesso gene presenti nel nostro corpo sono mutate, è molto probabile che la nostra salute risenta dell’effetto di tale mutazione. Nel caso in cui è mutata una sola copia del gene, la mutazione si dice dominante se provoca effetti sull’organismo, mentre si dice recessiva se deve essere presente in entrambe le copie del gene per far sentire il suo effetto. La maggior parte delle mutazioni note sono recessive; molte mutazioni dominanti però sono così deleterie per l’organismo che quando sono presenti in duplice copia risultano letali.
Una persona che possiede due copie identiche di un determinato gene, entrambe normali o entrambe mutate secondo la stessa mutazione, si dice
omozigote (per quella determinata mutazione di quel determinato gene).
Viceversa, una persona che possiede due copie diverse dello stesso gene (una normale e una mutata) è detta eterozigote (per quella determinata mutazione di quel determinato gene). Osservando un individuo normale non potremo mai sapere con certezza se si tratta di un omozigote normale oppure di un individuo portatore nel suo corredo genetico di una mutazione recessiva in una delle due copie di un determinato gene. Per appurarlo occorre analizzare a fondo il suo patrimonio genetico.
Si definisce fenotipo l’insieme delle caratteristiche biologiche palesi di un individuo e genotipo la sua effettiva costituzione genetica. Fra genotipo e fenotipo non sempre c’è una corrispondenza diretta.
Riferimenti bibliografici capitolo 1
[1] J.Patrick Fitch, Bahrad Sokhansanj, “Genomic Engineering: Moving Beyond DNA Sequence to Function”, Proceedings of the IEEE, Vol.88, No.12, pp 1949-1971, dicembre 2000.
[2] Vittorio Sgaramella, “Origine della vita e controllo dell’evoluzione”, Le Scienze dossier, Numero 15-Pimavera 2003, pp 16-21.
[3] Osvald T.Avery, Colin M. MacLeod, Maclyn McCarty, “Studies on the chemical of the substance inducing transformation of pneumococcal types”, The Journal of the Experimental Medicine, Vol. 79, No. 2, pp 137-158, 1 febbraio 1944.
[4] Lubert Stryer, “Biochimica”, Ed. Zanichelli, dicembre 1996.
[5] J.D.Watson, F.H.C.Crick, “A structure for Deoxyribose Nucleic Acid”, Nature, Vol. 171, pp 737-738, 2 aprile 1953.
[6] Richard P.Bowater, “DNA Structure”, Encyclopedia of the Human Genome/2003 MacMillan Publishers Ltd, Nature Publishing Group/
http://www.ehgonline.net/dnastructure.pdf.
[7] Carlo Alberto Redi, Maurizio Zuccotti, Silvia Garagna, “L’altro Genoma”, Le Scienze dossier, Numero 15-Pimavera 2003, pp 44-51.
[8] Bruce Alberts, “DNA replication and recombination”, Nature, Vol. 421, pp 431-435, 23 gennaio 2003.
[9] Reginald H.Garrett, Charles M.Grisham, “Biochimica”, Ed. Zanichelli, 1998
[10] Edoardo Boncinelli, “I nostri geni”, Einaudi Tascabili. Saggi 507, 1998.
[11] Primer on Molecular Genetics, DOE Human Genome 1991-92 Program Report, giugno 1992.
[12] Brian Hayes, “The invention of the Genetic Code”, American Scientist, Vol. 86, No. 1, pp 8-14, gennaio-febbraio 1998.
Tabelle:
Tabella 1: Carlo Alberto Redi, Maurizio Zuccotti, Silvia Garagna, “L’altro Genoma”, Le Scienze dossier, Numero 15-Pimavera 2003, pag. 48. Tabella 2: Edoardo Boncinelli, “I nostri geni”, Einaudi Tascabili.
Saggi 507, 1998, pag.20.
Tabella 3: Edoardo Boncinelli, “I nostri geni”, Einaudi Tascabili. Saggi 507, 1998, ricavata da pag. 22.
Figure:
Fig. 1.1: http://sigu.univr.it/sigu/sigu_didattica/stampa.php?sbj=dnastory. Collegato l’1 settembre 2003.
Fig. 1.2: J.D.Watson, F.H.C.Crick, “A structure for Deoxyribose Nucleic Acid”, Nature, Vol. 171, 2 aprile 1953, pag. 737.
Fig. 1.3: P.Bowater, “DNA Structure”, Encyclopedia of the Human Genome/2003 MacMillan Publishers Ltd, Nature Publishing Group.
Fig. 1.4: Primer on Molecular Genetics, DOE Human Genome 1991-92 Program Report, giugno 1992, elaborata da pag.6.
Fig. 1.5: elaborata da Bruce Alberts, “DNA replication and recombination”, Nature, Vol. 421, pp 431-435, 23 gennaio 2003.
Fig. 1.7: elaborata da considerazioni di Lubert Stryer, “Biochimica”, Ed. Zanichelli, dicembre 1996 e di Edoardo Boncinelli, “I nostri geni”, Einaudi Tascabili. Saggi 507, 1998.
Fig. 1.8: U.S. Department of Energy Human Genome Program.
http://www.ornl.gov/hgmis/graphics/slides/sciexepression.html.
Fig. 1.9: elaborata da Lubert Stryer, “Biochimica”, Ed. Zanichelli, dicembre 1996.
Fig. 1.10: elaborata da considerazioni di: Edoardo Boncinelli, “I nostri geni”, Einaudi Tascabili. Saggi 507, 1998.
Fig. 1.11: Edoardo Boncinelli, “I nostri geni”, Einaudi Tascabili. Saggi 507, 1998, ricavata da pag. 34.
![Figura 1.3 : struttura chimica del DNA. [6]](https://thumb-eu.123doks.com/thumbv2/123dokorg/5635264.69267/5.892.166.773.383.865/figura-struttura-chimica-dna.webp)
![Figura 1.4: possibili accoppiamenti fra le basi. [11]](https://thumb-eu.123doks.com/thumbv2/123dokorg/5635264.69267/6.892.171.769.184.633/figura-possibili-accoppiamenti-fra-basi.webp)
![Figura 1.5 : fasi del meccanismo di replicazione del DNA. [8]](https://thumb-eu.123doks.com/thumbv2/123dokorg/5635264.69267/10.892.90.825.334.925/figura-fasi-meccanismo-replicazione-dna.webp)
![Figura 1.6 : confronto fra le molecole di RNA e DNA. [4]](https://thumb-eu.123doks.com/thumbv2/123dokorg/5635264.69267/15.892.206.726.704.1139/figura-confronto-fra-le-molecole-di-rna-dna.webp)


![Tabella 3: il codice genetico [10]](https://thumb-eu.123doks.com/thumbv2/123dokorg/5635264.69267/19.892.166.771.531.1104/tabella-il-codice-genetico.webp)