Introduzione
1.2.a La struttura delle proteine della famiglia otd/otx
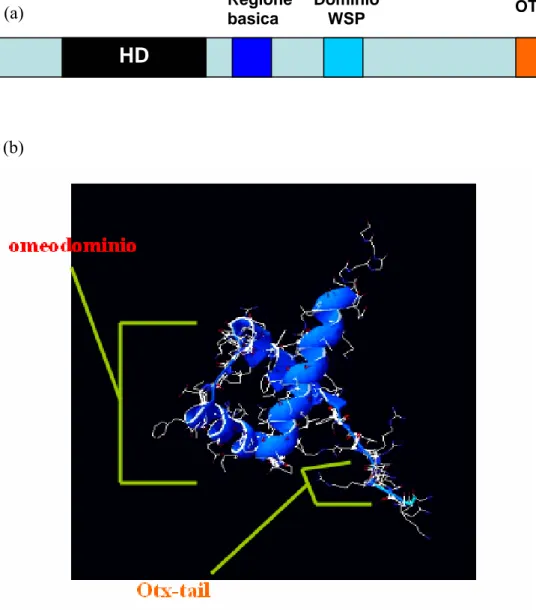
Le proteine della famiglia otd/otx sono fattori di trascrizione che presentano domini funzionali caratteristici. Sono presenti un omeodominio della classe bicoid, che determina la specificità di legame, una regione basica, un dominio WSP e un motivo amminoacidico caratteristico che viene detto otx-tail (Fig. 2a, 2b).Regione basica Dominio WSP OTX-tail (a) HD (b)
Figura 2: (a) Schema di una proteina della famiglia otd/otx con i domini tipici che la caratterizzano. Alcune
proteine della famiglia possiedono una ripetizione in tandem della otx-tail. (b) Rappresentazione della struttura tridimensionale di una proteina della famiglia otd/otx. Sono evidenziati l’omeodominio e la otx-tail.
Introduzione
L’omeodominio è una regione di 61 amminoacidi codificata da una sequenza di nucleotidi detta “homeobox”. Questa regione viene così chiamata perché è stata inizialmente caratterizzata nei geni Hom di Drosophila, le cui mutazioni determinano trasformazioni omeotiche, cioè cambiamenti nell’identità di un segmento corporeo in quella di un altro. L’omeodominio ha una struttura tridimensionale del tipo elica-giro-elica, formata da tre α-eliche, in cui l’elica 3 entra nel solco maggiore del DNA ed è responsabile del maggior numero di contatti con esso. In questo modo l’elica 3 garantisce la specificità di legame al DNA. I geni contenenti un homeobox sono stati distinti in due ampie classi: la classe dei geni HOX e quella dei geni correlati al gene
paired (prd) (Galliot et al., 1999; Galliot e Miller, 2000). Esistono tre tipi di
proteine Prd, che vengono distinte in base all’amminoacido posto in posizione 50 dell’omeodominio. Questo amminoacido è particolarmente importante perché da esso dipende l’affinità per la sequenza di riconoscimento sul DNA (Galliot et al., 1999). La classe Paired (cui appartengono, per esempio, i geni
pax) ha una serina in posizione 50 (S50) dell’omeodominio ed un secondo
dominio di legame al DNA, detto PAIRED domain, formato da 128 amminoacidi; esso è bipartito in due motivi HTH (un dominio PAI N-terminale ed uno RED C-N-terminale), in grado di legare il DNA, anche in modo cooperativo con l'omeodominio. Il secondo sottogruppo è costituito dai geni
paired-like, della classe di Bicoid, caratterizzati dalla presenza di una lisina in
posizione 50 (K50). Infine, i geni Q50 paired-like codificano per un
omeodominio con un residuo di glutammina nella solita posizione (Jun e Desplan, 1996). Nell’omeodominio delle proteine della classe otd/otx, la presenza di una lisina in posizione 50, corrispondente alla posizione 9 dell’elica di riconoscimento, è in grado di conferire un’alta affinità di legame al DNA verso la sequenza target TAATCC/T (Briata et al., 1999). A valle dell’omeodominio si trova una regione basica, che presenta numerosi residui di
Introduzione
lisina ed arginina, seguita da un motivo WSP. In Crx il motivo WSP contribuisce all’attivazione di geni target (Chau et al., 2000). Infine la porzione più vicina al C-terminale contiene un dominio che è comune a pressoché tutti i geni otd/otx, che per questo viene detto otx-tail (Furukawa et al., 1997). Questa regione nelle proteine otx dei vertebrati è ripetuta in tandem (Furukawa et al., 1997), mentre è assente in otd (Muller et al., 1999).