4. R
ISULTATI.
4.1. C
ONFRONTO DELLʼ
EFFICIENZA DI ESTRAZIONE E DELLʼ
EFFICIENZA DI AMPLIFICAZIONE DELLʼRNA
VIRALE ESTRATTO DAI CAMPIONI MEDIANTE DUE DIFFERENTI KIT COMMERCIALI.
Dal confronto tra le efficienze di estrazione dei campioni dellʼRNA virale estratto dagli epatopancreas delle diverse specie di molluschi bivalvi, addizionati con 10,0 μl della sospensione del controllo di processo (FCV), e di quella di amplificazione, ottenuta mediante contaminazione con 1,0 μl di RNA di NoV GII, è emerso quanto segue:
• Mytilus edulis
La media dellʼefficienza di estrazione dellʼRNA virale con il Kit 1 (QIAamp Viral RNA Mini Kit) su 24 campioni di M. edulis è stata del 15,80 (± 17,37) %, mentre la media dellʼefficienza di estrazione dellʼRNA virale sugli stessi campioni con il Kit 2 (NucliSENS® MiniMAG®) è stata del 20,41 ± 36,05 %. Lʼanalisi statistica non ha rilevato differenze significative tra i due Kit (P = 0,5753).
Per quanto riguarda lʼefficienza di amplificazione, i campioni di RNA virale estratto con il Kit 2 presentano unʼefficienza più alta (105,51 ± 40,13 %) rispetto a quelli estratti con il Kit 1 (71,18 ± 55,72 %) e la differenza tra le due è risultata significativa (P < 0,05).
I risultati dell'efficienza di estrazione e dellʼefficienza di amplificazione per la specie M. edulis sono riportati in tabella:
! "#! Codice % Efficienza Estrazione Kit 1 % Efficienza Estrazione Kit 2 % Efficienza Amplificazione Kit 1 % Efficienza Amplificazione Kit 2 1 2,66 0,00 74,77 65,98 2 3,60 0,29 163,58 89,50 3 7,94 0,07 125,70 92,02 4 16,16 0,40 77,91 50,35 5 13,22 0,13 184,03 82,36 6 24,74 26,90 0,00 52,85 18 39,25 49,85 117,28 174,11 19 67,87 164,24 93,95 186,61 20 42,36 72,49 140,44 131,95 21 0,03 0,40 17,43 126,58 22 23,73 19,69 35,60 133,79 23 36,22 28,83 30,35 175,32 24 27,64 19,29 56,25 80,66 25 1,97 28,04 9,81 57,04 26 24,74 35,74 0,60 158,01 30 0,02 18,25 143,40 135,66 33 21,54 21,11 35,60 127,46 63 2,50 0,00 9,47 61,99 64 1,29 1,65 81,23 95,93 65 6,61 0,04 72,20 77,92 66 2,14 0,04 66,43 104,97 67 7,09 0,66 42,04 89,50 69 0,00 0,88 0,00 86,45 70 5,79 0,74 130,17 95,26 Media 15,80 20,41 71,18a 105,51b DS 17,37 36,05 55,72 40,13 !
a, b: P < 0.05. Kit 1: QIAamp Viral RNA Mini Kit (Qiagen); Kit 2: NucliSENS® MiniMAG® (bioMerieux).
• Donax trunculus
Lʼanalisi statistica del confronto tra la media dellʼefficienza di estrazione dellʼRNA virale su 24 campioni di D. trunculus con i due diversi kit commerciali ha evidenziato una differenza significativa (P < 0,05), dove il Kit 2 ha una media più alta (12,44 ± 15,71 %) rispetto al Kit 1 (3,89 ± 8,88 %).
Anche per quanto riguarda lʼefficienza di amplificazione, il Kit 2 ha mostrato unʼefficienza media maggiore (54,74 ± 39,69 %) rispetto a quella del Kit 1 (13,98 ± 29,66 %) ed in questo caso lʼanalisi statistica ha evidenziato che tra le due medie esiste una differenza altamente significativa (P < 0,01).
I risultati dell'efficienza di estrazione e dellʼefficienza di amplificazione per la specie D. trunculus sono riportati in tabella:
Codice % Efficienza Estrazione Kit 1 % Efficienza Estrazione Kit 2 % Efficienza Amplificazione Kit 1 % Efficienza Amplificazione Kit 2 7 0,04 59,70 0,00 15,28 8 0,46 50,20 0,01 14,66 9 0,03 3,18 0,00 0,63 10 0,19 0,00 0,02 0,00 11 1,25 12,64 0,02 3,82 12 23,50 1,11 4,90 2,82 13 8,03 23,91 3,77 49,65 14 18,57 16,33 46,33 31,43 15 34,65 1,07 109,40 131,95 16 0,98 18,00 0,02 98,62 17 0,41 14,22 0,01 102,81
! "#! 31 0,37 9,00 0,00 11,74 32 2,4 32,21 0,00 77,92 34 0,16 12,29 32,99 85,26 35 0,07 12,55 13,97 69,26 36 0,07 9,58 13,87 94,61 58 0,01 0,04 0,00 85,86 59 0,00 0,02 0,00 82,36 61 1,98 0,19 9,21 25,70 62 0,04 0,00 2,65 65,07 72 0,01 6,63 0,57 57,83 75 0,00 14,32 0,00 26,79 79 0,07 0,66 95,93 86,45 82 0,08 0,82 1,78 93,30 Media 3,89a 12,44b 13,98A 54,74B DS 8,88 15,71 29,66 39,69
a, b: P < 0.05; A, B: P < 0,01. Kit 1: QIAamp Viral RNA Mini Kit (Qiagen); Kit 2: NucliSENS® MiniMAG® (bioMerieux).
• Crassostrea gigas
La media dellʼefficienza di estrazione dellʼRNA virale con il Kit 1 su 24 campioni di C. gigas è stata del 7,85 (± 10,01) %, mentre con il Kit 2 è stata leggermente inferiore (2,29 ± 2,58 %), ma anche in questo caso lʼanalisi statistica ha evidenziato una differenza significativa tra i due kit (P < 0,05).
Per quanto riguarda lʼefficienza di amplificazione, i campioni di RNA virale estratto con il Kit 1 presentano unʼefficienza più bassa (51,43 ± 39,67 %) rispetto a quelli
estratti con il Kit 2 (73,81 ± 40,42 %), ma lʼanalisi statistica non ha rilevato differenze significative tra le due medie (P = 0,05911).
I risultati dell'efficienza di estrazione e dellʼefficienza di amplificazione per la specie C. gigas sono riportati in tabella:
Codice % Efficienza Estrazione Kit 1 % Efficienza Estrazione Kit 2 % Efficienza Amplificazione Kit 1 % Efficienza Amplificazione Kit 2 41 1,38 4,96 28,52 65,52 42 0,06 3,27 9,28 47,30 48 6,34 0,00 6,25 93,95 49 0,00 1,09 2,32 0,17 50 0,00 0,13 0,00 38,16 52 4,84 0,44 29,12 69,74 53 0,57 1,93 4,27 0,48 54 0,50 0,04 3,08 59,87 55 4,67 0,99 30,35 93,95 56 0,51 3,68 2,37 0,09 68 8,97 0,03 38,16 17,92 71 18,70 7,89 115,67 89,50 73 41,21 1,09 39,78 85,86 74 8,60 4,38 107,92 93,30 76 6,39 1,11 67,83 115,67 83 3,52 0,07 73,71 100,70 84 6,84 1,18 77,38 104,97 85 8,97 0,61 87,06 94,61 86 29,54 0,69 49,65 75,26
! "#! 87 4,71 5,69 103,53 99,31 88 0,55 1,07 86,45 95,93 89 17,81 4,44 88,27 97,94 90 5,41 1,04 93,30 167,02 91 8,37 9,12 90,13 64,17 Media 7,85a 2,29b 51,43 73,81 DS 10,01 2,58 39,67 40,42
a, b: P < 0,05. Kit 1: QIAamp Viral RNA Mini Kit (Qiagen); Kit 2: NucliSENS® MiniMAG® (bioMerieux).
• Chamelea gallina
La media dellʼefficienza di estrazione dellʼRNA virale con il Kit 1 su 12 campioni di C. gallina è stata di 0,00 ± 0,01 % con un solo valore valido, mentre quella con il Kit 2 è stata leggermente superiore, con una media di 2,29 ± 2,90 %. Lʼanalisi statistica ha evidenziato una differenza altamente significativa tra i due kit (P < 0,01).
Per quanto riguarda lʼefficienza di amplificazione, i campioni di RNA virale estratto con il Kit 1 presentano una media molto più bassa (0,15 ± 0,53 %) rispetto a quelli estratti con il Kit 2 (36,04 ± 35,20 %) ed anche in questo caso lʼanalisi statistica ha rilevato una differenza altamente significativa tra i due kit (P < 0,01).
I risultati dell'efficienza di estrazione e dellʼefficienza di amplificazione per la specie C. gallina sono riportati in tabella:
Codice % Efficienza Estrazione Kit 1 % Efficienza Estrazione Kit 2 % Efficienza Amplificazione Kit 1 % Efficienza Amplificazione Kit 2 37 0,00 0,00 0,00 89,50
38 0,00 1,40 0,00 17,92 39 0,00 4,89 0,00 54,71 40 0,00 1,62 0,00 33,45 43 0,00 5,98 0,00 1,22 44 0,00 0,00 0,00 7,86 45 0,032 0,00 1,85 81,23 46 0,00 8,11 0,00 80,66 51 0,00 0,07 0,00 59,87 77 0,00 5,62 0,00 1,88 80 0,00 1,53 0,00 0,00 81 0,00 0,00 0,00 4,21 Media 0,00A 2,43B 0,15A 36,04B DS 0,01 2,90 0,53 35,20
A, B: P < 0,01. Kit 1: QIAamp Viral RNA Mini Kit (Qiagen); Kit 2: NucliSENS® MiniMAG® (bioMerieux).
4.2. C
ONFRONTO DELLʼ
EFFICIENZA DI ESTRAZIONE E DELLʼ
EFFICIENZA DI AMPLIFICAZIONE DELLʼRNA
VIRALE ESTRATTO MEDIANTE DUE DIFFERENTI KIT COMMERCIALI TRA LE DIVERSI MATRICI.
Al fine di valutare se le diverse matrici determinassero delle differenze nellʼestrazione dellʼRNA virale è stato effettuato un confronto dellʼefficienza di estrazione e dellʼefficienza di amplificazione con entrambi i kit tra le diverse specie analizzate. I campioni di C. gallina non sono stati considerati in questa tipologia di analisi, in quanto di numero inferiore rispetto agli altri.
! "#! Matrice % Efficienza Estrazione Kit 1 % Efficienza Estrazione Kit 2 % Efficienza Amplificazione Kit 1 % Efficienza Amplificazione Kit 2 Mytilus edulis 15,80 a 20,41a 71,18 a 105,51 a Donax trunculus 3,89 b 12,44b 13,98 b 54,74 b Crassostea gigas 7,85 b 2,29 b 51,43 a 73,81 b
a, b: P < 0.05. Kit 1: QIAamp Viral RNA Mini Kit (Qiagen); Kit 2: NucliSENS® MiniMAG® (bioMerieux).
• Il confronto dei valori dellʼefficienza di estrazione con il Kit 1 dei 24 campioni di M. edulis, D. trunculus e C. gigas ha mostrato una differenza altamente significativa (P < 0,01) tra le tre diverse matrici. In particolar modo al test per confronti multipli Student-Newman-Keuls, lʼefficienza più alta (15,80%) dei campioni di M. edulis è risultata significativa (P < 0,05) rispetto alle altre due matrici, che presentano unʼefficienza di estrazione rispettivamente di 3,89% e 7,85%.
• Il confronto tra i valori dellʼefficienza di estrazione con il Kit 2 dei 24 campioni di M. edulis, D. trunculus e C. gigas ha mostrato una differenza significativa (P < 0,05). Anche in questo caso la matrice M. edulis, che ha presentato unʼefficienza più alta (20,41%) rispetto alle altre due matrici (rispettivamente di 12,44% e 2,29%), è risultata significativa (P < 0,05) allʼesame statistico dei confronti multipli.
• Anche per quanto riguarda i valori dellʼefficienza di amplificazione ottenuta dallʼestrazione con il Kit 1, lʼanalisi della varianza ha rilevato differenze altamente significative (P < 0,01) tra le diverse matrici (M. edulis, D. trunculus e C. gigas). In questo caso i campioni di M. edulis con unʼefficienza media di amplificazione del 71,18% e quelli di C. gigas (51,43%) non hanno presentato al confronto multiplo differenze significative, mentre lʼefficienza del 13,98% dei campioni di D. trunculus è risultata significativamente più bassa (P < 0,05) rispetto alle due precedenti matrici.
• Così come per il Kit 1, i valori dellʼefficienza di amplificazione del Kit 2 hanno mostrato una differenza altamente significativa (P < 0,01) tra le tre matrici prese in esame. Al confronto multiplo lʼefficienza di amplificazione più alta (105,51%) dei campioni di M. edulis è risultata significativa (P < 0,05) rispetto ai valori medi di D. trunculus (54,74%) e di C. gigas (73,81%) che tra loro non hanno presentato differenze.
!
""!
4.3. C
ONFRONTO DELLʼ
EFFICIENZA DI ESTRAZIONE E DELLʼ
EFFICIENZA DI AMPLIFICAZIONE DELLʼRNA
VIRALE ESTRATTO MEDIANTE DUE DIFFERENTI KIT COMMERCIALI RISPETTO AI LIMITI DI ACCETTABILITÀ RIPORTATI IN PROCEDURA.
Matrice !"#$%$ &'$("$ )*+,"-."$)-.$ /00")"/.1*$("$ /2#3*1"-./$ 4%5$6$&'$("$ )*+,"-."$ *.*7"11*#"$ !"#$8$ &'$("$ )*+,"-."$)-.$ /00")"/.1*$("$ /2#3*1"-./$ 4%5$6$&'$("$ )*+,"-."$ *.*7"11*#"$ !"#$%$ &'$("$)*+,"-."$ )-.$/00")"/.1*$("$ *+,7"0")*1"-./$ 49:5$6$&'$("$ )*+,"-."$ *.*7"11*#"$ !"#$8$ &'$("$)*+,"-."$ )-.$/00")"/.1*$("$ *+,7"0")*1"-./$ 49:5$6$&'$("$ )*+,"-."$ *.*7"11*#"$ Mytilus
edulis #$%#&'! $#%#&(! $&%#&)! #&%#&*! Donax
trunculus +%#&)! $+%#&*! #%#&)! $,%#&*! Crassostrea
gigas $+%#&! $-%#&! $$%#&! $"%#&! Chamelea
gallina .%$#)! +%$#*! .%$#'! -%$#(!
a, b: P < 0.05; A, B: P < 0,01. Kit 1: QIAamp Viral RNA Mini Kit (Qiagen); Kit 2: NucliSENS® MiniMAG® (bioMerieux).
Nella tabella sono riportati, per ogni matrice, i campioni che secondo la procedura seguita soddisfano i limiti dellʼefficienza di estrazione (E.E. > 1%) e di amplificazione (E.A.> 50%). Lʼutilizzo del Kit 1 per lʼestrazione di M. edulis sembra soddisfare il requisito di accettabilità per lʼefficienza di estrazione in quasi tutti i campioni (21/24), mentre i campioni estratti con il Kit 2 soddisfano questo criterio solo per la metà di quelli analizzati (12/24), e la differenza tra i due risulta significativa (P < 0,05). Al
contrario, per la matrice D. trunculus, un numero maggiore di campioni che rientrano nel limite stabilito dellʼ1% per lʼefficienza di estrazione sono derivati dallʼestrazione con il Kit 2 (17/24), riportando una differenza con il Kit 1 altamente significativa (P < 0,01), con soli 7 campioni su 24 risultati dentro tale limite. Per i campioni di C. gigas invece i due kit sembrano avere unʼefficienza di estrazione simile per quanto riguarda il superamento del limite dellʼ1% (Kit 1: 17/24; Kit 2: 15/24), non riscontrando tra i due differenze significative (P = 0,759). Infine, anche i campioni di C. gallina, seppur analizzati in numero minore degli altri, presentano un risultato simile a D. trunculus; lʼestrazione con il Kit 1 infatti non ha permesso di raggiungere mai il limite indicato dalla procedura (0/12), mentre il Kit 2, sebbene abbia estratto in maniera soddisfacente solo 7 campioni su 12, risulta dare risultati con una differenza altamente significativa rispetto al primo (P < 0,01).
Per quanto riguarda il superamento del limite del 50% di efficienza di amplificazione indicato dalla procedura, complessivamente il Kit 2 ha dato i risultati migliori. Dei 24 campioni analizzati per le prime tre matrici, infatti, lʼestrazione con il Kit 2, dopo contaminazione dellʼRNA virale estratto con 1,0 μl di RNA di GII, ha permesso di soddisfare i limiti riportati in procedura a 24, 13 e 18 campioni contro i 14, 2 e 11 del Kit 1, rispettivamente per M. edulis, D. trunculus e C. gigas. Allʼanalisi statistica però, mentre la differenza riscontrata per i campioni di M. edulis e D. trunculs è risultata altamente significativa (P < 0,01), per C. gigas i due kit sembrano dare un risultato simile (P = 0,077). Per i campioni di C. gallina, anche lʼefficienza di amplicazione risulta simile a quella di estrazione, con 0 campioni che rientrano nel limite della procedura dopo lʼutilizzo del Kit 1 e con 5 su 12 nel caso del Kit 2, e la loro differenza è risultata significativa (P < 0,05).
!
"#!
4.4. Q
UANTIFICAZIONE E COSTRUZIONE DELLA RETTA DI TARATURA DELDNA
PLASMIDICO
.
In seguito alla purificazione del DNA plasmidico contenente lʼinserto sia per quanto riguarda NoV GI e GII che di HAV, si è proceduto alla quantificazione mediante lettura spettrofotometrica alla lunghezza dʼonda di 260 nm. I tre diversi plasmidi sono stati quantificati mediante diluizione 1:50 e si è potuto così calcolare la loro concentrazione.
I risultati sono riportati in tabella:
A 260 nm Concentrazione DNAplasmide (ng/μl) N° copie plasmide/μl Plasmide NoV GI 0,0590 147,5 3,7 x 1010
Plasmide NoV GII 0,0970 242,5 6,1 x 1010
Plasmide HAV 0,2549 637,25 1,3 x 1011
• NoV GI e NoV GII
I plasmidi per NoV GI e NoV GII sono stati diluiti con diluizioni scalari 1:10 e sottoposti a Real Time PCR. In questo modo è stato possibile calcolare una retta di taratura mediante i Ct medi delle varie diluizioni dei diversi plasmidi.
Plasmide NoV GI
Lʼefficienza della reazione di Real Time PCR per il plasmide di NoV GI è stata del 97,7%, con un R2 di 0,991, una Slope di -3,378 ed una y-intercetta di 42,185.
Diluizioni N° di copie plasmide/ μl Ct
10-8 1 x 102 35,77 10-7 1 x 103 31,93 10-6 1 x 104 28,53 10-5 1 x 105 24,61 10-4 1 x 106 22,54
!
"#!
Fluor PCR R Slope y-Intercept
Efficiency(%) Squared
FAM 97,7 0,991 -3,378 42,185
Plasmide NoV GII
Lʼefficienza della reazione di Real Time PCR per il plasmide di NoV GII è stata del 97,2%, con un R2 di 0,995, una Slope di -3,392 ed una y-intercetta di 42,259.
Diluizioni N° di copie plasmide/ μl Ct
10-7 1 x 103 31,81
10-6 1 x 104 29,20
10-4 1 x 106 21,47
Fluor PCR R Slope y-Intercept Efficiency(%) Squared
!
"#! • HAV
Per quanto riguarda lʼHAV è stata effettuata solo lʼanalisi in PCR classica di tipo qualitativo, in quanto non è stato possibile rilevare la fluorescenza emessa dalla sonda HAV150 e questo non ci ha permesso di mettere a punto unʼanalisi quantitativa in Real Time RT-PCR.
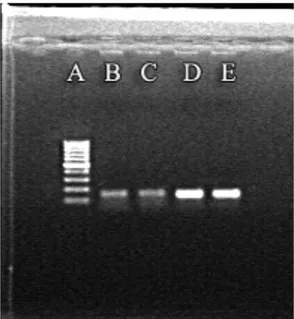
Il plasmide contenente lʼinserto per lʼHAV, sottoposto ad amplificazione con primer specifici per HAV, ha dato esito positivo. Come controllo di amplificazione è stato utilizzato lʼRNA virale di HAV dopo retrotrascrizione.
!
Figura 2. Amplificato del plasmide HAV. A: 100bp DNA Ladder; B e C: amplificato dellʼRNA virale dellʼHAV retrotrascritto ed amplificato; C e D amplificato del plasmide contenente lʼinserto per lʼHAV.
4.5. Q
UANTIFICAZIONE E COSTRUZIONE DELLA RETTA DI TARATURA DEL TRASCRITTO.
Successivamente il DNA plasmidico contenente lʼinserto di NoV GI e GII e HAV è stato trascritto in RNA ed anche lʼRNA è stato quantificato mediante lettura spettrofotometrica alla lunghezza dʼonda di 260 nm. I trascritti sono stati quantificati mediante diluizione 1:50 e si è potuto così calcolare la loro concentrazione.
I risultati sono riportati in tabella:
A 260 nm
Concentrazione RNA trascritto (ng/μl)
Trascritto NoV GI 2,05 4093,6
Trascritto NoV GII 1,433 2866
Trascritto HAV 1,699 3398
• NoV GI e GII
In seguito i trascritti di NoV GI e GII sono stati diluiti con diluizioni scalari 1:10 e sottoposti in doppio a Real Time RT-PCR. In questo modo è stato possibile quindi anche calcolare una retta di taratura mediante i Ct medi delle varie diluizioni dei diversi trascritti.
Trascritto NoV GI
Lʼefficienza della reazione di Real Time RT-PCR per il trascritto di NoV GII è stata del 100,2%, con un R2 di 0,996, una Slope di -3,316 ed una y-intercetta di 47,896.
!
"#!
Diluizioni N° di copie trascritto / μl Ct medio
10-10 1 x 105 31,88 ± 0,184 10-9 1 x 106 27,79 ± 0,177 10-8 1 x 107 24,07 ± 0,148 10-7 1 x 108 21,63 ± 0,115 10-6 1 x 109 17,84 ± 0,200 10-5 1 x 1010 14,50 ± 0,101 10-4 1 x 1011 11,86 ± 0,020
Fluor PCR R Slope y-Intercept Efficiency(%) Squared
FAM 100,2 0,996 -3,316 47,896
Trascritto NoV GII
Lʼefficienza della reazione di Real Time RT-PCR per il trascritto di NoV GII è stata del 98,7%, con un R2 di 0,994, una Slope di -3,354 ed una y-intercetta di 43,131.
Diluizioni N° di copie trascritto / μl Ct medio
10-10 1 x 105 26,51 ± 0,191 10-9 1 x 106 22,88 ± 1,061 10-8 1 x 107 19,03 ± 0,340 10-7 1 x 108 16,70 ± 0,029 10-6 1 x 109 12,95 ± 0,355 10-5 1 x 1010 9,49 ± 0,193
!
"#!
!
Fluor PCR R Slope y-Intercept
Efficiency(%) Squared
• HAV
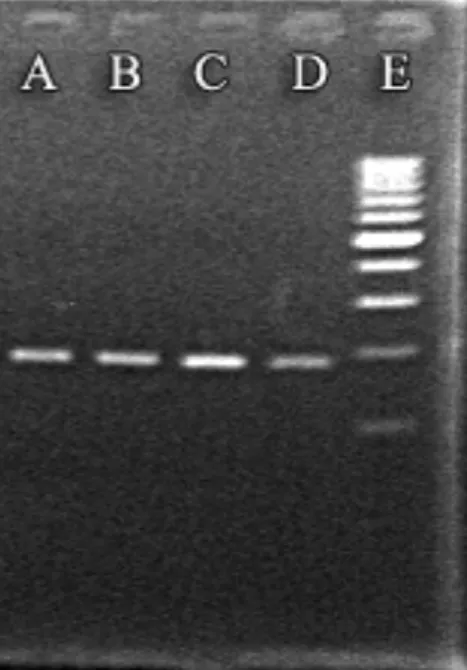
Per quanto riguarda il trascritto di HAV è stata effettuata lʼanalisi in RT-PCR. Il trascritto di HAV, sottoposto a retrotrascrizione ed amplificazione con primer specifici per HAV, ha dato esito positivo. Come controllo di retrotrascrizione e di amplificazione è stato utilizzato lʼRNA virale di HAV.
4.6. A
NALISI QUANTITATIVA PERN
OV
GI
EGII
IN ALCUNI CAMPIONI DI MOLLUSCHI BIVALVI.
Successivamente è stata effettuata lʼanalisi quantitativa di alcuni campioni già risultati positivi ad analisi qualitativa in Real Time RT-PCR, rispettivamente, per NoV GI e NoV GII; in questo modo è stato possibile valutare lʼutilizzo del trascritto nella quantificazione in Real Time RT-PCR di questi virus in campioni di molluschi bivalvi.
Figura 3. Amplificato del trascritto di HAV. E: 100bp DNA Ladder; A e B: amplificato dellʼRNA virale HAV; C e D amplificato del trascritto HAV
!
"##! Sia per NoV GI che per NoV GII sono stati analizzati tre diversi campioni estratti sia con il Kit 1 che con il Kit 2; i risultati sono mostrati di seguito:
NoV GI
Lʼefficienza della reazione di Real Time RT-PCR quantitativa per NoV GI è stata del 99,9%, con un R2 di 1,00, una Slope di -3,324 ed una y-intercetta di 42,381.
Fluor PCR R Slope y-Intercept Efficiency(%) Squared FAM 99,9 1,000 -3,324 42,381 Kit di estrazione RNA virale Codice campione Matrice % Efficienza Estrazione % Efficienza Amplificazione Ct N° copie RNA virale/ g campione Kit 1 34 D. trunculus 0,16 32,99 38,08 2520 Kit 1 35 D. trunculus 0,07 13,97 38,86 1479 Kit 1 36 D. trunculus 0,07 13,87 38,38 2057 Kit 2 34 D. trunculus 12,29 85,26 29,57 427800 Kit 2 35 D. trunculus 12,55 69,26 34,63 12900 Kit 2 36 D. trunculus 9,58 94,61 32,37 61200
!
"#$! NoV GII
Lʼefficienza della reazione di Real Time RT-PCR quantitativa per NoV GII è stata del 109,7%, con un R2 di 0,997, una Slope di -3,110 ed una y-intercetta di 41,838.
Fluor PCR R Slope y-Intercept Efficiency(%) Squared FAM 109,7 0,997 -3,110 41,838 Kit di estrazione RNA virale Codice campione Matrice % Efficienza Estrazione % Efficienza Amplificazione Ct N° copie RNA virale/ g campione Kit 1 30 M. edulis 0,02 143,40 30,25 686617 Kit 1 18 M. edulis 39,25 117,28 29,26 866629 Kit 1 20 M. edulis 42,36 140,44 31,64 246873 Kit 2 30 M. edulis 18,25 135,66 32,71 52500 Kit 2 18 M. edulis 49,85 174,11 36,34 3930 Kit 2 20 M. edulis 72,49 131,95 34,37 16260