2
.
MATERIALI E METODI
2.1 Raccolta dei campioni
Per indagare lo spettro fenotipico associato a mutazioni nel gene PCDH19, in questo studio sono stati selezionati 290 pazienti, 271 (più del 90%) erano di sesso femminile, con epilessia con esordio infantile, con crisi febbrili e afebbrili, generalizzate o focali, con o senza ritardo mentale e, ove presente, di severità variabile. Dall’anamnesi effettuata, alcuni tra i pazienti scelti avevano familiari con epilessia mentre gli altri erano casi sporadici.
Data la somiglianza fenotipica con la Sindrome di Dravet, molti dei probandi risultavano negativi per l’analisi di mutazione del gene SCN1A e per la ricerca di delezioni e duplicazioni dello stesso gene (effettuata con il Kit MLPA-MRC Holland).
Le informazioni cliniche relative ai pazienti e ai familiari sono state raccolte presso la Clinica di Neurologia Pediatrica dell’AOU Meyer di Firenze per le famiglie che si sono rivolte direttamente al nostro centro oppure sono state riferite dai medici dei pazienti seguiti in altri centri italiani ed europei.
Per procedere con le analisi sono stati richiesti e ottenuti i consensi informati dai genitori o tutori; lo studio era stato approvato dalla commissione etica.
Il materiale biologico utilizzato per le indagini molecolari pervenuto presso il laboratorio di Neurogenetica dell’AOU Meyer di Firenze, consisteva di campioni di DNA genomico o prelievi ematici in EDTA dei pazienti e ove disponibile dei rispettivi familiari. Il primo passaggio per il processamento dei campioni è stato la loro archiviazione nel database del nostro laboratorio conferendo ad ogni paziente un codice alfanumerico.
2.2 Analisi molecolare del gene PCDH19
2.2.1 Estrazione DNA da sangue periferico
Il sangue venoso periferico di ciascun individuo (5-10 ml) è stato prelevato in EDTA come anticoagulante ed è stato così processato: se il campione apparteneva al probando sono stati prelevati 1350 µl di sangue e sono stati trasferiti in una Eppendorf da 1,5 ml per l’estrazione del DNA tramite estrattore QIASymphony SP/AS (Qiagen); una identica aliquota è stata congelata come riserva a -20°C. Se il campione apparteneva ad un familiare del probando, la stessa procedura è stata effettuata utilizzando però 700 µl di sangue.
Il DNA dei probandi è stato eluito in 500 µl di buffer Qiagen ATE mentre quello dei familiari in 200 µl di buffer Qiagen ATE. I campioni ottenuti sono stati dosati allo spettrofotometro e conservati a 4°C nella banca di DNA del nostro laboratorio.
2.2.2 Quantificazione del DNA genomico
Il DNA genomico estratto con l’estrattore automatico Qiagen è stato dosato utilizzando lo spettrofotometro UV-Vis per micro-volumi NanoDrop 2000 (Thermo Scientific). Prima della misurazione dei campioni, è stato eseguito il bianco utilizzando il buffer Qiagen ATE. Poi è stato dosato ciascun DNA prelevandone 1,5 µl del totale. Lo spettrofotometro ha quantificato la concentrazione dell’acido nucleico e la qualità dell’estrazione. Il metodo spettrofotometrico sfrutta la capacità degli acidi nucleici di assorbire la luce UV con un massimo di assorbimento ad una lunghezza d'onda di 260 nm.
L'interferenza dovuta a contaminanti può essere evidenziata mediante il calcolo dei "rapporti". Il rapporto A260/A280 è usato per stimare la purezza degli acidi nucleici, dal momento che le proteine assorbono a 280 nm e che costituiscono la principale fonte di contaminazione. Un DNA puro dovrebbe avere un rapporto di circa 1.8, mentre un RNA puro dovrebbe dare un valore di approssimativamente 2.0. L'assorbanza a 230 nm riflette la
contaminazione del campione dovuta a sostanze come carboidrati, fenoli, peptidi o composti aromatici. Per campioni puri il rapporto A260/A230 dovrebbe essere circa 2.2.
2.2.3 Amplificazione mediante PCR delle regioni codificanti del gene PCDH19
Per il gene PCDH19 sono state identificate 3 isoforme diverse, due di queste costituite da 5 esoni (NM_020766.2, NM_001105243.1), la terza da 6 (EF676096.1). In questo studio è stata analizzata quest’ultima, che è l’isoforma più lunga.
Le sequenze relative alle coppie di primers utilizzate per amplificare i 6 esoni che compongono la regione codificante del gene PCDH19 (Accession Number: EF6760966.1), sono state scelte in modo da includere nell’analisi le sequenze introniche fiancheggianti gli esoni, a monte e a valle di questi. Lo scopo era quello di poter analizzare anche eventuali alterazioni a livello dei siti di splicing o vicini a questi e capaci di alterarli.
I primers sono stati disegnati utilizzando il software online Primer3 (versione 0.4.0) e sono stati testati attraverso tools bioinformatici di PCR in silico, predizioni di strutture secondarie degli oligonucleotidi scelti, eventuale presenza di polimorfismi noti interni alla sequenza oligonucleotidica. La sequenza dei primers utilizzati è riportata in tabella:
PCDH19_1AF 5’ GTCTCGGAGGGGTGTGGA 3’ PCDH19_1ER 5’ AGGAAACAAAAGCACCAGGA 3’ PCDH19_2F 5’ GAGAAGGATGCTGAATGCAC 3’ PCDH19_2R 5’ ACCCCCTCCTCTCCTAGAAA 3’ PCDH19_3F 5’ AACCCTCTGGGGTGCTTAAT 3’ PCDH19_3R 5’ ATCCAGCGAGCAGCTAAAGA 3’ PCDH19_4F 5’ CCTTGGGTTCCAATTCACTC 3’ PCDH19_4R 5’ AATGTTAAATCAAGCTTAGTTGCAG 3’ PCDH19_5F 5’ AGTGGGAACCTCTGTGCAAT 3’ PCDH19_5R 5’ CTGCTGAGGTGTGAGCTCTG 3’ PCDH19_6F 5’ AAAGTGCCATTTCCCACAAG 3’ PCDH19_6R 5’ GCCAAAGCTAATTTGCTCCA 3’ Tabella 2.1
Per l’amplificazione dei 6 esoni è stata utilizzata la FastStart Taq DNA Polymerase (Roche), una polimerasi Hot-Start, termostabile, capace di ridurre la formazione di ampliconi aspecifici e primer-dimers. Le temperature di annealing dei primers per ciascuna regione amplificata sono state selezionate in base alla formula standard: Tm=2(nA+nT)+4(nG+nC).
L’attività dell’enzima richiedeva una concentrazione di MgCl2 pari a 1,5 mM, dNTPs 0.25 mM, oligonucleotidi 0,5 uM, e Buffer Roche secondo protocollo. Per le regioni particolarmente ricche in GC è stata utilizzata una mix di reazione come sopra descritta ma con aggiunta del GC-buffer 5X (Roche).
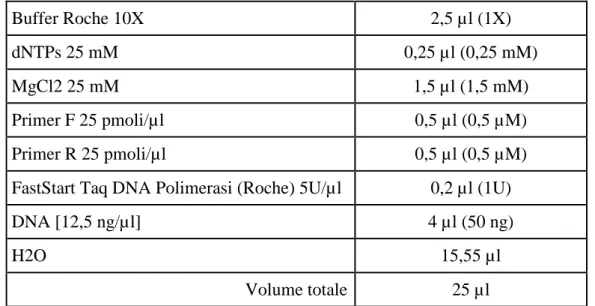
Dei 6 esoni, il primo, che era il più grande misurando più di 2 kb, è stato suddiviso in 5 parti per permetterne il sequenziamento, ma si è scelto di amplificarlo completamente con un’unica coppia di primers intronici per scongiurare l’amplificazione di eventuali pseudogeni o di altri geni ad elevata omologia di sequenza con questa regione altamente conservata di PCDH19. Inoltre essendo la sequenza ricca in GC, è stato aggiunto alla mix standard di reazione già descritta, il GC-buffer 5X (Roche). I protocolli di reazione sono di seguito riportati: Buffer Roche 10X 2,5 µl (1X) dNTPs 25 mM 0,25 µl (0,25 mM) MgCl2 25 mM 1,5 µl (1,5 mM) Primer F 25 pmoli/µl 0,5 µl (0,5 µM) Primer R 25 pmoli/µl 0,5 µl (0,5 µM)
FastStart Taq DNA Polimerasi (Roche) 5U/µl 0,2 µl (1U)
DNA [12,5 ng/µl] 4 µl (50 ng)
H2O 15,55 µl
Volume totale 25 µl
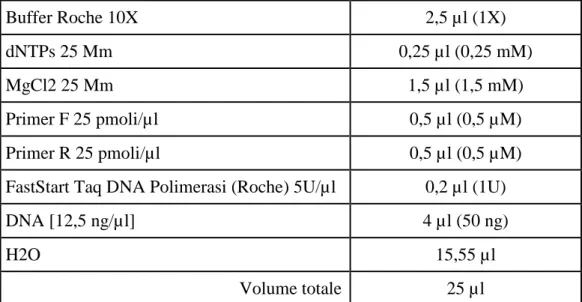
Buffer Roche 10X 2,5 µl (1X)
dNTPs 25 Mm 0,25 µl (0,25 mM)
MgCl2 25 Mm 1,5 µl (1,5 mM)
Primer F 25 pmoli/µl 0,5 µl (0,5 µM)
Primer R 25 pmoli/µl 0,5 µl (0,5 µM)
FastStart Taq DNA Polimerasi (Roche) 5U/µl 0,2 µl (1U)
DNA [12,5 ng/µl] 4 µl (50 ng)
H2O 15,55 µl
Volume totale 25 µl
Tabella 2.3. Protocollo utilizzato per l’amplificazione dell’esone 1
I programmi di amplificazione per tutti gli esoni sono riportati nelle tabelle seguenti: Denaturazione e attivazione dell’enzima 94°C 4 min 1 ciclo
Denaturazione 94°C 45 sec
Annealing 58°C 30 sec 35 cicli
Estensione 72°C 45 sec
Estensione finale 72°C 10 min 1 ciclo
Tabella 2.4. Programma di amplificazione usato per gli esoni 2 e 6
Denaturazione e attivazione dell’enzima 94°C 4 min 1 ciclo
Denaturazione 94°C 30 sec
Annealing 60°C 30 sec 35 cicli
Estensione 72°C 30 sec
Estensione finale 72°C 10 min 1 ciclo
Tabella 2.5. Programma di amplificazione usato per gli esoni 3,4 e 5
Denaturazione e attivazione dell’enzima 94°C 4 min 1 ciclo
Denaturazione 94°C 1 min
Annealing 62°C 30 sec 15 cicli
Estensione 72°C 2 min 30 sec
Denaturazione 94°C 1 min
Annealing 60°C 30 sec
Estensione 72°C 2 min 30 sec
20 cicli
Estensione finale 72°C 10 min 1 ciclo
Tutte le reazioni di PCR sono state effettuate mediante uno dei seguenti termociclatori presenti nel nostro laboratorio: GeneAmp 9700 (Applied Biosystem), 2720 thermal Cycler (Applied Biosystem), Triblock T3000 Thermocycler ( Biometra).
I prodotti di PCR sono stati analizzati su gel di agarosio all’1,8% in Tris-Borato-EDTA (TBE) 0,5X. La taglia dei frammenti è stata verificata mediante comparazione con marcatore di taglia molecolare.
2.2.4. Reazione di sequenza e analisi su sequenziatore automatico
I prodotti di PCR ottenuti sono stati purificati dai primers e nucleotidi in eccesso per poter essere utilizzati nella successiva reazione di sequenziamento. A tale fine è stato utilizzato ExoSAP-IT (Affymetrix), un prodotto contenente l’Esonucleasi I e la Fosfatasi alcalina. Il protocollo utilizzato prevedeva la preparazione di una mix contenente 0,5 µl di ExoSAP-IT, 1 µl di H2O e 1 µl di prodotto di PCR.
Successivamente i campioni così preparati sono stati incubati a 37°C per 15 minuti per la digestione e infine a 80°C per ulteriori 15 minuti per inattivare l’enzima. Il prodotto ottenuto è stato utilizzato per la reazione di sequenziamento.
Le reazioni di sequenziamento sono state eseguite utilizzando il prodotto BigDye Terminator V1.1 Cycle Sequencing Kit (Applied Biosystem, Foster City CA) basato sul metodo Sanger e su un particolare tipo di fluorescenza denominato Energy-Transfer. Le reazioni sono state effettuate in un volume di 10 µl, aggiungendo ai 2,5 µl totali di prodotto di PCR purificato il BigDye Terminator v1.1, il Sequencing Buffer 5X, il primer “forward” o “reverse” ad una concentrazione finale di 3,2 pmol/µl e l’H2O per arrivare al volume totale. I primers utilizzati sono gli stessi descritti nella tabella 2.1. L’unica eccezione riguarda l’esone 1 che a livello di reazioni di sequenza, data la notevole lunghezza, è stato suddiviso in 5 parti (A,B,C,D,E); i primers overlappanti utilizzati in questo caso sono riportati nella tabella seguente:
PCDH19_1AF 5’ GTCTCGGAGGGGTGTGGA 3’ PCDH19_1AR 5’ GCATTGTCGTTCAGGTCCTT 3’ PCDH19_1BF 5’ TCATGTCCAGCTCAATGGAA 3’ PCDH19_1BR 5’ TGGTGCCCTCGTCTGGAT 3’ PCDH19_1CF 5’ AGGTGACCGACTCCAATGAC 3’ PCDH19_1CR 5’ TTGTATTGGTCGTGCTGCTC 3’ PCDH19_1DF 5’ CCCTTTCGACTGCAGGAATA 3’ PCDH19_1DR 5’ ACCAGGTAGCCTATGCCAGA 3’ PCDH19_1EF 5’ CTCGACGTCAACGACAACAC 3’ PCDH19_1ER 5’ AGGAAACAAAAGCACCAGGA 3’
Tabella 2.7. Elenco primers utilizzati per il sequenziamento dell’esone 1
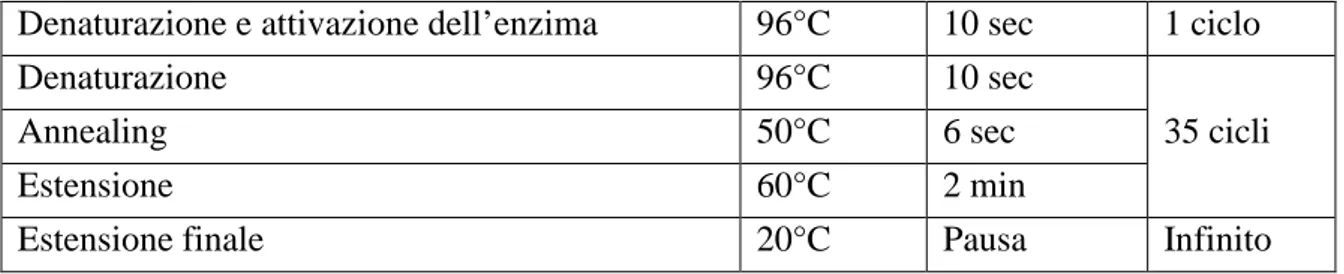
Il protocollo di sequenziamento, da noi denominato Seq50_35c è riportato nella tabella seguente:
Denaturazione e attivazione dell’enzima 96°C 10 sec 1 ciclo
Denaturazione 96°C 10 sec
Annealing 50°C 6 sec 35 cicli
Estensione 60°C 2 min
Estensione finale 20°C Pausa Infinito
Tabella 2.8. Programma di sequenziamento Seq50_35c
Tutte le reazioni di sequenziamento sono state effettuate mediante uno dei seguenti termociclatori presenti nel nostro laboratorio: GeneAmp 9700 (Applied Biosystem), 2720 thermal Cycler (Applied Biosystem), Triblock T3000 Thermocycler( Biometra).
I prodotti ottenuti sono stati successivamente purificati per rimuovere sali, primers e nucleotidi in eccesso, utilizzando prevalentemente due metodi. Uno prevedeva l’impiego di una resina Sephadex G-50, per purificazione su piastra tramite filtraggio. Prima della filtrazione, sono stati aggiunti 15 µl di H2O ai 10 µl di reazione per facilitare il recupero di un volume ottimale di prodotto da caricare nel sequenziatore automatico. Dopo una centrifugata di 4 minuti a 1000xg (SIGMA 4-15C, Qiagen) si ottenevano le reazioni di sequenza risospese in H2O e pronte per essere caricate nello strumento. L’altro metodo
prevedeva una precipitazione mediante sodio-acetato (NaOAc) ed etanolo: ai 10 µl di reazione si aggiungevano 2 µl di sodio acetato e 50 µl di etanolo al 100%. Si centrifugava 15 minuti a 15700xg e poi si toglieva il sovranatante e si lasciava asciugare. Seguiva un secondo lavaggio con 150 µl di etanolo al 70%, centrifugata di 5 minuti a 15700xg e successiva eliminazione del sovranatante. Il pellet (non visibile) veniva risospeso in formammide deionizzata Hi-Di Formamide (Applied Biosystem) evitando così il passaggio di denaturazione precorsa e ottenendo una notevole soppressione del segnale di background.
I prodotti di sequenza così purificati sono stati caricati sul sequenziatore automatico ABI 3130XL DNA Sequencer (Applied Biosystem) ed analizzati con 16 capillari in quarzo di lunghezza pari a 36 cm.
Le sequenze sono state poi analizzate con l’uso del programma SeqScape v2.5 (Applied Biosystem).
2.2.5.Real Time PCR: analisi Copy Number Variations (CNVs)
Per quantificare il numero di copie del gene PCDH19 presenti nel genoma, è stata utilizzato il kit TaqMan Copy Number Assay (Applied Biosystem) che combina la chimica TaqMan con la strumentazione Real-Time PCR. In linea generale il kit sopra menzionato è stato disegnato per individuare e misurare le CNVs nel genoma umano e murino. Le CNVs sono polimorfismi importanti associati a malattie genetiche tra cui il cancro, malattie immunitarie e patologie neurologiche.
Per il nostro studio sono state ordinate 6 probes TaqMan MGB (Applied Biosystem), specifiche per la regione cromosomica in cui mappa il gene PCDH19. Ogni sonda era cordinata di una coppia di primers non marcati. Le sonde TaqMan sono tutte marcate con il fluorocromo FAM all’estremità 5’, mentre il quencher non fluorescente e la molecola MGB sono attaccate all’estremità 3’ della sonda.
Data l’elevata specificità per la sequenza target, le sonde si appaiano alla regione complementare compresa tra i primers forward e reverse. Se l’ibridazione è avvenuta con successo, durante i cicli di amplificazione, l’attività 5’ esonucleasica della Taq polimerasi, rompendo la sonda interna e liberando il fluorocromo FAM, genera l’emissione di un segnale di fluorescenza. Poiché lo sviluppo della fluorescenza del reporter si ha solo se i primers e la sonda legano il DNA target, la specificità della PCR Real-Time è considerevolmente più alta rispetto alla PCR convenzionale.
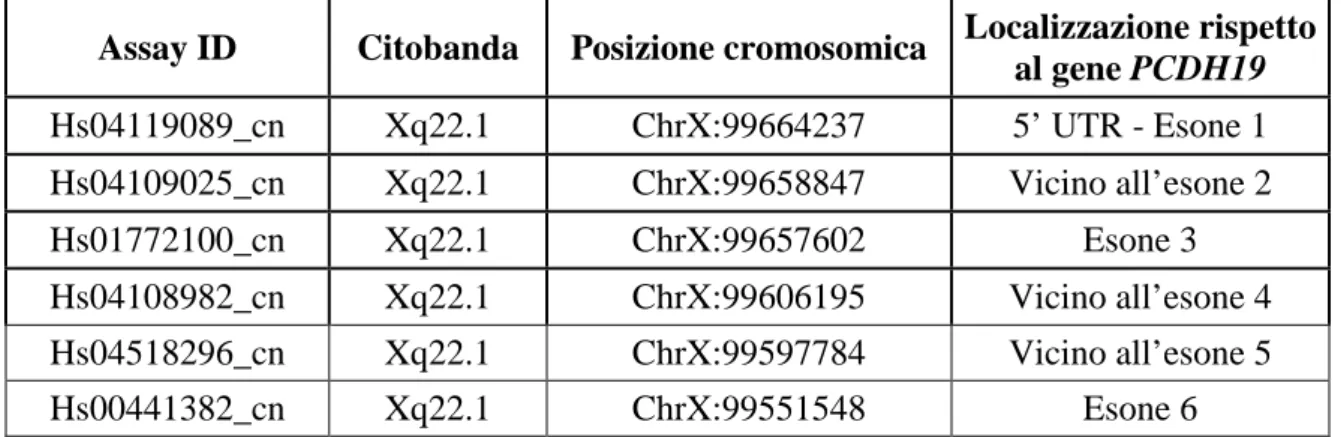
L’elenco dei saggi TaqMan utilizzati nel nostro studio per PCDH19:
Assay ID Citobanda Posizione cromosomica Localizzazione rispetto al gene PCDH19 Hs04119089_cn Xq22.1 ChrX:99664237 5’ UTR - Esone 1 Hs04109025_cn Xq22.1 ChrX:99658847 Vicino all’esone 2 Hs01772100_cn Xq22.1 ChrX:99657602 Esone 3 Hs04108982_cn Xq22.1 ChrX:99606195 Vicino all’esone 4 Hs04518296_cn Xq22.1 ChrX:99597784 Vicino all’esone 5 Hs00441382_cn Xq22.1 ChrX:99551548 Esone 6
Tabella 2.9. Informazioni relative ai saggi TaqMan Copy Number Assay 20X (Applied Biosystem) per il gene PCDH19
Il saggio TaqMan Copy Number Reference Assay per il gene RNase P era l’AssayID4403326 costruito sulla citobanda 14q11.2 da Applied Biosystem.
La quantificazione del numero di copie del gene PCDH19 nel genoma ottenuta nel nostro esperimento era una quantificazione relativa, non assoluta: il Ct della curva di amplificazione dei campioni testati è stato paragonato al Ct relativo ad un saggio di riferimento. La referenza utilizzata nel nostro esperimento era il gene RNase P, noto per essere presente in due copie nel genoma umano diploide. Il valore del Ct è stato scelto a livello della linea soglia in cui intersecavano le curve di tutti i campioni nella fase esponenziale. La differenza dei valori risultava nel ∆Ct, che successivamente veniva confrontato con il Ct relativo ad un campione di controllo (calibratore) di cui era noto il numero di copie del gene target, ottenendo così il valore del ∆∆Ct.
Per eseguire un esperimento di quantificazione relativa del numero di copie, era necessario impostare una Duplex Real-Time PCR in cui far amplificare contemporaneamente al gene target anche il gene di riferimento.
A tale scopo sono stati preparati 6 saggi, uno per ciascuna sonda relativa al gene target (PCDH19), ognuno comprendente anche la sonda relativa alla referenza (RNase P). Il kit TaqMan CNV prodotto da Applied Biosystem per il gene RNase P era comprensivo di una coppia di primers specifici e di una probe con al 5’ un reporter marcato con il fluorocromo VIC e al 3’ un quercer marcato con fluorocromo TAMRA.
Nella figura 2.10 sono mostrati i passaggi di una reazione di PCR in duplex, contenente contemporaneamente la sonda per il gene target e quella per la referenza, entrambi i saggi sono provvisti di Taq Dna Polimerasi con azione 5’ esonucleasica.
I soggetti da includere in questo primo gruppo di analisi, risultati negativi all’analisi di sequenza di PCDH19, erano 20 pazienti; tra questi era presente il padre di una paziente mutata, trovato eterozigote per la mutazione individuata nella figlia, allo scopo di capire se tale dato poteva trovare spiegazione nell’eventuale presenza di una duplicazione di tutto o parte del gene. Il campione usato come calibratore era un maschio sano noto per possedere una copia soltanto del gene target.
I 6 saggi totali eseguiti per ciascun paziente e per il campione calibratore sono stati tutti effettuati in triplice copia per ridurre l’errore dell’operatore. In ogni caso era previsto un NTC (No Template Control) cioè un campione non contenente DNA che aveva lo scopo di indicare il background di fondo e di individuare eventuali contaminazioni.
Prima di procedere con la reazione, tutti i DNA di interesse sono stati diluiti con Buffer TE 1X (Tris-EDTA) ad una concentrazione di 5 ng/ul.
L’esperimento è stato pianificato su piastre MicroAmp Optical Reaction Plate da 96 pozzetti. In ogni tubo è stata dispensata una mix di reazione costituita dai componenti elencati in tabella:
TaqMan Genotyping Master Mix, 2X 10 ul
TaqMan Copy Number Assay, 20X 1 ul
TaqMan Copy Number Reference Assay, 20X 1 ul
H2O nuclease-free 4 ul
DNA [5 ng/ul] 4 ul (20 ng)
Volume totale 20 ul
Tabella 2.11
Il kit TaqMan Genotyping Master Mix (Applied Biosystem) conteneva una AmpliTaq Gold DNA polimerasi UP (ultrapura) con azione 5’ esonucleasica, dNTPs e UDG (Uracil-DNA Glicosilasi).
Il protocollo di amplificazione utilizzato prevedeva un’incubazione a 95°C per 10 minuti per attivare l’AmpliTaq Gold Polimerasi, seguita da 40 cicli di denaturazione e annealing/elongazione rispettivamente alle seguenti condizioni: 95°C per 15 secondi e 60°C per 1 minuto.
Per l’analisi e l’interpretazione dei risultati è stato utilizzato lo strumento CFX 96 Real-time System (BIO-RAD) ed il relativo software CFX Manager Software Versione 1.5 (BIO-RAD).
2.3 X-inattivazione
Sono stati digeriti 500 ng di DNA genomico con incubazione a 37 ° C overnight (O/N) in un volume di 50 µl contenente 5 µl di Digestion Buffer Tango 10X e 1 µl di enzima HpaII (10 U / µl) (Roche Diagnostics, Mannheim, Germania), enzima metilasi sensibile che introduce due tagli nel locus Humara ove questo non risulti metilato.
Dopo la digestione, HpaII è stato inattivato a 65 ° C per 30 minuti. La PCR è stata eseguita sia sul DNA digerito che su quello non digerito con i primers specifici per il gene del recettore degli androgeni HUMARA, localizzato sul cromosoma X ed altamente polimorfico per la presenza di ripetizioni di triplette (CAG)12-30. La tipizzazione di tale locus permetteva di capire se il cromosoma X nelle femmine, che sono mosaici naturali, era stato inattivato in maniera casuale (rapporto da 50:50 a 80:20) o se c’era stata un’asimmetria quindi uno sbilanciamento di inattivazione tra cromosoma X paterno e cromosoma X materno (rapporto > 80:20). Il primer forward era marcato all’estremità 5’ con il fluorocromo FAM (6-carboxy-fluorescein). La polimerasi utilizzata in questo saggio era la FastStart Taq DNA Polymerase 5U/µl (Roche). I primers Humara utilizzati per il seguente saggio erano:
Forward: 5’ CCCCAGGCACCCAGAGGC 3’ Reverse: 5’ GAGAACCATCCTCACCCTGCT 3’.
Il protocollo di PCR è riportato nella seguente tabella (volume finale di 25 µl):
Denaturazione e attivazione dell’enzima 94°C 4 min 1 ciclo
Denaturazione 94°C 45 sec
Annealing 58°C 30 sec 28 cicli
Estensione 72°C 30 sec
Estensione finale 72°C 7 min 1 ciclo
Tabella 2.12.
La reazione di digestione è stata eseguita in parallelo, nelle stesse condizioni, anche sul DNA di un soggetto maschile come controllo: dato che il cromosoma X nel maschio non è metilato, l’assenza di amplificato era considerata una prova di una precedente digestione avvenuta con successo.
La PCR fluorescente è stata analizzata utilizzando come marker interno di lunghezze dei frammenti il GeneScan - 500 LIZ Size Standard (Applied Biosystems) marcato con il fluorocromo LIZ. Lo strumento utilizzato è stato il sequenziatore automatico a 16 capillari 3130XL ABI Prism (Applied Biosystems).
Gli elettroferogrammi sono stati analizzati con il software Peak Scanner v1.0 (Applied Biosystem).