La 4-ammino-4-deossicorismato sintasi (ADCS) catalizza il primo passo nella conversione del corismato e p-aminobenzoato, che è incorporato nell’acido folico. Sono stati progettati alcuni inibitori dell’ADCS come leads per una nuova classe di composti antimicrobici [3].
C’è una forte necessità di trovare nuove classi di antiinfettivi per combattere la resistenza acquisita ai farmaci da parte dei batteri e dei parassiti. Storicamente, gli antiinfettivi più utilizzati derivavano principalmente da prodotti naturali, sebbene anche farmaci non derivati da prodotti naturali sono normalmente impiegati nella pratica clinica (ad es., fluorochinoloni, sulfamidici). Il paradigma moderno per la scoperta di nuove classi di farmaci non di origine naturale si basa sulla scelta meticolosa del target del farmaco e sulle proprietà molecolari dei candidati a farmaci durante il loro sviluppo. Un’intelligente chimica combinatoria, in cui le librerie sono preparate usando le conoscenze della struttura della proteina target e le restrizioni sulle proprietà molecolari, contiene in sé una grande promessa circa una rapida scoperta del lead nello sviluppo degli antiinfettivi.
Nell’ultimo decennio, la chimica combinatoria e l’High-Throughput Screening (HTS, screening di una vasta serie di composti) hanno fatto grandi progressi nella scoperta di farmaci [22,23]. Con i metodi bead one-compound (OBOC), i ricercatori posseggono gli strumenti per creare vaste librerie per la scoperta di composti biologicamente attivi. Comunque, le difficoltà sorgono ancora a causa della natura sensibile del supporto solido, dalle sfide di sintetizzare chimicamente i tag di codificazione di un’unica struttura, dalle insidie nel testare le librerie on-bead [24, 25, 26], e dal processo di decodificazione della struttura [27, 28]. Metodi differenti possono essere riuniti in un protocollo coesivo per scoprire in modo efficiente gli inibitori usando i metodi OBOC. Per esempio, una libreria dovrebbe essere ottimizzata per
sfruttare la conoscenza del sito attivo/di legame. Per di più, un metodo di codifica della struttura dovrebbe essere implementato in modo che aiuti la chimica impiegata e che consenta un’identificazione rapida e affidabile dei ligandi legati alla superficie. Inoltre, l’eliminazione dei falsi positivi in uno screening one-bead è una necessità
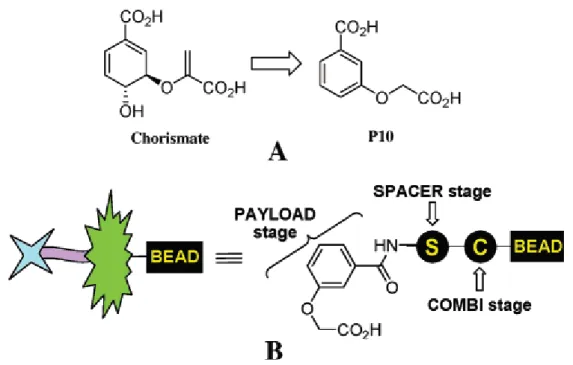
L’enzima bersaglio è la 4-ammino-4-deossicorismato sintasi (ADCS),com’è noto, costituisce un potenziale bersaglio per gli antimicrobici [3]. Il suo substrato è il corismato (Figura 9A).
Figura 9: (A) Struttura del corismato e dell’inibitore strutturalmente simile al corismato,
scelto per la sintesi della libreria. (B) Approccio alla progettazione della libreria utilizzando un derivato PAYLOAD.
E’ stata progettata una libreria che incorpora un analogo semplice del corismato (PAYLOAD) per dare la specificità del sito attivo connesso attraverso uno SPAZIATORE alle porzioni del COMBI. Questo progetto “di fase” è ispirato dai farmaci statinici che riducono il colesterolo e che inibiscono
l'idrossimetilglutaril-CoA-reduttasi. Essi contengono una porzione strutturalmente analoga all’idrossimetilglutarato, abbinata a quelle che sono completamente non correlate con il substrato affine. In realtà, esiste una letteratura recente che supporta questa strategia del progetto di base, che è fondata sulla teoria dell’inibitore analogo multisubstrato. La figura 9B illustra questo approccio, in cui le librerie sono progettate per sfruttare le opportunità di legame non soltanto all’interno, ma anche in prossimità del sito attivo dell’ADCS. Il PAYLOAD (analogo del corismato) e lo SPAZIATORE (lisina) sono costanti mentre la porzione COMBI [acido-(lisina-PAYLOAD)-aminoacido] varia.
Progettando queste librerie, i tag (codici) separabili dello spettro di massa del bromuro di cianogeno venivano scelti come metodo per un’identificazione rapida delle molecole presenti in superficie. Una preoccupazione relativa all’uso di tag di codifica con lo screening on-bead è che essi interagiranno con la proteina bersaglio, conducendo alla non identificazione dei composti del test quali ligandi. Questo problema può essere superato usando perline di resina bifunzionali, segregate topologicamente, poiché in un lavoro recente [28] è stato dimostrato che le strutture di resina e la densità dei ligandi legati alla superficie restringe le proteine all’esterno delle perline. In questo modo, confinare i tag di codifica all’interno delle perline espone le proteine soltanto ai ligandi di superficie in campioni on-bead.
Infine, un facile metodo per l’identificazione di high-throughput delle perline di resina legate all’ADCS (“hit”), era quello di utilizzare delle tinture fluorescenti attivate in commercio per etichettare la superficie dell’ADCS alle catene laterali della lisina [29]. Questo consente l’identificazione delle perline legate all’ADCS attraverso l’osservazione manuale sotto un microscopio a fluorescenza oppure tramite una selezione con COPAS (complex object parametric analyzer and sorter) automatizzato. L’applicazione originale di questa tecnica era limitata dall’antifluorescenza di fondo delle perline della libreria casuale, che portava a
dei falsi positivi. L’autofluorescenza è una proprietà inerente a tutte le librerie studiate. Dunque, la selezione tramite COPAS automatizzato prima dello screening con ADCS etichettata fluorescentemente veniva usata per rimuovere le perline che autofluorescevano. Lo strumento COPAS possiede il vantaggio ulteriore di identificare la regione dello spettro che ha la minore quantità di autofluorescenza, consentendo la rimozione di pochissime perline dalla libreria e la selezione dell’intensità di fluorescenza più appropriata.
Un’analisi cinetica significativa degli inibitori dell’ADCS richiede una comprensione del meccanismo cinetico dell’enzima. L’ADCS richiede Mg2+ per l’attività, che lega al carbossilato C1 del corismato e di diversi glutammati del sito attivo a livello della porzione finale del sito attivo esposta al solvente. Di conseguenza, si potrebbe pensare che l’ADCS (e, per estensione, gli altri enzimi omologhi) obbedisca a un meccanismo di ordine cinetico, con il corismato che si lega per primo e Mg2+ come secondo. I dati della cinetica confermano questo, come gli esperimenti di estinzione della fluorescenza con il corismato e gli inibitori artificiali. Sorprendentemente, perfino i semplici analoghi del corismato (come pure gli inibitori ulteriormente elaborati) sono competitivi sia verso il corismato sia verso l’Mg2+. Questa conoscenza ha permesso di dimostrare che gli inibitori PAYLOAD e PAYLOAD – SPAZIATORE si legano circa 60 volte più debolmente del corismato e che l’inibitore più potente si lega circa 1,5 volte più strettamente del corismato.
Progetto e sintesi del PAYLOAD e dello SPAZIATORE.
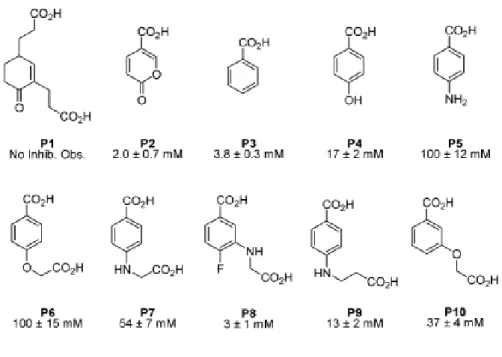
La Figura 10 presenta diversi composti del PAYLOAD che sono stati presi in considerazione per la sintesi della libreria. P2, P3, P4, e P5 erano disponibili da fonti commerciali. P6 [30], P7 [31], e P9 [32] erano sintetizzati secondo le procedure riportate in letteratura.
Figura 10: Valori di Kinei confronti di ADCS per potenziali strutture PAYLOAD.
P1 veniva sintetizzato secondo le procedure mostrate nello schema 1. La cicloaddizione di Dies-Alder del diene di Danishefsky modificato (P1a) con il metilacrilato ha prodotto una miscela diastereomerica dei cicloaddotti (P1b). L’esposizione di questi cicloaddotti a un complesso 10% HF-piridina in aceto nitrile ha portato a una miscela di enoni isomerici (P1c e P1c´). L’alchilazione vinilica della miscela di enoni con il metilacrilato avrebbe dovuto produrre P1d´, che avrebbe liberato P1´, il bersaglio effettivo di questa sintesi, per idrolisi dell’estere. In realtà si è avuta esclusivamente la formazione di P1d con
conseguente sintesi del nuovo PAYLOAD P1.
Schema 3: Sintesi di P1a
a
Reagenti e condizioni: (i) metil acrilato, toluene, t.a., 16 h; (ii) 10%HF/Py in CH3CN, t.a., 2 h;
(iii) metil acrilato, DBU, DMF, riflusso, 16 h; (iv) MeOH, t.a., 16 h.
Lo schema 4 descrive la sintesi di P8.
Schema 4: Sintesi di P8a
a
Reagenti e condizioni: (i) H2, Pd/C, EtOH, 16 h, t.a.; (ii) TEA, THF, N2, 50-60 °C, 16 h; (iii)
La riduzione di P8a a P8b ha consentito la N-alchilazione con tert-butil 2-bromoacetato per produrre P8c. La successiva saponificazione di P8c ha prodotto il PAYLOAD P8. P10 era scelto come PAYLOAD della libreria in ragione della sua facilità di sintesi, della sua semplice adattabilità alla sintesi in fase solida, e della sua somiglianza strutturale al corismato (per es., un acido carbossilico contenente una catena laterale).
Per questa libreria di prova, è stata scelta la lisina protetta semiortogonalmente (tipo Fmoc-Lis(Dde)-OH) come struttura sulla quale legare le fasi di PAYLOAD e COMBI. L’acido carbossilico della lisina era usato nella formazione del legame del peptide con un elemento di diversità sull’ amminoacido, e la α-ammina era N-acilata con un elemento di diversità sull’ acido carbossilico per liberare la fase COMBI. La ∈-ammina incorporava la fase di PAYLOAD attraverso l’N-acilazione. La strategia di accoppiamento richiedeva che l’acido aril-carbossilico del PAYLOAD P10 non fosse protetto, mentre la porzione dell’acido arilossi-acetico era protetta come tert-butil-estere. Come descritto nello schema 3, l’aril acido era protetto come metilestere poiché competeva nello spiazzamento SN2
del bromuro nello step di alchilazione del tert-butil-2-bromuroacetato. L’O-alchilazione di questo fenolo (1) conduceva a 2, e il trattamento seguente con idrossido di litio portava a una miscela del composto desiderato 3 e di un 33% del diacido P10. Un campione di P10 era ritrasformato nel composto 3 per trattamento con Boc anidride. Questa conversione finale era eseguita in alta resa, con completa selettività per l’acido carbossilico alifatico.
Sono stati sintetizzati altri tre altri derivati (4, 5 e 7) per rendere meno complessi i contributi di ogni inibitore di fase al legame. Il composto 4 era sintetizzato
Schema 5: Sintesi di P10 e altri derivatia
a
Reagenti e condizioni: (i) AcCl, MeOH, N2, riflusso, 8 h.; (ii) K2CO3, THF, N2, 4 h; (iii)
LiOH,H2O, THF, 6 h; (iv) (Boc)2° DMAP, CH3CN, N2, 0 °C, t. a., 16 h; (v) 20% piperidina in
DMF, 2 x10 min; (vi) 3, HOBt, DIC; (vii) Fmoc-Lys(Dde)-OH, HOBt, DIC; (viii) anidride acetica (10 equiv), DIEA (12 equiv), DMF; (ix) 2% idrazina in DMF, 2 x 10 min; (x) 2, HOBt, DIC; (xi) 6, NaCNBH3, 1% AcOH, TMOF; (xii) TFA/H20/TIS (95, 2.5, 2.5, v/v/v).
dal 3 usando una resina Rink ammide. Il composto 5 era sintetizzato per determinare il contributo dello SPAZIATORE di lisina all’affinità di legame. Per il composto 5 è stata utilizzata anche la sintesi in fase solida che avviene con alta resa e fornisce un composto altamente puro. Il composto 7 era sintetizzato per determinare se un ammino gruppo caricato positivamente vicino alla posizione normale dello ione Mg2+ avrebbe accresciuto il legame dell’inibitore. Un’amminazione riduttiva su fase solida era eseguita su 6, l’analogo aldeidico di 3, per liberare 7 puro con buona resa dopo una serie di passaggi efficaci.
Sintesi della libreria. La libreria in oggetto è stata preparata come descritto nello schema 4, usando una resina ammino-funzionalizzata segregata topologicamente. Tramite l’impiego di ~ 90% del totale dei gruppi funzionali disponibili per il tag codificante (ristretto all’interno della perlina), i segnali del tag MALDI-TOF (Matrix-Assisted Laser Desorption/Ionization-Time of Flight) sono stati rilevati con sicurezza.
Schema 6: Sintesi della libreriaa
a
Reagenti e conditioni: (i) H2O, 48 h; (ii) Alloc-OSu (0.1 equiv), DIEA, DCM/Et2O (55:45,
v:v), 30 min; (iii) Fmoc-MeOH, HOBT, DIC; (iv) 20% piperidina in DMF, 2 x 10 min; (v) N-Fmoc-3-(4-bromofenil)-β-alanine, HOBt, DIC; (vi) Fmoc-Arg(Pmc)-OH, HOBt, DIC; (vii) N-Fmoc-2,2’-etilendiossibis(etilamina)monosuccinamide, HOBt, DIC; (viii) Pd(PPh3)4 (0.24 equiv), PhSiH3 (20 equiv), DCM; (ix) split beads in 42 colonne; (x) library assembly (Fmoc-X1-OH, HOBt, DIC); (xi) mix beads; (xii) Fmoc-Lys(Dde)-OH, HOBt, DIC; (xiii) H2O, 24 h; (xiv) Fmoc-Osu (0.7 equiv), DIEA, DCM/Et2O (55:45, v:v), 1 h; (xv) (Boc)2O (1 equiv), DIEA,
DCM; (xvi) library assembly X2-CO2H, DIC, HOBt; (xvii) 2% idrazina in DMF; (xviii)
3-(2-tert-butoxy-2-oxoethoxy)benzoic acid (3), HOBt, DIC; (xix)
TFA/phenol/TIS/H2O/ethanedithiol (90/5/2/2/1, v/w/v/v/v).
La sintesi del tag codificante è stata effettuata in accordo con le procedure riportate in letteratura [27]. Lo spaziatore divisibile comprende quattro entità: (1) la metionina, che abilita la frammentazione della resina tramite il bromuro di cianogeno (metionina + bromuro di cianogeno → omoserina lattone) [33]; (2) la ß-bromoalanina, che facilita l’identificazione del tag tramite i pattern isotopici
del bromo nello spettro di massa [34]; (3) l’arginina; e (4) uno spaziatore. Gli ultimi due incrementano la solubilità del tag e il peso molecolare, che facilita l’analisi MALDI-TOF evitando i segnali di massa dovuti alla matrice. Le rimanenti trasformazioni indicate nello schema 4 completano la costruzione dei tag codificanti e la molecola bersaglio trovata sulla superficie della perlina.
I gruppi di protezione sul tag codificante (Fmoc ossia Fluorenil-metossi-carbonilico) e sulla superficie della perlina (Alloc ossia residuo di tipo allilico) vengono rimossi, e viene introdotto il primo elemento di diversità COMBI (amminoacido X1). Dopo, la lisina protetta semiortogonalmente viene attaccata al
tag codificante in crescita e alla molecola bersaglio. Le perline sono poi aumentate di volume in acqua per 24 h, e la superficie più esterna e una frazione del tag codificante sull’interno sono protetti con Fmoc (0,7 equivalenti), lasciando il centro della perlina con un’ammina libera che è protetta ortogonalmente con Boc. In questo modo, il volume più interno della perlina libera un tag che riporta il primo elemento di diversità COMBI (per esempio, X1).
Il secondo elemento di diversità COMBI viene introdotto dopo la deprotezione del Fmoc. Il PAYLOAD 3 è poi accoppiato all’ammina distale Dde (Dimetil-diossicicloesiliden-etil)-deprotetta. Il secondo tag codificante contiene la molecola completa bersaglio. L’intera superficie della molecola bersaglio può essere così identificata dal MALDI-TOF dopo la frammentazione data dal bromuro di cianogeno (CNBr) dei tag di codifica dalla resina. L’accoppiamento del PAYLOAD 3 e la rimozione di tutti i gruppi di protezione completa la sintesi della libreria codificata con spettrometria di massa (MS). L’incorporazione di 48 elementi amminoacidici per la diversità X1 e di 48 elementi acidi carbossilici per
la diversità ha dato come risultato X2 2304 composti test.
L’identificazione di ciascun elemento di diversità è compiuta con le equazioni seguenti:
X1 = M1 – 712 – (K accoppiato con P10)
X2 = M2 – M1 – 712 – (K accoppiato con P10)
In queste equazioni M1 e M2 sono segnali di massa MALDI-TOF, corrispondenti
ai segnali del primo e del secondo tag, ciascuno dei quali mostra un pattern di impronta bromo isotopica. Come test, parecchie perline vengono rimosse dalla libreria durante la sintesi e sequenziate per convalidare la sintesi della libreria. Queste perline liberavano gli attesi segnali MALDI-TOF a ogni punto di test.
Classificazione COPAS della libreria.
Le librerie combinatorie create usando metodi OBOC con composti aromatici contengono perline auto fluorescenti [3].
Screening della libreria.
L’enzima Alexa Fluor 488 marcato mostrava l’84% dell’attività dell’enzima non marcato, come determinato dal saggio dell’LDH-accoppiato (vedi sotto). Dopo un’ora di incubazione con l’enzima marcato, la libreria era osservata sotto un microscopio a fluorescenza in 15 batchs di circa 2500 perline ciascuno. In ogni batch, circa il 2% delle perline che fluoresceva con forza veniva isolato manualmente con una micropipetta; di queste, venivano selezionate 15 perline con la fluorescenza più brillante. Le perline “hit” erano lavate con guanidina cloridrato 8 M (per rimuovere l’ADCS) e acqua.
Analisi MALDI-TOF e ulteriore sintesi libera dalla resina.
Ogni perlina “hit” lavata era incubata individualmente con bromuro di cianogeno per frammentare i tag. L’analisi MALDI-TOF produceva segnali che consentivano l’identificazione dei ligandi di superficie per 11 delle 15 perline “hit” lavate e frammentate. Ciò corrisponde a una leggibilità al 75%, che è in accordo con i valori della letteratura [35]. Gli 11 composti identificati dal MALDI-TOF sono mostrati nella Figura 11.
Figura 11: Valori di Ki per i ligandi identificati con il test dell’ADCS Alexa Fluor 488
Da questi risultati, erano individuati due set di ligandi duplicati (L1 = L4; L3 = L8 = L11), suggerendo che la maggior parte della libreria era stata scrinata e che i ligandi identificati erano tra quelli si che legavano più strettamente. Questi ligandi esibivano un pattern strutturale distinto, ciascuno dei quali conteneva un gruppo idrofobico e uno idrofilo nella fase COMBI. I ligandi erano risintetizzati on-bead usando la resina Rink ammide e rilasciata per l’analisi cinetica. I ligandi risintetizzati erano purificati fino a una purezza >95% con HPLC.
Risultati cinetici.
I dati della velocità iniziale per determinare il meccanismo cinetico dell’ADCS sono stati ottenuti dalla misurazione dell’incremento nell’assorbanza a 290 nm dovuto alla formazione del PABA (Para-AminoBenzoic Acid – acido para-amminobenzoico) in un campione accoppiato con ADCL (Acid Deposition Critical-Load – carico critico di deposito acido) in eccesso. La Figura 12 mostra i risultati di questi esperimenti; i dati si accordano meglio all’equazione per un meccanismo sequenziale ordinato, con il corismato primo legante:
(2)
]
][
[
]
[
]
][
[
2 2 max 2 + ++
+corismato
corismato
Mg
K
K
Mg
corismato
V
Mg corismato vi =I dati nella Figura 12A forniscono le stime iniziali delle costanti cinetiche Kcorismato e KMg2+. Stime più accurate sono state ottenute dagli esperimenti nei
quali la concentrazione di un substrato era variata attraverso un ampio campo, mentre l’altra era mantenuta costante al suo valore stimato di KM. Questo dava
Kcorismato = 0,50 ± 0,05 mM e KMg2+ = 10 ± 1 µM.
Figura 12: Analisi cinetica di ADCS. (A) Plot doppio-reciproco che mostra l’effetto dedi Mg++ sul legame del corismato. Questo e altri dati (non mostrati) definitivamente dimostrano che il meccanismo cinetico per ADCS è ordinato, con il corismato che si lega per primo e Mg++ per secondo. Le concentrazioni del corismato sono 50, 100, 250, 375, and 650 µíM. (B) Plot doppio-reciproco che mostra che P10 è competitivo verso il corismato. Le concentrazioni di P10 sono 0, 10, 25, 40 mM. (C) Plot doppio-reciproco che mostra che P10 è competitivo verso Mg++. Le concentrazioni di P10 sono0, 15, 22.5, 30 mM.
Ciascun inibitore era analizzato contro l’ADCS variando le concentrazioni dell’inibitore attraverso un campo appropriato e mantenendo costante la concentrazione del corismato a 13 µM (il valore di KM alle condizioni di analisi).
La forte assorbanza sotto i 300 nm dovuta agli inibitori necessitava dell’uso di un secondo enzima di accoppiamento, l’LDH (lattato deidrogenasi). I tassi iniziali erano misurati seguendo la riduzione nell’assorbanza a 340 nm dovuta all’ossidazione di NADH (nicotin-amide-adenindinucleotide). I dati erano adattati alla seguente equazione di inibizione competitiva:
(3)
Come giustificazione di ciò, P9, P10, 4, 5, 7, e L5 erano soggetti ad un’ulteriore analisi cinetica, in cui le concentrazioni del corismato e dell’inibitore erano variate per ciascuno. L’analisi Lineweaver-Burk (doppio diagramma reciproco) dei dati rivelava che ogni composto era un inibitore competitivo dell’ADCS. I dati ottenuti per P10 sono presentati nella Figura 12B come esempio rappresentativo. Le analisi cinetiche erano effettuate per tutti i ligandi identificati. Tutti hanno valori Ki relativi al P10 (Figura 10).
Sorprendentemente, i composti P10, 4, 5, e L5 erano tutti competitivi per quanto riguarda l’Mg2+ come pure per il corismato. Come esempio rappresentativo, la Figura 12C illustra i dati ottenuti per P10. Veniva usato il saggio dell’LDH-accoppiato. Le concentrazioni dell’inibitore e dell’Mg2+ erano variate, mentre il corismato era mantenuto costante a 200 µM.
vi =
]
[
]
[
]
[
maxcorismato
K
I
K
corismato
V
i M
+
La via dello scichimato converte il glucosio nel corismato, il metabolita del punto di ramificazione per la biosintesi dei composti carbociclici aromatici nei sistemi biologici. Cinque vie metaboliche primarie partono dal corismato (Phe/Tyr, salicilato/siderofori, PABA/folati, Trp, e chinoni di trasferimento di elettroni), ma pure parecchi metaboliti secondari iniziano con il corismato [36]. Queste vie, inclusa la stessa via dello scichimato, sono assenti nell’uomo; i loro prodotti sono componenti essenziali della nostra dieta. L’attrattività di queste vie come bersagli antimicrobici è stata riconosciuta da tempo [37, 38]. Diversi farmaci/composti efficaci oggi in commercio agiscono su queste vie, incluso l’erbicida glifosato, i sulfamidici, e altri.
L’ADCS, l’antranilato sintasi (AS), e l’isocorismato sintasi (IS) sono tre enzimi meccanicisticamente omologhi utilizzanti il corismato e danno inizio rispettivamente alla biosintesi del PABA, del Trp e del salicilato. L’obbiettivo di scoperta di un farmaco antimicrobico è trovare un composto che sia un inibitore del legame di uno o più di questi enzimi, dal momento che ognuna di queste vie è critica per la patogenicità dei batteri [39, 40]. Un composto che ne inibisca più di uno avrà una probabilità radicalmente ridotta che si sviluppi la resistenza primaria per opera di una mutazione simultanea di due o più di questi enzimi. Sarebbe equivalente a un cocktail multifarmaco in una molecola singola di inibitore. L’alto grado di similarità tra questi enzimi rende la ricerca più facile. Le strutture di questi enzimi mostrano tutte che i siti attivi sono rimossi dal solvente ma non sono nascosti così in profondità nella proteina interna.
Sono stati progettati inibitori con un progetto “a fasi” nel quale si ha una fase di PAYLOAD che si dirige al sito attivo, una fase dello SPAZIATORE che si estende fuori dal sito attivo, e una fase COMBI che è il fulcro della chimica combinatoria e che dovrebbe essere in grado di rendere produttive le interazioni di legame con la superficie dell’enzima vicina al sito attivo.
Il primo punto era trovare un analogo stabile del corismato che sarebbe stato facilmente soggetto alla sintesi in fase solida e avrebbe diretto la libreria degli inibitori al sito attivo. Sulla base di precedenti studi, è stato definito che, una catena laterale contenente un gruppo carbossilato legato tramite uno o due carboni era essenziale per il legame stretto all’IS e all’AS [41, 42]. A questo scopo, sono stati esaminati i composti mostrati in Figura 10 quali candidati possibili. Il P10 era scelto come il miglior compromesso tra tutti questi requisiti. Il secondo componente del progetto è lo SPAZIATORE. Questa porzione sarà il bersaglio di diversificazione e di ottimizzazione attraverso la chimica combinatoria e lo screening con l’enzima marcato. Inizialmente è stata utilizzata la lisina come SPAZIATORE; la modellazione mostrava che questa è un’eccellente prima approssimazione alla lunghezza richiesta per andare fuori del sito attivo alla fessura della superficie, dove la porzione COMBI avrebbe avuto le opportunità per interazioni di serendipità con i residui vicino alla superificie. La lisina contiene anche i gruppi funzionali α-ammino e α-carbossilato che sono facilmente riconducibili all’elaborazione in fase solida con gli elementi combinatoriali.
Una serie di 48 acidi venivano scelti come elementi di diversità per l’acilazione delle α-ammine e una serie di 48 aminoacidi erano scelti come elementi di diversità per l’accoppiamento all’α-carbossilato della lisina, portando alla nostra libreria OBOC di 2304 composti. La codifica delle strutture era raggiunta con uno schema di etichettatura dello spettro di massa, impiegando perline di resina segregate topologicamente, che consentivano un’identificazione rapida e semplice della struttura dei composti del test.
Ottenuta la libreria, è stata preclassificata usando lo strumento COPAS per accrescere il segnale in rapporto al rumore per i composti “hit”, rimuovendo le perline autofluorescenti che interferiscono con l’identificazione delle perline ADCS-legate. Questa preclassificazione possiede un impatto maggiore sulla
nostra abilità di identificare le perline ADCS-legate con una bassa frequenza di falso positivo, poi il 6% delle perline veniva rimosso nella preclassificazione e circa il 2% delle perline preclassificate era incubato con quelle ADCS marcate che erano fluorescenti.
La Figura 11 mostra 11 strutture ottenute dalle 15 perline più luminose scelte in uno screen ADCS. Queste erano risintetizzate sulla resina a base Rink ammide, e le costanti di inibizione di quelle sufficientemente solubili per essere analizzate sono riportate sotto le strutture. Questi composti non sono strettamente legati con qualsiasi standard, ma ricoprono il ruolo veramente importante di dimostrare che il progetto dell’inibitore “staged” è una linea promettente di ricerca. Ognuno degli inbitori di fase saggiati ha una costante di inibizione sostanzialmente più bassa di P10, indicando che le porzioni dello SPAZIATORE e/o del COMBI forniscono significativi aumenti nell’affinità di legame.
L’inibitore del legame più stretto identificato è L5 con un valore di Ki di 360 µM.
I composti mostrati nella Figura 13 sono stati progettati e sintetizzati per rendere meno complessi i contributi all’affinità di legame da parte dei vari componenti.
Figura 13: Strutture e valori di Ki per inibitori elaborati e derivati
Sorprendentemente, la conversione di P10 in un ammide semplice (4) non aveva un effetto significativo sull’affinità di legame. Pure un’ulteriore elaborazione attraverso la formazione dell’ammide con il gruppo ∈-ammino dell’N-acetil-L lisina ammide (5) non altera in maniera significativa l’affinità di legame dell’inibitore basato sul P10, dimostrando che qualsiasi interazione di potenziale negativo tra la lisina e l’enzima è compensata da quelle positive e, cosa ancora più importante, che tutte le interazioni che guidano ad un incremento di circa 100 volte nell’affinità di legame di L5 paragonato al P10 sono totalmente dovute alle interazioni fatte tra la porzione COMBI e la superficie dell’enzima prossimale al sito attivo.
Il meccanismo cinetico dell’ADCS non è stato descritto in letteratura. Ma è importante comprendere se il legame del corismato e dell’Mg2+ obbedisca a un meccanismo casuale o a un meccanismo sequenziale ordinato così che l’effetto
delle loro concentrazioni sul legame dell’inibitore possa essere verificato. I risultati degli studi [3] mostrano che l’ADCS obbedisce a un meccanismo cinetico strettamente regolato con il primo legame del corismato e quello dell’Mg2+ come secondo:
Le analisi di piena inibizione di parecchi inibitori, inclusi il P10, il 4, il 5 e l’L5, mostrano che essi sono tutti competitivi rispetto sia al corismato sia all’Mg2+. I risultati per il P10 sono evidenziati nella Figura 12B. Questo era atteso per gli ultimi composti, ma non per il P10. Si tratta di uno stretto analogo del corismato, il cui gruppo C1 carbossilato è un legante bidentato per Mg2+ nel complesso attivo dell’enzima [10].
Dato che il P10 è strutturalmente abbastanza differente dal corismato per precludere il legame all’Mg2+, allora il confronto corretto per il legame non è quello al corismato nel complesso ternario con l’enzima e Mg2+, ma piuttosto quello al corismato nel complesso binario con il solo enzima. Questo valore,
Kcorismato nel meccanismo cinetico regolato, è risultato essere 0,5 mM, abbastanza
più grande del valore di 13 µM misurato in precedenza per il corismato in presenza di Mg2+ 5 mM [43]. Perciò, il P10 si lega circa 60 volte più debolmente del corismato per dare l’ equivalente complesso con l’enzima. L’inibizione competitiva nei confronti sia del corismato sia dell’Mg2+ per mezzo di analoghi
E + corismato
K
corismatoE · corismato
+ 2
Mg
K
E · corismato ·Mg2+ Kcat E + ADC + Mg2+
semplici del corismato con l’ADCS, ma non con l’antranilato sintasi o l’isocorismato sintasi, può spiegare i precedenti studi sull’inibizione che mostravano che gli analoghi del corismato, in genere, si legano più strettamente a questi ultimi due enzimi, in confronto all’ADCS in presenza di Mg2+ [42]. L’inibizione competitiva del P10 riguardo sia al corismato sia all’Mg2+ fornisce una spiegazione soddisfacente riguardo al perché la conversione del P10 nel suo derivato ammidico 4 non incrementa in modo drastico la costante di inibizione. Ci si potrebbe aspettare che la conversione del gruppo carbossilato all’ammide ostacolasse il legame energeticamente favorevole all’ Mg2+ se il P10, infatti, permettesse il legame all’Mg2+. L’osservazione che la conversione all’ammide semplice possiede un effetto energetico insignificante è un’ulteriore prova che l’Mg2+ non si lega al complesso ADCS-P10. Inoltre, questo suggerisce pure che la carica negativa del gruppo aril carbossilato non contribuisce in modo significativo al proprio legame (o che il gruppo ammidico fornisce le interazioni energeticamente equivalenti).
Valori simili di Ki per il composto 5 paragonati al P10 e al 4 implicano che la
porzione di SPAZIATORE della lisina non va incontro né a buone né a cattive interazioni (tutt’al più sono uguali) con l’enzima mentre attraversa il sito di legame Mg2+ sulla superficie dell’enzima. Questo risultato positivo dimostra che l’inquinamento sterico del linker è prontamente accomodata. La lisina come SPAZIATORE era stata scelta per la sua facilità di sintesi. Uno sforzo combinatorio mirato a ottimizzare la combinazione SPAZIATORE-PAYLOAD ha la potenzialità di generare un’energia di legame sostanziale dalla porzione dello SPAZIATORE, dati i diversi glutammati (che formano la maggior parte del sito di legame dell’Mg2+) e altri gruppi polari in questa regione. Retrospettivamente, veniva fatto uno sforzo iniziale per catturare una parte di questa energia di legame con la sintesi di 7. Era deludente scoprire che la conversione dell’ammide di 5 alla benzilammina di 7 non generava sostanziali interazioni di legame addizionali. Questo può essere dovuto alla posizione
subottimale del gruppo amminico a carica positiva che era inteso catturare una parte dell’energia di legame di cui dispone Mg2+. Tuttavia l’ottimizzazione dello SPAZIATORE tramite un programma combinatorio non basato sui peptidi potrebbe rimediare a questo.
Infine, era delineato l’effetto del linker della resina sull’affinità dell’ADCS per i composti dei test resina-legati. La Figura 13 mostra le strutture dei derivati L3 e L5, dove un analogo strutturale del linker è attaccato al carbossilato usato per l’attacco della resina. Sia per L3 che per L5, l’inclusione dell’analogo del linker aveva soltanto un effetto minore sull’affinità del composto stesso nel test per l’ADCS. Questo risultato gratificante dimostrava che l’attacco covalente del composto del test alla resina tramite il linker aveva effetti deleteri trascurabili sull’affinità dell’enzima per questo.
Questi risultati hanno dimostrato l’applicabilità di un paradigma generale per la scoperta di un farmaco, basato sulla chimica combinatoria e sul progetto di un inibitore “di fase”. La grande maggioranza degli enzimi legano i loro substrati nel sito attivo che è vicino alla superficie della proteina e hanno un canale aperto che guida al solvente. Una porzione simile al substrato che diriga una molecola al sito attivo dell’enzima, persino se essa ha da sola un’affinità relativamente bassa, può essere elaborata per avvantaggiarsi delle interazioni di serendipità che possono condurre ad un’affinità elevata. L’uso dell’enzima fluorescentemente marcato per individuare i composti dei test legati alla superficie permetteva, infine, un’automazione totale di questa procedura semplice di screening tramite una preclassificazione, basata sul COPAS, della libreria, seguita dalla classificazione delle reazioni di legame dell’enzima, in cui le perline diventano fluorescenti a causa dell’associazione dell’enzima ai ligandi di superficie.