Universit`a di Pisa
FACOLT`A DI SCIENZE MATEMATICHE FISICHE E NATURALI Corso di Laurea Specialistica in Biodiversit`a ed Evoluzione
Analisi paleogenetiche condotte sui reperti umani rinvenuti
nella necropoli romana del Frizzone (Lucca)
Candidato:
Anna Sandionigi
Matricola 264303
Relatori:
Prof. Francesco Mallegni
Prof. David Caramelli
Correlatore:
Dott. Alessandro Manfredini
Universit`a di Pisa
FACOLT`A DI SCIENZE MATEMATICHE FISICHE E NATURALI Corso di Laurea Specialistica in Biodiversit`a ed Evoluzione
Analisi paleogenetiche condotte sui reperti umani rinvenuti
nella necropoli romana del Frizzone (Lucca)
Candidato:
Anna Sandionigi
Matricola 264303
Relatori:
Prof. Francesco Mallegni
Prof. David Caramelli
Correlatore:
Dott. Alessandro Manfredini
S O M M A R I O
In generale, si pensa che la diversità genomica entro e fra popolazio-ni moderne sia frutto dei cambiamenti demografici e delle pressiopopolazio-ni evolutive che hanno interessato i loro antenati preistorici e proto-storici
(Ammerman e Cavalli Sforza,1984; vonHaeseler, A. et al., 1995;
Ri-chards, M.B. et al., 2000; Torroni, A. et al., 2001; Underhill, P.A. et
al., 2001). L’ipotesi principale è che i processi che hanno determinato la variabilità genetica complessiva di una nazione intera, come l’Italia, debbano essere avvenuti quando la densità di popolazione era bassa, cioé in tempi molto remoti (Piazza, A. et al., 1988). Finora però, non è stato possibile verificare pienamente questa assunzione, proprio perché non esistono molti dati sulle popolazioni antiche e quelli che esistono sono in parte contrastanti.
Dati recenti sulla popolazione Etrusca (Vernesi, C. et al.,2004) fanno pensare che ci sia una discontinuità genetica tra questa e le attuali popo-lazioni che abitano la medesima area; inoltre da uno dei pochi studi su larga scala disponibili sul DNA antico è stato dimostrato come un dra-stico cambiamento nel pool genico mitocondriale possa richiedere an-che solo pochi secoli (Wang, L. et al.,2000). Alla luce di quanto esposto appare perciò necessario raccogliere dati su altre popolazioni antiche, e paragonare gli individui moderni ed antichi in varie località e in tempi differenti al fine eseguire uno studio genetico diacronico e sincronico. Per questo motivo nasce il progetto “Eredità genetica dell’Italia Antica” in cui questa tesi è inserita.
Dai 13 inumati analizzati in questo studio provenienti dalla necropoli del Frizzone in provincia di Lucca (II sec. a.C., I sec. d.C.), sono state ricostruite 8 sequenze complete, attraverso tecniche di antropologia mo-lecolare /paleogenetica, della lunghezza di 360 bp della regione HVR-I (16024-16383), la regione di controllo (D-Loop) del DNA mitocondriale (mtDNA).
Il confronto di queste sequenze mitocondriali attraverso il tempo e lo spazio dovrebbe permettere di ricostruire uno scenario più realistico di quelli che erano i rapporti fra i lucchesi antichi, fra essi e le altre popolazioni italiche antiche e le popolazioni che vivono ora nelle stesse aree.
Lo scopo ultimo di questo progetto è introdurre una dimensione tem-porale nello studio della variabilità genetica, che ci permetta di com-prendere con quanta rapidità le linee mitocondriali vengano rimpiazza-te attraverso il rimpiazza-tempo, e quindi che livelli di continuità genealogica esi-stano fra popolazioni che hanno occupato la stessa regione geografica in diversi intervalli di tempo.
I N D I C E
1 i n t r o d u z i o n e 1 1.1 Il DNA antico 1
1.2 Il popolamento dell’Europa 4 1.3 Le popolazioni antiche italiane 9 1.4 Il caso delle popolazioni lucchesi 13
1.5 Studi genetici sulla popolazione Etrusca 15 1.6 Gli scavi del Frizzone 17
2 m a t e r i a l i 21
2.1 Provenienza dei campioni 21 2.2 Il DNA mitocondriale 25 3 m e t o d i 29
3.1 Il problema delle contaminazioni 29 3.1.1 La degradazione dell’ aDNA 29 3.1.2 I Golden Criteria 34
3.1.3 Il recupero dei reperti 35
3.2 Analisi di laboratorio. Metodologie applicate 36 3.2.1 Il laboratorio 36
3.2.2 La pulizia del campione ed estrazione del DNA 38 3.2.3 La PCR 39
3.2.4 Il controllo dei prodotti di PCR 42 3.2.5 Il clonaggio 44
3.2.6 Il sequenziamento 47
3.2.7 La ricostruzione della sequenza consenso 48 4 m e t o d o l o g i e d i a n a l i s i d e i d a t i 51
4.1 Indici di diversità standard e genetica 51
4.1.1 Analisi a livello intra-popolazionistico 51 4.1.2 Analisi a livello inter-popolazionistico 54 4.2 Software per la visualizzazione grafica 56
4.2.1 MultiDimensional Scaling (MDS) 56 4.2.2 Median-Joining Network 57
4.3 I dati analizzati 57
viii i n d i c e
5 r i s u l t a t i e d i s c u s s i o n e 61
5.1 Indici di diversità genetica nei campioni lucchesi 62 5.2 Confronti tra le popolazioni antiche e moderne 67 6 c o n c l u s i o n i e p r o s p e t t i v e f u t u r e 75
1
I N T R O D U Z I O N E
1.1 i l d n a a n t i c o
La ricostruzione a livello molecolare della storia della nostra specie si era basata, fino a poco tempo fa, unicamente sullo studio di DNA mo-derno campionato in popolazioni attuali. Gli studi fondati sulla moltitu-dine di dati ricavati cercavano di evidenziare le potenziali relazioni tra queste popolazioni al fine di poter formulare delle ipotesi sui plausibili processi storici che hanno portato all’attuale variabilità genetica.
Le inferenze che si possono ottenere dall’analisi della struttura dei geni attuali si basano su supposti teorici non necessariamente certi e che non possono essere verificati, non esistendo, infatti, dati relativi alle popolazioni antiche coinvolte in questi processi.
Negli ultimi anni, grazie allo sviluppo di nuove tecnologie, è stato possibile raccogliere non solo i dati provenienti da popolazioni attuali, ma anche informazioni di pari qualità da popolazioni antiche utiliz-zando resti archeologici. La scienza che si occupa di investigare questo campo è chiamata Archeo-Antropologia Molecolare. La possibilità di ricostruire l’evoluzione molecolare (definita come un processo storico mediante il quale il DNA, portatore dell’informazione genetica di tutti gli esseri viventi, accumula mutazioni neutre o dovute alla selezione naturale) attraverso una comparazione diacronica delle sequenze nu-cleotidiche di gruppi umani moderni e antichi, diventa un potente stru-mento per tentare di rispondere a domande comuni sia agli antropologi che agli archeologi.
Per “DNA antico” (aDNA = ancient DNA) si intendono i residui di materiale genetico estraibili da qualsiasi tipo di materiale biologico, di-stinto per età, origine e stato di conservazione, quali ossa, resti mummi-ficati animali o vegetali, preparati istologici, manufatti e coproliti.
2 i n t r o d u z i o n e
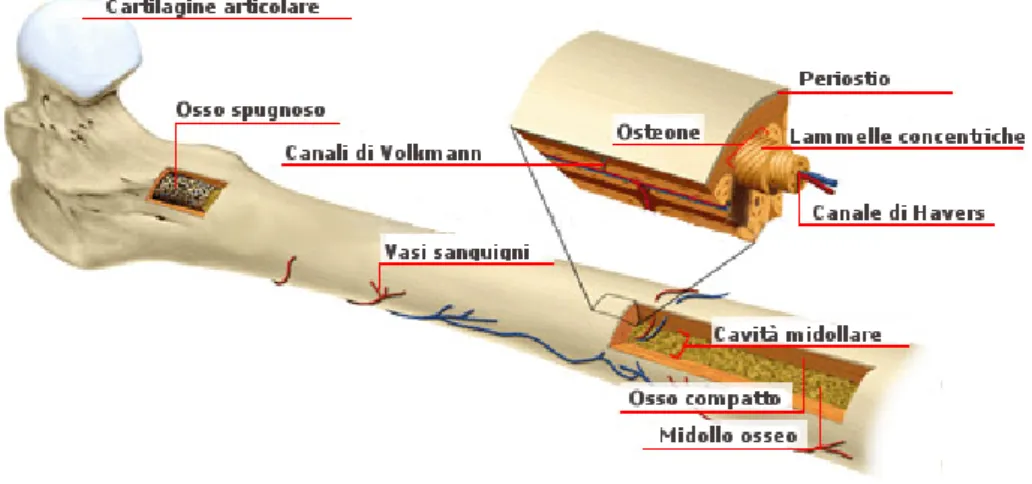
Figura 1: Struttura ossea.
Il DNA che è possibile recuperare dai resti scheletrici antichi si com-pone principalmente di materiale estratto dal tessuto osseo.
Le ossa sono composte da due componenti (Figura1). Una è la com-ponente cellulare - formata principalmente da osteociti, cellule che han-no completato il loro sviluppo e che sohan-no comprese nelle lacune dove, protetti dalla matrice interstiziale calcificata vanno incontro solo a de-gradazione autolitica, essendo meno soggetti all’azione di enzimi litici provenienti da microrganismi o da altri elementi cellulari.
L’altra componente è la matrice, composta da fibre di collagene inter-calate con cristalli di idrossiapatite.
Uno dei due principali tipi di sistemi genici utilizzati negli studi mo-lecolari, sulla storia e l’evoluzione umana, è indubbiamente il DNA mi-tocondriale (mtDNA), l’altro è il cromosoma Y. Il motivo di tanto succes-so è dovuto ai suoi numerosi vantaggi. In primo luogo è presente in un
1.1 il dna antico 3
gran numero di copie (circa 103-104 genomi per cellula) e a differenza di quanto avviene per i geni nucleari, di cui si hanno solo due copie per cellula, esiste un’elevata probabilità di ritrovarne qualche copia anche in campioni molto degradati, come possono essere i resti antichi. Un altro vantaggio è che le sequenze di DNA mitocondriale accumulano mutazioni rapide e costanti nel tempo, che non alterano le funzioni del mitocondrio e sono di fatto, da considerarsi neutre cioè non soggette all’eliminazione della selezione naturale.
Grazie a questa caratteristica il DNA mitocondriale può essere con-siderato un ottimo orologio molecolare il cui rapido ticchettio è fonda-mentale per individuare cambi genetici caratterizzati da tempi evolutivi brevi.
Il tasso di evoluzione medio per l’intera molecola di mtDNA è stato stimato a partire da quei gruppi tassonomici per i quali erano dispo-nibili i tempi di divergenza ottenuti da fossili, da dati biodemografici o proteici. Questo tasso è risultato valido per molti raggruppamenti e pari all’1-2% di basi mutate per milione di anni.
Un altro fondamentale attributo di questo sistema genico, che lo ha re-so tanto importante negli studi filogenetici riguardanti l’uomo, è la sua trasmissione ereditaria per via unicamente materna, senza fenomeni quali la ricombinazione e la segregazione.
Partendo da questi assunti si è potuta formulare l’ipotesi dell’ “Eva” mitocondriale secondo cui, la linea di discendenza mitocondriale è do-vuta ad una sola antenata che ha trasmesso il suo mtDNA inalterato eccetto per mutazioni casuali (Wilson e Cann, 1992).
Partendo dal presupposto, infatti, che il numero di differenze nelle sequenze di mtDNA che separa due individui è direttamente propor-zionale al tempo a partire dal quale essi hanno condiviso la stessa an-tenata, é possibile costruire alberi filogenetici che riflettono la storia genealogica di un individuo, di una popolazione o di una specie da parte materna. Il grado di somiglianza diminuisce più si risale lungo l’albero, fino ad includere tutta l’umanità.
4 i n t r o d u z i o n e
1.2 i l p o p o l a m e n t o d e l l’europa
In base ai tempi di coalescenza calcolati alle radici dei primi aplogruppi presenti in Europa, è stato stimato che circa 40.000 anni fa, nel Paleoli-tico Superiore europeo, Homo sapiens iniziò la sua diffusione sul conti-nente, arrivando a colonizzare tutte le aree libere dai ghiacci in breve tempo.
Successivamente, durante il massimo picco dell’ultima glaciazione circa 18.000 anni fa, la presenza umana si restrinse alle zone più cal-de cal-del sud (rifugi glaciali) e solo più tardi, con il ritirarsi cal-dei ghiacci, riapparse più a nord (Mesolitico, 11.000-7.000 anni fa).
A seguito delle prime testimonianze di sviluppo dell’agricoltura e della nascita dei primi insediamenti permanenti nel Vicino Oriente cir-ca 10.000 anni fa, in una nuova fase culturale denominata Neolitico, i manufatti tipici di tale periodo si diffusero gradualmente verso ovest e verso nord lungo le medesime rotte della precedente colonizzazione paleolitica.
Non sono documentati successivi cambiamenti demografici che ri-guardano l’Europa nel suo complesso, perciò da un punto di vista ge-netico, la struttura degli attuali europei dovrebbe riflettere gli effetti o della prima colonizzazione paleolitica, o della riespansione mesoliti-ca, o della diffusione della cultura neolitimesoliti-ca, oppure di tutti questi tre processi insieme (Caramelli, 2009).
Il concetto di storia genetica dell’Europa è stato studiato da Luca Lui-gi Cavalli Sforza e collaboratori negli anni ’70, quando per la prima vol-ta si è provato ad applicare i dati genetici a studi di carattere
archeologi-co (Ammerman e Cavalli Sforza, 1984). I ricercatori assunsero come
ipotesi di partenza che l’incremento demografico nelle zone di origine dell’agricoltura avesse promosso una costante e regolare espansione de-mica, motivata dalla ricerca di nuove terre da coltivare e che dal Medio Oriente (la regione della “mezzaluna fertile”), dove l’agricoltura si svi-luppò circa 10.000 anni fa, partì l’espansione degli agricoltori-pastori verso Nord-Ovest.
I dati archeologici testimoniano l’arrivo dell’agricoltura nei Paesi Bal-tici e nelle isole Britanniche, le ultime ad essere raggiunte dal
movi-1.2 il popolamento dell’europa 5
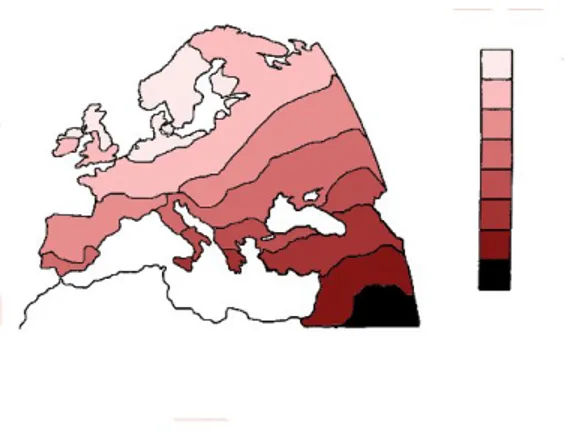
Figura 2: Cline Sud-Est Nord-Ovest di diffusione della cultura neolitica.
mento migratorio, fra i 5.500 e 4.200 anni fa. L’espansione verosimil-mente comportava via via commistione con popolazioni preesistenti, di dimensioni numericamente piuttosto ridotte, discendenti dei cacciatori-raccoglitori paleolitici.
I dati genetici ottenuti con i polimorfismi proteici e i polimorfismi del DNA nucleare supportano bene questo quadro: l’analisi combina-ta delle variazioni di frequenze alleliche a molti loci mostra un chiaro gradiente genetico, cioè variazioni graduali e regolari di frequenze ge-niche (clini) da Sud-Est a Nord-Ovest. L’andamento del gradiente gene-tico è sovrapponibile all’andamento della diffusione dell’agricoltura in Europa.
I dati pubblicati negli ultimi dieci anni, ottenuti con mtDNA e inte-grabili con quelli ricavati dall’analisi del cromosoma Y, sono piu’ com-plessi da interpretare e la distribuzione degli aplogruppi non mostra un chiaro andamento clinale.
Un quadro filogeografico europeo è stato ottenuto attraverso lo studio dei sistemi non-ricombinanti, soprattutto con l’uso dell’analisi del fon-datore che permette di datare la colonizzazione di una regione tramite la tecnica dell’orologio molecolare.
Applicando a questo principio i dati della variabilità mitocondriale europea, M.B. Richards (Richards, M.B. et al., 1996, 1998) ha calcola-to che circa tre quarti dei moderni aplogruppi micalcola-tocondriali deriva da
6 i n t r o d u z i o n e
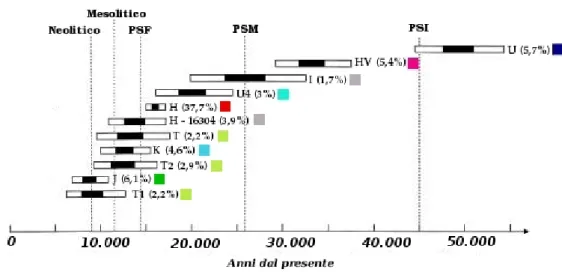
Figura 3: Età degli 11 fondatori principali degli aplogruppi mitocondriali europei; le barre bianche e nere indicano gli intervalli di confidenza delle età stimate al 95% e al 50%; i quadrati colorati a lato delle percentuali si riferiscono alla Figura 4 nella pagina successiva e alla Figura5 nella pagina8. Modificato
da (Richards, M.B. et al.,2000) .
solo 11 antenati di cui solo un piccolo gruppo è riconducibile al Neoli-tico, a partire da 7.000 anni fa, mentre la maggior parte sembra essersi originata tra i 15.000 e i 50.000 anni fa, nel Paleolitico Superiore.
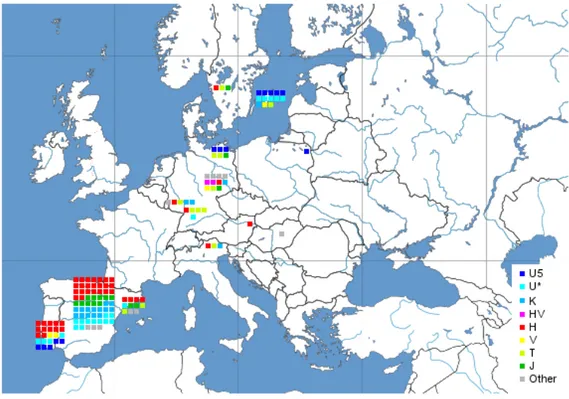
Gli aplogruppi mitocondriali, che contribuiscono maggiormente alla prima componente principale sono H, pre-V e U5, concentrati al polo europeo, e pre- HV1 e U1, concentrati al polo del Vicino Oriente.
L’analisi del fondatore suggerisce che l’aplogruppo U5 apparve in Europa molto presto, circa 45.000 anni fa, mentre gli aplogruppi H e pre-V arrivarono durante il Paleolitico Superiore medio e si riespansero dopo il picco dell’ultima glaciazione (18.000 anni fa).
L’aplogruppo H è il più frequente sia in Europa che nel Vicino Orien-te, ma la maggiore rappresentatività si trova nell’Europa occidentale (circa 45-50% in quasi tutte le popolazioni, 60% nei Baschi) rispetto al Vicino Oriente (circa 25-30%), e ha quindi un andamento opposto a quello che ci si dovrebbe aspettare dalla diffusione demica.
1.2 il popolamento dell’europa 7
Viceversa, i principali fondatori neolitici dovrebbero essere stati mem-bri degli aplogruppi J e T1, e nessuno dei due è un fattore determinante nella prima componente principale (Caramelli, 2009), il loro apporto è infatti stimato non superiore al 20%.
Per supportare meglio il modello del popolamento europeo, sono state effettuati studi utilizzando il cromosoma Y.
La componente autoctona europea è stata identificata come aplogrup-po R* mentre quella neolitica del Vicino Oriente come aplogrupaplogrup-po J, in
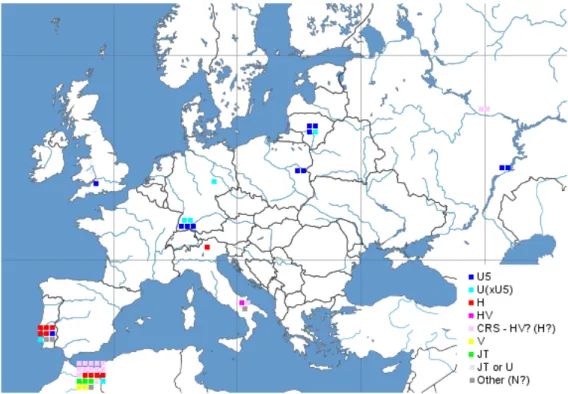
Figura 4: Mappa della distribuzione degli aplogruppi mitocondriali in Europa re-lativa al Paleolitico. E’ da notare la mancanza di mtDNA H, l’aplogrup-po europeo più diffuso oggi, nella maggior parte o forse anche in tutti i campioni dell’ Europa Centrale/Nord/Est, invece dominata da U5. D’al-tra parte, H è presente in maniera dominante nel Portogallo e nel Ma-rocco pre-neolitico e il suo precursore, HV, è stato scoperto in reperti di età Gravettiana in Italia. http://leherensuge.blogspot.com/2009/10/ european-ancient-mtdna-in-sequential.html
8 i n t r o d u z i o n e
analogia con il marcatore neolitico nel DNA mitocondriale. L’aplogrup-po J mostra un gradiente clinale simile a quello della prima comL’aplogrup-ponen- componen-te principale dei marcatori classici, mentre l’ aplogruppo R* è molto comune nell’Europa occidentale e decresce spostandosi verso Est.
Volendo quindi delineare un quadro generale del periodo Neolitico, si può vedere che per quanto riguarda il DNA mitocondriale il mag-giore apporto è a livello del sudest Europeo, nelle regioni centrali e nel nord, al contrario delle regioni mediterranee dove sembra essere stato minore (Richards, M.B. et al., 2000). Diversamente gli aplogruppi del
Figura 5: Mappa della distribuzione degli aplogruppi mitocondriali in Euro-pa relativa al Neolitico. L’alta presenza in territorio basco dell’a-plogruppo H mostra una certa continuità con le popolazioni pre-Neolitiche similmente alla condizione Portoghese. Così come la presen-za diffusa di U5. Appaiono in questa mappa le componenti J e T1 assenti in quella Paleolitica.http://leherensuge.blogspot.com/2009/10/ european-ancient-mtdna-in-sequential.html
1.3 le popolazioni antiche italiane 9
cromosoma Y riferiti al Neolitico sono molto comuni proprio nella zo-na mediterranea e nell’Europa centrale, ma sono praticamente assenti in nord Europa e nelle Isole Britanniche (Semino, O. et al., 2000). Que-sti dati suggeriscono l’ipotesi per cui la colonizzazione dell’Europa sia stata caratterizzata da una serie di movimenti non bilanciati di gruppi di soli uomini o sole donne (Perlès, 2001; Wilson, J.F. et al.,2001).
1.3 l e p o p o l a z i o n i a n t i c h e i t a l i a n e
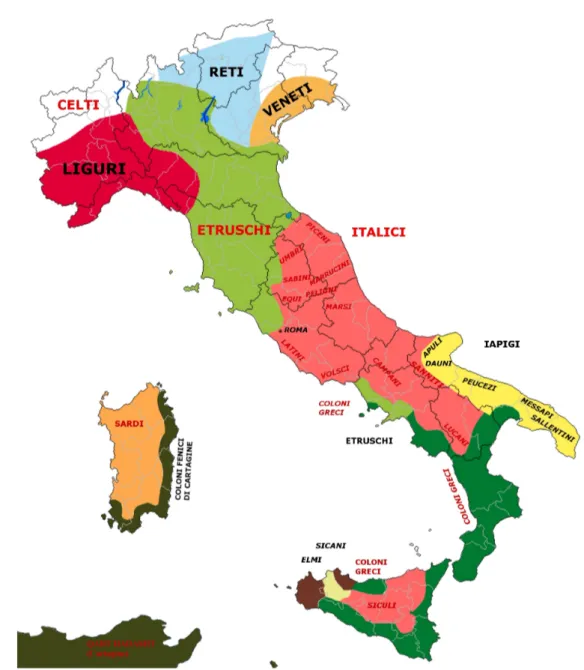
L’ampia eterogeneità geografica del suolo italiano portò, fin dalle epo-che antiepo-che, a una forte differenziazione, nell’origine, nella lingua e nel tipo di cultura, tra le stirpi presenti sul territorio.
L’Italia antica prima della conquista romana si caratterizza infatti per la presenza di popolazioni indoeuropee, stanziatesi nella penisola a partire dal II millennio a.C., che vanno sotto il nome di Italici.
Nell’Italia Nord orientale il nucleo preponderante è costituito dai Ve-neti che già al volgere del II millennio e per tutto il I svilupparono la loro civiltà.
Nell’odierno Lazio, tra l’Appenino e la costa, erano stanziati i La-tini, i Volsci e gli Equi; mentre dall’altra parte dell’Appennino, fino a sconfinare nelle odierne Umbria e Marche, i Sabini, gli Umbri, i Marsi, i Peligni, i Marrucini, i Piceni. Più a Sud, in parziale corrispondenza della fascia montuosa appenninica tra gli odierni Abruzzo e Campania, gli Osci; nell’odierna Puglia gli Iapigi, e nell’odierna Basilicata e Calabria i Lucani e i Bruzi.
Il quadro dell’Italia si completa con due popoli di origine non in-doeuropea, i Liguri, abitanti una vasta regione tra Arno e Rodano e gli Etruschi, che dall’Etruria (Toscana e alto Lazio) si espansero fino alla Pianura Padana. Bisogna inoltre considerare le più recenti migrazioni galliche nel Nord Italia e della fiorente colonizzazione ellenica della Magna Grecia.
In un quadro così complesso si comprende come il popolamento del-l’Italia sia sempre stato un argomento molto dibattuto sopratutto a li-vello multidisciplinare. In particolare recenti studi a carattere genetico
10 i n t r o d u z i o n e
condotti sulla popolazione italiana attuale sembrano dimostrare come l’Italia sia un mosaico di gruppi etnici ben differenziati.
L’ipotesi maggiormente accertata è che la romanizzazione dell’Ita-lia e dell’Europa non abbia cambiato in modo sostanziale la struttura genetica delle popolazioni conquistate.
A fronte di tutto questo, anche se i dati sulle popolazioni antiche sono pochi e in parte contrastanti, si ritiene che la diversità genomica entro e fra popolazioni moderne sia frutto dei cambiamenti demografici e delle pressioni evolutive che hanno agito a livello degli antenati preistorici e protostorici (Ammerman e Cavalli Sforza,1984;Richards, M.B. et al.,
2000;Torroni, A. et al., 2001). Il pensiero comune, anche se non ancora
verificato, è che i processi che hanno determinato la variabilità genetica complessiva di una nazione intera, come l’Italia, debbano essere avve-nuti quando la densità di popolazione era bassa, quindi in tempi molto remoti.
Al contrario della maggior parte dell’Europa, l’Italia sembra essere ca-ratterizzata da una continuità tra le popolazioni del Paleolitico medio superiore e quelle del Paleolitico tardo superiore (Leighton, 1999),non andando incontro ad uno spopolamento durante l’ultima glaciazione e mantenendo una sostanziale parte delle linee mitocondriali e del NRY (Non-Recombining Y-chromosome= porzione non ricombinante del cromo-soma Y) del Paleolitico medio superiore.
In questi anni sono state effettuate indagini su alcune popolazioni ita-liche tra cui la popolazione Etrusca (VIII-III sec. a.C.) (Vernesi, C. et al., 2004), quella Picena (VIII-IV a.C.) (Cappellini, 2003) e quella Nuragica (XI-IX sec. a.C.) (Sanna, 2003). Secondo gli studi condotti sull’insieme dei dati ricavati, la notevole omogeneità rilevata all’interno del gruppo dei reperti neolitici e la stretta relazione evidenziata in tutte le anali-si inter popolazionistiche effettuate tra il gruppo dei paleolitici e quello dei neolitici, induce a pensare che il substrato di partenza da cui si sono originate le diverse comunità del periodo neolitico italiano fosse quello paleo-mesolitico autoctono.
Secondo il modello proposto, gli individui appartenenti a questi pri-mi gruppi originari sono andati poi incontro ad un progressivo
pro-1.3 le popolazioni antiche italiane 11
cesso di neolitizzazione, cambiando il loro modo di sostentarsi, il loro rapporto con l’ambiente e anche i rapporti con gli altri gruppi.
È possibile che alcune di queste comunità con il tempo siano anda-te incontro a progressivi fenomeni di isolamento dovuti a particolari localizzazioni geografiche e successivamente allo sviluppo di espres-sioni culturali molto peculiari. Due esempi possono essere il fenomeno sviluppatosi nell’Italia centrale tirrenica, una zona circondata da monta-gne, e della Sardegna, un’isola, che hanno dato origine rispettivamente alla popolazione etrusca e a quella nuragica. Il prolungato isolamento di questi gruppi umani potrebbe aver determinato, attraverso fenomeni come la deriva genetica, la loro peculiare struttura popolazionistica.
Nelle altre regioni della penisola si ipotizza sia intervenuto un di-verso processo evolutivo. Le varie comunità del Neolitico si sono man-tenute probabilmente più in contatto le une con le altre a causa delle minori barriere geografiche, ricevendo probabilmente l’apporto di una seconda componente genetica dall’esterno; un esempio potrebbe esse-re l’arrivo di individui dal Vicino Oriente come pesse-revisto dall’ipotesi di Ammerman e Cavalli Sforza (Ammerman e Cavalli Sforza, 1984). Dalle analisi di genetica delle popolazioni infatti, sia il gruppo dei re-perti paleolitici che quello dei neolitici, mostrano una certa relazione con le popolazioni dell’Armenia e dell’Azerbajan. Queste regioni si trovano esattamente sulla rotta che si ipotizza fosse utilizzata dai va-ri gruppi per entrare in Europa, sia durante il peva-riodo Paleolitico che successivamente nel neolitico.
Il flusso genico determinato nel tempo da questi interscambi avrebbe reso più omogenee le popolazioni italiche originatesi da queste comu-nità meno isolate. La variabilità genetica osservata nella popolazione picena, che risulta essere molto vicina a quella delle popolazioni attua-li, potrebbe essere una conseguenza di questi meccanismi. Se questa ipotesi venisse accertata, allora potremmo pensare di poter applicare lo stesso modello alle altre popolazioni italiche. Seguendo sempre questa linea si deduce che dalla riespansione di queste popolazioni, dopo me-scolamenti, si sarebbe originato il pool genico delle popolazioni italiche attuali (Lari, 2004).
12 i n t r o d u z i o n e
Figura 6: I popoli presenti nella penisola italica, secondo le ipotesi storiche, nel periodo intorno al V sec. a.C..
1.4 il caso delle popolazioni lucchesi 13
1.4 i l c a s o d e l l e p o p o l a z i o n i l u c c h e s i
Il paleoambiente che caratterizzava la piana di Lucca nei tempi antichi era molto diverso rispetto alla moderna situazione fortemente antro-pizzata. Le scoperte relative ad insediamenti del Paleolitico, del Me-solitico, del Neolitico e di numerose grotticelle sepolcrali eneolitiche, testimoniano l’antica frequentazione umana dell’area lucchese.
I più antichi manufatti litici risalgono al paleolitico inferiore (35.000 anni fa). Nella parte sud-orientale della pianura lucchese sono state ritrovate tracce di Homo neanderthalensis, prima, e di Homo sapiens, dopo, risalenti alla Glaciazione del Würm (18.000-10.000 anni fa). In questo periodo la conca lucchese compresa tra gli alvei dei fiumi Serchio e Arno, si presentava ricca di dossi alluvionali che negli ultimi millenni facilitarono lo sviluppo di insediamenti umani (Mencacci e Zecchini,
1982).
La loro presenza nella piana non è costante nel tempo. I manufat-ti limanufat-tici ritrovamanufat-ti tesmanufat-timoniano l’esistenza di culture preistoriche sino al Bronzo medio, dopo di che scompaiono le tracce di frequentazione che si ripresentano nel VIII secolo a.C. (Villanoviano) per poi proseguire ininterrottamente con gli Etruschi e i Romani.
I secoli non rappresentati da testimonianze archeologiche furono pro-babilmente caratterizzati da un peggioramento delle condizioni clima-tiche, intensificazione delle piogge, che determinarono l’allagamento della piana spingendo i gruppi umani verso le alture.
Nuove attestazione umane riapparvero probabilmente a partire dal XIV-XII secolo a.C., quando avvenne una deviazione fluviale del Paleo-serchio, che si aprì una seconda via attraverso le strette di Ripafratta permettendo il deflusso delle acque dalla piana. La nuova rotta del fiu-me verso sud-ovest determinò radicali trasformazioni ambientali che influenzarono l’abitabilità della zona (Mencacci e Zecchini, 1982).
Purtroppo tracciare un quadro chiaro delle vicende del popolamen-to dell’area è veramente difficile vispopolamen-to la quasi popolamen-totale mancanza sia di reperti biologici che culturali.
Le poche informazioni suggeriscono che intorno all’anno 1000 a.C. tribù germaniche provenienti dal nord Europa presero stabile
dimo-14 i n t r o d u z i o n e
ra nella pianura che si estendeva ad ovest del lago di Bietina, percor-sa dai corsi d’acqua Serchio e Ozzeri (Giovannini, 2003). L’etimologia del toponimo Lucca, non avrebbe la radice etrusca, ligure o celtoligure, ‘Luck’, luogo paludigno, come si pensava in un primo momento ( Man-cini,1975), ma sarebbe di origine indoeuropea, ‘Lawk’ successivamente latinizzato in ‘Luca’ da lucus: luogo ombroso e cupo come il sottobosco.
A partire dal IV secolo a.C incominciò la romanizzazione dell’area Toscana. Questo evento portò a uno spopolamento degli insediamenti estruschi collocati a sud della regione a favore di nuovi insediamenti nel nord con la nascita di nuovi centri (Pisa, Firenze, Fiesole, Volterra, Lucca ecc.) (Francovich, 1999).
Dopo il 180 a.C. Lucca divenne ufficialmente colonia latina. La po-polazione Etrusca e Ligure ancora presente sul territorio della piana, venne assorbita dalla nuova presenza romana che insieme alle opere di bonifica iniziò la costruzione di una grande rete di strade consolari che collegavano la regione a Roma e all’Italia settentrionale.
In questo modo, grazie alla posizione strategica di nodo viario, Lucca assunse un ruolo di primaria importanza nei rapporti militari, ammini-strativi e commerciali.
Una strada consolare, probabilmente esistente già nel II-I sec. a.C., detta Cassia o Clodia, collegava la regione da un lato ad Arezzo e di qui a Roma e dall’altro alla viabilità costiera. Verso nord, attraverso la valle del Serchio, si snodava la via Lucca - Parma. Altre strade permet-tevano ancora di raggiungere la costa: verso Pisa, aggirando i Monti Pisani, e verso Luni, attraverso la Val Freddana, la via Faetina metteva in comunicazioni l’Etruria con l’Emilia (Riparbelli, 1982).
L’avvicendarsi di genti diverse e il ruolo estremamente strategico fan-no di Lucca, anche durante i secoli successivi un luogo di stallo per le genti provenienti da Nord. Durante le invasioni barbariche protrattesi durante tutto il V e il VI secolo d.C. Lucca diventa la sede del potere della Tuscia longobarda.
Per registrare una stasi dei grandi flussi migratori in Toscana si dovrà aspettare l’XI secolo; infatti, è proprio dopo l’anno Mille che gli studiosi individuano gli elementi di continuità ancora presenti nella struttura della popolazione attuale (Francovich, 1999).
1.5 studi genetici sulla popolazione etrusca 15
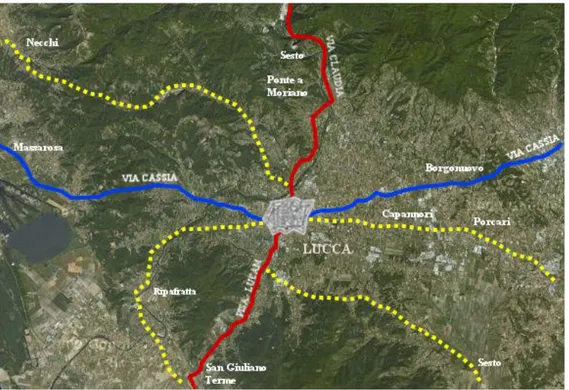
Figura 7: Lucca romana: rete viaria primaria e secondaria. La linea blu indica la Via Cassia, la rossa Via Claudia. Modificata da (Riparbelli,1982).
Appare evidente che le lontane origini delle genti che hanno popolato la piana lucchese sono miste e profondamente segnate dalle diverse dominazioni.
1.5 s t u d i g e n e t i c i s u l l a p o p o l a z i o n e e t r u s c a
Fra le popolazioni che si sono avvicendate nella piana di Lucca un chia-ro rilievo storico l’ha avuto la popolazione Etrusca. Gli scavi condotti tra il 1994 e il 1996 nel Comune di Porcari, lungo il corso dell’antico Auser (ad oggi il fiume Serchio), tra il Palazzaccio e Via Santa Lucia, avevano portato alla luce resti di una frequentazione etrusca già riconducibile al VI sec. a.C..
16 i n t r o d u z i o n e
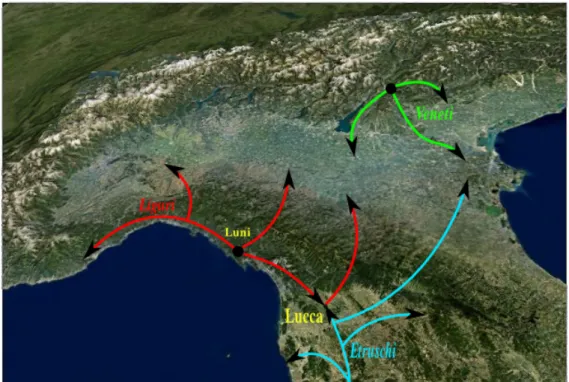
Figura 8: Situazione Pre-Romana del Centro-Nord Italia (circa V sec. a.C.). Con le freccie sono segnate le popolazioni che sono venute a contatto con la popolazione Etrusca.
La cultura etrusca si è sviluppata in centro Italia dall’VIII sec. a.C.. Poco si sa della sua origine e i documenti che ci sono pervenuti di epo-ca romana non fanno chiarezza su questo enigma. Le analisi e gli studi effettuati sulla lingua e la cultura etrusca fanno pensare a una popo-lazione non-Indoeuropea stanziatasi nella penisola prima della nascita della cultura Romana. Gli antichi storici parlano di una migrazione in massa di una popolazione dall’Anatolia o dalle Alpi, ma gli archeologi attuali escludono a priori questa possibilità.
Dai primi studi paleogenetici era emerso che solo due aplotipi, tra i più comuni in Europa, erano condivisi tra la popolazione moderna e quella antica, ciò ci induce a ipotizzare una scomparsa delle sequenze etrusche nel corso del tempo. Quest’ultimo risultato può essere anche dovuto alla particolare struttura sociale di queste popolazioni: infatti
1.6 gli scavi del frizzone 17
tutti gli scheletri che sono stati analizzati provengono da tombe molto ricche di corredi funebri. Tali strutture sono notoriamente appannaggio di una elite, quindi questi studi possono considerarsi limitati a quella parte della popolazione che costituiva le classi sociali più alte. Inoltre si era riscontrata una certa similarità tra la popolazione Turca moderna e gli Etruschi, questo probabilmente a causa degli scambi commerciali intrapresi con alcune popolazioni dell’Asia Minore (Caramelli, 2009;
Vernesi, C. et al.,2004).
Dalle analisi dei dati basate sulla teoria della coalescenza (
Guima-raes, S. et al., 2009) emerge che è esplicitamente dimostrabile una
di-scontinuità genetica tra gli Etruschi e i Toscani moderni. Questa potreb-be dipendere da vari fenomeni: un estinzione degli aplogruppi carat-terizzanti la popolazione, dovuta ad una serie di forti flussi migratori o ad una migrazione di massa in questo caso causata dall’invasione Romana.
Le conclusioni possono essere quindi due. La prima è che gli antena-ti dei Toscani moderni non siano gli Etruschi e la seconda è che non ci siano prove di una relazione a causa del fatto che non si siano trovate sequenze moderne rappresentanti quelle antiche. Le domande sull’ori-gine e le relazioni filogenetiche del popolo etrusco rimangono ancora aperte e ci si chiede che cosa sia accaduto al popolo etrusco dopo il I secolo a.C..
1.6 g l i s c a v i d e l f r i z z o n e
Durante le fasi di analisi di scavo, nell’area compresa tra il Comune di Capannori e il Comune di Porcari, già fonte, fin dai primi anni ’90, di importanti testimonianze di frequentazione etrusca e romana, emersero tra il 1997 e il 2002, in specifico nell’area denominata localmente “a Casa del Lupo”, al catasto “il Frizzone”, un palinsesto di opere agrarie dall’età romana a quella medievale.
La continua esplorazione dell’area, interessata dalla nuova costruzio-ne del casello autostradale, ha permesso di far affiorare la prima costruzio- necro-poli d’età romana individuata nella fascia orientale della piana centu-riata di Lucca. Questa scoperta potrebbe far luce e fornire dati utili sui
18 i n t r o d u z i o n e
costumi e l’organizzazione degli spazi sepolcrali, per lo più ignoti, del-l’insediamento nella piana lucchese dalla tarda età repubblicana fino ai primi del II sec. d.C.. Inoltre potrebbe fornire importanti informazioni sull’organizzazione del territorio.
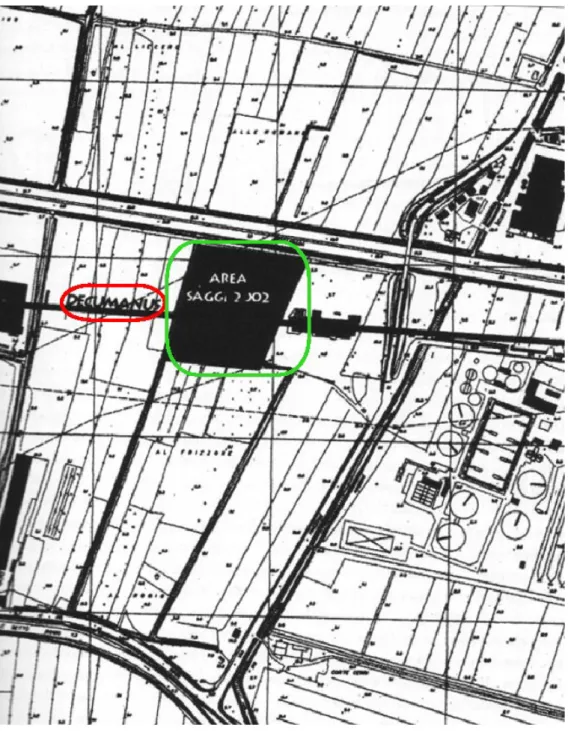
Analizzando l’area in cui si trova la necropoli e la centuriazione luc-chese, si può affermare che un decumanus attraversava l’area dei saggi e che l’area sepolcrale si trova esattamente a ridosso di quest’ultimo. Questo dato conferma la presenza di antichi tracciati agricoli poi sosti-tuiti da spazi silvo-palustri in epoca tardo antica e altomedievale, anche a causa delle continue esondazioni fluviali.
Secondo l’ipotesi per cui le vie che escono dalle porte cittadine sono anche a Lucca, l’asse di distribuzione delle necropoli urbane, allora nel-la piana i limites centuriali svolgono molto probabilmente lo stesso ruo-lo, catalizzando nel tempo le sepolture delle comunità che vivono nelle fattorie distribuite nel territorio. Secondo l’analisi stratigrafica l’area se-polcrale, nella prima metà del II sec. d.C., esaurisce la sua funzione cambiando, dopo qualche decennio, destinazione d’uso con una ripre-sa di utilizzo agricolo da parte di nuovi coloni completamente ignari dell’antico ruolo funerario.
1.6 gli scavi del frizzone 19
Figura 9: L’area del Frizzone a Capannori. Il cerchio rosso evidenzia l’asse del decumanus, in verde l’area dei saggi di scavo che vanno dal 1997 al 2002.
2
M A T E R I A L I
2.1 p r o v e n i e n z a d e i c a m p i o n i
Il materiale a disposizione per questo studio, costituito principalmente da frammenti ossei e dentari, è rappresentato da un gruppo di 13 in-dividui appartenuti a genti probabilmente vissute nella piana lucchese tra il II sec. a.C. e il I sec. d.C.. Durante le fasi di scavo, i reperti ossei non hanno subito nessun tipo di trattamento di pulizia o di restauro e il materiale destinato alle analisi in laboratorio è stato maneggiato da un’unica persona che ha provveduta alla sua catalogazzione e messa in disparte.
L’area interessata dalle operazioni di scavo, copre una superficie di circa 2 ettari ed è stata indagata con un reticolato diagnostico formato da trincee disposte su un quadrettato con lato di 25 metri. Le trincee erano larghe 2 m e profonde 1,5 rispetto al piano della campagna attuale e numerate progressivamente da est a ovest (trincea A, trincea B, etc.) e da nord a sud (trincea I, trincea II, etc.).
Lo scavo ha permesso di identificare una successione stratigrafica composta dai seguenti livelli.
• Un livello argilloso plastico (imbibito d’acqua). Comprendente il suolo agricolo, archeologicamente sterile, e presumibilmente ri-conducibile a periodi medievali.
• Un livello costituito da argilla limosa. Segnalante un evento al-luvionale inquadrabile all’interno di un arco cronologico ampio, compreso, all’incirca tra il III sec. a.C. e l’età medievale. I manu-fatti ritrovati sono di età romana. Le sepolture di interesse per questo studio sono attribuite a questo livello. In particolare sono stati ritrovati deposizioni ad incenerazioni in corrispondenza del-la trincea II, mentre in una più vasta area sepolcrale si sono potute
22 m a t e r i a l i
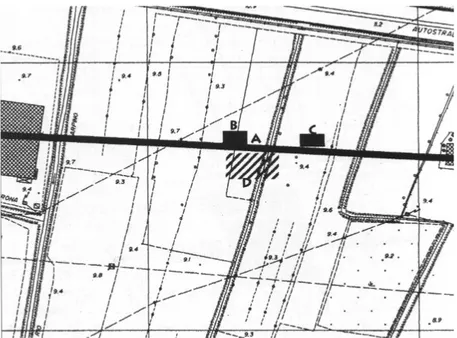
Figura 10: L’area di scavo. Le lettere indicano rispettivamente: D l’area agricola inte-ressata da un reticolato di fossati, A l’asse del decumanus, B l’area delle sepolture, C l’area di deposizioni ad incenerazioni.
individuare due fasi di tumulazioni: una più recente, comprenden-te le deposizioni di resti incinerati, ed una più antica, consiscomprenden-tencomprenden-te in inumazioni in fosse terragne. In base ai pochi reperti di corre-do ritrovati, gli inumati presenti nella fossa terragna dello scavo si possono attribuire alla fine del II sec. a.C., mentre le deposizioni ad incenerazioni sono attribuibili ad un periodo compreso tra la fine del I sec. a.C. ed il II sec. d.C..
• Un livello limoso con componente sabbiosa. In esso sono presenti i resti di due tumulazioni, tipologicamente e cronologicamente at-tribuibili alla fase delle deposizioni per inumazione documentate nello strato precedente. Costituisce un evento alluvionale che si può considerare concluso nel corso del III sec. a.C..
La stratigrafia ottenuta documenta, sommariamente, le dinamiche evolutive dell’ambiente e delle interazioni tra questo e le attività degli
2.1 provenienza dei campioni 23
Figura 11: Schematizzazione della stratigrafia rilevata durante lo scavo delle trincee II e IV.
antichi abitanti della zona. Le caratteristiche chimico-fisiche dei livelli documentati lasciano intuire un’alternarsi tra la successione di alcuni eventi alluvionali, attribuibili a ripetute esondazioni fluviali di un brac-cio del fiume Auser che hanno interessato la zona e lo sfruttamento dell’area per fini antropici quali attività agricole e sepolcrali.
Il primo e più antico nucleo di deposizioni è stato individuato in trincea IV, dove sono stati recuperati i resti di 10 inumati caoticamente deposti in un area ristretta. Ad oggi non sono ancora state effettuate le analisi antropometriche e la determinazione del sesso. Dai pochi dati archeologici emersi si è potuto dedurre che l’area sepolcrale è stata
24 m a t e r i a l i
Figura 12: (a) Sepoltura inumato 1-2. (b) Sepoltura inumato 37. (c) Area di scavo vista dall’alto.
utilizzata in momenti diversi, riconducibili nello specifico a due fasi. La prima più antica riferibile alla fine del II sec. a.C., ed una più recente attribuibile alla metà del II sec. d.C..
La prima fase è data da quattro inumati (inumati 1-4) individuati nel-la porzione più occidentale delnel-la necropoli, mentre nel-la fase attibuita alnel-la metà del I sec. d.C., rappresentata da cinque inumazioni (inumati 29, 33-35, 37-38), è localizzata a ridosso dei limiti più occidentali dell’area delle incenerazioni, dove si è rilevata una parziale sovrapposizione tra le fasi di utilizzo della necropoli ad incenerazione e ad inumazione.
Lo spazio destinato alle inumazioni è ristretto a poche decine di metri quadrati, concentrato lungo il limite orientale della superficie di scavo. Un tratto caratterizzante di questa modalità di sepoltura sembra essere
2.2 il dna mitocondriale 25
la scarsa cura impiegata, gli inumati infatti, non sono isorientati e si pre-sentano con una disposizione a tratti caotica. La quasi totale mancanza di corredo funerario, come già accenato in precedenza, potrebbe essere associata a un basso status sociale dei defunti, con eccezione forse della coppia di inumati 1 e 2, entrambi in possesso di un anello con castone lavorato.
Un’altra ipotesi, e forse la più plausibile, suggerisce l’esigenza di ese-guire tumulazioni in tempi celeri, per ragioni non rilevabili, ma presu-mibilmente attribuibili più al contesto politico che attraversa la penisola in quel momento, sconvolta da guerre civili, che non a quello sociale degli inumati (Ciampoltrini, G. et al., 2005).
I restanti tre campioni (Trincea II-US 34, Trincea IV-US 66, Trincea IV-US 72) sono emersi durante gli scavi nell’area di deposione ad ince-nerazione. Il rituale di seppellimento era già stato riscontrato in altre aree di scavo, come quella di San Ponziano (Ciampoltrini,1998), e do-veva consistere in una cerimonia molto lunga e articolata. Dal tipo di corredo e dall’ordine con cui sono state effettuate le sepolture emerge chiaramente che si è difronte a un altro periodo storico rispetto a quello delle depozioni ad inumazione.
In Tabella1 è riportato l’elenco dei campioni, la datazione presunta, il sito di provenienza e il distretti ossei recuperati per le analisi.
2.2 i l d n a m i t o c o n d r i a l e
Come già accennato nel capitolo precedente oggi risulta possibile, at-traverso l’analisi di vari marcatori, effettuare studi molecolari per com-prendere la storia e le dinamiche che hanno interessato la nostra spe-cie durante tutto il suo processo evolutivo e i rapporti tra le varie popolazioni che da essa si sono originate.
Tra questi il più utilizzato quando si parla di DNA antico, è il DNA contenuto nei mitocondri (mtDNA). Il DNA mitocondriale presenta in-fatti alcune peculiarità che lo rendono adatto allo studio di campioni antichi.
Per comprendere meglio i motivi che hanno reso il DNA mitocon-driale lo strumento più utilizzato nel settore della micro-evoluzione
26 m a t e r i a l i
CAMPIONE DATAZIONE SITO PROVENIENZA FRAMMENTO OSSEO
Inumato 1 II a.C. Necropoli Frizzone tibia, femore, calotta, dente Inumato 2 II a.C. Necropoli Frizzone calotta, femore
Inumato 3 II a.C. Necropoli Frizzone calotta, denti, coste ,femore Inumato 4 II a.C. Necropoli Frizzone calotta, femore
Inumato 29 I d.C. Necropoli Frizzone calotta, fibula, tibia
Inumato 33 I d.C. Necropoli Frizzone radio, ulna, femore, calotta Inumato 34 I d.C. Necropoli Frizzone calotta, denti, femore, tibia Inumato 35 I d.C. Necropoli Frizzone coste, omero
Inumato 37 I d.C. Necropoli Frizzone calotta, denti, costa, femore Inumato 38 I d.C. Necropoli Frizzone calotta, denti, femore, perone Trincea
II-US 34
II d.C. Necropoli Frizzone fram. ossei
Trincea IV-US 66
II d.C. Necropoli Frizzone fram. ossei
Trincea IV-US 72
II d.C. Necropoli Frizzone fram. ossei
Tabella 1: Inumati presi in esame durante lo studio, la datazione presunta, il sito di provenienza e i frammenti ossei utilizzati per le analisi.
molecolare e in particolare negli studi sul DNA antico se ne illustrano brevemente le caratteristiche principali.
Il genoma mitocondriale umano è contenuto nei mitocondri, organelli citoplasmatici di origine endosimbiontica la cui funzione primaria è di fornire energia alla cellula attraverso la fosforilazione ossidativa.
Come nella maggior parte degli animali multicellulari, esso è piccolo, con un’organizzazione genica compatta e non presenta elementi come introni e sequenze ripetute presenti nel DNA nucleare, pertanto risulta facilmente caratterizzarbile.
E’ formato da molecole circolari chiuse di DNA a doppia elica lunghe 16.569 bp (basis pair= paia basi), che corrispondono allo 0,25% del geno-ma totale di un individuo. I due filamenti differiscono per la composi-zione in basi. Il filamento pesante H (heavy=pesante) è ricco in residui
2.2 il dna mitocondriale 27
Figura 13: Struttura schematizzata del DNA mitocondriale.
guaninici, mentre quello leggero L (light=leggero) in residui citosinici. Si è constatato che è in grado di replicarsi autonomamente e di codi-ficare per 37 geni, un numero ridotto rispetto ai circa 20.000 codificati dal DNA nucleare. L’ordine sequenziale è identico nell’uomo come nel topo. E’ composto da 2 geni per l’RNA ribosomiale (12S 16S), 22 geni per gli RNA transfer necessari per la sintesi proteica mitocondriale e 13 geni che codificano per polipeptidi di quattro subunità enzimatiche e della fosforilazione ossidativa: le subunità I, II, III della citocromo ossidasi C, e le subunità 6 e 8 dell’adenosintrifosfatasi (ATPasi), il cito-cromo b e le 7 subunità dell’NADH deidrogenasi della catena respira-toria (Wallace et al., 1987). Per questo motivo una mutazione avversa in uno qualsiasi di questi geni causa gravissime infermità sopratutto di carattere nervoso.
Come si è riscontrato per il genoma nucleare, le varie regioni che compongono l’mtDNA (codificanti e non) presentano tassi di evoluzio-ne differenti (Cann et al., 1984). I tratti che possiedono il tasso di so-stituzione più basso sono i geni per i tRNA e gli rRNA, mentre i tratti
28 m a t e r i a l i
più variabili sono le regioni non codificanti (es. D-loop), i cui tassi di divergenza sono da 1 a 4-5 volte maggiori rispetto a quelli dei geni che codificano per le proteine.
La regione D-loop consiste in circa 1122 bp e di essa fanno parte le regioni ipervariabili I e II (HVRI e HVRII), il segmento I si colloca tra le posizioni nucleotidiche 16.024 e 16.365 ed il segmento II tra le posizioni 73e 340. In totale queste regioni si estendono per circa 900 bp localizzati vicino all’origine del genoma mitocondriale.
Nel DNA mitocondriale inoltre esistono meno meccanismi di ripa-razione rispetto a quelli presenti nel DNA nucleare, questo compor-ta quindi un compor-tasso di mucompor-tazione più elevato dovuto alla perdicompor-ta del-la capacità di proof-reading deldel-la polimerasi mitocondriale durante del-la replicazione.
La regione I risulta essere il segmento più informativo per confronti tra popolazioni che si siano separate in tempi evolutivi recenti, con-tiene infatti una regione lunga 110 bp chiamata “intervening sequence” che sembra variare per lunghezza e composizione in basi in maniera specie-specifica. Le HVRs rivelano il 3% della variabilità fra gli indivi-dui, e presentano un gran numero di siti polimorfici distribuiti in modo disomogeneo secondo cluster chiamati “punti caldi” (hot spot).
La sequenza del mtDNA è nota da oltre 20 anni (Anderson et al., 1981) e fu ottenuta a partire da un soggetto inglese. Oggi comune-mente viene indicata come “sequenza di riferimento di Cambridge” (CRS=Cambridge Reference Sequence) e viene utilizzata come riferimen-to per determinare le varianti polimorfiche del mtDNA, classificate in aplotipi (combinazione di alleli a loci concatenati) e a loro volta rag-gruppati in aplogruppi (gruppo di aplotipi di cui si ipotizza un’origine comune, grazie alla condivisione di mutazioni caratteristiche general-mente ad evoluzione lenta) che tendono a essere circoscritti a differenti aree geografiche e a differenti popolazioni umane, la cui analisi permet-te di ricostruire gli spostamenti antichi dell’uomo attraverso i continenti e le varie regioni del mondo.
3
M E T O D I
3.1 i l p r o b l e m a d e l l e c o n t a m i n a z i o n i
3.1.1 La degradazione dell’ aDNA
Il recupero del DNA fossile pone numerosi problemi a causa dell’insta-bilità chimica degli acidi nucleici. Dopo la morte di un organismo, il DNA contenuto nelle sue cellule va incontro ad una serie di processi degradativi, causati dalle rotture che interessano i legami nucleotidici e che comportano frammentazioni, alterazioni e delezioni irreparabili
(Lindahl,193). Gli acidi nucleici si decompongono spontaneamente in
soluzione, anche in condizioni fisiologiche, per effetto dell’idrolisi. Tut-tavia, mentre la cellula viva, avendo elaborato processi di riparazione del DNA (grazie all’intervento di enzimi quali la glicosidasi, la polime-rasi e la ligasi) può rimediare a questi danni, una cellula morta non è più in grado di far fronte ai processi di degradazione. Già dalla morte di un organismo, si attivano molti fenomeni che danneggiano, frammenta-no ed alteraframmenta-no le molecole di DNA. Infatti, in un mezzo acquoso, quali possono esse i liquidi di decomposizione, il DNA subisce soprattutto due tipi di attacco: un deterioramento chimico (idrolisi ed ossidazione) al quale si aggiunge un deterioramento di tipo enzimatico (autolisi e decomposizione batterica).
I deterioramenti chimici altereranno la struttura del filamento di DNA e l’idrolisi ne determina una rottura.
Il legame carbonio-azoto (C-N) tra le basi e gli zuccheri è particolar-mente fragile e la sua rottura determina la perdita progressiva delle basi azotate. Generalmente le purine (la guanina in particolare) si liberano rispetto alle pirimidine con una velocità di depurinazione di 20 volte inferiore le une rispetto alle altre.
30 m e t o d i
Figura 14: Sotto sezione trasversale di ossa archeologiche non trattate. Visione al mi-croscopio a trasmissione. (a) Tessuto osseo in buono stato di preservazio-ne, la freccia indica una microfrattura. (b) Tessuto osseo in cattivo stato di preservazione (Guarino et al.,2006).
In presenza di un sito abasico, in cui cioè una base si è staccata dal deossiribosio, il filamento è indebolito e va rapidamente incontro a rot-tura, poiché i residui di deossiribosio privi delle basi sono in equilibrio tra la loro originaria forma ciclica e stabile, e una forma lineare aldei-dica e reattiva. La reazione d’idrolisi è catalizzata dagli acidi, motivo per cui in condizioni di pH debole avviene un deterioramento più rapi-do del DNA, inoltre, è influenzata dalla temperatura che, se molto alta, può favorire una riduzione da 5 a 10 volte della velocità del processo. Quando un osso viene seppellito in un terreno con pH neutro o legger-mente basico la struttura piatta dell’idrossiapatite cambia e si avvicina a quella dell’idrossiapatite pura, cioè a forma di ago. Questo cambia-mento diminuisce la solubilità dell’osso perchè diminusce il rapporto superficie/volume (Berna, F. et al., 2004). L’adsorbimento del DNA su idrossiapatite dimezza la velocità di depurinazione contribuendo a ren-dere più probabile il recupero di materiale genetico a partire dalle ossa fossili.
Anche i gruppi amminici delle basi sono suscettibili di idrolisi, che può provocare deaminazione. Nel DNA a singolo filamento la velocità di deaminazione è simile per pirimidine e purine; la conformazione a doppia elica invece offre maggiore stabilità per le pirimidine (l’emivita
3.1 il problema delle contaminazioni 31
di un residuo citosinico in una molecola a doppio filamento a 37° e pH 7.4 è mediamente 30.000 anni). Per questi motivi la depurinazione può essere considerata come il principale meccanismo di degradazione del DNA in soluzione (Lindahl e Nyberg, 1972). D’altronde disidratare il DNA per impedire l’idrolisi sarebbe impossibile poichè le molecole d’acqua sono indispensabili per mantenere la struttura stessa del DNA, che una volta disidratato sarebbe ancora più vulnerabile.
L’ossidazione costituisce la forma principale di modificazione post mortem del DNA: nella cellula il nucleo è povero di ossigeno mentre do-po la morte, quando la membrana nucleare si rompe, ne contiene molto di più. L’ossigeno, attraverso la produzione di radicali liberi idrossi-lici, converte la guanina in 8-idrossi-guanina; così gli attacchi ossida-tivi danneggiano contemporaneamente sia le basi (in particola modo Timina e Citosina) che perdono un doppio legame e la loro originaria struttura ad anello, andando così incontro a frammentazione (Lindahl
e Andersson, 1972), sia gli zuccheri impedendo l’accoppiamento dei
nucleotidi danneggiati con quelli del filamento opposto.
Come già accennato prima sia variazioni di pH, che temperature ele-vate costituiscono un ostacolo alla conservazione del DNA: in partico-lar modo queste ultime, oltre a mettere alla prova i legami a idrogeno e le forze di Van der Waals, favoriscono il proliferare di batteri, muf-fe e altri organismi che contaminerebbero in modo molto consistente il DNA antico, degradandolo e rendendone particolarmente problematica l’analisi.
È comune opinione degli studiosi che la decomposizione cominci al momento della morte, immediatamente dopo di essa, anche se la visi-bilità esterna dei suoi effetti sia relativamente successiva. In questa fase la decomposizione è causata principalmente da due fattori: autolisi (la suddivisione dei tessuti dai propri prodotti chimici interni del corpo ed enzimi) e putrefazione propriamente detta (scissione degli elementi co-stitutivi dei tessuti operata dai batteri). L’ autolisi comincia subito dopo la morte di un organismo: la membrana nucleare si rompe ed i compo-nenti del citoplasma e del nucleo si mescolano. I vari enzimi, le nucleasi, normalmente isolate dal nucleo possono allora agire e “digeriranno” il DNA, fenomeno favorito ad una temperatura di 37 C°. Il
funzionamen-32 m e t o d i
Figura 15: Danni riportabili dal DNA antico. Con le frecce sono indicati i siti in cui le molecole di DNA subiscono modificazioni chimiche che ne determinano la degradazione. Le principali sono la depurinazione, la deaminazione ed i danni ossidativi (modificato da (Hofreiter, M. et al.,2001a).
to post mortem delle nucleasi cessa quando terminano all’interno della cellula le scorte di ossigeno. I micro organismi velocemente iniziano a fare il resto. Il processo è identico a quello autolitico con la liberazione di esonucleasi ed endonucleasi che deteriorano le molecole restanti.
Come si è visto, la maggior parte dei danni post mortem avvengono come rotture della doppia elica e modificazione ossidativa del nucleoti-de, impedendo, in entrambi i casi, la successiva replicazione enzimati-ca. Tuttavia, modificazioni minori della sequenza, come nel caso della deamminazione idrolitica e della depurinazione, permettono il corretto funzionamento della polimerasi e si manifestano con un numero limi-tato di basi variate all’interno dei cloni sequenziati (Krings, M. et al.,
1997).
I prodotti di amplificazione del aDNA sono caratterizzati comune-mente da errori, con una prevalenza generale verso le transizioni di
3.1 il problema delle contaminazioni 33
C-G in T-A ed una inferiore di A-T in G-C (Hansen, A. et al., 2001;
Hofreiter, M. et al., 2001b; Gilbert, M.T.P. et al., 2003). Il numero alto
di transizioni C-G in T-A sembra essere dovuto all’alto tasso di deam-minazione idrolitica della citosina ad uracile ed a timina. Nei primi anni del secondo millennio si è così provveduto a creare dei modelli di distribuzione dei danni post mortem a livello del DNA mitocondriale umano.
In uno studio condotto da Gilbert nel 2003 è stata analizzata la di-stribuzione del danno post mortem nel DNA mitocondriale recuperato, amplificato e clonato a partire da 37 campioni umani antichi; le sequen-ze dei cloni ottenuti sono stati paragonati ad una selezione di dati già pubblicati. Dopo aver calcolato il tasso relativo di danno per le posizio-ni del nucleotide all’interno della prima regione ipervariabile umana (HVRI) e dei geni dell’unità secondaria della Citocromo Ossidasi III, at-traverso il confronto dei loci danneggiati intra e tra le regioni, è stato rilevato che esistono degli hot spots di danno che, nella HVRI, sono cor-relati con siti noti per avere in vivo alti tassi di mutazione. Per contro, la sottoregione HVRI, con funzione strutturale conosciuta, come MT5, ha in vivo tassi di mutazione più bassi diminuendo così i tassi di danno post mortem. I dati post mortem, inoltre, hanno identificato una possibile sottoregione funzionale della HVRI, chiamata a “bassa-diversità 1“,con la mancanza di danno della sequenza. La quantità di danni post mor-tem osservata nelle regioni mitocondriali di codificazione risultava così significativamente più bassa che nell’HVRI.
La distribuzione di questi siti, regolarmente danneggiati negli esse-ri umani, è molto simile a quella osservata nelle normali sostituzioni evolutive, stando a significare che il danno dell’aDNA può generare sequenze artefatte che imitano i cambiamenti evolutivi previsti. Sulla base dello spettro dei danni post mortem osservati, è stato quindi intro-dotto un semplice metodo per l’identificazione di eventuali errori per l’attribuzione dei corretti aplogruppi (Gilbert, M.T.P. et al.,2003)
Nonostante questi accorgimenti, il tempo ed il modo in cui avven-gono i danni post mortem rimanavven-gono non del tutto compresi per poter progettare efficaci trattamenti enzimatici di riparazione in vitro per lo studio del DNA antico. Il continuo incremento di dati disponibili, e
34 m e t o d i
recuperati da una vasta gamma di substrati, potrà permettere di verifi-care le varie ipotesi al fine di determinare esattamente il filo specifico dell’origine degli eventi di danno osservati (Gilbert, M.T.P. et al., 2007).
Di recente è stato anche presentato un modello (Ho, S.Y. et al., 2007) che, usando uno studio di simulazione, valuta la quantità dei danni nei dati del DNA e rappresenta i relativi effetti in una struttura filogenetica bayesana. I risultati indicano che gli alti tassi di mutazione, precedente-mente valutati dai set di dati del aDNA, non sono completaprecedente-mente rela-tivi ai siti di danno della sequenza, ma sono probabilmente dovuti ad altri fattori quale, per esempio, la persistenza dei polimorfismi transito-ri. Tale modello sembra essere particolarmente utile per la collocazione dei limiti superiori di credibilità sulla quantità di danno di sequenza in fase di allineamento, motivo per cui questo modello potrebbe essere utilizzato sia per i futuri studi sul DNA antico, ma anche per la valu-tazione degli errori di ordinamento nel DNA moderno (Mallegni e
Lippi, 2009).
3.1.2 I Golden Criteria
I problemi legati agli studi sull’aDNA dati sia dalla struttura in sè della molecola, che da problemi legati alla metodologia di indagine, devo-no essere limitati il più possibile. Questo in parte è attuabile grazie a un rigoroso protocollo d’indagine basato principalmente su quelli che vengono definiti i Golden Criteria (Cooper e Poinar, 2000) che sono:
• separazione fisica delle aree di lavoro nelle fasi di pre e post amplificazio-ne;
• inserimento di controlli negativi in fase di estrazione e amplificazione; • valutazione del grado di preservazione biochimico delle molecole antiche; • amplificazione di frammenti corti di DNA;
3.1 il problema delle contaminazioni 35
Figura 16: Situazione ideale di scavo.
3.1.3 Il recupero dei reperti
La collaborazione fin dalle prime fasi di scavo tra antropologo moleco-lare e il resto dello staff è fondamentale. Se si decide di effettuare uno studio molecolare su determinati campioni è utile pianificare l’azione fin dall’inizio, in modo da individuare subito il campione destinato alle analisi.
La metodologia migliore è quella di assicurarsi che appena il reper-to viene riportareper-to in superficie, si manipoli con guanti e mascherina (sarebbe appropriata anche una tuta sterile), si ponga in sacchetti ste-rili, si controlli lo stato di umidità dell’osso e si provveda a un’utile asciugatura schedandolo adeguatamente (è indicato, nel caso il cam-pione provenga da grotte o ghiacciai porlo in un contenitore termico isolato con ghiaccio, non deve infatti risentire di forti cambiamenti di temperatura).
36 m e t o d i
Al fine di far arrivare in laboratorio il maggior numero di informa-zioni bisogna prelevare anche un campione o più (nel caso di evidenti differenze) del terreno di giacitura così da permettere una possibile analisi di organismi presenti nel terreno ma anche una possibile analisi dei processi di diagenesi. Nel caso di utilizzo di consolidanti per l’a-sporto di terreno è bene conoscerne la composizione in modo da poter asportare prima il reperto di interesse.
È da evitare la pulizia con acqua e, anche se sporco di terra, il campio-ne andrebbe consegnato al laboratorio senza essere pulito. Per elimina-re eventuali incrostazioni calcaelimina-ree bisogneelimina-rebbe usaelimina-re metodi meccanici anziché acidi in quanto questi ultimi distruggono il DNA.
Andrebbero privilegiati nella scelta dei campioni distretti scheletrici ricchi in tessuto osseo compatto e poco importanti per lo studio morfo-metrico come coste e falangi. Per le ossa lunghe è possibile ricavare un tassello a forma di cuneo che non pregiudichi le analisi morfometriche, da estrarre con un seghetto monouso o sterilizzato dopo ogni utilizzo. Per i denti è possibile prelevare il campione dalla parte interna usando un microcarotatore, avvantaggiando così l’analisi molecolare, in quanto è la dentina che contiene il DNA meglio conservato, e nel contempo evitando di alterare la morfologia del reperto in modo eccessivo.
Il campione deve pervenire velocemente al laboratorio di analisi con i dati relativi a tutte le persone che l’hanno manipolato (la cosa migliore è che sia una sola e che in ogni caso sia disposta a sottoporsi a tipizzazio-ne). Qualora non sia possibile inviarlo immediatamente al laboratorio di analisi del DNA, il campione andrebbe conservato in freezer a -20°
C (Pruvost, M. et al., 2007).
3.2 a n a l i s i d i l a b o r a t o r i o. metodologie applicate
3.2.1 Il laboratorio
Il lavoro di analisi del DNA antico è stato effettuato preso il labora-torio di Antropologia Molecolare dell’Università di Firenze, mentre la successiva analisi dei dati ottenuti si è svolta presso il Dipartimento di Biologia ed Evoluzione dell’Università di Ferrara.
3.2 analisi di laboratorio. metodologie applicate 37
Figura 17: Planimetria del laboratorio dove si sono svolte le analisi (Laboratorio di Antropologia, Università di Firenze).
Come già affermato precedentemente, per poter effettuare le analisi sul DNA antico e ottenere dati autenticabili è necessaria la divisione delle aree di lavoro. Per questo motivo il laboratorio dedicato a queste analisi deve essere strutturato, e tutte le attività svolte al suo interno ge-stite, nel rispetto dei criteri sopra citati. Il laboratorio di Antropologia Molecolare dell’Università di Firenze è pertanto organizzato secondo lo schema in Figura 17. Nella zona PRE-amplificato vengono svolte tutte le fasi di lavoro precedenti all’amplificazione del DNA: lo stoccaggio, la pulizia e la preparazione del campione, l’estrazione del DNA e la preparazione della miscela di reazione per la PCR. Mentre nella zona POST-amplificato vengono effettuate le analisi elettroforetiche, il clo-naggio e la reazione di sequenza. Questi due settori sono fisicamente separati, gli strumenti e tutti gli oggetti presenti nella zona POST non
38 m e t o d i
devono assolutamente mai essere introdotti nella zona PRE, così come gli operatori, una volta stati nella zona POST non possono più accedere alla zona PRE nell’arco della stessa giornata, per evitare di contaminare l’ambiente ed i campioni.
Ogni superficie di lavoro viene sistematicamnte sterilizzata mediante ipoclorito di sodio o nel caso vengano svolte attività con un alto potere contaminante (pulizia del campione ed estrazione del DNA, prepara-zione della mix per la PCR) vengono irradiate mediante raggi UV a 254 nm che inducono la formazione di dimeri di timina e la conseguente distruzione del DNA potenzialmente contaminante.
3.2.2 La pulizia del campione ed estrazione del DNA
Per poter procedere alla fase di estrazione è necessaria una fase pre-liminare di pulizia del campione. Entrambe le operazioni si svolgono sotto una cappa aspirante previamente sterilizzata dopo ogni utilizzo con raggi UV a 254 nm. Durante tutta la durata dell’esperimento l’o-peratore deve necessariamente indossare una tutta sterile o un camice monouso, una mascherina e un paio di guanti sterili che devo essere cambiati ogni qualvolta si tratti un campione diverso.
Per pulire il campione ed eliminare un possibile strato contenente DNA esogeno, si procede legivando la superficie dell’osso con carta abrasiva o nel caso di sporco di maggiori dimensioni con un bisturi mo-nouso. In seguito ogni superficie del frammento osseo viene irradiata ortogonalmente per 45 minuti con raggi UV a 254 nm.
La base di partenza dell’estrazione sono le polveri ricavate dal cam-pione pulito, per farle viene utilizzato un mortaio di ceramica prece-dentemente pulito, sempre con ipoclorito di sodio, etanolo assoluto e acqua bidistillata sterile.
Per l’estrazione del DNA è sufficiente una quantità di polvere pari a 0.5 o 1 gr. La polvere ricavata in eccesso può essere conservata a -20°C in tubi Falcon sterili per successive estrazioni o analisi.
La fase di estrazione consta di vari passaggi. La prima è una lisi, che si ha aggiungendo sodio-dodecil-fosfato (SDS) che rompe le membrane cellulari e ne rilascia il contenuto. Segue poi l’aggiunta di EDTA
(com-3.2 analisi di laboratorio. metodologie applicate 39
posto chimico: etilediammina tetra-acetato) che chela gli ioni magnesio riducendo ulteriormente l’integrità della parete. Utilizzando il fenolo si eliminano tutti i componenti che non siano il DNA.
La polvere d’osso viene introdotta in una provetta da 15 ml steriliz-zata a raggi γ (Cellbio) e successivamente incubata su un piano rotante per 10 min a T ambiente con 2.2 volumi di SSC 1X (NaCl 0.15 M e ci-trato di sodio 0.015 M, pH 7.2) e 0.5% (v/v) di SDS 10%. Si aggiunge poi un volume di fenolo saturo in Tris-HCl 1 M a pH 8 e la soluzione viene fatta ruotare per 45 minuti al termine dei quali ha inizio la clas-sica estrazione fenolo-cloroformio per eliminare la maggior parte delle molecole organiche (Sambrook e Russell, 2001). Il fenolo denatura le proteine, il cloroformio in seguito rimuove le tracce di fenolo.
Il DNA così estratto viene purificato con Centrex UF-2 Schleicher & Schuell per togliere alcuni degli eventuali inibitori presenti (ad es. acidi umici, tannini e composti di Mallard che inibiscono l’attività della Taq polimerasi) e concentrato utilizzando una matrice di silice (MERmaid® SPINTM Kit, Bio 101 Inc. La Jolla, CA), basata sull’adsorbimento del DNA, ed etanolo, seguendo in entrambi i passaggi le istruzioni delle case produttrici . Al termine dell’operazione si ottengono 50 µl di eluito contenenti il DNA pronto per l’amplificazione. A tutte le fasi è appli-cato anche un controllo negativo, contenente tutti i reagenti ma non la polvere d’osso, che verrà in seguito amplificato per verificare l’assenza di contaminazioni nei reagenti.
3.2.3 La PCR
La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l’acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione (amplificazione) di frammenti di acidi nucleici dei quali si conoscano le sequenze nucleo-tidiche iniziali e terminali. L’amplificazione mediante PCR consente di ottenere in vitro molto rapidamente la quantità di materiale genetico necessaria per le successive applicazioni.
Per il processo di amplificazione è necessaria una soluzione tampo-nata di:
40 m e t o d i
• 33,2 µl di H2O
• 4 µl di MgCl2;
• 5 µl di KCl Tris Buffer 10x;
• 1µl di ciascun primer relativo alla sequenza che si intende ampli-ficare;
• 3,5 µl di miscela di nucleotidi;
• 0,3 µl dell’enzima Taq polimerasi ad alta fedeltà; • 2 µl di DNA da amplificare (DNA stampo).
Il tutto per un volume finale di 50 µl. I primers sono lunghi 20-30 bp e la sequenza è specifica per il frammento del DNA indicato per la rea-zione di ampificarea-zione. Si deduce che la perfetta complementarietà tra questi e il DNA bersaglio, e l’unicità di questo all’interno del genoma, influenzano l’efficienza della reazione di PCR (Brownie, J. et al.,1997).
La regione bersaglio per il nostro studio è l’HVR-I della regione D-Loop del DNA mitocondriale, lunga 360 bp. A causa della degradazio-ne del DNA antico, e quindi della sua presunta frammentarietà, non è consigliato amplificarla interamente in un unico passaggio, per cui l’H-VR I è stata suddivisa in tre tratti over-lappanti (sovrapponibili l’uno con l’altro) identificati dai seguenti primers (Caramelli, D. et al, 2003):
• L15995(5’-CTACAGGTGGTCAAGTATTTATGGT-3’) H16132(5’CCACCATTAGCACCCAAAG-3’) per il primo; • L16107(5’-CGCTATGTATTTCGTACATTACTGC-3’)
H16261(5’-TGGTATCCTAGTGGGTGAGG-3’) per il secondo; • L16247 (5’-GATTTCACGGAGGATGGT-3’)
3.2 analisi di laboratorio. metodologie applicate 41
Figura 18: Preparazione di una Mix di PCR sotto cappa aspirante (Genesphere PBI).
La mix di PCR viene preparata all’interno di una cappa aspirante (Ge-nesphere PBI) che permette, da parte dell’operatore l’inserimento solo delle mani e che viene sterilizzata dopo ogni utilizzo tramite raggi UV a 254nm per evitare contaminazioni. L’operatore deve indossare camice, mascherina, cuffia e guanti sterili per tutta la durata dell’esperimen-to. Al fine di minimizzare il rischio di contaminazione, tutti i reagenti utilizzati sono aliquotati in ambiente sterile, questo per non utilizzare troppe volte i reagenti e in modo che ogni esperimento abbia i propri. La mix viene equamente distribuita per tutti i campioni inserendo inol-tre due controlli, uno negativo con tutti i reagenti senza il DNA per verificare l’assenza di contaminazioni, ed uno positivo con DNA mo-derno, per verificare la riuscita della reazione ed avere un riferimento con cui confrontare gli eventuali risultati ottenuti dai campioni. L’ag-giunta del DNA moderno al controllo positivo viene effettuata in una stanza diversa da quelle della PCR e dell’estrazione.
Tutti i tubini risultanti (uno per ogni campione più i due controlli) vengono inseriti in un termal cycler, la macchina che permette di auto-matizzare la reazione di PCR impostando parametri quali le temperatu-re di denaturazione, appaiamento, estensione e gli intervalli di tempo
42 m e t o d i
necessari per la reazione.
Dopo una prima fase a 94°C per 10 minuti in cui si ha l’attivazio-ne dell’enzima AmpliTaq Gold PE seguono 50 ripetizioni del seguente schema:
• denaturazione eseguita in genere a 94°C per 45 secondi;
• annealing (appaiamento): ad una temperatura di 54,5°C per 60 secondi;
• estensione per 60 secondi a 72°C.
Il cicli si concludono con uno step di estensione finale per 5 minuti a
72°C (Mullis e Faloona, 1987).
3.2.4 Il controllo dei prodotti di PCR
L’elettroforesi è una tecnica analitica e separativa basata sul movimento di particelle elettricamente cariche immerse in un fluido per effetto di un campo elettrico applicato mediante una coppia di elettrodi al fluido stesso. Le particelle si spostano verso il catodo se hanno carica positiva e verso l’anodo se hanno carica negativa. Nel caso specifico del DNA, l’elettroforesi permette di separare frammenti di DNA in base alla loro misura sfruttando la capacità di questa molecola, carica positivamente, di migrare verso il polo negativo.
Durante le analisi per questa tesi, il controllo dei prodotti di PCR, è stato effettuato tramite un gel di agarosio al 2% su lastra di plexiglass caricando 12 µl di amplificato. Le maglie del gel vengono attraversate dai frammenti di DNA in corsa nel campo elettrico, che si separeran-no in base alla propria lunghezza. Più un frammento sarà corto, mesepareran-no resistenza incontrerà tra le maglie e più migrerà lontano. Al gel viene aggiunto Bromuro di Etidio (EtBr) che intercalandosi tra le basi di DNA forma il complesso DNA-EtBr che risulta fluorescente alla luce UV, per-mettendo così di visualizzare il risultato della PCR, posizionando il gel su un transilluminatore UV al termine della corsa elettroforetica.