Workshop
Costruire dispositivi
con un
NanoLEGO bio-inorganico
26 gennaio 2016
Dalle 9.00 alle 15.45
ENEA Casaccia - Sala Mimose
Info:
Selene Baschieri 6612
Cristina Cantale 6602
Claudia Dalmastri 3196
Chiara Lico 3946
Piero Morales 6082
Workshop: ”Costruire disposi1vi con un NanoLEGO bio-‐inorganico" 26 gennaio 2016 ore 9.00 Sala Mimose
Agenda della giornata
Sessione an1meridiana, ore 9.00 precise: tema1ca e specifiche competenze
Ore 9.00 Sistemi e componen1 nanometrici
Gendarme: Selene Baschieri
9.00 Piero Morales Verso una nanotecnologia ibrida bio-‐inorganica
9.15 Claudia Dalmastri Nanostru=ure di DNA come "breadboards” per bioele=ronica e fotonica 9.35 CrisDna Cantale SoFwares di proge=azione e "rendering" di nanostru=ure di DNA
9.50 Angiola Desiderio Repertori molecolari per selezionare ed oJmizzare pepDdi ad alta specificità di legame
10.05 CrisDna Capodicasa AnDcorpi ed aptameri come “bio-‐conne=ori” ad alta specificità 10.20 Chiara Lico Virus: da agenD patogeni a nanoparDcelle
10.35 Massimo Cristofaro Rece=ori olfaJvi negli inseJ come componenD di una stru=ura bio-‐ele=ronica
Ore 10.50 Caffè
Ore 11.10 L’nterfaccia bio-‐inorganica: dal nano al macro
Gendarme: Theo Dikonimos
11.10 Nicola Lisi Carbonio esagonale come interfaccia ele:rica:a;vità di ricerca sul grafene in
Casaccia
11.25 Francesco Buonocore EsperimenD in silico per studiare la stru=ura e la dinamica di sistemi biologici e Caterina Arcangeli autoassemblanD e di interfacce bio-‐inorganiche
11.55 Serena Gagliardi Plasmonica e nano-‐spe=roscopia
12.10 Walter Vastarella Nanosensori e nanosuperfici per biosensori ele=rochimici
12.25 Luigi Quercia Smart Sensing: dallo sviluppo di chemosensori alla sensorisDca biomimeDca
12.40-‐13.45 Pausa pranzo
Sessione pomeridiana, ore 13.45: prospeSve
Moderatore: Piero Morales
Discussione sulle prospe;ve di questa temaAca di ricerca: quali competenze interne, quali esterne, quali finanziamenA, quale personale, quali ricadute, quali utenA finali
Presentazione di idee applicaAve per favorire collaborazioni tra discipline diverse finalizzate alla stesura di proge; per lo sviluppo di nano-‐disposiAvi da uAlizzare in diversi se:ori quali sensorisAca, ele:ronica, informaAca, con ricadute anche in campo medico (diagnosAca), farmaceuAco e alimentare (sicurezza). Tu; i partecipanA sono quindi invitaA a presentare eventuali idee e proposte che verranno discusse nel corso della sessione pomeridiana.
Costruire dispositivi con un
"nano-‐LEGO" bio-‐inorganico
Un workshop con questo titolo si è tenuto il 26 genaio 2016 presso il Centro ENEA della Casaccia. Vi hanno partecipato biologi, fisici, chimici, ingegneri che hanno messo sul tavolo le rispettive competenze per valutare alcune nuove modalità di costruzione e organizzazione della materia su scala molecolare; l'obiettivo è individuare un modo economico, flessibile, modulare, ed energeticamente assai più sostenibile degli attuali metodi usati per l'assemblaggio di dispositivi elettronici e informatici. Nel percorso che è stato delineato, dalla conoscenza delle strutture molecolari alla costruzione di dispositivi con molteplici applicazioni, si può partire dall’osservazione della capacità della natura di autoassemblare atomi e molecole in forme geometriche spesso dai colori accattivanti; e si osserva come alla semplicità delle strutture cristalline inorganiche, corrisponda una grande variabilità di morfologie delle strutture organiche; variabilità la cui origine va ricercata da un lato nella minore intensità delle forze che governano le conformazioni delle macromolecole organiche che formano il mondo vivente, dall'altro nella loro struttura a base polimerica che consente una molteplicità di configurazioni. I fisici hanno coniato il termine di "soft matter" per questo tipo di materia, che facilmente si auto-‐organizza in fogge diverse, e che spesso, come nei processi di biomineralizzazione o di adesione di muffe o batteri, mostra anche la capacità di interazioni tenaci e specifiche verso materiali inorganici. Rispetto alle sintesi di composti inorganici, la sintesi della materia soffice richiede poca energia e, per la materia vivente, procede secondo un progetto preciso, scritto nel "linguaggio" del DNA, macromolecola "soffice" a doppia elica. Accoppiare queste proprietà delle biomolecole, così come altre funzionalità evolute di specifiche molecole organiche, a quelle del materiale inorganico apre nuove, interessantissime prospettive nella scienza dei materiali.
Nel corso della giornata, è stato presentato il potenziale utilizzo innovativo del DNA, basato sulle stesse peculiari proprietà molecolari che ne fanno il sistema universale di informazione genetica e che rendono possibile anche le sue applicazioni come nano-‐materiale per la costruzione di "breadboards" per nano-‐dispositivi. Si tratta di uno straordinario modo di costruire impalcature "soffici" su scala nanometrica, dalle forme più disparate, una sorta di
"Meccano" molecolare basato sull'appaiamento di sequenze appartenenti a catene diverse di
DNA opportunamente programmate. Oltre ai risultati ottenuti nel corso di sperimentazioni effettuate nei laboratori ENEA in collaborazione con altri gruppi (Oak Ridge, e Aharus), sono stati riportati i necessari approcci metodologici, dalla biochimica alla bioinformatica per l'utilizzo e l'upgrade di opportuni softwares per programmare le sequenze di DNA.
Nel progettare appaiamenti di sequenze di DNA, è possibile lasciarne alcuni tratti liberi, a disposizione di ulteriori sequenze da accoppiare in seguito per poter dotare le impalcature di specifiche funzioni in locazioni predeterminate. In questo gioco di assemblaggi un ruolo chiave vengono ad avere dei "connettori" specifici, quali anticorpi (proteici) e aptameri (di acidi nucleici) che immobilizzano specifiche componenti molecolari in locazioni predeterminabili con una precisione di pochissimi nanometri sull'impalcatura di DNA. E, tra i diversi tipi di "connettori" ci sono anche piccoli peptidi che presentano una forte adesione specifica per diverse superfici inorganiche; questi ulteriori connettori, analizzati, sia sperimentalmente che per mezzo di simulazioni computazionali, permettono da un lato di autoassemblare sulla breadboard di DNA nanocomponenti inorganiche come particelle conduttrici o quantum dots inorganici, dall'altro di immobilizzare le nostre "nano-‐ breadboards" in locazioni indirizzabili di un più ampio sistema inorganico, in una logica di organizzazione gerarchica funzionale all'interfacciamento delle nano-‐breadboards con i sensi umani. Sono state analizzate anche nanostrutture naturali prefabbricate, come i virus, che possono essere connesse alle impalcature progettate da noi, in modo organizzato secondo una
logica funzionale alla costruzione di nanodispositivi. In generale, l'uso di componenti prefabbricate ci sposta dalla logica costruttiva del Meccano verso quella del Lego, e cioè verso un gioco ancora di connessioni, ancora modulare, ma con molte più possibilità.
Nel corso della giornata sì è discusso anche di elettronica molecolare. L'utilizzo di singole molecole organiche di riconosciute proprietà semiconduttrici nella nanoelettronica ha subito nell'ultimo decennio una impasse legata al mancato superamento di due problemi principali: l'insufficiente precisione, a livello molecolare, delle pur avanzatissime tecniche di nanofabbricazione artificiale, e la difficoltà di iniettare cariche da nanoelettrodi a stato solido in singoli sistemi organici aromatici e coniugati. Mentre l'autoassemblaggio basato su DNA offre nuove prospettive alla soluzione del primo problema, i recenti studi sul carbonio in forma grafitica bidimensionale (grafene) e le simulazioni di peptidi selettivi per questa forma del carbonio offrono prospettive altrettanto valide alla soluzione del secondo problema. Sono state quindi analizzate le capacità di sintesi per Chemical Vapour Deposition, e trasferimento di grafene su opportuni substrati, sviluppate presso i laboratori dell'ENEA e si è analizzata la possibilità di un patterning litografico del grafene in dispositivi bio-‐inorganici.
Oltre alla connessione elettrica basata su grafene, si è parlato anche di connettori fotonici come interfaccia tra i nanodispositivi e i sistemi di rivelazione, ovvero dell’uso di fotoni per scambiare segnali, come avviene ad esempio per le reazioni chimiche associate a un segnale fluorescenza. Se disponessimo di sorgenti di luce particolarmente intense e strettamente localizzate, potrebbe essere vantaggiosamente sfruttato il debole segnale di diffusione anelastica della luce: lo scattering Raman, che è l'impronta più caratteristica e specifica della composizione e della conformazione molecolare. Oggi è possibile realizzare nanoantenne metalliche autoorganizzate su breadboards di DNA, che sfruttando la risonanza plasmonica di nanoparticelle di metalli nobili, emettono luce nella regione visibile e possono fornire segnali Raman sufficientemente intensi anche da singole molecole localizzate nel cosiddetto "campo vicino" (cioé < della lunghezza d'onda della luce) dell'antenna. Questo tipo di interfacciamento è favorito dallo sviluppo della "plasmonica" (lo studio delle eccitazioni elettroniche collettive di un materiale che producono un dipolo oscillante con conseguente emissione di luce).
I biosensori, logico sbocco del percorso "biomimetico" verso la realizzazione economica e sostenibile di nano-‐dispositivi sono un problema tecnologico aperto. Anche se conosciamo innumerevoli sensori presenti in natura (per luce, temperatura, umidità, pH), la traduzione dei segnali molecolari dei sistemi biologici in segnali elettrici utilizzabili resta spesso problematica, in particolare per dispositivi nanometrici. Alcuni biosensori naturali, come le cellule delle antenne degli insetti hanno tuttavia sensibilità straordinarie e possono avere applicazioni pratiche molto utili e dirette. La tecnica della elettroantennografia, per esempio, sorprendente nella sua semplicità e affidabilità, si basa sull'intero meccanismo cellulare (micrometrico), che è più facile da tradurre in un segnale utilizzabile rispetto al meccanismo molecolare (nanometrico). Integrare quest'ultimo nei nanodispositivi su base di DNA potrebbe rappresentare un formidabile avanzamento e la relazione sulle possibilità offerte dai sensori entomologici è stata di grande stimolo.
Le ultime due relazioni della mattinata di workshop sono state dedicate all'illustrazione del lavoro in corso per il miglioramento delle prestazioni di sensori e biosensori, con un accento particolare al problema dell'aumento del rapporto superficie/volume mediante l'impiego di materiali nanostrutturati. Agli interessanti risultati ottenuti si aggiunge la padronanza di una tecnologia più consolidata nella parte di trasduzione del segnale elettrico ed elettrochimico. Questa ricerca, più direttamente orientata all'applicazione può beneficiare fortemente delle nanotecnologie "bio-‐templated", così come lo sviluppo di queste ultime può avvalersi delle tecnologie della prima. In generale, le sinergie rilevate nel corso della giornata tra i diversi gruppi di ricerca che si sono incontrati sono estremamente promettenti.
Workshop
"Costruire dispositivi con un
NanoLEGO bio-‐inorganico"
Casaccia, 26 gennaio 2016, ore 9.00, Sala Mimose
Piero Morales
(SSPT-‐PROMAS-‐MATPRO. Tel.: 06 3048 6082/6350; e-‐mail: [email protected])
Verso una nanotecnologia ibrida bio-‐inorganica e autoassemblante
Gli sviluppi della nanoelettronica e della microtecnologia degli ultimi dieci anni, unitamente ai grandi progressi della biochimica e della biologia molecolare, e alla recente visione del DNA come materiale per nanofabbricazioni programmabili e componibili (un nano-‐LEGO molecolare), ci permettono di ipotizzare la possibilità di ottenere dispositivi autocostruiti, con costi energetici e produttivi trascurabili. Alle necessarie nanotecnologie artificiali su materiali inorganici potremo sostituire o affiancare sempre di più delle nanotecnologie organiche o biologiche autoassemblanti, e dei meccanismi di accoppiamento meccanico, elettrico e fotonico tra il nano-‐mondo a base di silicio e quello a base di carbonio.
La giornata di workshop cercherà di far emergere un potenziale coordinamento tra la ricerca biologica e biochimica da un lato, e le nanotecnologie artificiali dall'altro, per progettare e costruire materiali e dispositivi sostenibili.
Claudia Dalmastri
(SSPT-‐BIOAG-‐SOQUAS. Tel.: 06 3048 3196; email: [email protected])
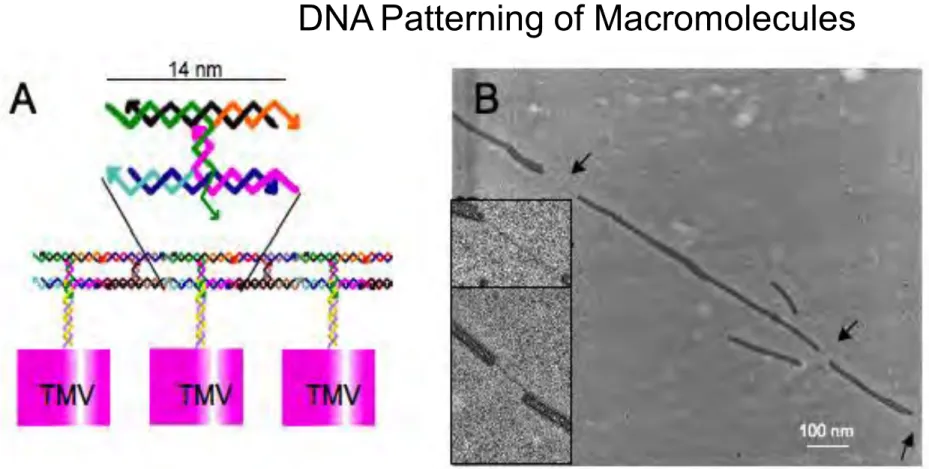
Nanostrutture di DNA come "breadboards" per bio-‐elettronica e fotonica
Le stesse proprietà molecolari del DNA che ne fanno il sistema universale di informazione genetica rendono possibile anche le sue applicazioni come nano-‐materiale per la costruzione di breadboards per nano-‐dispositivi. Infatti, l’appaiamento specifico tra le basi nucleotidiche che, nella cellula vivente, determina la struttura a doppia elica e garantisce l’esatta replicazione ed espressione del DNA, permette anche di costruire in laboratorio strutture complesse di DNA in cui forma e dimensione dipendono dall’autoassemblaggio di sequenze nucleotidiche programmate.
Esistono due approcci principali per la costruzione di nano-‐strutture di DNA. Il primo, sviluppato da Winfree e LaBean, si basa sull’appaiamento tra oligonucleotidi sintetici a livello di siti specifici. La programmazione prevede il disegno di regioni complementari per il riconoscimento specifico tra coppie di oligonucloeotidi predefiniti e la formazione di segmenti a doppia elica solo in queste regioni prescelte, in modo da generare una struttura a tessere (“tiles”, ognuna ottenuta da 9 oligonucleotidi) delle dimensioni desiderate. Il secondo approccio, ideato da P. Rothemund, consiste nella formazione di strutture di DNA a partire da un’unica molecola di DNA a singolo filamento di origine naturale. Attraverso il legame con brevi oligonucleotidi complementari a regioni selezionate del DNA del batteriofago M13, questo viene ripiegato su se stesso per dare origine a strutture di forma diversa, da cui la definizione di “origami” in analogia con l’arte giapponese.
Entrambi i sistemi sono stati studiati nei laboratori dell’ENEA. Gli aspetti tecnici e le relative difficoltà operative, i risultati ottenuti, le problematiche emerse, le prospettive applicative saranno oggetto della presentazione.
Cristina Cantale
(SSPT-‐BIOAG-‐SOQUAS. Tel.: 06 3040 6601; e-‐mail: [email protected])
Softwares di progettazione e "rendering" di nanostrutture di DNA
La possibilità di usare il DNA per applicazioni nanotecnologiche ha richiesto lo sviluppo di strumenti di software dedicato. Alcuni di questi software sono liberamente disponibili altri sono commerciali.
In questo modo sono state disegnate le sequenze per le giunzioni delle eliche alla base delle “tiles”, per definire le “sticky-‐end” necessarie al loro assemblaggio e per predire i prodotti della reazione di auto assemblaggio di un’insieme di filamenti di DNA.
Sono anche stati sviluppati 3D Editors per il disegno di strutture di DNA.
Il disegno di DNA origami ha richiesto ulteriori strumenti di calcolo in grado di gestire il disegno di un grande numero di filamenti unici di DNA che si organizzano su un filamento in una forma ben definita.
Verranno analizzati alcuni di questi softwares, con particolare attenzione al disegno di DNA origami.
Angiola Desiderio
(SSPT-‐BIOAG-‐BIOTEC. Tel.: 06 3048 4176; e-‐mail: [email protected])
Repertori molecolari per selezionare e ottimizzare peptidi ad alta specificità di legame
Alcune molecole biologiche hanno la proprietà di controllare finemente i processi cellulari in virtù della loro capacità di riconoscere in maniera altamente specifica un’ampia varietà di superfici bersaglio su scala nanometrica. Sfruttando questa abilità è stato dimostrato che alcuni peptidi (brevi catene di aminoacidi) sono in grado di legare selettivamente diversi materiali inorganici.
Peptidi con caratteristiche definite possono essere selezionati e prodotti con sistemi biotecnologici, che sfruttano la possibilità di montare sequenze aminoacidiche sulla superficie di virus (Phage Display Libraries). La possibilità di introdurre attraverso questo sistema variabilità di sequenza in maniera combinatoriale permette di costruire repertori molecolari ad altissima complessità, utilizzabili anche per migliorare l‘affinità e/o la specificità di legame di peptidi per una data superficie.
Questo strumento biotecnologico permette quindi di ottenere specifici nanoleganti per una ampia varietà di applicazioni, molte delle quali ancora da esplorare.
Cristina Capodicasa
(SSPT-‐BIOAG-‐BIOTEC. Tel.: 06 3048 6558; e-‐mail: [email protected])
Anticorpi ed aptameri come “bio-‐connettori” ad alta specificità
Gli anticorpi sono proteine complesse prodotte dal sistema immunitario animale che
riconoscono e neutralizzano tutto ciò che viene considerato estraneo dall’organismo, a partire da microrganismi infettivi (come batteri, funghi, virus) fino a singole molecole (proteine, tossine etc.). La capacità di legare molecole diverse con estrema specificità è stata sfruttata negli anni per molteplici scopi applicativi. Anticorpi specifici contro un target d’interesse possono essere ottenuti mediante selezione in vivo, utilizzando il sistema immunitario animale, oppure mediante una selezione in vitro da repertori sintetici di anticorpi ricombinanti.
Un' alternativa più recente alle proteine anticorpali è rappresentata dagli aptameri che sono invece acidi nucleici a singolo filamento caratterizzati da una specifica struttura tridimensionale e che sono in grado anch’essi di legare molecole di varia natura. Gli aptameri, al contrario degli anticorpi, si possono ottenere unicamente mediante selezione in vitro. La specificità e la versatilità di legame degli anticorpi e degli aptameri rendono queste molecole efficaci strumenti per applicazioni biotecnologiche e terapeutiche.
Nell’ambito della sensoristica, queste molecole di pochi nanometri possono essere immobilizzate sulla superficie diuna nanostruttura di DNA, funzionando poi come “connettorii” in grado di catturare e/o posizionare in modo specifico componenti ulteriori del sensore o molecole d’interesse.
Chiara Lico
(SSPT-‐BIOAG-‐BIOTEC. Tel.: 06 3048 3946; e-‐mail: [email protected] )
Virus: da agenti patogeni a nanoparticelle
I virus sono agenti infettivi in grado di replicarsi solo all' interno di cellule (animali, vegetali o batteriche) in maniera altamente ospite-‐specifica. Al di fuori del loro specifico ospite i virus possono essere considerati come “nano-‐oggetti” strutturalmente molto semplici perché costituti da un guscio (capside) di dimensioni comprese tra i 20 e i 900 nm con scheletro proteico e morfologia e complessità variabili, che racchiude il genoma (RNA o DNA). Grazie a queste caratteristiche i virus stanno trovando applicazione in settori di ricerca anche molto distanti tra loro. Nanoparticelle virali di diversa natura (ad esempio virus vegetali) mediante funzionalizzazione delle proteine capsidiche (ad esempio “ingegnerizzazione” con peptidi opportuni o derivatizzazione chimica) sono stati utilizzati per mettere a punto saggi diagnostici, come veicolo di antigeni o farmaci, o per ottenere la deposizione controllata di materiali di interesse su scala nanometrica.
Massimo Cristofaro
(SSPT-‐BIOAG-‐PROBIO. Tel.: 06 3048 3480; e-‐mail: [email protected]) Recettori olfattivi negli insetti come componenti di una struttura bio-‐elettronica
Mentre per altri mammiferi noti per l’elevata percezione degli odori, come il cane, la differenza è data dal numero di recettori e dalle differenze fisiologiche dell’organo, un discorso a parte va fatto per gli insetti, in cui l’organo olfattivo è morfologicamente e funzionalmente diverso. Esso è costituito dalle antenne, in ognuna delle quali sono presenti diversi tipi di unità olfattive (sensilli), ciascuna contente da uno a più neuroni olfattivi (ORN). Inoltre, oltre ai recettori olfattivi usuali, gli insetti dispongono di un sistema trasduzionale alternativo che sfrutta un gruppo di proteine (ORCO) che, attivando le proteine binding (OBP) precedentemente legate alla molecola volatile, ne permettono la connessione con lo specifico ricettore di membrana, posizionato sul dendride dell’ORN. Si ipotizza che gli insetti abbiano aumentato in modo esponenziale il numero di ricettori olfattivi e che utilizzino queste proteine come meccanismo molecolare selettivo che gli consente di determinare la presenza di una molecola volatile anche a concentrazioni molto basse.
Inoltre, gli insetti –come i cani-‐ sono dotati di capacità di apprendimento, ovvero possono imparare a riconoscere un odore “inusuale”. Tale capacità è stata già utilizzata con alcuni insetti sociali (le api) per scopi antiterroristici. Su questo tema ci siamo precedentemente confrontati con altri ricercatori, per realizzare dei sistemi di monitoraggio biologici e/o bio-‐ tecnologici per la georeferenziazione di dispostivi bellici interrati (land-‐mines).
L’obiettivo che ci poniamo, nell’ambito delle nanotecnologie, è quello di confermare che anche il riconoscimento di molecole “diverse” sia basato su una componente molecolare specifica: se l’ipotesi è corretta questo permetterebbe di sintetizzare una nuova classe di biosensori, con una componente proteica ricavata dall’analisi molecolare del complesso di proteine e recettori di membrana deputato al riconoscimento di specifici odori “acquisiti”.
Nicola Lisi
(SSPT-‐PROMAS-‐MATPRO. Tel.: 06 3048 4180; e-‐mail: [email protected])
Carbonio esagonale come interfaccia elettrica: attività di ricerca sul grafene in Casaccia
La deposizione chimica da fase vapore, o CVD, è una tecnica molto potente che permette la crescita di strati di carbonio con spessore atomico. La dimensione dei depositi è limitata esclusivamente dalla dimensione dei reattori e dei substrati, ed è quindi possibile la deposizione di campioni di grandi dimensioni. La crescita avviene su fogli metallici sottili, come il rame o il nichel. La microstruttura dei depositi può essere controllata variando le condizioni di crescita, per ottenere depositi con caratteristiche funzionali diverse: grafene (monocristallo o cristalli grandi, ibridizzazione sp2, singolo strato), Few Layer Graphene o FLG (cristalli grandi, ibridizzati sp2, da 2 a 10 strati), grafene nanocristallino (cristalli piccoli, ibridizzati sp2, singolo strato), grafene funzionalizzato (parziale ibridizzazione sp3, gruppi funzionali sopra la superficie), grafene drogato (atomi diversi, p.e. N, sostituiti al C nel reticolo).
Dopo il CVD il grafene viene separato dal substrato metallico per il suo successivo utilizzo, e trasferito sul substrato di destinazione con processi a bassa temperatura. Questo è un passaggio critico per il mantenimento dell’integrità della struttura e delle proprietà del materiale, ma anche un elemento essenziale dell’interesse per il grafene e per gli altri
materiali 2D.
La scelta del tipo di grafene e quindi del processo di crescita e del metodo di trasferimento viene effettuata in base all’applicazione a alla caratterizzazione scelta.
Le applicazioni industriali del grafene CVD non sono dietro l’angolo, per i costi elevati e per le proprietà elettroniche che non raggiungono ancora i valori desiderati, ma anche perché i materiali 2D vengono ad alterare il “paradigma” ed occorre tempo perché emergano metodi, processi e soprattutto dispositivi che ne sfruttino le potenzialità. Presso i laboratori della Casaccia, in collaborazione con Portici, abbiamo studiato l’applicazione fotovoltaica in combinazione con assorbitori di Silicio.
Francesco Buonocore e Caterina Arcangeli
(SSPT-‐PROMAS-‐MATPRO. (CA) Tel.: 06 3048 6898; e-‐mail [email protected]; (FB) Tel.:06 3048 6445; e-‐mail [email protected])
Esperimenti in silico per studiare la struttura e la dinamica di sistemi biologici autoassemblanti e delle interfacce bio-‐inorganiche
Il rapido avanzamento nel campo della tecnologia HPC ha permesso di introdurre nella scienza odierna l’esperimento computazionale meglio noto come esperimento in silico. Quest’ultimo si avvale di una modellistica teorica di base e della sua implementazione in un algoritmo con cui simulare il comportamento di un sistema. In base all’efficienza dell’algoritmo è possibile investigare sistemi più realistici e via via più complessi. Gli esperimenti in silico possono raggiungere condizioni molto vicine a quelle sperimentali, e a volte possono essere confrontati direttamente con essi. Quando ciò accade, le simulazioni diventano uno strumento molto potente non solo per capire e interpretare gli esperimenti a livello microscopico ma anche studiare fenomeni
non accessibili sperimentalmente o che implicano esperimenti costosi.
Nel nostro intervento vi illustreremo alcuni esempi pratici di un approccio computazionale che combina modelli ab initio e simulazioni classiche per studiare la struttura e la dinamica delle interfacce bio-inorganiche e dei sistemi biologici autoassemblanti.
Serena Gagliardi
(SFN-‐TECFIS. Tel.: 06 3048 4299; e-‐mail: [email protected])
Plasmonica e nanospettroscopia
L’interazione del campo elettromagnetico con materiali metallici e nanostrutturati viene descritta introducendo i concetti di plasmone e di plasmone localizzato [1].
Sfruttando le peculiari proprietà ottiche dei plasmoni, è stato possibile nel corso degli ultimi 20 anni sviluppare tecniche di nanospettroscopia. Nella nanospettroscopia l’interazione in campo vicino viene utilizzata per localizzare la radiazione di sonda al di sotto del limite diffrazione della luce, ovvero per aumentare anche di diversi ordini di grandezza i segnali e ottenere informazioni da singole molecole. Tra le più diffuse tecniche di spettroscopia che sfruttano la plasmonica ci sono il surface-‐enhanced fluorescence (SEF), surface-‐enhanced Raman scattering (SERS) [2], e la spettroscopia Raman di tipo tip-‐enhanced [3].
Se è vero che gli studi sulla plasmonica sono iniziati più di 20 anni fa, d’altro canto solo un più lento progresso anche tecnologico consente un utilizzo in modo controllato di queste tecniche, che fino ad oggi sono state sviluppate ed utilizzate spesso in modo fenomenologico. La morfologia delle nano particelle metalliche, la forma degli aggregati, e le loro distanze determinano le proprietà ottiche dei plasmoni di superficie; pertanto la capacità di sintesi di nanoparticelle singole e di controllo dell’aggregazione, e disposizione su pattern ben definiti, ad esempio attraverso opportune funzionalizzazioni, permette pertanto di avere probe spettroscopici ovvero metamateriali per applicazioni diverse con caratteristiche modellabili e controllate [4, 5].
Nel breve intervento verranno illustrati i fondamenti e le possibili applicazioni dei concetti accennati sopra.
Walter Vastarella
(SSPT-‐PROTER-‐BIOGEOC. Tel.: 06 3048 6883; e.mail: [email protected])
Nanosistemi e nanosuperfici per biosensori elettrochimici
Il contributo sarà incentrato principalmente sull’uso di sistemi nanostrutturati e superfici nano-‐dimensionate per applicazioni in biosensoristica. Nella fattispecie saranno illustrate le enormi potenzialità che offrono i sistemi nano-‐sagomati (membrane, molecular imprinted polymers, sistemi micellari) per costruire in modo semplice, con processi bottom-‐up, supporti come detector elettrochimici o per microelettronica.
Saranno illustrate varie tipologie di nanoelettrodi e nanotubi, funzionalizzati e non, per biosensori enzimatici ed immunosensori. Seguendo i recenti studi e sviluppi di dispositivi innovativi multiparametrici, sempre con sensoristica di tipo elettrochimico (microarray, lab on chip), si arriverà ad esplorare il camop degli aptasensori (DNA biosensors). Una valutazione generale sui vantaggi delle nanotecnologie nei biosensori sarà utile per aiutare a capire quali futuri sviluppi ed investimenti potranno risultare utili a rilanciare il settore della bioanalitica.
Luigi Quercia
(SSPT-‐USER-‐SITEC. Tel.: 06 3048 3243 ; e-‐mail: [email protected])
Smart Sensing: dallo sviluppo di chemosensori alla sensoristica biomimetica
Nel corso degli ultimi 20 anni sono state sviluppate diverse tipologie di chemosensori basati su ossidi metallici, film polimerici e materiali inorganici nanostrutturati per la realizzazione di nasi elettronici in grado di rivelare miscele di gas e composti organici volatili. Le principali criticità da affrontare con questi materiali sono la selettività e la sensibilità dei dispositivi ottenuti. Lo sviluppo recente delle bionanotecnologie rende possibile investigare nell’ambito della sensoristica biomimetica per la realizzazione di oggetti e dispositivi che traggono ispirazione dalla natura. Materiali sensibili basati sul sistema olfattivo biologico possono consentire la rilevazione di basse concentrazioni di composti volatili organici grazie alla loro elevata selettività e sensibilità, ottime caratteristiche di biocompatibilità, biodegradabilità, fornendo la base per la realizzazione di dispositivi sensoriali dal ridottissimo consumo energetico e rapidi tempi di risposta. La porta di accesso a questo “mondo affascinante” è una opportuna nanotecnologia ibrida bio-inorganica.
Costruire disposi,vi con un nanoLEGO bio-‐inorganico
Verso una nanotecnologia ibrida bio-‐
inorganica
Perché un diverso modo di
costruire
L’ele?ronica è dovunque, e siamo sempre più
affama, di tante nuove “funzioni”, sempre più
concentrate…
Lo schema di uno smartphone e uno zoom sui suoi
componen5. La CPU in basso a destra con5ene milioni di
transistors
Due immagini di fabbriche di chips ele=ronici in camera pulita
(a sinistra) e di assemblaggio di telefoni cellulari (a destra)
danno un’idea visiva delle risorse impiegate nell’ele=ronica e
I.T.
Microele?ronica e nanotecnologie sono i
processi produLvi che richiedono più
energia di tuL
La maggior parte dei
disposi5vi ele=ronici
odierni ha richiesto
per la fabbricazione
più energia di quanta
ne useranno in tre
anni di loro vita
media.
Guardate i numeri:
10
13
-‐10
8
J/kg!!
N.B. in termini di energia occorre
mol5plicare x 3
Fonte: Williams A. United Na5ons Univ. Tokio 2008
Per fabbricare un computer
servono 7.3 x10
9Joule di
… e il mo,vo di questa spaventosa richiesta di
energia sta nella tecnologia di fabbricazione “top-‐
down” per fare miliardi di ques, disposi,vi
Decine di diversi processi in serie su miliardi di disposi5vi, decine
di migliaia di mq di impian5 in camera pulita, materiali ultrapuri,
acidi e basi, alte temperature….
A present day
L’ odierna ele?ronica non è sostenibile….
…. ma possiamo farne a meno?
L’abbiamo inventata per avere da, a portata di mano molto
più velocemente:
l’ele?ronica a stato solido è un milione di volte più veloce del
nostro pensare…
C’è una soluzione? Possiamo inventarci un modo di fabbricare
disposi,vi velocissimi a basso costo?
Da qualche anno, crediamo di sì
In realtà, la storia era già iniziata 40 anni fa…
Arieh Aviram e Mark Ratner, 1974
Una catena coniugata di poli5ofene tra due nanoele=rodi:
il polimero costa poco, le corren5 sarebbero molto basse, e….
se potessimo montarlo facilmente…
“A strategic plan for molecular electronics”
Bella idea, ma…
… non sappiamo fabbricare nanoele=rodi a distanza
controllata al nanometro, e abbastanza uniformi e so`li,
e la corrente “blu” (tunnel tra gli ele=rodi) può essere
confrontabile con quella “rossa” (through molecule). E
poi…
…possono cambiare gli angoli e
l’isolamento al conta?o… e molto altro
… e così dopo 40 anni è ben lungi dall’essere
economicamente e commercialmente appe5bile.
In sostanza:
•
Le tecnologie della microele=ronica a stato solido non sono
abbastanza precise per un matching con singole molecole
organiche;
•
Le energie necessarie per inie=are cariche da un metallo a
una singola molecola organica sono talmente alte da
modificare l’interfaccia in modo casuale;
•
I cos5 energe5ci restano al5ssimi perché sempre di
tecnologie ar5ficiali “top-‐down” si tra=a.
Per il momento, niente ele?ronica molecolare…
e allora… madre natura ci offre qualche spunto:
sa fabbricare nanostru?ure più complesse, più precise
e più piccole dei nostri tri-‐gates, a cos, energe,ci
bassissimi…
E.T.?
No, virus T4
… ma ormai lo si sa quasi fare; con un sistema
modulare a base di DNA
Vaso azteco?
No,nanostru6ura di DNA 3-‐
dimensionale
Amme?endo di saperlo fare bene, dobbiamo conne?erci con
una parte, rido?a ma per ora indispensabile, di ele?ronica
convenzionale. Vogliamo un’organizzazione gerarchica delle
componen, su diversi livelli di “breadboards”
La breadboard inorganica in alto a sinistra è all’incirca il massimo che
sappiamo fare con la litografia, ma possiamo spingerci più avan5 con la
densità a=accandoci le nano-‐breadboards di DNA; sulle quali, a loro volta,
si autoassemblano le componen5 proteiche o altri sistemi più complessi.
Come nel LEGO
Inorganica,
Au/SiO
2
Organica,
pr
oteine su DNA
(Gothelf group,
2010)
Insomma vogliamo stabilire un modo
di fabbricare nanometrico,
economico, modulare, interfacciabile
con lo stato solido (che oltre che
veloce è anche molto affidabile).
Come possiamo contribuire a questa idea?
Questo è lo scopo di questo workshop
Claudia Dalmastri
e
Cris5na
Cantale
mostrano come
•
si possono
disegnare
e
autoassemblare
semplici
nanostru=ure di DNA da
usare come “breadboards”
per componen5 proteiche,
con una densità di circa 1/30
nm
2
•
si possono agganciare e
sospendere queste nano-‐
schedine a nanoele=rodi
metallici
Angiola Desiderio
,
Caterina
Arcangeli e Francesco Buonocore
affrontano, l’una dal punto di
vista
sperimentale
, gli altri da
quello
teorico-‐computazionale
un diverso 5po
di aggancio
seleBvo
,
basato su cariche
ele=riche e conformazioni di
pep5di, tra nanostru=ure
organiche e superfici
inorganiche; o, viceversa, di
nanostru=ure inorganiche
(quantum dots) su archite=ure
organiche
Cris5na Capodicasa ci parla dell’uso di
an5corpi
(proteine) e di
aptameri
(acidi nucleici) come
“
conne6ori”
da montare sulle “breadboards” per
mol5plicare le possibilità di autoassemblaggio di
componen5
Chiara Lico
sposta
l’a=enzione sull’uso di
virus
ba=eriofagi e
vegetali
come
componen5
biologiche
più complesse e
preassemblate, da
immobilizzare sulle
“breadboards”, o da
usare come nanowires o
nanoantenne.
Massimo Cristofaro
allarga ulteriormente la
prospe`va della componen5s5ca a
sistemi più
complessi
da usare come sensori ultra sensibili, gli
antennomeri degli inse`; inoltre ci dà un primo
esempio di interfacciamento grossolano ma
efficace, verso l’estrazione di un segnale ele=rico
dal processo di riconoscimento molecolare
Sistemi e componen, nanometrici
Dal nano al macro
Nicola Lisi
racconta
schema5camente i progressi
fa` sulla sintesi e il
trasferimento di foglie` di
grafene
e perché ques5 siano
par5colarmente u5li per
interfacciare ele=ricamente
la componen5s5ca organica
con nanowires metallici e con
l’ele=ronica convenzionale
Sistemi e componen, nanometrici
Dal nano al macro
Serena Gagliardi
mostra la prospe`va di
un diverso interfacciamento tra il macro
e il nano, quello che si può o=enere
a=raverso l’emissione controllata di luce
su dimensioni più piccole di quelle della
sua lunghezza d’onda; e quindi in grado
di interagire perfino con singole
molecole. E di converso mostra come le
possibilità offerte dagli origami di DNA di
posizionare nanopar5celle metalliche con
precisione nanometrica, perme=a di
o=enere nano-‐eme`tori di luce
(nanoantenne) in modo potenzialmente
ben controllabile
Walter Vastarella
illustra le tecnologie
dei
micro-‐ e
nanobiosensori
sviluppate in Casaccia
e le più recen5
metodologie di
rivelazione del segnale
ele=rochimico
Luigi Quercia
si sposta
verso le applicazioni
più industriali,
illustrando la ricerca
sui chemosensori e
biosensori e le
applicazioni di un naso
ele=ronico
commerciale al se=ore
ortofru`colo
Buon diver,mento!!
Nel pomeriggio discuteremo di
prospe`ve, di applicazioni, di proge`
e…
di come reperire le risorse economiche
per questo lavoro
Claudia Dalmastri
e
Cris.na
Cantale
•
mostrano come si possono
disegnare
e
autoassemblare
semplici nanostru5ure di DNA
da usare come “breadboards”
per componen. proteiche, con
una densità di circa 1/30 nm
2
•
e come si possono agganciare
e sospendere queste nano-‐
schedine a nanoele5rodi
metallici
Nanostru1ure di DNA
come "breadboards" per
bioele1ronica e fotonica
Claudia Dalmastri
Breadboards
Da micro a nano
Perché il DNA?
Perché usare materiale organico per costruire
disposi.vi ele5ronici?
Perché il DNA ha proprietà molecolari uniche di
autoassemblaggio e potenziale adesione a infinite
stru1ure molecolari
Stru1ura a doppia elica, Watson e Crick (1953)
DNA: la doppia elica
Il DNA è “nano”