3. Materiali e metodi
3.1 Linea cellulare
La linea cellulare BSMC (Clonetics® Normal Human Bronchial Smooth Muscle
Cells) (Figura 12) è costituita da cellule di muscolatura liscia bronchiale umana. Le cellule crescono in adesione, si presentano morfologicamente con una aspetto affusolato ed hanno un tempo di duplicazione di circa 24 ore.
Fig. 12. BSMC in coltura
Le cellule sono state coltivate nel mezzo SmGM®-2 (Lonza) contenente
glucosio (5,5 mM), FBS (5%), gentamicina (50 µg/ml), anfotericina-B (50 ng/ml), insulina (5 µg/ml), fattore di crescita fibroblastico basico umano (hFGF- B) (2 ng/ml) e fattore di crescita epidermico umano (0,5 ng/ml).
La crescita ed il mantenimento in coltura delle cellule sono stati realizzati in fiasche T75 e T25 ad una temperatura costante di 37°C, in un’atmosfera controllata al 5% di CO2. Le cellule sono state mantenute in crescita
esponenziale fino al 70-80% di confluenza e successivamente sub-coltivate in rapporto 1:3.
3.2 Modello in vitro di desensibilizzazione omologa
Il modello di desensibilizzazione omologa del recettore β2, è stato realizzato
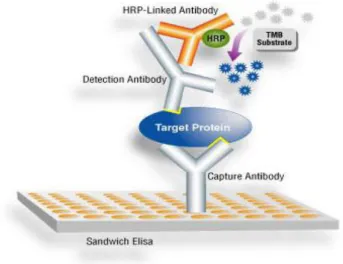
mediante l’esposizione di cellule BSMC per 24 h ad una concentrazione 1 µM di salbutamolo. La valutazione del grado di desensibilizzazione è stata effettuata mediante la misurazione dei livelli intracellulari di cAMP, monitorati con il saggio ELISA “Cyclic AMP EIA kit” (Cayman Chemical company, USA) (Figura 13).
Fig. 13. Funzionamento del saggio ELISA
3.3 Analisi RT-PCR
L’espressione dei recettori β2 e dei PPAR è stata valutata mediante tecnica PCR
seminate in fiasche T25 in presenza o meno di un trattamento farmacologico. L’RNA totale è stato estratto mediante RNeasy Mini kit (Qiagen, Valencia, CA, USA). La concentrazione ed il grado di purezza dell’RNA totale è stato misurato mediante l’assorbimento a 260 nm e la valutazione del rapporto dei valori ottenuti alle due lunghezze d’onda 260 nm e 280 nm, impiegando uno spettrofotometro GeneQuant pro (Biochrom, UK). La qualità del materiale di partenza era indicato da un rapporto di densità ottica 260/280 nm > 1,9. L’integrità dell’RNA è stata inoltre verificata mediante elettroforesi su gel di agarosio all’1 %.
Per ogni campione, è stato retrotrascritto 1 µg di RNA totale mediante il QuantiTect Reverse Transcription kit (Qiagen, Valencia, CA, USA), mentre l’amplificazione del cDNA è stata realizzata mediante il kit “Masterscript RT- PCR System” (Qiagen, Italia). I primers per il recettore β2, PPAR-γ e per il gene
housekeeping GAPDH sono stati disegnati mediante il software Primer-Blast (NCBI) e sintetizzati da Sigma Genosys (Cambridge, UK). Le sequenze dei primers utilizzate sono state: ACCAGGAAGCCATCAACTG-3′ (F) e 5′-GAAGACCATGATCACCAGGGG-3′ (R) per il recettore β2-adrenergico;
5′-TTCAGAAATGCCTTGCAGTG-3′ (F) e 5′-CACCTCTTTGCTCTGCTCCT-3′ (R) per il PPARγ; ATGACTTCCAAGCTGGCCGT-3′ (F) e CCTCCTTCAAAAACTTCTCCACACC-3′ (R) per l’interleuchina-8; 5′-TGAAACCCACTCCAAACACA-3′ (F) e 5′-AACTGATGCGTGAAGTGCTG-3′ (R) per l’enzima COX-2; 5′-GTGAAGGTCGGAGTCAACG-5′-AACTGATGCGTGAAGTGCTG-3′ (F) e 5′-GGTG AAGACGGCCAGTGGACTC-3′ (R) per l’enzima GAPDH.
3.4 Analisi EMSA (Electrophoretic Mobility Shift Assay)
Gli estratti nucleari (5 µg) sono stati preincubati per 20 minuti in ghiaccio con 2 µg di poli (dI-dC) e successivamente per 30 minuti a temperatura ambiente, con la sonda oligonucleotidica PPAR marcata con gamma [32P] ATP (100,000 – 150,000 cpm; Perkin Elmer) in buffer di legame (50% glicerolo, 10 mM Tris– HCl, pH 7.6, 500 mM KCl, 10 mM EDTA, 1 mM ditiotreitolo) in volume finale di 20-30 µl. L'oligonucleotide consensus per PPAR′ (5- CAAAACTAGGTCAAAGGTCA-3′) è stato ottenuto da Santa Cruz Biotechnology. Il complesso sonda–proteina è stato separato mediante corsa elettroforetica su gel nativo 5% polyacrylamide in 0.5X TBE buffer (100mM Tris–HCl, 100 mM boric acid, 2mM EDTA) a 150 V in ghiaccio. Il gel è stato poi essiccato e le bande radioattive individuate mediante autoradiografia.
3.5 Isolamento dei monociti e formazione delle MP
I monociti sono stati isolati dal sangue periferico di donatori. Ogni campione è stato diluito 1:1 con PBS/EDTA 2 mM, miscelato con ¼ del volume di una soluzione di Dextran T500 al 4% e lasciato sedimentare per 30 minuti a temperatura ambiente, per eliminare tutti gli eritrociti presenti. Il surnatante è stato poi centrifugato per 10 minuti a 200 g. Il pellet ottenuto è stato risospeso in 30 ml di PBS/EDTA, portato a volume con 15 ml di soluzione Ficoll-Hypaque, e centrifugato per 30 minuti a 350 g a temperatura ambiente. Le
seminate in piastre da 24 pozzetti per 18 ore a 37°C in modo da ottenere circa 106 cellule per pozzetto. La produzione di microparticelle è stata indotta trattando i monociti con lo ionoforo del calcio, A23187, alla concentrazione di 12 µM in RPMI 1640 per 15 minuti a 37°C. Le cellule sono state poi centrifugate a 14.000 g per 5 minuti a temperatura ambiente per rimuovere i monociti ed i frammenti cellulari di grosse dimensioni che possono essersi formati durante la stimolazione.
3.6 Misurazioni dei livelli di IL-8
Le cellule sono state seminate in piastre da 96 pozzetti alla densità di 5.000 cellule per pozzetto. Lo schema di trattamento è stato lo stesso impiegato per la valutazione dei livello di cAMP (paragrafo precedente). La superficie di poliestere di ogni pozzetto di una piastra da 96 è stata ricoperta con 100 µl di anticorpo, specifico per la proteina di interesse, diluito in coating buffer e incubato a 4°C. Il giorno dopo ogni pozzetto è stato bloccato con 300 µl di Assay buffer per 1 ora a temperatura ambiente. Passata un’ora è stato eliminato l’Assay buffer, sono stati aggiunti 100 µl di ogni campione diluito in Assay buffer ed è stata costruita la curva di calibrazione attraverso la quale è stato possibile valutare la concentrazione di IL-8. Immediatamente dopo, ai campioni ed allo standard sono stati aggiunti 50 µl di anticorpo biotinilato specifico per la proteina in esame diluito in Assay Buffer. La piastra è stata incubata a temperatura ambiente per 2 ore in condizioni di lenta agitazione. Dopo tre
lavaggi con 300 µl di Wash Buffer sono stati aggiunti 100 µl/pozzetto di Streptavidin-HRP diluito 1:400 in Assay Buffer e la piastra è stata incubata per 30 minuti a temperatura ambiente. Dopo ulteriori cinque lavaggi con Wash Buffer, sono stati aggiunti 100 µl del substrato cromogeno 3, 3’, 5, 5’ tetrametil benzidina (TMB) e la piastra è stata incubata per 5 minuti a temperatura ambiente, dopo è stata bloccata la reazione con 100 µl di acido solforico 1.8 N, ed è stata effettuata la lettura a 450/650 nm mediante spettrofotometro.
3.7 Misurazione livelli di PGE2
La misurazione dei livelli di PGE2 è stata effettuata mediante il saggio ELISA, Prostaglandin E2 EIA KIT ( Cayman, USA). Circa 5000 cellule per pozzetto sono state seminate in piastre da 96 wells ed incubate overnight per permetterne l’adesione. A fine trattamento, un’aliquota di surnatante è stata prelevata e trasferita nella piastra contenente l’anticorpo primario e successivamente lasciata ad incubare overnight a 4 °C. Dopo aver effettuato una serie di lavaggi è stato addizionato l’anticorpo secondario. In seguito ad un ulteriore periodo di incubazione di circa 90 minuti si assiste ad una variazione colorimetrica delle soluzioni, indice di avvenuta reazione. La misurazione dei livelli di prostaglandina è stata effettuata mediante lettura spettrofotometrica a 405 nn.
3.8 Analisi del ciclo cellulare
Le cellule sono state seminate in dischi Petri da 100 mm alla densità di 105
cellule/ml e trattate con una soluzione composta da salbutamolo e rosiglitazone alla concentrazione di 0.5 µM rapporto 1:1. Terminato il trattamento, le cellule sono state lavate 2 volte con PBS ed il DNA è stato marcato con propidio ioduro (25 µg/mL), RNasi (1 mg/mL) e Nonidet-P40 (0.1%). I campioni sono stati posti in ghiaccio per 30 minuti ed analizzati in citofluorimetria mediante un FACScan (Becton-Dickinson, San José, CA, USA). I dati sono stati elaborati con i software CELLQuest e ModWt (Verity Software, Topsham, ME, USA).
3.9 Analisi dei dati
I risultati sono stati presentati come media ± SEM di esperimenti indipendenti effettuati in triplicato. I dati sono stati analizzati mediante l’utilizzo del software Prism (Graphpad Software, San Diego, CA, USA). L’analisi statistica per le differenze tra le medie è stata condotta mediante il metodo dell’analisi della varianza (t Student per dati appaiati e ANOVA). Valori di p inferiori a 0,05 sono stati considerati statisticamente significativi.