3. RISULTATI
3.1. Diversità genetica all’interno dei campioni
L’analisi per valutare la struttura genetica di A. antennatus è stata effettuata su sequenze delle regioni parziali di DNA mitocondriale della subunità I della citocromo ossidasi (514 bp) della subunità 16S dell’RNA ribosomiale (547 bp), ricavate da 141 individui di Aristeus antennatus provenienti dalle tre località considerate. Dall’allineamento delle sequenze nucleotidiche sono risultati 21 aplotipi per il 16S e 26 aplotipi per la COI. Per quanto riguarda i siti polimorfici rappresentano complessivamente il 4% per il 16S ed il 5% per la COI. In linea generale, risulta una diversità aplotipica medio-alta e valori di diversità nucleotidica bassi (Tab. 3.1, Fig. 3.1).
Tabella 3.1. Aristeus antennatus. Stime di diversità genetica:. n: taglia del campione; Nh: numero di aplotipi; Np:numero di siti polimorfici; h: diversità aplotipica; π: diversità nucleotidica.
Località gene n Nh Np h π Calella 16S 60 10 10 0.359 ±0.079 0.0008 ±0.0002 COI 59 13 18 0.395 ±0.082 0.0016 ±0.0004 16S+COI 59 21 28 0.589 ±0.078 0.0012 ±0.0003 Genova 16S 42 8 7 0.570 ±0.073 0.0013 ±0.0002 COI 44 12 16 0.589 ±0.085 0.0038 ±0.0008 16S+COI 42 17 23 0.724 ±0.075 0.0026 ±0.0004 Palermo 16S 39 8 8 0.410 ±0.099 0.0010 ±0.0003 COI 37 5 8 0.338 ±0.096 0.0016 ±0.0006 16S+COI 36 10 15 0.552 ±0.097 0.0013 ±0.0004 Totale 16S 141 21 22 0.445 ±±±±0.051 0.0010 ±±±±0.0001 COI 140 26 26 0.447 ±±±±0.054 0.0023 ±±±±0.0004 16S+COI 137 44 47 0.624 ±±±±0.050 0.0017 ±±±±0.0002
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9
Calella Genova Palermo
h
16s COI 16S+COI
Fig. 3.1. Istogramma della diversità aplotipica (h) di A. antennatus tra le località considerate.
La conversione delle sequenze nucleotidiche della regione COI in sequenze ammioacidiche, utile per verificare la presenza di pseudogeni, ha rivelato solo una mutazione non sinonima in un individuo di Genova, cioè che determina la sostituzione di un amminoacido con un altro, in posizione 130 (Fig. 3.2). Una Valina (Val, V) è stata sostituita da una Isoleucina (Ile, I), amminoacido comunque dalle caratteristiche fisico- chimiche simili.
Tab. 3.2. Sequenza amminoacidica della regione parziale della COI, in colore rosso l’amminoacido sostituito. 10 20 30 40 50 60 PVLYQHLFWFFGHPEVYILILPAFGMISHIISQESGKKEAFGTLGMIYAMLAIGVLGFV 70 80 90 100 110 VWAHHMFTVGMDVDTRAYFTSATMIIAVPTGIKIFSWLGTLHGTQLNYSPSLLWAL 120 130 140 150 160 170 GFVFLFTVGGLTGVVLANSSIDIILHDTYYVVAHFHYVLSMGAVFGIFAGIAHWFP I
3.2. Diversità genetica tra i campioni
3.2.1. F- statisticaI valori di FST ottenuti per i campioni di Aristeus antennatus hanno un intervallo
che varia da -0.00188 a 0.07238 e sono riportati in Tab. 3.2. Il valore più alto è stato riscontrato tra i campioni di Genova e quelli di Calella per il gene 16SrRNA. Il valore negativo riscontrato tra i campioni di Palermo e Calella per il gene 16SrRNA è da considerarsi uguale a 0 e deriva dalle correzioni apportate all’indice originario di Wright (1921, 1931) da Weir e Cockerham (1984) per rendere la stima indipendente dalle dimensioni del campione.
Tab. 3.3. Valori di Fst a coppie calcolati per le tre località campionate per il gene 16SrRNA in
A.antennatus
Calella Genova Palermo
Calella - - -
Genova 0.072** - -
Palermo -0.001*** 0.057** -
ns non significativo, * P<0.05, ** P<0.01, *** P<0.001
Tab. 3.4.Valori di Fst a coppie calcolati per le tre località campionate per il gene COI in A.antennatus
Calella Genova Palermo
Calella - - -
Genova 0.040* - -
Palermo 0.005ns 0.025ns -
Tab. 3.5. Valori di Fst a coppie calcolati per le tre località campionate per il gene COI+16S in
A.antennatus
Calella Genova Palermo
Calella - - -
Genova 0.053** - -
Palermo 0.002*** 0.035* -
3.2.2. Distanza genetica
I valori di distanza genetica sono stati calcolati, a partire dalla regione formata dalle due sequenze unite (COI+16S), secondo il metodo di Tamura & Nei (1993) sia tra gli individui all’interno dei campioni sia tra i campioni. A livello inte-individuale il valore più alto è stato registrato tra gli individui di Genova con un valore doppio rispetto agli altri due campioni presi in esame (Tab. 3.6).
Tab.3.6. Valori di distanza genetica di Tamura e Nei (1993) tra individui di A.antennatus all’interno dei campioni. L’errore standard è stato calcolato utilizzando “bootstrapping” con 1000 pseudorepliche.
D Errore standard
Calella 0.0012 0.0003
Genova 0.0026 0.0007
Palermo 0.0013 0.0004
Per quanto riguarda la distanza genetica tra i tre campioni, i valori ottenuti sono dello stesso ordine di grandezza di quelli precedentemente visti a livello inter- individuale. In particolare i valori tra i campioni Genova-Calella e Genova-Palermo risultano essere uguali e maggiori rispetto alla distanza genetica ottenuta tra Palermo e Calella (Tab.3.2).
Tab 3.7. Valori della distanza genetica di Tamura e Nei (1993) tra i campioni di A.antennatus. (al di sotto della diagonale). L’errore standard (al di sopra della diagonale) è stato calcolato utilizzando “bootstrapping” con 1000 pseudorepliche.
Calella Genova Palermo
Calella - 0.0005 0.0003
Genova 0.0020 - 0.0005
3.2.3 Analisi della varianza molecolare (AMOVA)
L’AMOVA è stata eseguita considerando solamente due livelli: 1) varianza tra individui all’interno dei campioni e 2) varianza tra campioni. Per entrambi i geni e per il caso dei geni uniti la varianza è risultata risiedere prevalentemente a livello inter-individuale con valori percentuali piuttosto alti che variano tra il 95.5% e 93.7%. L’AMOVA ha fornito anche il valore della Φ-statistica, analoga all’F-statistica, quindi indice di eterogeneità genetica all’interno dei vari livelli gerarchici. I valori di Φ-statistica sono risultati significativi al test di permutazione con 1000 repliche (Tab. 3.8, 3.9, 3.10)
Tab. 3.8. Risultati dell’analisi della varianza molecolare (AMOVA) in Aristeus antennatus per la regione COI. (Excoffier et al., 1992).
Tab 3.9. Risultati dell’analisi della varianza molecolare (AMOVA) in Aristeus antennatus per la regione 16S (Excoffier et al., 1992).
Fonte della variaza gl Componente della varianza Percentuale della Varianza Φ-statistica P Tra campioni 2 0.01617 2.7 Tra individui all’interno
del campione 137 0.57872 97.3
ΦST = 0.027 0.011
Fonte della varianza gl Componente della varianza
Percentuale della varianza
Φ-statistica P
Tra campioni 2 0.012820 4.5 Tra individui all’interno
del campione
138 0.27192 95.5
Tab. 3.10. Risultati dell’analisi della varianza molecolare (AMOVA) in Aristeus antennatus per la regione costituita dalle due sequenze unite (COI+16S) (Excoffier et al., 1992)
Fonte della varianza gl Componente della varianza
Percentuale della varianza
Φ-statistica P
Tra campioni 2 0.03058 3.5 Tra individui all’interno
del campione
134 0.85499 96.5
3.2.4. Network
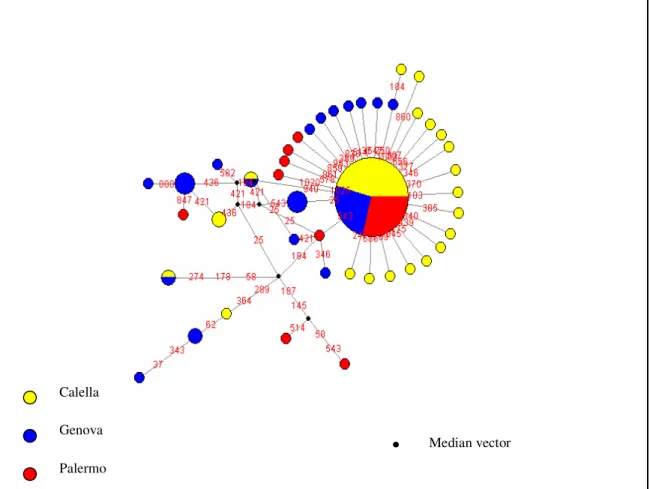
I tre network elaborati con la tecnica del “median-joining” non sono risultati completamente concordanti. Quello del 16S è caratterizzato dalla struttura “a manubrio” (Avise, 2000), costituito cioè da due aplotipi comuni ritrovati in tutti e tre i campioni che sono diversi per una sola mutazione; a questi aplotipi ne sono legati altri a frequenza bassa (1 e 2) separati dall’aplotipo comune da una o, al massimo, due mutazioni (Fig. 3.2). Il network ottenuto per la COI, invece mostra una struttura più complessa con un aplotipo nettamente più frequente rispetto agli altri (Fig. 3.3). La caratteristica comune ai due network è la presenza di “star phylogeny”, cioè un aplotipo comune molto frequente da cui si separano per una o poche mutazioni aplotipi poco frequenti. Nel network ottenuto con le due regioni mitocondriali unite, viene mantenuta la complessità rilevata con la COI, inoltre la presenza di una star phylogeny è accentuata rispetto ai network delle singole regioni (Fig. 3.4.). Da rilevare che in tutti e tre i network, l’aplotipo comune è costituito da una proporzione pressochè costante di individui appartenenti ai tre diversi campioni. Dai network non emergono separazioni di aplotipi che abbiano un significato geografico, con gli individui di tutte e tre le località rappresentati in tutte le parti del network (Figg. 3.2, 3.3, 3.4).
Fig 3.2. Network degli aplotipi del gene 16SrRNA rilevati in 141 individui di A. antennatus ottenuto con la tecnica del median-joining
Calella Genova Palermo
Fig 3.3. Network degli aplotipi del gene COI rilevati in 141 individui di A. antennatus ottenuto con la tecnica del median-joining
Palermo
Median vector Genova
Fig 3.4. Network degli aplotipi del gene COI+16S rilevati in 141 individui di A. antennatus ottenuto con la tecnica del median-joining
Calella Genova Palermo
3.2.5. Stime di flusso genico
La stima di flusso genico calcolata per A. antennatus, per tutti e tre i modelli presi in considerazione, risulta maggiore di 4, ritenuto valore al di sopra del quale viene considerata una condizione di panmissia (Tab. 3.11).
Tab 3.11. Stime di flusso genico in A.antennatus
Nei 1982
γst: 0.037
Nm: 12.9Lynch and Crease 1990 Nst: 0.033 Nm: 14.8
3.2.6. Isolamento da distanza
Per verificare la presenza di “isolamento da distanza” è stata effettuata l’analisi di regressione tra le distanze nautiche e i valori a coppia di FST (Tab. 3.12). È stato
inoltre applicato il test di Mantel alle matrici di distanza genetica (FST) e di distanza
nautica (in miglia nautiche) (Fig. 3.5)
Tab. 3.12. Sopra la diagonale: distanze nautiche (in miglia marine); sotto la diagonale: valori di FST per
ciascuna coppia dei 3 campioni di A. antennatus analizzati
Calella Genova Palermo
Calella - 299 512
Genova 0,010 - 418
Palermo -0,001 0,019 -
Il test di Mantel ha dato un valore di Z = 10.797 con un valore associato di probabilità pari a P = 0.665. La non significatività del test ha consentito di accettare l’ipotesi zero di indipendenza delle due matrici, cioè di escludere la presenza di isolamento da distanza.
3
.3 Aspetti di demografia storica
La distribuzione “mismatch” ha fornito grafici con curve finalizzate all’analisi degli aspetti demografici; in particolare quello dell’espansione demografica recente. Per il gene 16S le curve ottenute presentano un andamento che coincide quasi perfettamente alla curva attesa, prevista per una recente espansione demografica (Fig. 3.6). Nei casi della COI e delle sequenze unite COI+16S la distribuzione ha fornito una curva che si distacca leggermente, solo per pochi punti, dalla curva attesa (Fig. 3.7, 3.8). Le deviazioni dal modello dell’espansione demografica recente risultano significative soltanto in due casi: la curva relativa alla COI nel campione di Genova e quella relativa alle due sequenze unite nel campione di Calella (Tab. 3.13). Complessivamente, il totale dei campioni risulta seguire il modello dell’espansione recente.
Il test R2di Ramos-Onsins & Rozas (2002) è risultato significativo (P < 0.05) in tutti i
casi presi in considerazione, ad esclusione del campione di Calella per il gene 16S, dove però il valore di probabilità (P = 0.070) è di poco superiore al livello di significatività. I test dell’FS
di Fu (1997) e della D di Tajima (1998) hanno fornito significatività in tutti i casi (Tab. 3.13).
Tab. 3.13. Risultati dei test: SSD, D di Tajima, FS di Fu e R2 di Ramos-Onsins & Rozas. La significatività dei
valori è stata ottenuta con 10000 permutazioni
Località mt region SSD P D P FS P R2 P Calella 16S 0.001 0.860 -2.218 0.000 -10.623 0.000 0.041 0.070 COI 0.007 0.522 -2.434 0.000 -11.369 0.000 0.034 0.008 16S+COI 0.170 0.001 -2.568 0.000 -28.727 0.000 0.025 0.000 Genova 16S 0.009 0.175 -1.554 0.037 -4.890 0.000 0.058 0.052 COI 0.445 0.001 -1.510 0.042 -4.319 0.000 0.059 0.045 16S+COI 0.046 0.341 -1.644 0.039 -26.506 0.000 0.052 0.014 Palermo 16S 0.001 0.939 -1.934 0.012 -6.251 0.000 0.055 0.034 COI 0.023 0.243 -1.659 0.023 -0.937 0.000 0.061 0.040 16S+COI 0.013 0.368 -2.054 0.002 -28.189 0.000 0.051 0.000 Total 16S 0.004 0.658 -1.934 0.017 -29.291 0.000 0.019 0.024 COI 0.158 0.255 -1.868 0.022 -26.842 0.000 0.023 0.007 16S+COI 0.076 0.237 -2.089 0.014 -27.807 0.000 0.017 0.000