3.3.2 Strumenti
monolitici
per elettroforesi capillare
con rivelatore di fluorescenza integrato
La maggior parte dei microstrumenti per elettroforesi capillare sono stati fabbricati su substrati di vetro, usando fotomoltiplicatori posizionati fuori dal chip per la rivelazione di fluorescenza indotta da laser
(tecnica LIF, “Laser-Induced Flourescence”). I vantaggi della
miniaturizzazione sono quindi ridotti in quanto questi sistemi necessitano di apparecchiature di alcuni ordini di grandezza più grandi per la rivelazione [35].
L’uso di tecniche di rivelazione su chip può rendere realizzabili strumenti di diagnostica del DNA portatili. Una strada per realizzare questo obiettivo è quella di usare tecniche di rivelazione nano-ottiche.
Mentre gli schemi di rivelazione elettrochimica sono ancora sotto sviluppo14, quelli di fluorescenza sono molto attraenti, in quanto molti degli attuali protocolli biochimici già includono marcatori fluorescenti (per esempio, come visto precedentemente, il protocollo di sequenziamento con il metodo di Sanger). Quindi esperimenti realizzati su chip possono essere inseriti in protocolli già esistenti senza cambiamenti da un punto di vista biochimico e la rivelazione per fluorescenza può essere usata per analisi su chip. Inoltre sistemi più complessi per analisi del DNA richiedono piattaforme versatili che dovrebbero contenere strumenti come scaldatori, sensori di temperatura, capillari, camere di reazione, sensori di rivelazione del DNA e valvole.
14Per approfondire tale argomento:
• A.T. Wolley, K. Lao, A.N. Glazer & Richard A. Mathies, “Capillary Electrophoresis Chips with Integrated Electrochemical Detection”, Anal.Chem., 1998, Vol. 70, pp 684-688. • R.S. Martin, A.J. Gawron & S.M.Lunte, “Dual-Electrode Electrochemical Detection for
Poly(dimethysiloxane)-Fabricated Capillary Electrophoresis Microchips”, Anal.Chem., 2000, Vol. 72, pp 3196-3202.
Gli strumenti microfabbricati con superficie in materiale plastico sono indicati per queste applicazioni, anche a causa del loro basso costo di fabbricazione e del loro alto potenziale di integrazione [36].
Questi strumenti sono costruiti a partire dalla formazione di capillari per elettroforesi con pareti sottili di “Parylene C” su un substrato piano. Gli strumenti basati sul parylene possono essere integrati con scaldatori, rivelatori, valvole e altri componenti microfluidici. Inoltre i sistemi di parylene sono biocompatibili e hanno una fluorescenza di fondo molto bassa che li rende adatti ad analizzare il DNA.
Solitamente come matrice di setaccio per analisi del DNA viene usato l’acrilamide in quanto offre una alta risoluzione di separazione. A differenza del vetro, i polimeri come il parylene, a causa della sua struttura molecolare, hanno una alta permeabilità alle molecole di ossigeno (circa 7.5 x 10-14 kg/m s Pa, da paragonarsi alla permeabilità del vetro di circa 7.5 x 10-20 kg/m s Pa). Ciò pone un problema in quanto l’acrilamide (e gel simili) in presenza di ossigeno polimerizzano. Per evitare tale inconveniente, è necessario creare una barriera di rivestimento per la parete interna del canale di elettroforesi che isoli il parylene dal gel di separazione. Il materiale di barriera solitamente scelto è l’allumina (Al2O3) in quanto soddisfa requisiti quali trasparenza, bassa fluorescenza di fondo, alta resistività, biocompatibilità e buona adesione ai polimeri.
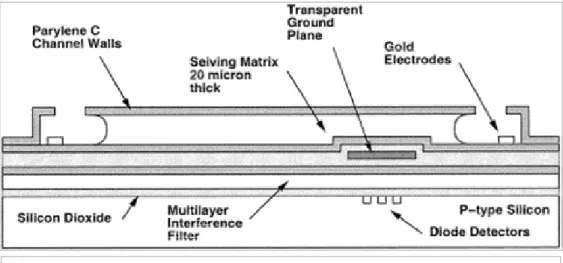
In figura 3.30 è presentato lo schema della sezione trasversale di uno strumento monolitico per elettroforesi capillare con rivelatore di fluorescenza integrato realizzato con il metodo di surface
micromachining [37].
Lo strumento fondamentalmente consiste di fotodiodi, che rilevano i segnali di fluorescenza, realizzati su un substrato di silicio, di un filtro di interferenza ottica, che impedisce all’eccitazione della luce di inibire la rivelazione della fluorescenza, di un “ground plane” trasparente, del canale
per elettroforesi, realizzato con un sottile film plastico di parylene C, e di elettrodi.
Il successo dell’integrazione di strumenti microfluidici ottici ed elettronici necessita di alcuni strati di isolamento [38].
L’isolamento chimico è necessario per impedire che gli ioni presenti nella soluzione di separazione raggiungano i rivelatori, che sono sensibili agli ioni. L’isolamento chimico è fornito dalla parete inferiore del canale di elettroforesi che ha uno spessore di 5 µm.
Inoltre, i fotodiodi devono essere dielettricamente isolati dalle alte tensioni necessarie durante la fase di separazione per evitare interferenze con la loro risposta. Tale isolamento è anche necessario perché le alte tensioni possono causare inversioni nel substrato di silicio vicino al fotodiodo causando alti livelli di rumore. Questo isolamento è fornito in parte dallo spessore di 8 µm costituito della parete di parylene e del filtro ottico. Principalmente però l’isolamento dagli alti campi elettrici è ottenuto usando un “ground plane”; esso deve essere trasparente in quanto deve permettere la trasmissione del segnale ottico fluorescente.
Figura 3.30: schema della sezione trasversale dello strumento per elettroforesi. Le componenti più rilevanti di tale strumento sono i fotodiodi, il filtro di interferenza
Il “ground plane” trasparente è realizzato usando un sottile strato di AZO (“Aluminum Zinc Oxide”)15, posizionato tra la parte superiore del filtro ottico e la parte inferiore del canale.
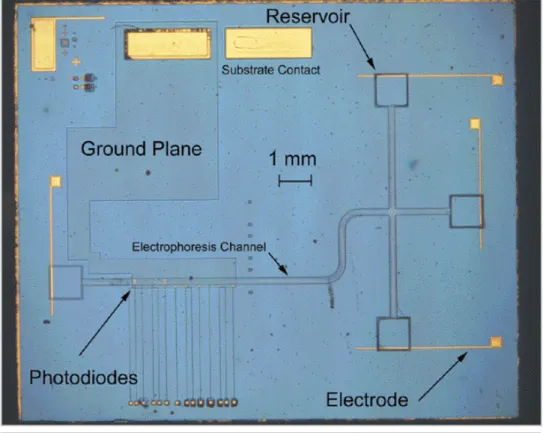
La figura 3.31 mostra lo strumento per elettroforesi microfabbricato con rivelatore di fluorescenza su chip. Il canale è largo 200 µm e la sua lunghezza totale è di circa 1.3 cm.
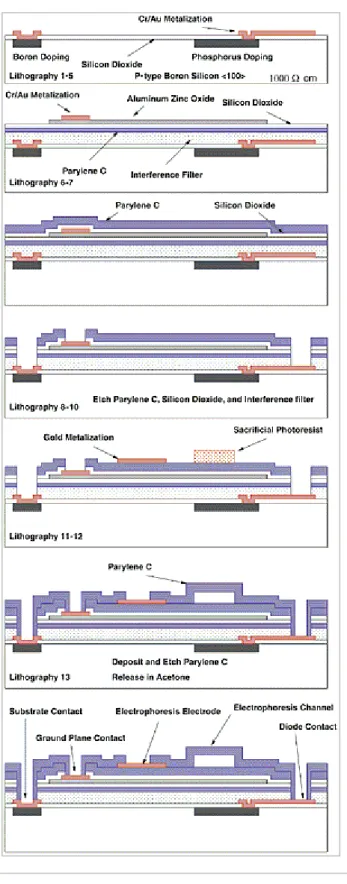
Il processo di fabbricazione dello strumento per elettroforesi è costituito da 16 passi litografici, tre dei quali sono necessari per il rivestimento interno del canale di parylene C con la barriera di allumina.
15 L’ossido di zinco (ZnO) è un materiale usato come strato conduttore trasparente per le sue
buone proprietà ottiche ed elettriche. La sua conduttività elettrica è controllata da vacanze di ossigeno e/o zinco interstiziale, che agisce da donatore di tipo n. La sua resistività è ulteriormente abbassata dopando con elementi come alluminio e boro. Gli strati di ZnO dopati con alluminio (AZO) hanno quindi alta trasparenza nel campo del visibile e bassa resistività e sono adatti per
Figura 3.31: “micrograph” ottico dello strumento per elettroforesi. Su un singolo chip sono integrati: fotodiodi, filtro di interferenza, “ground plane” trasparente,
In figura 3.32 è mostrato il flusso di processo semplificato usato per la costruzione dello strumento.
Il wafer di partenza è un substrato di silicio con resistività di 1000 Ω cm, drogato con boro, di tipo p e con orientazione <100>.
Per prima cosa sui wafer viene fatto un impianto di ioni boro (dose 1 x 1012 cm-2) a 8.02 x 10-15 J, per prevenire l’inversione del substrato sotto le metallizzazioni vicino alla superficie. Nella fase iniziale del processo, nel substrato di silicio viene costruito il fotodiodo usando due passi successivi di impiantazione ionica (impiantazione di tipo n di ioni fosforo a 8.02 x 10-15 J, a una dose di 5 x 1014 cm-2, e impiantazione di tipo p per il contatto di substrato di ioni boro a 4.8 x 10-15 J, a una dose di 1 x 1015 cm-2) realizzati usando due diverse maschere di fotoresist. Dopo aver tolto le maschere usate per l’impiantazione, viene fatto crescere un ossido di passivazione termico di 0.2 µm a 900°C, necessario per isolare il substrato, e quindi sono realizzati i contatti con i fotodiodi, deponendo per evaporazione in una camera a vuoto prima cromo (0.05 µm) e poi oro (0.2 µm) sul substrato.
Le metallizzazioni Cr/Au sono quindi modellate usando una maschera di fotoresist e un bagno corrosivo per il metallo.
Quindi viene deposto sulla sommità del wafer il filtro di interferenza; esso consiste di circa 20 strati alternati di SiO2 e TiO2, per uno spessore totale di circa 3 µm. Sopra il filtro viene deposto uno strato di 2.5 µm di parylene C, che serve come strato di passivazione per il filtro, per mantenere l’integrità delle proprietà ottiche. Dopo aver deposto per
sputtering un sottile strato di SiO2 (0.18 µm), viene deposto, sempre per sputtering, lo strato di AZO di 0.15 µm che, successivamente, viene opportunamente modellato. Il contatto con lo strato AZO viene realizzato deponendo uno strato di Cr/Au (0.02 µm/0.08 µm) con la tecnica liftoff.
Figura 3.32: flusso di processo semplificato per la fabbricazione dello strumento per elettroforesi. Non sono indicati i 3 passi necessari per il
posizionamento della barriera di allumina all’interno del canale di parylene. [37]
Un secondo strato di SiO2 è deposto per sputtering sopra l’AZO; esso serve sia per passivare l’AZO sia per fornire una eccellente adesione allo strato di parylene C che gli viene deposto sopra. Questo strato di parylene è spesso 4.8 µm ed è la parte inferiore del canale per elettroforesi.
Successivamente sono realizzati i fori di contatto incidendo gli strati di parylene (tramite Reactive Ion Etching con plasma di O2) e SiO2 (attacco con soluzione di BHF), in modo da avere accesso alla metallizzazione del fotodiodo e quella dell’AZO.
Quindi sono modellati sul parylene gli elettrodi in oro (spessore 0.1 µm) per l’elettroforesi con la tecnica liftoff ed è deposto il fotoresist sacrificale. Infine la parte superiore del canale è formata deponendo uno strato di parylene di 5.3 µm e quindi opportunamente sagomato con un attacco RIE con plasma di O2 usando un fotoresist spesso. In quest’ultimo passo sono anche realizzati i serbatoi per l’elettroforesi.
La fase di preparazione alla separazione elettroforetica consiste nel caricamento della matrice setaccio e nel riempimento dei serbatoi e del canale di separazione con una soluzione buffer (operazioni standard in ogni separazione elettroforetica). Un campione di DNA viene quindi marcato fluorescentemente con “SYBR Green I”, intercalando un marcatore ogni cinque coppie di basi di DNA (più la catena di DNA è lunga e più marcatori fluorescenti contiene). Circa 0.2 µl di DNA campione sono sufficienti per l’analisi e sono caricati nel serbatoio “Sample”. In questo tipo di strumento, il campo elettrico applicato durante la separazione è di 110 V/cm.
In figura 3.33, è rappresentato uno schema di rivelazione di fluorescenza su chip. In tale schema un amplificatore stabilizzato a chopper viene usato per misurare la corrente del fotodiodo, mentre un LED blu viene utilizzato come sistema di eccitazione con un filtro passa banda a 450 ± 27 nm. I dati sono campionati a una frequenza di 64 Hz e registrati su PC per essere elaborati. Le separazioni nel canale elettroforetico possono
inoltre essere osservate con opportuni microscopi e registrate su nastro video (in fig. 3.35 un esempio di immagine video).
L’efficienza quantica relativa del fotodiodo, combinata all’azione del filtro di interferenza sul chip, è mostrata in figura 3.34.
Si osserva che il filtro blocca la luce di eccitazione fino a circa
495 nm; al di sotto di tale
lunghezza d’onda, il fotodiodo non emette fotoelettricamente elettroni. L’efficienza massima del fotodiodo si ha nello spettro del rosso.
Lo strumento analizzato è stato usato per effettuare separazioni elettroforetiche di frammenti di DNA di diverse lunghezze, contenenti un quantitativo di marcatori proporzionale al numero di coppie di basi (bp) presenti nel frammento. L’effettiva distanza dall’iniettore al rivelatore è di 0.9 cm.
Figura 3.33: sistema di rivelazione di fluorescenza. [37]
Figura 3.34: efficienza quantica relativa del fotodiodo con il filtro di interferenza
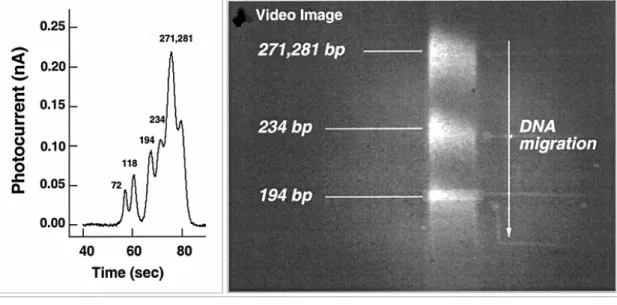
L’elettroferogramma e l’immagine video di figura. 3.35 mostrano i risultati della separazione avvenuta: si osserva che DNA di dimensioni maggiori, più luminosi, generano nel fotodiodo correnti maggiori.
In questo tipo di sistema è problematico raggiungere alte risoluzioni di separazione. Infatti, in realtà, il rivelatore ha un ha una superficie di rilevazione più grande del fotodiodo a causa della diffusione dei portatori; per questo motivo il rilevatore raccoglie la fluorescenza di più molecole di DNA contemporaneamente, rendendo difficile la loro distinzione. Tale problematica è accentuata dal fatto che il LED fornisce una eccitazione di area larga e che la fluorescenza emessa dalle molecole di DNA è isotropica. La combinazione di questi eventi rende la fluorescenza visibile a 200 µm dal rivelatore causando, nel caso di molecole di DNA di lunghezza simile, fenomeni di sovrapposizione di fluorescenza (con conseguenti picchi nella corrente rilevata). Una possibile soluzione al problema può essere quella di usare sorgenti con piccole aree di eccitazione (come per esempio laser) e realizzare rilevatori di piccole dimensioni. Tali soluzioni sono attualmente in studio.
Figura 3.35: esempio di separazione elettroforetica effettuata nello strumento per elettroforesi capillare integrato. A sinistra elettroferogramma realizzato usando il rivelatore
3.4 Progetto di “Lab On Chip” monolitico su
silicio in grado di realizzare amplificazione e
analisi di campioni di DNA
La PCR ha rivoluzionato la biologia molecolare ed è al centro di molti sforzi attuali per documentare e capire le diversità genetiche umane.
La PCR tipicamente richiede un ciclo termico per sottoporre gli stampi di DNA in modo sequenziale a denaturazione (a temperatura di 94ºC), annealing dei primer (in un range di temperatura variabile fra 50 e 60 ºC, a seconda della sequenza di primer usati) e passi di estensione (a 72ºC)(argomento già trattato nel paragrafo 2.5).
Sebbene siano stati descritti una grande varietà di sistemi in grado di eseguire cicli termici per l’amplificazione dei campioni con PCR, la maggior parte delle apparecchiature commerciali di ciclo termico mettono insieme un relativamente massiccio blocco di metallo combinato o con array di tubi di plastica o con micropiani multistrato, realizzando singole reazioni di PCR [40, 41]. La grande massa termica di questi strumenti limita le velocità massime di transizione della temperatura ottenibili, con un significativo incremento del tempo richiesto per completare la PCR ciclica e inoltre si ha una accelerazione delle reazioni collaterali parassite che possono compromettere il corretto svolgimento del processo di PCR.
Negli ultimi anni la domanda crescente per più alte efficienze di sistemi che realizzano la PCR ha condotto a pressioni non solo per avere più alte velocità di transizione delle temperature, ma anche per la riduzione del volume di reazione e l’integrazione con altre operazioni necessarie per l’analisi del DNA. È quindi iniziata la sperimentazione sia per applicare
materiali ancora poco utilizzati in ambito biomedico sia per la realizzazione di dispositivi microfluidici che integrino più funzionalità contemporaneamente. Le proprietà fisiche del silicio rendono tale materiale ideale per applicazioni microfluidiche, in quanto il silicio ha buona conduttività termica, che consente il mantenimento di una temperatura uniforme su aree molto grandi, e bassa capacità termica, che permette la realizzazione di cicli termici molto rapidi (proprietà indispensabile per la realizzazione di processi di amplificazione come la PCR). Inoltre il silicio permette l’integrazione di controlli elettronici sullo stesso dispositivo.
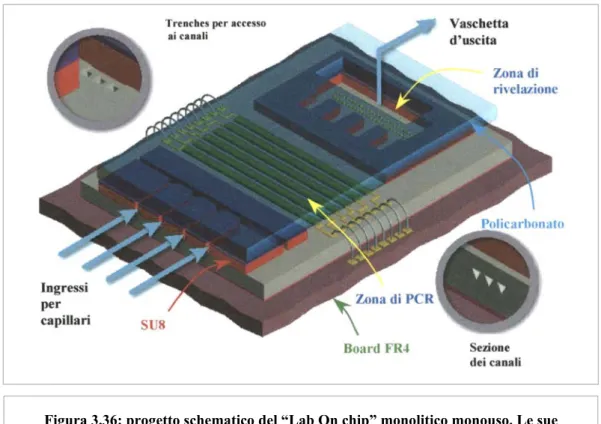
Il chip monolitico di figura 3.36 in silicio è stato progettato per svolgere fasi di analisi del DNA quali l’amplificazione, mediante PCR, e la rivelazione dei campioni, usando la tecnica della ibridazione [42, 43].
L’amplificazione è realizzata “ciclando” una miscela di reazione (che contiene il campione di DNA da amplificare, i primer, i quattro dNTP e l’enzima DNA polimerase) attraverso i tre passi di temperatura (ciclo
Figura 3.36: progetto schematico del “Lab On chip” monolitico monouso. Le sue dimensioni sono indicativamente di 24.3 mm x 14 mm. [42]
termico) visti precedentemente. Le temperature di 94ºC e 72ºC sono fisse, mentre la temperatura di annealing è variabile e dipende dalla sequenza dei primer. Una tipica reazione di PCR è composta di 20-40 cicli termici, in modo da preparare un quantitativo sufficiente di DNA per l’analisi mediante ibridazione.
L’idea di base è quella di creare canali in cui la miscela di reazione possa scorrere lentamente e, durante l’avanzamento del liquido, operare i vari cicli di PCR; fra l’ingresso e la zona di rivelazione nei canali vengono quindi create più zone con differenti temperature per consentire più cicli termici. Inoltre deve essere previsto un ultimo ciclo prima della rivelazione in cui il DNA da analizzare (già amplificato) sia denaturato (l’analisi avviene per ibridazione e quindi il DNA da analizzare deve essere a singola catena, cioè “ssDNA”).
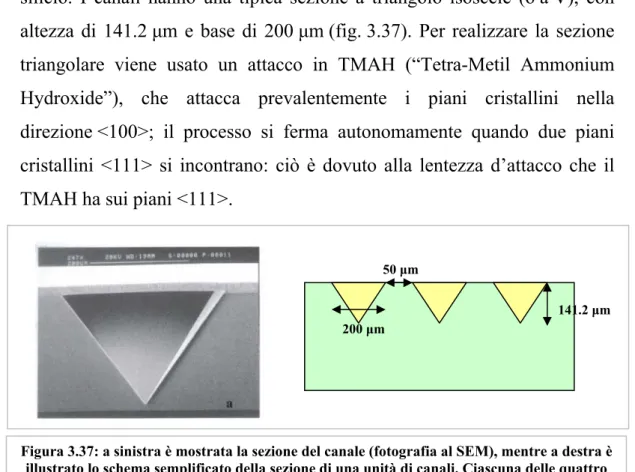
In questo microsistema la PCR avviene in canali sepolti nel wafer di silicio. I canali hanno una tipica sezione a triangolo isoscele (o a V), con altezza di 141.2 µm e base di 200 µm (fig. 3.37). Per realizzare la sezione triangolare viene usato un attacco in TMAH (“Tetra-Metil Ammonium Hydroxide”), che attacca prevalentemente i piani cristallini nella direzione <100>; il processo si ferma autonomamente quando due piani cristallini <111> si incontrano: ciò è dovuto alla lentezza d’attacco che il TMAH ha sui piani <111>.
200 µm
141.2 µm 50 µm
Figura 3.37: a sinistra è mostrata la sezione del canale (fotografia al SEM), mentre a destra è illustrato lo schema semplificato della sezione di una unità di canali. Ciascuna delle quattro
Vengono realizzate quattro unità, ciascuna delle quali contiene tre canali (fig. 3.37). I canali nelle unità sono distanti 50 µm, mentre i gruppi sono distanti fra loro 1 mm e sono lunghi 12 mm. Tale realizzazione permette di ottenere una distribuzione uniforme del liquido di reazione nei canali e un buon collegamento con i capillari esterni.
Sono inoltre stati studiati differenti trattamenti per la superficie interna dei canali. Ciò è stato necessario in quanto sul silicio non trattato i prodotti della PCR scorrono lentamente e sono di bassa qualità. Dopo la passivazione del silicio con 1 µm di ossido termico, i prodotti e la qualità sono stati considerati buoni. Sono state provate anche altre passivazioni, come per esempio la deposizione di parylene C o di nitruro di silicio, che hanno fornito buoni risultati di PCR e riproducibilità, ma si sono dimostrati incompatibili con la parte restante del processo di fabbricazione. Di conseguenza, l’ossido termico sulle pareti del canale è il miglior compromesso tra vincoli biologici e tecnologici.
La creazione di canali sepolti permette di lasciare la superficie del wafer libera per altri processi tecnologici come disegno fotolitografico di resistori di polisilicio (necessari per creare le zone a differenti temperature), connessioni elettriche (in questo caso di alluminio), fabbricazione degli elettrodi destinati a contenere le sonde per l’ibridazione e deposizione di resine.
L’obiettivo è quello di scaldare e misurare la temperatura direttamente sopra la zona dei canali e quindi dei resistori riscaldatori sono posizionati sopra i canali. La figura 3.38 mostra il progetto del sistema di riscaldamento realizzato su un chip di prova (detto “Chip Termico”). Su questo chip gli elementi riscaldanti in polisilicio sono stati ottimizzati per soddisfare le richieste termiche della reazione di PCR con una tensione di ingresso di 12 V. I resistori devono essere posizionati in modo da ottenere sul chip un riscaldamento il più uniforme possibile.
In questa realizzazione vengono anche usati quattro resistori in alluminio come sensori di temperatura: essi controllano che il riscaldamento sul chip sia uniforme. La misurazione di temperatura viene effettuata sfruttando la variazione del valore dei resistori in funzione della temperatura stessa. I resistori hanno infatti una struttura a quattro terminali, in modo che in due di essi venga fatta scorrere una corrente mentre sui restanti due terminali venga letta la tensione. La misura di resistenza è semplicemente data dal rapporto tra il valore della tensione letta ai capi del resistore e quello della corrente forzata. Un circuito esterno di controllo provvede a stabilizzare la temperatura del chip al valore desiderato.
Per la creazione di parte della vaschetta di uscita e degli imbocchi di ingresso dei capillari per il trasporto del liquido biologico che subirà la PCR, viene usato un materiale chiamato SU8 (fig. 3.36). Si tratta di una resina che, oltre a essere economica e biocompatibile, può essere modellata semplicemente mediante processo litografico. Vengono così realizzate
I canali che contengono la miscela di reazione scorrono sotto questa
zona centrale.
Figura 3.38: progetto del sistema di riscaldamento. Le tre colonne di sei elementi ciascuna sono i resistori riscaldatori; i quattro
sensori di temperatura sono posizionati su ciascun lato delle precedenti colonne. [43]
quattro entrate differenti, rendendo possibile l’effettuazione di quattro reazioni PCR indipendenti usando gli stessi cicli di temperatura.
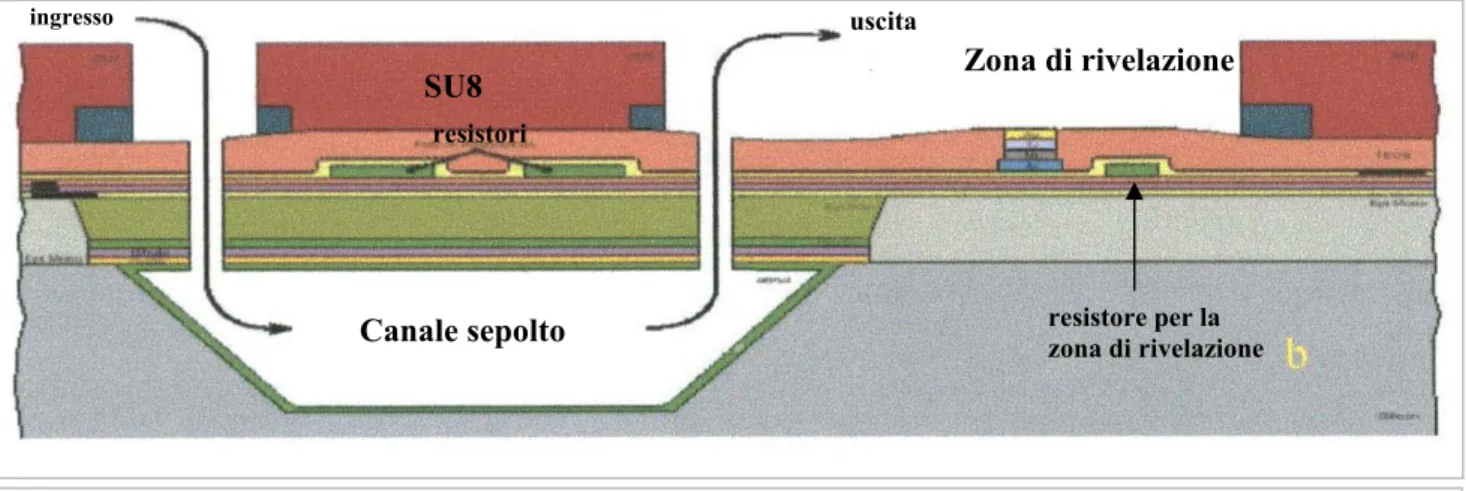
In figura 3.39 è riportata la sezione longitudinale del Lab on Chip. In essa viene indicato il percorso del liquido biologico nel canale sepolto sopra il quale sono posizionati i resistori riscaldatori e sopra di essi l’SU8. L’SU8 inoltre delimita la zona di rivelazione in cui vengono posizionate le sonde per l’ibridazione. Questa struttura viene isolata dall’esterno con una copertura di policarbonato (PC), trasparente e biocompatibile, che viene opportunamente attaccato con delle colle, anch’esse biocompatibili (in figura 3.36 è visibile la copertura di policarbonato).
Per spingere ulteriormente il rendimento del chip, sono state progettate sul prototipo due zone di temperatura indipendenti: una zona che cicla la temperatura sopra i canali per realizzare l’amplificazione (ottenuta con uno schema di resistori simile a quello visto precedentemente) e una seconda zona che ha una temperatura fissa di ibridazione. In questo modo vengono assicurati un flusso continuo di amplificazione e un protocollo di analisi.
La rivelazione del DNA amplificato è realizzata per ibridazione con una catena (sonda) precedentemente immobilizzata sul substrato.
Figura 3.39: schema della sezione longitudinale del prototipo di Lab on Chip. Viene indicato il percorso del liquido biologico dall’ingresso nel dispositivo fino alla zona dove avviene la rivelazione. [42]
Canale sepolto Zona di rivelazione SU8 ingresso uscita resistori resistore per la zona di rivelazione
Successivamente la visualizzazione dei risultati avviene mediante la stimolazione delle sonde con luce ultravioletta.
Le sonde solitamente rappresentano particolari sequenze di nucleotidi che possono rappresentare geni o parti di essi; esse sono collegate a degli elettrodi mediante una molecola di collegamento (linker). Gli elettrodi sono integrati nella zona di rivelazione e sono composti da strati di differenti materiali (alluminio, titanio e oro); la figura 3.40 mostra come essi appaiono in fotografia: l’oro è il materiale più superficiale e quindi è l’unico visibile.
L’inserimento delle sonde di DNA sugli elettrodi è un processo irreversibile ed è realizzato con polimerizzazione elettrochimica di acidi nucleici modificati con pirolo (molecola linker). Applicando una tensione opportuna all’elettrodo rivestito d’oro, i gruppi pirolo
copolimerizzano formando polipirolo, che si attacca all’elettrodo. In figura 3.41 è schematizzata la procedura di aggancio di sonde differenti su due diversi elettrodi. L’operazione avviene in due passi distinti. Inizialmente, l’attivazione dell’elettrodo 1 consente l’aggancio delle sonde desiderate (rosse in figura) esclusivamente su tale elettrodo. Come già visto nel caso dei microarray è indispensabile avere conoscenza del corretto posizionamento delle sonde agganciate per una giusta interpretazione dei
Figura 3.40: fotografia al SEM dei pad nella zona di rivelazione su cui vengono
posizionate le sonde di DNA. Gli elettrodi per l’aggancio delle sonde in
risultati una volta avvenuta l’ibridazione fra la sonda sull’elettrodo e il DNA da analizzare.
La procedura di aggancio delle sonde sull’elettrodo 2 è identica a quella vista precedentemente; l’aggancio delle sonde di tipo 2 non deve alterare l’aggancio precedente. Per verificare la bontà dell’integrazione delle sonde, un liquido “test”, dopo essere stato amplificato mediante PCR ed etichettato, viene trasportato nella zona delle sonde; è stato verificato che effettivamente le sonde reagiscono solo con la catena di DNA a loro complementare.
In figura 3.42 è
Figura 3.41: illustrazione del processo MICAM eseguito per il posizionamento delle sonde sui differenti elettrodi. Il pirolo è indicato con il cerchio mentre le linee curve,
rosse e blu, indicano differenti sonde di DNA. [43]
Elettrodo 1 Elettrodo 2
Elettrodo 1 Elettrodo 2 Pirolo + sonda 1
Pirolo + sonda 2
Figura 3.42: fotografia al SEM che mostra l’avvenuta ibridazione del liquido “test” con le
sonde a lui complementari posizionate sugli elettrodi. [42]
mostrata una parte della zona di rivelazione: la visualizzazione dei risultati avviene stimolando le sonde con luce ultravioletta e l’avvenuta ibridazione è visivamente identificabile (pallino bianco in figura).
Per una corretta realizzazione di uno strumento come quello appena visto, è necessario riuscire a operare una PCR continua (o dinamica), ovvero è necessario riuscire a effettuare i vari cicli di PCR mentre la miscela di reazione avanza nei canali; ciò implica che il liquido, istante per istante, arrivi nella zona di rivelazione con una temperatura che è variabile fra i 54 ºC e i 94 ºC. Contemporaneamente, per realizzare una ibridazione continua, è necessario che, durante tutti i cicli di PCR, tutta la zona dove sono presenti le sonde sia a temperatura costante e stabile (fig. 3.43); solo dopo l’effettuazione dell’ultimo ciclo di PCR, il liquido della vaschetta d’uscita (zona di rivelazione) viene portato a 94 ºC per la denaturazione finale (per circa 2 min) e infine a 54 ºC per l’ibridazione (per circa 1 h).
La riuscita di tali operazioni impone la necessità di avere un isolamento termico fra la zona di PCR e la zona di rivelazione e inoltre è necessario avere un controllo attivo anche della zona di rivelazione (ovvero
Temperatura Tempo 20-30 cicli di PCR 94 ºC 72 ºC 54 ºC Temperatura sonde, 50-60 ºC 15 s 15 s 15 s
Figura 3.43: condizioni per la zona canali e la zona sonde per ottenere PCR e ibridazione continua; con la linea nera è indicata la temperatura nella zona dei canali, mentre con la linea
l’esigenza di avere un resistore nella zona di rivelazione che agisce indipendentemente da quelli della zona dei canali, fig. 3.39). In questo modo vengono assicurati un flusso continuo di amplificazione, mediante PCR, e un protocollo di analisi, mediante ibridazione.
L’isolamento termico deve essere realizzato con estrema accuratezza: è infatti necessario ottenere uno scarto termico fra zona canali e zona sonde anche fino a circa 40 ºC, nello spazio di qualche millimetro. L’obiettivo è di ottenere tale isolamento cercando di aumentare la resistenza termica tra zona canali e zona sonde creando, sul retro del dispositivo, una serie di alette di dispersione del calore (zona “trench”) e introducendo due pozzi di calore, uno sotto la zona canali e l’altro sotto la zona sonde. Lo scopo dei due pozzi di calore è differente: il pozzo di calore posto sotto la zona dei canali, serve a conferire al sistema velocità durante i cicli di PCR e a garantire un flusso termico uniforme dalla superficie del chip verso il suo interno (ovvero verso la zona canali); il secondo pozzo, posizionato sotto la zona di rivelazione, serve a portar via calore proveniente dalla zona canali.
L’alettatura realizzata sul retro del wafer, in pratica, è un microdissipatore di calore integrato su silicio. Infatti la zona alettata aumenta la superficie immersa nel fluido attorno al dispositivo e con cui
Figura 3.44: uno dei quattro scavi realizzati sulla parte posteriore del wafer di silicio necessari per ottenere una buona dissipazione di calore. [42]
esso scambia il calore. Solitamente il fluido è aria che è sottoposta a convezione con conseguente aumento della dissipazione di calore generato dal dispositivo. Gli scavi delle quattro alette necessarie per la dissipazione sono realizzati con un attacco in TMAH. In figura 3.44 è mostrato uno degli scavi ottenuti sul retro del wafer di silicio.
Il modello teorico della struttura così realizzata mostra che portando la temperatura nella zona dei canali a 94 ºC (temperatura massima del ciclo PCR), con il resistore della zona delle sonde (zona “probe”) disattivato, effettivamente ciascuno scavo abbassa la temperatura di circa 10 ºC (fig. 3.45 in alto). Inoltre, attivando il resistore della zona delle sonde, può essere ottenuta ogni temperatura tra 50 e 60 ºC, che è l’intervallo di temperatura ottimale per l’ibridazione.
Con la simulazione dello stato dinamico (fig. 3.45 in basso) si verifica che il ciclo rapido della zona dei canali ha un impatto molto piccolo sulla zona delle sonde. Nelle simulazioni, il resistore della zona delle sonde non è attivato e la temperatura in tale zona oscilla intorno a 33 ºC. Attivando l’elemento riscaldante, la temperatura può aumentare tra 50 e 60 ºC.
Il prototipo analizzato dimostra che esiste la possibilità di realizzare strutture microfabbricate in grado di
Temperatura (ºC) Trench zone Probe zone 0 Distanza (mm) 24 94 88 76 64 52 40 Channel zone 96 76 56 36 16 0 40 80 Probe zone Channel zone Temperatura (ºC) Tempo (s)
Figura 3.45: in alto, profilo di temperatura della zona dei canali, della zona delle alette e della zona
delle sonde; la zona “trench” funziona effettivamente da dissipatore di calore. In basso:
simulazioni termiche in stato dinamico. Dopo aver portato la temperatura a 94 ºC, sono imposti
alla zona di canale rapidi cicli termici tra 94 ºC e 52 ºC. È evidente che la zona delle sonde
svolgere più operazioni di analisi del DNA. È prevedibile ipotizzare per questo tipo di dispositivi difficoltà di interfacciamento con il “macro-mondo”, in particolare nella preparazione e iniezione di volumi inferiori al microlitro in maniera indipendente in ciascun canale, per poter eseguire più PCR in parallelo. Da un punto di vista biologico, è problematica l’estrazione del DNA e la preparazione della miscela di PCR in volumi inferiori al microlitro.
Riferimenti bibliografici capitolo 3
[1] Tibor Chován, András Guttman, “Microfabricated devices in biotechnology and biochemical processing”, TRENDS in Biotechnology, Vol. 20, No. 3, pp 116-122, 3 marzo 2002.
[2] Bruno Murari, “Micro-Electro-Mechanical System: A New Adventure in the Semiconductor World”, Forum ADEMIS, Parigi, 18 novembre 1999.
[3] Bruno Murari, “Is Micromachining Still a Dream or an Industrial Reality?”, STMicroelectronics.
[4] Eric S.Lander, “Array of hope”, Nature genetics supplement, Vol. 21, gennaio 1999, pp 3-4, 1999 Nature America Inc.
[5] Manjula Kurella e altri, “DNA Microarray Analysis of Complex Biologic Processes”, Journal of the American Society of Nephrology, Vol. 12, 2001, pp 1072-1078.
[6] D. Gillespie, S. Spiegelman, “A quantitative assay for DNA-RNA hybrids with DNA immolised on a membrane”, J. Mol. Biol., Vol. 12, pp 829-842, 1965.
[7] David J.Duggan, Michael Bittner, Yidong Chen, Paul Meltzer & Jeffrey M.Trent, “Expression profiling using cDNA microarray”, Nature genetics supplement, Vol. 21, gennaio 1999, pp 10-14, 1999 Nature America Inc.
[8] Vivian G.Cheung, Michael Morley, Francisco Aguilar, Aldo Massimi, Raju Kucherlapati & Geoffrey Childs, “Making and reading microarray”, Nature genetics supplement, Vol. 21, gennaio 1999, pp 15-19, 1999 Nature America Inc.
[9] O.Sheils, S.Finn & J.O’Leary, “Nucleic acid microarray: an overview”, Current Diagnostic Pathology (2003), No. 9, pp 155-158, 2003 Elsevier Science Ltd.
[10] Christian Stratowa, Roger Abseher, “Microarrays in Disease Diagnosis and Prognosis”, Encyclopedia of the human genome/
2003 Mcmillan Publishers Ltd, Nature Publishing group/
www.ehgoline.net.
[11] C.F. Edman, D.E. Raymond, D.J.Wu, E. Tu, R.G. Sosnowski, W.F. Butler, M. Neremberg & M.J. Heller, “Electric Field directed nucleic acid hybridization on microchips”, Nucleic Acids Research, Vol. 25, No. 24, pp 4097-4914, 1997.
[12,13] Dal sito internet della Nanogen: http://www.nanogen.com. In particolare:
http://www.nanogen.com/products/nanochip_micro.htp e
http://www.nanogen.com/products/workstation.htm .
[14] Edwin Southern, Kalim Mir & Mikhail Shchepinov, “Molecular interactions on microarrays”, Nature genetics supplement, Vol. 21, gennaio 1999, pp 5-9, 1999 Nature America Inc.
[15] Ronald C. McGlennen, “Miniaturization Technologies for Molecular Diagnostics”, Clinical Chemistry, Vol. 47, No. 3, pp 393-402, 2001. [16] Sito internet Affimetrix: http://www.affymetrix.com.
In particolare consultare (collegato il 20 ottobre 2003):
http://www.affymetrix.com/technology/manufacturing/index.affx.
[17] Robert J. Lipshutz, Stephen P.A. Fodor, Thomas R. Gingeras & David J. Lockart, “High density synthetic oligonucleotide arrays”, Nature genetics supplement, Vol. 21, gennaio 1999, pp 20-24, 1999 Nature America Inc.
[18] P.F. Xiao, N.Y. He, Q.G. He, Z.C. Liu, Z.H. Lu, “Soft litography for oligonucleotide arrays fabrication”, maggio 2001, pp 3104-3107, 2001 IEEE.
[19] “Agilent SurePrint Technology”, gennaio 2003, Agilent Technologies 2003, pubblicazione numero: 5988-8171EN. Ind. internet :
www.chem.agilent.com/temp/rad6DEC9/00039489.pdf. Collegato il
[20] S.B. Fulmer-Smentek, “Performance comparison of Agilent’s 60-mer and 25-mer in situ synthesized oligonucleotides microarrays”, Agilent Technologies 2003, pubblicazione numero: 5988-5977EN.
Ind. internet:
www.chem.agilent.com/temp/radAE3E3/00042209.pdf. Collegato il
22 ottobre 2003.
[21] Stephen C. Jacobson J. Michael Ramsey: “Integrated Microdevice for DNA Restriction Fragment Analysis”, Analytical Chemistry, Vol. 68, No. 5, pp 720-723, 1 marzo 1996.
[22] Dieter Schmalzing, Aram Adourian, Lance Koutny, Liuda Ziaugra, Paul Matsudaria & Daniel Ehrlich, “DNA sequencing on Microfabricated Electrophoretic Devices”, Analytical Chemistry, Vol. 70, No. 11, pp 2303-2310, 1 giugno 1998.
[23] Adam T. Woolley & Richard A. Mathies, “Ultra-High-Speed DNA Sequencing Using Capillary Electrophoresis Chips”, Analytical Chemistry, Vol. 67, No. 20, pp 3676-3680, 15 ottobre 1995.
[24] Ho Jin Jung, Young Chan Bae, “New Silanisation Coating for DNA Fragment Analysis by Capillary Electrophoresis”, Journal of
Polymer Science: Part A: Polymer Chemistry, Vol. 40,
pp 1405-1420, 2002, 2002 Wiley Periodicals Inc.
[25] Shaorong Liu, Yining Shi,William W. Ja & Richard A. Mathies, “Optimization of High-Speed DNA Sequencing on Microfabricated Capillary Electrophoresis Channels”, Analytical Chemistry, Vol. 71, No. 3, pp 566-573, 1 febbraio 1999.
[26] D. Schmalzing, L. Koutny, A. Adourian, P. Belgrader, P. Matsudaira & D. Ehrlich, “DNA typing in thirty seconds with a microfabricated device”, Proc.Natl.Acad.Sci.USA, Vol. 94, pp 10273-10278, settembre 1997, Genetics. Ind. internet (collegato il 27 ottobre 2003): http://www.pnas.org/cgi/content/full/94/19/10273.
[27] L. Koutny, D. Schmalzing, T.A. Taylor & M. Fuchs, “Microchip Electrophoretic Immunoassay for Serum Cortisol”, Analytical Chemistry, Vol. 68, No. 1, pp 18-22, 1 gennaio 1996.
[28] Y. Sci, P.C .Simpson, J.R. Scherer, D. Wexler, C. Skibola, M.T. Smith, & Richard A. Mathies, “Radial Capillary Array Electrophoresis Microplate and Scanner for High-Performance Nucleic Acid Analysis”, Analytical Chemistry, Vol. 71, No. 23, pp 5354-5361, 1 dicembre 1999.
[29] I.L. Medintz, B.M. Paegel, Richard A. Mathies, “Microfabricated capillary array electrophoresis DNA analysis system”, Journal of Chromatografy, 924 (2001), pp 265-270.
[30] B.M. Paegel, C.A. Emrich, G.J. Wedemayer, J.R. Scherer & Richard A. Mathies, “High throughput DNA sequencing with a microfabricated 96-lane capillary array electrophoresis bioprocessor”, Proc. Natl. Acad. Sci. (PNAS), Vol. 99, No. 2, pp 574-579, 15 gennaio 2002. Ind internet (collegato il 15 ottobre 2003): http://www.pnas.org/cgi/content/full/99/2/574.
[31] B.M. Paegel, L.D. Hutt, P.C. Simpson & Richard A. Mathies, “Turn Geometry for Minimizing Band Broadening in Microfabricated Capillary Electrophoresis Channels”, Analytical Chemistry, Vol. 72, No. 14, pp 3030-3037, 15 luglio 2000.
[32] B.M. Paegel, S.H.I. Yeung & Richard A. Mathies, “Microchip Bioprocessor for Integrated Nanovolume Sample Purification and
DNA Sequencing”, Analytical Chemistry, Vol. 74, No. 19,
pp 5092-5098, 1 ottobre 2002.
[33] B.M. Paegel, R.G. Blazej & Richard A. Mathies, “Microfluidic devices for DNA sequencing: sample preparation and electrophoretic analysis”, Current Opinion in Biotechnology, 2003, 14: 42-50.
[34] C.A. Emrich, H. Tian, I.L. Medintz & Richard A. Mathies, “Microfabricated 384-Lane Capillary Array Electrophoresis Bioanalyzer for Ultrahigh-Throughput Genetic Analysis”, Analytical Chemistry, Vol. 74, No. 19, pp 5076-5083, 1 ottobre 2002.
[35] P.R. Selvaganapathy, E.T. Carlen & C.H. Mastrangelo, “Recent Progress in Microfluidic Devices for Nucleic Acid and Antibody Assays”, Proceedings of the IEEE, Vol. 91, No. 6, giugno 2003, 2003 IEEE.
[36] Jun Zheng, C. Mastrangelo, M.A. Burns & D.T. Burke, “Single Stranded DNA Separations in Oxide-Passivated Thin-Walled Plastic Microchannels”, 2nd Annual International IEEE-EMBS Special Topic Conference on Microtechnologies in Medicine & Biology, Poster 226, pp 442-446, 2-4 maggio 2002, 2002 IEEE.
[37] J.R. Webster, M.A. Burns, D.T. Burke & C.H. Mastrangelo, “Electrophoresis system with integrated on-chip fluorescence detection”, Art. No. 0-7803-5273-4/00, pp 306-310, 2000 IEEE.
[38] J.R. Webster, M.A. Burns, D.T. Burke & C.H. Mastrangelo, “Monolithic Capillary Electrophoresis Device with Integrated Fluorescence Detector”, Analytical Chemistry, Vol. 73, No. 7, pp 1622-1626, 1 aprile 2001.
[39] S.H. Jeong, S. Kho, D. Jung, S.B. Lee, J.H. Boo, “Deposition of aluminum-doped zinc oxide films by RF magnetron sputtering and study of their surface characteristics”, Surface and Coatings Technologies, 174-175, 2003, pp 187-192, 2003 Elsevier Science. [40] Seth Stern, Carlton Brooks, Michelle Strachan, Anne Kopf-Sill,
J.Wallace Parce, “Microfluidic Thermocyclers for Genetic
Analysis”, 2002 Inter Society Conference on Thermal Phenomena, Art. No. 0-7803-7152-6/02, pp 1033-1038, 2002 IEEE.
[41] Deirdre Meldrum, “Automation for Genomics, Part One: Preoaration for Sequencing”, Genome Research, Vol. 10, No. 8, pp 1081-1092, agosto 2000. Ind. Internet (collegato il 15 ottobre 2003):
http://www.genome.org/cgi/content/full/10/8/1081.
[42] M. Torchia, “Progetto e realizzazione di un Lab-On-Chip integrato su silicio con particolare enfasi all’isolamento termico”, Tesi di Laurea Università di Pisa, 2001.
[43] A. Fuchs, U. Mastromatteo e altri, “A silicon Lab-On-Chip for integrated sample preparation by PCR and DNA analysis by hybridization”, 2nd Annual International IEEE-EMBS Special Topic Conference on Microtechnologies in Medicine & Biology, Poster 130, pp 227-231, 2-4 maggio 2002, 2002 IEEE.
Figure:
Fig. 3.1 e 3.2: dal sito della STMicroelectronics: http://www.st.com. Fig. 3.3: http://microarray1k.aecom.yu.edu/. Collegato il 16 ottobre 2003. Fig. 3.4: http://cmgm.stanford.edu/pbrown/mguide/tips.html. Collegato il
19 ottobre 2003.
Fig. 3.5: David J.Duggan, Michael Bittner, Yidong Chen, Paul Meltzer & Jeffrey M.Trent, “Expression profiling using cDNA microarray”, Nature genetics supplement, Vol. 21, gennaio 1999, pp 10-14, 1999 Nature America Inc, elaborata da pag 11.
Fig. 3.6: dal sito dalla Nanogen: http://www.nanogen.com; in particolare: figura in alto:
http://www.nanogen.com/technology/core_technology.htm;
figura in basso: http://www.nanogen.com/technology/index.htm. Collegato il 7 novembre 2003.
Fig. 3.7: dal sito della Nanogen:
http://www.nanogen.com/product/nanochip_micro.htm. Collegato il
7 novembre 2003.
Fig. 3.8: dal sito internet della Affimetrix:
http://www.affymetrix.com/corporate/media/image_library/low_res/
single_feature.jpg. Collegato il 18 ottobre 2003.
Fig. 3.9: figura a sinistra:
http://www.affymetrix.com/corporate/media/image_library/low_res/
photolitography.jpg. Collegato il 18 ottobre 2003.
figura a destra:
http://www.affymetrix.com/corporate/media/image_library/low_res/
photolithographicmask.jpg. Collegato il 18 ottobre 2003.
Fig. 3.10: dal sito della Affimetrix:
http://www.affymetrix.com/technology/manufacturing/index.affx.
Collegato il 20 ottobre 2003.
Fig. 3.11: collegato il 18 ottobre all’indirizzo:
http://www.affymetrix.com/corporate/media/image_library/low_res/ hgu133plus.jpg
Fig. 3.12 collegato il 18 ottobre all’indirizzo:
http://www.affymetrix.com/corporate/media/image_library/low_res/ hybridization_of_tagged_probes.jpg
Fig. 3.13: collegato il 18 ottobre all’indirizzo:
http://www.affymetrix.com/corporate/media/image_library/low_res/t agged_and_untagged.jpg
Fig. 3.14: Ind. internet :
www.chem.agilent.com/temp/rad6DEC9/00039489.pdf. Collegato il
22 ottobre 2003.
Fig. 3.15 e 3.16: elaborate da pag. 3677 dell’articolo:
Adam T. Woolley & Richard A. Mathies, “Ultra-High-Speed DNA Sequencing Using Capillary Electrophoresis Chips”, Analytical Chemistry, Vol. 67, No. 20, pp 3676-3680, 15 ottobre 1995.
Fig. 3.17: elaborata da: D. Schmalzing, L. Koutny, A. Adourian, P. Belgrader, P. Matsudaira & D. Ehrlich, “DNA typing in thirty seconds with a microfabricated device”, Proc.Natl.Acad.Sci.USA, Vol. 94, pp 10273-10278, settembre 1997, Genetics.
Ind. internet (collegato il 27 ottobre 2003):
http://www.pnas.org/cgi/content/full/94/19/10273.
Fig. 3.18: elaborata da pag. 20 dell’articolo: L. Koutny, D. Schmalzing, T.A. Taylor & M. Fuchs, “Microchip Electrophoretic Immunoassay for Serum Cortisol”, Analytical Chemistry, Vol. 68, No. 1, pp 18-22, 1 gennaio 1996.
Fig. 3.19: figura in alto: Analytical Chemistry, Vol 68, No 1, pp 18-22, 1 gennaio 1996 (stesso articolo della figura 3.16).
Figura in basso: Shaorong Liu, Yining Shi,William W. Ja & Richard A. Mathies, “Optimization of High-Speed DNA Sequencing on Microfabricated Capillary Electrophoresis Channels”, Analytical Chemistry, Vol. 71, No. 3, pp 566-573, 1 febbraio 1999.
Fig. 3.20 e 3.24: Y. Sci, P.C .Simpson, J.R. Scherer, D. Wexler, C. Skibola, M.T. Smith, & Richard A. Mathies, “Radial Capillary Array Electrophoresis Microplate and Scanner for High-Performance Nucleic Acid Analysis”, Analytical Chemistry, Vol. 71, No. 23, pp 5354-5361, 1 dicembre 1999.
Fig. 3.21: B.M. Paegel, C.A. Emrich, G.J. Wedemayer, J.R. Scherer & Richard A. Mathies, “High throughput DNA sequencing with a microfabricated 96-lane capillary array electrophoresis bioprocessor”, Proc. Natl. Acad. Sci. (PNAS), Vol. 99, No. 2, pp 574-579, 15 gennaio 2002. Ind. internet (collegato il 15 ottobre 2003): http://www.pnas.org/cgi/content/full/99/2/574.
Fig. 3.22 (elaborata) e 3.23: B.M. Paegel, L.D. Hutt, P.C. Simpson & Richard A. Mathies, “Turn Geometry for Minimizing Band Broadening in Microfabricated Capillary Electrophoresis Channels”,
Analytical Chemistry, Vol. 72, No. 14, pp 3030-3037,
15 luglio 2000.
Fig. 3.25 e 3.29: C.A. Emrich, H. Tian, I.L. Medintz & Richard A. Mathies, “Microfabricated 384-Lane Capillary Array Electrophoresis Bioanalyzer for Ultrahigh-Throughput Genetic Analysis”, Analytical Chemistry, Vol. 74, No. 19, pp 5076-5083, 1 ottobre 2002.
Fig. 3.26 e 3.27: B.M. Paegel, C.A. Emrich, G.J. Wedemayer, J.R. Scherer & Richard A. Mathies, “High throughput DNA sequencing with a microfabricated 96-lane capillary array electrophoresis bioprocessor”, Proc. Natl. Acad. Sci. (PNAS), Vol. 99, No. 2, pp 574-579, 15 gennaio 2002. Ind. internet (collegato il 15 ottobre 2003): http://www.pnas.org/cgi/content/full/99/2/574.
Fig. 3.28: elaborata da pag. 5094 dell’articolo: B.M. Paegel, S.H.I. Yeung & Richard A. Mathies, “Microchip Bioprocessor for Integrated Nanovolume Sample Purification and DNA Sequencing”, Analytical Chemistry, Vol. 74, No. 19, pp 5092-5098, 1 ottobre 2002.
Fig. 3.30, 3.31, 3.34 e 3.35: J.R. Webster, M.A. Burns, D.T. Burke & C.H. Mastrangelo, “Monolithic Capillary Electrophoresis Device with Integrated Fluorescence Detector”, Analytical Chemistry, Vol. 73, No. 7, pp 1622-1626, 1 aprile 2001.
Fig. 3.32 e 3.33: J.R. Webster, M.A. Burns, D.T. Burke & C.H. Mastrangelo, “Electrophoresis system with integrated on-chip fluorescence detection”, Art. No. 0-7803-5273-4/00, pp 306-310, 2000 IEEE.
Fig. 3.36, 3.39, 3.40, 3.42, 3.44: M. Torchia, “Progetto e realizzazione di un Lab-On-Chip integrato su silicio con particolare enfasi all’isolamento termico”, Tesi di Laurea Università di Pisa, 2001.
Fig. 3.38, 3.41, 3.45: A. Fuchs, U. Mastromatteo e altri, “A silicon Lab-On-Chip for integrated sample preparation by PCR and DNA analysis by hybridization”, 2nd Annual International IEEE-EMBS Special Topic Conference on Microtechnologies in Medicine & Biology, Poster 130, pp 227-231, 2-4 maggio 2002, 2002 IEEE.
![Figura 3.33: sistema di rivelazione di fluorescenza. [37]](https://thumb-eu.123doks.com/thumbv2/123dokorg/5635270.69271/8.892.181.762.155.425/figura-sistema-di-rivelazione-di-fluorescenza.webp)