1. I
NTRODUZIONE.
1.1. V
IRUS COINVOLTI NELLE INFEZIONI ALIMENTARI.
Lo studio dei dati epidemiologici sulle infezioni alimentari degli ultimi anni ha evidenziato unʼimportanza crescente dei virus enterici, anche a seguito del miglioramento delle tecniche di analisi che permettono di attribuire a questi agenti patogeni molti episodi epidemici prima classificati ad eziologia sconosciuta. Fino allʼinizio degli anni 70, infatti, le gastroenteriti virali nell'uomo erano diagnosticate solo sulla base di criteri epidemiologici (Kaplan et al., 1982) a causa della scarsa sensibilità e disponibilità di metodi per l'identificazione dei virus enterici che non permettevano la loro determinazione diretta negli alimenti e, talvolta, neanche da campioni fecali (Atmar et al., 1995).
Lʼaumento delle gastroenteriti virali è stato correlato anche allʼincremento numerico dʼindividui a rischio in alcuni settori di popolazione, quali anziani ed immunocompromessi, ed anche lo stress rappresenta un fattore predisponente. Infatti, individui sani sottoposti a situazioni di particolare pericolosità, quali soldati in teatri di guerra, si sono dimostrati più suscettibili a questo tipo di patologie (Ahmad, 2002). Lʼincremento degli scambi internazionali di prodotti alimentari ha contribuito inoltre alla rapida diffusione di malattie trasmesse per queste vie, incluse quelle ad eziologia virale, comʼè stato segnalato sia dallʼOrganizzazione Mondiale della Sanità (Motarjemi et al., 1995) che dal Codex Alimentarius (Codex Committee on Food Hygiene, 1999).
!
"#! I virus enterici replicano nellʼintestino umano e possono essere presenti nelle feci di individui infetti con concentrazioni che possono arrivare fino a 1010 particelle virali per grammo (Feachem et al., 1983; Yates et al., 1988; Gerba 2000; Skinhoj et al., 1981; Tassopoulos et al., 1986). Le feci disperse nelle acque fognarie possono provocare la contaminazione delle acque superficiali, incluse quelle marine, poiché i processi di trattamento dei liquami non sono generalmente capaci di rimuovere completamente i virus (Gilgen et al., 1997). Questi possono sopravvivere a lungo in acqua (Abad et al., 1997; Carducci et al., 1996; Enriquez et al., 1995; Koopmans and Duizer, 2004; Payment 1998), in quanto protetti dai trattamenti di disinfezione, dall'azione inattivante della temperatura, del pH e dei raggi UV ed infine dall'antagonismo microbico grazie a due fenomeni che sembrano essere alla base della loro persistenza e della loro diffusione nell'ambiente: l'aggregazione e l'adsorbimento (Sobsey et al., 1980, Croci et al., 1992).
Il primo corrisponde alla formazione di aggregati formati da 2-10 unità virali chiamate "Clumps" che garantiscono una maggiore resistenza delle singole particelle alle condizioni ambientali avverse (Mbithi et al., 1991), mentre il secondo è legato al grande potenziale di adsorbimento che questi virus hanno nei confronti di qualunque tipo di materiale particolato (organico ed inorganico), come particelle argillose, silicati, batteri, cellule algali e particolati organici, che aumenta la loro sopravvivenza nelle acque d'estuario e nei sedimenti spesso grazie alla creazione di una vera e propria barriera fisica. Così protetti riescono a sopravvivere per lunghi periodi nell'ambiente marino e possono essere trasportati dalle correnti anche a grande distanza dai punti di scarico (Dowd et al., 1998).
Nella tabella 1.1. sono riportati i diversi virus patogeni che possono essere ritrovati in ambiente acquatico.
Diverse prove sperimentali hanno evidenziato che i virus sono più resistenti dei batteri ai comuni trattamenti di bonifica, compresa la clorazione (Duizer et al., 2004; Gulati et al., 2001), ed è stato dimostrato che sopravvivono fino a 130 giorni in acqua di mare, quindi più a lungo dellʼEscherichia coli che è utilizzato come indicatore di contaminazione fecale sia dell'acqua che dei molluschi (Gary, 1988). Nel caso di utilizzo di acque contaminate per scopi irrigui o nellʼallevamento di molluschi bivalvi, i virus contaminano gli alimenti che consumati crudi o poco cotti possono causare malattia nel consumatore.
Diversamente dai batteri, i virus non si moltiplicano negli alimenti né producono tossine in quanto parassiti intracellulari obbligati e quindi incapaci di replicare al di fuori della cellula ospite; per questo motivo nei diversi prodotti alimentari possono Tabella 1.1. Classificazione, metodi di rilevamento e tipizzazione dei virus patogeni presenti negli alimenti. (Koopmans and Duizer, 2004)
!
"#! essere semplicemente veicolati e perdere la loro infettività (Cliver, 1997; Appleton, 2000; Koopmans et al., 2002; Koopmans and Duizer, 2004; Fleet et al., 2000). La dose infettante per contro è molto bassa: generalmente tra 1 e 10 unità virali (Leclerc et al., 2002, Ward et al., 1984) e quindi il rischio dʼinfezione da virus presenti negli alimenti a parità di livello di esposizione risulta molto più alto di quello per i batteri. Si conoscono oltre 120 virus enterici classificati in diverse specie in base alle loro caratteristiche morfologiche, chimiche, fisiche, antigeniche e genetiche (Rao et al., 1986; Durkop,1992; Wilhelmi et al., 2003; Phan et al., 2007; Panda et al., 2007; Hansman et al., 2007) ma non tutti sono stati correlati con casi di malattia nellʼuomo trasmessi attraverso lʼacqua o gli alimenti.
I virus enterici responsabili di infezioni alimentari comprendono:
• Virus che provocano gastroenteriti: Rotavirus, Adenovirus tipo 40 e 41 e due generi di Calicivirus enterici umani: i Norovirus (NoV) e i Sapovirus (SV).
• Virus dellʼepatite a trasmissione oro-fecale: virus dellʼEpatite A (Hepatitis A Virus, HAV) e virus dellʼEpatite E (Hepatitis E Virus, HEV).
• Virus che si replicano nellʼintestino umano ma provocano patologie in altri organi, quali il sistema nervoso centrale o il fegato (Enterovirus) (Koopmans et al., 2002, De Medici et al., 2005, Koopmans and Duizer, 2004).
In diverse specie animali sono state isolate varianti geneticamente differenti da quelle umane. I NoV sono stati isolati frequentemente sia nei bovini che nei suini; tali ceppi sono però geneticamente ben distinti da quelli isolati nellʼuomo e non è mai stata documentata la trasmissione alla specie umana (Sugieda et al., 1998; Sugieda et al., 2002). Anche i virus dellʼEpatite E isolati nei suini sono geneticamente molto simili a quelli isolati da casi clinici umani, anche se è da sottolineare che lʼisolamento di HEV in questi animali è abbastanza comune anche in quei paesi dove lʼHEV è raramente
diagnosticato nellʼuomo (Worm et al., 2002). Lʼalta prevalenza di anticorpi anti-HEV è stata riportata nel sangue di donatori da regioni non endemiche, e questo potrebbe essere legato alla trasmissione zoonotica del virus. Virus HEV-correlati sono stati riportati nel suino, nel cervo e nel cinghiale, come anche in roditori e pollame. La trasmissione diretta è stata evidenziata dallʼanimale allʼuomo attraverso il consumo di carne di cervo poco cotta o da fegato di cinghiale crudo. Le persone che consumano prodotti suini contaminati o sono coinvolti nellʼallevamento del maiale sono
potenzialmente a rischio di infezione da HEV
(www.who.it/vaccine_research_/disease/zoonotic/en/index2.html).
Quanto detto suggerisce che il rischio di trasmissione zoonotica dei virus enterici è relativamente basso, anche se non può essere completamente escluso. Cʼè quindi la necessità, anche in relazione alla flessibilità genetica che tutti i virus a RNA normalmente dimostrano di avere, di monitorare continuamente la loro presenza nellʼambiente per evidenziare qualsiasi cambiamento del loro comportamento (Duizer et al., 2004).
1.2. N
OROVIRUS.
1.2.1.
C
ENNI STORICI E TASSONOMIA.
I Norovirus, detti anche “Small Round Structured Viruses”, SRSV, o “Norwalk-like Viruses”, dalla prima identificazione in unʼepidemia in una scuola elementare nel 1968 a Norwalk, Ohio, USA, sono il prototipo dei Calicivirus (calice dal latino calix = calice, coppa) (Leuenberger et al., 2007; Rabenau et al., 2003).
!
"#! Inizialmente descritti sulla base del loro aspetto al microscopio immuno-elettronico come dei Picornavirus o Parvovirus (Kapikian et al., 1972), vennero più tardi inseriti in una nuova famiglia creata appositamente: le Caliciviridae.
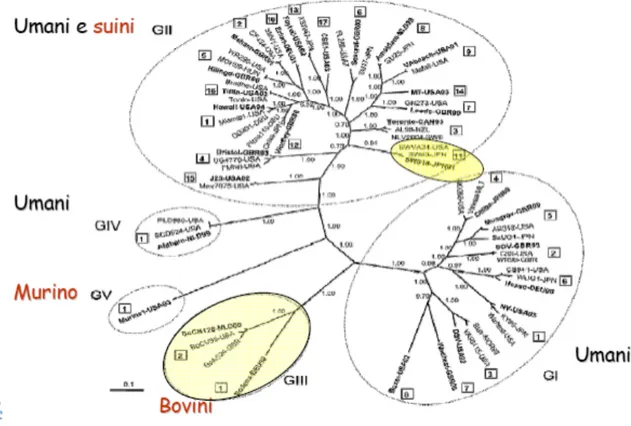
La famiglia Caliciviridae, virus a RNA a singola catena, poliadenilato, con senso positivo, è divisa in quattro generi: Norovirus, Saporovirus, Lagovirus e Vesivirus (Costantini et al., 2006; Ozawa et al., 2007).
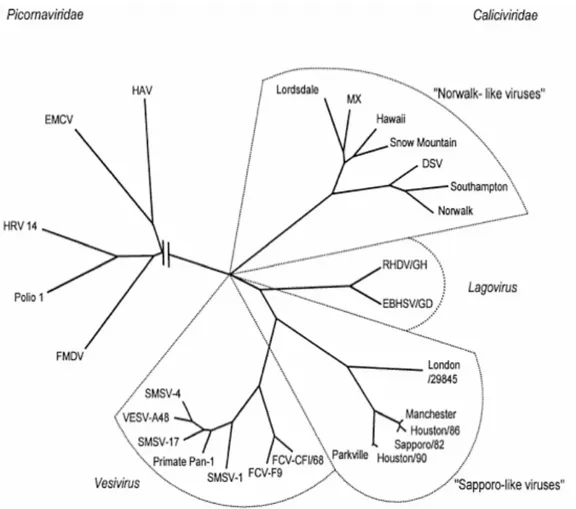
In figura 1.2. è illustrato lʼalbero filogenetico costruito mediante lʼanalisi delle sequenze nucleotidiche dellʼintero gene della proteina capsidica VP1 (ORF2) dei virus di cui si sospettava lʼappartenenza alla Famiglia Caliciviridae. In evidenza è rappresentata la distanza della famiglia Caliciviridae da Picornaviridae. Allʼinterno della famiglia Caliciviridae, inoltre, si distinguono filogeneticamente i quattro rami dei generi Norovirus (Norwalk-like virus NLV), Lagovirus, Vesivirus e Sapovirus (Sapporo-like virus SLVs).
I distinti rami presenti all'interno del genere Lagovirus rappresentano le 2 specie di questo genere: il virus della malattia emorragica del coniglio (RHDV) e il virus della
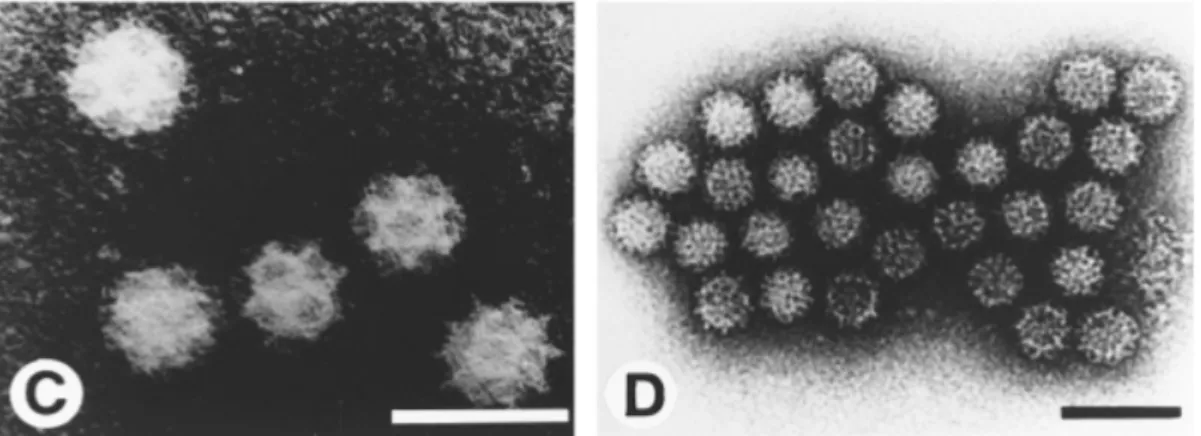
Figura 1.1. C e D fotografia in contrasto negativo di Norwalk-like Virus di estratti di feci umane. La barra rappresenta 50 nm (Green et al., 2000).
sindrome della lepre marrone Europea (EBHSV). Anche nel genere Vesivirus si evidenziano due rami distinti ai quali appartengono il calicivirus della specie felina (FCV) e il virus dellʼesantema vescicolare dei suini (SMSV). Sono presenti anche altri rami all'interno di generi NLV e SLVs.
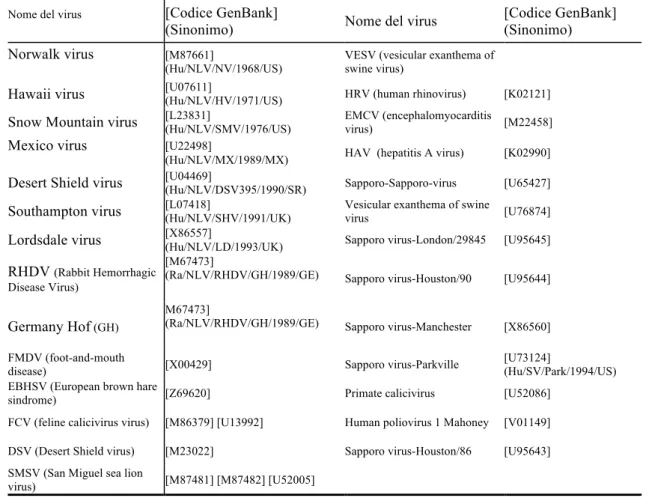
I codici dei ceppi analizzati in GenBank per la realizzazione di questa analisi sono riportati in tabella 1.2.: il codice è seguito dal codice di identificazione utilizzato dallʼInternational Committee on Taxonomy of Viruses (Specie in cui il virus è stato trovato/Genere/Sigla del virus/anno di ritrovamento/Nazione di isolamento).
Figura 1.2. Albero filogenetico che rappresenta i 4 generi della Caliciviridae e la loro distanza da Picornaviridae (Berke and Matson, 2000).
!
"#!
Nome del virus [Codice GenBank]
(Sinonimo) Nome del virus
[Codice GenBank] (Sinonimo) Norwalk virus [M87661]
(Hu/NLV/NV/1968/US)
VESV (vesicular exanthema of swine virus)
Hawaii virus [U07611] (Hu/NLV/HV/1971/US) HRV (human rhinovirus) [K02121]
Snow Mountain virus [L23831] (Hu/NLV/SMV/1976/US) EMCV (encephalomyocarditis virus) [M22458]
Mexico virus [U22498]
(Hu/NLV/MX/1989/MX) HAV (hepatitis A virus) [K02990]
Desert Shield virus [U04469] (Hu/NLV/DSV395/1990/SR) Sapporo-Sapporo-virus [U65427]
Southampton virus [L07418] (Hu/NLV/SHV/1991/UK) Vesicular exanthema of swine virus [U76874]
Lordsdale virus [X86557] (Hu/NLV/LD/1993/UK) Sapporo virus-London/29845 [U95645]
RHDV (Rabbit Hemorrhagic Disease Virus)
[M67473]
(Ra/NLV/RHDV/GH/1989/GE) Sapporo virus-Houston/90 [U95644]
Germany Hof (GH)
M67473]
(Ra/NLV/RHDV/GH/1989/GE) Sapporo virus-Manchester [X86560] FMDV (foot-and-mouth
disease) [X00429] Sapporo virus-Parkville
[U73124]
(Hu/SV/Park/1994/US) EBHSV (European brown hare
sindrome) [Z69620] Primate calicivirus [U52086]
FCV (feline calicivirus virus) [M86379] [U13992] Human poliovirus 1 Mahoney [V01149] DSV (Desert Shield virus) [M23022] Sapporo virus-Houston/86 [U95643] SMSV (San Miguel sea lion
virus) [M87481] [M87482] [U52005]
Tabella 1.2. Codici GenBank dei ceppi utilizzati per lʼalbero filogenetico dei Virus di cui si sospettava lʼappartenenza alla Famiglia Caliciviridae. (Katayama et al., 2002).
Geneticamente i Norovirus possono essere divisi in cinque genogruppi (GI, GII, GIII, GIV e GV) in base alla regione N-terminale (regione C) della regione del capside VP1 e alla sequenza della regione RdRp (Bull et al., 2006; Castilho et al., 2006; Lodder and Husman, 2004).
I NoV che colpiscono lʼuomo appartengono ai genogruppi I, II e IV, i quali possono essere rispettivamente suddivisi in 8, 19 e 1 genotipi sulla base della topologia dellʼalbero filogenetico e sulla sequenza del gene del capside (Costantini et al., 2006). Non esistendo però un sistema di genotipizzazione unificato per i NoV, i genogruppi possono essere suddivisi in vario numero di genotipi (Vainio and Myrmel, 2006). Inizialmente sulla base della regione D identificata allʼinterno della sequenza VP1, il
genogruppo I è stato suddiviso in 7 genotipi ed il genogruppo II in 12 (Vinjè et al., 2004), mentre utilizzando la diversità nella sequenza nucleotidica del gene del capside N terminus/Shell, il genogruppo I è stato suddiviso in 14 genotipi ed il GII in 17 (Kageyama et al., 2004).
Al genogruppo II appertengono anche i NoV che infettano il suino, identificati in Giappone, Europa e Stati Uniti, ed appartenenti a tre diversi genotipi (Costantini et al., 2006) mentre al genogruppo IV appartengono anche quelli che colpiscono i carnivori domestici e selvatici, identificati come GIV.2 (Martella et al., 2008).
!
"#! I NoV del genogruppo III, che colpiscono il bovino e sono stati identificati in Germania, Inghilterra e Stati Uniti, sono stati suddivisi in 2 genotipi (Costantini et al., 2006). Il genogruppo V presenta un solo genotipo cui appartengono i NoV murini (Martella et al., 2008).
1.2.2.
I
LV
IRUS.
NoV è un virione di forma sferica, privo di envelope, di piccole dimensioni, 25-35 nm di diametro (Leuenberger et al., 2007), e possiede un genoma a RNA a singola catena, poliadenilato, di senso positivo lungo 7.400 – 7.700 nucleotidi (Bull et al., 2006, Castilho et al., 2006).
Il capside contiene 90 dimeri che formano un involucro dal quale protrudono 90 capsomeri simili ad archi a livello locale dei doppi assi. Gli archi sono disposti in modo tale da formare, nelle posizioni tre e cinque delle pieghe icosaedriche, larghe depressioni simili a calici, da cui il nome Caliciviridae. (Green et al., 2001)
Figura 1.4. X-ray structure of the Norwalk virus capsid, with the inset showing details of the structure of the subunits.
The different colors represent different regions of the capsid protein.
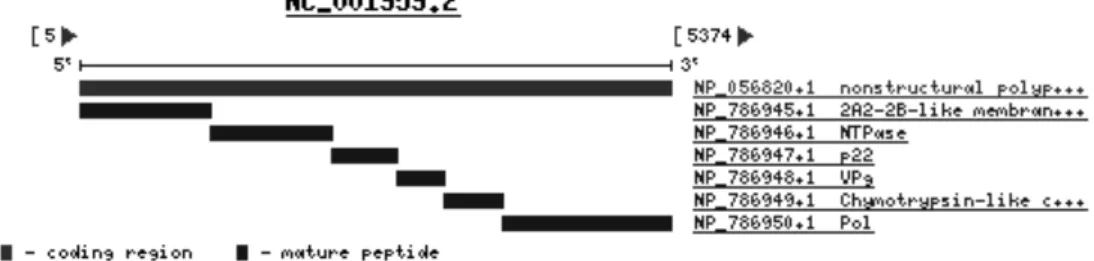
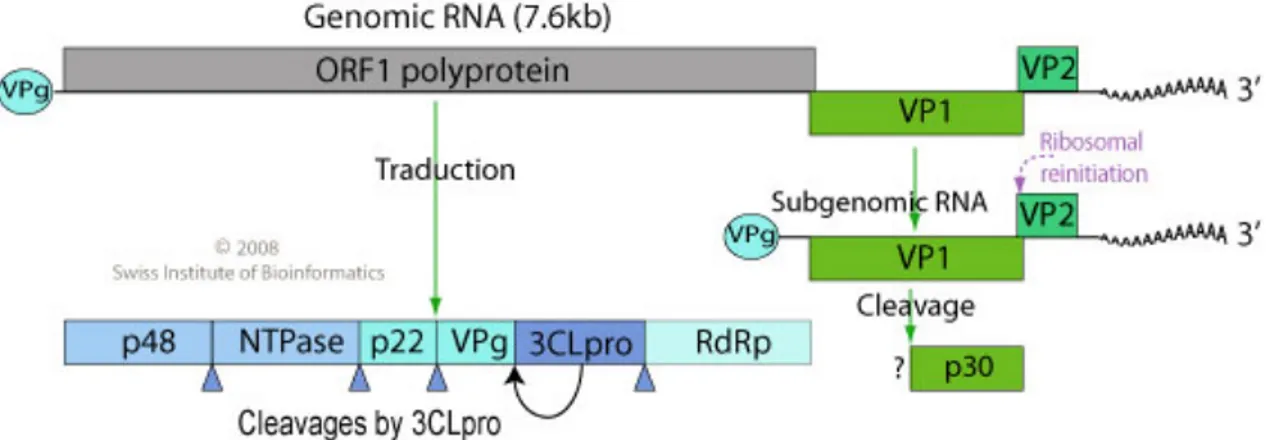
Il genoma dei NoV è diviso in tre open reading frame (ORFs) (figura 1.5.):
• ORF1 codifica una grande poliproteina, approssimativamente 1700 amminoacidi. Essa subisce un cleavage proteolitico che produce proteine virali, omologhe a proteine di altri virus a RNA a singola catena, che includono nucloeside trifosfatasi, proteasi 3C-like, RNA polimerasi-RNA dipendente (RdRp o POL);
Figura 1.5. Schematic of the complete NV genome, indicating the location of the nonstructural protein cleavage products within the ORF1 polyprotein, the viral structural proteins VP1 and VP2 encoded by ORF2 and ORF3, respectively, and the 5ʼ and 3ʼ untranslated regions (Asanaka et al., 2005).
Figura 1.6. Rappresentazione schematica della sequenza codificante della ORF1, sono rappresentati i punti di taglio (cleavage) che generano le sei proteine mature. ORF1 codifica per un precursore poliproteico che viene tagliato dopo la traduzione dalla proteasi in sei proteine non strutturali: p48, NTPasi, p22, la proteina VPg (genome-linked viral protein), proteasi (pro) e la RNA polimerasi RNA-dipendente (RdRp). La proteasi taglia nei siti mostrati nellʼimmagine dallʼinterruzione della retta (GenID accession no. 1491970).
!
"#!
• ORF2 codifica la proteina capsidica maggiore VP1 di 550 amminoacidi; 90 dimeri di questa proteina del capside formano la struttura T=3 icosaedrica;
• ORF3 codifica un basso numero di copie della proteina capsidica minore VP2, il cui ruolo preciso non è chiaro, sebbene sia stata suggerita una funzione nellʼup-regolazione dellʼespressione della VP1 e nella stabilizzazione come proteina istone-like nel complesso capside-RNA. (Bull et al., 2006; Castilho et al., 2006; Clarke and Lambden, 2001; Siebenga et al., 2008)
La regione più conservata del genoma dei NoV comprende le regioni C-terminale di ORF1 e la regione dellʼN-terimale di ORF2 (Bull et al., 2006).
Figura 1.7. Rappresentazione schematica della sequenza codificante della ORF2, che genera la proteina capsidica (GenID accession no. 1491972).
Figura 1.8. Rappresentazione schematica della sequenza codificante della ORF3 (a), che genera la proteina capsidica minore VP2. (GenID accession no. 1491971).
1.2.3.
C
ICLO VITALE.
Nonostante la diffusione mondiale e lʼalto livello dʼincidenza non si conosce molto della biologia del Norovirus, del suo ciclo vitale e dei suoi punti critici per un eventuale trattamento (Cox et al., 2009). Non esistendo ad oggi dei sistemi di coltura cellulari efficaci per la replicazione dei NoV che colpiscono lʼuomo, viene considerato, come modello per comprendere la replicazione, il ciclo vitale, la patogenesi e la risposta immunitaria dellʼospite, il Murine Norovirus-1 (MNV-1) (Wobus et al., 2004; Karst et al., 2003).
Wobus et al. hanno dimostrato infatti che MNV-1 ed il Norovirus umano hanno molte caratteristiche simili, tra cui il genoma e la sua organizzazione e funzione, la grandezza del virione (da 28 a 35 nm), la modalità di trasmissione oro-fecale e la sintomatologia provocata (Wobus et al., 2006). Inoltre, allʼinterno dei 5 genogruppi, il norovirus murino è considerato lʼunico ceppo in grado di replicare in colture cellulari di mammifero, così da divenire un eccellente candidato come modello sperimentale del norovirus umano (Wobus et al., 2004; Karst et al., 2003).
Wobus et al. nel 2004 hanno dimostrato che MNV-1 replica nelle linee cellulari ematopoietiche, incluse le cellule RAW 264.7, nelle cellule del midollo spinale che derivano da macrofagi e nelle cellule dendritiche. Altre linee cellulari di macrofagi e cellule dendritiche, che permettono la replicazione di questo virus, sono le IC21, P388D1, WBC264-9C e JAWSII. Le cellule RAW 264.7 rappresentano il modello più ampiamente utilizzato di linee cellulari immortalizzate per gli studi di MNV.
Il virus attacca i recettori della cellula ospite, penetra per endocitosi recettore mediata, perde il suo rivestimento (Uncoating) e rilascia il genoma virale nel citoplasma.
!
""! La proteina VPg viene rimossa dallʼRNA virale, permettendo la traduzione di ORF1 in una poliproteina di cui fanno parte alcune proteine non strutturali coinvolte nella trascrizione stessa dellʼRNA. RNA a singolo filamento (Single strand RNA ssRNA) di senso negativo complementari al genoma vengono sintetizzati usando come modello lʼRNA genomico e viene sintetizzato nuovo RNA genomico usando lʼRNA di senso negativo come modello, così come lʼRNA sub-genomico codifica per proteine strutturali. La fase finale è lʼassemblaggio delle particelle virali e il loro rilascio (figura 1.9.).
1.2.4.
E
PIDEMIOLOGIA.
I NoV sono i patogeni più comunemente isolati sia in casi sporadici che in episodi epidemici di gastroenterite, responsabili di più del 90% delle gastroenteriti non batteriche e di circa il 50% dellʼepidemie di gastroenterite nel mondo (Lopman et al., 2008; Patel et al., 2008). Si stima che il 12% dei casi di gastroenterite severa nei bambini al di sotto dei 5 anni di età ed il 12% dei casi moderati e medi di diarrea nelle
Figura 1.9. RNA genomico di Norovirus: lineare, monopartitico, a singolo filamento di senso positivo, di dimensioni comprese tra 7.3 e 8.3 kb. Allʼestremità 5ʼ una proteina virale (VPg) è legata covalentemente, mentre il terminale 3ʼ è poliadenilato. (http://www.expasy.ch/viralzone/all_by_species/194.html).
persone di tutte le età siano imputabili ai NoV (Patel et al., 2008). In studi condotti nei bambini sotto i 5 anni, la prevalenza di infezioni severe da NoV nei paesi in via di sviluppo è risultata comparabile a quella nei paesi industrializzati (12%) (Patel et al., 2008). Si stima, infatti, che ogni anno i NoV causino 64.000 episodi di diarrea che richiede ospedalizzazione e 900.000 visite cliniche di bambini nei paesi industrializzati e fino a 200.000 decessi di bambini sotto i 5 anni di età nei paesi in via di sviluppo (Patel et al., 2008).
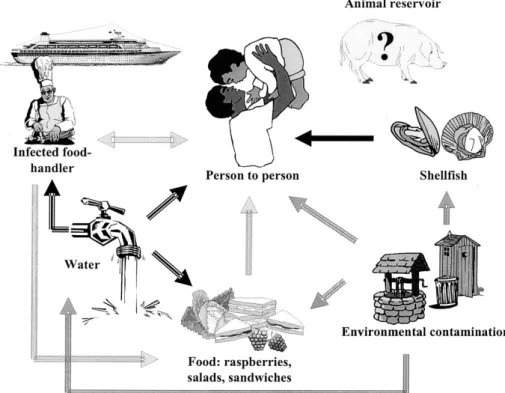
I NoV sono stati rinvenuti in campioni ambientali (per esempio nei liquami trattati e non trattati) così come in alimenti contaminati quali ostriche, frutti di mare, sandwich, insalata, frutti di bosco e perfino in cubetti di ghiaccio (da Silva et al., 2007; Ozawa et al., 2007). Bassi livelli di trasmissione si possono verificare mediante approvvigionamento idrico per contaminazione delle falde o delle acque superficiali. Le acque dolci e marine infatti sono soggette a contaminazione a causa dellʼintensa attività umana che determina apporti continui in aree specifiche. Lʼingestione accidentale di acque ricreative contaminate può portare ad episodi di gastroenterite sporadici (La Rosa et al., 2007), ma sono soprattutto i molluschi filtratori che vivono o vengono allevati in acque contaminate che possono diventare una fonte di contagio; si pensa infatti che i NoV possano essere concentrati nei bivalvi (La Rosa et al., 2007; Nishida et al., 2007). Lʼinfezione primaria risulta dallʼingestione di alimenti o acqua contaminati da feci infette, mentre lʼinfezione secondaria si ha per contatto diretto da persona a persona, aerosol prodotto durante il vomito, fomiti e persone addette alla preparazione dei cibi infetti (La Rosa et al., 2007).
In virtù di queste modalità di trasmissione i NoV possono rappresentare un problema in ambienti in cui si trovano gruppi di persone a stretto contatto; lʼinfezione può
!
"#! essere infatti velocemente trasmessa mediante feci e vomito (Lopman et al., 2008). Casi epidemici possono quindi verificarsi in ambienti di comunità come ospedali, navi da crociera, scuole e ristoranti (Ozawa et al., 2007).
La contaminazione ambientale è frequente a causa dellʼalta resistenza del virus, che può persistere sulle superfici per diverse settimane ed ad alte temperature fino a 60 °C (Leuenberger et al., 2007; Rabenau et al., 2003).
Figura 1.10. Description: The transmission of Noroviruses and Sapoviruses is thought to occur mainly through fecal-oral routes. Several authors have suggested that human caliciviruses may be transmitted by aerosols contaminated with vomit from infected individuals. Outbreaks resulting from contamination of water in community or family water systems have been documented, but waterborne outbreaks resulting from contamination of municipal water systems rarely occur. Food-borne outbreaks resulting from consumption of contaminated food such as uncooked shellfish and precooked foods, such as salad, ham and sandwiches, are common occurrences (Moreno-Espinosa et al., 2004).
Da un punto di vista molecolare è stato evidenziato che, sebbene sia ampia la diffusione dei genogruppi I e II nei campioni dei liquami, la malattia umana è causata soprattutto dai genotipi del genogruppo II (La Rosa et al., 2007). Non se ne conosce la ragione ma una possibile spiegazione è rappresentata da differenze nelle proprietà biologiche come la virulenza, la via di trasmissione, o la stabilità del virus nellʼambiente (La Rosa et al., 2007). Da uno studio epidemiologico condotto in Francia, è emerso che il genogruppo I, più resistente nellʼambiente del GII, è responsabile del 78% delle infezioni alimentari e raro nei casi sporadici (Bon et al., 2005), anche se estese epidemie legate a GI.1 e GI.3 sono state evidenziate sia in adulti che in bambini confermando lʼimportanza di questo genogruppo nelle epidemie nelle comunità (Neonen et al., 2008).
Il genogruppo II invece è predominante nelle infezioni ospedaliere e nelle case di cura (73%) ed è raro nelle infezioni da ingestione di molluschi o legate alle acque contaminate.
La continua ed alta circolazione di questo genogruppo nella popolazione potrebbe essere spiegata invece da una trasmissione soprattutto per contatto diretto tra persona e persona (Bon et al., 2005). Anche altri studi confermano la predominanza del GII in tutto il mondo con una prevalenza del 70-90% in Germania, Olanda, Francia, Regno Unito, Canada e Stati Uniti (Bon et al., 2005; Ike et al., 2006). Allʼinterno del GII, il genotipo GII.4, responsabile delle epidemie europee del 2002 e 2004, dal 2002 sembra essere il genotipo predominante (Ike et al., 2006; Vainio and Myrmel, 2006). Questo sembrerebbe attribuibile allo sviluppo di nuove varianti di questo genogruppo che avrebbero acquisito uno o più vantaggi rispetto a quelle precedentemente circolanti. Uno di questi vantaggi potrebbe essere dato da un più
!
"#! ampio range di legame agli antigeni di istocompatibilità di questo genotipo, oltre che da altri fattori che potrebbero includere una più alta stabilità delle particelle e quindi una maggior capacità di sopravvivere fuori dallʼospite, un alto tasso di replicazione e altri fattori che comunque vanno ulteriormente investigati (Siebenga et al., 2008).
Numerose caratteristiche dei NoV, quali lʼalta infettività, lʼestrema stabilità, la resistenza ai comuni disinfettanti e la capacità di causare malattie invalidanti, hanno portato al loro inserimento nella categoria B della classificazione dei bioagenti. (Karst, 2010).
Non è ancora ben chiaro il ruolo giocato dagli animali come serbatoio, tuttavia la presenza e la diffusione di ceppi ricombinanti di Bovine Norovirus (BoNov) e Porcine Norovirus (PoNoV) pone lʼattenzione non solo sulle infezioni umane ma anche su quelle animali (Costanti et al., 2006).
Nel periodo 2000-2010 il consumo di molluschi bivalvi è stato allʼorigine di numerosi focolai di gastroenteriti da NoV in Europa (Le Guyader et al., 2006; Nenonen et al., 2009). Tra marzo 2006 e marzo 2010 (Baert et al., 2009) attraverso il “Rapid Alert System for Food and Feed” (RASFF) sono state effettuate 19 notifiche di allerta relative alla presenza di Norovirus in ostriche. La maggior parte dei casi (17 su 19) si è verificata nel periodo compreso tra gennaio ed aprile, in cui si ha un aumento dei consumi legato ad unʼelevata qualità del prodotto (https://webgate.ec.europa.eu/rasff-window/portal/).
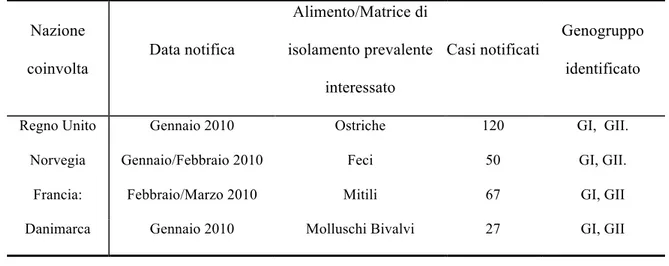
Nel 2010 diversi paesi Europei sono stati interessati contemporaneamente da focolai di gastroenterite da Norovirus. Da gennaio a marzo 2010, lʼEuropean Centre for Disease Prevention and Control (CEPCM) è stato infatti informato attraverso il “Food
and Waterborne Diseases and Zoonoses Surveillance Network” (FWD) dellʼinsorgenza di focolai legati al consumo di ostriche in 5 paesi UE: Regno Unito (UK), Norvegia, Francia, Svezia e Danimarca. In totale sono stati segnalati 334 casi distribuiti in 65 piccoli gruppi.
Nei diversi episodi è stata rilevata la presenza di Norovirus GI e GII, riscontro comune in focolai correlati a consumo di ostriche, che riflette una contaminazione ambientale (Le Guyader et al., 2006; Bon et al., 2005; Symes et al., 2007). In alcuni focolai Bon et al. (2005) hanno identificato fino a 7 diversi ceppi di norovirus, il che contrasta con la teoria della trasmissione da persona a persona, nei quali generalmente è in causa un genotipo unico.
Studi sulle epidemie correlate al consumo di molluschi bivalvi ed allʼacqua contaminata hanno infatti chiaramente dimostrato che, per singolo evento epidemico, sono stati identificati diversi genogruppi (da uno a sei), mentre negli episodi in cui il contagio avviene per trasmissione diretta è di solito coinvolta una singola variante. Questa contaminazione multipla dei molluschi bivalvi e dellʼacqua, ormai ben documentata, rifletterebbe la contaminazione fecale da NoV (Bon et al., 2005). Anche uno studio effettuato in Italia su molluschi bivalvi venduti al dettaglio nel periodo 2005-2008 ha evidenziato come il 12,1 % dei campioni esaminati presentasse contaminazione da NoV GII, varianti GII.4/2004 e GII.b/Hilversum, circolanti nello stesso lasso di tempo in Italia (Terio et al., 2010).
! "#! Nazione coinvolta Data notifica Alimento/Matrice di isolamento prevalente interessato Casi notificati Genogruppo identificato Regno Unito Gennaio 2010 Ostriche 120 GI, GII.
Norvegia Gennaio/Febbraio 2010 Feci 50 GI, GII. Francia: Febbraio/Marzo 2010 Mitili 67 GI, GII Danimarca Gennaio 2010 Molluschi Bivalvi 27 GI, GII
Tabella 1.2. Diffusione nei diversi paesi europei epidemia del 2010
(http://www.eurosurveillance.org/images/dynamic/EE/V15N12/Norovirus_Fig.jpg)
1.2.6.
A
SPETTI CLINICI.
È dimostrato che per causare l'infezione è sufficiente una dose di 10-100 particelle, ed ad essa segue un periodo di incubazione di 15-50 ore (dose dipendente), con una
Figura 1.11. Rappresentazione dei casi registrati nelle prime settimane del 2010.
(http://www.eurosurveillance.org/images/dynamic/EE/V15N12/Norovirus_F ig.jpg)
media di 12-48 ore (Caul, 1996; Leuenberger et al., 2007). Lʼinfezione può essere asintomatica o sintomatica ma nel secondo caso la malattia presenta una sintomatologia moderata ed autolimitante in 12-60 ore (Caul, 1996; Leuenberger et al., 2007; Straub et al., 2007). I sintomi caratteristici sono vomito, di solito esplosivo, diarrea, crampi addominali, febbre lieve o assente, mal di testa, mialgia e malessere generale (Caul, 1996; Prato, 2004; La Rosa et al., 2007; Leuenberger et al., 2007). La mortalità in assenza di fattori concomitanti è estremamente rara, ma può verificarsi nelle persone immunocompromesse (Caul, 1996; Leuenberger et al., 2007; Straub et al., 2007).
La capacità del NoV di rimanere infettante dopo il passaggio attraverso lo stomaco implica che sia acido resistente (Caul, 1996; Leuenberger et al., 2007). La replicazione virale, infatti, avviene nelle cellule epiteliali del piccolo intestino, come sembrano dimostrare le biopsie effettuate da volontari infettati, dove si è evidenziato allargamento ed appiattimento dei villi, sebbene lʼevidenza diretta della maturazione virale non sia stata ancora messa in evidenza (Caul, 1996; Schreiber et al., 1974; Schreiber et al., 1973; Blacklow et al., 1976; Dolin et al., 1975; Troeger et al., 2009; Karst, 2010). La mucosa dell'intestino mostra alterazioni legate allo stato infiammatorio accompagnate da temporaneo malassorbimento (all'incirca per due settimane). Non sono state messe in evidenza lesioni a stomaco e colon (Blacklow, 1971).
Lo sviluppo di tecnologie molecolari nella diagnosi di questi virus ha, inoltre, messo in evidenza infezioni da NoV in casi di encefalopatia, coagulazione intravascolare, enterite necrotica, sindrome del colon irritabile post-infettiva, convulsioni infantili benigne (Karst, 2010) e, da un recente studio, sembra emergere che potrebbero
!
"#! essere coinvolti come concausa nello sviluppo della sindrome di Crohn (Simmons, 2010).
La risposta immunitaria al virus è complessa e non del tutto nota: alcuni individui sviluppano gastroenterite, alcuni sviluppano un'infezione asintomatica mentre altri non risultano infetti pur essendo stati esposti al virus (Lopman et al., 2008).
Questo indicherebbe il ruolo dell'immunità acquisita così come della resistenza innata all'infezione (Lopman et al., 2008).
Nella suscettibilità giocano infatti un ruolo importante anche fattori genetici dell'ospite, che determinano la presenza/assenza di recettori virali. Questi recettori, antigeni del gruppo dellʼistocompatibilità, mostrano patterns di legame specifici/ceppo virale, determinando l'abilità del virus ad infettare le cellule dell'ospite (Siebenga et al., 2008).
Esiste un'immunità a breve termine che dura da giorni a settimane, mentre non è nota un'immunità a lungo termine dopo una singola infezione (Leuenberger et al., 2007). La frequente esposizione può stimolare un'immunità duratura, ma anche i fattori genetici sembrano giocare un ruolo importante. Gli anticorpi preesistenti non sarebbero in grado di proteggere dalla reinfezione con lo stesso genotipo (Siebenga et al., 2008). Gli alti livelli di diversità genetica tra i differenti genogruppi e persino tra i genotipi dello stesso genogruppo, che derivano dall'alto tasso di mutazioni e da eventi di ricombinazione, contribuiscono all'ampio grado di diversità antigenica (Siebenga et al., 2008). La diversa risposta immunologica al patogeno sembra comunque trovare spiegazione in un'immunità di tipo locale più che con la produzione di anticorpi circolanti (Leuenberger et al., 2007).
1.2.7.
M
ETODI DI RILEVAZIONE E DIAGNOSI.
Particelle virali di NoV furono per la prima volta rilevate nel 1972 mediante immuno-elettromicroscopia a trasmissione (TEM) e per lungo tempo tale metodica è stata accettata come “gold standard method” per la diagnosi di NoV (Rabenau et al., 2003). I vantaggi della microscopia elettronica per la visualizzazione diretta delle particelle virali nei campioni sono il rilevamento di qualsiasi agente virale presente in concentrazione sufficiente (almeno 105 - 106 particelle/ml) ed una rapida diagnosi (3 ore dal ricevimento del campione al laboratorio). Dʼaltra parte, oltre a richiedere una strumentazione specifica e costosa ed un operatore specializzato che spenda un tempo considerevole per campione, la sua bassa sensibilità può ostacolare la diagnosi, oltre a non consentire lʼidentificazione oltre il livello di genere (Rabenau et al., 2003).
Il test ELISA (Enzyme-Linked Immunosorbent Assays) per la diagnosi di NoV in campioni fecali è stato messo a punto nel 1985 da Gary e colloboratori; da allora, sono stati prodotti diversi kit commerciali come il NoroCheck™ ELISA Kits (Kim Laboratories Inc.), lʼIDEIA Norwalk-like virus (Dakocytomation Ltd., Ely, United Kingdom) e il Ridascreen Norwalk-like virus enzyme immunoassay (R-Biopharm AG, Darmstadt, Germany). In uno studio condotto per comparare questi ultimi due kit alla RT-PCR è stata dimostrata una sensibilità rispettivamente del 38% e 36% ed una specificità del 96% e 88% nei confronti dei NoV (de Bruin et al., 2006), mentre un altro studio riporta per lʼIDEIA Norwalk-like virus una sensibilità del 55,5% e una specificità del 98,3% sempre rispetto alla RT-PCR (Richards et al., 2003). Il rilevamento dei NoV nelle feci mediante lʼELISA è una metodica veloce (di solito 6 ore e meno di un giorno lavorativo), economica e di facile esecuzione ed è quindi
!
"#! utilizzabile per screening di un ampio numero di campioni (Rabenau et al., 2003). A causa della bassa sensibilità però, i campioni fecali negativi al test ELISA dovrebbero essere ritestati mediante RT-PCR (de Bruin et al., 2006). I limiti di questa metodica includono il basso limite di rilevamento (104 - 105 particelle di virus per ml di campione), quindi sono utilizzabili nelle fasi acute e nei primi giorni di malattia, e limiti legati alla disponibilità di antigene virale naturale per testare i sieri e sviluppare i reagenti (Moe et al., 2004). Con lo sviluppo di particelle NoV-like ricombinanti sono stati fatti molti progressi nello sviluppo di metodiche Enzyme Immunoassay per il rilevamento di immunoglobuline A (IgA), IgG e IgM nel siero e IgA nelle feci, ed è stata messa a punto anche una metodica per il rilevamento di IgA e IgG nella saliva (Moe et al., 2004).
1.2.8.
M
ETODI COLTURALI.
La ricerca sui NoV, incluso lo sviluppo di strategie di prevenzione e controllo, è stata ostacolata dallʼimpossibilità di far crescere questi virus su colture cellulari se non mediante ardui sforzi (Wobus et al., 2005). Staub e collaboratori sono riusciti a propagare in coltura cellulare NoV GI e GII su un modello di epitelio intestinale umano differenziato utilizzando una linea cellulare embrionale umana di epitelio intestinale fatta crescere in 3 dimensioni su biglie porose ricoperte di collagene I e simulando lʼambiente fluido in un bioreattore “Rotating wall-vessel” (Straub et al., 2007). In conclusione le conoscenze sulla coltivazione dei NoV non permettono al momento attuale di utilizzare questa metodica per la rilevazione del virus.
1.2.9.
M
ETODI DI RILEVAMENTO MOLECOLARE.
Il sequenziamento e lo studio dellʼorganizzazione del genoma dei NoV umani risalgono agli inizi degli anni 90 (Lambden et al., 1993; Matsui et al., 1991; Xi et al., 1990). Il clonaggio di larghe porzioni del genoma dei NoV e la disponibilità di sequenze nucleotidiche per le regioni genomiche che codificano lʼRNA polimerasi (RdRP o POL) (Xi et al., 1990) e una proteina immunogenica (IP) (Matsui et al., 1991), la proteina del capside codificata dalla regione ORF2, hanno aperto la strada al rilevamento dei NoV mediante ibridizzazione con sonde cDNA, e allo sviluppo di metodiche più sensibili per il rilevamento dei NoV nei campioni fecali, come la RT-PCR (De Leon et al., 1992).
Già dai primi studi, sebbene si utilizzasse una metodica di PCR classica e rilevamenti mediante sonde marcate o gel dʼagarosio, la RT-PCR aveva dimostrato una sensibilità maggiore rispetto alle metodiche fino ad allora utilizzate per la diagnostica dei NoV nei campioni fecali (De Leon et al., 1992; Matsui et al., 1991; Xi et al., 1992). In quegli anni è stata messa a punto anche la metodica di RT-PCR per il rilevamento di NoV nelle ostriche mediante PCR con i primer della regione RdRP e rilevamento in gel dʼagarosio (Atmar et al., 1993). La chiave per unʼefficiente reazione di RT-PCR è il trovare sequenze conservate da usare come primer ma la grande diversità delle sequenze del genoma dei NoV lʼhanno resa una sfida impegnativa (Kageyama et al., 2003). Nel 2003 Kageyama e collaboratori hanno messo a punto una Real Time RT-PCR quantitativa per i NoV in campioni fecali utilizzando la sequenza della giunzione ORF1-ORF2 che sembra essere la regione maggiormente conservata e che permette una genotipizzazione dei genogruppi GI e GII mediante il sequenziamento dei prodotti di amplificazione della PCR (Kageyama et al., 2003). Nello stesso studio,
!
"#! confrontando le diverse metodiche precedentemente utilizzate, è stata rilevata una sensibilità del 99% per la Real Time RT-PCR quantitativa con i primer disegnati sulla giunzione ORF1-ORF2, del 77% con la RT-PCR convenzionale per RdRp e dellʼ83% per la regione del capside N/S (Kageyama et al., 2003). Ad oggi diversi gruppi di ricercatori hanno messo a punto diversi protocolli di Real Time RT-PCR quantitativa per la ricerca di NoV in feci (Kageyama et al., 2004; Schmid et al., 2004; Ueki et al., 2007), in campioni ambientali (da Silva et al., 2007; Hewitt et al., 2007) ed alimentari (Jothikumar et al., 2005; Loisy et al., 2005; Myrmel et al., 2004; Nishida et al., 2003; Nishida et al., 2007; Oogane et al., 2008), e sono stati fatti studi comparativi di alcuni di questi protocolli (La Rosa et al, 2007) ma non vi è ancora uniformità internazionale sulle metodiche utilizzate. Lo stato dellʼarte per la diagnostica dei NoV mediante PCR vede studi per la messa a punto di Multiplex (Triplex) Real Time, per il rilevamento e la distinzione di NoV GI, GII e GIII in campioni fecali e ambientali mediante primer disegnati sulla giunzione ORF1-ORF2 (Wolf et al., 2007), la combinazione di tecniche immunomagnetiche associate alla Real Time RT-PCR per il rilevamento dei NoV nel cibo (Park et al., 2008), e lo sviluppo di Loopamp (loop-mediated isothermal amplification) kit per il rilevamento dei NoV in campioni fecali (Iturriza-Gòmara, 2008).
1.3. HAV.
1.3.1.
I
L VIRUS.
Il virus dellʼEpatite A (HAV) (figura 1.1.) appartiene alla famiglia Picornaviridae ed è stato classificato come unico rappresentante del genere degli Hepatovirus (Francki et al., 1991; Costa-Mattioli et al., 2003).
!
!
Questo virus ha dimensioni di circa 27-32 nm, capside icosaedrico, privo di envelope, a simmetria cubica, formato da 32 subunità o capsomeri e contiene un singolo filamento di RNA a polarità positiva di circa 7,5 kb (Koff, 1998). Come tutti i Picornavirus, il suo genoma può essere diviso in tre distinte regioni (figura 1.13.):
• Una regione non codificante allʼestremità 5′ (5ʼNTR) che costituisce circa il 10% del genoma a cui è covalentemente legata una proteina virale (VPg) che interviene nella replicazione. Tale regione presenta numerose strutture secondarie che contengono segnali per il riconoscimento e per il legame da parte dei ribosomi dellʼospite al genoma virale. Inoltre i nucleotidi compresi tra 1 e 150 sembrerebbero necessari per lʼinizio della sintesi di nuovo RNA virale (formazione del complesso replicativo).
• Un lungo open reading frame (ORF) che codifica tutte le proteine virali.
• Una corta regione non codificante allʼestremità 3′ (3ʼNTR), seguita da una coda poli-A, di 40-80 nucleotidi (Nainan et al., 2006).
!
"#! Le funzioni delle proteine e dei polipeptidi dellʼHAV sono mostrate nella tabella 1.3. La regione 5ʼNTR risulta la più conservata dellʼintero genoma ed è stata molto utilizzata per sviluppare la maggior parte dei metodi di analisi proposti per la diagnosi in differenti substrati ambientali e clinici (Sanchez et al., 2007; Nainan et al., 2006). La meno conservata è la regione codificante per la proteina virale 1 (VP1) che per questo motivo è stata ampiamente studiata per comprendere lʼevoluzione molecolare e per definire la distribuzione geografica dellʼHAV (Robertson et al., 1992).
La genotipizzazione dellʼHAV viene effettuata comparando la percentuale di identità delle basi nella congiunzione tra la VP1 e la poliproteina non strutturale 2A (VP1/2A) del genoma anche se, secondo alcuni autori, la comparazione di solo questo tratto (circa 168 basi) non è sufficiente; inoltre dovrebbe essere confrontato tutto il gene per comprendere lʼevoluzione molecolare dellʼHAV (Costa-Mattioli et al., 2002).
Tabella 1.3. Funzioni delle proteine e dei polipeptidi del virus dellʼEpatite A (Nainan et al., 2006).
!
"#! Considerando lʼidentità delle basi nella congiunzione VP1/2A sono stati identificati sette genotipi (I-VII), diversi per più del 15% della sequenza genomica come riportato in figura 1.14. I Genotipi I e III sono stati suddivisi in due subgenotipi che sono diversi per non più del 7,5% delle posizioni delle basi.
Alla variabilità a livello nucleotidico non consegue un equivalente grado di variazione a livello amminoacidico. Grazie a questo basso livello di diversità antigenica tutti i virus di HAV sono dello stesso sierotipo e lʼimmunità ottenuta a seguito di unʼinfezione, anche asintomatica, persiste tutta la vita verso tutti i genotipi umani.
I primati risultano i soli ospiti naturali dellʼEpatite A: i virus appartenenti ai genotipi I, II, III e VII sono stati isolati nellʼuomo, mentre quelli appartenenti ai genotipi IV, V e VI sono stati isolati in scimmie in cattività che mostravano una malattia simile allʼEpatite umana (Robertson et al., 1992; Costa-Mattioli et al., 2003; Arankalle et al., 2009).
1.3.2.
M
ODALITÀ DI INFEZIONE.
Il virus penetra nella cellula ospite per endocitosi (Morace, 2000) dopo essersi legato al recettore specifico sulla membrana cellulare (figura 1.15.).
!
"#! L'RNA viene tradotto nella lunga poliproteina che successivamente viene tagliata dalla proteinasi virale (Gauss-Muller et al., 1984).
Parte dell'RNA virionico viene usato come stampo per la replicazione che avviene attraverso la sintesi intermedia di un RNA a polarità negativa, su cui viene poi sintetizzato lʼRNA virionico, utilizzando la polimerasi virale 3D e la proteina VPg, in un complesso replicativo a livello delle membrane cellulari. Nel complesso replicativo intervengono anche le proteine 2B, 2C, 3AB e 3C (o 3ABC). (Gauss-Muller et al., 1984)
Le proteine strutturali si assemblano per formare un capside che avvolge l'RNA. Le particelle virali complete vengono liberate all'esterno della cellula in vescicole citoplasmatiche che si rompono, rilasciando il virus, quando vengono a contatto con i sali e gli acidi biliari. Durante la replicazione virale non si osserva il blocco della sintesi proteica cellulare e tutte le sue fasi non provocano danni visibili alla cellula, contrariamente a quanto avviene nel caso degli altri Picornavirus (Gauss-Muller et al., 1984).
1.3.4.
E
PIDEMIOLOGIA.
Anche se in teoria una sola particella virale è in grado di determinare l'infezione, lo sviluppo di uno stato di malattia clinica e il suo decorso dipendono da numerosi altri fattori, quali lo stato immunitario, lʼetà dell'ospite, le eventuali patologie intercorrenti e la virulenza del microrganismo. Lʼingestione di alimenti contaminati o, direttamente, ma più raramente, di microscopiche particelle di materiale fecale provenienti dai malati, sono i modi più comuni con cui si determina e si trasmette lʼinfezione (Lemon, 1997; Tjon et al., 2005).
La relazione tra lʼetà in cui avviene lʼinfezione e lʼespressione clinica della malattia è caratteristica dellʼepidemiologia dellʼHAV: in pratica, aumentando la prima (lʼetà) aumenta anche la seconda (lʼespressione clinica). Tra i casi di Epatite A riportati in USA, la percentuale di quelli mortali aumenta dallo 0,2% tra i bambini di 5-14 anni allʻ1,8% tra gli adulti di oltre 50 anni. (Guidance for the Prevention and Control of Hepatitis A Infection, HPA, 2009)
LʼHAV ha una distribuzione mondiale (figura 1.16.) ed ogni anno si manifestano circa 1,5 milioni di casi clinici di Epatite A nel mondo (CDC, 2002).
I Paesi Nordici mostrano la percentuale più bassa dʼinfezione con unʼincidenza di circa il 15%. In Nord America ed in Europa una piccola parte della popolazione viene infettata in età infantile, mentre la maggioranza rimane suscettibile durante lʼetà Figura 1.16. Endemicità (molto bassa, bassa, intermedia, medio alta ed alta) dellʼHAV nel mondo (CDC, 2002).
!
"#! adulta (CDC, 2002). Nel Nord America le epidemie colpiscono soprattutto giovani adulti, ma anche bambini delle più basse classi socio-economiche; sono state anche riportate epidemie tra gli omosessuali e tra coloro che fanno uso di stupefacenti. (Tjon et al., 2005; Ballesteros et al. ,1996; Villano et al., 1997)
Lʼepidemiologia dellʼHAV negli Stati Uniti è fondamentalmente cambiata grazie allʼutilizzo del vaccino: prima della sua distribuzione (iniziata nel 1995-1996) lʼincidenza dellʼEpatite A era stata prevalentemente ciclica, con picchi che si ripetevano ogni 10-15 anni. Negli Stati Uniti durante il periodo 1980-1995 sono stati riportati annualmente dal Centers for Disease Control (CDC) approssimativamente da 22.000 a 36.000 casi di Epatite A (9,0-14,5 casi per 100.000 abitanti), ma studi sullʼincidenza della malattia nella popolazione indicano che il numero effettivo dʼinfezioni dovrebbe essere ben più alto. Si stima che il numero medio di infetti per anno sia stato di oltre 270.000 durante il periodo 1980-1999, ossia circa 10 volte il numero di casi denunciato. LʼAdvisory Committee on Immunization Practices stima che ogni anno negli Stati Uniti 100 persone muoiano a causa di complicazioni epatiche attribuite allʼinfezione da HAV (ACIP, 2006).
Lʼepidemiologia dellʼEpatite A nei Paesi in via di sviluppo è differente da quella dei Paesi industrializzati; nei primi infatti lʼincidenza della malattia negli adulti è relativamente bassa a causa dellʼesposizione in età infantile e la maggior parte degli individui sopra i diciotto anni mostra unʼimmunità che persiste tutta la vita. In Africa, in America Latina ed in parte dellʼAsia, la maggioranza delle infezioni si verificano in età infantile e molti paesi di queste aree continentali sono caratterizzati da unʼalta endemicità. (Tufenkeji 2000; Lee et al., 2008; Barzaga, 2000; Tanaka, 2000)
In alcuni paesi asiatici, essendo migliorati sia gli standard igienici che le condizioni socio-economiche, si è avuta una riduzione dellʼesposizione verso lʼHAV nei bambini
con diminuzione dellʼendemicità. In altre aree in cui le condizioni socio-economiche non sono molto migliorate, lʼendemicità rimane invece medio-alta (David, 2004). Nei paesi del Sud-Est asiatico le modalità di origine e di diffusione delle epidemie di HAV sono alquanto differenti nei diversi stati, con frequenti variazioni regionali allʼinterno di essi. Le epidemie si originano più frequentemente attraverso alimenti e acqua contaminati e comunemente avvengono in scuole, centri di soggiorno, ospedali, ristoranti (Barzaga, 2000).
Tabella 1.4. Tassi annuali/100.000 per età sesso ed area geografica delle epatiti virali acute nel 2007 (SEIEVA, 2003-2008).
!
""! Lʼinfezione da HAV rappresenta la forma di epatite più diffusa in Italia (tabella 1.5.) ove ancora oggi rappresenta un importante problema di sanità pubblica poiché, anche se lʼendemia è progressivamente diminuita, in questi ultimi anni vi sono state aree nelle regioni meridionali (Campania, Puglia e Sicilia) in cui si sono periodicamente ripetute epidemie (ad intervalli di due – quattro anni).
Nel 1984 presso l'Istituto Superiore di Sanità (ISS) è stato creato il Sistema Epidemiologico Integrato dellʼEpatite Virale Acuta (SEIEVA), un sistema informativo grazie al quale è possibile una valutazione dell'incidenza delle epatiti acute in Italia e la messa a punto di idonee strategie di prevenzione. Lʼattività di sorveglianza del SEIEVA ha avuto un ruolo rilevante nellʼidentificazione delle modalità di trasmissione dei virus epatici e nel controllo di focolai epidemici grazie ai vari soggetti che partecipano attivamente alla sorveglianza: lʼIstituto Superiore di Sanità, le Regioni e le ASL.
Per calcolare i tassi dʼincidenza il SEIEVA impiega le notifiche inviate dalle Regioni e dalle ASL utilizzando un questionario “ad hoc" per elaborare i dati clinici ed i fattori di rischio. I tassi dʼincidenza indicano negli ultimi anni una tendenza in diminuzione dei casi notificati, essendo passati da un valore di 10 per 100.000 nel 1985 ad un valore di 2 per 100.000 nel 2007 (SEIEVA 2007) (tabella 1.5.). Nel corso del 1997 a causa dellʼepidemia verificatasi in Puglia, la notifica dei casi di Epatite A ha evidenziato un tasso di incidenza particolarmente elevato (19 per 100.000) (Germinario et al., 2000; Chironna et al., 2002).
Tabella 1.5. Tassi di incidenza (x 100.000) dellʼepatite A per fasce di età nel periodo 1985-2007. (SEIEVA, 2003-2008).
La fascia di popolazione maggiormente colpita risulta quella degli adulti, in cui la più probabile fonte di esposizione allʼHAV è rappresentata dal consumo di frutti di mare e da viaggi in aree endemiche (tabella 1.6.).
Tabella 1.6. Casi notificati di Epatite A con fattore di rischio identificato. (SEIEVA, 2003-2008)
!
"#!
1.3.5.
P
ATOGENESI E SINTOMATOLOGIA.
La probabilità di manifestare sintomi clinici aumenta con lʼetà in cui avviene lʼinfezione, infatti la malattia risulta più lieve nei bambini piuttosto che negli adulti, fino a diventare più severa nelle persone anziane, nelle quali lʼospedalizzazione e lʼesito infausto diventano più probabili (Ciocca, 2000).
Nei bambini di età inferiore ai 6 anni la maggior parte delle infezioni decorre in modo asintomatico e, nel caso si presentino i sintomi, di solito sono lievi e non specifici (Mayer et al., 2010). Nei bambini più grandi e negli adulti, lʼinfezione è invece di solito sintomatica, con il caratteristico ittero che si presenta nella maggior parte dei casi. (Mayer et al., 2010; Abarca et al., 2011)
Allʼinfezione segue un periodo dʼincubazione variabile tra 2 ed 8 settimane (Glikson et al., 1992; Sjogren et al., 1987), al cui termine si manifesta una fase prodromica di 2-10 giorni con caratteristici sintomi clinici: malessere, nausea, anoressia, astenia, mialgie, cefalea, febbre moderata, fino a pervenire alla fase conclamata, caratterizzata dallʼiperbilirubinemia, dallʼittero e dallʼemissione di urine color marsala e di feci chiare (epatite itterica) (CDC, 2009; eMedicineHealth, 2007).
Le lesioni anatomiche consistono in genere in una congestione epatica, con moderata epatomegalia e fenomeni degenerativi parcellari degli epatociti, fino alla necrosi cellulare di questi ultimi. La bilirubina invade il circolo ematico anziché defluire normalmente nelle vie biliari. Le cellule istiocitarie dei sinusoidi epatici aumentano di numero e di volume. Le transaminasi sieriche aumentano fino al punto da raggiungere, durante la fase itterica, valori molto elevati proprio a causa della necrosi delle cellule epatiche infette e del passaggio di enzimi epatici nel torrente ematico. (CDC, 2009; eMedicineHealth, 2007) Trascorse 3-4 settimane le
transaminasi e la bilirubina tornano ai valori normali ed i sintomi si attenuano fino a scomparire (eMedicineHealth, 2007).
Alcune volte lʼepatite acuta può provocare una necrosi massiva del fegato e pertanto il danneggiamento consistente delle funzioni epatiche con la successiva morte del malato. Questa forma di epatite, detta fulminante, risulta molto rara e si verifica in meno dellʼ1,5% dei casi di epatite acuta (Akriviadis and Redeker, 1989; Previsani et al., 2004).
Tuttavia in molti altri casi, al termine del periodo di incubazione, la malattia evolve in forma anitterica o del tutto inapparente (Previsani et al., 2004; Hadler et al., 1980), come si può notare dalla tabella 1.7., in cui vengono mostrati i possibili esiti di unʼinfezione da HAV.
Dalla figura 1.17. si può notare che lʼHAV compare nel sangue dei pazienti e nelle feci circa due settimane dopo lʼinfezione e prima della comparsa dei sintomi clinici (v. Tabella 1.7. Possibili esiti di unʼinfezione da HAV (SEIEVA, 2003-2008)
!
"#! anche tabella 1.8.) (Brundage et al., 2006). Le feci sono molto infettanti poiché contengono unʼaltissima quantità di virus (fino a 109 virioni/g) per circa 2 settimane dopo la comparsa dei sintomi clinici (Skinhoj et al., 1981; Tassopoulos et al., 1986).
1.3.6.
M
ETODI DI RILEVAZIONE E DIAGNOSI.
LʼHAV è stato per la prima volta rilevato in estratti di feci al microscopio elettronico utilizzando un antisiero omologo (Feinstone et al., 1973) e particelle virali dello stesso tipo sono state osservate nel siero e nel fegato di persone infettate sperimentalmente con HAV (Provost et al., 1975). Lʼantigene HAV è stato rilevato nei campioni fecali, colture cellulari e campioni ambientali utilizzando test radioimmunologici ed immunoenzimatici (Hollinger et al., 2001; Moritsugu et al., 1976). Tuttavia lʼidentificazione dellʼantigene nel sangue è stata difficile (Hollinger et al., 2001), in quanto la fibronectina ha la possibilità di legarsi allʼHAV e mascherare i determinanti antigenici necessari per il rilevamento immunologico (Seelig et al., Tabella 1.8. Dettaglio degli eventi clinici che si manifestano nel corso dellʼinfezione da HAV (SEIEVA, 2003-2008).
!
"#! 1984). I polipeptidi del capside dellʼHAV e lʼRNA virale sono stati rilevati in complessi di IgM circolanti isolati da scimpanzé infettati sperimentalmente (Margolis et al., 1988).
La risposta immunitaria umorale gioca un ruolo fondamentale nella diagnosi dellʼinfezione da HAV e nella differenziazione dellʼepatite A dagli altri tipi di epatite virale. Ad oggi sono disponibili in commercio una serie di test per la rilevazione delle
IgM, che compaiono circa 4 settimane dopo lʼinfezione e persistono per molti mesi,
(Cuthbert 2001; Dignani et al., 2003; Jindal et al., 2002; Poddar et al., 2002; Margolis et al. 1988; Stapleton 1995), e delle IgG, che compaiono nel sangue poco dopo le IgM e permangono per tutta la vita (Skinhoj et al. 1977). Gli anticorpi IgG sono protettivi contro tutti i ceppi di HAV, dal momento che esiste solamente un sierotipo del virus (Skinhoj et al. 1977).
Le IgM, IgA e IgG anti-HAV sono solitamente presenti al momento della comparsa dei sintomi. Dal momento che l'epatite dovuta allʼinfezione da HAV è clinicamente indistinguibile dalla malattia causata da altri virus dell'epatite (HBV, HCV, HDV e
HEV), si sono resi necessari test sierologici per fare la diagnosi (Stapleton, 1995).
Dal punto di vista diagnostico, le IgM anti-HAV sono state utilizzate come indicatori
primari dellʼinfezione acuta (Cuthbert, 2001); si tratta principalmente di anticorpi rivolti
contro le proteine del capside. Sono stati valutati diversi metodi per la rilevazione di
questo specifico virus, tra cui tecniche radioimmunologiche (Purcell et al., 1976),
colorazione immunochimica (Huang et al., 1979), test ELISA (enzyme-linked
immunosorbent assay) (Delem, 1992), immunoblotting (Wang et al., 1996) e dot blot
immunogold filtration (Shao et al., 2003). In commercio sono disponibili test ELISA
I saggi diagnostici disponibili in commercio sono configurati in modo tale che, sebbene gli anticorpi IgM possano essere presenti per un lungo periodo di tempo, le minori concentrazioni, rilevate da 4 a 6 mesi dopo lʼinizio dellʼinfezione, non
producono un risultato positivo (Stapleton, 1995). Inoltre questi test rilevano anticorpi
IgM anti-HAV solo per un breve periodo di tempo in persone a cui è stato
recentemente somministrato il vaccino per lʼepatite A (Wiedmann et al., 2003).
In precedenza lʼinfezione da HAV era diagnosticata mediante il rilevamento di anticorpi IgG anti-HAV, mentre i saggi attualmente disponibili in commercio rilevano gli anticorpi totali anti-HAV (IgG e IgM). La presenza di anticorpi totali anti-HAV e l'assenza di IgM anti-HAV può essere utilizzata, infatti, per distinguere tra infezioni pregresse e recenti.
Anticorpi diretti verso proteine strutturali del capside sono prodotti a seguito di immunizzazione con il vaccino. Solo una piccola percentuale (dallʼ8 al 20%) delle
persone vaccinate ha una risposta transiente di IgM anti-HAV (Lemon, 1993;Shouval
et al., 1993; Sjogren et al., 1991) mentre IgG anti-HAV sono prodotte da tutte le persone immunizzate con successo (Lemon, 1993; Shouval et al., 1993; Sjogren et al., 1991). Tuttavia, sempre che non siano modificati, i test commerciali disponibili per gli anticorpi totali anti-HAV non sono abbastanza sensibili da rilevare la concentrazione di anticorpi in una percentuale significativa di persone immunizzate,
!
"#!
1.3.7.
M
ETODI COLTURALI.
LʼHAV è stato coltivato in diversi tipi di cellule di origine umana e non, tra cui cellule
di rene della scimmia verde africana (Daemer et al., 1981; Wang et al., 1986) e
cellule fetali renali di scimmia rhesus (Flehmig, 1980). In contrasto con la maggior
parte dei Picornavirus, lʼHAV di origine umana richiede un lungo periodo di adattamento prima di riuscire a crescere in coltura cellulare e, una volta adattato, produce unʼinfezione persistente e si attenua, comʼè stato dimostrato sperimentalmente dal fatto che non provoca più malattia se inoculato nei primati (Feinstone et al., 1983). Inoltre sono prodotte concentrazioni relativamente più basse di virus e di antigene virale rispetto agli altri Picornavirus. Le mutazioni nellʼacido
nucleico virale possono giocare un ruolo importante nellʼadattamento dellʼHAV in
coltura cellulare (Cohen et al., 1989; Emerson et al., 1991; Emerson et al., 1993; Funkhouser et al., 1994) e nella sua attenuazione (Emerson et al., 2002; Feinstone et al., 1983). LʼHAV replica nelle colture cellulari senza segni dʼinfezione con effetto citopatico e senza danni apparenti alla cellula ospite. A causa della mancanza di un effetto citopatico in coltura cellulare, le analisi immunologiche hanno lo scopo di
rilevare lʼantigene HAV (Siegl et al., 1984). I metodi comunemente utilizzati per
quantificare lʼinfettività includono test di radioimmunofluorescenza (Lemon et al.,
1983), test di fluorescenza, test radioimmunologici in situ (Siegl et al., 1984) ed ibridazione in situ (Jiang et al., 1987).
1.3.8.
M
ETODI DI RILEVAMENTO MOLECOLARE.
Le tecniche di rilevamento degli acidi nucleici sono più sensibili rispetto ai test immunologici dellʼantigene virale per il rilevamento dellʼHAV in campioni di diversa origine (ad esempio campioni fecali, ambientali o cibo). LʼHAV è stato rilevato mediante diverse tecniche molecolari, ma lʼamplificazione dellʼRNA virale mediante RT-PCR è attualmente il metodo più sensibile e più utilizzato per la rilevazione dellʼRNA dellʼHAV. La purificazione dellʼRNA virale dai campioni clinici ed ambientali è il primo passo fondamentale in RT-PCR, e spesso sono necessari kit di estrazione differenti a seconda della tipologia del campione da cui si vuole estrarre lʼRNA virale. Uno dei primi protocolli per lʼestrazione dellʼRNA virale da campioni di feci o da siero prevedeva il trattamento con proteinasi K seguito da estrazione fenolo-cloroformio e precipitazione in etanolo (Nainan et al., 1991; Robertson et al., 1991). In seguito sono diventati commercialmente disponibili per lʼestrazione dellʼRNA e degli acidi nucleici totali prodotti che utilizzano guanidinio tiocianato-fenolo-cloroformio (Chomczynski, 1993) che hanno aumentato la sensibilità e la specificità di rilevamento dellʼHAV (de Paula et al., 2003; Hutin et al., 1999; Nainan et al., 2005). Sono state utilizzate sfere magnetiche rivestite con anticorpi anti-HAV (Jothikumar et al., 1998) per separare il virus da potenziali inibitori della trascrittasi inversa e della PCR, i quali si ritrovano molto spesso nei campioni ambientali e fecali. Per il rilevamento di HAV sono stati applicati protocolli di estrazione virale automatizzati che permettono una maggiore velocità di analisi, sono riproducibili ed affidabili ed hanno una sensibilità di rilevazione simile a quella dei metodi di estrazione manuale. (Lee and Prince, 2001; CDC).
!
"#! Lʼefficienza di trascrizione inversa di RNA a cDNA può essere ridotta dalla presenza di inibitori nel materiale di partenza; la messa a punto di trascrittasi inversa senza attività RNasi (RNasi H) ha permesso di aumentare lʼefficienza di estrazione, anche se enzimi non ingegnerizzati continuano ad essere utilizzati per lʼidentificazione di unʼampia varietà di target, incluso lʼHAV, con alta specificità e sensibilità (Nainan et al., 1996; Stahlberg et al., 2004a; Stahlberg et al., 2004b). Anche se gli enzimi termostabili possono migliorare lʼefficienza della RT, riducendo la formazione di strutture secondarie e migliorando lʼappaiamento, non è stato valutato il loro effetto sulla rilevazione di HAV. Primer specifici o casuali possono essere utilizzati come stampo per la reazione di RT (Stahlberg et al., 2004a; Stahlberg et al., 2004b).
La Nested PCR, dove i prodotti ottenuti dal primo step di PCR vengono utilizzati come base per un secondo step di PCR, è stata utilizzata per amplificare HAV da campioni clinici ed ambientali in cui si prevede che la carica virale sia bassa (Bower et al., 2000; Di Pinto et al., 2003; Gilgen et al., 1997; Li et al., 2004; Nainan et al., 1996). Lʼanalisi dei prodotti di PCR con ibridazione con sonda ha anche dimostrato un aumento della sensibilità di rilevazione.
Lo sviluppo di one-step RT-PCR, in cui la trascrizione inversa e la reazione di PCR sono accoppiate, ha notevolmente ridotto i tempi della procedura (Di Pinto et al., 2003; Li et al., 2004). Tuttavia questo metodo sembra ridurre la sensibilità di rilevazione fino a 1 unità logaritmiche rispetto ai due step RT-PCR (Nainan et al., 2006).
Multiplex RT-PCR, in cui sono amplificate le sequenze genomiche di più di un organismo, offre il modo più efficace per rilevare più agenti in campioni clinici ed ambientali rispetto alle tradizionali RT-PCR di amplificazione per ogni singolo agente.
Questo metodo è stato sviluppato per la rilevazione simultanea di HAV e HEV e di rotavirus, poliovirus e HAV.
1.4. I
MOLLUSCHI COME VEICOLO Dʼ
INFEZIONE DEI VIRUS ENTERICI.
I bivalvi o lamellibranchi sono molluschi appartenenti alla classe Bivalvia e si nutrono mediante la filtrazione di ampi volumi di acqua; in questo modo possono concentrare fitoplancton, parassiti, batteri o virus presenti nelle colonne dʼacqua (Rehnstam-Holm and Hernroth, 2005).
I molluschi ed i loro derivati sono responsabili di malattie alimentari in tutto il mondo; la diffusione delle epidemie legate al loro consumo dipende principalmente dalle condizioni climatiche e dalle abitudini alimentari (Scoging, 1992). Si stima, infatti, che siano coinvolti in epidemie legate al cibo per il 2% in Gran Bretagna, per lʼ11% negli Stati Uniti, per il 20% in Australia e per il 70% in Giappone, dove è grande tradizione il consumo di frutti di mare crudi (Scoging, 1992). LʼEpatite A è lʼinfezione più diffusa legata al consumo di bivalvi, ma negli ultimi sono state rilevate altre forme gastroenteriche ad eziologia virale come le infezioni da Adenovirus sierotipo 40 e 41 e da Norovirus (NoV) (Rehnstam-Holm and Hernroth, 2005). La clorazione per la disinfezione delle acque di scarico non risolve il problema, in quanto i NoV e lʼHAV sono in grado di resistere alle elevate concentrazioni di cloro, che inattivano invece le cellule vegetative batteriche (Rippey, 1994).
Essendo ben chiaro a livello comunitario che lʼuso di Escherichia coli come indicatore è un metodo insufficiente e che questi standard batterici non danno garanzie sufficienti rispetto alla presenza di virus e di batteri del genere Vibrio