Enzimi che utilizzano corismato come target multiplo di un singolo inibitore: verifica di un modello a tre fasi
La resistenza antibiotica ai farmaci, oggi un problema globale della sanità, è aumentata notevolmente negli ultimi decenni [1]. Il tasso di infezioni da Staphilococcus aureus (MRSAα ) resistenti alla meticillina è raddoppiato tra il 2000 e il 2005. Negli Stati Uniti, queste infezioni, causano più morti dell’AIDS. La bassa redditività e il breve arco di vita funzionale degli antibiotici ha avuto come risultato la costante diminuizione nella ricerca e nello sviluppo da parte dell’industria. Di conseguenza, solo due classi di antibiotici sono stati introdotte dal 1967: gli oxazolidinoni (2000) e i lipopeptidi (2003) [2].
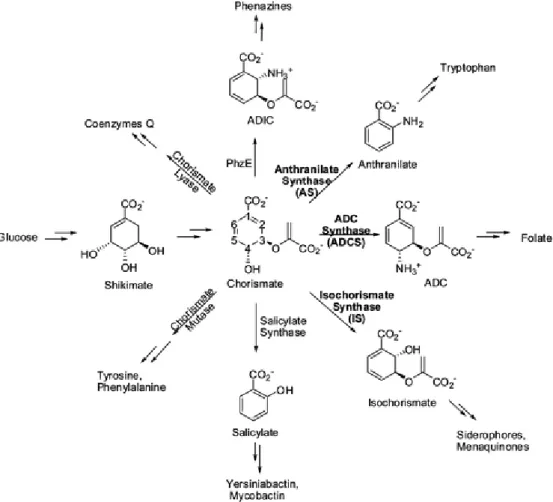
Alcuni autori hanno sviluppato un approccio semplificativo per scoprire potenziali “lead” antimicrobici: con il metodo “one-bed-one-compound” (OBOC), sono state create vaste librerie combinatoriali con enzima marcato a fluorescenza [3]. E’ stata progettata una piccola (2304 elementi) libreria, “proof-of-concept”, basata su peptidi per esaminare un campione di enzimi omologhi che utilizzano il corismato: la 4-amino-4-deossicorismato sintasi (ADCS), la isocorismato sintasi (IS), l’antranilato sintasi (AS), e la salicilato sintasi (SS). ADCS, IS, AS, e SS catalizzano i primi step nella formazione di folato, enterobactina (un composto chelante del ferro, i.e. sideroforo), triptofano e
micobactina (un sideroforo da Mycobacterium tuberculosis), rispettivamente nei batteri e nei parassiti apicomplexa (Figura 1).
Figura 1: Il corismato è un composto centrale nel metabolismo di batteri, piante, funghi e parassiti apicomplexa. Gli enzimi che utilizzano il corismato sono essenziali per la sopravvivenza e la virulenza di questi organismi.
Gli enzimi che utilizzano corismato sono ottimi target farmacologici antimicrobici per diverse ragioni: (1) catalizzano il primo importante step nella formazione di numerosi metaboliti critici per la sopravvivenza e/o virulenza (Figura 1), (2) sono strutturalmente e meccanicisticamente omologhi, suggerendo così la possibilità che un farmaco possa inibire molteplici vie metaboliche, (3) sono presenti solo nei batteri, nelle piante, nei funghi e nei parassiti apicomplexa (e.g., Plasmodium, Toxoplasma, e Cryptosporidium) e assenti negli umani.
Inoltre, questi enzimi e le relative vie metaboliche non sono stati pienamente impiegati come obiettivi farmacologici. Un esempio di target riuscito di queste vie metaboliche sono i farmaci sulfamidici che inibiscono la biosintesi del folato attraverso l’inibizione della diidropteroato sintasi (DHPS) che avviene due step a valle di ADCS.
L’omologia strutturale di ADCS, IS, AS, e SS, comprendente l’interno del sito attivo quasi identico, ha suggerito l’obiettivo di sviluppare un composto che avrebbe inibito più di uno di questi enzimi. Il paradigma di inibizione multitarget da parte di un singolo composto è divenuto sempre più importante negli ultimi anni [3]. Questa strategia è stata sfruttata per scoprire trattamenti per malattie quali HIV-AIDS, cancro, depressione, e infezioni da Mycobacterium tubercolosis [4,5]. Un farmaco che inibisca più enzimi sarebbe altamente auspicabile per la sua maggiore efficacia, anche a modeste affinità di legame, dovute all’inibizione di più pathway metabolici che si combinerebbero con una lenta crescita. Inoltre, la maggior difficoltà per lo sviluppo di resistenza primaria (i.e., mutazione degli enzimi target per superare l’inibizione), che richiederebbe numerose mutazioni simultanee e spontanee, rende questa idea interessante. Tuttavia, meccanismi alternativi di resistenza (e.g., pompe di deflusso o modificazioni covalenti) rimarrebbero problematiche.
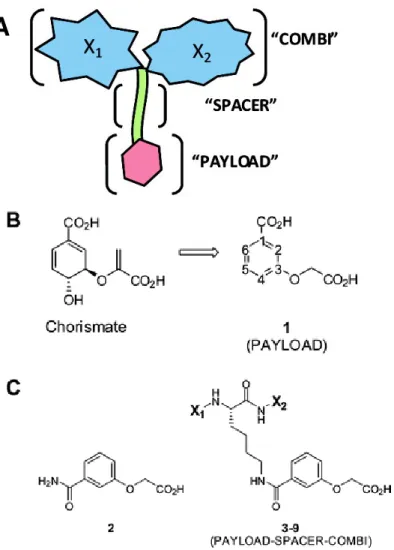
Il modello di inibizione proposto dagli Autori [6], ispirato alle comuni statine, farmaci utilizzati per abbassare il colesterolo, include tre fasi: PAYLOAD, SPACER, e COMBI, come illustrato nella Figura 2. Il PAYLOAD è un’ imitazione di corismato, creata per guidare il composto verso il sito attivo dell’enzima. Lo SPACER collega insieme le fasi PAYLOAD e COMBI e si estende al di fuori del sito attivo verso la regione superficiale delle proteine esposta al solvente. La fase COMBI si ha laddove gli elementi di diversità combinatoria vengono introdotti nella libreria.
Figura 2: (A) La strategia di progettazione di inibitori impiega tre "tappe": PAYLOAD, SPACER e COMBI. X1 e X2 rappresentanot I due elementi di diverstà presenti in ciascun membro della libreria combinatoriale. (B) Il composto 1 PAYLOAD che mima il corismato è stato progettato per dirigere gli inibitori al sito attivo. (C) 2 è stato sintetizzato e saggiato per paragonare direttamente i risultati della inibizione degli inibitori 3−9, elaborati completamente, con il PAYLOAD 1.
Schreiber e collaboratori prima hanno introdotto il concetto di librerie combinatorie condizionate e hanno utilizzato questa tecnica per scoprire ligandi per il dominio SH3 della fosfatidilinositolo 3-chinasi e, in seguito, per la istone deacetilasi [7].
Questa strategia è simile al paradigma di screening farmacologico basato sul frammento, dove i sottocomponenti dell’inibitore finale vengono identificati e ottimizzati individualmente, poi collegati in uno stadio successivo. L’approccio basato sul frammento ha lo svantaggio riconosciuto che il processo di collegamento dei frammenti può essere problematico. Al contrario, il metodo impiegato in questo studio affronta questo ostacolo significativo collegando i frammenti fin dall’inizio. In particolare, viene consentito dallo screening di libreria OBOC ad elevato parallelismo e dalla tecnologia di identificazione “hit” utilizzata in questo approccio.
In un precedente lavoro, lo screening di libreria OBOC che impiegava ADCS nella selezione dell’ hit ha portato all’identificazione e alla caratterizzazione cinetica di sette inibitori di ADCS [3]. I risultati presentati in esso hanno stabilito l’efficacia del paradigma generale per l’inibizione dell’enzima che utilizza corismato basato sulla chimica combinatoria e un progetto d’inibizione a fasi. Inoltre gli stessi autori hanno dimostrato [6] che la fase COMBI interagisce con residui di superficie prossimi al sito attivo e porta a una maggiore affinità di legame per gli enzimi. Inoltre hanno dimostrato il loro obiettivo centrale, inibire più di un enzima con lo stesso composto. I sette inibitori individuati nello screen ADCS, così come i sei loro derivati, sono stati saggiati anche rispetto a IS e AS. Generalmente, gli inibitori caratterizzati in precedenza con ADCS si legano più strettamente a IS e AS. Cinque degli inibitori mostrano inibizione micromolare di ADCS, IS, e AS, e il miglior composto del binding complessivo inibisce ADCS, IS, e AS con valori di Kirispettivamente di 720, 56 e 80 µM.
Inoltre, il sono state studiate due importanti caratteristiche del modello tramite sintesi e saggi biologici di quattro nuovi composti rispetto a ADCS, IS, e AS. Per prima cosa, la lunghezza ottimale della fase SPACER è stata verificata variandone la lunghezza. Quindi, la possibilità della fase COMBI di essere principalmente responsabile dei risultati di inibizione è stata dimostrata con la sintesi e il saggio di composti solo-COMBI e solo-COMBI-SPACER. Si è
scoperto che questi inibiscono minimamente e non specificamente AS e ADCS,confermando così che tutte e tre le fasi sono necessarie per un’inibizione ottimale.
Infine, i risultati di inibizione sono presentati nel contesto delle proprietà cinetiche e strutturali di ADCS, IS, e AS, che sono enzimi dipendenti da Mg²+. I meccanismi cinetici impiegati da questi enzimi spiegano e rendono conto dei modelli di inibizione osservati nei confronti del corismato e del cofattore Mg²+.
Meccanismi cinetici di AS e IS. Sono stati misurati i tassi iniziali delle reazioni
nelle quali sono state variate le concentrazioni del corismato e su un Mg²+ ad ampio range. Entrambe le serie di dati di AS e IS sono state nel complesso più adatte al’equazione 1, che descrive un meccanismo cinetico casuale.
Schema 1:
vi={Vmax[chorismate] [Mg²+]}/
{KchorismateK Mg²+ + αKchorismate[Mg²+]
Lo schema 1 descrive gli equilibri e i parametri cinetici che corrispondono all’equazione (1). I valori costanti di legame per corismato e Mg²+ sono stati determinati dall’analisi di regressione non lineare. Il meccanismo cinetico di ADCS e i valori costanti di legame del substrato sono stati descritti precedentemente [3].
L’affinità di IS per il corismato è molto bassa in assenza di Mg²+ , come dimostrato dagli elevati valori di Kcorismato relativi a αKcorismato. Il legame tra
corismato e Mg²+ altamente cooperativo, mentre per AS il legame dei suoi substrati all’enzima libero dipende molto meno dalla presenza dell’altro rispetto a IS e ADCS.
Il meccanismo cinetico casuale impiegato da AS e IS contrasta con il meccanismo cinetico organizzato osservato per ADCS [3]. Nel caso di ADCS, il corismato si lega per primo e segue poi il magnesio. Il legame di corismato a ADCS provoca un’ ampia modificazione conformazionale e a sostegno di questo, esistono sostanziali differenze tra le forme “aperte” e “chiuse” delle strutture cristalline ai raggi x di ADCS [8]. Tre residui di glutammato, Glu302, Glu436, e Glu439, situati appena sopra il sito di legame putativo del corismato, sono strettamente conservati tra ADCS, AS e IS e sono indicati per coordinare Mg²+. La distanza tra due di essi, Glu302 e Glu439, riflette l’ampiezza del movimento delle proteine tra le forme aperta (25.3 Å) e chiusa (6.2 Å) di ADCS. Poiché il legame Mg²+ non è completamente formato nella struttura “aperta”, un meccanismo cinetico casuale non è possibile per ADCS.
AS e IS, invece, subiscono un cambiamento conformazionale minore sul legame di corismato e ciò è supportato da dati cristallografici [9]. Nel caso di AS, si possono paragonare le differenze strutturali tra la forma “chiusa” AS di Serratia mercescens [9], e la forma “aperta” AS di Salmonella typhimurium [10], (questi due isoenzimi di AS sono proteine quasi identiche). La distanza tra Glu302 e Glu439 nella forma aperta e nella forma chiusa cambia rispettivamente da 10.7 a
6.8 Å. Il cambiamento nella distanza tra questi analoghi residui di glutammato in IS è di grandezza simile: 9.9 Å (aperta) contro 5.9 Å (chiusa).
Poichè il sito di legame del magnesio in AS e IS si forma in modo più completo in assenza di corismato, è possibile per i due substrati legarsi indipendentemente l’uno dall’altro. I dati cinetici e strutturali di AS e IS supportano un meccanismo nel quale il corismato e Mg²+ si legano in maniera cooperativa e i dati di inibizione per ADCS, AS, e IS supportano ulteriormente questa conclusione.
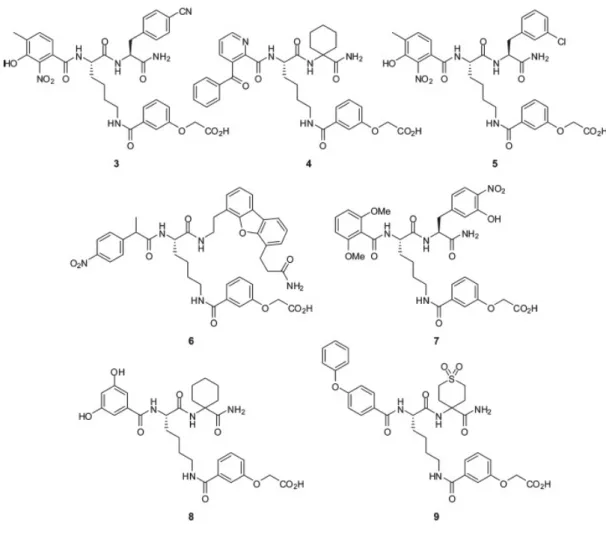
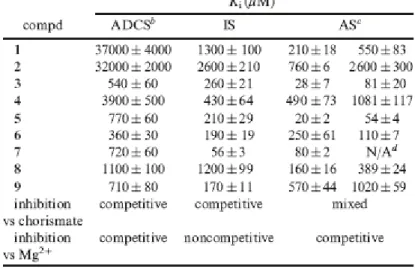
Risultati inibizione. Gli inibitori (3-9) identificati sono stati risintetizzati [3] e
saggiati verso IS e AS (Figura 3) ed i risultati (insieme ai dati per i composti 1 e
2), sono mostrati in Tabella 1. In particolare, tutti gli inibitori (tranne 8) si legano
più strettamente con IS e AS di quanto non facciano con ADCS. Il miglior inibitore di IS è 7 con un valore di Ki di 56 µM; mentre il composto 5 è un
inibitore di AS con un valore di Kidi 20 µM.
Tabella 1: Inibizione di ADCS, IS e AS da parte dei Composti della Libreria identificati in un ADCS On-Bead Screen. ɑ Le costanti di inibizione riflettono l’inibizione rispetto al legame con
il corismato. ƅ Dati presi da rif 3. c Valori di K
i ‘,elencati nell’ultima colonna, riflettono il legame
inibitore al complesso [E • corismato]. d Non applicabile perché il composto 7 ha mostrato
inibizione pura non competitiva rispetto al corismato.
Il confronto dell’attività inibitoria da parte di 3-9 con quella del PAYLOAD è con il derivato ammidico 2 perché lo SPACER e il PAYLOAD sono connessi da un legame ammidico. L’inibizione di IS da parte di 7 è 46 volte più forte di 2, e il composto 5 lega 38 volte più strettamente AS rispetto a 2. Paragonati a ADCS, gli inibitori 3-9 si legano più strettamente a AS e IS, tuttavia il grado al quale la fase COMBI migliora l’inibizione è generalmente minore per IS e AS che per ADCS. Il valore di Ki di 6, miglior inibitore di ADCS, è 360 µM, 89 volte meglio
rispetto a 2. L’impatto maggiore di COMBI sul legame ADCS può essere collegato ad un più ampio cambio conformazionale che avviene sul legame del corismato, relativo a IS e AS.
Per AS, la mancanza di inibizione competitiva verso il corismato è risultata inaspettata, infatti nessuno dei legami inibisce in modo competitivo il legame del corismato; 1-6, 8 e 9 mostrano un’inibizione mista e 7 è un inibitore non
competitivo. Data la somiglianza tra il corismato 1 e 2, ci si aspetterebbe che questi inibitori siano competitivi contro il corismato. I valori di Ki riportati in
Tabella 1 riflettono il legame dell’inibitore al complesso [AS*corismato]. Tranne il composto 6, gli inibitori si legano più strettamente all’enzima libero piuttosto che al complesso [AS*corismato]. Lo schema 2 rende conto del meccanismo cinetico casuale di AS.
Schema 2:
L’inibizione competitiva rispetto al Mg²+ indica che questi composti si legano vicino al sito attivo di AS. (Tale risultato conferma che il complesso [AS*corismato*inibitore] è cataliticamente inattivo perché è necessario Mg²+ per
la catalisi). Nella struttura cristallina a raggi X AS di S. marcenses, un omologo quasi identico di AS di S. typhimurium, il magnesio è collocato nel sito attivo immediatamente prossimo al gruppo carbossilato C1 dell’acido benzoico analogo del corismato. Inoltre, tre residui di glutammato circondano e coordinano Mg²+. Gli inibitori affini probabilmente si legano vicino ai residui di
glutammato analoghi per inibire competitivamente il legame del Mg²+ ma, proporre l’esatta collocazione dell’inibitore nel legame con AS, sarebbe difficile.
Negli studi di inibizione di IS, invece, ogni inibitore ha inibito competitivamente il corismato e non competitivamente il magnesio. I diversi schemi di inibizione rispetto a ogni substrato sono una conseguenza del meccanismo cinetico casuale impiegato da IS e AS. Esistono siti di legame adiacenti separati per corismato e Mg²+, e gli inibitori si legano solo a uno di questi siti, permettendo all’altro substrato di legarsi.
Lunghezza ottimale di SPACER. La lisina è stata scelta inizialmente come
elemento SPACER (spaziatore) per la nostra libreria dopo che studi di docking con la struttura cristallina [17] di ADCS hanno indicato che la sua catena laterale di 4 atomi di carbonio è un’ottima lunghezza. Per dimostrare questa deduzione, i composti 10 e 11 sono stati sintetizzati e saggiati rispetto a ADCS, IS, e AS (Figura 4).
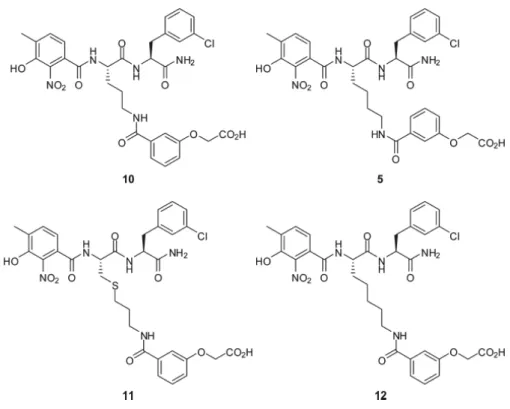
Figura 4: I composti 10, 5, 11, e 12 sono costituiti da elementi SPACER diversi: L-ornitina, L-lisina, S-propil-L-cisteina, e L-α-omolisina, rispettivamente. Questi composti sono costituiti da frazioni identiche di PAYLOAD e COMBI.
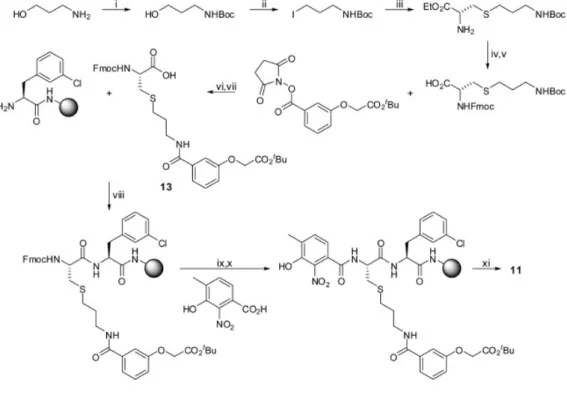
L’analogo 10 è stato preparato con L-ornitina, in modo che lo SPACER contenga un carbonio in meno rispetto alla lisina. A causa dell’alto costo e della scarsa disponibilità di un precursore L-α-omolisina, è stato sintetizzato invece 11 (un analogo isosterico di 12) (Figura 5).
Figura 5:Sintesi del composto 11. Reagenti e condizioni: (i) Boc2O, Et3N, DCM riflusso, per
una notte; (ii) I2, PPh3, 1H-imidazolo, DCM, rt, 3,5 h; (iii) Cs2CO3, DMF, metilestere
dell’idrocloride L-cisteina, rt, per una notte; (iv) LiOH, MeOH, rt, 2h; (v) Fmoc-OSu, THF, Na2CO3, 0°C, 1 h; (vi) TFA:DCM (3:7), rt, 30 min; (vii) DIPEA, H2O/ACN, rt, 7 h; (viii) HOBt,
DIC; (ix) 20% piperidina in DMF, 2 * 10 min; (x) HOBt, DIC; (xi) TFA/H2O/TIS (95, 2.5,
Le costanti di inibizione ottenute dal saggio cinetico di 5, 10, e 11 sono riportate in Tabella 2.
Tabella 2: L’effetto della lunghezza dello SPACER sull’inibizione di ADCS, IS e AS. ɑ I
valori di Ki riflettono l’inibizione rispetto al corismato. ƅ Ogni inibitore ha inibito
competitivamente il corismato e l’Mg2+. cOgni inibitore ha mostrato inibizione competitiva
verso il corismato e inibizione non competitiva verso Mg2+ . d Ogni inibitore ha mostrato
inibizione mista verso il corismato e ha inibito competitivamente l’Mg2+. eNessuna inibizione
osservata sopra 1000 µM; saggiare oltre 1000 µM non è stato possibile a causa degli strumenti e delle limitazioni del saggio.
Le lunghezze di SPACER corrispondono alla distanza tra il carbonio carbonilico di ammide del PAYLOAD e il carbonio α dell’aminoacido (e.g., ornitina, lisina, cisteina) con la sua catena laterale nella conformazione estesa. I valori di Ki
riflettono l’inibizione rispetto al corismato. La determinazione del meccanismo di inibizione (i.e., competitivo versus non competitivo) ha seguito un processo simile a quello descritto per gli inibitori 3-9.AS e ADCS sono più sensibili alla lunghezza dello SPACER. Uno SPACER più corto diminuisce fortemente il legame con ADCS, e uno SPACER più lungo è più nocivo per il legame con AS. Comunque è difficile interpretare l’effetto della lunghezza dello SPACER sull’inibizione di AS perché questi composti non sono inibitori competitivi del corismato. IS è stato il meno sensibile alla lunghezza dello SPACER; la
differenza nell’inibizione da parte di 5, 10 e 11 è stata minima. Nonostante un piccolo miglioramento nell’inibizione di IS da parte di uno SPACER più lungo (11), questo vantaggio non controbilancia il prezzo pagato con ADCS per lo SPACER più lungo. I risultati in Tabella 2 supportano la deduzione tratta dalla struttura cristallina che la lisina fornisca la lunghezza di SPACER ottimale per gli inibitori ADCS.