Appendice A
Strutture per l’organizzazione dei dati
A.1 kd-tree
Nella parte di ricerca del punto più vicino, necessaria per il funzionamento del nostro algoritmo, è
stato implementato un metodo per l’organizzazione e la ricerca dei punti, che permette di
velocizzare le operazioni. Data una nuvola di punti, la ricerca di quello più vicino ad un punto
arbitrario, avviene di solito misurando le distanze di tutti gli altri rispetto a quest’ultimo e
scegliendo quello che ha la distanza minore. In questo modo però l’ordine di grandezza del tempo
necessario alla ricerca sarà di tipo O(N) (ordine N, se N è il numero di punti da controllare),
l’utilizzo dei kd-tree invece, permette di escludere automaticamente i punti più lontani ed il tempo
per la ricerca scende all’ordine O(logN), anche se per costruirlo occorre O(NlogN).
Esistono diversi tipi di kd-tree a seconda dell’uso che se ne deve fare. Quello usato in questo lavoro
è descritto sotto ed è un tipo adatto a strutture con grande numero di dati.
Il nome indica che si tratta di strutture ad albero binario con “k” dimensioni. Nel nostro caso k = 3
sono le coordinate dei punti nello spazio che descrivono la nuvola.
I kd-tree con k = 3, lavorano dividendo lo spazio 3D in zone sempre più piccole con dei piani
perpendicolari ai tre assi del sistema di riferimento, fino a che una zona arriva a contenere un
numero di punti massimo stabilito.
Le porzioni di spazio vengono suddivise ogni qualvolta i punti in esse contenuti superano il numero
consentito. La suddivisione avviene secondo un piano diretto perpendicolarmente ad uno degli assi
coordinati, questo asse ogni volta cambierà ciclicamente all’interno di ogni suddivisione.
In questo modo la nuvola di punti viene ricorsivamente divisa in due parti, separate da un piano.
Ognuna delle due parti rappresenta un ramo del kd-tree mentre il piano (identificato con l’asse ad
esso perpendicolare e la coordinata lungo di esso) rappresenta il nodo. Il processo va avanti
separatamente all’interno delle singole suddivisioni cambiando ciclicamente l’asse rispetto al quale
il piano è perpendicolare. Il tutto si arresta quando all’interno delle suddivisioni sono contenuti un
numero massimo di punti scelto dall’utente.
Alla fine del procedimento lo spazio risulta diviso in tanti parallelepipedi contenenti ognuno una
serie di punti molto vicini tra loro che non superano la quantità massima consentita, queste
suddivisioni (chiamate foglie) rappresentano la parte finale dell’albero ossia quella che contiene
effettivamente i punti.
La costruzione di un kd-tree in due dimensioni è schematizzata in figura 1.
Figura 1 – Costruzione di un kd-tree nel piano per 4 punti, con numero massimo di punti nella suddivisione pari a 2. Nella struttura ad albero, i rettangoli con altri collegamenti sono nodi, quelli da dove non partono altri collegamenti sono foglie e le linee di interconnessione sono i rami.
Come si vede, ogni volta che si aggiunge un punto, si scorre all’interno dell’albero confrontando
ogni volta una delle coordinate del punto con quella caratteristica del nodo (il confronto avviene ad
esempio con la coordinata x, se quella del nodo è più grande del punto si va nel ramo sinistro,
contrariamente in quello destro). Se la zona dove è inserito il nuovo punto, supera il numero
massimo di punti consentito, questa viene divisa a metà secondo un piano perpendicolare ad uno
degli assi che cambia orientamento in modo ciclico (in questo caso a due dimensioni, si divide
ciclicamente con una linea secondo x e y). Ogni nodo conserva la direzione di divisione ed il valore
alla quale questa avviene.
l
1l
8A
l
2l
3l
4l
5l
7l
6l
9l
10C
B
E
D
J
I
K
H
F
G
D
G
F
E
A
C
B
I
H
J
K
l5
l1
l9
l6
l3
l10
l7
l4
l8
l2
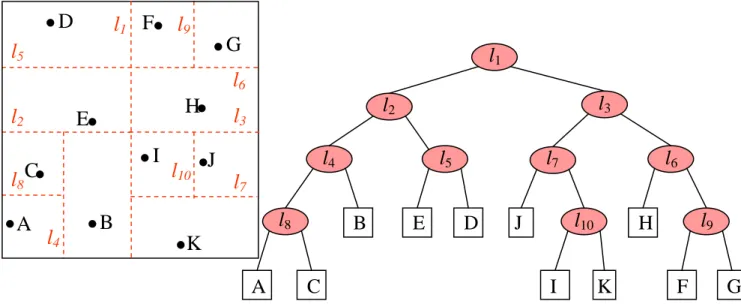
Figura 2 – Struttura di un kd-tree punti nel caso piano, con un numero massimo di punti nelle foglie pari a 1.
In figura 2 e 3 sono schematizzati rispettivamente una suddivisione nel piano (con la relativa
struttura ad albero del kd-tree) e la procedura di ricerca del punto più vicino ad un arbitrario punto
q. Quando all’algoritmo di ricerca viene richiesto di trovare il punto più vicino ad uno di coordinate
note, questo scorre all’interno dell’albero confrontando ogni volta la coordinate del punto con
quella caratteristica di ogni nodo ed entrando in una zona piuttosto che un’altra. Quando arriva nella
suddivisione finale contenente i punti, allora misura effettivamente le distanze da tutti i punti
presenti all’interno e prende il più vicino (nel nostro esempio il punto è uno solo).
l
1l
8A
l
2l
3l
4l
5l
7l
6l
9l
10C
B
E
D
J
I
K
H
F
G
D
G
F
E
A
C
B
I
H
J
K
l5
l1
l9
l6
l3
l10
l7
l4
l8
l2
q
Appendice B
Software adoperato
Matlab® Version 7 (Release 14) - The MathWorks, Inc.
http://www.mathworks.com
Geomagic Studio® 6 – Raindrop Geomegic, Inc
http://www.geomagic.com/
EDS® Imageware® Versione 10 - Structural Dynamics Research Corporation
http://www.sdrc.com
Microsoft Word (Microsoft Office) - Microsoft Corporation.
Appendice C
Codici sviluppati
C.1 Codice per allineamento a coppie
Metodo punto-piano
function [modmat, info] = minimize_lsqnonlin_9(X, A, cptol); global ptpass; % global cptol; cloud2 = X'; cptol = cptol*10; [X, A] = bounding_box(X', A'); X = X'; A = A'; % tempmodpts = curvature_sample_manual_final(X, 75, 0.1, 1, 3, 10, 0.33); % tempfixpts = curvature_sample_manual_final(A, 75, 0.1, 1, 3, 10, 0.33); tempmodpts = uniform_sample_for_cs_unif_final(X, 0.005); % tempfixpts = uniform_sample_for_cs_unif_final(A, 0.33); % tempmodpts = X; tempfixpts = A; [tmp, tmp, TreeRoot] = kdtree(tempfixpts, []); R = eye(3); T = zeros(3,1); count = 1;
while count < 6 % Controllo sulle iterazioni
ptpass = newpoint2planeass_8(TreeRoot, tempmodpts, cptol); rId = eye(3);
t(1:3,1) = zeros; r = rodrigues(rId); Mrt = cat(1, r, t);
Dist = distanze_pt_piano_8(Mrt);
% Settiamo le opzioni in modo che permettano di passare
% all'algoritmo di Gauss-Newton, più efficace del LM quando il residuo % è piccolo, quindi se la registrazione è già accettabile in
% partenza
options = optimset('LargeScale', 'off', 'LevenbergMarquardt', 'off'); [nMrt, mindist, residual, exitflag, info(count)] =
lsqnonlin(@distanze_pt_piano_8, Mrt, [] , [], options);
% La variabile output 'info' ci da' informazioni sulle singole % chiamate della funzione LSQNONLIN
Mrt = nMrt; minmat = nMrt;
trm = minmat(4:6,1);
rotmodpts = rotm*tempmodpts';
modpts = [rotmodpts(1,:)+trm(1); rotmodpts(2,:)+trm(2); rotmodpts(3,:)+trm(3)]; tempmodpts = modpts'; R = rotm*R; T = rotm*T + trm; ptpass = []; count = count + 1;
% Decremento dinamico del parametro cptol if isequal(count,2) cptol = cptol*0.6; elseif isequal(count,3) cptol = cptol*0.6; elseif isequal(count,4) cptol = cptol*0.6; elseif isequal(count,5) cptol = cptol*0.55; end end kdtree([],[],TreeRoot); modmat = R * cloud2;
modmat = [modmat(1,:)+T(1); modmat(2,:)+T(2); modmat(3,:)+T(3)]'; % save info_minimize_alg info;
save modmat.asc modmat -ASCII;
Codici appoggio
Associazione dei piani:
function ptpass = newpoint2planeass_8(TreeRoot, X, cptol); global ptpass;
% global cptol;
% X matrice dei query points % A matrice dei fit planes
% [tmp, tmp, TreeRoot] = kdtree(A, []); apc = 1;
numass = size(X,1);
assplns(1:numass,7) = zeros; for i = 1:numass
[PtsInNeighborhood, Dist, IDX] = kdrangequery(TreeRoot, X(i,:), cptol); if size(PtsInNeighborhood,1)>2 % Se trova meno di 3 punti non associa il piano
[x0, vers] = lsplane(PtsInNeighborhood);
d = -x0(1)*vers(1)-x0(2)*vers(2)-x0(3)*vers(3); % Coefficiente 'd' della rappresentazione parametrica del piano
assplns(apc,:) = [X(i,:) vers' d]; apc = apc+1;
end end
fin = apc-1;
Distanze punto-piano
function Dist = distanze_pt_piano_8(Mrt); global ptpass;
global cptol;
points = ptpass(:,1:3); nump = size(points,1); k = 1:nump;
% Coefficienti a,b,c,d dei piani
% secondo vettori colonna lunghi quanto il numero dei piani a = ptpass(:,4); b = ptpass(:,5); c = ptpass(:,6); d = ptpass(:,7); r = rodrigues(Mrt(1:3,1)); t = Mrt(4:6,1); Xrt = r(1,1)*points(k,1)+r(1,2)*points(k,2)+r(1,3)*points(k,3)+t(1); Yrt = r(2,1)*points(k,1)+r(2,2)*points(k,2)+r(2,3)*points(k,3)+t(2); Zrt = r(3,1)*points(k,1)+r(3,2)*points(k,2)+r(3,3)*points(k,3)+t(3); % Formula per la distanza punto-piano
NumDist = a(k).*Xrt+b(k).*Yrt+c(k).*Zrt+d(k); DenDist = sqrt(a(k).^2+b(k).^2+c(k).^2); Dist = (NumDist./DenDist);
Metodo punto-punto
function [minmat, modmat] = minimize_points_distance_lsqnonlin2(X, A, cptol); % X matrice dei query points (mobile)
% A matrice dei corrispondenti (fissa) global indA; global indX; global tempfixpts; global tempmodpts; global cptol; % cptol = cptol*10; count = 1; indpts = 0; tempfixpts = A; tempmodpts = X; [tmp, tmp, TreeRoot] = kdtree(A, []); numX = size(X,1); corr = zeros(numX,3);
R = eye(3); T = zeros(3,1); stop_iter = 0;
old_minmat = zeros(6,1);
while ~isequal(stop_iter,1) % Controllo sulle iterazioni for i=1:numX
[ PtsInNeighborhood, Dist, IDX ] = kdrangequery( TreeRoot, tempmodpts(i,:), cptol ); if ~isempty(PtsInNeighborhood) [Y,ind] = min(Dist); corr(i,:) = [IDX(ind),i*ones,Y]; end
% [PtsInNeighborhood, Dist, IDX] = kdrangequery(TreeRoot, tempmodpts(i,:), tol);
% if ~isempty(PtsInNeighborhood)
% [cdist, ordcand] = sort(Dist,1); % scanord = ordcand(1);
% selpt = PtsInNeighborhood(scanord,:); % indpts = indpts+1;
% asspts(indpts,:) = [selpt X(i,:)]; % end end [I,J,indA] = find(corr(:,1)); [I,J,indX] = find(corr(:,2)); rId = eye(3); t(1:3,1) = zeros; r = rodrigues(rId); Mrt = cat(1, r, t); % dst = distanze_pt_pt(Mrt); % iter = 1; % while iter < 3 eval(['display(''Iterazione N.' num2str(count) ''')']);
options = optimset('LargeScale', 'off', 'LevenbergMarquardt', 'off'); [nMrt, mindist] = lsqnonlin(@distanze_pt_pt, Mrt, [] , [], options);
% [nMrt, mindist] = fminsearch(@sumsquaredist_points_distance, Mrt); Mrt = nMrt; % iter = iter + 1; % end minmat = nMrt; rotm = rodrigues(minmat(1:3,1)); trm = minmat(4:6,1); rotmodpts = rotm*tempmodpts';
modpts = [rotmodpts(1,:)+trm(1); rotmodpts(2,:)+trm(2); rotmodpts(3,:)+trm(3)];
tempmodpts = modpts'; % ptpass = [];
% % Decremento dinamico del parametro cptol % if isequal(count,5) % cptol = cptol*0.6; % elseif isequal(count,10) % cptol = cptol*0.6; % elseif isequal(count,15) % cptol = cptol*0.6; % elseif isequal(count,20) % cptol = cptol*0.6; % end
% Controllo condizione di convergenza if ~isequal(count,1)
stop_iter = 1; end for i=1:6 incr_contr = abs((minmat(i)-old_minmat(i))/abs(minmat(i))); if incr_contr>0.01 stop_iter = 0; end end old_minmat = minmat; R = rotm*R; T = rotm*T + trm; count = count + 1; end kdtree([],[],TreeRoot); inimat = X; modmat = R * inimat';
modpts2 = [modmat(1,:)+T(1); modmat(2,:)+T(2); modmat(3,:)+T(3)]'; modmat2 = modpts2;
save modmat2.asc modmat2 -ASCII;
Codici appoggio
Distanze punto-punto
function dst = distanze_pt_pt(Mrt); global tempfixpts global tempmodpts; global cptol; global indA; global indX; AA = tempfixpts(indA,:); XX = tempmodpts(indX,:); nump = size(XX,1); r = rodrigues(Mrt(1:3,1)); t = Mrt(4:6,1); k = 1:nump; Xrt = r(1,1)*XX(k,1)+r(1,2)*XX(k,2)+r(1,3)*XX(k,3)+t(1); Yrt = r(2,1)*XX(k,1)+r(2,2)*XX(k,2)+r(2,3)*XX(k,3)+t(2); Zrt = r(3,1)*XX(k,1)+r(3,2)*XX(k,2)+r(3,3)*XX(k,3)+t(3); dst = sqrt((AA(:,1)-Xrt(:)).^2+(AA(:,2)-Yrt(:)).^2+(AA(:,3)-Zrt(:)).^2);C.2 Codice per allineamento multiplo
function Ini_MW11
%%% Parte input (richiesta n° di nuvole e caricamento) nuvpts = input ('Numero delle nuvole da immettere ... '); cptol = input ('Valore del parametro TOL ... ');
for nn = 1:nuvpts
eval(['nuvola_corr = input(''Nuvola_' num2str(nn) ' ... '', ''s'');']); % Riga OK
% pin_ask = input('Pin? S/N [N]: ... ', 's');
eval(['nuvola_' num2str(nn) '.data = load(nuvola_corr);']); % % Condizione di pinning
% if ~isempty(pin_ask)
% eval(['nuvola_' num2str(nn) '.pin = 1;']); % end
end
%%% Inizializza i campi "nuvola_j.virtual_mates.nuvola_n" ossia i %%% virtual mates della nuvola opposta, "nuvola_j.vm_data_on.nuvola_k" %%% ossia i punti della nuvola da accoppiare ai virtual mates ed il campo %%% che indica l'insieme delle nuvole 'merged'
for vmi=1:nuvpts for vmi2=1:nuvpts
if ~isequal(vmi, vmi2)
eval(['nuvola_' num2str(vmi) '.virtual_mates.nuvola_' num2str(vmi2) ' = [];']);
eval(['nuvola_' num2str(vmi) '.vm_data_on.nuvola_' num2str(vmi2) ' = [];']);
eval(['nuvola_' num2str(vmi) '.merge_field = [];']); end
end end
temp_max_overlap = 0; % Inizializza il contatore di max overlap %%% Conteggio della nuvola con massimo numero di overlap
for vm = 1:nuvpts
eval(['nuvola_' num2str(vm) '_overlap = 0;']); for nbc = 1:nuvpts
if ~isequal(vm, nbc)
eval(['ovch = overlap_check_1(nuvola_' num2str(vm) '.data, nuvola_' num2str(nbc) '.data);']);
if ovch == 1
eval(['nuvola_' num2str(vm) '_overlap = nuvola_' num2str(vm) '_overlap + 1;']);
eval(['temp_over = nuvola_' num2str(vm) '_overlap;']); if temp_over > temp_max_overlap temp_max_overlap = temp_over; max_overlap_cloud = vm; end end end end end
%% Fine conteggio numero max overlap
%%% Calcola tutti i virtual_mates relativi delle nuvole che si intersecano for vm = 1:nuvpts
for nbc = 1:nuvpts
if ~isequal(vm, nbc)
eval(['ovch = overlap_check_1(nuvola_' num2str(vm) '.data, nuvola_' num2str(nbc) '.data);']);
if ovch == 1
eval(['nuvmob = nuvola_' num2str(vm) '.data;']); eval(['nuvfix = nuvola_' num2str(nbc) '.data;']); % Computa allineamento pairwise
t_modmat = minimize_lsqnonlin_9(nuvmob, nuvfix, cptol); % Prende i punti dalla matrice che verranno usati per il % riallineamento
[tmp, tmp, TreeRoot] = kdtree(nuvfix, []); % Costruisce un kdtree
tempmodpts = t_modmat; numX = size(tempmodpts,1); corr = zeros(numX,3); for i=1:numX
[ PtsInNeighborhood, Dist, IDX ] = kdrangequery( TreeRoot, tempmodpts(i,:), cptol ); if ~isempty(PtsInNeighborhood) [Y,ind] = min(Dist); corr(i,:) = [IDX(ind),i*ones,Y]; end end [I,J,indA] = find(corr(:,1)); [I,J,indX] = find(corr(:,2));
kdtree([],[],TreeRoot); % Svuoto il kdtree
% Prendo 5000 punti corrispondenti delle due nuvole per poi immagazzinarli
% nella struttura "nuvola_n.virtual_mates.nuvola_k" ed
% i rispettivi indici della nuvola_k, nella struttura % denominata nuvola_k.data_ind_vm.nuvola_n, saranno % queste distanze poi ad essere minimizzate
vm_ind = ceil(length(indX)*rand(5000,1));
if isempty(find(vm_ind == 0)); % Controllo sull'overlap effettivo
vm_nuv_mob = t_modmat(indX(vm_ind),:); vm_data2 = nuvmob(indX(vm_ind),:);
eval(['nuvola_' num2str(nbc) '.virtual_mates.nuvola_' num2str(vm) ' = vm_nuv_mob;']);
eval(['nuvola_' num2str(vm) '.vm_data_on.nuvola_' num2str(nbc) ' = vm_data2;']);
% Queste strutture devono muoversi solidarmente % alla nuvola end end end end end
%%% Fine calcolo virtual_mates
%%% Mette tutte le nuvole nel dormant_set for k = 1:nuvpts
eval (['dormant_set(' num2str(k) ') = {''nuvola_' num2str(k) '''};']); end
%%% Trasferisco la nuvola con più overlap nell'active_set
eval(['active_set = {''nuvola_' num2str(max_overlap_cloud) '''};']); % %%% Tolgo la nuvola dal dormant_set
while ~isempty(dormant_set) % Condizione di fine
nuvdor = size(dormant_set,2);
nuvact = size(active_set,2);
%%% Calcolo degli overlap delle nuvole nel d_s rispetto all' a_s temp_nom = 0; % Inizializza il contatore temporaneo di max overlap for d = 1:nuvdor
tnom = char(dormant_set(d)); eval([ tnom '_overlap = 0;']); for a = 1:nuvact
dor_cld = char(dormant_set(d)); act_cld = char(active_set(a));
eval(['covck = overlap_check_1(' dor_cld '.data, ' act_cld '.data);']);
if covck == 1
eval([ tnom '_overlap = ' tnom '_overlap + 1;']); end
end
eval(['nom = ' tnom '_overlap;']); if nom > temp_nom temp_nom = nom; ind_temp_nom = d; cmo = char(dormant_set(d)); end end
%%% Inserisce il curr nell'active_set
eval(['active_set(size(active_set,2)+1) = {''' cmo '''};']); %%% Rimuove il curr dal dormant_set
eval(['dormant_set(' num2str(ind_temp_nom) ') = [];']);
%%% Inserisce il primo elemento nella coda 'queue.push(curr)' queue(1) = {cmo};
%%%
while ~isempty(queue)
%%% Prende la nuvola dalla fine della coda
nuvmob = char(queue(end)); % 'Converte' dalla coda di nomi (celle), in matrice
% %%% Controllo della condizione di pinning
% eval(['pin_cond = ' char(queue(end)) '.pin;']);
% if pin_cond == 0 % Se la nuvola non è bloccata si procede all'allineamento
eval(['mer_f_contr = ' nuvmob '.merge_field;']);
%%% Procedura di individuazione dei 'vicini' nell'active_set if isempty(mer_f_contr)
%%% CASO DELLA NUVOLA SINGOLA
nbors = []; % Inizializza la lista dei vicini nuvact2 = size(active_set,2);
for ii = 1:nuvact2
sc_contr = active_set(ii); sc2_contr = queue(end);
if ~isequal(sc_contr, sc2_contr);
eval(['oc = overlap_check_1(' char(queue(end)) '.data, ' char(active_set(ii)) '.data);']);
if oc == 1
eval(['r_oc = ' char(queue(end)) '.vm_data_on.' char(active_set(ii)) ';']);
if ~isempty(r_oc)
temp_nbor = active_set(ii); nbors = [nbors, temp_nbor]; temp_nbor = [];
end end end end
num_nbors = size(nbors,2);
%%% Individuazione dei 'virtual mates'
virt_mates = []; % Inizializza la matrice dei virtual mates for nbn = 1:num_nbors % Immagazzina tutti i virtual_mate delle nuvole adiacenti
t_v_m = []; % Inizializza la matrice dei temporary virtual mates t_nuvnbr = char(nbors(nbn));
%%% V_M della nuvola sui neighbors
eval(['v_m_nuvmob = ' char(queue(end)) '.vm_data_on.' t_nuvnbr ';']);
%%% V_M dei neighbors sulla nuvola
eval(['v_m_nuvnbr = ' t_nuvnbr '.virtual_mates.' char(queue(end)) ';']);
if ~isempty(v_m_nuvmob) & ~isempty(v_m_nuvnbr) t_v_m = cat(2, v_m_nuvmob, v_m_nuvnbr); virt_mates = cat(1, virt_mates, t_v_m); end
end else
%%% CASO DELLE NUVOLE 'MERGED'
nbors = []; % Inizializza la lista dei vicini nuvact2 = size(active_set,2);
eval(['mer_f_count = size(' nuvmob '.merge_field,2);']); for ii = 1:nuvact2
s_c_contr = queue(end);
if ~isequal(s_c_contr, active_set(ii)) % Controlla che non calcoli la stessa nuvola
check_m = 1;
for m_f_c = 1:mer_f_count sc_contr = active_set(ii);
eval(['sc2_contr = ' char(queue(end)) '.merge_field(' num2str(m_f_c) ');']);
if isequal(sc_contr, sc2_contr); % Controlla che le nuvole non siano già 'merged'
check_m = 0; end
end
if check_m == 1
eval(['oc = overlap_check_1(' char(sc2_contr) '.data, ' char(active_set(ii)) '.data);']);
if oc == 1
eval(['r_oc = ' char(sc2_contr) '.vm_data_on.' char(active_set(ii)) ';']);
if ~isempty(r_oc)
temp_nbor = active_set(ii); nbors = [nbors, temp_nbor]; temp_nbor = []; end end end end end
%%% Procedura di assegnazione dei virtual mates num_nbors = size(nbors,2);
%%% Individuazione dei 'virtual mates'
virt_mates = []; % Inizializza la matrice dei virtual mates %%% Virtual mates della nuvola indicata nel 'queue(end)'
for nbn = 1:num_nbors % Immagazzina tutti i virtual_mate delle nuvole adiacenti
t_v_m = []; % Inizializza la matrice dei temporary virtual mates t_nuvnbr = char(nbors(nbn));
eval(['v_m_nuvmob1 = ' char(queue(end)) '.vm_data_on.' t_nuvnbr ';']);
%%% V_M dei neighbors sulla nuvola corrente
eval(['v_m_nuvnbr1 = ' t_nuvnbr '.virtual_mates.' char(queue(end)) ';']);
end
%%% Virtual mates delle nuvole solidali a quella corrente for nmcs = 1:mer_f_count
for nbn = 1:num_nbors % Immagazzina tutti i virtual_mate delle nuvole adiacenti
t_v_m = []; % Inizializza la matrice dei temporary virtual mates
t_nuvnbr = char(nbors(nbn));
eval(['t_nuvmrg = ' char(queue(end)) '.merge_field(' num2str(nmcs) ');']);
if ~isequal(t_nuvnbr, char(t_nuvmrg)) %%% V_M della nuvola sui neighbors
eval(['v_m_nuvmob2 = ' char(t_nuvmrg) '.vm_data_on.' t_nuvnbr ';']);
%%% V_M dei neighbors sulla nuvola
eval(['v_m_nuvnbr2 = ' t_nuvnbr '.virtual_mates.' char(t_nuvmrg) ';']);
v_m_nuvmob = cat(1, v_m_nuvmob1, v_m_nuvmob2); v_m_nuvnbr = cat(1, v_m_nuvnbr1, v_m_nuvnbr2); if ~isempty(v_m_nuvmob) & ~isempty(v_m_nuvnbr) t_v_m = cat(2, v_m_nuvmob, v_m_nuvnbr); virt_mates = cat(1, virt_mates, t_v_m); end end end end end %%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%% if ~isempty(virt_mates) virt_mates2 = virt_mates;
[pp_minmat, point_point_modmat] = MW_min_pts_dist2(virt_mates2); mob_mat = point_point_modmat;
fix_mat = virt_mates2(:,4:6);
[point_plane_modmat, p_pl_minmat] = minimize_lsqnonlin_10(mob_mat, fix_mat, cptol);
%%% Concateno le 2 matrici di rototraslazione MR1 = rodrigues(pp_minmat(1:3,1)); MT1 = pp_minmat(4:6,1); MR2 = rodrigues(p_pl_minmat(1:3,1)); MT2 = p_pl_minmat(4:6,1); MRR = MR1*MR2; MTT = MR2*MT1 + MT2;
minmat2 = cat(1, rodrigues(MRR), MTT);
%%% Questa sotto è la matrice di rototraslazione 'totale' rotm = rodrigues(minmat2(1:3,1));
trm = minmat2(4:6,1);
%%% Svuoto la matrice dei virtual mates virt_mates = [];
virt_mates2 = [];
%%% Applico lo spostamento alla nuvola corrente e tutte le sue %%% strutture
eval(['temp_data = ' char(queue(end)) '.data;']); rotmodpts = rotm*temp_data';
modpts = [rotmodpts(1,:)+trm(1); rotmodpts(2,:)+trm(2); rotmodpts(3,:)+trm(3)];
mod_data = modpts';
eval([ char(queue(end)) '.data = mod_data;']);
%%% Applico lo spostamento ai virtual_mate rispetto alle altre %%% nuvole, della nuvola in questione
for nvc = 1:nuvpts
eval(['s_c_c = {''nuvola_' num2str(nvc) '''};']); if ~isequal(queue(end), s_c_c)
eval(['e_c_vm = ' char(queue(end)) '.virtual_mates.nuvola_' num2str(nvc) ';']); if ~isempty(e_c_vm) t_rt_vm = e_c_vm; vmrotmodpts = rotm*t_rt_vm'; rt_vm = [vmrotmodpts(1,:)+trm(1); vmrotmodpts(2,:)+trm(2); vmrotmodpts(3,:)+trm(3)]; eval([char(queue(end)) '.virtual_mates.nuvola_' num2str(nvc) ' = rt_vm''' ';']); end end end
%%% Applico lo spostamento ai punti scelti della nuvola che andranno a
%%% minimizzare la loro distanza con i loro virtual mates sulla
%%% nuvola adiacente
for rnvc = 1:nuvpts
eval(['s_c_c = {''nuvola_' num2str(rnvc) '''};']); if ~isequal(queue(end), s_c_c)
eval(['re_c_vm = ' char(queue(end)) '.vm_data_on.nuvola_' num2str(rnvc) ';']); if ~isempty(re_c_vm) rt_rt_vm = re_c_vm; rvmrotmodpts = rotm*rt_rt_vm'; nrt_vm = [rvmrotmodpts(1,:)+trm(1); rvmrotmodpts(2,:)+trm(2); rvmrotmodpts(3,:)+trm(3)]; eval([char(queue(end)) '.vm_data_on.nuvola_' num2str(rnvc) ' = nrt_vm''' ';']); end end end
%%% Applico lo spostamento alle eventuali nuvole solidali a quella %%% corrente e tutte le loro strutture
eval(['nuv_corr_m = ' char(queue(end)) '.merge_field;']); if ~isempty(nuv_corr_m)
eval(['cl_mer = size(' char(queue(end)) '.merge_field, 2);']); for n_cl_mer = 1:cl_mer
eval(['t_curr_merg = ' char(queue(end)) '.merge_field(' num2str(n_cl_mer) ');']);
curr_merg = char(t_curr_merg);
%%% Applico lo spostamento alla nuvola corrente e tutte le sue
%%% strutture
eval(['temp_data = ' curr_merg '.data;']); rotmodpts = rotm*temp_data';
modpts = [rotmodpts(1,:)+trm(1); rotmodpts(2,:)+trm(2); rotmodpts(3,:)+trm(3)];
mod_data = modpts';
eval([ curr_merg '.data = mod_data;']);
%%% Applico lo spostamento ai virtual_mate rispetto alle altre
%%% nuvole, della nuvola in questione for nvc = 1:nuvpts
eval(['s_c_c = {''nuvola_' num2str(nvc) '''};']); if ~isequal(curr_merg, char(s_c_c))
eval(['e_c_vm = ' curr_merg '.virtual_mates.nuvola_' num2str(nvc) ';']); if ~isempty(e_c_vm) t_rt_vm = e_c_vm; vmrotmodpts = rotm*t_rt_vm'; rt_vm = [vmrotmodpts(1,:)+trm(1); vmrotmodpts(2,:)+trm(2); vmrotmodpts(3,:)+trm(3)];
eval([ curr_merg '.virtual_mates.nuvola_' num2str(nvc) ' = rt_vm''' ';']);
end end end
%%% Applico lo spostamento ai punti scelti della nuvola che andranno a
%%% minimizzare la loro distanza con i loro virtual mates sulla
%%% nuvola adiacente for rnvc = 1:nuvpts
eval(['s_c_c = {''nuvola_' num2str(rnvc) '''};']); if ~isequal(curr_merg, char(s_c_c))
eval(['re_c_vm = ' curr_merg '.vm_data_on.nuvola_' num2str(rnvc) ';']); if ~isempty(re_c_vm) rt_rt_vm = re_c_vm; rvmrotmodpts = rotm*rt_rt_vm'; nrt_vm = [rvmrotmodpts(1,:)+trm(1); rvmrotmodpts(2,:)+trm(2); rvmrotmodpts(3,:)+trm(3)];
eval([ curr_merg '.vm_data_on.nuvola_' num2str(rnvc) ' = nrt_vm''' ';']); end end end end end %%%
queue(end)= []; % Tolgo l'ultima nuvola dalla coda %%% Merging dei neighbors
ok_merge = 0; if num_nbors > 1
for mi = 1:num_nbors for mi2 = 1:num_nbors if ~isequal(mi, mi2)
eval(['mer_contr = ' char(nbors(mi)) '.merge_field;']);
eval(['num_mer = size(' char(nbors(mi)) '.merge_field, 2);']);
if ~isempty(mer_contr) ok_merge = 1;
for mer_count = 1:num_mer % Qui controllo se sono già 'merged'
eval(['same_merge_cont1 = ' char(nbors(mi)) '.merge_field(' num2str(num_mer) ');']);
same_merge_cont2 = nbors(mi2); if isequal(same_merge_cont1, same_merge_cont2)
ok_merge = 0; end
end
if ok_merge == 1 % Qui applica il merge m_count = num_mer + 1;
eval([ char(nbors(mi)) '.merge_field(' num2str(m_count) ') = {''' char(nbors(mi2)) '''};']);
end else
ok_merge = 1;
m_count = num_mer + 1;
eval([ char(nbors(mi)) '.merge_field = {''' char(nbors(mi2)) '''};']); end end end end end if ok_merge == 1 queue(end+1) = nbors(1); end else queue(end) = []; end end end for sf = 1:nuvpts
eval(['t_s_data = nuvola_' num2str(sf) '.data;']);
eval(['save nuvola_' num2str(sf) '_reg.asc t_s_data -ASCII;']); end
Codici appoggio
Rilevamento delle sovrapposizioni
function [answ] = overlap_check(data1, data2); % ---% Controlla la sovrapposizione delle nuvole %
% data1: 3 x m % data2: 3 x n
% ---answ = 1;
% [data1, data2] = bounding_box(data1, data2); % Questo per evitare sovrapposizioni 'virtuali' delle nuvole
xmin1 = min(data1(:,1)); ymin1 = min(data1(:,2)); zmin1 = min(data1(:,3)); xmin2 = min(data2(:,1)); ymin2 = min(data2(:,2)); zmin2 = min(data2(:,3));
xmax1 = max(data1(:,1)); ymax1 = max(data1(:,2)); zmax1 = max(data1(:,3)); xmax2 = max(data2(:,1)); ymax2 = max(data2(:,2)); zmax2 = max(data2(:,3));
if ((xmin2 > xmax1) | (xmax2 < xmin1)) | ((ymin2 > ymax1) | (ymax2 < ymin1)) | ((zmin2 > zmax1) | (zmax2 < zmin1))
answ = 0; end
Distanze punto-punto tra coppie virtuali
function [minmat2, modmat] = MW_min_pts_dist2(virt_mates); % X matrice dei query points (mobile)
% A matrice dei corrispondenti (fissa) global n_tempmodpts; global n_tempfixpts; global asspts; count = 0; indpts = 0; asspts = virt_mates; n_tempmodpts = asspts(:,1:3); n_tempfixpts = asspts(:,4:6); % [tmp, tmp, TreeRoot] = kdtree(tempfixpts, []); numX = size(asspts,1); % corr = zeros(numX,3);
% asspts(numX, 6) = zeros; % Iniz. punti corrispondenti R = eye(3);
T = zeros(3,1);
while count < 20 % Controllo sulle iterazioni % for i=1:numX
% [ PtsInNeighborhood, Dist, IDX ] = kdrangequery( TreeRoot, n_tempmodpts(i,:), cptol ); % if ~isempty(PtsInNeighborhood) % [Y,ind] = min(Dist); % corr(i,:) = [IDX(ind),i*ones,Y]; % end % %
% % [PtsInNeighborhood, Dist, IDX] = kdrangequery(TreeRoot, tempmodpts(i,:), tol);
% % if ~isempty(PtsInNeighborhood)
% % [cdist, ordcand] = sort(Dist,1); % % scanord = ordcand(1);
% % selpt = PtsInNeighborhood(scanord,:); % % indpts = indpts+1;
% % asspts(indpts,:) = [selpt X(i,:)]; % % end
% end % [I,J,indA] = find(corr(:,1)); % [I,J,indX] = find(corr(:,2)); rId = eye(3); t(1:3,1) = zeros; r = rodrigues(rId); Mrt = cat(1, r, t); dst = distanze_pt_pt2(Mrt); % iter = 1; % while iter < 3
options = optimset('MaxFunEvals', 5000, 'MaxIter', 2000);
[nMrt, mindist] = lsqnonlin(@distanze_pt_pt2, Mrt, [] , [] , options);
% [nMrt, mindist] = fminsearch(@sumsquaredist_points_distance, Mrt); Mrt = nMrt; % iter = iter + 1; % end minmat = nMrt; rotm = rodrigues(minmat(1:3,1)); trm = minmat(4:6,1); rotmodpts = rotm*n_tempmodpts';
modpts = [rotmodpts(1,:)+trm(1); rotmodpts(2,:)+trm(2); rotmodpts(3,:)+trm(3)]; n_tempmodpts = modpts'; % ptpass = []; R = rotm*R; T = rotm*T + trm; count = count + 1; end % kdtree([],[],TreeRoot); inimat = asspts(:,1:3); modmat = R * inimat';
modpts2 = [modmat(1,:)+T(1); modmat(2,:)+T(2); modmat(3,:)+T(3)]'; %%% Aggiunte per 'esportare' la matrice i rototraslazione
rot_part = rodrigues(R); trasl_part = T;
minmat2 = cat(1, rot_part, trasl_part); % save modmat2.asc modmat2 -ASCII;
Distanze punto-piano per punti corrispondenti virtuali
function [modmat, minmat] = minimize_lsqnonlin_10(X, A, cptol); global ptpass; % global cptol; cloud2 = X'; cptol = cptol*10; [X, A] = bounding_box(X', A'); X = X'; A = A'; % tempmodpts = curvature_sample_manual_final(X, 75, 0.1, 1, 3, 10, 0.33); % tempfixpts = curvature_sample_manual_final(A, 75, 0.1, 1, 3, 10, 0.33); tempmodpts = X;
tempfixpts = A;
[tmp, tmp, TreeRoot] = kdtree(tempfixpts, []); R = eye(3);
T = zeros(3,1); count = 1;
while count < 4 % Controllo sulle iterazioni
ptpass = newpoint2planeass_8(TreeRoot, tempmodpts, cptol); rId = eye(3);
t(1:3,1) = zeros; r = rodrigues(rId); Mrt = cat(1, r, t);
Dist = distanze_pt_piano_8(Mrt);
% Settiamo le opzioni in modo che permettano di passare
% all'algoritmo di Gauss-Newton, più efficace del LM quando il residuo % è piccolo, quindi se la registrazione è già accettabile in
% partenza
options = optimset('LargeScale', 'off', 'LevenbergMarquardt', 'off', 'MaxFunEvals', 2000);
[nMrt, mindist, residual, exitflag, info(count)] = lsqnonlin(@distanze_pt_piano_8, Mrt, [] , [], options);
% La variabile output 'info' ci da' informazioni sulle singole % chiamate della funzione LSQNONLIN
Mrt = nMrt; minmat = nMrt;
rotm = rodrigues(minmat(1:3,1)); trm = minmat(4:6,1);
rotmodpts = rotm*tempmodpts';
modpts = [rotmodpts(1,:)+trm(1); rotmodpts(2,:)+trm(2); rotmodpts(3,:)+trm(3)]; tempmodpts = modpts'; R = rotm*R; T = rotm*T + trm; ptpass = []; count = count + 1;
% Decremento dinamico del parametro cptol if isequal(count,2) cptol = cptol*0.5; elseif isequal(count,3) cptol = cptol*0.5; elseif isequal(count,4) cptol = cptol*0.45; % elseif isequal(count,5) % cptol = cptol*0.5; end end kdtree([],[],TreeRoot); modmat = R * cloud2;
modmat = [modmat(1,:)+T(1); modmat(2,:)+T(2); modmat(3,:)+T(3)]'; rot_part = rodrigues(R);
trasl_part = T;
minmat = cat(1, rot_part, trasl_part); % save info_minimize_alg info;