CAPITOLO III
RISULTATI I:
3
ANALISI INTRA-POPOLAZIONISTICA
Il profilo genetico della popolazione spagnola, oggetto di questa tesi, è stato analizzato a livello intra-popolazionistico secondo tre distinti approcci di studio come spiegato nel capitolo introduttivo.
3.1. ANALISI I: UN APPROCCIO STORICO-POLITICO
Sono state analizzate 9 popolazioni, ma la popolazione delle Canarie è stata eliminata dalle analisi comparative viste le modeste dimensioni e l’impossibilità di accorpare i dati sui campioni provenienti da tale zona con qualcuna delle altre popolazioni per ovvie ragioni di isolamento geografico. Le popolazioni studiate sono riportate nella tabella 3.1.1.
L’intero campione è stato tipizzato, come già detto in precedenza, per 5 sistemi VNTR e STR, ma dal momento che per il marcatore D17S5 è stato riscontrato un forte difetto di eterozigosi, i dati relativi a tale locus sono stati esclusi dalle analisi comparative. Su tale sistema ci si soffermerà più avanti.
Popolazione D1S80 D1S111 D8S566 D8S591 D17S5 Andal-Extr 82 84 82 56 76 Arag-LaRio 52 54 52 46 54 Astur-Galiz 46 58 58 30 52 Catal-Bale 56 86 88 60 80 Murc-Valen 38 46 40 24 46 Canaria___ 16 18 18 14 16 Casti-Manc 98 100 88 66 78 Canta-Leon 64 70 60 46 54 Navar-Basc 72 78 76 60 78 Totale (2N) 524 594 562 402 534
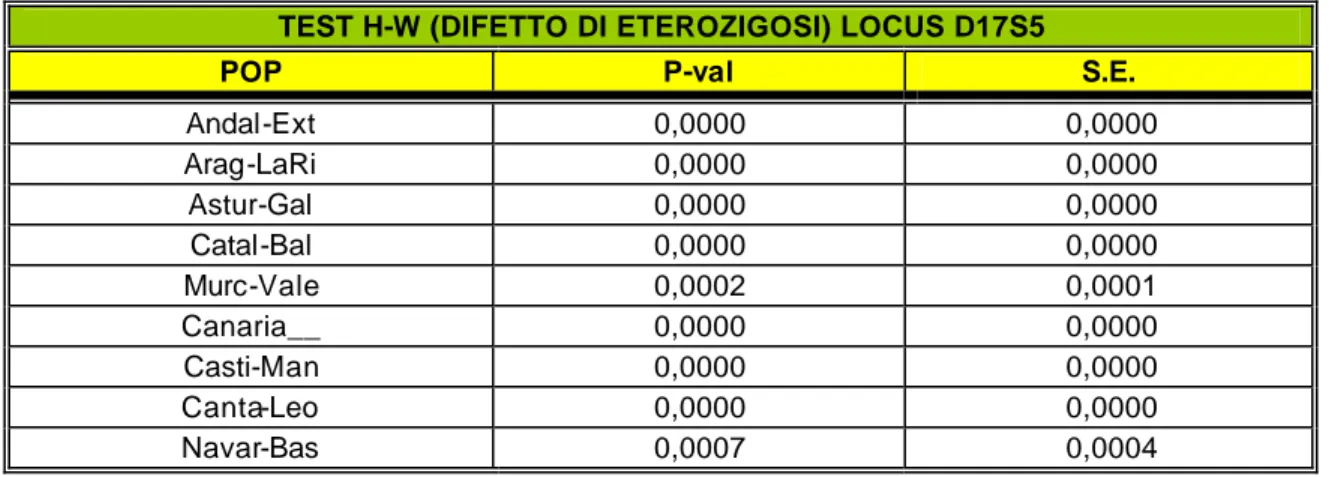
TEST H-W (DIFETTO DI ETEROZIGOSI) LOCUS D17S5
POP P-val S.E.
Andal-Ext 0,0000 0,0000 Arag-LaRi 0,0000 0,0000 Astur-Gal 0,0000 0,0000 Catal-Bal 0,0000 0,0000 Murc-Vale 0,0002 0,0001 Canaria__ 0,0000 0,0000 Casti-Man 0,0000 0,0000 Canta-Leo 0,0000 0,0000 Navar-Bas 0,0007 0,0004
Tabella 3.1.2.: Test di Hardy-Weinberg condotto nell’ipotesi di difetto di eteroigosi
Prima di analizzare i dati complessivi per tutti i loci, è stato effettuata un’analisi locus per locus al fine di riordinare le informazioni fornite da ciascuno di essi.
3.1.1 ANALISI LOCUS PER LOCUS
Per quanto riguarda il sistema D17S5, si può vedere dalla tabella A.1. in appendice, con i valori di P che danno misura della divergenza dall’equilibrio di Hardy-Weinberg, che tutte le popolazioni analizzate mostrano valori altamente significativi con P uguale o vicino allo zero. Tutti i campioni analizzati mostrano infatti un forte difetto di eterozigosi, come si può osservare dai valori di P per lo stesso test condotto nell’ipotesi di eccesso di omozigosi riportati in tabella 3.1.2. Visto qua nto osservato, non risulta né interessante né utile svolgere un’analisi genica o genotipica per tale locus, le cui anomalie verranno prese in considerazione nel capitolo successivo.
Per il sistema D1S80 sono stati osservati 73 genotipi. In tabella A.2. sono riportate le frequenze genotipiche. Si conclude che il genotipo più frequente è la combinazione degli alleli 18 e 24 ossia la combinazione fra gli alleli più frequenti nel complesso del campione spagnolo e della popolazione europea in generale. All’interno della popolazione andalusa la maggior parte dei genotipi sono combinazioni dell’allele 24. All’interno della popolazione Catalana sono più frequenti gli omozigoti 24-24, genotipo
che indica un certo grado di isolamento, ma comunque dato dalla forma allelica più frequente nel complesso della popolazione europea (Sciacca et al. 2004; Smolyanitsky 2003; Kondopoulou et al. 1999, Drmic et al. 1998; Alessio, 1998; Inoko, 1998; Sepulchre, 1995; Lareu, 1993; Pontes, 1993). La presenza di genotipi con alleli con un maggior numero di ripetizioni (e.g. 39-41) è un aspetto singolare della popolazione Basca. Non si rileva una particolare distribuzione genotipica se non per il fatto che i genotipi più frequenti sono le combinazioni dell’allele 24.
La tabella A.1. in appendice mostra i valori di P per il test esatto per valutare eventuali deviazioni dall’equilibrio di Hardy-Weinberg. Applicata la correzione di Bonferroni (Pc=0,005) a questi valori, tutte le popolazioni risultano in equilibrio ad eccezione del
campione della Castiglia che si discosta in modo molto significativo dall’equilibrio (P=0,0007). La popolazione castigliana presenta un forte difetto di eterozigosi.
Dalle tavole di frequenza allelica in appendice (Tabella A.3.) si osservano, per il sistema D1S80, 25 alleli con una media di 14 alleli per popolazione. Il locus si presenta pertanto abbastanza informativo. Gli alleli più frequenti sono quelli con 24 e 18 ripetizioni con frequenza del 24,6% e 33,4% rispettivamente. L’andamento allelico è bimodale così come descritto anche in altri studi (Sciacca et al. 2003; Das et al. 2002; Maviglia e coll. 2001; Mastana et al. 2000; Drmic e coll. 1998) con un picco a livello dell’allele 18 ed un altro a livello dell’allele 24 come mostrato in figura 3.1.1. Sono stati osservati alleli rari presenti solo in alcune popolazioni; ad esempio l’allele 15 si osserva solamente nella popolazione Catalana (Catal- Bale) così come gli alleli 35 e 36. L’allele 41 è presente solamente nella popolazione Basca (Navar-Basc).
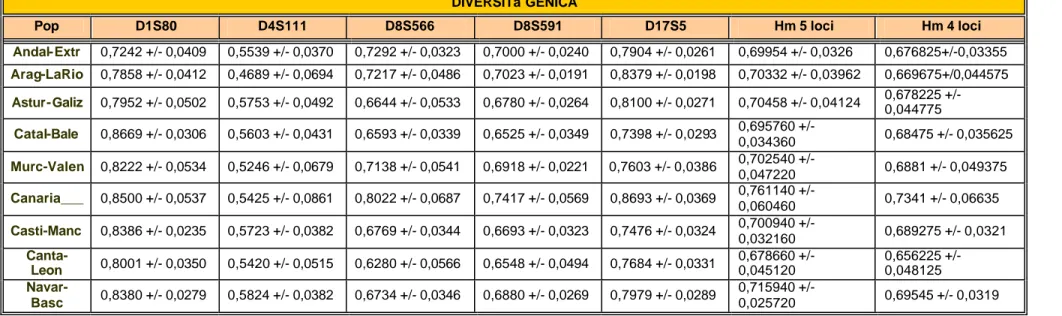
Dall’analisi della diversità genica (“Gene Diversity”; tabella 3.1.3), calcolata secondo Nei (1987), si osserva che i valori più alti in assoluto riguardano la popolazione catalana ed i più bassi la popolazione andalusa. I valori di diversità genica decrescono dalla regione catalana verso le regioni da questa più distanti sia a Nord che a Sud. L’unica eccezione è rappresentata della popolazione basca che possiede dei valori alti di diversità genica, ma è separata dalla catalogna da una regione di valori bassi data dal campione aragonese (Ara- LaRi). La media dei valori di variabilità genica è del 79%, il sistema D1S80 si rivela pertanto molto informativo.
La differenziazione genica per questo locus, calcolata per tutte e 9 le popolazioni, è significativa. Come si può osservare dalla tabella A.4 in appendice, con l’analisi di differenziazione genica a coppie di popolazioni attraverso l’uso delle catene di Markov, si ottengono valori di significatività (P<0,05) fra le seguenti coppie di popolazioni:
Figura 3.1. 1:Andamento allelico del sistema D1S80. Alleli Obs -20 0 20 40 60 80 100 120 140 160 180 200 allele 17 19 21 23 25 27 29 31 33 35 37 39 Alleli Obs
Andalusia (Andal- Ext) e Catalogna (Catal-Bale), Andalusia (Andal-Ext) e Murcia (Murc-Valen), Andalusia (Andal-Ext) e Castiglia (Casti-Man), Andalusia (Andal- Ext) e Paesi Baschi (Navar-Bas), Galizia (Astur-Gal) e Murcia (Murc-Vale), Catalogna (Catal-Bal) e Castiglia, Catalogna e Paesi Baschi, Paesi Baschi e Murcia. È molto alto il livello di significatività fra la popolazione Basca e la popolazione Catalana (P=0,004), valore da sottolineare vista la vicinanza geografica fra le due regioni e l’isolamento genetico dei Baschi riportato nella maggior parte della letteratura per questa popolazione.
La tabella A.5, riportata in appendice, mostra i valori di P per la differenziazione genotipica calcolata a coppie di popolazioni. Questi risultati confermano la situazione descritta dall’analisi della differenziazione genica a coppie.
In tabella A.6. è mostrata la distribuzione genotipica per il sistema D4S111. I genotipi più comuni osservati nell’insieme delle popolazioni sono il genotipo 2-3 ed il genotipo 2-2, con frequenza del 33% e del 34% rispettivamente. In realtà, come si può osservare, in 4 popolazioni su 9 il genotipo più frequente è il 2-3. Non è possibile individuare un particolare pattern di distribuzione genotipica.
La distribuzione genotipica osservata non è in accordo con una situazione di equilibrio secondo Hardy-Weinberg, ma, applicando la correzione di Bonferroni, si osserva che tutte le popolazioni analizzate risultano in equilibrio.
Dall’analisi della diversità genica, calcolata con il metodo di Nei (1987) e descritta in tabella 3.1.3, la media è del 55%. I valori più alti in assoluto riguardano la popolazione basca, i più bassi quella aragonese.
Sono stati osservati 3 alleli. L’allele più frequente (60,8%) è l’allele 2 (8 ripetizioni). L’andamento allelico è chiaramente unimodale come si può dedurre dall’osservazione dei dati sulle frequenze alleliche riportati in tabella A.7. in appendice.
Nell’analisi di differenziazione genica a coppie di popolazioni per questo locus, solamente la distanza fra la popolazione dell’Andalusia (Andal- Ext) e la popolazione della Castiglia (Casti-Man) raggiunge valori di significatività (P< 0,05) (tabella A.8). La differenziazione genotipica in tabella A.9. conferma questa situazione.
Nell’analisi del sistema D8S566 sono stati osservati 15 genotipi diversi nelle 9 popolazioni analizzate con una media di 7 per campione. Le frequenze genotipiche sono riportate in tabella A.10. Il genotipo 3-4 è il più frequente in assoluto in tutte le popolazioni, con un valore medio del 30% e rappresenta addirittura il 46% del
campione galiziano. Altri genotipi con frequenza medio alta sono le combinazioni degli alleli 2, 3 e 4. La distribuzione dei genotipi osservati è in equilibrio di Hardy-Weinberg in tutte e 9 le popolazioni (tabella A.1).
La frequenza allelica è mostrata in tabella A.11. Sono stati osservati 6 alleli. L’allele più frequente è quello con 19 ripetizioni (allele 4; frequenza: 41,5%) ed il secondo più frequente quello con 18 ripetizioni (allele3; frequenza: 35,6%). L’andamento allelico è unimodale con valori di frequenza solo leggermente più alti per gli alleli più corti. In tabella 3.1.3 è sono mostrati i valori di diversità genica calcolati come indicato nel paragrafo dedicato ai metodi statistici. L’eterozigosi media è del 69%, il sistema risulta pertanto abbastanza informativo. Non si osserva una particolare distribuzione dei valori di diversità genica.
La tabella A.12. in appendice mostra i valori di significatività della differenziazione genica a coppie di popolazioni. La differenziazione genica è significativa fra la popolazione delle Canarie (Canaria_) e le popolazioni Catalana e Basca e tra la popolazione catalana e quella della regione di Murcia. La differenziazione genotipica a coppie di popolazioni differenzia in modo significativo la popolazione catalana dalla popolazione di Murcia, come si può vedere in tabella A.13.
Nell’analisi dei dati ottenuti dal marcatore D8S591, sono stati rilevati 12 ge notipi. Il genotipo più frequente in tutte le popolazioni analizzate è la combinazione allelica 1-3 (30%). All’interno della popolazione della regione di Murcia e Valencia il genotipo omozigote 3-3 ed il genotipo 1-3 presentano lo stesso valore di frequenza (21%). I dati sulle frequenze genotipiche sono ripotati in tabella A.14 in appendice.
In 2 delle 9 popolazioni si osserva una deviazione dall’equilibrio di Hardy-Weinberg che viene corretta applicando il metodo di Bonferroni (Pc=0,005).
Per il sistema D8S591 sono stati osservati 5 alleli. Come si può osservare in tabella A.15. i più frequenti sono gli alleli 1 e 3 con 10 e 12 ripetizioni rispettivamente (relative frequenze: 43% e 29,8%) e, in generale, sono molto più comuni gli alleli con minor numero di ripetizioni. La distribuzione allelica è unimodale con un picco in corrispondenza dell’allele 1, la curva di distribuzione ha una pendenza elevata evidenziando la notevole differenza di frequenza fra alleli con poche ripetizioni da quelli con 13 o 14 ripetizioni della sequenza (alleli 4 e 5). Solamente all’interno della popolazione delle Canarie l’allele 5 raggiunge una frequenza più elevata, circa sei volte
DIVERSITà GENICA
Pop D1S80 D4S111 D8S566 D8S591 D17S5 Hm 5 loci Hm 4 loci
Andal-Extr 0,7242 +/- 0,0409 0,5539 +/- 0,0370 0,7292 +/- 0,0323 0,7000 +/- 0,0240 0,7904 +/- 0,0261 0,69954 +/- 0,0326 0,676825+/-0,03355 Arag-LaRio 0,7858 +/- 0,0412 0,4689 +/- 0,0694 0,7217 +/- 0,0486 0,7023 +/- 0,0191 0,8379 +/- 0,0198 0,70332 +/- 0,03962 0,669675+/0,044575 Astur-Galiz 0,7952 +/- 0,0502 0,5753 +/- 0,0492 0,6644 +/- 0,0533 0,6780 +/- 0,0264 0,8100 +/- 0,0271 0,70458 +/- 0,04124 0,678225 +/- 0,044775 Catal-Bale 0,8669 +/- 0,0306 0,5603 +/- 0,0431 0,6593 +/- 0,0339 0,6525 +/- 0,0349 0,7398 +/- 0,0293 0,695760 +/- 0,034360 0,68475 +/- 0,035625 Murc-Valen 0,8222 +/- 0,0534 0,5246 +/- 0,0679 0,7138 +/- 0,0541 0,6918 +/- 0,0221 0,7603 +/- 0,0386 0,702540 +/- 0,047220 0,6881 +/- 0,049375 Canaria___ 0,8500 +/- 0,0537 0,5425 +/- 0,0861 0,8022 +/- 0,0687 0,7417 +/- 0,0569 0,8693 +/- 0,0369 0,761140 +/- 0,060460 0,7341 +/- 0,06635 Casti-Manc 0,8386 +/- 0,0235 0,5723 +/- 0,0382 0,6769 +/- 0,0344 0,6693 +/- 0,0323 0,7476 +/- 0,0324 0,700940 +/- 0,032160 0,689275 +/- 0,0321 Canta-Leon 0,8001 +/- 0,0350 0,5420 +/- 0,0515 0,6280 +/- 0,0566 0,6548 +/- 0,0494 0,7684 +/- 0,0331 0,678660 +/- 0,045120 0,656225 +/- 0,048125 Navar-Basc 0,8380 +/- 0,0279 0,5824 +/- 0,0382 0,6734 +/- 0,0346 0,6880 +/- 0,0269 0,7979 +/- 0,0289 0,715940 +/- 0,025720 0,69545 +/- 0,0319
tanto quella osservata nelle altre popolazioni.Dalla tabella di diversità genica si rileva un valore medio di eterozigosi del 68%. In tabella A.16. si può osservare che le sole regioni che hanno un differenziamento genotipico significativo fra loro sono Cantabria (Canta-Leo) e Aragona (Arag-LaRi). La stessa coppia di popolazioni mostra valori significativi di differenziamento genico (tabella A.17).
3.1.2 DIVERSITÀ GENICA
Al fine di realizzare un confronto più rigoroso ed analitico delle popolazioni considerate è stato quantificato l’indice di diversità genica media per ogni sistema esaminato e quindi la diversità media. Per ciascun valore è stato inoltre calcolato il relativo errore standard secondo Nei (1987). Come si può osservare nella tabella 3.1.3 i valori più alti riguardano il locus D1S80 ed i più bassi il locus D4S111 che è, infatti, il meno polimorfico.
Nonostante nella stessa tabella siano riportati anche i dati di eterozigosità media su cinque loci, non sono stati presi in considerazione, vista la presenza dei dati sul sistema D17S5 che, come visto, presenta un fortissimo difetto di eterozigosi. In un confronto a 4 loci, la variabilità più alta in assoluto, escludendo dall’analisi il piccolo campione della Canarie, è quella riguardante la popolazione basca. La variabilità più bassa in assoluto riguarda invece la popolazione cantabrica. I valori sono per lo più simili, ma si può descrivere un gradiente decrescente dalla regione basca verso il sud e l’ovest.
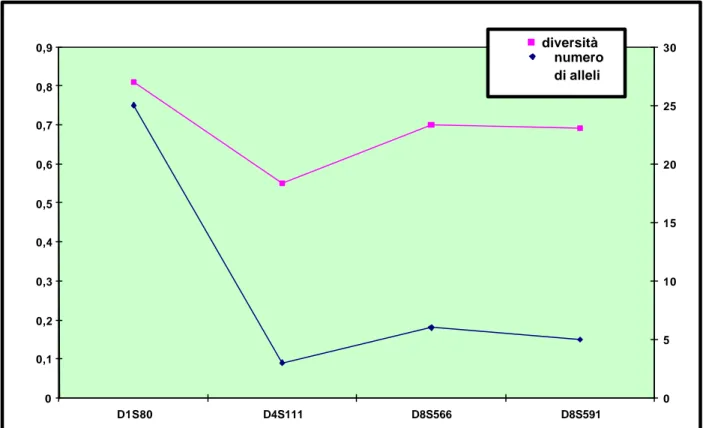
È da osservare come l’alta variabilità del locus D1S80 sia da mettere in relazione all’alto numero di alleli di questo sistema a differenza del locus D4S111 che appare essere il meno informativo (figura 3.1.2.).
3.1.3 ANALISI DEGLI APLOTIPI E DELLE DISTANZE GENETICHE
L’analisi aplotipica è stata condotta usufruendo solamente di quattro dei cinque sistemi tipizzati (D1S80, D4S111, D8S566, D8S591), visto il forte livello di omozigosi riscontrato per il marcatore D17S5.
Si osserva che la maggior parte degli aplotipi più comuni sono risultato delle combinazioni degli alleli più frequenti nel complesso dell’intero campione.
Figura 3.1.2.: Andamento della diversità in relazione al numero di alleli
Ogni singola popolazione mostra un numero di forme aplotipiche consistente, da un minimo di 36 della regione di Murcia e Valencia ad un massimo di 78 in Castiglia La Mancia. La distribuzione delle frequenze aplotipiche all’interno di ogni singola popolazione e, in una veduta generale, nel complesso del campione in analisi, è molto omogeneo. Gli aplotipi più diffusi mostrano frequenze tra il 3,4% (Galizia)ed il 7,1% (Aragona- La Rioja). Gli aplotipi più frequenti differiscono principalmente per gli alleli in posizione 3 e 4, cioè per i sistemi microsatelliti (D8S566 e D8S591). La posizione del primo locus è rappresentata sempre dagli alleli 18 e 24, i più frequenti nella totalità delle popolazioni caucasoidi (Sciacca et al. 2004, Kondopoulou et al. 1999, Drmic et al. 1998). Fa eccezione un aplotipo dei più frequenti (3,4%) fra la popolazione galiziana che si differenzia proprio in posizione del marcatore D1S80 (34,3,4,3). Gli aplotipi più frequenti nella totalità del campione spagnolo sono l’aplotipo 18,2,3,1 ed il 24,2,4,1. Data la bassa frequenza di tutti gli aplotipi ed il fatto che siano per lo più combinazioni degli alleli più frequenti di ciascun locus nell’insieme degli Europei, non è possibile inferire niente sulla struttura genetica della popolazione spagnola.
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9 D1S80 D4S111 D8S566 D8S591 0 5 10 15 20 25 30 diversità numero di alleli
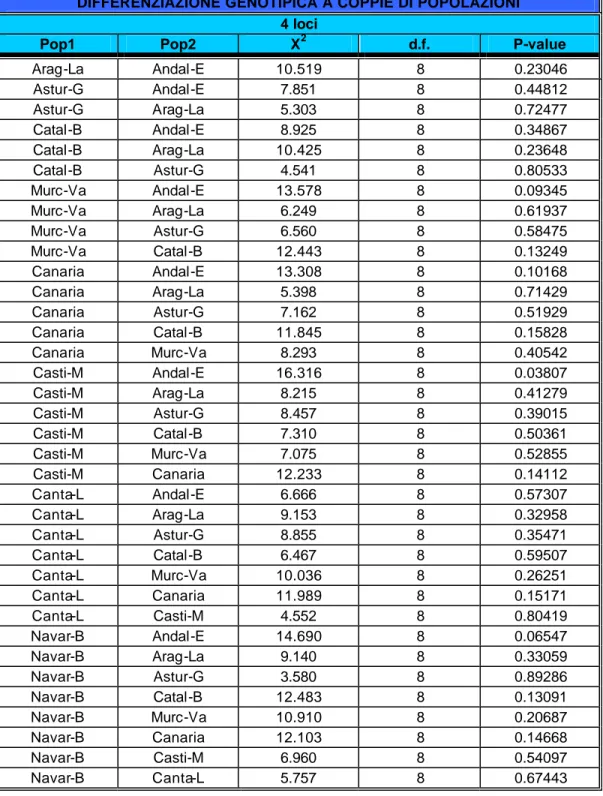
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI 4 loci
Pop1 Pop2 X2 d.f. P-value
Arag-La Andal-E 10.519 8 0.23046 Astur-G Andal-E 7.851 8 0.44812 Astur-G Arag-La 5.303 8 0.72477 Catal-B Andal-E 8.925 8 0.34867 Catal-B Arag-La 10.425 8 0.23648 Catal-B Astur-G 4.541 8 0.80533 Murc-Va Andal-E 13.578 8 0.09345 Murc-Va Arag-La 6.249 8 0.61937 Murc-Va Astur-G 6.560 8 0.58475 Murc-Va Catal-B 12.443 8 0.13249 Canaria Andal-E 13.308 8 0.10168 Canaria Arag-La 5.398 8 0.71429 Canaria Astur-G 7.162 8 0.51929 Canaria Catal-B 11.845 8 0.15828 Canaria Murc-Va 8.293 8 0.40542 Casti-M Andal-E 16.316 8 0.03807 Casti-M Arag-La 8.215 8 0.41279 Casti-M Astur-G 8.457 8 0.39015 Casti-M Catal-B 7.310 8 0.50361 Casti-M Murc-Va 7.075 8 0.52855 Casti-M Canaria 12.233 8 0.14112 Canta-L Andal-E 6.666 8 0.57307 Canta-L Arag-La 9.153 8 0.32958 Canta-L Astur-G 8.855 8 0.35471 Canta-L Catal-B 6.467 8 0.59507 Canta-L Murc-Va 10.036 8 0.26251 Canta-L Canaria 11.989 8 0.15171 Canta-L Casti-M 4.552 8 0.80419 Navar-B Andal-E 14.690 8 0.06547 Navar-B Arag-La 9.140 8 0.33059 Navar-B Astur-G 3.580 8 0.89286 Navar-B Catal-B 12.483 8 0.13091 Navar-B Murc-Va 10.910 8 0.20687 Navar-B Canaria 12.103 8 0.14668 Navar-B Casti-M 6.960 8 0.54097 Navar-B Canta-L 5.757 8 0.67443
Tabella 3.1.4.: Approccio storico-politico: differenziazione genotipica a coppie di popolazioni
calcolata sulle frequenze genotipiche dei 4 loci (D1S80, D4S111, D8S566, D8S591) con il metodo di Fisher (1954).
La tabella 3.1.4 mostra i valori di differenziazione genotipica a coppie di popolazioni calcolati con i dati complessivi dei 4 loci con il metodo di Fisher (1954). La differenziazione genotipica è significativa (P<0,05) solo fra la popolazione andalusa e quella castigliana. Il test esatto di differenziazione fra popolazioni, basato sulle frequenze aplotipiche e calcolato con il metodo di Goudet (1996), è altamente significativo (P<0,01) nel complesso delle popolazioni. Lo stesso test applicato a coppie di popolazioni mostra differenze significative fra le seguenti coppie di popolazioni: andalusa (Andal-Extr) e catalana (Catal-Bale); andalusa e murciana (Murc-Valen); aragonese (Arag-LaRio) e galiziana (Astur-Gali); aragonese e catalana; aragonese e murciana; aragonese e canaria (Canarie); galiziana e murciana; galiziana e castigliana; catalana e murciana e catalana e castigliana. I valori di P per questa analisi sono riportati in figura 3.1.3.
La tabella 3.1.5, riportata qui sotto, mostra la distanza genetica della corda (Dc) fra le popolazioni analizzate. Escludendo i dati relativi alla popolazione delle Canarie, la distanza genetica media calcolata su tutte le popolazioni è di 0,140. la popolazione che dista maggiormente dalle altre è la popolazione di Murcia, come si può infatti osservare dalla rappresentazione grafica nel MDS lungo la prima dimensione. La popolazione che, invece, mostra i minori valori di distanza genetica dalle altre è la popolazione galiziana. Applicando il procedimento MDS alla matrice delle distanze genetiche ottenute con il metodo Dc si ottiene il grafico riportato in figura 3.1.4.
La rappresentazione grafica del MDS è stata condotta escludendo la popolazione delle Canarie ed il locus D17S5. Si osserva che, considerando la prima dimensione, tutte le popolazioni rientrano all’interno dello stesso cluster ad esclusione della regione di Murcia-Valencia. La seconda dimensione accorpa più uniformemente tutte le popolazioni. L’indice di stress assume il valore di 0,122 e fa considerare il grafico attendibile rispetto ai rapporti di distanza tra le popolazioni e dunque accettabile.
Approccio storico-politico: 4 loci 9 popolazioni
AND ARA GAL CAT MUR CAN CAS SAN BA Andalusia AND
Aragona ARA 0.174 Galizia GAL 0.152 0.150 Catalogna CAT 0.156 0.181 0.140 Mursia MUR 0.194 0.162 0.155 0.187
Isole Canarie CAN 0.249 0.229 0.243 0.256 0.273
Castiglia la Mancia CAS 0.171 0.162 0.161 0.129 0.170 0.272 Cantabria SAN 0.139 0.162 0.156 0.138 0.164 0.271 0.127
Paesi Baschi BAS 0.154 0.174 0.123 0.150 0.191 0.251 0.133 0.142
Tabella 3.1.5.: Matrice delle distanze genetiche calcolate con il metodo della corda (Cavalli-Sforza e
Figura 3.1.3: Approccio storico-politico: test di differenziazione a coppie di popolazioni con i dati su 4 loci.
AND ARA GAL CAT MUR CAN CAS SAN BAS AND ARA 0.06836+-0.0129 GAL 0.07147+-0.0109 0.04847+-0.0051 CAT 0.00023+-0.0002 0.00746+-0.0017 0.24332+-0.0296 MUR 0.00124+-0.0008 0.02923+-0.0054 0.04798+-0.0060 0.00013+-0.0001 CAN 0.24531+-0.0269 0.03679+-0.0022 0.75912+-0.0176 0.57352+-0.0269 0.04174+-0.0042 CAS 0.74694+-0.0163 0.31391+-0.0299 0.03802+-0.0058 0.01300+-0.0053 0.00243+-0.0010 0.27558+-0.0132 SAN 0.24916+-0.0168 0.08016+-0.0077 0.23972+-0.0188 0.07121+-0.0144 0.00189+-0.0010 0.23052+-0.0187 0.94525+-0.0072 BAS 0.12743+-0.0216 0.46402+-0.0214 0.33132+-0.0117 0.11167+-0.0213 0.01517+-0.0056 0.54761+-0.0280 0.11442+-0.0158 0.06160+-0.0113
Figura 3.1.4.: Rappresentazione del Multidimensional Scaling ottenuto dalla matrice di distanze genetiche Dc basate sulle frequenze degli aplotipi a 4 loci. Stress=0,1223. Castiglia-Mancia Aragona-La Rioja Asturia-Galizia Andalusia-Extremadura Murcia-Valencia Cantabria-Leon Catalogna-Baleari Navarra-Paesi Baschi -1,2 -0,6 0 0,6 1,2 -1,5 -1 -0,5 0 0,5 1 1,5 Dimensione 1 Dimensione 2
3.2. ANALISI II: UN APPROCCIO GEOGRAFICO
Sono state analizzate le 5 popolazioni indicate nella tabella 3.2.1 in base alla geografia del territorio, secondo quanto spiegato nell’introduzione.
Anche in questo caso l’intero campione è stato tipizzato per i 5 sistemi VNTR e STR, ma i dati sul locus D17S5 sono stati esclusi dall’analisi finale.
3.2.1 ANALISI LOCUS PER LOCUS
Per il sistema D17S5, anche in questo caso, tutte le popolazioni mostrano un forte difetto di eterozigosi con P=0,000 ossia altamente significativo (tabella A.18., in appendice).
In questo secondo approccio analitico, per il locus D1S80, sono stati rilevati 80 genotipi fra 528 individui. Come si può vedere in tabella A.19 il numero dei genotipi oscilla fra 15 (popolazione dell’Andalusia) e 38 (popolazione della Meseta). I genotipi più frequenti nella totalità del campione sono il 18-24 ed il 18-18, rispettivamente in 3 e 2 popolazioni. In ciascuna popolazione la frequenza del genotipo più frequente è in media del 16% ad eccezione dell’Andalusia che mostra una frequenza del 35% per il genotipo 18-24.
Applicando la correzione di Bonferroni (Pc=0,01), le popolazioni analizzate risultano
tutte in equilibrio di Hardy-Weinberg ad eccezione della popolazione della Meseta per il locus D1S80. Popolazione D1S80 D4S11 D8S566 D8S591 D17S5 Zona Cantabrica 124 144 142 94 138 Zona Pirenaica 52 64 64 50 66 Zona Mediterranea 76 104 98 60 96 Meseta 220 226 204 168 192 Andalusia 56 60 60 36 56 Totale 528 598 568 408 548
In tabella A.20 sono riportati i valori di frequenza allelica. Si osservano18 alleli differenti. Gli alleli più frequenti sono sempre quelli con 18 e 24 ripetizioni, con frequenza del 24% e del 33% rispettivamente . Anche in questo caso l’andamento allelico è bimodale. Gli alleli con il più alto numero di ripetizioni (39 e 41) sono stati osservati solo nella Zona Cantabrica, come ci si può attendere per popolazioni isolate dal resto della penisola da alte catene montuose quali la cordigliera Cantabrica.
I valori di diversità genica, mostrati in tabella 3.2.2, sono compresi fra il 77% ed l’85%, valori rilevati rispettivamente per l’Andalusia e la Zona Mediterranea. La popolazione della Meseta presenta il secondo valore più basso ed è possibile osservare un gradiente decrescente di variabilità genica dalla zona Mediterranea verso la zona centro – meridionale della Spagna.
In tabella A.21. sono forniti i valori di P di differenziazione genotipica a coppie di popolazioni. Il locus in esame fornisce valori significativi di differenziazione genotipica tra la popolazione della Zona Cantabrica e quella della Zona Mediterranea, ancora tra la prima e la Meseta e tra la coppia Zona Pirenaica-Meseta. Il differenziamento genotipico fra le popolazioni cantabra e mediterranea è altamente significativo con un valore di P=0,002.
La differenziazione genica a coppie di popolazioni, in tabella A.22., conferma questa situazione.
Per il sistema D4S111 la distribuzione genotipica è mostrata in tabella A.23. in appendice. Sono presenti 6 genotipi nel complesso della popolazione spagnola. Il genotipo più frequente è l’omozigote 2-2, ad eccezione delle popolazioni della regione Cantabrica e dell’Andalusia nelle quali predomina il genotipo 2-3. Il genotipo 2-2 ha frequenza massima nella Meseta (42%) e il valore mostra un gradiente decrescente verso il Nord ed il Sud della Penisola. Il genotipo 3-3 ha una frequenza del 10% in Andalusia, almeno di due volte maggiore di quella registrata per le altre popolazioni. Il genotipo 3-4 è presente solamente tra la popolazione della Meseta.
La distribuzione genotipica è in equilibrio di Hardy-Weinberg in tutte le popolazioni, previa applicazione della correzione di Bonferroni (Pc).
Sono stati osservati 4 alleli, come si può vedere dalla tabella delle frequenze alleliche in appendice (tabella A.23.). Il più frequente è l’allele 3 con 9 ripetizioni e la distribuzione allelica è unimodale. L’allele con 10 ripetizioni (allele 4) è presente solo nella popolazione della Meseta con una frequenza dello 0,4%. Come detto in precedenza
nella parte introduttiva, questa è una forma allelica rara già osservata in uno studio sulla popolazione australiana (rif. Bibliogr.).
Si può osservare che i valori di diversità genica sono in generale relativamente bassi, ma mostrano un evidente gradiente decrescente dal nord al sud da un valore massimo di diversità di 0,57 in Cantabria ad uno minimo di 0,53 in Andalusia. (Tabella 3.2.2). I risultati del calcolo di differenziamento genotipico a coppie di popolazioni, ottenuto sulla base delle frequenze genotipiche mostra la separazione fra la Zona Pirenaica e l’Andalusia e fra l’Andalusia e la Meseta. (Tabella A.25. in appendice). La differenziazione genica a coppie di popolazioni (tabella A.26) distingue la Zona Pirenaica dall’Andalusia e quest’ultima dalla Meseta, elemento indicativo vista la vicinanza geografica delle due regioni.
Per il sistema D8S566 sono stati osservati 14 genotipi, come in tabella A.27. la popolazione con il maggior numero di genotipi diversi è la popolazione della Meseta (12 genotipi), quella con il minor numero l’Andalusia (7 genotipi). Il genotipo più frequente, all’interno di tutte le popolazioni, è la combinazione allelica 3-4. La sua frequenza più bassa riguarda la popolazione dei Pirenei (24%). Si osserva un gradiente di frequenza decrescente di questo genotipo da Nord a Sud (36% nella Zona Cantabrica e 28% in Andalusia).
La distribuzione genotipica è in equilibrio di Hardy-Weinberg in ciascuna della popolazioni in esame senza eccezione alcuna e senza correzioni sui va lori di P ottenuti con il test di probabilità.
Dalla tabella A.28, che riporta le frequenze alleliche osservate per questo sistema, si nota che sono stati rilevati 6 alleli. Il più frequente è l’allele 4 di 19 ripetizioni e la distribuzione allelica è perfettamente unimodale ossia l’insieme delle forme alleliche con un numero intermedio di ripetizioni è il più rappresentativo. In due popolazioni su sei (Zona Cantabrica e Zona Mediterranea) gli alleli 3 e 4 hanno pressoché la stessa frequenza (40,4% e 38-40% rispettivamente), mentre fra la popolazione della Meseta l’allele 4 ha una frequenza un terzo più elevata di quella dell’allele 3. L’allele con 21 ripetizioni della sequenza (allele 6) è presente solo nella Meseta, ossia fra le popolazioni del centro della penisola. Le frequenze non mostrano nel complesso un gradiente geografico particolare.
I valori di diversità genica oscillano fra il 74% ed il 66%, il sistema si presenta pertanto abbastanza informativo. Non si osserva alcun particolare gradiente se non che i valori di
variabilità più bassa riguardano la popolazione Cantabrica, una popolazione isolata dalle catene montuose che la separano dal resto della Penisola.
Le tabelle A.29 e A.30 mostrano i valori di P per la differenziazione genotipica e genica a coppie di popolazioni. Nessuna delle coppie mostra valori di significativa differenziazione sia genotipica che genica.
In tabella A.31 è riportata la distribuzione genotipica per il sistema D8S591. Nel complesso i genotipi osservati sono 12; la Zona Cantabrica è quella nella quale si osserva la maggior diversità genotipica (10 forme su 12), inoltre in questa regione si osserva una frequenza rilevante del genotipo 3-5 (2,9%), presente in altre due popolazioni (Zona Mediterranea e Meseta), ma con frequenze di almeno un terzo minori. Questi due valori possono essere dovuti all’isolamento geografico delle popolazioni dei Monti Cantabrici e dei Pirenei. Il genotipo omozigote 4-4 è presente solo nella popolazione del Sud della Penisola (Andalusia). La regione che mostra il minor numero di genotipi è la Zona Pirenaica.
La distribuzione dei genotipi osservati, previa correzione di Bonferroni (Pc), è in
equilibrio di Hardy-Weinberg in tutte le popolazioni in esame.
In tabella A.32. sono riportate le distribuzioni alleliche. Sono stati osservati 5 alleli, il più frequente dei quali è quello con 10 ripetizioni (allele 1; frequenza: 42,9%). L’allele 5, il meno frequente, ha una frequenza leggermente più elevata nella regione Cantabrica. L’andamento allelico è bimodale, visto che il secondo allele più frequente è quello con 12 ripetizioni ossia l’allele 3; questo accade in tutte le popolazioni analizzate.
Ancora una volta, per avere una misura del grado di informatività del sistema, analizziamo i valori di diversità genica calcolati secondo Nei (1987). I valori sono abbastanza uniformi per tutte le popolazioni con una media di 0,68. Il marcatore D8S591 risulta dunque essere abbastanza informativo, ma meno rispetto all’altro polimorfismo STR usato in questo studio. Il valore di variabilità genica più alto riguarda la popolazione Andalusa (72%), mentre quello più basso la popolazione pirenaica (63%). Il sistema polimorfico in esame delinea una situazione di diminuzione graduale della variabilità genica dal Sud verso il Nord della Spagna.
La differenziazione genotipica e la genica a coppie di popolazioni, i cui risultati sono riportati nella tabelle A.33 ed A.34. rispettivamente, in appendice, non è significativa in nessun caso.
Diversità genica Popolazioni D1S80 D4S111 D8S566 D8S591 Hm 4 loci Zona Cantabrica 0,8325+/-0,0232 0,5751+/-0,0296 0,6664+/-0,0278 0,6781 +/- 0,0183 0,688025+/-0,024725 Zona Pirenaica 0,8469+/-0,0317 0,5610+/-0,0513 0,7290+/-0,0355 0,6378 +/- 0,0373 0,693675+/-0,03895 Zona Mediterranea 0,8505+/-0,0258 0,5497+/-0,0403 0,6887+/-0,0355 0,6882 +/- 0,0192 0,694275+/-0,0302 Meseta 0,8125+/-0,0187 0,5554+/-0,0274 0,6771+/-0,0217 0,6799 +/- 0,0172 0,681225+/-0,02125 Andalusia 0,7760+/-0,0422 0,5333+/-0,0438 0,7492+/-0,0361 0,7247 +/- 0,0246 0,695800+/-0,036675
3.2.2 DIVERSITÀ GENICA
Al fine di realizzare un confronto più rigoroso ed analitico delle popolazioni considerate, anche a questo secondo livello di analisi è stato quantificato l’indice di diversità genica media per ogni sistema esaminato e quindi la diversità media con il relativo errore standard. Come spiegato nella sezione relativa ai metodi statistici, i valori di diversità genica (Gene Diversity) e relativo errore sono stati stimati con il metodo di Nei (1974, 1987). Come si può osservare nella tabella 3.2.2 i valori più alti riguardano il locus D1S80 ed i più bassi il locus D4S111 che è infatti il meno polimorfico. In un confronto a 4 loci la variabilità più alta in assoluto è quella riguardante la popolazione dell’Andalusia. La variabilità più bassa in assoluto riguarda invece la popolazione della Meseta. La media dei valori di variabilità a 4 loci è 0,69. Il valore di informatività è dunque abbastanza alto per il totale del campione oggetto di questo studio, ma i valori sono molto omogenei nelle varie popolazioni (68-69%).
3.2.3 ANALISI DEGLI APLOTIPI E DELLE DISTANZE GENETICHE
L’analisi aplotipica è stata condotta usufruendo dei dati di 4 dei 5 loci analizzati vista la forte deviazione dall’equilibrio di HardyWeinberg per il polimorfismo D17S5.
Nella popolazione della regione Cantabrica sono stati osservati ben 104 aplotipi, il più frequente ha frequenza del 4% (aplotipo 18,2,3,1). Lo stesso aplotipo presenta il valore di massima frequenza all’interno della popolazione della Zona Mediterranea (frequenza: 2,8%). Nella Zona Pirenaica presenta massima frequenza (4,5%) un aplotipo che non è comune nelle altre popolazioni, è l’aplotipo 18,3,3,3. La Zona Pirenaica è quella con un numero minore di varianti aplotipiche: solo 52 contro le 143 presenti nella popolazione della Meseta. L’aplotipo più comune in quest’ultima è l’aplotipo 24,2,4,1 (frequenza:5%) tra i più frequenti nella totalità del campione analizzato. L’Andalusia mostra un aplotipo caratteristico della regione (18,2,2,1), con la frequenza più alta all’interno della popolazione andalusa (4,8%).
La tabella 3.2.3 mostra i risultati del test di differenziazione genotipica a coppie di popolazioni ottenuti in base alle frequenze genotipiche complessive di tutti i sistemi
DIFFERENZIAZIONE GENOTIPICA A COPPIE DI POPOLAZIONI 4 loci
Pop1 Pop2 X2 d.f. P-value
Pirenaica Cantabrica 9.396 8 0.31000 Mediterranea Cantabrica 11.938 8 0.15397 Mediterranea Pirenaica 6.611 8 0.57912 Meseta Cantabrica 14.073 8 0.07987 Meseta Pirenaica 8.308 8 0.40394 Meseta Mediterranea 5.297 8 0.72546 Andalusia Cantabrica 13.098 8 0.10852 Andalusia Pirenaica 10.520 8 0.23041 Andalusia Mediterranea 7.935 8 0.43989 Andalusia Meseta 10.081 8 0.25942
Tabella 3.2.3.: Approccio geografico: differenziazione genotipica a coppie di popolazioni calcolata sulle
frequenze genotipiche di 4 loci.
utilizzati. La differenziazione non è significativa, ossia le distribuzioni osservate risultano essere sostanzialmente omogenee. Il test esatto di differenziazione fra popolazioni, basato sulle frequenze aplotipiche e calcolato con il metodo di Goudet (1996), è invece significativo (P=0,03) nel complesso delle popolazioni. Lo stesso test applicato a coppie di popolazioni, riportato in figura 3.2.1, mostra differenze significative fra la popolazione della Zona Cantabrica e la popolazione della Meseta e tra quest’ultima e la popolazione della Zona mediterranea.
Figura 3.2.1.: Approccio geografico: test di differenziazione a coppie di popolazioni con i dati
su 4 loci.
CANTA PIREN MEDIT MESET ANDAL
CANTA
PIREN 0.21287+-0.0132
MEDIT 0.21443+-0.0324 0.07757+-0.0115
MESET 0.00112+-0.0006 0.16700+-0.0266 0.00005+-0.0001
ANDAL 0.40713+-0.0342 0.45559+-0.0266 0.52421+-0.0386 0.22141+-0.0324
La tabella 3.2.4, riportai valori di distanza genetica della corda (Cavalli-Sforza e Edwards, 1964) fra le popolazioni analizzate. La distanza media è 0,104. La popolazione della Meseta è quella che in media dista meno da tutte le altre (distanza=0,095), dato che ci si può facilmente spiegare alla luce vista non solo della
storiche che fanno di questa zona il centro di amalgama dell’intera nazione spagnola. La popolazione che più si separa dalle altre, in modo molto marcato è la popolazione andalusa (distanza=0,118). Le distanze maggiori in assoluto sono quelle fra l’Andalusia e le regioni del Nord e le minori quelle fra la Meseta e la Cantabria e fra la Meseta e la Zona Mediterranea ossia fra la regione del centro e le zone costiere.
Il metodo MDS è stato applicato alle distanze genetiche escludendo i dati relativi al sistema D17S5. La sua rappresentazione grafica è data in figura 3.2.2. Si nota la presenza di un cluster principale dato dalle popolazioni del centro (Meseta), del versante mediterraneo e di quello atlantico. Le popolazioni della zona pirenaica e del sud (Andalusia), invece, si collocano ai due estremi della prima dimensione, distanti dall’insieme suddetto.
Tabella 3.2.4.: Approccio geografico: matrice delle distanze genetiche calcolate con il metodo
della corda (Cavalli-Sforza e Edwards, 1964).
CANTA PIREN MIDIT MESET ANDAL
CANTA
PIREN 0,106944
MIDIT 0,082639 0,10625
MESET 0,088889 0,099306 0,08125
Meseta Andalusia Cornice cantabrica Zona mediterranea Zona pirenaica -1,2 -0,6 0 0,6 1,2 -1 -0,5 0 0,5 1 1,5 Dimensione 1 Dimensione 2
3.3. ANALISI III: UN APPROCCIO LINGUISTICO
A questo livello di studio sono state analizzate 9 popolazioni in base a criteri di tipo linguistico, le popolazioni in esame sono riportate in tabella 3.3.1. Anche in questo caso l’intero campione è stato tipizzato per i 5 sistemi VNTR e STR, ma i dati sul locus D17S5 sono stati esclusi dall’analisi finale.
3.3.1 ANALISI LOCUS PER LOCUS
Con questo terzo approccio analitico, per il sistema D1S80, sono stati osservati 25 alleli, con una media di 13 alleli per popolazione. La popolazione con il minor numero di alleli è quella di lingua galiziana, mentre quella di lingua basca mostra 16 forme alleliche distinte. La distribuzione è riportata in tabella A.35. Gli alleli più frequenti sono il 18 ed il 24. L’allele 18 raggiunge il valore di frequenza massimo nella regione di lingua (dialetto) aragonese (32%) ed il minimo nella regione di lingua basca (20,6%). La popolazione di lingua valenciana mostra il valore di frequenza minimo per l’allele 24. La popolazione di lingua basca è l’unica che possiede gli alleli con il più alto numero di ripetizioni (39 e 41). La distribuzione degli alleli nelle diverse popolazioni vede perlopiù favorite le forme alleliche con un numero intermedio di ripetizioni.
Popolazione D1S80 D4S111 D8S566 D8S591 D17S5 Galiziano 38 50 50 22 44 Basco 68 72 70 54 70 Catalano 38 62 64 44 56 Valenciano 46 54 48 28 54 Aragonese 56 60 56 52 64 Castigliano 74 80 70 54 64 Castigliano_sud 76 76 78 60 70 Murciano 46 52 52 38 48 Andaluso 64 68 66 42 62 Totale (2N) 506 573 554 394 532
Tabella 3.3.1: Approccio linguistico: database generale delle popolazioni analizzate. È riportato
La distribuzione genotipica è in equilibrio di Hardy-Weinberg in tutte le popolazioni analizzate previa correzione di Bonferroni (Pc=0,005). Tabella A.36.
La differenziazione genotipica a coppie di popola zioni, mostrata in tabella A.37 in appendice, distingue, ampiamente all’interno della soglia di significatività, la popolazione di lingua basca da quelle di lingua catalana e valenziana (dialetto del catalano), la popolazione di lingua catalana da quella che parla castigliano e la popolazione di lingua galiziana da quelle che parlano murciano e castigliano.
La differenziazione genica a coppie di popolazioni è riportata in tabella A.38 in appendice e risulta significativa per le seguenti coppie: Galiziano-Castigliano, Galiziano-Murciano, Basco-Catalano, Basco-Valenciano, Basco-Andaluso, Catalano-Castigliano e Valenziano-Catalano-Castigliano.
In tabella A.39 si trovano i valori di frequenza genotipica del polimorfismo D4S111. Sono stati osservati 6 genotipi dei quali il più frequente è l’omozigote 2-2 ed il secondo più frequente al combinazione allelica 2-3 con rispettiva frequenza del 34% e del 33%. La distribuzione del genotipo 2-2 mostra un gradiente decrescente di frequenza dalla popolazione di lingua aragonese (frequenza: 47%) verso il resto delle popolazioni con una distribuzione a raggiera che ha il suo minimo fra la popolazione di lingua murciana (frequenza: 23%) . Infatti il genotipo 2-2 non è il più comune all’interno di tutti i campioni analizzati: nelle popolazioni basca, galiziana, murciana e andalusa il genotipo 2-3 è il più frequente. Questa combinazione allelica mostra i valori più alti di frequenza fra le popolazioni più meridionali (Andaluso con frequenza 0,44) e quelle più settentrionali (Galiziano con frequenza 0,44) ed i più bassi nella popolazione di lingua castigliana (frequenza 0,23).
La distribuzione è in accordo con la situazione di equilibrio di Hardy-Weinberg. La tabella A.40 riporta le frequenze dei 3 alleli osservati. L’allele più frequente è l’allele 2 (8 ripetizioni), presente nel 60% dell’intero campione analizzato. Il secondo allele più frequente, quello con 9 ripetizioni (allele 3), ha una frequenza di ben tre volte minore rispetto all’allele 2 (24%).
La differenziazione genica a coppie di popolazioni non distingue in modo significativo nessuna popolazione dalle altre. La differenziazione genotipica, ugualmente a coppie di popolazioni, conferma questa situazione. Le tabelle A. 41 e A. 42 riportano questi risultati.
Per il sistema D8S566, i valori di frequenza genotipica sono mostrati i tabella A.43. Sono stati osservati 16 genotipi il più frequente dei quali è, come atteso, quello dato dalla combinazione dei due alleli più comuni. Gli alleli rilevati sono 6; le loro frequenze si possono le ggere in tabella A. 44. Il più frequente è l’allele 4 con frequenza del 40%. In realtà, anche se nell’intero campione è questo l’allele più frequente, in cinque popolazioni su nove l’allele 3 è predominante. Nella popolazione di galiziana l’allele 3 raggiunge addirittura lo 0,5 di frequenza. L’allele 6 si ritrova solamente nelle popolazioni che parlano Murciano e Aragonese.
I risultati del test di Hardy-Weinberg in tabella A.36 mostrano che la popolazione è in equilibrio.
La differenziazione genotipica e quella genica a coppie di popolazioni non sono significative in nessun caso. Tabelle A. 45 e A. 46.
Per il sistema D8S591 la tabella con le frequenze genotipiche è riportata in tabella A. 47 in appendice. Sono stati osservati 12 genotipi. In media ogni popolazione possiede 8-9 forme genotipiche distinte ad eccezione della popolazione di lingua galiziana per la quale sono state descritte solo 5 forme genotipiche diverse. Questa popolazione mostra una bassa variabilità genotipica anche negli altri loci analizzati. Il genotipo più frequente è la combinazione allelica 1-3 (frequenza: 31%). Questo accade in tutte le popolazioni analizzate ad eccezione della popolazione di lingua valenciana che mostra più genotipi omozigoti 1-1 (frequenza: 22%).
Come si può notare in tabella A.48 sono stati rilevati 5 alleli. L’allele più frequente è l’1 (43%). L’allele 5 è presente in 4 popolazioni su 9 con frequenze basse (dell’ordine di 0,015) ad eccezione della popolazione di lingua basca che mostra una frequenza di 0,043. L’allele 4 raggiunge il livello massimo di frequenza all’interno della popolazione che parla dialetto andaluso e il livello minimo in quella di lingua basca. Gli alleli 1 e 2 hanno frequenze distribuite in modo omogeneo nei gruppi analizzati.
La differenziazione genotipica a coppie di popolazioni non distingue significativamente nessun gruppo dagli altri. La differenziazione genica non mostra valori di significatività nell’intero campione né, con l’analisi a coppie di popolazioni, si osservano diversità significative. Differenziazione genotipica e genica a coppie di popolazioni sono riportate nelle tabelle A. 49 e A. 50 in appendice.
Anche a questo livello analitico, è stato applicato il metodo MDS alla matrice delle distanze genetiche ottenute con il metodo della corda. La rappresentazione grafica del MDS in figura 3.3.1, evidenzia la presenza di un cluster centrale di lingua castigliana attorno al quale “orbitano” tutte le altre lingue e dialetti della Spagna.
3.3.2 ANALISI SEPARATA PER I DUE TIPI DI POLIMORFISMI
Al fine di comprendere al meglio l’apporto informativo fornito all’analisi dai due sistemi molecolari utilizzati è stata condotta un’analisi separata per i minisatelliti ed i microsatelliti. Popolazione D8S566 D8S591 Galiziano 38 50 Basco 68 72 Catalano 38 62 Valenziano 46 54 Castigliano 74 80 Andaluso 64 68 Totale 328 386
Tabella 3.3.2 : Database delle popolazioni analizzate con l’uso dei marcatori minisatelliti
Popolazione D1S80 D4S111 Galiziano 22 44 Basco 54 70 Catalano 44 56 Valenciano 28 54 Castigliano 54 64 Andaluso 42 62 Totale 244 350
Figura 3.3.1: Rappresentazione del Multidimensional Scaling ottenuto dalla matrice di distanze genetiche Dc basate sulle frequenze degli aplotipi a 4 loci. Stress=0,1163. Murciano Basco Catalano Castigliano sud Castigliano Andaluso Aragonese Valenziano Galiziano -1,2 -0,6 0 0,6 1,2 -2 -1,5 -1 -0,5 0 0,5 1 1,5 2 Dimensione 1 Dimensione 2
Quest’analisi separata per i due sistemi segue anch’essa un approccio linguistico. Per rendere più fine lo studio a questo livello, sono state considerate solamente 6 popolazioni rappresentanti le quattro lingue principali (Basco, Castigliano, Catalano e Galiziano) ed i due dialetti più caratterizzati linguisticamente (Andaluso del Castigliano e Valenziano del Catalano) (tabelle 3.3.2 e 3.3.3).
3.3.3 DIVERSITÀ GENICA
È stato calcolato l’indice di diversità genica di ciascuna popolazione con il relativo errore standard secondo Nei (1987), separatamente per i sistemi VNTR e per i sistemi STR con lo scopo di quantificare il grado di risoluzione di questi in ciascuna popolazione e quindi nell’intero campione analizzato (tabella 3.3.4).
I valori di massima variabilità sono principalmente quelli calcolati sui dati dei VNTR anche se i valori degli STR non si discostano di molto. Di solito i microsatelliti sono più informativi rispetto ai minisatelliti e sono preferiti negli studi di antropologia molecolare, ma in questo caso, l’alto polimorfismo del sistema D1S80 conferisce al gruppo VNTR questa proprietà. La variabilità più alta in assoluto riguarda la popolazione di lingua basca che mostra il secondo valore più alto anche tra i dati dei microsatelliti. La variabilità più bassa in assoluto riguarda invece la popolazione di lingua galiziana, che però mostra valori intermedi per i marcatori minisatelliti.
DIVERSITÀ GENICA
Pop Microsatelliti Minisatelliti
Galiziano 0.8827 +/- 0.0257 0.9314 +/- 0.0180 Basco 0.9242 +/- 0.0114 0.9476 +/- 0.0111 Catalano 0.9000 +/- 0.0187 0.9191 +/- 0.0178 Valenziano 0.8910 +/- 0.0208 0.9336 +/- 0.0173 Castigliano 0.9025 +/- 0.0233 0.8953 +/- 0.0240 Andaluso 0.9381 +/- 0.0101 0.9118 +/- 0.0217
Tabella 3.3.4.: Indici di diversità genetica e relativo errore standard secondo Nei (1987) nelle
I sistemi microsatelliti
I test di differenziamento fra popolazioni in base ai dati aplotipici è stato condotto secondo Goudet, J., M. Raymond, T. de Meeüs and F. Rousset, 1996. I risultati mostrano valori che differenziano in modo significativo (P<0,05) i galiziani dai catalani, i galiziani dai castigliani, i baschi dai valenziani, ed ancora i valenziani dai castigliani. Tali differenziazioni rispondono a quanto atteso per popolazioni quali i galiziani, i catalani ed i baschi, genti con lingua propria ed una forte identità culturale. La matrice con i valori di P per questo test è mostrata in figura 3.3.2. Gli aplotipi più comuni all’interno di tutte le popolazioni in esame sono le combinazioni degli alleli più frequenti di ognuno dei due loci. L’aplotipo 1-3 è il più frequente in quattro popolazioni su sei. La popolazione di lingua castigliana mostra il massimo di frequenza (20%) per l’aplotipo 1-4, che ha invece il 10% o meno di frequenza fra le altre popolazioni. Fra la popolazione di lingua catalana l’aplotipo 4-4 è il più frequente (19%).
Tutte le popolazioni analizzate mostrano i valori attesi di eterozigosi ad eccezione della popolazione di lingua catalana che si discosta significativamente dal valore atteso per il sistema D8S591. La popolazione mostra eccesso di eterozigosi. La popolazione andalusa mostra invece, sempre per lo stesso locus, difetto di eterozigosi. È stata calcolata la distanza genetica tra coppie di popolazioni mediante la formula della distanza della corda di C.Sforza-Edwards (1964). I valori ottenuti sono riportati nella tabella 3.3.5.
GAL BAS CAT VAL CAS AND
Galiziano GAL Basco BAS 0.17446+-0.0110 Catalano CAT 0.04956+-0.0056 0.85405+-0.0063 Valenziano VAL 0.55398+-0.0101 0.03102+-0.0033 0.14012+-0.0057 Castigliano CAS 0.00368+-0.0005 0.27446+-0.0186 0.77139+-0.0082 0.00219+-0.0004 Andaluso AND 0.62393+-0.0122 0.54311+-0.0074 0.35987+-0.0133 0.10328+-0.0068 0.16856+-0.0118
Figura 3.3.2.: Test di differenziazione aplotipica a coppie di popolazioni con i dati sui sistemi
6 popolazioni (loci STR)
GAL BAS CAT VAL CAS AND Galiziano GAL Basco BAS 0.138 Catalano CAT 0.152 0.086 Valenciano VAL 0.067 0.160 0.170 Castiglian CAS 0.141 0.119 0.112 0.147 Andaluso AND 0.148 0.157 0.161 0.126 0.119
Tabella 3.3.5 : Matrice di distanze genetiche calcolate con il metodo della corda
(Cavalli-Sforza e Edwards, 1964)
La rappresentazione grafica del MDS (Figura 3.3.3), messa a punto con il procedimento MDS applicato alle distanze genetiche Dc, non ci permette di dedurre un particolare pattern genetico sulla popolazione spagnola analizzata. Visti i valori di variabilità i loci microsatelliti utilizzati sono informativi, ma è necessario avere dati su un maggior numero di loci.
Figura 3.3.3 : Rappresentazione del MDS ottenuto dalla matrice di distanze genetiche Dc basate sulle frequenze degli aplotipi dei microsatelliti. Stress=0,0006. Basco Catalano Andaluso Castigliano Valenziano Galiziano -1,2 -0,6 0 0,6 1,2 -2 -1,5 -1 -0,5 0 0,5 1 1,5 2 Dimensione 1 Dimensione 2
I sistemi minisatelliti
I test di differenziamento fra popolazioni in base ai dati aplotipici è stato condotto secondo Goudet, J., M. Raymond, T. de Meeüs and F. Rousset, 1996. I risultati, riportati in figura 3.3.4., mostrano valori che differenziano in modo significativo (P<0,05) la popolazione di lingua catalana da quelle di lingua basca, valenziana, castigliana e Andalusa. Il valenziano è un dialetto del catalano, ma le due popolazioni sono sempre rimaste geneticamente isolate l’una dall’altra.
Gli aplotipi più frequenti sono il 2-18 ed il 2-24, ossia la combinazione degli alleli più frequenti di entrambi i polimorfismi in analisi. Le popolazioni catalana e valenziana sono in difetto di eterozigosi per il locus D1S80.
È stata calcolata la distanza genetica tra coppie di popolazioni mediante la formula della distanza della corda di C.Sforza-Edwards (1964) riportata nella tabella 3.3.6.
Alle distanze genetiche Dc è stato applicato il procedimento MDS. La rappresentazione grafica del MDS (Figura 3.3.4) non ci permette di dedurre un particolare pattern genetico sulla popolazione spagnola analizzata. Probabilmente è dovuto al fatto che solo uno dei due marcatori utilizzati (il D1S80) sia realmente informativo.
GAL BAS CAT VAL CAS AND
Galiziano GAL Basco BAS 0.10376+-0.0137 Catalano CAT 0.21926+-0.0103 0.00000+-0.0000 Valenziano VAL 0.53740+-0.0114 0.06706+-0.0118 0.02222+-0.0034 Castigliano CAS 0.08100+-0.0122 0.40718+-0.0155 0.00000+-0.0000 0.07454+-0.0080 Andaluso AND 0.22561+-0.0131 0.24558+-0.0100 0.00017+-0.0002 0.15882+-0.0159 0.51154+-0.0126
Figura 3.3.4.: Test di differenziazione aplotipica a coppie di popolazioni con i dati sui sistemi
2 loci 6 popolazioni (VNTR)
GAL BAS CAT VAL CAS AND Galiziano GAL Basco BAS 0.216 Catalano CAT 0.176 0.236 Valenziano VAL 0.202 0.247 0.180 Castiglian CAS 0.202 0.171 0.175 0.177 Andaluso AND 0.228 0.215 0.211 0.238 0.176
Tabella 3.3.6 : Matrice di distanze genetiche calcolate con il metodo della corda
Basco Catalano Andaluso Castigliano Valenziano Galiziano -1,2 -0,6 0 0,6 1,2 -2 -1,5 -1 -0,5 0 0,5 1 1,5 2 Dimensione 1 Dimensione 2