1. INTRODUZIONE
Il lavoro di questa tesi è un’indagine sulla genetica di popolazioni e sulla filogeografia del riccio di mare edule Paracentrotus lividus Lamarck, 1816 (Echinodermata, Echinoidea). Nel presente lavoro è stata analizzata la struttura genetica della specie in rapporto alle sue capacità di dispersione e/o l’esistenza di parziali o totali barriere geografiche, fisiche ed ecologiche al flusso genico, che possono portare a relativo isolamento delle popolazioni.
Un altro obiettivo del lavoro, di carattere applicativo, riguarda la pesca di P. lividus, che viene esercitata sia a livello amatoriale che professionale. Il prelievo di questa specie può raggiungere localmente livelli molto intensi e rivestire anche un notevole interesse economico (Palacín et al., 1997; Baroli et al., 2006), tanto da indurre alla costituzione di impianti di acquacoltura e maricoltura specializzati. Con il presente lavoro si è effettuato un tentativo di identificazione degli stock di P. lividus e di valutare lo stato di “salute genetica” della specie. I marcatori genetici molecolari possono infatti fornire importanti informazioni per la valutazione degli stock (Carvalho & Hauser, 1994; Grant & Bowen, 1998; Cassens et al., 2003) e di conseguenza consentono, unitamente ai risultati di studi demografici e di dinamica di popolazione, una gestione delle risorse pescabili più corretta e responsabile.
1.1. Il genere Paracentrotus Mortensen, 1903
Il genere Paracentrotus è uno dei due generi di echinoidi che presentano piastre ambulacrali polipore (l’altro è Loxechinus, distribuito in America meridionale), presenta tubercoli secondari grandi, apparato apicale con piastre terminali di solito esterne, membrana peristomiale con scarse piastrine, valve dei pedicellari globiferi con un dentello per lato, pedicellari tridattili di uno o due tipi (Tortonese, 1965). È stato a lungo confuso con il genere Strongylocentrotus Brandt, che appartiene ad un’altra famiglia (Strongylocentrotidae) ed è assente nel Mediterraneo. Al genere Paracentrotus sono state attribuite specie fossili del Miocene e Pliocene, ma attualmente il genere è bispecifico, con P. lividus e P. gaimardi.
1.2. Biologia ed ecologia di Paracentrotus lividus Lam., 1816 Sinonimi:
Echinus lividus Lamarck, 1816. Echinus saxatilis Tiedemann, 1816. Echinus vulgaris Blainville, 1825. Echinus purpureus Risso, 1826. Toxopneustes lividus Heller, 1868.
Strongylocentrotus lividus A Agassiz, 1872-74; Ludwig, 1879; Koehler, 1883; Carus, 1885; Checchia-Rispoli, 1906; Lo Bianco, 1909.
Paracentrotus lividus Mortensen, 1903; Koehler, 1921; Ranson, 1925; HL Vatova, 1928; Tortonese, 1933; Dieuzeide, 1935; Cherbonnier, 1965.
Secondo quanto riportato da Tortonese (1965), P. lividus (Fig. 1.1) si ritrova nel Mar Mediterraneo ed in prossimità delle coste dell’Oceano Atlantico nord-orientale, dall’Irlanda e dalla Scozia fino al Marocco, alle Canarie, a Madeira e alle Azzorre. Si riscontra dalla superficie fino a 80 m di profondità, ma più frequentemente viene osservato entro i 20 m. È specie tipicamente infralitorale, vive sugli scogli, sui
bassifondi sassosi e rocciosi con alghe, nelle praterie di Posidonia oceanica; preferisce ambienti riparati dalla luce e ricchi di vegetazione. In P. lividus il contorno del corpo è di regola circolare, ma può tendere ad una forma pentagonale. Uno dei caratteri morfologici più variabili è il colore degli aculei, che può andare dal nero al violetto, a varie tonalità di verde, al bruno, al rossiccio. Sono stati anche osservati individui di colore chiarissimo, quasi bianchi. La sua attitudine a scavarsi nicchie nella roccia, che in questo modo può apparire addirittura alveolata quando questi echinoidi vi alloggiano in gran numero, si manifesta molto più in Atlantico che in Mediterraneo. P. lividus può vivere in ambienti caratterizzati da imprevedibilità ambientale, quali i porti e gli ambienti salmastri. Questo echinoide suole ricoprirsi di corpi estranei (pietruzze, conchiglie, pezzetti di alghe), che trattiene per mezzo dei pedicelli; il significato biologico di questo comportamento non è del tutto compreso ed esperienze di laboratorio volte ad indagare questo aspetto hanno messo in evidenza una correlazione positiva nei confronti sia dell’esposizione alle radiazioni UV (Verling et al., 2002), che della quantità di sedimento fine sospeso nell’acqua (Richner & Milinski, 2000). P. lividus si nutre di alghe e di foglie di Posidonia, ma il cibo animale non è escluso. L’attività alimentare avviene soprattutto durante la notte. È considerato da molti Autori come fondamentale nella strutturazione delle comunità fitali in maniera diretta, ma anche delle comunità zoobentoniche (Sammarco, 1980; Himmelman et al., 1983). L’attività alimentare può causare la rimozione totale di letti di alghe, evitando il sopravvento di specie favorite dal punto di vista biologico nella competizione per lo spazio, e può creare zone nude che possono essere colonizzate da organismi incrostanti o sessili, favorendo una maggiore articolazione dei popolamenti bentonici di fondi duri in generale (Sala et al., 1998). Esperimenti volti a verificare l’effettiva importanza dei popolamenti di P. lividus e dell’altro echinoide Arbacia lixula L. sulle comunità di fondi duri, hanno evidenziato come l’effetto degli echinoidi sia di difficile interpretazione: è stato suggerito che ci possano essere notevoli discrepanze nell’entità e nella natura di tali effetti in relazione alle caratteristiche trofiche e, più in generale, ecologiche delle singole località prese in esame (Benedetti Cecchi et al., 1998; Bulleri et al., 1999). P. lividus compie spostamenti con ritmo nictemerale, principalmente allo scopo di cibarsi: di notte si porta allo scoperto sulle rocce, si avvicina alla superficie e, allo stato giovanile, si
arrampica sulle foglie di Posidonia.
La riproduzione prevede fecondazione esterna, con la liberazione in acqua dei gameti; cellule uovo e spermatozoi possono sopravvivere 24h o poco più, almeno in condizioni sperimentali, e la probabilità che avvenga fecondazione diminuisce drasticamente dopo le prime ore di vita (Levitan et al., 1991; L. Kozinkova, comunicazione personale). La larva, un echinopluteo (Fig. 1.2), è liberamente natante e planctontrofica, può vivere da 20 fino a 40 giorni prima di fissarsi al substrato e compiere la metamorfosi che infine comporta il passaggio da simmetria bilaterale a pentamera (Fig. 1.3) e l’assunzione dell’aspetto che tutti conoscono. Lo sviluppo sessuale sembra essere raggiunto intorno al terzo dei presunti nove anni di vita (Crapp & Willis, 1975).
Fig. 1.2: Fasi dello sviluppo dell’echinopluteo di Paracentrotus lividus.
Fig. 1.3: Metamorfosi del Pluteo in Paracentrotus lividus.
La maturazione delle gonadi sembra essere indotta da un fotoperiodo minore di 12 ore (Shpigel et al., 2004); per il Mediterraneo è stata rilevata la presenza di uno (Zavodnik, 1987; Byrne, 1990; Lozano et al., 1995) o due (Fenaux, 1968; Crapp & Willis, 1975; Pedrotti, 1993) picchi di maturazione delle gonadi a seconda delle località geografiche analizzate, ma non è chiaro se alla maturazione segua un’effettiva liberazione dei gameti o se, ancora più importante, le larve originate riescano ad insediarsi e svilupparsi correttamente. I plutei si ritrovano infatti nel plancton durante tutto l’arco dell’anno, e due studi condotti alle isole Medas (Catalogna, Spagna) sull’efficacia dell’insediamento hanno rilevato un solo picco di reclutamento in primavera, risultante dal più rapido e consistente picco di maturazione gonadica dei mesi invernali (Hereu et al., 2004), oppure due picchi, uno in primavera ed uno in autunno, dei quali il primo più consistente (Tomas et al., 2004). La maturazione delle gonadi è indipendente dalla temperatura dell’acqua, la liberazione in massa dei gameti, invece, sarebbe innescata solo in seguito ad una soglia minima di temperatura (Barnes et al., 2001). Questo sarebbe il motivo di crolli demografici di notevole entità sulle coste irlandesi che sembrano trovare una spiegazione basata sulla presenza di eventi ENSO (El Nino Southern Oscillation). I fenomeni ENSO sono in grado di modificare il corso della Corrente del Golfo, che normalmente arriva a lambire le coste più occidentali di Irlanda e Gran Bretagna con le sue acque sensibilmente più calde; quando il normale regime idrodinamico viene alterato, la temperatura superficiale delle acque ne risente in maniera sensibile. Secondo Barnes et al. (2001), in alcuni anni gli effetti dei fenomeni ENSO sarebbero tali da impedire il raggiungimento della soglia minima di “spawning” di P. lividus, impedendo il reclutamento di intere coorti di larve. La natura del substrato non sembra essere, invece, importante ai fini del reclutamento (Hereu et al., 2004), anche se potrebbe poi influire, a seconda delle sue caratteristiche, sulla maggiore o minore protezione nei confronti della predazione o dei fenomeni fisici o atmosferici e quindi, in ultima analisi, sulla mortalità (Hereu et al., 2005). Le cause della mortalità post-insediamento non sono attualmente chiarite ma hanno un ruolo fondamentale nel plasmare la densità delle popolazioni di P. lividus: a fronte di un reclutamento
massiccio, infatti, la mortalità post-insediamento arriva al 75% nei primi sei mesi di vita (Sala & Zabala, 1996) ed al 99% al termine del primo anno (Lopez et al., 1998). Un altro importante fattore di mortalità, almeno nel Mediterraneo, sembra essere la predazione da parte di pesci (Coris julis e Diplodus spp.) (Sala, 1997; Guidetti & Mori, 2005), che sembrerebbe meno importante in ambiente atlantico (Barnes & Crook, 2001). Anche la mortalità dovuta alla pesca dilettantistica e professionale, quando viene praticata intensamente, può assumere una notevole entità, ma ciò è spesso limitato alle zone più superficiali e a luoghi facilmente accessibili da terra o nelle vicinanze di piccole marinerie (Guidetti et al., 2004). Negli ultimi anni, anche in Toscana, precisamente sul litorale di Massa, la fioritura di dinoflagellati bentonici si è rivelata un’importante fonte di mortalità a livello locale. I dinoflagellati infatti sono noti produttori di tossine che possono causare danni di vario grado negli echinoidi che li ingeriscono insieme al cibo. Questi danni vanno dalla perdita degli aculei fino addirittura alla morte, che può colpire moltissimi individui in brevissimo tempo. Sono state registrate anche intossicazioni in soggetti umani che si erano alimentati di ricci contenenti tossine accumulate nei tessuti (Sansoni et al., 2003).
1.3. La variabilità genetica all’interno delle specie
In ambiente marino la diversità genetica è distribuita su scale spaziali diverse, che vanno dai pochi metri alle migliaia di chilometri (Procaccini & Maltagliati, 2003). La distanza fisica esistente fra gli organismi può influenzare il livello dei rapporti genetici esistenti fra essi ed agire come fattore gerarchizzante. I pattern di diversità genetica osservati variano in rapporto ai cicli vitali ed alla capacità di dispersione delle specie: generalmente, specie con alte capacità di dispersione tendono ad essere meno geneticamente strutturate, grazie all’azione omogeneizzante del flusso genico (Procaccini & Maltagliati, 2003). È da notare, comunque, che recentemente sono state trovate eccezioni a questa regola (Swearer et al. 1999; Luttikhuizen et al., 2003; Taylor & Hellberg, 2003).
La variabilità genetica rappresenta la base necessaria per i futuri cambiamenti evolutivi delle specie ed i livelli di diversità genetica nelle popolazioni possono fornire le prove di eventi evolutivi avvenuti in tempi passati più o meno recenti (Weir, 1990).
L’evoluzione è, in ultima analisi, il risultato di cambiamenti successivi nei tipi e nelle frequenze dei geni nelle popolazioni; ciò determina e rispecchia l’adattamento degli organismi e delle popolazioni all’ambiente. La variabilità genetica presente nelle popolazioni naturali è il risultato dell’interazione di fattori genetici, biologici, ecologici ed evolutivi che agiscono in sinergia sulle specie. Le modalità di ingresso di nuova variabilità genetica in una popolazione sono ritenute classicamente la mutazione, che agisce su scale di tempo relativamente grandi ed il flusso genico, che può permettere l’ingresso di nuovi geni mediante l’incrocio degli individui nativi con gli immigranti (Hartl & Clark, 1993). Le popolazioni naturali di una determinata specie, che appartengono a regioni geograficamente distinte, presentano quasi sempre differenze nella distribuzione dei geni al loro interno, ciò indica la presenza di strutturazione genetica in quella specie. La struttura genetica attuale delle specie è il risultato di processi storici e delle forze di tipo microevolutivo (Hartl & Clark, 1993). La struttura genetica delle specie e, quindi, i meccanismi evolutivi e le strategie adattative da esse adottate, possono essere conosciuti analizzando la distribuzione dei geni all’interno delle popolazioni.
Nonostante esistano vari metodi per valutare la struttura genetica di una specie questi forniranno informazioni solo su un piccolo campione, sia perché il numero di individui analizzati rispetto alla taglia della popolazione sarà limitato, sia perché l’informazione sarà limitata a quella fornita dai marcatori genetici utilizzati (Procaccini & Maltagliati, 2003). I dati ottenuti saranno quindi soggetti ad errore di campionamento per quanto riguarda i parametri di variabilità genetica in senso assoluto, ma saranno estremamente utili per analisi comparative tra le popolazioni. Le stime del grado di relazione dei geni tra individui e tra popolazioni possono essere utilizzate come approccio alla comprensione dell’azione relativa delle forze relative che agiscono sulla specie considerata. Ogni stima può essere valutata grazie all’esistenza di vari modelli teorici semplificativi sviluppati dalla nascita della genetica di popolazioni ad oggi; tali modelli si fondano su assunzioni che approssimano le caratteristiche genetiche delle popolazioni naturali. Il classico modello di Hardy-Weinberg, per esempio, è basato su una popolazione di taglia infinita e panmittica, con tasso di mutazione pari a zero ed assenza di fenomeni come migrazione e selezione. Una popolazione con caratteristiche simili è ovviamente inesistente in
natura, ciò nonostante il modello di Hardy-Weinberg è tutt’oggi di grande utilità; può essere utilizzato, per esempio, per ipotizzare la presenza di fenomeni selettivi oppure di effetto Wahlund (1928).
La distribuzione geografica della variabilità genetica entro una specie è regolata da una serie di fattori che possono essere riuniti in due categorie principali:
1. fattori intrinseci a particolari specie che determinano differenti livelli di flusso genico (per esempio capacità di dispersione, comportamento riproduttivo, specificità di habitat);
2. fattori estrinseci, di tipo geologico, fisico e climatico che possono essere a lungo termine (modifica dell’habitat per movimenti tettonici, avanzamento o scioglimento dei ghiacci, variazioni del livello del mare) ed a breve termine (perdita di habitat ad opera dell’uomo, introduzione di specie alloctone).
L’importanza relativa della selezione naturale e della deriva genetica è stata a lungo dibattuta in passato dagli evoluzionisti (Lewontin, 1991; Avise, 1994). Negli ultimi anni le problematiche relative a tale dibattito, per ora irrisolto, sono state praticamente abbandonate e l’attenzione è stata spostata verso lo studio della struttura genetica delle specie sulla base della distribuzione geografica delle popolazioni ed anche dei loro rapporti filogenetici (Avise, 2000). Negli studi di questo tipo ci si attiene alla teoria della neutralità (Kimura, 1982) e si assume che la selezione a livello locale sia una causa secondaria e trascurabile nei modelli di variabilità genetica osservati. La selezione naturale viene rimessa in gioco nel momento in cui i modelli osservati non si adattino a quelli attesi o a quelli teorici.
1.4. Il flusso genico
Il flusso genico si realizza in proporzione alle possibilità di incontro e quindi di scambio genico tra individui o gruppi di individui di una determinata specie. Le capacità di movimento caratteristiche dei vari stadi della vita di un organismo, gameti, embrioni, larve ed adulti, condizionano uno dei principali fattori microevolutivi che agiscono sulla struttura genetica di una specie, definendo il grado di isolamento genetico tra le popolazioni che la compongono, favorendo o sfavorendo il differenziamento tra le stesse. L’isolamento geografico ed il flusso genico tra
popolazioni determinano il potenziale per la differenziazione genetica e, in ultima analisi, per la speciazione, dovuta all’isolamento riproduttivo. La differenziazione genetica tra popolazioni può avvenire ad opera della selezione naturale, per caratteristiche ecologiche locali, oppure per gli effetti stocastici della deriva genetica. In quest’ottica, l’analisi della struttura genetica delle specie e del flusso genico tra le popolazioni è un gradino essenziale per la comprensione degli effetti della selezione e della deriva genetica nel produrre i modelli geografici di differenziazione genetica.
Le stime di flusso genico possono essere ottenute tramite metodi diretti o indiretti (Slatkin, 1985): i primi sono rappresentati essenzialmente dai metodi di marcatura e ricattura, che possono essere usati solo per misurare gli spostamenti degli stadi adulti e fornire stime dirette sul numero di migranti tra varie località. Questi metodi presentano, comunque, alcune controindicazioni in quanto potrebbero non individuare migrazioni occasionali a lungo raggio, oppure perché non consentono di stabilire la fitness dei migranti, cioè se ai fenomeni migratori seguano poi fenomeni riproduttivi. I metodi diretti inoltre non possono essere utilizzati per la maggior parte delle specie per motivi di ordine pratico. Le misure indirette, basate sull’uso di alcuni parametri relativi alla struttura genetica delle popolazioni, permettono di indagare i livelli storici di flusso genico che hanno originato la variabilità genetica osservata.
1.5. La filogeografia
A differenza degli studi filogenetici classici, che hanno alla base fenomeni macroevolutivi, come ad esempio la speciazione, la genetica di popolazioni e la filogeografia si occupano di processi inter-individuali e di popolazione che sono in funzione di meccanismi microevolutivi (ad es. flusso genico, mutazione, selezione naturale), che determinano la distribuzione spazio-temporale della diversità genetica di una specie. In particolare, la filogeografia è una disciplina che riguarda i principi ed i processi che governano la distribuzione geografica delle linee genealogiche, specialmente quelle all’interno delle specie, oppure quelle tra specie molto affini (Avise, 2000). La filogeografia tratta le componenti storiche e filogenetiche della distribuzione spaziale delle linee genealogiche. In altri termini, il tempo e lo spazio sono considerati in maniera unitaria ed in questo spazio ideale vengono riportate le
linee genealogiche oggetto di studio (Avise, 2000). 1.6. Cenni sull’analisi dei dati filogeografici
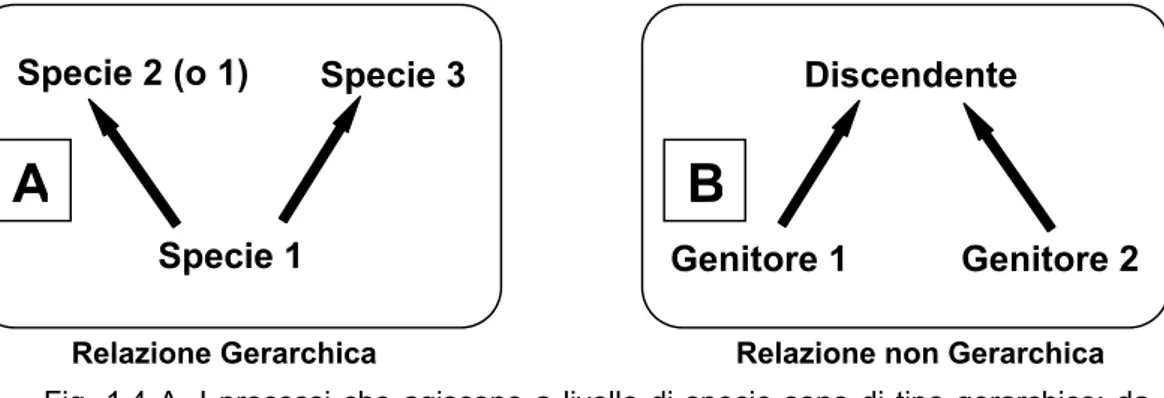
L’esplosione delle tecniche di biologia molecolare avvenuta negli scorsi decenni ha portato all’accumulo di una considerevole quantità di informazioni genetiche sulle popolazioni naturali. Allo stesso tempo, i progressi teorici sulla genetica di popolazioni, e specialmente la teoria della coalescenza, hanno permesso lo sviluppo di validi strumenti per l’analisi di dati a livello intraspecifico. Sono stati sviluppati diversi metodi di tipo filogenetico per analizzare al meglio le caratteristiche uniche dei dati intraspecifici. I rapporti evolutivi che si stabiliscono ai livelli superiori ed inferiori fino a quello di specie differiscono per natura: la relazione tra due geni campionati in individui appartenenti a specie differenti sono di tipo gerarchico (filogenesi sensu stricto), perché sono il prodotto dell’isolamento riproduttivo e della separazione tra popolazioni su lunga scala temporale; durante i primi stadi del processo di speciazione la mutazione e la deriva genetica portano alla fissazione di alleli differenti e infine a pool genici non sovrapponibili. Per contro, le relazioni esistenti tra geni campionati in individui della stessa specie (tokogenesi) non sono di tipo gerarchico, perchè sono il risultato di fenomeni di riproduzione sessuale, di eventi di mutazione relativamente recenti e di ricombinazione (Fig. 1.4).
Specie 1
Specie 2 (o 1) Specie 3 Discendente
Genitore 1 Genitore 2
Relazione Gerarchica Relazione non Gerarchica
A
B
Fig. 1.4 A. I processi che agiscono a livello di specie sono di tipo gerarchico: da una specie ancestrale originano due specie discendenti. B. I processi che agiscono all’interno di una specie sono di tipo non gerarchico: due individui contribuiscono con i loro geni alla formazione di un nuovo individuo. Modificato da Posada & Crandall, 2001.
I metodi tradizionalmente utilizzati a livello sopraspecifico come la maximum likelihood, la maximum parsimony e la minimum evolution, non tengono conto del fatto che a livello di popolazione vengono violati alcuni dei loro assunti di base. Questo si concretizza spesso in uno scarso potere di risoluzione ed un’inadeguata rappresentazione delle relazioni genealogiche a livello intraspecifico. Inoltre la minore divergenza genetica riscontrabile tra individui della stessa specie implica una scarsità di quegli aspetti caratterizzanti utilizzati nelle analisi filogenetiche, diminuendone il potere statistico (Posada & Crandall, 2001).
Nelle popolazioni naturali la maggior parte degli aplotipi esistenti sono presenti in molte copie identiche, originatesi per replicazione del DNA mitocondriale (mtDNA) e successiva eredità materna. Quando una di queste copie subisce una mutazione, originando un nuovo aplotipo, è estremamente improbabile che le altre copie mutino a loro volta, determinando la scomparsa dell’aplotipo ancestrale; quindi ci si aspetta che l’aplotipo ancestrale sia ancora rappresentato nella popolazione, e possa essere campionato insieme agli aplotipi che ne derivano. I tradizionali metodi filogenetici sono basati su alberi che si dividono in maniera dicotomica, ma la persistenza dell’aplotipo ancestrale contemporaneamente alla presenza di molti aplotipi da esso derivanti produce ramificazioni multiple non rappresentabili come semplici biforcazioni. Detto ciò, si può prevedere che più antico sarà un aplotipo, maggiore sarà il numero di aplotipi derivati ad esso connessi. I processi evolutivi che intervengono a livello di popolazione, come la ricombinazione tra geni, l’ibridazione di differenti linee genealogiche e l’omoplasia, generano relazioni di tipo reticolato che non sono ammesse dai metodi tradizionali e possono essere male interpretati (Posada & Crandall, 2001).
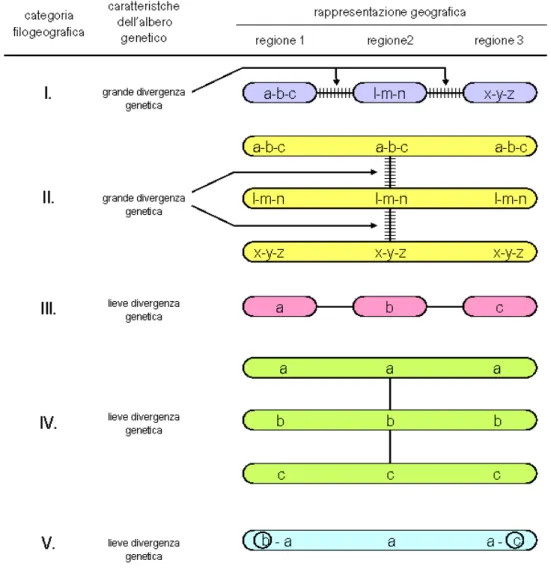
Avise (2000) propone una classificazione in cinque categorie principali delle disposizioni possibili e più tipiche riscontrabili nei network e negli alberi ottenibili con l’analisi filogeografica del mtDNA (Fig. 1.5). Le categorie descritte non sono necessariamente da ritenersi come reciprocamente esclusive; inoltre non rappresentano che una piccola frazione delle numerose possibilità ottenibili con dati reali:
Fig. 1.5. Categorie filogeografiche proposte da Avise et al. (2000). Le forme comprendono particolari aplotipi mitocondriali (contraddistinti da lettere) o gruppi di aplotipi affini le cui distribuzioni geografiche sono evidenziate. Gli aplotipi sono connessi in network filogenetici connessi da linee che rappresentano una mutazione ciascuna a meno che non siano presenti barrette trasversali che indicano ciascuna un’ulteriore mutazione.
I. Alberi profondi, linee genealogiche principali in allopatria. In questo caso si riscontrano aplogruppi circoscritti, separati da un numero di mutazioni relativamente grandi. In altre parole, differenze genetiche evidenti distinguono profonde divisioni allopatriche nelle varie linee genealogiche. Una struttura più fine può essere presente a scala minore, all’interno delle regioni. Questa tipica distribuzione può essere il frutto di barriere al flusso genico: col tempo popolazioni in allopatria finiscono per occupare rami ben distinti e separati di un albero filogenetico intraspecifico. Le differenze genetiche possono riflettere
l’accumularsi di nuove mutazioni, successive alla separazione delle popolazioni e/o l’assortimento casuale a partire da un pool genico iniziale molto polimorfico. Soprattutto in specie ad ampia diffusione e bassa dispersione e flusso genico, l’estinzione di aplotipi intermedi può contribuire in maniera consistente all’immagine finale di profonda separazione. Dati di questo tipo necessitano comunque di conferme provenienti da altri marcatori (es. da altri geni), oppure da altre fonti di informazioni biogeografiche come la paleontologia o l’anatomia comparata, la morfometria ecc. Diversamente non è possibile stabilire se il pattern non sia il frutto di caratteristiche intrinseche al marcatore scelto.
II. Alberi profondi, linee genealogiche principali in simpatria. In questo caso si presentano separazioni evidenti tra i cladi di un albero, con le principali linee genealogiche distribuite indistintamente su una vasta area. In teoria simili organizzazioni possono presentarsi in specie con grande dimensione efficace (Ne) ed elevato flusso genico in tempi remoti su scala evolutiva. In seguito, alcune linee genealogiche anche molto distanti filogeneticamente possono accidentalmente conservarsi, mentre molte altre, intermedie alle prime, possono perdersi nel tempo per una graduale erosione. Una selezione stabilizzante può facilitare l’insorgere di simili situazioni, favorendo la lunga sopravvivenza di alcune linee filetiche, per quanto poco rappresentate. Comunque, la maggior parte delle specie che presentano una simile disposizione, hanno probabilmente subito un rimescolamento secondario tra popolazioni che si sono evolute in allopatria e che sono quindi soggette a diverse condizioni ambientali, selezione, deriva genetica ecc. (Avise et al., 1984; Kim et al., 1998); spesso, a conferma dell’ipotesi dell’origine allopatrica, esistono riscontri indipendenti provenienti da altre discipline come la paleontologia. Tipica di questa disposizione degli aplotipi è anche una distribuzione mismatch bimodale (vedi paragraf1 3.3 e 4.3).
III. Alberi poco separati, linee genealogiche principali in allopatria. In questo caso la maggior parte o tutti gli aplotipi sono molto affini, ma comunque hanno una precisa distribuzione geografica. Questo implica che il flusso genico sia stato sufficientemente lento, in relazione alle dimensioni della popolazione, da consentire il prevalere di alcune linee genealogiche, oppure che si sia verificata deriva genetica (o selezione diversificante), tale da produrre divergenza
genetica tra popolazioni che erano comunque in contatto in tempi storicamente recenti. La categoria III può degradare nella IV e nella V, a seconda dei livelli storici di flusso genico tra le popolazioni, che comunque non risulterebbe mai ostacolato da barriere biogeografiche persistenti. Specie che presentano una disposizione di tipo III su scala locale possono presentare ad una più ampia scala una disposizione del tipo I.
IV. Alberi poco separati, linee genealogiche principali in simpatria. Questo pattern è atteso nel caso di specie con un elevato flusso genico, ma un modesto o basso Ne, le cui popolazioni non abbiano subito l’effetto a lungo termine di barriere biogeografiche. In molti casi queste specie hanno subito fenomeni di espansione demografica, in seguito ad espansione del loro areale. Il risultato è un albero con rami corti, molti dei quali contengono individui indiscriminatamente diffusi su tutto l’areale della specie. Per specie di recente espansione o con un basso numero di fondatori, un altro risultato tipicamente atteso è quello della “star phylogeny”, cioè un pattern ottenibile con i network con un aplotipo ancestrale (o affine all’ancestrale) molto diffuso che si trova al centro della rappresentazione e gli aplotipi derivati più recentemente che sono connessi in maniera indipendente e con rami corti. Tipica è anche la disposizione a “dumbbell”, con più esplosioni a stella connesse da rami molto lunghi. A seconda dei pattern di dispersione correlati all’espansione della popolazione, queste specie possono mostrare in generale una disposizione di tipo I o di tipo II.
V. Alberi poco separati, varia distribuzione delle linee genealogiche. È un pattern intermedio tra il III e il IV e prevede linee genealogiche ad ampia diffusione più altre linee, affini alle prime, con diffusione locale. Questo tipo di risultato è il frutto di scarso flusso genico tra popolazioni comunque strettamente legate dal punto di vista storico. Gli aplotipi più comuni sono di solito ancestrali per la specie, mentre gli aplotipi più rari sono da considerarsi come derivati. Quindi, gli aplotipi rari condivisi da gruppi di individui o di popolazioni rappresentano potenziali marcatori per cladi di recente evoluzione.
1.7. Il DNA mitocondriale (mtDNA)
Come è noto, nelle cellule il DNA ha localizzazioni differenti. Il DNA è presente nel nucleo (nDNA), nei mitocondri (mtDNA) e, nel caso delle piante, nei cloroplasti (cpDNA) e nei plastidi (pdDNA). Sia negli animali che nelle piante il DNA organellare è generalmente ereditato per via materna. Il DNA mitocondriale, in particolare, viene considerato come non ricombinante, anche se questa caratteristica è stata messa in dubbio da osservazioni di processi ricombinativi a carico del mtDNA; inoltre è stata individuata una ricombinasi mitocondriale il cui ruolo non è ancora del tutto chiaro (Lightowlers et al., 1997).
Il mtDNA è rappresentato da una singola molecola circolare costituita da circa 16000 paia di basi (bp), è un genoma estremamente compatto, ed è costituito principalmente da regioni codificanti prive di introni e non separate tra loro da sequenze interposte. In ogni cellula sono contenute numerose copie di mtDNA e generalmente queste copie sono identiche (omoplasmiche). A volte però ci possono essere due o tre genomi mitocondriali diversi (eteroplasmici) in uno stesso individuo; l’eteroplasmia può derivare da nuove mutazioni insorte a livello del mtDNA, dal “pool” di mitocondri presenti nella cellula uovo da cui deriva l’individuo (Moritz, 1987), in alcune specie può essere, invece, la conseguenza di una trasmissione biparentale del mtDNA (Zouros, 1992).
Molti studi dimostrano che il mtDNA è più soggetto a mutazione di quello nucleare anche perché più esposto ad agenti mutageni: il mitocondrio è la sede della fosforilazione ossidativa ed è quindi a contatto con i radicali liberi dell’ossigeno prodotti durante alcune reazioni (Avise, 2000). Inoltre, gli enzimi atti al metabolismo dei contaminanti, dei farmaci, e delle sostanze tossiche in genere, sono situati sulla membrana mitocondriale, perciò queste sostanze si concentrano a livello dei mitocondri, così come i prodotti del loro metabolismo, talvolta più pericolosi delle sostanze di origine. Tutte le sostanze tossiche e mutagene vengono facilmente in contatto con il mtDNA. Da sottolineare è anche il fatto che il mtDNA non è circondato da particolari membrane, ma si trova libero all’interno del mitocondrio, strettamente associato alla membrana mitocondriale interna; non è, inoltre, organizzato in nucleosomi e non è associato a proteine istoniche (Wallace, 1994).
Nonostante l’esposizione diretta ai contaminanti, il mtDNA possiede limitate capacità di riparazione rispetto al nDNA. Non è in grado per esempio di riparare i dimeri di pirimidina; inoltre va incontro a molti più cicli di replicazione, ed ha quindi una maggiore probabilità di accumulare errori dovuti all’inserzione di basi scorrette durante i processi duplicativi, visto anche che la DNA-polimerasi mitocondriale non possiede l’attività esonucleasica 3’-5’. La frequenza di errore della DNA-polimerasi mitocondriale è di circa 10-6 per nucleotide incorporato, ovvero da tre a cinque ordini di grandezza superiore rispetto agli errori commessi dalla DNA-polimerasi nucleare, che è provvista della subunità che svolge la funzione di correzione di bozze e che presenta una frequenza di errore di 10-9-10-11 per nucleotide incorporato (Cooper et al., 1995).
Rispetto al genoma nucleare, quello mitocondriale è molto meno esteso, però è presente in molte più copie per cellula, tanto che il numero di copie di mtDNA presente in un’unica cellula può essere dell’ordine delle migliaia. Dato questo enorme numero di geni mitocondriali presenti in ogni cellula, ci si potrebbe aspettare che una mutazione che insorga in una singola molecola di mtDNA abbia scarsa probabilità di essere fissata e di conseguenza il tasso di mutazione sia molto basso; in realtà il genoma mitocondriale possiede un elevato indice di fissazione, tanto che può essere considerato un “hot-spot” per le mutazioni. Una possibile spiegazione per l’elevato tasso di fissazione è che si verifichi una sorta di collo di bottiglia nelle popolazioni di mtDNA, mediante il quale viene selezionata solo una parte delle molecole di mtDNA destinate ad essere successivamente duplicate (Lightowlers et al., 1997). La combinazione tra instabilità del mtDNA e un elevato indice di fissazione fa sì che il tasso di mutazione del mtDNA sia molto elevato: circa dieci volte maggiore di quello riscontrabile in sequenze equivalenti del genoma nucleare (Brown et al., 1979). Quindi, il mtDNA evolve molto in fretta e presenta un tasso di divergenza tra sequenze che va dal 2 al 4% per milione di anni (Cann et al., 1987).
Di norma le regioni codificanti hanno tassi di mutazione più bassi, mentre le regioni non codificanti presentano tassi di mutazione più alti. Il differente tasso di mutazione delle varie regioni permette di usare il mtDNA per l’analisi della diversità genetica sia a livello intra- che inter-specifico (Procaccini & Maltagliati, 2003). Il mtDNA è comunemente usato come marcatore molecolare per la sua versatilità ed
affidabilità sia nei vertebrati che negli invertebrati. Non bisogna, comunque, dimenticare alcuni inconvenienti che i marcatori mitocondriali possono presentare, quali ad esempio, l’eteroplasmia, o la presenza di pseudogeni, cioè inserzioni di geni mitocondriali all’interno del genoma nucleare (Zhang & Hewitt, 1996). La presenza di geni mitocondriali nel genoma nucleare e la loro successiva amplificazione nelle analisi di studi filogeografici e di genetica di popolazioni possono alterare sostanzialmente i risultati e condurre a conclusioni errate. Un metodo per verificare la presenza di pseudogeni è quello di separare il genoma mitocondriale da quello nucleare, per esempio attraverso una centrifugazione che permetta la separazione dei mitocondri dal nucleo. Successivamente, si possono amplificare con gli stessi primer i due genomi per vedere, in primo luogo se il frammento mitocondriale viene amplificato anche nel DNA nucleare e, in caso affermativo, se esistono differenze tra le sequenze ottenute dai mitocondri e quelle nucleari, in modo da poter riconoscere ed escludere queste ultime quando si presentino specificamente come contaminanti tra i dati mitocondriali.
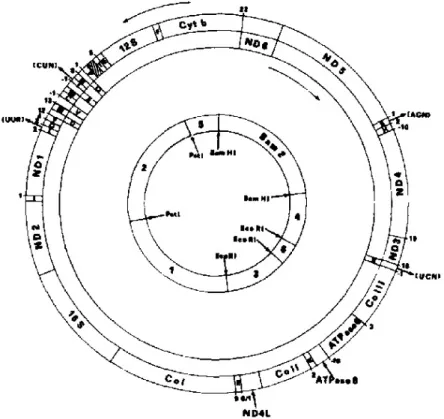
In Paracentrotus lividus, il genoma mitocondriale misura 15697 bp, codifica per due RNA ribosomali (rRNA), ventidue RNA di trasferimento (tRNA) e dodici RNA messaggeri (mRNA) che codificano per tredici subunità dei complessi respiratori della membrana interna del mitocondrio (Cantatore et al., 1989) (Fig. 1.6). L’arrangiamento dei geni differisce da quello riportato per altri animali: i geni per gli rRNA 16S e 12S, sono separati da circa 3.300 basi contenenti i geni ND1 e ND2 ed un insieme di 15 tRNA. La sequenza che codifica per l’ND4L (NADH deidrogenasi, subunità 4L) non è contenuta nell’mRNA dell’ND4 ma possiede un suo specifico mRNA localizzato tra il tRNA per l’arginina ed il gene COII. La regione non codificante di maggiore entità, situata nell’insieme dei geni per i tRNA, è lunga solo 132 pb, ma contiene sequenze omologhe a quelle della regione di controllo (D-Loop) dei vertebrati. Altre regioni non codificanti, tutte molto corte, sono inframezzate al genoma e contengono sequenze consenso ricche di A e T molto conservate, che probabilmente hanno un ruolo nella trascrizione o nella sintesi degli RNA (Fig. 1.6).
Su base puramente teorica, è previsto l’uso delle regioni codificanti, caratterizzate da tasso di mutazione più basso, per l’analisi della diversità genetica a livello interspecifico, riserbando le regioni non codificanti, come l’origine di
replicazione, per indagini intraspecifiche. Tuttavia esistono molte eccezioni a questa regola: per esempio Grant e Bowen (1998) hanno usato il gene Cyt b nelle sardine dei generi Sardina e Sardinopsis; Cassens et al. (2003) hanno indagato la genetica di popolazioni di Lagenorhyncus obscurus tramite lo studio delle sequenze del gene Cyt b; la sequenza del gene COI è stata invece usata per uno studio filogeografico sugli echinoidi del genere Tripneustes (Lessios et al., 2003).
La sequenza della regione di controllo in P. lividus ammonta a poche decine di basi ed è quindi inutilizzabile per i nostri fini; inoltre in un lavoro di recente pubblicazione, Duran et al. (2004) hanno utilizzato con successo la sequenza del gene per la COI (di 644 bp) per indagare la struttura genetica di P. lividus in Atlantico e lungo le coste spagnole mediterranee. Nel presente studio è stato preso in considerazione il gene che codifica per citocromo b (Cyt b), un marcatore ampiamente impiegato in studi filogeografici su larga scala (Avise, 2000).
Fig. 1.6. Organizzazione generale del genoma mitocondriale di P. lividus. Il cerchio più interno mostra i siti di restrizione per BamHI, EcoRI, e PstI. I due cerchi più esterni mostrano l’ordine dei geni. Le frecce orientate poste all’interno e all’esterno dei cerchi indicano la direzione della trascrizione dei rispetivi geni.
1.8. Caratteristiche genetiche di Paracentrotus lividus
È nota per P. lividus una certa variabilità fenotipica: nello specifico sono riportate in bibliografia “popolazioni” di individui di taglia significativamente inferiore in relazione alla presenza di stress ambientale (Pancucci et al. 1993). Arculeo et al. (1998) hanno studiato la variabilità genetica, utilizzando l’elettroforesi degli alloenzimi, in campioni distinguibili dal punto di vista morfologico provenienti da due località del Mediterraneo: alcuni animali erano adulti di taglia piccola del Golfo di Amvrakikos (Grecia), altri, di dimensioni usuali, provenivano dal Golfo di Palermo. L’elevata stima di flusso genico e il basso valore di distanza genetica rilevati tra questi campioni hanno portato gli Autori a respingere l’ipotesi di due popolazioni separate.
La scarsa variabilità genetica, come osservato in altri invertebrati, può essere legata al grado di imprevedibilità ambientale; d’altro canto un alto grado di variabilità genetica è invece proprio degli echinodermi tipici degli ambienti profondi, tipicamente più stabili ed uniformi (Murphy et al., 1976; Matsuoka et al., 1993). La relazione tra caratteri morfologici e stress ambientale, che caratterizza P. lividus del Golfo di Amvrakikos, non ha una base genetica (Arculeo et al., 1998). Differenze fenotipiche possono non riflettere differenze genetiche, ma rappresentare plasticità che riflette un adattamento a condizioni trofiche locali. Questi risultati sono in accordo con quelli riscontrabili per popolazioni di altri invertebrati marini con larva planctonica (Hunt & Ayre 1989; Solé-Cava et al. 1994).
Recentemente Duran et al. (2004) hanno utilizzato la sequenza del gene mitocondriale codificante per la subunità I della citocromo ossidasi (COI) in 127 individui di P. lividus campionati in sei località atlantiche e sei mediterranee, per studiare la struttura genetica della specie. I risultati di questo lavoro hanno evidenziato un alto livello di diversità genetica all’interno dei campioni, ma una scarsa strutturazione della specie, dati che suggeriscono un notevole flusso genico tra le varie località e scarsa divergenza genetica tra individui del bacino Atlantico e quelli del Mediterraneo. È comunque risultata significativa la differenziazione genetica riscontrata tra i due bacini.
Napoli, al fine di indagare la strutturazione della specie ad una scala spaziale relativamente piccola. Questi Autori, come era da attendersi, non hanno rilevato alcuna strutturazione genetica alla scala considerata.
1.9. Cenni sulla pesca di Paracentrotus lividus
Molte specie di echinoidi sono pescate attivamente in varie regioni del mondo per la delicatezza del sapore delle loro gonadi. Questo tipo di pesca è stata praticata fino a pochi decenni fa da un ristretto numero di persone con metodi artigianali ed il prodotto della pesca era normalmente venduto localmente (Boudouresque, 1987). In tempi più recenti, principalmente per l’aumento di domanda di mercato, la pesca degli echinoidi ha subito un notevole incremento, determinando fenomeni di “overfishing” in diverse specie di interesse commerciale (FAO, 1996) e inducendo l’introduzione di misure restrittive e di controllo come quote massime di cattura giornaliera, taglia minima di commercializzazione, rotazione dei siti di prelievo ed istituzione di centri per l’allevamento.
La pesca di Paracentrotus lividus è regolamentata dal Decreto Ministeriale del 12 gennaio 1995. Questa normativa si compone di cinque articoli e prevede una serie di limiti qui riassunti: l’articolo 1 stabilisce che la pesca di P. lividus è consentita ai pescatori subacquei professionisti ed ai pescatori sportivi per mezzo della raccolta manuale in immersione o grazie all’uso di specchio, asta e rastrello. L’articolo 2 fissa per i pescatori professionisti un limite di 1000 esemplari al giorno ed un limite di 50 esemplari al giorno per i pescatori sportivi. L’articolo 3 fissa la taglia minima degli esemplari catturabili a 7 cm comprese le spine. L’articolo 4 stabilisce il divieto alla pesca di P. lividus nei mesi di maggio e giugno. L’articolo 5 stabilisce le sanzioni che scaturiscono dall’inosservanza dei precedenti articoli, ai sensi degli articoli 15-a e 26 della legge n° 963 del 14 luglio 1965. In Sardegna, nell’area marina protetta Penisola del Sinis – Isola di Mal di Ventre (Oristano), è stata approvata l’ordinanza n° 94 del 12 novembre 2003, che prevede norme leggermente differenti, tra cui il rilascio del “tesserino di prelievo del riccio”, sul quale annotare quotidianamente dati relativi alle catture, il divieto di utilizzare autorespiratori per la raccolta, che quindi può avvenire solo da natante o in apnea, e una taglia minima di raccolta diversa: 50 mm senza
spine. Queste misure, moderatamente più restrittive, preservano gli individui al di sotto dell’isobata dei 5 m, e potrebbero garantire il ripopolamento delle aree sfruttate (Baroli et al., 2006). È da rimarcare il fatto che per questo tipo di pesca, come per molti altri, le leggi siano scarsamente applicate (Guidetti et al., 2004) e manchi un controllo diretto ed efficace sul territorio, soprattutto nei confronti dei pescatori sportivi.
La complessità delle relazioni ecologiche che connettono gli echinoidi con il resto dell’ecosistema bentonico rendono difficile l’interpretazione degli effetti della pesca di questi animali sulle comunità. Guidetti et al. (2004) hanno confrontato vari parametri, tra cui la densità di individui per unità di superficie, il diametro medio degli individui, la biomassa totale, relativi ai popolamenti di P. lividus e Arbacia lixula, di cui solo il primo oggetto di pesca, lungo la costa della Puglia. Lo studio è stato svolto prendendo in esame aree notoriamente sfruttate, perché raggiungibili da terra o perché prossime a piccoli porti, ed aree difficilmente accessibili, e quindi utilizzabili come controllo. Dai risultati emerge che il principale effetto della pesca su P. lividus è una diminuzione della biomassa totale (da tre a sette volte) dovuta alla sottrazione dalle aree impattate di tutti gli animali di dimensioni superiori alla taglia minima di commercializzazione. Per A. lixula è stato rilevato che le dimensioni medie degli individui rimangono costanti al variare dei siti, mentre la densità aumenta laddove P. lividus è soggetto a pesca. Ciò suggerisce l’esistenza di una sorta di vantaggio nella competizione per le risorse dato dalla minore biomassa di P. lividus e di una possibile compensazione ecologica da parte di A. lixula non riscontrata in lavori precedenti (Benedetti Cecchi et al., 1998; Bulleri et al., 1999).