Sezione 3 – Capitolo II
Caratterizzazione morfologica e molecolare di
Frontonia salmastra Dragesco & Dragesco-Kernéis, 1986
e dei simbionti ad essa associati
Parte Prima
Introduzione
II.1.1: Caratteristiche generali del gen. Frontonia
Il genere Frontonia EHRENBERG 1833 (cl. Oligohymenophorea DE PUYTORAC ET AL. 1974, ord. Peniculida FAURÉ-FRÉMIET IN CORLISS 1956) comprende numerose specie di protozoi ciliati, caratterizzati da una ciliatura somatica abbondante, uniformemente distribuita, e da una tipica sutura post-orale che si estende lungo la faccia ventrale posteriormente al citostoma. La ciliatura orale, nettamente distinta da quella somatica, consiste in una membrana paraorale sul lato destro e organelli composti, quali membranelle adorali e tre peniculi – donde il nome dell’ordine – sul lato sinistro, provvisti di quattro o cinque cinezie i cui cinetosomi appaiono coalescere. Le cinezie vestibulari, presenti in numero variabile da specie a specie, sono composte di trichinetidi.
Il genere risulta piuttosto omogeneo, ed in certi casi la diagnosi specifica è di difficile precisazione: in certi casi, i soli criteri morfologici sono apparsi insufficienti per definire le specie con sicurezza (Dragesco and Dragesco-Kernéis, 1986). Recentemente, l’applicazione di metodologie molecolari per l’analisi filogenetica di tale genere ne ha rivelato la probabile parafilia (Andreoli et al., 2006).
- Batteri simbionti in Frontonia
I dati disponibili in letteratura riguardo ad eventuali simbionti procariotici associati a
Frontonia spp. sono scarsi (Fokin and Schweikert, 2003). Tali simbiosi potrebbero in effetti
essere particolarmente rare: ad esempio, il diverso regime alimentare di Frontonia spp., basato prevalentemente su alghe unicellulari, potrebbe sfavorire l’assunzione di batteri infettanti durante la fagocitosi (Fokin and Schweikert, 2003): questa, infatti, sembra essere la via abitualmente seguita dai batteri infettanti per raggiungere la propria sede nell’ospite
(Görtz, 2003)1. È tuttavia possibile che scarsità di simbiosi segnalate possa dipendere anche
dal basso numero di studi sinora compiuti, a sua volta causato dalla difficile coltivabilità di questi peniculidi in condizioni di laboratorio (vd. oltre), e dalla conseguente difficoltà di caratterizzare tanto le specie in esame quanto gli eventuali batteri simbionti. D’altro canto, le specie di ciliati facilmente coltivabili del gen. Paramecium, appartenente al medesimo ordine di Frontonia e filogeneticamente affine a quest’ultimo, sono invece molto studiate, e numerose associazioni simbiotiche vi sono state descritte (es. Görtz, 2003). È pertanto possibile che indagini ulteriori su Frontonia spp. possano rivelarsi interessanti nello studio dei rapporti simbiotici fra ciliati e procarioti.
Sono stati ad oggi descritti due simbionti, rispettivamente di Frontonia sp., specie marina non identificata, e di F. leucas, dulciacquicola (Fokin, 2004).
La prima associazione simbiotica fra batteri e ciliati del gen. Frontonia è stata descritta da Rosati et al. nel 2001. La specie di appartenenza dell’ospite non è stata determinata: le caratteristiche morfologiche riportate (Rosati et al., 2001) sono riconducibili a quelle descritte per F. leucas, che tuttavia è specie prettamente dulciacquicola (es. Fokin, 2004). L’organismo simbionte era presente, all’interno di vacuoli citoplasmatici, in tutti gli individui della popolazione analizzata: si trattava di un batterio flagellato, affusolato (in certi casi sono stati segnalati esemplari incurvati), dalla lunghezza di 5-6 μm, dotato di doppia membrana; i flagelli consentivano a questo organismo una certa motilità se portato al di fuori della cellula ospite (Rosati et al., 2001). La simbiosi si è mantenuta stabile per almeno un mese: per questo ed altri motivi (es. la presenza di movimenti di metabolici, quali vescicole e granuli di glicogeno, fra il citoplasma dell’ospite ed i batteriosomi) gli autori hanno concluso che tale rapporto “non è relazione occasionale [bensì] un’associazione stabile, molto probabilmente vantaggiosa per entrambi i partner” (Rosati et al., 2001). La reale natura di questa simbiosi non è comunque stata determinata (Rosati et al., 2001).
Nel 2003 è stato descritto un secondo simbionte di Frontonia, presente nella specie dulciacquicola F. leucas (Fokin and Schweikert, 2003). I batteri risultavano collocati all’interno del nucleoplasma macronucleare di una certa percentuale (variabile stagionalmente, mai nulla) di individui della popolazione oggetto di studio: si presentavano di forma allungata, dalla lunghezza di 5-16 μm (probabilmente variabile a seconda della fase del ciclo vitale), strutturalmente G-; capsidi virali risultavano presenti al loro interno (Fokin and Schweikert, 2003). Non sembrava possedere killer-effect, né era in grado di infettare, in condizioni di laboratorio, cellule di F. leucas prive di simbionte (Fokin and Schweikert, 2003). Esperimenti di ibridazione in situ hanno determinato la sua appartenenza alla cl.
1 La presenza di numerose associazioni simbiotiche, mutualistiche o dispensabili, descritte in ciliati
prettamente algivori quali ad es. Euplotes spp. (es. Görtz, 2003 ; Vannini et al., 2004) porta a ritenere che tali simbiosi possano istaurarsi indipendentemente dal regime alimentare dell’ospite.
Alphaproteobacteria; non è stata effettuata la caratterizzazione molecolare (Fokin and
Schweikert, 2003).
Il presente studio, durante il quale sono stati individuati due simbionti presenti in F.
salmastra, uno dei quali è stato caratterizzato morfologicamente e molecolarmente,
contribuisce ad aumentare il numero di dati disponibili sui rapporti simbiotici esistenti in tale genere, fornendo inoltre alcuni strumenti molecolari utilizzabili per indagini future.
Sezione 3 – Capitolo II
Parte Seconda
Risultati dell’analisi
II.2.1: Isolamento e messa in coltura di F. salmastra
Esemplari di F. salmastra sono stati individuati in tutti i campionamenti effettuati nello Stagno 1. Benché soggetta a fluttuazioni numeriche, la specie appare sempre presente in tale ambiente. Tuttavia, all’interno dei campioni si estingue dopo breve tempo dalla raccolta. Le cellule destinate ad effettuare tentativi di coltivazione sono state prelevate sia da sedimento campionato in data 02-10-2005 (Salinità 10‰) che da campioni raccolti in data 16-10-2005 (Salinità 4‰). Le cellule sono state isolate in acqua di mare sintetica a salinità corrispondente a quella del campione.
Sono state eseguite prove di alimentazione al fine di individuare il nutrimento ottimale per tali organismi. Sono stati testati come fonte di cibo l’alga verde Dunaliella salina, batteri amilotrofi (incentivandone la crescita con riso bollito), l’alga bruna Pheodactylum
tricornutum (tre repliche per ciascun trattamento; cellule provenienti da campioni del
02-10-2005: la salinità delle colture algali somministrate – 33‰ – è stata corretta tramite diluizione). Risultati positivi sono stati ottenuti esclusivamente con l’alga bruna.
Sono stati effettuati 24 tentativi di stabilire colture monoclonali di tali organismi. Le cellule isolate per l’effettuazione di tali tentativi provenivano dal campione raccolto in data 16-10-2005. A ciascun organismo è stata somministrata come alimento l’alga bruna, allevata in acqua di mare sintetica a salinità 5‰ in modo da evitare il ricorso alla diluizione.
L’allevamento in condizioni di laboratorio di questa specie si è presentato difficoltoso. Durante le fasi iniziali, le colture cellulari si accrescono a ritmi elevati, per poi stabilizzarsi; repentine estinzioni possono però causare la morte dell’intera popolazione in circa due-tre giorni. Tali estinzioni avvengono senza preavviso, e non è stato possibile determinarne la causa scatenante. I sintomi iniziali sono la riduzione della motilità cellulare, finché tutti gli organismi precipitano, pressoché immobili, sul fondo del recipiente di coltura. Dopo circa 24h le cellule presentano vacuoli contrattili anormalmente rigonfi, ed assenza totale di
movimento. La lisi cellulare sopraggiunge dopo circa 48h dalla cessazione del movimento e
dopo alcune ore dalla comparsa del rigonfiamento dei vacuoli. È stato ipotizzato che la ragione di tali sintomi possa essere una disfunzione a carico della membrana plasmatica, con
conseguente cessazione del movimento ciliare e problemi di osmoregolazione. Non siamo tuttavia stati in grado di capirne le cause. È stato osservato che alcuni esemplari, trasferiti in mezzo sterile, isosmotico col mezzo di coltura, durante le fasi iniziali della sindrome, riacquistavano motilità e potevano essere recuperati; tale trattamento non sortiva però alcun effetto nella maggior parte dei casi, così come se attuato durante le fasi finali di tali crisi (cellule ferme, vacuoli rigonfi). Al contempo, è stato possibile appurare che i fenomeni di estinzione insorgevano quasi sempre dopo aver sottoposto le cellule ad un cambio repentino del contenitore e/o del mezzo di coltura (es. causato da trasferimento della coltura in recipienti più capienti contenenti o meno nuovo mezzo). È stato quindi deciso di continuare il trasferimento delle colture di F. salmastra sino al raggiungimento di un volume compreso fra 20 e 50 ml, raggiunto il quale il recipiente non sarebbe più stato cambiato. È stato evitato l’accumulo di volume in eccesso, derivante dall’aggiunta delle soluzioni di alghe, rimovendo periodicamente il mezzo tramite pipetta Pasteur o per rovesciamento degli strati superficiali della coltura stessa (di norma contenenti poche cellule). In tal modo la frequenza degli eventi di estinzione si è abbassata sensibilmente (dall’83,4% sui 24 ceppi iniziali al 25% sugli 8 ceppi sopravvissuti sino all’introduzione del trattamento).
È stato possibile ottenere tre colture monoclonali (FS 81/1, FS 82/2, FS 83/2) stabili nel tempo per un periodo di otto-nove mesi. Tali colture sono state mantenute in cella termostatata a 19±1°C, con alternanza luce-buio di 12 ore, e cibate con Pheodactylum
tricornutum a salinità 5‰ con cadenza settimanale. Date le precarie condizioni di
allevamento, non è stato tuttavia possibile ottenere grandi quantità di cellule, cosicché gli esperimenti sono stati condotti in gran parte su pochi esemplari per ciascun ceppo.
Il metodo di allevamento individuato consente di ottenere colture monoclonali stabili per periodi di tempo medio-lunghi, ma non scongiura il rischio di estinzione improvvisa del ceppo. Sono pertanto richieste ulteriori ricerche per determinarne la causa. Alcune ibridazioni in situ con sonde ad ampio spettro, compiute su esemplari raccolti nelle fasi avanzate della crisi, hanno escluso che esse potessero essere provocate da eubatteri parassiti, come inizialmente ipotizzato. Curiosamente, anche la seconda specie appartenente al gen.
Frontonia ritrovata nella pozza (cfr. Parte Prima: Introduzione, presente capitolo) è risultata
soggetta alle medesime crisi, sebbene forse con una frequenza leggermente più bassa. È possibile che l’allevamento delle specie alolimnobie del gen. Frontonia si presenti difficoltoso a causa di specifici adattamenti all’ambiente salmastro sviluppati da tali organismi, che non è stato possibile riprodurre in condizioni di laboratorio.
II.2.2: Caratterizzazione morfologica
L’identificazione morfologica della specie è stata condotta sulla base della forma, della lunghezza in vivo delle cellule e sull’analisi dell’apparato macronucleare. I numerosi
tentativi di sottoporre le cellule ad impregnazione argentica hanno dato scarsi risultati. Sono state impiegate cellule prelevate direttamente da campione e, in un secondo momento, anche cellule appartenenti al ceppo FS 81/1 (in seguito rivelatosi identico a FS 82/2 ed FS 82/3). L’osservazione in vivo ha mostrato organismi di dimensioni medie di circa 150 μm, di forma affusolata, più slargata nella parte apicale, leggermente compresse in senso dorsoventrale sebbene prive di una netta differenziazione fra i due lati. Il vacuolo contrattile è collocato in posizione marginale nei pressi della regione mediana del corpo cellulare: negli esemplari in buona salute risulta poco evidente (vd. sotto). Il numero di cinezie somatiche appare compreso fra 80 e 100 (la scarsa qualità delle impregnazioni non ha permesso una stima più precisa di tale dato). Il citostoma è collocato a circa due quinti della lunghezza della cellula: si presenta come una fessura longitudinale, di forma triangolare, dalla lunghezza di circa 18-20 μm. La sutura post-orale, caratteristica del genere, appare ben evidente, e si sviluppa lungo gran parte della superficie ventrale. Numerose tricocisti fusiformi sono presenti nella regione ectoplasmatica, immediatamente al di sotto della superficie corticale.
L’infraciliatura orale appare, nelle sue linee generali, simile a quanto descritto per la specie dulciacquicola F. leucas EHRENBERG 1838 o per quella marino-salmastricola F. marina FABRE-DOMERGUE 1891 (Dragesco and Dragesco-Kernéis 1986). Sul lato destro sono visibili i tre peniculi, pressappoco delle dimensioni del citostoma. Sul lato sinistro sono tuttavia presenti due cinezie paraorali e cinque vestibulari: tali caratteristiche non coincidono con quanto riportato né per F. marina né per F. leucas (Dragesco and Dragesco-Kernéis 1986: per dettagli cfr. Parte Terza: Discussione dei risultati ottenuti, presente capitolo). L’apparato nucleare si compone di un macronucleo di forma ellittica, dal diametro medio di 30 μm, collocato a circa un terzo dell’asse cellulare a partire dall’estremità anteriore del ciliato. I micronuclei sembrano essere numerosi; per le loro piccole dimensioni (circa 2 μm) e la collocazione generalmente vicina al macronucleo sono però difficili da osservare. La specie è stata identificata come Frontonia salmastra DRAGESCO AND DRAGESCO-KERNÉIS 1986.
II.2.3: Caratterizzazione molecolare
E’ stata caratterizzata la sequenza del gene codificante per il 18S rRNA a partire da una estrazione di DNA condotta su circa 20 cellule del ceppo FS 83/2. Dato l’esiguo numero di cellule, l’estrazione è stata effettuata utilizzando il kit “Nucleospin® Plant” (cfr. Sezione 2: Materiali e Metodi).
Per la reazione di PCR sono stati impiegati i primer F82penic (5’-GAA ACT GCG AAT GGC TC-3’) e R1513karyo (5’-TGA TCC ATC CGC AGG TTC-3’). L’inserimento di un innesco
specifico per i peniculidi ha ridotto i rischi di amplificazioni aspecifiche e/o contaminazioni; il secondo innesco utilizzato è specifico per i karyorelictidi, ma è in grado di appaiarsi anche
alla maggior parte delle sequenze di peniculidi. L’annealing è avvenuto a 57° C; l’estensione è stata prolungata per 2’ a 72° C.
Il sequenziamento parziale (primer R1052) ha dimostrato che la sequenza risultava diversa da tutte le altre sequenze di Frontonia spp. depositate nel data base di riferimento. È stato pertanto caratterizzato l’intero gene per il 16S rRNA, tramite altre due reazioni di sequenziamento utilizzando i primer R536 ed F919, in modo da ottenere una sequenza di circa 1400 nucleotidi. Tale sequenza è stata inserita in un albero filogenetico.
La sequenza di F. salmastra risulta collocata in posizione basale rispetto a quella di F.
leucas, che, al momento dell’inserimento, presenta le maggiori affinità con essa. In seguito, è
stata caratterizzata una sequenza di Frontonia sp. presentante una percentuale del 100% di identità con la sequenza di FS 83/2. Gli esemplari erano stati raccolti in un ambiente salmastro presso Marina di Massa (MC).
L’analisi filogenetica è stata effettuata mediante l’elaborazione di alberi basati sul criterio di parsimonia e sul Neighbor-joining method. La sequenza di F. salmastra si colloca in posizione basale rispetto al clade comprendente quella di F. leucas in entrambi gli alberi così ottenuti: in quello realizzato con il Parsimony criterion, tale posizione è supportata da un valore di bootstrap del 92% (con 1000 pseudorepliche).
Per verificare che gli altri ceppi isolati dal medesimo campione fossero effettivamente della stessa specie, sono state ottenute le sequenze parziali del gene per il 18S rRNA anche dei ceppi FS 81/1 e FS 82/2. Data la scarsa quantità di DNA iniziale, dovuta al basso numero di cellule impiegate, è stato necessario ricorrere a procedure di eminest onde ottenere quantità di amplificato sufficiente per il sequenziamento. Sono di seguito riportati i risultati di tali esperimenti.
Reazione Ceppo Primer Annealing t Estensione Esito Sequenza
F82penic I FS81/1 R1513karyo 57°C 2’ Prod. Non Visibile F82penic II (nest I) FS81/1 R1280penic 57°C 1’45’’ Positivo (R1052) Come FS 83/2 F82penic III FS82/2 R1513karyo 57°C 2’ Prod. Non Visibile F82penic IV (nest III) FS82/2 R1280penic 57°C 1’45’’ Positivo (R1052) Come FS 83/2
Il marcatore molecolare utilizzato indica che FS 81/1, FS 82/2 ed FS 83/2 appartengono alla medesima specie. Tale conclusione è supportata dai dati morfologici raccolti, che non evidenziano differenze significative fra ceppo e ceppo. La caratterizzazione del gene per il 18S rRNA di FS 81/1 ed FS 82/2 non è pertanto stata completata.
II.2.4: Individuazione e caratterizzazione del simbionte principale
Già durante le osservazioni preliminari in vivo, mediante microscopio ottico, di esemplari di
F. salmastra, era stato osservata una certa percentuale di individui caratterizzati dal
possedere un macronucleo più grande del normale e di forma rotondeggiante anziché ellissoidale. Tale percentuale era circa del 40% nella popolazione presente nei campioni del 02-10-2005 e del 20-25% in quella dei campioni del 12-10-2005. L’osservazione al microscopio a contrasto interferenziale ha evidenziato la presenza di oggetti fusiformi, in numero variabile da individuo a individuo, all’interno di tutti i macronuclei presentanti forma anomala. Tali oggetti sono stati estratti dal nucleo ed osservati a 1000x. Sono stati identificati come cellule dalla lunghezza media di 20 μm – ma sono stati osservati esemplari anche di 30 μm –, dalla forma allungata, slargata centralmente ed affusolata verso le due estremità, con un rigonfiamento più o meno marcato in corrispondenza della regione centrale. È stata altresì osservata la presenza di compartimentalizzazione del citoplasma, che in quasi tutti gli esemplari osservati appariva diviso in una zona compatta, opaca, nella parte anteriore della cellula, ed una regione traslucida collocata posteriormente. Ibridazioni in situ eseguite utilizzando la sonda universale per eubatteri Eub338 hanno confermato la natura batterica delle cellule. Le caratteristiche morfologiche osservate, unite alla collocazione macronucleare di tali oggetti, hanno suggerito l’ipotesi che potesse trattarsi di forme infettanti di un batterio appartenente al genere Holospora.
Alcuni degli esemplari in cui era stata osservata la presenza di oggetti endonucleari, provenienti da campioni raccolti il 16-10-2005, sono stati recuperati ed isolati nel tentativo di ottenere colture monoclonali di cellule infette. Tali tentativi hanno dato esito fallimentare per le già citate difficoltà di allevamento della specie; l’unico ceppo monoclonale di cellule infette (FS 96/1, con una percentuale di infezione del 32% al quinto mese di allevamento) è andato perduto dopo 5 mesi di coltura in seguito ad un evento di estinzione. Prove di mantenimento di piccole popolazioni di cellule infette non hanno parimenti avuto successo. È stato deciso di condurre gli esperimenti per la caratterizzazione del simbionte a partire da cellule infette prelevate direttamente dalla popolazione naturale. Le tre colture monoclonali precedentemente avviate (FS 81/1, FS 82/2, FS 83/2), sottoposte a screening, non sono risultate infette da Holospora sp.
Per avere una conferma immediata di quanto ipotizzato sulla base dei dati morfologici raccolti, ovvero che si trattasse di forme infettanti di Holospora sp., sono state eseguite ibridazioni in situ impiegando direttamente una sonda specifica per il gen. Holospora (Holosp1: Amann et al., 1991). Le cellule infette sono state prelevate dalla popolazione presente nei campioni raccolti il 02-10-2005. L’ibridazione ha dato esito positivo, confermando l’ipotesi precedentemente formulata. È stato inoltre possibile individuare anche le forme riproduttive, precedentemente non osservabili in vivo al microscopio a contrasto interferenziale in virtù delle loro piccole dimensioni. Si tratta di cellule di forma ellissoidale, a volte bastoncellare, dalla lunghezza media di 2-2,5 μm, presenti in numero variabile all’interno del macronucleo dell’ospite. I macronuclei potevano contenere quasi esclusivamente tali forme riproduttive o presentare sia le une che le altre; raramente sono stati osservati macronuclei in cui prevaleva la forma infettante.
I dati morfologici e di distribuzione così raccolti sono stati confrontati con quelli delle altre specie di Holospora precedentemente descritte: H. obtusa (HAFKINE 1890) GROMOV and OSSIPOV 1981, H. undulata (HAFKINE 1890) GROMOV and OSSIPOV 1981, H. elegans (HAFKINE 1890) PREER and PREER 1982, H. caryophila PREER and PREER 1982, H.
acuminata OSSIPOV 1980, H. bacillata FOKIN and SABANEYEVA 1993, H. curvata FOKIN
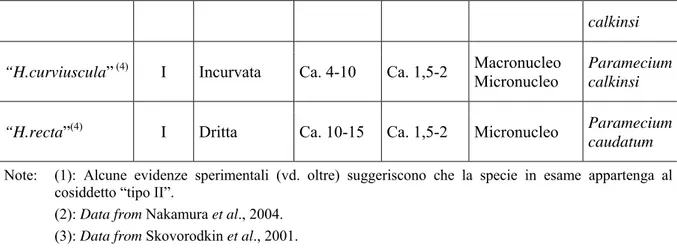
and SABANEYEVA 1993, H. curviuscula BORCHSENIUS ET AL. 1983, H. recta FOKIN 1991. La Tabella II.1 fornisce un raffronto sinottico di tali dati.
SPECIE “Tipo” Forma F.I. Lunghezza
F.I. (μm) Lunghezza F.R. (μm) Localizzazione Infezione Ospite (sp.)
[in esame] ? (1) Fusiforme Ca. 10-30 Ca. 2-2,5 Macronucleo Frontonia salmastra
H.obtusa(2) I Dritta Ca. 11-13 Ca. 1,5-2 Macronucleo Paramecium
caudatum
H.undulata(3) I Spirale Ca. 10-15 Ca. 2-2,5 Micronucleo Paramecium
caudatum
H.elegans(4) I Dritta Ca. 5-20 Ca. 1,5-2 Micronucleo Paramecium
caudatum
H.caryophila(4) II Dritta Ca. 5-6 Ca. 1-3 Macronucleo P. caudatum
P. biaurelia
“H.acuminata” (4) I Dritta Ca. 5-8 Ca. 2-2,5 Micronucleo Paramecium
bursaria
“H.bacillata” (4) II Bastoncellare Ca. 5-17 [n.r.] Macronucleo P. calkinsi
P. woodruffi
calkinsi
“H.curviuscula” (4) I Incurvata Ca. 4-10 Ca. 1,5-2 Macronucleo Micronucleo Paramecium
calkinsi
“H.recta”(4) I Dritta Ca. 10-15 Ca. 1,5-2 Micronucleo Paramecium
caudatum
Note: (1): Alcune evidenze sperimentali (vd. oltre) suggeriscono che la specie in esame appartenga al cosiddetto “tipo II”.
(2): Data from Nakamura et al., 2004. (3): Data from Skovorodkin et al., 2001. (4): Data from Görtz and Schmidt, 2005.
Tabella II.1: Confronto fra le specie di Holospora attualmente descritte (prima colonna). Le specie fra virgolette non sono attualmente convalidate. In part., è possibile che le specie H. recta ed H. elegans siano in realtà differenti morfotipi della medesima specie (Görtz, 2003). La seconda colonna riporta il “tipo”, con riferimento alla modalità di rilascio delle forme infettanti dall’ospite (vd. sopra). Sono poi riportate le caratteristiche morfologiche della infettante (F.I.; terza colonna) e la lunghezza della forma infettante e riproduttiva (F.R.; quarta e quinta colonna). La sesta colonna indica la localizzazione dell’infezione nell’apparato nucleare dell’ospite. L’ultima colonna a destra riporta il nome della specie di ciliato che costituisce l’ospite naturale del simbionte.
I dati morfologici relativi all’aspetto della forma infettante ed alle sue dimensioni non corrispondono a quelli riportati per nessun’altra specie di Holospora. Il tasso di infezione medio (non riportato in tabella) sembra differire anch’esso dalla media generale per le infezioni da Holospora sp. riscontrabile nelle popolazioni naturali dei ciliati ospiti (circa del 10%: Görtz, 1996).
- Caratterizzazione molecolare
Il gen. Holospora appartiene alla cl. Alphaproteobacteria GARRITY, BELL AND LILBURN 2005 (Amann et al., 1991). È stato pertanto possibile ottimizzare le procedure per l’amplificazione tramite PCR del gene per il 16S rRNA del simbionte a partire da un estratto di DNA genomico da cellule infette di Frontonia salmastra, prelevate direttamente da popolazione (campione raccolto il 16-10-2005). Sono stati utilizzati i primer αF19a ed αR1517, entrambi specifici per tale classe. Procedendo da cellule estratte direttamente da campione, sommariamente sciacquate in acqua distillata sterile e non sottoposte a digiuno (tale operazione viene invece normalmente effettuata per consentire la digestione di eventuali frammenti di DNA batterico contenuto nei vacuoli alimentari), è stato necessario aumentare le condizioni di stringenza della reazione in modo da minimizzare l’amplificazione di contaminanti. È stata pertanto effettuata una reazione di touchdown PCR, con temperature di annealing di 63, 57 e 50° C. Il prodotto di reazione è stato sequenziato con i primer F343, R515 e F785, in modo da caratterizzare l’intero gene. La sequenza ottenuta è stata inserita in un albero filetico. Essa è risultata collocata all’interno della
famiglia Holosporaceae GÖRTZ AND SCHMIDT 2005 (ord. Rickettsiales GIESZCZYKIEWICZ 1939).
L’elaborazione di alberi filogenetici basati sul Parsimony criterion (cfr. Sezione 2: Materiali
e Metodi) è stata inizialmente effettuata con una selezione di 17 sequenze, di cui 11
appartenenti a generi compresi nella fam. Holosporaceae o ad organismi filogeneticamente correlati ad essa, e 5 alla fam. Rickettsiaceae PINKERTON 1936, utilizzate come outgroup. Sono stati impiegati un filtro di classe “termini” ed uno specifico per α-proteobatteri. La lunghezza relativa dei rami è stata riparametrata sulle differenze fra le sequenze analizzate. L’albero ha mostrato la stabile associazione (bootstrap value 100%, con 1000 pseudorepliche) con l’unica sequenza di Holospora allora depositata in banca dati, ovvero H.
obtusa (Amann et al., 1991). È emersa però una la scarsa stabilità delle posizioni relative del
clade comprendente Holospora sp. (in esame), H. obtusa ed il loro sister group (due sequenze di batteri a vita libera già presenti in data base) all’interno delle Holosporaceae (bootstrap value 58%, con 1000 pseudorepliche); in particolare, l’associazione con il clade comprendente il gen. Caedibacter è apparsa poco stabile. Sono stati perciò disegnati altri due alberi, basati sul medesimo criterio, utilizzando una diversa selezione di sequenze (42 sequenze, di cui 18 appartenenti alla fam. Holosporaceae e 24 alla fam. Rickettsiaceae). È stato applicato, nel primo caso, la medesima selezione di filtri utilizzata in precedenza; nel secondo caso, il solo filtro specifico per α-proteobatteri. La lunghezza relativa dei rami è stata riparametrata sulle differenze fra le sequenze analizzate. Gli alberi sono risultati differire di pochissimo, presentando la medesima topologia e lievi differenze nella lunghezza dei rami. In entrambi, la sequenza in esame si collocava in posizione basale rispetto al clade comprendente le sequenze di Holospora obtusa e di H. undulata (di recente caratterizzazione e pertanto non inclusa nella precedente selezione). Tale associazione è apparsa stabile (bootstrap value 100%, con 1000 pseudorepliche). Il clade costituito da Holospora spp. e dalla sequenza di un α-proteobatterio a vita libera (UniAlp16) è risultato debolmente associato a quello comprendente il gen. Caedibacter (bootstrap value 54%, con 1000 pseudorepliche).
Sono stati realizzati, con la medesima selezione di specie, due alberi elaborati con il
Neighbor-joining method. La combinazione di filtri impiegata è quella riportata per il Parsimony criterion. La topologia di entrambi gli alberi appare leggermente diversa: il clade
del gen. Holospora risulta qui collocato in posizione basale rispetto a quello comprendente
Caedibacter spp. e la sequenza del batterio a vita libera (UniAlp16) precedentemente
associato con Holospora spp. Le distanze relative fra i nodi di separazione dei diversi cladi appaiono comunque molto ridotte, rivelando la scarsa significatività di tali associazioni. Sono stati calcolati due alberi mediante il criterio di Maximum-likelihood. La selezione di specie e la combinazione di filtri utilizzata sono le stesse riportate per gli altri sistemi di
elaborazione. Gli alberi risultanti presentano una topologia molto simile a quanto osservato per quelli basati sugli algoritmi di parsimonia. Un esempio di albero filogenetico ottenuto con questa selezione di specie è riportato al termine del presente capitolo (Figura II.1).
È stata calcolata la matrice di similarità per la configurazione di 17 sequenze precedentemente utilizzata per la realizzazione del primo albero di parsimonia. La matrice ha indicato una percentuale di identità del 93,74% fra la sequenza del 16S rDNA della specie in esame e quella di H. obtusa.
- Indagine ultrastrutturale
Alcuni esemplari di F. salmastra infetti, prelevati da campioni raccolti in data 16-10-2005, sono stati fissati per l’ottenimento di preparati per il microscopio elettronico a trasmissione (T.E.M.). Scopo di questo esperimento è stato l’ottenimento di dati riguardanti l’ultrastruttura del batterio simbionte.
Sono stati osservati numerosi batteri all’interno del macronucleo dell’ospite, sia in forma riproduttiva che infettante. Le forme riproduttive mostrano la struttura interna tipica dei batteri Gram-negativi, con una parete peptidoglicanica compresa all’interno di due membrane fosfolipidiche. Non sembrano presenti altre compartimentalizzazioni, né inclusioni citoplasmatiche od altre strutture cellulari complesse. Le forme infettanti presentano invece una zona periplasmatica elettrondensa, contenente una matrice densa ed uniforme, di natura proteica, nettamente separata dalla regione citoplasmatica più chiara. Da notare che, nella maggior parte degli esemplari osservati, l’estensione di tale zona periplasmatica è molto ridotta rispetto a come essa si presenta nelle grandi forme infettanti osservate in vivo. È possibile che le cellule osservate tramite microscopio a trasmissione si trovassero a stadi più precoci del differenziamento cellulare, quando l’accumulo di peptidi in tale comparto non è ancora stato completato, oppure che si trattasse di esemplari giunti recentemente all’interno del nucleo, le cui proteine periplasmatiche fossero già state rilasciate durante il trafficking vacuolare, in procinto di differenziarsi in forme riproduttive. Durante l’osservazione al T.E.M. sono state più volte identificate forme infettanti al di fuori del nucleo cellulare. Tali forme erano circondate da membrana e situate in diverse zone del citoplasma. Data la lontananza dal citostoma di alcuni di tali vacuoli, è stato ritenuto poco plausibile che si trattasse di forme infettanti fagocitate, dirette verso il macronucleo. È invece possibile che le cellule osservate stessero lasciando il macronucleo per poi essere rilasciate nell’ambiente. Tale modalità di rilascio delle forme infettanti (ciclo “di tipo II”: cfr. Parte
Terza: Discussione dei risultati ottenuti, presente capitolo), considerata meno evoluta
rispetto alla creazione del cluster di forme infettanti in concomitanza con la cariodieresi nucleare dell’ospite (Görtz, 2003), accomuna un gruppo di specie di Holospora, fra cui H.
È pertanto possibile che anche il simbionte di F. salmastra presenti un ciclo vitale di “tipo II”. Ulteriori verifiche sperimentali sono però richieste per chiarire le effettive modalità di rilascio delle forme infettanti da parte di questo organismo.
II.2.5: Individuazione e caratterizzazione del simbionte secondario
Gli esperimenti di ibridazione in situ condotti su cellule provenienti da popolazioni, che hanno portato all’individuazione hanno rivelato la presenza di un secondo organismo procariotico simbionte di F. salmastra, localizzato nel citoplasma delle cellule ospiti. La percentuale di infezione, in queste prime ibridazioni, non è stata determinata: risultava comunque sensibilmente più bassa di quella registrata per l’infezione da Holospora sp. (vd. sopra). Il simbionte non sembrava presentare caratteristiche morfologiche particolari: le cellule apparivano di forma rotondeggiante, leggermente allungata, dalla lunghezza approssimativa di 2-3 μm, distribuiti in numero variabile (nell’ordine delle centinaia di individui) nel citoplasma dell’organismo ospite. Sono state condotte ibridazioni in situ sui ceppi FS 81/1, FS 82/2, FS 83/2 ed FS 96/1 (quest’ultimo ceppo è andato in seguito perduto) al fine di individuare l’eventuale presenza del simbionte. Tali ibridazioni hanno dato esito positivo, sebbene la percentuale di infezione risultasse piuttosto bassa. Ibridazioni preliminari con sonde ad ampio spettro sono state effettuate sui medesimi ceppi in modo determinare il gruppo tassonomico d’appartenenza dell’organismo. Sono state parimenti testate la sonda specie-specifica CC23a (Caedibacter caryophilus), la sonda genere-specifica Roseo645 (Roseobacter spp.) e la sonda Roseogroup_530 (specifica per batteri filogeneticamente vicini al gen. Roseobacter). Date le difficoltà di allevamento di F.
salmastra precedentemente esposte, i vetrini dei ceppi FS 81/1, FS 82/2 ed FS 83/2 sono stati
realizzati con bassi numeri di cellule (5-10): l’inevitabile distacco di alcune di esse (o tutte) durante la procedura sperimentale non ha talvolta consentito di osservare l’esito dell’ibridazione, o di trarne conclusioni significative.
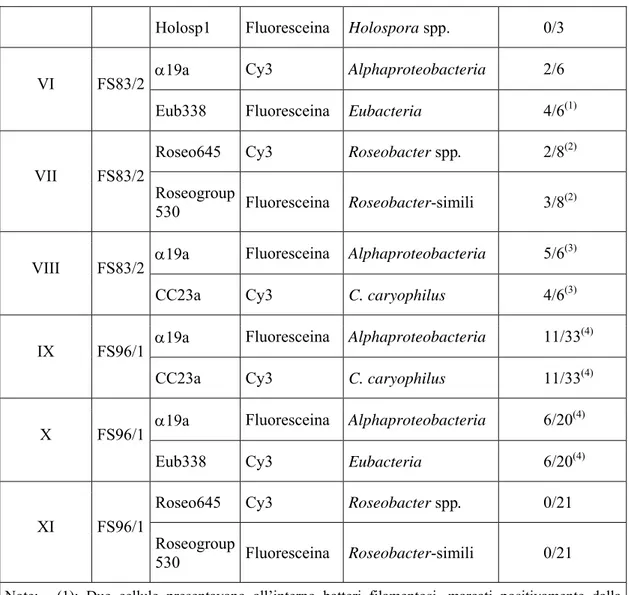
La Tabella II.2 riassume gli esiti di tali esperimenti.
Ibridazione Ceppo Sonda Fluorocromo Specificità Cellule Positive
α19a Cy3 Alphaproteobacteria 1/2
II FS82/2
Holosp1 Fluoresceina Holospora spp. 0/2
α19a Cy3 Alphaproteobacteria 1/2
IV FS83/2
Holosp1 Fluoresceina Holospora spp. 0/2
Holosp1 Fluoresceina Holospora spp. 0/3
α19a Cy3 Alphaproteobacteria 2/6
VI FS83/2
Eub338 Fluoresceina Eubacteria 4/6(1)
Roseo645 Cy3 Roseobacter spp. 2/8(2)
VII FS83/2
Roseogroup
530 Fluoresceina Roseobacter-simili 3/8(2) α19a Fluoresceina Alphaproteobacteria 5/6(3)
VIII FS83/2
CC23a Cy3 C. caryophilus 4/6(3)
α19a Fluoresceina Alphaproteobacteria 11/33(4)
IX FS96/1
CC23a Cy3 C. caryophilus 11/33(4)
α19a Fluoresceina Alphaproteobacteria 6/20(4)
X FS96/1
Eub338 Cy3 Eubacteria 6/20(4)
Roseo645 Cy3 Roseobacter spp. 0/21
XI FS96/1 Roseogroup
530 Fluoresceina Roseobacter-simili 0/21
Note: (1): Due cellule presentavano all’interno batteri filamentosi, marcati positivamente dalla sonda Eub338 ma non da α19a. In due cellule è stato osservato il simbionte secondario, positivo ad entrambe le sonde.
(2) In due cellule le sonde hanno evidenziato gli stessi batteri: disposti a catenella all’interno del citoplasma in un caso, di forma coccoide ed adesi alla superficie esterna del ciliato nell’altro. In una terza cellula, la sonda Roseogroup_530 ha legato un batterio simbionte citoplasmatico molto abbondante all’interno dell’ospite. È probabile che in questo caso la sonda abbia legato aspecificamente il simbionte secondario.
(3): Batteri positivi alla sonda α19asono stati individuati in cinque cellule; in tre casi il basso numero di esemplari individuati non permette di identificarli con il simbionte secondario. Due cellule presentavano invece l’infezione del simbionte secondario, in centinaia di esemplari, marcato dalla sonda α19a. La sonda CC23a risultava legata in modo specifico a batteri adesi alla superficie delle cellule; in alcuni casi marcava batteri presenti all’interno della cellula, ma non il simbionte secondario individuato nelle due cellule.
(4): Tutti i simbionti individuati sono stati riconosciuti appartenenti al gen. Holospora. La sonda CC23a ha marcato aspecificamente questi ultimi.
Tabella II.2: Ibridazioni in situ per l’identificazione del simbionte secondario di F. salmastra. L’ultima colonna riporta il numero di cellule, espresso come cellule positive sul totale di cellule osservate, al cui interno sia stato individuato un qualsiasi numero di batteri marcati in modo specifico dalla sonda.
Dalle ibridazioni è emersa la presenza occasionale di altri tipi di batteri all’interno delle cellule di F. salmastra. Tali infezioni appaiono tuttavia molto sporadiche all’interno della popolazione. Il simbionte secondario precedentemente descritto è risultato positivo alla
sonda specifica per gli α-proteobatteri. Dato il basso numero di cellule osservate, la stima della percentuale di infezione ricavabile per ciascun ceppo è poco attendibile; appare tuttavia minore di quella osservata nelle popolazioni naturali di F. salmastra durante le ibridazioni preliminari, suggerendo che tale simbionte possa andare perduto nel tempo.
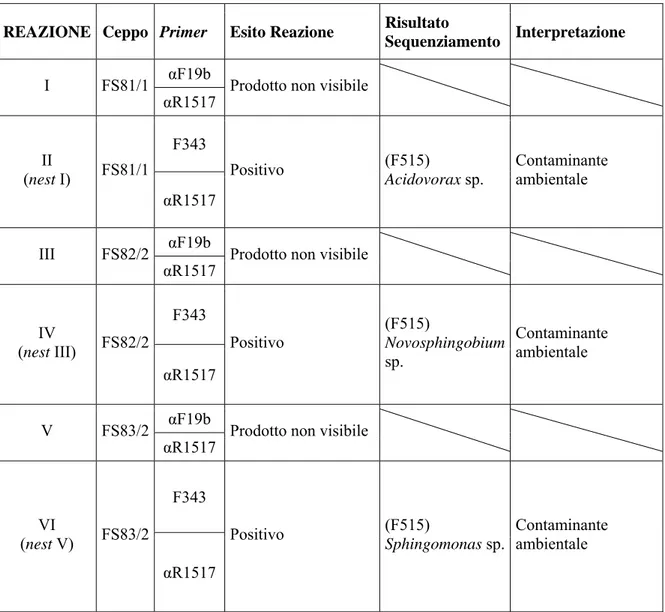
Sono stati effettuati una serie di tentativi di amplificazione del 16S rDNA del simbionte secondario a partire da estrazioni di DNA di cellule dei ceppi FS 81/1, FS 82/2 ed FS 83/2 (in cui era stata precedentemente accertata l’assenza del simbionte principale). Sono stati impiegati inneschi specifici per gli α-proteobatteri ed elevate condizioni di stringenza (touchdown PCR) allo scopo di evitare l’amplificazione di eventuale DNA contaminante. È stato fatto ricorso a procedure di eminest per incrementare la quantità di prodotto ottenuto da ciascuna reazione. Le reazioni effettuate ed i risultati ottenuti sono riportati nella Tabella II.3.
REAZIONE Ceppo Primer Esito Reazione Risultato
Sequenziamento Interpretazione
αF19b I FS81/1
αR1517 Prodotto non visibile F343 II (nest I) FS81/1 αR1517 Positivo (F515) Acidovorax sp. Contaminante ambientale αF19b III FS82/2
αR1517 Prodotto non visibile F343 IV (nest III) FS82/2 αR1517 Positivo (F515) Novosphingobium sp. Contaminante ambientale αF19b V FS83/2
αR1517 Prodotto non visibile F343 VI (nest V) FS83/2 αR1517 Positivo (F515) Sphingomonas sp. Contaminante ambientale
Tabella II.3: Tentativi di amplificazione della gene codificante per il 16S rRNA del simbionte secondario di F. salmastra. La penultima colonna riporta la specie la cui sequenza presenta la maggiore affinità con quella di nuova caratterizzazione, fra quelle presenti all’interno della banca dati di riferimento (NCBI GenBank).
La discussione e l’interpretazione di tali risultati sono riportati nella parte successiva del presente capitolo. La sequenza del simbionte secondario non è stata caratterizzata. È stato accertata la sua appartenenza alla cl. Alphaproteobacteria. La bassa percentuale di infezione nei ceppi ha probabilmente limitato la quantità di DNA-stampo presente nel campione di partenza, mentre le tecniche di amplificazione utilizzate hanno selezionato positivamente DNA batterico proveniente da contaminanti ambientali.
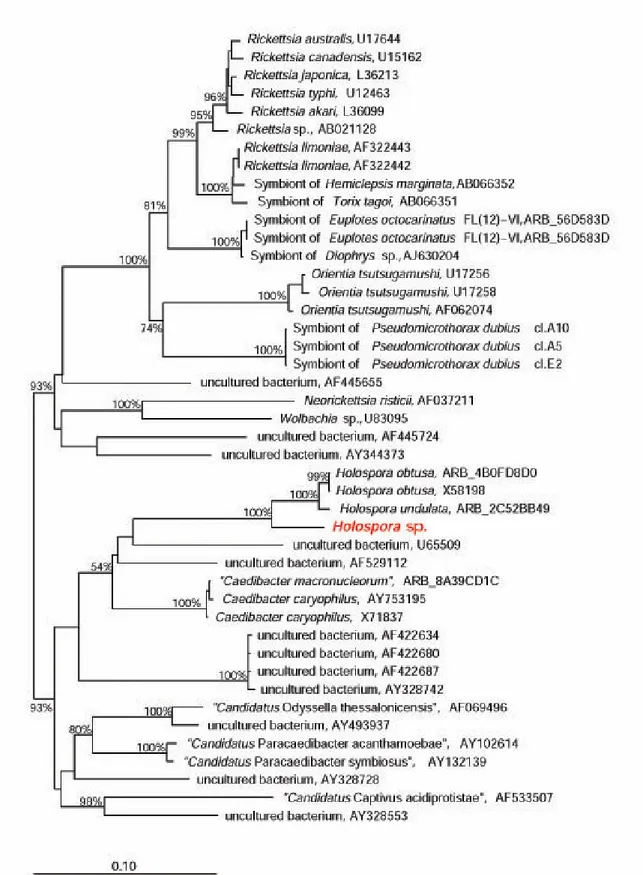
Figura II.1: Cladogramma filogenetico di una selezione di specie di α-proteobatteri appartenenti alle famiglie Rickettsiaceae ed Anaplasmataceae (clade in alto), ed Holosporaceae (clade in basso) dell’ordine Rickettsiales (per dettagli si rimanda al presente capitolo). È stata evidenziata la collocazione della sequenza di Holospora sp. simbionte di F. salmastra. I valori di bootstrap riportati sono calcolati su 1000 pseudorepliche. La lunghezza della componente orizzontale rappresenta la distanza evolutiva fra le sequenze: la barra misurante la scala corrisponde a 10 cambiamenti nucleotidici ogni 100 posizioni.
Sezione 3 – Capitolo II
Parte Terza
Discussione dei risultati ottenuti
II.3.1: Caratterizzazione morfologica e molecolare
L’identificazione morfologica della specie è risultata piuttosto difficile, soprattutto a causa della impossibilità di ottenere esemplari correttamente colorati con la tecnica dell’impregnazione argentica. È stato supposto (Fokin, pers. comm.) che ciò possa dipendere da caratteristiche peculiari della struttura corticale del ciliato in esame, che impediscono la corretta precipitazione dell’argento (la tecnica è comunque di difficile esecuzione, ed è ugualmente possibile che gli esperimenti siano falliti per cause indipendenti dalla natura delle cellule: per dettagli cfr. Sezione 2: Materiali e Metodi).
La maggior parte dei caratteri morfologici osservati in tale specie coincidono con quanto descritto per le specie F. leucas EHRENBERG 1838 e F. marina FABRE-DOMERGUE 1891 (Dragesco and Dragesco-Kernéis, 1986). In particolare, risultano molto simili a quanto descritto per F. leucas la morfologia del corpo cellulare, le dimensioni, le posizioni relative di macronucleo, vacuolo contrattile e citostoma (le descrizioni di F. leucas presentano tuttavia notevoli differenze fra loro, suggerendo una grande variabilità intraspecifica: Dragesco and Dragesco-Kernéis, 1986). Coincide con F. marina per le dimensioni, l’aspetto e la posizione del macronucleo e l’habitat marino-salmastro (F. leucas è invece una specie prettamente dulciacquicola).
Alcune differenze con le specie anzidette sono state tuttavia notate a livello dell’infraciliatura orale. F. leucas presenta una cinezia paraorale e tre (Carey, 1992) o quattro (Dragesco and Dragesco-Kernéis, 1986) cinezie vestibulari; F. marina possiede normalmente sei cinezie vestibulari ed un’unica cinezia paraorale (Dragesco and Dragesco-Kernéis, 1986). Negli esemplari osservati, le cinezie vestibulari appaiono essere cinque: tale carattere è tuttavia suscettibile di variazioni all’interno della stessa specie e potrebbe non costituire un marcatore morfologico significativo (Dragesco and Dragesco-Kernéis, 1986). Sono state osservate due cinezie paraorali: la gran parte delle specie appartenenti al gen.
Frontonia, incluse le due anzidette, ne possiede invece una sola (cfr. ad es. Carey, 1992).
Tale carattere è invece presente nella specie F. salmastra. Inoltre, gli organismi in esame condividono con essa sia la struttura generale del corpo cellulare (forma, dimensioni,
posizione di nucleo e citostoma) che le abitudini alolimnobie. La specie è stata pertanto descritta come F. salmastra DRAGESCO AND DRAGESCO-KERNÉIS 1986.
F. salmastra è stata ritrovata e descritta originariamente in ambienti salmastri dell’Africa
intertropicale (Dragesco and Dragesco-Kernéis, 1986), e sino ad oggi non era mai stata segnalata in Italia. Successivamente al ritrovamento del 02-10-2005, organismi riconducibili a quelli osservati sono stati segnalati in uno stagno costiero salmastro (Salinità 5‰) nei pressi di Marina di Massa (MC) ed in un ambiente iperalino (Salinità 49‰) lungo le coste pugliesi presso Campomarino (TA) (Fokin, pers. comm). L’identificazione con gli organismi in esame è stata effettuata su base morfologica per gli esemplari pugliesi (Fokin, pers.
comm), e morfologico-molecolare per quelli provenienti da Marina di Massa (Andreoli et al.,
2006.). Nel caso venisse confermata l’identificazione con F. salmastra, tale specie risulterebbe decisamente coarse-grained per quanto riguarda le caratteristiche di salinità dell’ambiente. La popolazione oggetto del presente studio vive all’interno di un range di salinità compreso fra il 14‰ ed il 5‰ (osservazione riferita al solo periodo in esame: 10-2005 – 06-2006).
La sequenza del gene codificante per il 18S rRNA ottenuta da tali organismi è risultata diversa da tutte le altre sequenze di Frontonia spp. presenti all’interno del data base di riferimento. Essa risulta comunque inserita all’interno del clade comprendente il gen.
Frontonia, in posizione basale rispetto alla sequenza di F. leucas. L’analisi filogenetica
effettuata ha rivelato l’associazione stabile della sequenza caratterizzata al clade comprendente quella di F. leucas, benché basalmente posta rispetto a quest’ultimo: tale associazione appare ben supportata da buoni valori di bootstrap. Come già accennato (cfr.
Parte Prima: Introduzione, presente capitolo), studi recenti hanno evidenziato la probabile
parafilia del gen. Frontonia (Andreoli et al., 2006): l’analisi di un maggior numero di sequenze appare dunque necessaria per definire con maggiore sicurezza le posizioni relative delle varie specie attualmente classificate in tale genere.
II.3.2: Caratterizzazione del simbionte principale
I batteri simbionti endonucleari presenti nella popolazione in esame sono stati identificati come α.proteobatteri appartenenti al genere Holospora.
- Cenni sulle caratteristiche del gen. Holospora
Tutte le specie batteriche comprese nel gen. Holospora (HAFKINE 1890) GROMOV AND OSSIPOV 1981 (cl. Alpharoteobacteria GARRITY, BELL AND LILBURN 2005; ord.
Rickettsiales GIESZCZYKIEWICZ 1939) ad oggi conosciute sono in grado di moltiplicarsi
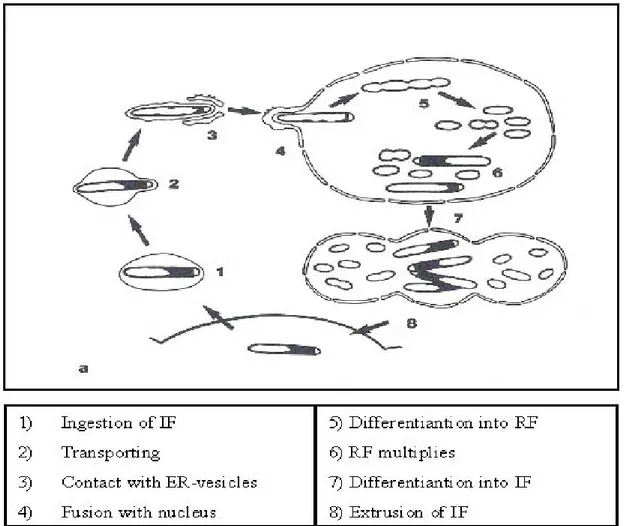
Paramecium MÜLLER 1773 (cl. Oligohymenophorea, ord. Peniculida). Il loro ciclo vitale è complesso e unico fra i procarioti. Comprende due forme: una grande forma infettante, capace di raggiungere il nucleo dell’ospite dopo essere stata ingerita da questi, ed una forma riproduttiva, di norma piccola e morfologicamente poco differenziata, in grado di riprodursi per fissione binaria all’interno del nucleo nonché di dare origine alle nuove forme infettanti. Queste vengono in seguito rilasciate dalla cellula ospite: le modalità di tale rilascio variano da specie a specie e potrebbero costituire un carattere tassonomico significativo. Un breve schema del ciclo vitale di Holospora sp. è riportato in Figura II.2; La Figura II.3 illustra invece l’organizzazione-tipo di una generica forma infettante (le immagini sono riportate al termine del presente capitolo). La cellula presenta le due membrane cellulari tipiche dei batteri Gram-negativi. Lo spazio periplasmatico compreso fra di esse occupa, nelle fasi precoci dell’infezione, più della metà della lunghezza cellulare, e contiene una matrice densa, di natura proteica, distribuita prevalentemente ad una estremità della cellula, mentre lo spazio citoplasmatico, meno denso, viene a localizzarsi presso l’altra estremità, determinando nella forma infettante una struttura polarizzata. Lo spazio periplasmatico contiene diverse proteine di membrana stadio-specifiche, che fuoriescono dalla cellula durante le prime fasi dell’infezione e prendono rapporto con le strutture cellulari dell’ospite. Il ruolo di tali proteine sembra essere quello di impedire la digestione del batterio e far dirigere il fagosoma verso l’apparato nucleare dell’ospite, all’interno del quale la forma infettante sarà rilasciata (Görtz, 2003). La forma infettante, per il resto, è fisiologicamente inattiva: costituisce una sorta di stadio di resistenza simile alle spore di altri procarioti (da cui il nome del genere), solitamente in grado di sopravvivere nell’ambiente per alcuni mesi. Una volta introdotta nel nucleo dell’ospite, essa si frammenta in 4-10 parti, originando le più piccole forme riproduttive. La struttura di tali forme ricorda quella di un tipico batterio G-, dalla forma bastoncellare e privo di evidenti accumuli proteici nel periplasma. A differenza delle forme infettanti, le forme riproduttive presentano un’alta affinità al materiale cromatinico, e permangono all’interno del nucleo durante le divisioni cellulari (Skovorodkin
et al., 2001). Esse si riproducono all’interno nel nucleo dell’ospite; in seguito, alcune di esse
aumentano di dimensioni e si differenziano in nuove forme infettanti. Nella modalità di rilascio più nota, queste forme si portano al centro, in corrispondenza del connecting piece del nucleo in divisione, formando un raggruppamento compatto (cluster). Avvenuta la cariodieresi, tale cluster viene trasportato verso il citoprocto, dal quale le forme infettanti vengono rilasciate nell’ambiente. L’altra modalità di rilascio, considerata più primitiva (Görtz, 2003), non prevede il raggruppamento delle forme infettanti, che lasciano il macronucleo, all’interno di vacuoli, in modo indipendente dalle fasi del ciclo cellulare dell’ospite; non è noto, in questo secondo caso, come avvenga l’effettiva liberazione delle spore all’esterno. Le specie di Holospora che utilizzano quest’ultima modalità di rilascio
vengono dette “di tipo II”, mentre quelle presentanti il raggruppamento delle forme infettanti ed il rilascio in corrispondenza della divisione nucleare, di “tipo I”.
Sono state descritte, ad oggi, nove specie di Holospora, di cui soltanto quattro sono ufficialmente convalidate. Come precedentemente sottolineato, si tratta di simbionti obbligati di ciliati del gen. Paramecium, in grado di riprodursi nel macronucleo o nel micronucleo dell’ospite. L’infezione è organulo-specifica ed anche specie-specifica: i simbionti possono essere artificialmente trasferiti da una specie ospite all’altra, ma sopravvivono per un tempo limitato, dopodiché vengono espulsi dal nucleo e digeriti (Fokin et al., 2005). Il significato funzionale di tale rapporto appare lungi dall’essere chiarito. Secondo alcuni autori (es: Görtz, 2003; Görtz and Schmidt, 2005) le specie di Holospora sono da considerarsi a tutti gli effetti parassiti intracellulari di Paramecium, intendendo per “parassitismo” una associazione non-mutualistica in cui l’uno dei contraenti ricava beneficio dallo sfruttamento dell’altro (adapted from Görtz, 2003; il parassitismo è talvolta considerato come una forma di predazione: Begon et al., 1996). Le caratteristiche che accomunerebbe tali specie ai parassiti “tradizionali” sarebbero l’alta infettività (verificata in condizioni di laboratorio: Restif and Kaltz, 2006), il rilascio regolare nell’ambiente di forme specializzate ai fini della propagazione dell’infezione, ed il fatto che, in almeno un caso (infezione da H. elegans in P.
caudatum), è stato accertato che il procariote inibisca la funzionalità del micronucleo del
ciliato, sede dell’infezione (Görtz, 1996), determinando una sorta di “castrazione” dell’organismo ospite simile a quanto si osserva nelle infezioni di alcune specie di trematodi o di cirripedi ai danni di crostacei decapodi (Riedl, 1991). La morte dell’ospite in conseguenza all’infezione da Holospora appare un evento raro e fortuito, sebbene possa accadere che un accumulo eccessivo di forme infettanti all’interno dell’apparato nucleare dell’ospite possa provocare la lisi della membrana nucleare (Görtz, 2003): in generale, il protista non appare danneggiato dalla presenza del batterio, anche se ciò non costituisce una prova che non si tratti ugualmente di un rapporto parassitico. Studi recenti hanno tuttavia evidenziato che, almeno in certi casi, l’ospite possa ottenere dei benefici dalla presenza del simbionte. È noto che la presenza di H. obtusa può modificare il normale tasso di espressione di almeno sei geni coinvolti nei processi di signaling intracellulare, trascrizione e metabolismo aerobio dell’ospite P. caudatum (Nakamura et al., 2004); in certi casi (Hori and Fujishima, 2003) è stato dimostrato che il simbionte è in grado di indurre alti livelli basali di espressione dei geni codificanti per le Heat Shock Proteins Hsp60 e Hsp70: ciò conferisce un preadattamento vantaggioso per l’ospite, che diviene in grado di sopravvivere a bruschi passaggi di temperatura (da 25°C a 35°C) che invece danneggiano le cellule non infette, i cui livelli basali di espressione di HSP sono più bassi. In questo caso, quindi, l’ospite trae un possibile vantaggio dalla presenza del simbionte (anche se la spesa metabolica necessaria alla trascrizione dei geni HSP è indubbiamente maggiore negli individui infetti: Hori and
Fujishima, 2003). Tale caratteristica non rientra fra quelle di un rapporto di tipo parassitico. La natura del rapporto fra Holospora sp. e l’ospite ciliato resta pertanto non chiara.
Come precedentemente affermato, tutte le specie di Holospora sinora riconosciute valide (o comunque caratterizzate in modo esaustivo) risultano essere simbionti obbligati di ciliati del gen. Paramecium. Esistono tuttavia alcune segnalazioni riguardanti la presenza di batteri endonucleari Holospora-simili, per morfologia e ciclo vitale, in ciliati appartenenti a generi diversi da Paramecium quali Stentor multiformis, Stentor polymorphus (Görtz and Wiemann, 1987), Zoothamnium pelagicum (Laval, 1970), Trithigmostoma cucullus (Görtz and Maier, 1991), Spirostomum sp. (Görtz, unpubl.), Spirostomum cfr. minus (Fokin, pers. comm.). Nessuno di questi batteri è stato caratterizzato molecolarmente; nel caso di Spirostomum cfr.
minus, l’ibridazione con la sonda Holospora-specifica Holosp1 ha dato esito negativo
(Fokin, pers. comm.). Per alcuni autori (Görtz, 2003), tali osservazioni indicherebbero che la presenza di simbionti del gen. Holospora non sarebbe ristretta a Paramecium spp. Occorrerebbero tuttavia verifiche di tipo molecolare al fine di chiarire i reali rapporti filogenetici fra questi simbionti e le specie di Holospora attualmente caratterizzate.
- Identificazione e caratterizzazione del simbionte principale
Il batterio Holospora-simile trovato in Frontonia salmastra rappresenta il primo caso di caratterizzazione molecolare di un simbionte del genere Holospora che svolge il proprio ciclo vitale all’interno di un ospite ciliato non appartenente al gen. Paramecium. La sequenza ottenuta risulta possedere una percentuale di similarità (93,74%) è inferiore a quella convenzionalmente ritenuta sufficiente per poter stabilire l’identità fra specie batteriche (97%): è possibile confermare che si tratta di due specie differenti. L’analisi filogenetica della sequenza di 16S rDNA ottenuta lo colloca stabilmente all’interno del clade comprendente le altre due specie di Holospora sinora caratterizzate. Tale associazione risulta infatti stabile in tutti gli alberi filogenetici realizzati, basati su differenti algoritmi. In particolare, per gli alberi realizzati mediante il Parsimony criterion è stato possibile calcolare i valori di bootstrap relativi ai nodi di tale associazione: le specie sono associate in tutte le 1000 pseudorepliche (bootstrap value 100%), dimostrando la stabilità di tale risultato. La similarità fra le tre specie appare piuttosto alta, a giudicare dalle distanze riportate negli alberi calcolati con il Neighbor-joining method. All’interno di tale clade, il simbionte di F.
salmastra si colloca basalmente rispetto al cluster formato dalle altre due specie (H. obtusa e H. undulata), risultando la più dissimile fra le tre. Il dato molecolare concorda con le
peculiarità già osservate in questa specie, relative soprattutto alle dimensioni della forma infettante, all’infettività ed al ciclo vitale, probabilmente di “tipo II” (le altre due specie presentano entrambe un ciclo di “tipo I”). La percentuale di similarità calcolata fra la sequenza del simbionte in esame e quella di H. obtusa è comunque inferiore al 97%,
convenzionalmente ritenuta il valore-soglia per poter confermare l’identità fra specie batteriche: la sequenza ottenuta non appartiene pertanto a nessuna delle specie precedentemente caratterizzate. I dati molecolari sin qui raccolti suggeriscono l’ipotesi che tale organismo appartenga ad una nuova specie del gen. Holospora. È possibile che l’associazione con Frontonia abbia contribuito a differenziare, anche molecolarmente, tale
Holospora dalle altre specie congeneri simbionti di Paramecium. Tuttavia, la pressione
selettiva che agisce sugli organismi simbionti obbligati è differente, e spesso più bassa, di quanto avviene per gli organismi a vita libera; conseguentemente, i primi possono accumulare mutazioni e divergere fra loro più rapidamente di questi ultimi (Görtz, 1996). Tali considerazioni rendono i dati ottenuti tramite marcatori molecolari di più difficile interpretazione.
La stabilità dell’associazione del clade “Holospora” con quello comprendente il gen.
Caedibacter appare invece debole. I valori di bootstrap calcolati per gli alberi ottenuti con
algoritmi di parsimonia risultano bassi (54%), e la distanza fra i nodi da cui tali cladi originano, calcolata con il Neighbor-joining, è poco significativa. Ciò può significare un basso grado di parentela fra questi due generi. Alternativamente, è possibile che tale risultato sia artefattuale: possibili cause sono la presenza di regioni ipervariabili all’interno della sequenza del 16s rDNA di Caedibacter spp., il basso numero di sequenze di organismi di entrambi i generi ad oggi disponibili per il confronto e la considerazione sopra riportata che ”l’orologio molecolare dei simbionti può girare più in fretta” (Görtz, 1996). Ulteriori analisi sono richieste per definire con maggiore precisione le posizioni relative di tali cladi all’interno della famiglia Holosporaceae.
- Considerazioni sulla natura dell’associazione fra ospite e simbionte
I risultati sin qui ottenuti non consentono di formulare ipotesi valide circa la natura del rapporto fra F. salmastra ed Holospora sp. Sono tuttavia possibili alcune considerazioni. La percentuale di infezione registrata nella popolazione naturale presente nell’ambiente monitorato appare variabile nel tempo, anche di diversi punti percentuali. Al momento non è possibile stabilire se tale variazione abbia un andamento ciclico oppure sia stocastica: si può comunque ritenere plausibile che uno o più fattori ambientali possano influenzarne l’andamento. Verificare simili ipotesi costituirà un possibile sviluppo del presente lavoro (cfr. Sezione 5: Conclusioni e Prospettive Future). Tale percentuale appare comunque superiore a quella media riportata per le infezioni da Holospora in popolazioni naturali di
Paramecium spp. (circa 10%: Görtz, 2003), suggerendo una infettività particolarmente
elevata del batterio e/o una minore resistenza del ciliato.
La presenza del simbionte non sembra danneggiare funzionalmente l’ospite, almeno per quanto concerne la sua attività vegetativa. L’infezione è stata scoperta in un ceppo
monoclonale allevato per oltre cinque mesi, il che indica che le cellule infette mantengono la capacità di dividersi (si suppone che l’infezione fosse presente nell’individuo da cui è originato il ceppo). Si ignora se le cellule infette siano o meno in grado di effettuare con successo la riproduzione sessuale: durante il periodo di svolgimento del presente studio, non sono del resto mai stati individuati esemplari di F. salmastra in coniugazione. Apparentemente, il comportamento degli individui infetti e non infetti, osservati in condizioni di laboratorio, non differisce.
Non sono state effettuate ricerche volte a determinare se la presenza del simbionte inducesse una qualche alterazione nella normale espressione genica dell’ospite, come si verifica in P.
caudatum (Nakamura et al., 2004); è quindi impossibile stabilire se la presenza del simbionte
conferisce un preadattamento favorevole all’ospite (conferendo resistenza agli shock termici, o decrementandone la palabilità, e così via), o se al contrario l’infezione comporti una spesa metabolica maggiore e sia conseguentemente debilitante. È stato tuttavia osservato che, dopo il prelievo del campione e la sua collocazione in laboratorio, la percentuale di cellule infette inizia a diminuire rapidamente, e l’infezione scompare nel giro di poche settimane. Ciò prova ulteriormente la dispensabilità del simbionte per la sopravvivenza dell’ospite in condizioni di laboratorio. Non è tuttavia chiaro il motivo per cui l’infezione scompare. Solitamente, i batteri del genere Holospora mantengono la propria infettività in condizioni di laboratorio, e si diffondono rapidamente nelle colture: è possibile raggiungere percentuali di infezione del 100% (Görtz, 2003). Tale dato è riferito a colture pure: è possibile che, all’interno del campione prelevato, si vengano a creare condizioni ambientali che favoriscono l’abbassarsi della prevalenza dell’infezione. Tuttavia, anche le percentuali registrate nell’unico ceppo monoclonale infetto erano più basse di quanto riportato in letteratura. Apparentemente, il batterio in questione possiede una capacità infettante, in condizioni naturali, superiore a quella media delle specie congeneri, ma, a differenza di esse, non è in grado di mantenersi nel campione o in coltura pura.
II.3.3: Considerazioni sul simbionte secondario
Durante le ibridazioni in situ con sonde ad ampio spettro sono stati osservati più volte batteri presenti all’interno del citoplasma, solitamente in basso numero. Tali organismi presentavano caratteristiche morfologiche differenti fra loro: forme e dimensioni differenti, diversa organizzazione in strutture pluricellulari (ammassi, filamenti, catenelle), differente positività alle sonde gruppo-specifiche utilizzate. I batteri osservati non sembravano essere contenuti in fagosomi: si presentavano infatti sparpagliati all’interno del citoplasma; quelli organizzati in catenelle o filamenti si sviluppavano liberamente all’interno del citoplasma dell’ospite. Del resto, il gen. Frontonia comprende quasi esclusivamente specie algivore (Fokin, pers. comm.): effettivamente, i vacuoli alimentari individuati in vivo e in esemplari
ibridati di F. salmastra sono risultati contenere quasi esclusivamente alghe unicellulari, sia negli esemplari provenienti da coltura che in quelli raccolti direttamente da campione, mentre la presenza di batteri in tali vacuoli appare sporadica (ibridazioni di confronto realizzate su cellule di Paramecium calkinsi, specie sia algivora che batterivora, hanno consentito un paragone diretto con quanto osservato per F. salmastra). Tali dati suggeriscono che vari tipi di organismi procarioti possono occasionalmente stabilire rapporti di tipo simbiotico con questa specie.
L’organismo definito come “simbionte secondario” si differenzia dai casi precedentemente esposti per la sua abbondanza all’interno delle cellule infette, che possono ospitarne centinaia di esemplari. La percentuale di infezione appare però soltanto leggermente superiore a quanto registrato per gli altri batteri individuati (in totale, di circa il 6% nelle colture cellulari mantenute per alcuni mesi), e sembra decrescere con il passare del tempo e/o col mantenimento delle cellule in condizioni di laboratorio. È pertanto possibile che, anche in questo caso, si tratti di una simbiosi occasionale, dispensabile per l’ospite nelle condizioni di allevamento adottate e soggetta ad andare rapidamente perduta. Le percentuali di infezione riscontrate in natura risultano più basse di quanto registrato per Holospora sp. (Fokin, pers. comm.), suggerendo che anche il tasso di infezione della popolazione naturale sia più basso.
Le ibridazioni con sonde classe-specifiche hanno rivelato l’appartenenza di tale organismo alla cl. Alphaproteobacteria. L’ibridazione positiva ottenuta con la sonda genere-specifica Roseogroup_530 è stata interpretata come artefattuale. Nel caso specifico, la medesima sonda aveva legato, oltre al simbionte secondario, anche numerosi batteri esternamente posti rispetto alla cellula, in quantità tale da far ritenere improbabile che essi rientrassero tutti nel gen. Roseobacter o in gruppi ad esso filogeneticamente correlati; inoltre, la sonda
Roseobacter-specifica Roseo645, utilizzata in combinazione con la precedente, non aveva
marcato il simbionte secondario. Tutte le altre sonde genere-specifiche utilizzate hanno dato esito negativo. L’organismo bersaglio è stato marcato in modo non ambiguo dalle sole sonde Eub338 (eubatteri) ed α19a (α-proteobatteri).
Dai tentativi di caratterizzazione molecolare, effettuati mediante sequenziamento diretto del prodotto di amplificazione dell’estratto di DNA di cellule appartenenti ai tre ceppi monoclonali disponibili, sono state ottenute tre sequenze di 16S rDNA. In particolare, dai campioni di FS 83/2 e FS 82/2 sono state ottenute due sequenze di batteri sfingomonadidi (cl. Alphaproteobacteria), appartenenti rispettivamente ai generi, filogeneticamente molto vicini, Sphingomonas e Novosphingobium. Entrambi i generi comprendono specie di batteri a vita libera, saprobionti, comuni in ambienti stagnanti: non si segnalano specie simbionti (Balkwill et al., 2004). È stato ritenuto poco plausibile che si trattasse delle sequenze dei simbionti secondari: tale considerazione è stata avvalorata dalla discreta percentuale di
dissimilarità delle sequenze stesse, che ne consente l’assegnazione a due generi differenti. L’ipotesi che i due ceppi possedessero ciascuno un diverso tipo di simbionte, filogeneticamente correlato all’altro ed entrambi appartenenti ad una famiglia nella quale non sono mai state segnalate specie simbionti è stata ritenuta poco parsimoniosa, in termini di assunti a priori, ed è stata scartata. Successivamente, la sequenza parziale di un batterio appartenente al gen. Sphingomonas è stata ottenuta durante il tentativo di sequenziamento di
Holospora undulata simbionte di Paramecium caudatum. Poiché in tale coltura non è mai
stata segnalata la presenza di un simbionte secondario citoplasmatico, è apparso evidente che si trattasse di un batterio contaminante a vita libera. La coltura di P. caudatum è vecchia di molti mesi (Fokin, pers. comm.), e non proviene dal medesimo ambiente in cui è stata campionata F. salmastra: è possibile che Sphingomonas spp. sia un batterio molto comune ed in grado di resistere per lungo tempo nelle colture cellulari, oppure che la contaminazione sia avvenuta, in tempi recenti, nel laboratorio in cui sono conservate le colture di F.
salmastra. Tali considerazioni avvalorano l’ipotesi che Sphingomonas sp. (e per estensione
anche Novosphingobium sp.) non rappresenti il simbionte secondario, bensì un contaminante ambientale presente nelle colture cellulari di F. salmastra e P. caudatum.
Dal ceppo FS 81/1 è stata ottenuta una sequenza di Acidovorax sp. (Burkholderiales,
Comamonadaceae). Poiché tale famiglia appartiene alla classe Betaproteobacteria (Willems
and De Vos, 2002), risulta evidente che la sequenza caratterizzata non appartiene all’organismo osservato. Inoltre, all’interno di questo genere, non si segnalano specie simbionti di ciliati.
Figura II.2: Ciclo vitale (di “tipo I”) di un batterio del gen. Holospora (adapted from Görtz, 1996).
Figura II.3 (a fianco): Schema dell’organizzazione generale di una forma infettante di Holospora sp. Sono distinguibili il citoplasma batterico (C), traslucido, fortemente polarizzato, internamente posto rispetto agli altri comparti cellulari e circondato dalla membrana plasmatica interna (IM); lo spazio periplasmatico (P), contenente sostanze proteiche elettrondense, separato dallo spazio extracellulare dalla parete e dalla membrana esterna (OM); la struttura di aspetto ialino posta sull’apice cellulare (T). Durante le fasi precoci dell’infezione avviene il rilascio di alcune delle proteine periplasmatiche: il volume dello spazio periplasmatico viene progressivamente a ridursi, mentre il citoplasma, precedentemente localizzato presso il polo opposto, torna ad espandersi verso l’interno della cellula. In questa fase, le proteine rilasciate interagiscono con le membrane dell’ospite, impedendo l’acidificazione del fagosoma ed indirizzando lo stesso verso l’apparato nucleare. La fusione della membrana del fagosoma con quella nucleare consentirà la penetrazione del batterio all’interno del nucleo, dove avverrà la differenziazione in forme vegetative (adapted from Görtz, 1996).