M
M
A
A
T
T
E
E
R
R
I
I
A
A
L
L
I
I
e
e
M
15
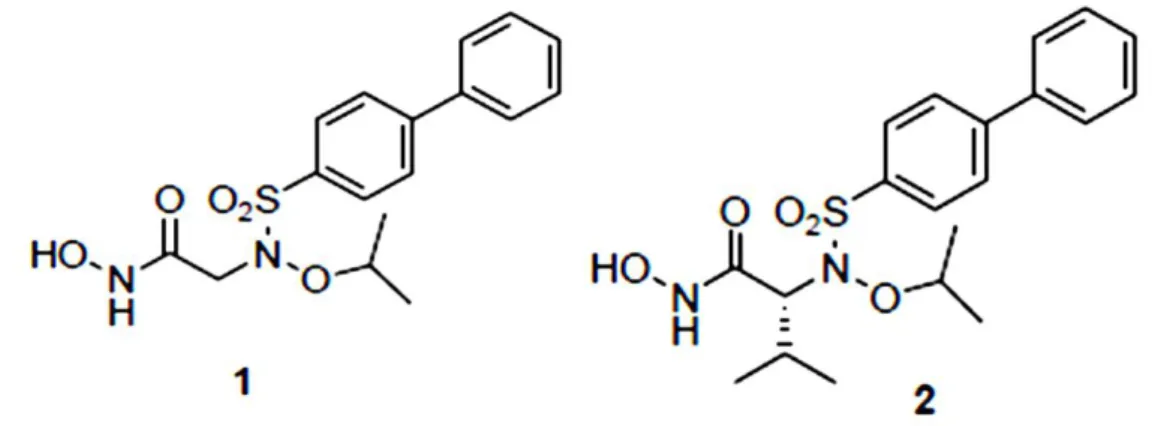
6. Inibitori della MMP-2
In questo lavoro di tesi sono stati saggiati due potenti inibitori della
MMP-2, il composto 1 e il composto 2, appartenenti alla famiglia degli N-O-isopropil sulfonammide idrossammati, sintetizzati nel laboratorio
diretto dal Professor Rossello, presso il Dipartimento di Scienze Farmaceutiche dell’Università di Pisa (fig.3).
Fig 3. Strutture chimiche dei composti 1 e 2.
Come standard di riferimento è stato utilizzato il composto CGS_27023A appartenente alla famiglia di idrossammati sulfonammidici terziari (Fig.4).
16
7. Coltura cellulare
La linea cellulare di glioblastoma umano U87MG è stata fornita dall’Istituto Nazionale per la Ricerca sul Cancro(ICLC) di Genova.
Le cellule U87MG (1 x106) sono state seminate in piastre di 10 cm e la coltura è avvenuta in mezzo completo sterile costituito da:
RPMI 1640 (Cambrex Bio Science Walkersville, Inc.; Walkersville, MD) addizionato di siero fetale bovino (FBS) al 10%;
L-glutammina 2 mM;
100 U/ml di penicillina;
100 mg/ml di streptomicina;
1% di amminoacidi non essenziali (GIBCO, Milano Italia).
La coltura è stata mantenuta nell’incubatore a 37°C in atmosfera umidificata composta da 5% di CO2 e 95% di O2.
Dopo 24 h il mezzo è stato sostituito con 2 ml di mezzo completo privo di siero .
17
8. Zimografia delle MMPs
La zimografia è una tecnica elettroforetica utilizzata per identificare su gel di poliacrilammide, in condizioni non denaturanti, l’attività proteolitica di enzimi separati sulla base del loro peso molecolare. Questa tecnica permette di evidenziare sia la forma pro-enzimatica che la forma attiva della MMP-2, in quanto hanno differenti pesi molecolari. Inoltre l’elettroforesi garantisce un’efficiente separazione di questo enzima da eventuali TIMPs.
La zimografia consiste nell’impiegare gel di poliacrilammide che è stato polimerizzato incorporando il substrato della MMP-2, cioè gelatina. Inoltre, la corsa elettroforetica viene effettuata in presenza di sodio dodici solfato (SDS), un agente denaturante che conferisce alle proteine una carica negativa, in modo tale da permetterne la separazione in seguito all’applicazione di un campo elettrico. Alla corsa elettroforetica è fatta seguire una prima fase di rinaturazione con Triton X-100, il quale rimuove i residui SDS presenti nel gel di poliacrilammide. In seguito il gel è incubato in condizioni di temperatura idonea e in presenza di cofattori, in modo da favorire l’attività gelatinolitica dell’enzima. La tecnica si conclude con la colorazione del gel, seguita da decolorazione, fino alla comparsa di bande bianche che rappresentano l’esito della digestione del substrato di gelatina da parte dell’enzima.
Questa tecnica è molto sensibile in quanto permette di evidenziare concentrazioni di enzima nell’ordine dei picogrammi.
Al fine di valutare il grado di inibizione esercitato dai composti 1, 2 e da CGS_27023A sull’attività di MMP-2, il mezzo privo di siero delle cellule U87MG in coltura è stato raccolto, centrifugato per rimuovere eventuali detriti cellulari e il sovranatante è stato incubato per 3 h a temperatura
18
ambiente con i composti 1, 2 o CGS_27023A alle concentrazioni corrispondenti alle loro IC50 (12 nM, 1 nM e 25 nM rispettivamente).
Il sovranatante così ottenuto è stato sottoposto ad un dosaggio proteico utilizzando un agente colorimetrico, il Biorad rosso (metodo di Bradford): tramite l’utilizzo di una retta di taratura, costruita con concentrazioni note di albumina, è possibile risalire alla concentrazione proteica di una soluzione effettuando una lettura spettrofotometrica a 595 nm.
Preparazione dei gel di poliacrilammide:
Gel di impaccamento (Upper gel): è il gel all’interno del quale vengono costituiti i pozzetti per il caricamento dei campioni. Ha la funzione di concentrare il campione proteico in una sottile banda prima che inizi la corsa elettroforetica.
Gel di separazione (Lower gel): è il gel dove avviene la separazione delle proteine in base al peso molecolare e dove viene incorporata la gelatina (Sigma-Aldrich, St. Louis, MO, USA) che rappresenta il substrato enzimatico. E’ un gel di poliacrilammide al 10% contenente SDS denaturante.
La polimerizzazione dei gel avviene mediante una reazione di catalisi radicalica che prevede l’aggiunta di un iniziatore di reazione, l’ammonio persolfato (APS), e di un catalizzatore, N,N,N’,N’-tetrametilendiammina (TEMED).
In seguito, 2 µg di proteine da ciascun campione sono stati addizionati della soluzione tampone 5X e caricati nel gel.
La soluzione tampone 5X contiene: - Tris-HCl 0,5 M pH 6.8
- SDS 2% che denatura le proteine fornendo loro una carica negativa; - Glicerolo 15%, utilizzato come addensante per favorire il
19
- Blu di bromofenolo, colorante inerte che rappresenta il fronte di migrazione.
La corsa elettroforetica viene effettuata nel tampone di corsa 1X (Tris-HCl, glicina, SDS, pH 8.6) ad amperaggio costante e viene fermata quando la linea blu esce dal fondo del gel.
Il gel viene prelevato e, a seguito dell’eliminazione del gel di impaccamento mediante una spatolina, vengono effettuati due lavaggi con Triton X-100 al 2,5% per 30 minuti. Successivamente il gel viene incubato per circa 19 ore a 20°C nel tampone di incubazione (Tris-HCl 50mM pH 7.5, CaCl2 10mM, NaCl 150 mM, Briji 35 0,05%).
Il giorno successivo il gel viene rimosso dal tampone di incubazione e viene immerso in una vaschetta contenente la soluzione colorante (Coomassie Brilliant Blue R-250 0,25%, metanolo 50%, acido acetico 10% in H2O MilliQ). Dopo 15 minuti, il gel viene trasferito in un’altra vaschetta
contenente la soluzione decolorante (metanolo 30% e acido acetico 10% in H2O MilliQ) dove viene lasciato finché non appaiono le bande chiare. Se
necessario l’operazione di lavaggio viene ripetuta più volte.
Le bande chiare che appaiono dopo la decolorazione corrispondono all’attività di digestione della gelatina da parte delle MMP-2, la quale può essere quantificata mediante il software che permette l’analisi densitometrica dell’immagine (ImageJ, National Institutes of Health, Bethesda, Maryland, USA).
20
9.
Studio degli effetti dei composti 1 e 2 sull’mRNA di MMP-2
Per valutare l’espressione genica della MMP-2, cellule U87MG non trattate e cellule U87MG trattate per 24 h con i composti in studio sono state lisate e usate per valutare i livelli di mRNA della MMP-2 mediante Real-time PCR.
9.1 Estrazione di RNA totale
L’estrazione di RNA è stata effettuata utilizzando il Qiagen Rneasy®
Mini Kit 50 (Qiagen, Hilden, Germany).
Il processo di estrazione dell' RNA consiste nel trattare il pellet di cellule con un tampone di lisi, contenente un detergente che rompe le membrane cellulari, e con una proteasi, un enzima necessario per la digestione delle componenti cellulari di natura proteica. Questa miscela viene successivamente trasferita in colonnine contenenti una membrana di gel di silice che lega selettivamente l'RNA, lasciando passare i polisaccaridi e le proteine. Gli acidi nucleici si legano, infatti, in modo specifico e reversibile alla superficie dei materiali silicati in presenza di sali, i quali determinano la rottura della struttura organizzata dell’acqua che circonda le molecole di RNA, favorendone l’assorbimento.
L’acido nucleico viene quindi eluito tramite lavaggi con un opportuno tampone a bassa concentrazione salina, permettendo l’ottenimento di un eluato di RNA puro.
Prima di procedere con l'estrazione, tutto il materiale necessario per le operazioni è stato immerso in una soluzione di NaOH 0,1 M per almeno 12 ore, in modo da eliminare le RNAsi presenti sui materiali, le quali, essendo in grado di degradare le molecole di RNA, andrebbero a compromettere la riuscita dell'estrazione di RNA integro.
21
Il materiale, prima dell'utilizzo, è stato lavato con acqua demineralizzata e asciugato.
L’estrazione di RNA è stata eseguita sulla base del protocollo fornito dalla ditta:
il pellet di cellule è stato sospeso in un tampone di lisi, preparato estemporaneamente, costituito da 600 µl di buffer RLT e da 6 µl di una soluzione di β-mercaptoetanolo
la sospensione è stata agitata vigorosamente con l'ausilio di un vortex per 1 minuto
alla sospensione sono stati aggiunti 600 µl di etanolo al 70%
la soluzione è stata trasferita in un apposito tubo raccoglitore,
provvisto di colonnina interna, e sottoposta a centrifugazione a 9300 g (10000 rpm; in microcentrifuga modello 5451D con rotore F45-24-11) per 15 secondi
è stato scartato il filtrato, sono stati aggiunti 700 µl di Buffer RW1 (tampone di lavaggio) ed è stata effettuata una centrifugazione a 9300 g (10000 rpm) per 15 secondi
è stato scartato nuovamente il filtrato e, dopo aver cambiato il tubo raccoglitore, alla colonnina sono stati aggiunti 500 µl di Buffer RPE (tampone di lavaggio) ed è stata effettuata una centrifugazione a 9300 g (10000 rpm) per 15 secondi
è stato scartato il filtrato, sono stati aggiunti 500 µl di Buffer RPE ed è stata effettuata una centrifugazione a 9300 g (10000 rpm) per 2 minuti
è stato scartato il filtrato ed è stata nuovamente fatta una
centrifugazione a 9300 g (10000 rpm) per 2 minuti
dopo aver cambiato il tubo raccoglitore, sono stati aggiunti 30 µl di acqua RNAsi-free ed è stata effettuata una centrifugazione a 9300 g (10000 rpm) per 1 minuto. Il filtrato ottenuto costituisce il
22
primo eluato e contiene RNA altamente concentrato (Fig.5).
23
9.2 Retrotrascrizione dell’RNA in cDNA
La retrotrascrizione è un processo attraverso cui un singolo filamento di RNA è retrotrascritto nel corrispondente DNA (cDNA) mediante l’utilizzo dell’enzima trascrittasi inversa, un primer, i precursori deossiribonucleotidi trifosfato (dNTP) e un inibitore delle RNA-polimerasi.
La trascrittasi inversa sintetizza una catena di DNA complementare allo stampo di mRNA, utilizzando i dNTPs. L’innesco per l’azione della trascrittasi inversa è rappresentato dal primer, il quale riconosce una sequenza di mRNA a lui complementare e vi si appaia.
Per la retrotrascrizione ha una notevole importanza la coda di circa 200 residui di adenina (coda poli-A) presente all’estremità 3’-OH degli mRNA eucariotici maturi e loro caratteristica peculiare. Incubando l'RNA totale estratto da una cellula con l'enzima trascrittasi inversa, i dNTPs, un tampone e ioni bivalenti Mg2+, in presenza di oligo(dT), un primer di circa venti residui di timina, è possibile dare il via alla formazione di un filamento di cDNA (DNA copia). Il primer, secondo le regole di complementarietà, riconosce specificatamente la coda di poli-A presente esclusivamente negli mRNA. In questo modo solo l' mRNA, l'unico che ha l'oligo(dT) appaiato, verrà retrotrascritto in cDNA; questo processo di appaiamento è definito annealing. Successivamente l’oligo(dT) fornisce l’OH iniziale che la trascrittasi inversa usa per allungare la catena usando come stampo il trascritto; questa seconda fase è detta di allungamento
24
Fig.6 Schema della retrotrascrizione [ adattata da www.8e.devbio.com ].
Per la retrotrascrizione è stato utilizzato il Qiagen Quantitect® Reverse Transcriptase Kit (Qiagen, Hilden, Germany).
La retrotrascrizione dell' RNA è stata eseguita sulla base del protocollo fornito dalla ditta (Fig.7):
in una eppendorf da 1,5 ml sono stati messi - 6 µl di RNA
- 4 µl di gDNA - 18 µl di H2O
e lasciati in incubazione a 42°C per 2 minuti per degradare l'eventuale DNA genomico presente
il campione cosi ottenuto è stato suddiviso in 2 eppendorf, ciascuna contenente 14 µl della soluzione precedentemente preparata
in una delle due eppendorf sono stati aggiunti 6 µl di master mix così composto:
- 1 µl di enzima Quantiscript Reverse Transcriptasi (retrotrascrittasi)
25
- 1 µl di RT Primer Mix
- 4 µl di Quantiscript RT Buffer
nella seconda eppendorf sono stati invece aggiunti 6 µl del master mix precedente, con la differenza che al posto di 1 µl di enzima è stato messo 1 µl di acqua RNAsi-free
entrambe le soluzioni sono state incubate a 42°C per 15 minuti e successivamente a 95°C per 3 minuti.
26
9.3 Real-time PCR
La real-time PCR è una variante della PCR (reazione a catena della polimerasi) che, mediante l’utilizzo di un colorante fluorescente, permette di quantificare i livelli di DNA o di cDNA durante la fase esponenziale dell’amplificazione.
Come la PCR tradizionale, per ottenere la polimerizzazione sono necessari:
il DNA genomico o il cDNA
l’enzima polimerasi, in particolare la Taq polimerasi, una variante termostabile ricavata dal batterio termofilo
Thermus aquaticus
una coppia di primers, cioè oligonucleotidi sintetici lunghi 18-30 nucleotidi complementari alle sequenze a monte e a valle del frammento che si desidera amplificare. Costituiscono l’innesco per l’attività della DNA polimerasi. I due primers non dovrebbero avere tratti di sequenza complementari tra di loro in modo da evitare la possibilità di dimerizzazione e la conseguente riduzione della loro concentrazione nella miscela di reazione.
E’ inoltre importante determinare la loro temperatura di denaturazione:
Tm (temperatura di melting) = 2 x (A+T) + 4 x (G+C)
e avere informazioni sulla loro temperatura di appaiamento:
27 ioni magnesio (Mg2+), la cui concentrazione è un fattore importante per l’attivazione della DNA polimerasi: infatti in assenza di un’adeguata concentrazione di magnesio libero è inattiva, mentre quando è in eccesso può aumentare il livello di amplificazione aspecifica
nucleotidi trifosfato (dATP – dGTP – dTTP – dCTP), la cui concentrazione non deve essere inferiore ai 200 µM, sia per impedire che la frequenza di incorporazioni errate aumenti fortemente, sia per riuscire a mantenere elevata la loro concentrazione anche negli ultimi cicli di amplificazione
un tampone salino nel quale far avvenire la reazione. La procedura di PCR consiste in:
o Ia
fase: denaturazione del DNA. Avviene ad una temperatura intorno a 95°C e serve a separare le due eliche complementari o IIa
fase: annealing (appaiamento). Avviene a temperature variabili comprese tra i 40 e i 68 °C, a seconda della sequenza di DNA da amplificare dalle caratteristiche dei primers. In questa fase avviene l’appaiamento dei primers con la loro sequenza complementare
o IIIa
fase: allungamento. Avviene a 72°C ad opera della DNA polimerasi, la quale utilizza i primers come innesco per costruire una copia complementare alla sequenza di interesse. Queste tre fasi vengono ripetute per un numero di cicli variabile, tale da portare all’amplificazione esponenziale del frammento di interesse (2n
, dove n è il numero di cicli).
La real-time PCR differisce dalla PCR classica per l’aggiunta, nella miscela di reazione, di un colorante che emette fluorescenza, la quale viene rilevata da un apposito detector e permette di seguire l’amplificazione in tempo
28
reale. L’aumento dell’emissione di fluorescenza viene riportato in un grafico in relazione al numero di cicli della PCR (Fig.8)
Fig.8 Modello di curva della real-time PCR. Rappresenta la variazione di fluorescenza
(ΔRn) in funzione del numero di cicli di PCR. Riadattata da www.ncbi.nlm.nih.gov › ProbeDB › Technologies.
Il numero di cicli di PCR in cui il segnale fluorescente è al di sotto del limite misurabile dallo strumento viene definito Baseline e in base alla variabilità di quest’ultimo viene scelto un livello di fluorescenza (Threshold) al di sopra del quale il segnale misurato può essere usato per definire il Threshold cycle (Ct) di un campione.
Il Ct è definito come il numero di cicli di PCR necessari affinché la fluorescenza misurata sia maggiore del Threshold e risulta il parametro fondamentale per ottenere dati accurati e riproducibili.
Segue poi la fase esponenziale che rappresenta la fase di massima amplificazione della sequenza di interesse e, infine, la fase di plateau, nella quale viene raggiunta la saturazione, per cui non è possibile avere un aumento dell’amplificato e di conseguenza della fluorescenza.
29
Il parametro utilizzato per avere una quantificazione dei livelli di DNA o di cDNA presenti nel campione in esame è il Ct, il quale è inversamente proporzionale alla quantità di templato iniziale.
Nella quantificazione relativa non viene misurata la concentrazione assoluta del templato, ma viene valutato l’aumento o la riduzione dell’espressione di un dato campione, paragonato a un controllo. I dati ottenuti sono normalizzati utilizzando un gene di riferimento costitutivamente espresso (gene housekeeping). Il metodo comunemente usato per la quantificazione relativa è il metodo del 2-ΔΔCt, dove:
ΔCt = Ct gene target – Ct gene housekeeping ΔΔCt = ΔCt gene target – ΔCt gene controllo
Tale metodo è valido quando l’efficienza di amplificazione è circa 2 ed è uguale in tutti i campioni. Quando questa situazione non è verificata, viene utilizzato il metodo di Pfaffl che tiene conto anche delle singole efficienze di amplificazione.
Ratio = (𝐸𝑡𝑎𝑟𝑔𝑒𝑡 )
𝛥𝐶𝑡 𝑡𝑎𝑟𝑔𝑒𝑡 (𝐶𝑇𝑅𝐿 −𝑡𝑟𝑎𝑡𝑡𝑎𝑡𝑜 )
(𝐸𝐻𝐾 )𝛥𝐶𝑡 𝐻𝐾 (𝐶𝑇𝑅𝐿 −𝑡𝑟𝑎 𝑡𝑡𝑎𝑡𝑜 )
Dove:
Etarget = efficienza di amplificazione del gene target
30
La real-time PCR è stata eseguita utilizzando Brilliant® II SYBR® Green (Stratagene, State HWY, USA), un mix che contiene:
Taq Polimerasi;
dNTP;
ioni Mg2+; tampone; SYBR® Green.
Il SYBR® Green è un colorante fluorescente che si lega esclusivamente al DNA a doppio filamento, mentre non si lega al DNA a singolo filamento
(Fig.9). Il segnale emesso dal SYBR® Green è rilevabile solo dopo un determinato numero di cicli PCR, quando cioè l’amplificazione è significativa.
31 1)
2)
3)
Fig.9 Meccanismo d’azione del colorante fluorescente SYBR® Green. 1) All’inizio del
processo di amplificazione, la miscela di reazione contiene DNA denaturato, primers e
la molecola fluorescente. 2) Dopo l’annealing dei primers, si legano poche molecole fluorescenti alla doppia elica. 3) Durante l’elongazione si verifica un aumento di fluorescenza che corrisponde all’ aumento del numero di copie dell’amplicone.
32
Per valutare l’aumento o la riduzione dell’espressione del gene della MMP-2 in seguito a trattamento con in composti in studio, abbiamo utilizzato il metodo della quantificazione relativa di Pfaffl, paragonando i livelli di mRNA delle MMP-2 estratto da cellule trattate rispetto ad un controllo. La normalizzazione è stata effettuata utilizzando come gene housekeeping la β-glucoronidasi (GUSB).
Le sequenze dei primers utilizzati per l’amplificazione sono le seguenti:
MMP-2 forward, 5’-CTGAAGGACACACTAAAGAAGATG-3’; MMP-2 reverse, 5’-GGAAGGCACGAGCAAAGG-3’;
GUSB forward, 5’-TGGTATAAGAAGTATCAGAAGCC-3’; GUSB reverse, 5’-GTATCTCTCTCGCAAAAGGAAC-3’.
L’analisi è stata effettuata in doppio e ciascun campione, contenente un volume finale di 25 µl, era costituito da:
cDNA 2 µl
Brillant® II SYBR® Green 12,5 µl MMP-2 for (8,28 µM) 0,91 µl MMP-2 rev (7,76 µM) 0,97 µl
H2O 8,62 µl
La real-time PCR, effettuata mediante Rotor-Gene 6 (Corbett Research, Sydney, Australia) ha previsto una fase iniziale a 95 °C per 15 min, seguita da 45 cicli costituiti ciascuno da:
Denaturazione a 94 °C per 15 secondi;
Annealing a 55 °C per 30 secondi;
Estensione a 72 °C per 30 secondi.
GUSB for (7,38 µM) 1,02 µl
GUSB rev (8,68 µM) 0,86 µl
o p
33
10.
Test ELISA
Il test ELISA (Enzyme-Linked Immunosorbent Assay) è un metodo di analisi immunologica che permette di analizzare particolari proteine nei fluidi biologici.
Per la determinazione della concentrazione di MMP-2 rilasciata dalle cellule nel loro sovranatante è stato utilizzato il RayBio® Human MMP-2 ELISA kit (RayBiotech Inc, Norcross, GA), con il quale è possibile determinare sia la forma pro che la forma attiva dell’enzima. Questo saggio utilizza un anticorpo specifico per la proteina umana MMP-2 legato sul fondo dei pozzetti di una piastra da 96 pozzetti. Aliquote degli standard e dei campioni trattati con MMPI vengono aggiunte nei pozzetti e le MMP-2 presenti si legano agli anticorpi immobilizzati sul fondo. In seguito a lavaggi dei pozzetti, effettuati per allontanare i componenti non legati, viene aggiunto un anticorpo anti MMP-2 umane legato a biotina. Dopo ulteriori lavaggi necessari per eliminare gli anticorpi biotinilati che non si sono legati, viene aggiunta streptavidina coniugata con perossidasi di rafano (HRP) che interagisce con la biotina. Dopo ulteriori lavaggi, viene aggiunta una soluzione di 3,3’,5,5’-tetrametilbenzidina (TMB), substrato dell’enzima, e l’intensità della colorazione che si sviluppa è direttamente proporzionale alla concentrazione di MMP-2 legata all’anticorpo. La soluzione Stop fa virare il colore da blu a giallo e l’intensità della colorazione viene letta a 450 nm utilizzando uno spettrofotometro (Wallac Victor 2, 1420 Multilabel Counter, Perkin-Elmer). Tramite una retta di taratura, costruita con i valori di assorbanza ottenuti dagli standards, è possibile risalire alla concentrazione di MMP-2 presente nei nostri campioni utilizzando il valore di assorbanza ottenuto.
Il valore minimo di concentrazione di MMP-2 misurabile con questo kit è pari a 3.5 ng/ml.
34 Preparazione dei reagenti:
Tutti i reagenti e i campioni sono stati tenuti a temperatura ambiente prima dell’utilizzo.
Nel kit sono presenti:
- Item A (piastra da 96 pozzetti rivestiti con anticorpi specifici per MMP-2 umane)
- Item E (tampone) è stato diluito 1:5 con H2O per soluzioni
- Item C (standard) è stato spinnato brevemente e poi sono stati aggiunti 400 µl di Item E 1x per preparare uno standard a concentrazione 2 µg/ml. Il tutto è stato mixato gentilmente per sciogliere la polvere. A partire da questa soluzione standard sono state effettuate diluizioni in serie (Fig.10)
Fig.10 Schema delle diluizioni in serie per la preparazione dello standard.
Dal manuale d’uso di RayBio®
Human MMP-2 ELISA Kit.
- Item B (soluzione di lavaggio concentrata 20x) è stato diluito in H2O per soluzioni ottenendo Item B 1x
- Item F (anticorpo anti MMP-2 legato a Biotina) è stato spinnato prima dell’uso ed in seguito sono stati aggiunti 100 µl di Item E 1x, ottenendo così Item F concentrato. Questa soluzione è stata diluita 80 volte con Item E 1x prima dell’uso.
35
- Item G (streptavidina con HRP) è stato diluito 22.000 volte con Item E
- Item H (substrato per l’enzima costituito da 3,3’,5,5’-tetrametilbenzidina in tampone)
- Item I (soluzione di stop costituita da acido solforico)
Procedura:
1. 100 µl di standards a diversa concentrazione, e 100 µl di campioni sono stati aggiunti in ciascun pozzetto della piastra che è stata poi lasciata in incubazione per 2,5 ore a temperatura ambiente in leggera agitazione
2. La soluzione è stata scartata e i pozzetti sono stati lavati 4 volte con Item B 1x (300 µl per pozzetto)
3. Nei pozzetti sono stati aggiunti 100 µl di Item F diluito e sono stati incubati 1 ora a temperatura ambiente in leggera agitazione
4. La soluzione è stata scartata e sono stati ripetuti i lavaggi come allo step 2
5. Sono stati aggiunti 100 µl di Item G diluito e il tutto è lasciato incubare per 45 minuti a temperatura ambiente e sotto leggera agitazione
6. La soluzione è stata scartata e sono stati ripetuti i lavaggi dello step 2
7. Sono stati aggiunti 100 µl di Item H in ciascun pozzetto al buio e sono stati incubati per 30 minuti a temperatura ambiente in leggera agitazione
8. Sono stati aggiunti 50 µl di Item I ed è stata immediatamente effettuata la lettura a 450 nm al Wallac Victor 2.
36
11. Analisi dei dati
Le analisi statistiche sono state eseguite tramite test ANOVA (post hoc test di Bonferroni) usando il programma di statistica GraphPad Prism
(GraphPad Software, versione 4.0; San Diego, CA). I risultati sono stati ritenuti significativi quando il valore p < 0.05. Infine, tutti i valori sono stati presentati come le medie ± errore standard della media (SEM), derivate da esperimenti indipendenti.