3.1 Studio del trascritto del gene Leafy
Cotyledon1-Like (HaL1L)
Lo studio del trascritto del gene Leafy Cotyledon1-like (HaL1L) è stata condotto tramite due diverse tecniche: RT-PCR relativa ed ibridazione in situ. La RT-PCR Relativa, con la quale è stato stimata la quantità di trascritto in diversi tipi di materiale vegetale, consiste nella co-amplificazione, nella stessa reazione di PCR, del cDNA di interesse e di un controllo interno (cDNA di un gene costitutivamente espresso), utilizzando due coppie di oligonucleotidi specifici, con Tm (temperature di appaiamento) analoghe. Questa tecnica è idonea ad evidenziare la presenza di trascritti rari o poco abbondanti come quelli dei fattori di trascrizione. I frammenti da amplificare, del cDNA d’interesse e del controllo interno, devono essere compatibili tra loro, ovvero non devono formarsi prodotti ibridi dovuti al legame tra i due amplificati, e nella co-amplificazione non devono essere prodotte bande aggiuntive rispetto a quelle attese. Affinché i dati ottenuti dalla RT-PCR relativa siano significativi, la reazione di PCR deve essere terminata quando i prodotti, sia del controllo interno che del cDNA di interesse, risultino evidenziabili e siano ancora all’interno della fase esponenziale. Per i nostri campioni, il numero dei cicli scelto è 26 e in alcuni casi 30. Il gene costitutivo utilizzato è quello della ß-actina, e la coppia di oligonucleotidi ACT3 e ACT5 (Tabella 2.2) permette l’amplificazione di un frammento di cDNA lungo 243 pb, distinguibile, ma non troppo diverso, dal frammento, di 175 pb, ottenuto con gli oligonucleotidi CHI-REL e CHI-TcDNA, per i cDNA di HaL1L (Tabella 2.2). Le quantità relative per ogni prodotto di PCR sono state quantificate mediante la scansione diretta del gel di Agarosio (2%) con il densitometro “UVP Image Store 5000” (Ultra Violet Product Ltd, Cambridge,
England) equipaggiato con l’UVP GelBase-GelBlot TM Windows Software. La presenza del trascrtto per L1L è stata localizzata, in sezioni longitudinali e/o trasversali di diversi tessuti presi in considerazione, con esperimenti d’ibridazione in
situ. L'ibridazione in situ (ISH) è una tecnica che consente di individuare trascritti
specifici, nelle loro localizzazioni, cellulari e/o tissutali. Il segnale d’espressione, dipende dal legame tra l’RNA messaggero del gene d’interesse e la sonda marcata. Questa ultima è costituita da una specifica sequenza ad RNA, sonda antisenso, complementare al trascritto da studiare in situ e marcata con metodo non radioattivo. L’eventuale presenza di falsi segnali positivi è valutata, in ciascun esperimento di ISH, con l’uso di una sonda senso costituita da una sequenza identica al trascritto del gene da studiare.
3.1.1 Studio del trascritto del gene HaL1L in embrioni
zigotici di Helianthus annuus
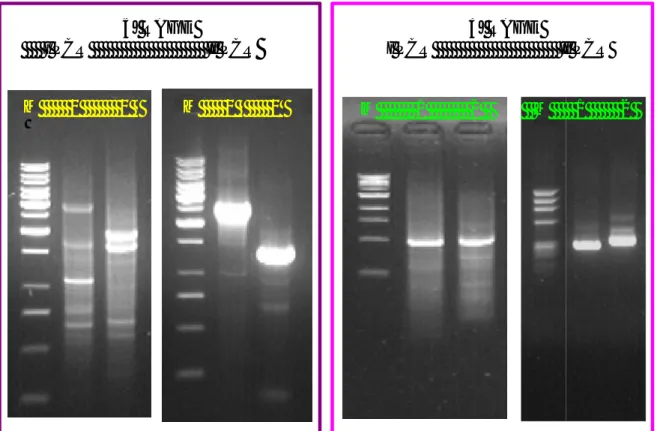
Inizialmente, il livello del trascritto per L1L è stata valutato, mediante RT-PCR Relativa, in embrioni zigotici poiché, in letteratura, il ruolo del gene L1L, isolato per la prima volta in Arabidopsis thaliana, è stato associato all’embriogenesi. L’analisi è stata condotta in embrioni di Helianthus annuus a quattro diversi stadi di sviluppo: 5, 10, 21 e 28 giorni dall’impollinazione (GDI). I risultati ottenuti, riportati in figura 3.1, evidenziano che è presente, a 26 cicli di PCR, in tutti gli stadi di sviluppo considerati, sebbene a livelli diversi.
L’analisi statistica ha evidenziato che la quantità del trascritto, molto elevato negli embrioni di 5 e 10 giorni, decresce in maniera consistente negli stadi di sviluppo embrionale più avanzati presi in considerazione (Figura 3.1 A). Il livello significativamente più elevato è stato rilevato negli embrioni zigoti allo stadio di sviluppo di 5 giorni dall’impollinazione, mentre non si riscontra una differenza significativa tra gli embrioni di 21 e 28 giorni.
In tessuti ovarici di girasole non si osserva la presenza di trascritti di HaL1L mentre, sebbene a livelli molto bassi, trascritti del gene sono accumulati in ovuli non fecondati (Figura 3.1 B).
A
C
Fig. 3.1. Livello di espressione relativa di HaL1L stimato mediante RT-PCR.
(A) L’amplificazione dei trascritti per HaL1L e ß-actina è stata condotta usando 5 µg di RNA totale estratto da embrioni zigotici a stadi diversi di sviluppo (giorni dall’impollinazione: GDI). (B) Ovuli (O) e ovari (OV) non fertilizzati un giorno prima dell’apertura dei fiori.
(C) Il livello di espressione del trascritto HaL1L, normalizzato rispetto al trascritto della β-actina è stimato misurando la fluorescenza dei prodotti di PCR separati in gel di agarosio (2%) colorato con bromuro di etidio. I valori in B seguiti dalla stessa lettera non sono significativamente differenti (P = 0,05) in accordo al test di Tukey.
Embrioni zigotici (GDI)
5 g 10 g 21 g 28 g
β-actina (243 pb) HaL1L (175 pb)O OV
β-actina (243 pb) HaL1L (175 pb)26 cicli
30 cicli
a a b c 0 20 40 60 80 100 5 d 10 d 21 d 28 dStadio di embrioni zigotici (GDI)
Trascritti di HaL1L (%)
5g 10g 21g 28g
Allo scopo di determinare la localizzazione tessuto specifico di HaL1L sono state effettuate ibridazioni in situ su sezioni longitudinali di embrioni di Helianthus annus a diversi stadi di sviluppo. I materiali utilizzati sono stati:
§ embrioni immaturi in stadi precoci di sviluppo (Fig. 3.2 e 3.3); § embrioni a 5 giorni dall’impollinazione (Fig. 3.4 e 3.5);
§ embrioni a 10 giorni dall’impollinazione (Fig. 3.6 e 3.7).
La localizzazione del trascritto HaL1L è visualizzato sostanzialmente a livello di dell’embrione, dell’endosperma, del sospensore, del tegumento interno e delle cellule del tappeto.
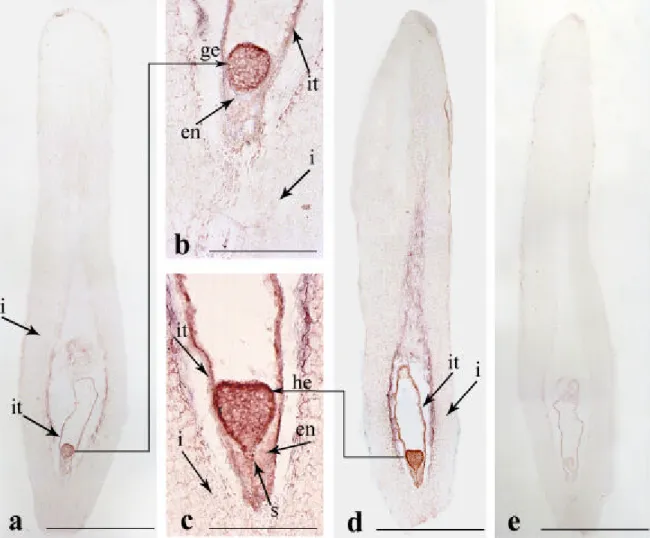
A stadi precoci come quello globulare e a cuore l’mRNA è stato prevalentemente ritrovato nelle cellule dei tessuti esterni dell’embrione (Fig. 3.2 b,c) anche se, nello stadio a cuore, vi è un evidente segnale rilevabile nel sospensore e nelle cellule del tappeto (Fig. 3.2 c,d). L’esame a maggiore ingrandimento della sezione longitudinale del seme di circa 5 giorni dalla data di impollinazione evidenzia il segnale di ibridazione a livello di specifici domini cellulari, le cellule del tappeto e le cellule dell’endosperma (Fig. 3.4 e 3.5).
A 7 giorni dall’impollinazione, i trascritti HaL1L sono distribuiti attraverso l’embrione (Fig. 3.3 a), ma il segnale è più evidente nelle cellule apicali del tegumento interno e delle cellule del tappeto.
Un’analoga localizzazione cellulare del segnale, indicativo dell’espressione di
HaL1L, è stata individuata anche nelle sezioni longitudinali del seme dopo circa 10
giorni dalla data di impollinazione (Fig. 3.6). Tuttavia, in questo ultimo campione, è evidente un segnale marcato d’espressione nella regione calazale (finestra calazale), che rimane attiva, con la funzione di veicolare nutrimenti dallo sporofito sia al gametofito femminile che all’embrione (Fig. 3.7 c, d) (Forino et al., 2000).
Fig. 3.2. Ibridazione in situ di HaL1L mRNA in ovuli di Helianthus annuus a differenti stadi di sviluppo. Tutte le sezioni sono state incubate per tre giorni.
(a-b) Sezioni longitudinali di ovuli di girasole con embrione zigotico allo stadio globulare. (c-d) Sezioni longitudinali di ovuli di girasole con embrione allo stadio a cuore.
(e) Controllo negativo ibridato con sonda senso RNA HaL1L. Scala delle barre = 1 mm in a, d ed e; 250 µm in b e c.
ge, embrione globulare; he, embrione a cuore; en, endosperma; i, tegumento; it, cellule del tappeto; s, sospensore.
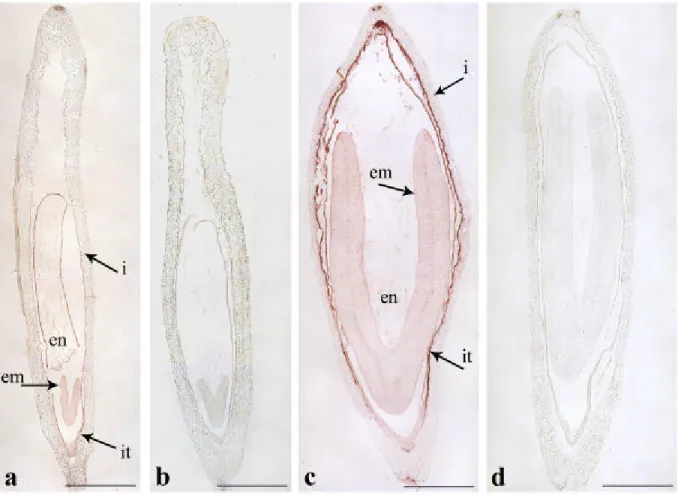
Fig. 3.3 ibridazione in situ di HaL1L mRNA in ovari di Helianthus annuus a differenti stadi di sviluppo.
(a-b) Sezioni longitudinali di ovari di girasole a 5 giorni dopo l’impollinazione (GDI) con embrione zigotico allo stadio cotiledonare.
(c-d) Sezioni longitudinali di ovari di girasole a 10 giorni dopo l’impollinazione (GDI) con embrione zigotico allo stadio cotiledonare avanzato.
Da notare che i trascritti sono accumulati nell’embrione, nell’endosperma nelle cellule del tegumento e del tappeto.
I controlli negativi utilizzando sonde RNA HaL1L sono mostrate in b e d, l’ibridazione RNA antisenso HaL1L è mostrata in a e c.
Tutte le sezioni sono state incubate per 3 giorni. Scala delle barre = 1 mm.
Fig. 3.4 a, b, c. Sezioni longitudinali di 3 diversi embrioni dopo 5 giorni dalla impollinazione (5 GDI) ibridate con la sonda HaL1L antisenso. In d è riportata l’ibridazione con la sonda HaL1L senso. Scala delle barre: 500 µM.
Le frecce indicano: (em) embrione, (ed) endosperma, (it) cellule del tappeto. em en it en it em em it en
a
d
c
b
Fig. 3.5. a. Sezione longitudinale di un seme con embrione di circa 5 giorni dalla data di impollinazione (5 GDI) ibridata con sonda HaL1L antisenso. (em) embrione zigotico allo stadio di torpedine avanzata. (en) endosperma.
Nel riquadro laterale b, particolare a maggiore ingrandimento per valutare il segnale di ibridazione a livello dei vari domini cellulari: (it) cellule del tappeto; (en) endosperma, (i) tegumento. 60µm
it
en
b
1 mm
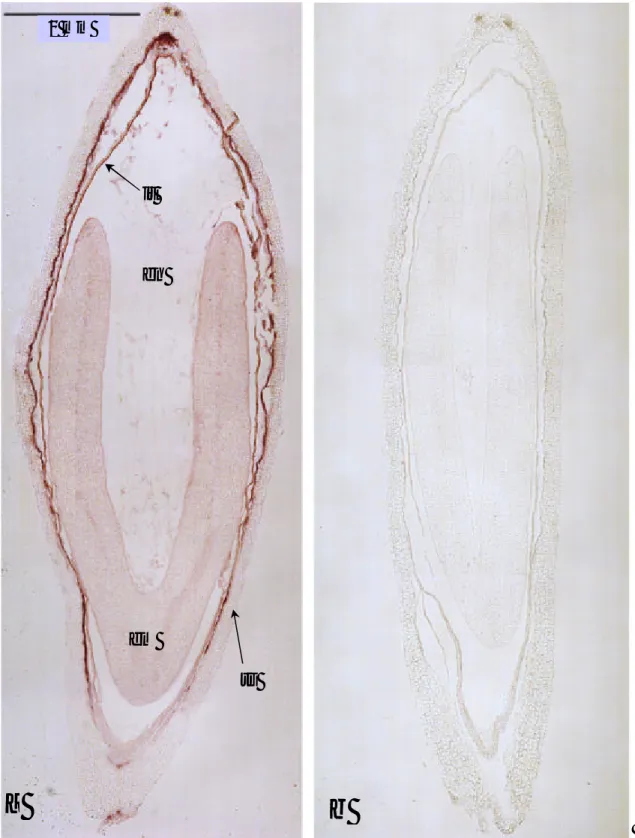
Fig. 3.6. a. Sezione longitudinale di un seme con embrione di circa 10 giorni dalla data di impollinazione (10 GDI) ibridata con sonda HaL1L antisenso. (em) Embrione zigotico allo stadio cotiledonare; (en) endosperma; (ts) tegumenti del seme; (it) cellule del tappeto.
In b, analoga sezione ibridata con sonda HaL1L senso. en it 1 mm em ts
a
b
a
em i ite
b
em i itf
g
d
c
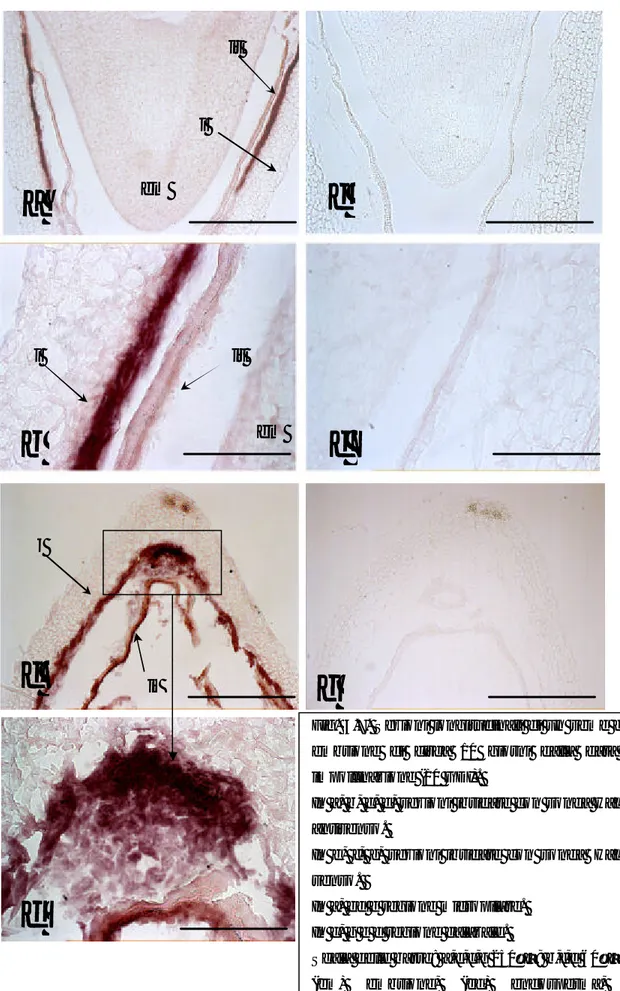
it iFig. 3.7. Sezioni longitudinali di un seme con embrione di circa 10 giorni dalla data di impollinazione (10 GDI).
In a, b, c, d, sezioni ibridate con sonda HaL1L antisenso.
In e, f, g, sezioni ibridate con sonda HaL1L senso.
In a, ed e regione micropilare. In c, g e d regione calazale.
Scala delle barre: a,e,c,g 250µM; b,f,d 60µM (em) embrione, (ed) endosperma, (i) tegumento.
3.1.2 Studio del trascritto del gene HaL1L in diversi
organi di Helianthus annuus
Dopo aver accertato la localizzazione del trascritto di HaL1L a livello dell’embrione zigotico e del seme, era interessante esaminare la sua localizzazione negli stadi post-embrionali di sviluppo. In particolare, l’RNA, utilizzato per condurre l’RT-PCR, è stato estratto dai seguenti organi di H. annuus: foglie cotiledonari di 10 giorni, foglie giovani (2 cm di lunghezza), foglie espanse (15 cm di lunghezza), fusto di piante allo stadio di bottone fiorale, radici di piante allo stadio di bottone fiorale e infiorescenze immature (capolini a 15 giorni dall’antesi). Sulla base di quanto accertato negli embrioni zigotici è stato deciso di condurre una prima serie di RT-PCR relativa utilizzando 26 cicli di amplificazione, ma con tali condizioni non si è evidenziata l’amplificazione del frammento di HaL1L in nessuno degli organi analizzati (dato non mostrato). Aumentando gradualmente il numero dei cicli è stata accertata la presenza del messaggero del gene HaL1L nei cotiledoni, nelle foglie espanse e nelle infiorescenze immature (Fig. 3.8). Tuttavia, come appare evidente dalla Figura 3.8, la banda relativa al trascritto HaL1L è risultata sempre molto flebile rispetto a quella della ß-actina. Per questi campioni non è stato possibile misurare il livello d’espressione, e quindi effettuare l’analisi statistica, anche perché la banda pertinente alla ß-actina non è più all’interno della fase esponenziale. Visto il basso livello di espressione accertato con l’RT-PCR, si è ritenuto poco opportuno effettuare, sui medesimi organi vegetali, gli esperimenti di ibridazione in situ. I risultati ottenuti lasciano, infatti, ipotizzare che i trascritti siano presenti in questi organi in quantità inferiori ai limiti di risoluzione permessi dalla tecnica ISH.
Fig. 3.8. Amplificazione dei trascritti per HaL1L e ß-actina, mediante RT-PCR, condotta usando 5 µg di RNA totale estratto da embrioni zigotici al decimo giorno dall’impolliazione (GDI) e dai seguenti tipi di organi: cotiledoni, foglie giovani, foglie espanse, fusto, radici ed infiorescenze immature. I tra scritti per HaL1L e ß-actina sono separati in gel di agarosio (2%) colorato con etidio bromuro. Le amplificazioni sono effettuate con diversi numeri di cicli in un intervallo compreso tra 26 e 45.
ORGANI
EZ M M M M M
COTILED. FOGLIE G . FOGLIE E. FUSTO RADICI INFIORES.
NUMERO CICLI
26 34 37 40 45 34 37 40 45 34 37 40 45 34 37 40 45 34 37 40 45 34 37 40 45 β-actina
3.2 Isolamento del DNA del gene HaL1L
3.2.1 Genome Walking
Il gene LEAFY COTYLEDON1-LIKE (HaL1L) è stato interamente isolato e sequenziato nella specie Helianthus annuus per mezzo del Walking on the Genome che si avvale della tecnica della RAGE (Rapid Amplification Genomic End).
Le informazioni derivate dalla mappa di restrizione della sequenza del cDNA precedentemente ottenuto sono state utilizzate per condurre analisi preliminari tramite ibridazioni su Southern Blot. Come prima fase è necessario infatti individuare opportuni enzimi di restrizione che taglino all’esterno della sequenza di DNA nota e che generino frammenti idonei, per dimensione, ad essere amplificati tramite PCR. I prodotti della digestione di una serie di enzimi di restrizione sono stati quindi analizzati mediante ibridazione con due differenti sonde, SONDA1 e SONDA3, capaci di evidenziare i frammenti contenenti rispettivamente le estremità 5’ e 3’ della regione codificante l’HaL1L. Nella Figura 3.9A è mostrata l’elettroforesi su gel di agarosio di una serie di digestioni con endonucleasi e nella Figura 3.9B le rispettive ibridazioni con le sonde suddette.
Sono risultati idonei gli enzimi DraI e SspI per l’analisi dell’estremità 5’ che danno rispettivamente frammenti di 2400 pb e 1800 pb, mentre per l’analisi dell’estremità 3’ sono stati utilizzati gli enzimi DraI, SspI e HpaI che danno rispettivamente frammenti di 1300, 1000, e 3000 pb.
Fig. 3.9. (A) Gel di agarosio mostrante i tagli effettuati sul DNA genomico dagli enzimi di restrizione per la selezione dei frammenti adatti alla RAGE. M = Marker di pesi molecolari. (B) Sviluppo della lastra del Southern blot. Ibridazione con la SONDA3 per la 3’RAGE; in rosso sono indicati i frammenti generati da DraI: 1300 pb (per 3’RAGE); in azzurro sono indicati i frammenti generati da SspI: 1000 pb (per 3’RAGE).
Ibridazione con la SONDA1 per la 5’RAGE. In rosso sono indicati i frammenti generati da DraI: 2400 pb (5’RAGE) e 1300 pb (3’RAGE); in azzurro sono indicati i frammenti generati da SspI: 1800 pb (5’RAGE) e 1000 pb (3’RAGE).
M PvuII DraI EcoRV StuI SspI Sty + M PvuII
PvuII DraI EcoRV StuI SspI StyI+ PvuII StyI+
PvuII PvuII DraI EcoRV StuI SspI
SONDA1 SONDA3 1300 pb 1300 pb 1000 pb 2400 pb 1800 pb 1000 pb
A B
Fig. 3.10. Elettroforesi su gel di agarosio (1,5%) colorato con Bromuro di Etidio. In giallo: Corsia 1, DNA amplificato nella reazione di 5’RAGE tagliato con enzima DraI; Corsia 2, DNA amplificato nella reazione di 5’RAGE tagliato con enzima SspI;
M = Marker di pesi molecolari.
In verde: Corsia 1, DNA amplificato nella reazione di 3’RAGE tagliato con enzima DraI; Corsia 2, DNA amplificato nella reazione di 3’RAGE tagliato con enzima SspI;
M = Marker di pesi molecolari.
Sulla sequenza del cDNA sono stati anche scelti gli oligonucleotidi specifici, In7, 1RAGE CDS, 3RAGE3 e 3RAGE2, mostrati nella Tabella 2.2 e utilizzati in coppia con i primer Adaptor primer 1 e Adaptor primer 2 nelle due successive PCR dove lo stampo iniziale è rappresentato dal DNA genomico opportunamente digerito e legato con gli adattatori secondo la procedura descritta in Materiali e Metodi. Nella seconda PCR sono stati ottenuti amplificati delle dimensioni attese conformi alle indicazioni
5’ RAGE
I PCR II PCR
3’ RAGE
I PCR II PCR
derivate dall’ibridazione su Southern blot, che sono stati eluiti da gel, clonati e sequenziati (Fig. 3.10).
La sequenza completa ottenuta con la tecnica della 5’-3’RAGE è mostrata nella Fig. 3.11. Essa conferma la sequenza del cDNA per l’HaL1L acquisita precedentemente e indica che sul DNA genomico la regione di 645 pb codificante il peptide è interrotta da un introne di 864 pb che rispetta la regola GT/AG nei siti di splicing e che è posizionato dopo il nucleotide +102, quando la numerazione dei nucleotidi della sequenza genomica inizia con il putativo sito di inizio della trascrizione (TSS).
L’allineamento, ottenuto con il programma ClustalW, della sequenza genomica di
H. annuus con la corrispondente regione del cromosoma V di Arabidopsis thaliana,
evidenzia, inoltre, che la sequenza ottenuta per l’H. annuus comprende una porzione dei geni a monte e a valle del gene HaL1L e risulta conservata tra specie diverse la posizione relativa dei geni collocati nella regione esaminata. Analisi effettuate con i programmi Fasta e Blast mostrano infatti che HaL1L di H. annuus è preceduto da una sequenza omologa a quella di un fattore di trascrizione legante il DNA, presente al 5’ dell’AtL1L di Arabidopsis thaliana (codice di accesso: At5G47660.1), mentre al 3’ del gene in entrambe le specie è posizionata una RNA binding/tRNA metiltransferasi, contenente un dominio tRNA (guanina-N1-)-metiltransferasi (codice di accesso per
A. thaliana: AT5G47680.1). Per quanto riguarda invece le lunghezze delle regioni
intergeniche solo il gene a valle è posizionato ad una distanza paragonabile: 353 pb in H. annuus e 354 pb in A. thaliana; viceversa in H. annuus il gene a monte di L1L è ad una distanza maggiore, 1601 pb, che in A. thaliana, 424 pb. La lunghezza delle regioni intergeniche sono calcolate in riferimento ai codoni di inizio e fine delle regioni codificanti i relativi peptidi, non essendo note per l’A. thaliana le lunghezze dei rispettivi trascritti.
TGAGGAAACCGGCAAATTGCAAGAACCAGTCGAGGAGTCCGTAAACAAGAAAAAGAGGAAGAGAAAATCAAGGAGGAAGATGGAGTTGTTTGTCGAAAGCATGATGAGAACAGT AATCCAAAAACAGGAAGAAATGCATAAACAGTTGATCGAGATGTTGGAACAGAAAGAAAAAGAAAGCATCATGCGCGAACAAGCATGGAAGAAACAAGAGATCGCTAGAGCAAA GAGGGACGAACACGCTAGAAAACAACACATATCGCAAAGCCTCGCTCTCATTTCTTTCATCAAAAACACATTGGGCCAAGAGATCCAAATCCCAGATTCTCTCGACATACAAGT TGATAATCAGAATCATCAAAGTCAAGAAAAGTCAACGTATGATGATATAGAAATGCATAACGTTGACGAATATGAGACGGACACGAATACCAAAAGATGGCCGAAATCTGAAGT GCAAGCGCTTATAACAGTTAGAGTTGCTCTGAATCAGAAGTTTAACGGGAAGATACCGAAAGCGTCTATTTGGGACGAAGTTGCTGCTGGTTTATCCAGTATGGGTTATAACCG GACCCCGAAAAAGTGCAAAGAGAAATGGGAAAACATCAACAAGTATTACAGAAGAACGATGGAGAAAGGGAAAGGATAGTAAATCATGCGCGTATTTCAGTGAGTTGGAAATGT TGCATAAAACCGGCTTTATCTCGTCAACTTCGCATGATGAAGATCACAGCATCAAGGTTGTTTCGTCAAATTCTCATGACGAGGATCAAAGCATTAAGGTTGTTTCATCAAACT CTCATGATGAGGGTCAAAACATGAAGGTTATTTCATCGGACTCTCTTGATGAGGATCAAAGCATGAAGGTTATGTCATCAAACTCTCTTGATGAGGATCAAAGGATCAAGGTCT TGCACTAGAATTCAAATATTGAGACGGGTTTGCGATTATGTTCTTATGACTGTATCAAATTTTGTCCAAGTGTAGACTTGATGAACTTATTTTGTTGTATCGTTGTCATAGAAG TAGAAGAAATGTTGTTCAATTTGAAATCTCATGGCAGATTTCCTTTAGTTTTAATAGACTTTTGACTTTGTGAAATTAAATGCTCAAAACACCATAACGTAATGGTAAAGTTAT AGTTTTTCAACTGACCCACTTCTTTATGTCTCTTGCTATTCTGTTTGGCATTAAAAAATCCAATGTCCATCAACTTAATTTTGATATACATTTGTCTTTGGACATGTTTAAGAT ACATGTAGATATAAAATTATTTTTCAAAATGTCAAGTTGCCTCTTAAATAGTTAAAATGGACAACCCTTAATTAAAATGACACTCTGCGAAAAACGTTGACACATGAAATCCCA CATTAACAACATCGTCTGTATGCCTATGATTATAGGCTAGTGACATCTTGGGGGTGAGATAAGACTTTTGGACCACTAGGTCCTGGGTTCAAATGGGTTTTTCCCAGATTTATT GAGTTTCCTCCTGAATTAGTGTATAGCCGTTATAGCCTAGTGGAGATGGATATGATCGGGTGGTTCCGCTGGTGGCACGATGATACTCCAGTGGTCCGTCAGTGATCCTAACTT GCCGTTCAAAAAAAACATCGTCTGTATATATAGCCTCTGCACATGTAACAGTTGCGCCACTAACCAATGGTTGTCGTTGACACGTTTGATACGCGATATTAAAAAATGAGTTGA GAGATTATTTTACACAAATCAAGGAAATATGTGATAATTTTTAATGGTATAATTAACCTTGTCTTAAAATGGATTTTCATTATTATGAAATAATTTAGACATTCTTGTGATATA TGCATCATCCAAGGTCCTAAAGACATTTTCTTAATAAAAACAAAGATTAAATCATAAAAAAAACATTAAATTATGATTAAATCTGCCAACTAACTATTGATTGGATTAAGAAAT ATAGAACGACACAGGAGGAGGCATGGACCATGTAGATCCAAAAGTCATAAATCAAAGTAATTAAAGTGCATGCATTAGTCAAAGAGGGTGGTGGTCAAATCAAATGCACCTTAA CCAAAAACACTCAATGCTAACCAACTTTGGTAGCTCCGGCGAATACCCGGTTCTTGTTCACTGAGCCACCATGCCACGTGTCCTCACCACCCCACCGGCCCACCACCATCAGCA -38 GCAGCTCCTTATATTATCTCCTCCTCACTTTGTTTTCAATTTCTAGAGAGAGACAATTCCCTCCCTTAGAGAGAGAAACATATGGAACGTGGAGGAGGTTTCCATGGCTACCAC +77
AGGCTCCCCATCCACCCTACATCTGGTACACATACACATATATACTCATACACATTCATATATATATATAATGTTTCTTTAACTTTATGGGATAGTTAACCTCTTTCTGCATCT GCATTGGCCTTTTTATTCTTCATCTGCAATATCATGTTTTCTTGCTATGTTTTTCTTTTATATATATATATATGTGTGTGTATATATATAATATAGATACATATATATACACAT CGGTGAACGAAAGGGGTCAAGAGGGTTTGCTGATCGCTGACCACTAGGCGTCTTCGTTATTCTAAGAAGTTCTGGTTCCGCCACTGGATGTATACCATACACGTACGTATAGTT ATTATGTTTGGTGTTGGAACAAGATCAAGAAATTTTTTTTTTACATTTTTAAAAGATTCTTTAATTTTTACTGTTTTGAGGATTCATAACTACTTTTACAGGCAATCTTCTGAC ACATAGACTCAGGCTAAGCTATTATTGATTGTTTTTAAGGTATCTTATGTTGATTTGTAAGCAGTTTTAACTTGAAACAACAAGATTTCTTGGAGTTTTAAGTGTGATTGATTG TATACAACAATGGTGGTCTGTTTGATCTGATTTAGGTTGTTGAATCTGAGTGAGATGTGTTAGTGACAGTGTGTGTTCGAATGTGTGTGGAAGGAGGACAAATATTGTAGAACA AGTGGCATGCATGCAAGAATATTACTCTCTTGTTTTGTCCCCACGAATCTGTCATTTTTGCACACTTCACTGGTTTTTGTTTTCACCATTTAACTTTCTTTTTTTAATTATTAC TACAATGAATATATGTATATATGATGTCATTAATATGATGTTATATATTCTTTTTAGTTTTATAATTTTTTTTTTATTTTTTTGATCTTAGGGATGCAACAGCCGGATATGAAG CAGAAGCAACCAGACACAACCAACACCACATCTACAGAAGACAATGAGTGCATTGTTAGAGAGCAAGACCGCTTTATGCCGATAGCAAACGTGATCAGGGTCATGCGAAAGATC TTGCCACCTCACGCGAAGATCTCTGACGACGCAAAAGAAACGATCCAAGAATGTGTTTCCGAGTACATTAGTTTTGTGACAGGTGAAGCGAATGACCGTTGCCAACGCGAGCAG AGAAAGACTATCACAGCTGAAGATGTGTTGTGGGCTATGAGCAAACTCGGGTTTGATGACTATATCGAACCGTTGACTGTGTATCTTCATCGCTACAGGGAGTTTGATGGGGGT GAGCGTGGGTCGATAAGGGGTGAGCCGCTTGTCAAGAGGGCTGCGGCTACTGCTGACCCTGGTCCGTTTGGTATGGGTCCGTTTGTGCCTGGTTCTCACATGGGTCATCATAAT GGGTTCTTTGGTCCTGCTAGCATTGCTGGTTTCTTTAAGGACCCGTCGAGTGCGGCTGGCCAGTCTGGGCCTGCTGGTTTCGAGCCGTATGCTCAGTGTAAAGACTAACTGGTT TCGATTTTCGAGCCGTGTGTTGGTTGGTTTGTTTTTTTTTCTTGTTTGTTTGTCTTCTTGGTCTTGGAGCAACAAATTACATTGGGTCAAACTTTGGCAAGGCATGTAAAAACT GACACTCTCCATCTACTGGTAGTAATCTGCTTTTCTTCCTAACTTTTATTTTATGAATTTGGAGTGTTTAAGATTTAACAGTTCCGTTATTATTAGTATTTTTTTAACATTTAA AAATATCAATTAATGATTAAAAATGTTTAAATAATATTTTCCTAACATTTAACCCGGTATTATTGGTGTTTTTTGAAACGATGCTTGCTTTCTCTTCCAAAATTTGTGCCATCG CCCTAGCCTGGATTTGTCGAGAAAGACAAAATGAATGGCTAGTTACTAACCTACTCGACGCGGGAAGGGATTTACAACGTCTCTGATCTGAAGAACTACAGCTTTTTGTATTCA
CA costituisce il putativo sito di inzio della trascrizione (TSS) di HaL1L, dove A è il primo nucleotide trascritto. La numerazione delle basi dell’intera sequenza mostrata, inizia da questo dimero: -1/+1. In arancio sono le regioni 5’ e 3’ UTR dell’mRNA. In violetto è indicato il CDS di HaL1L. ATG e TAA costituiscono rispettivamente i codoni di inizio e fine della traduzione. In azzurro e corsivo è indicato l’introne. GT/AG sono le estremità 5’ e 3’ dell’introne. In verde chiaro è indicato una porzione di gene simile a AT5G47680.1 di A. thaliana con orientamento inverso rispetto a HaL1L. CTA: codone di fine della traduzione.
3.3 Identificazione del promotore
3.3.1 Elementi caratterizzanti il promotore
La regione del promotore del gene HaL1L è stata individuata manualmente, in accordo con le sequenze consenso caratterizzanti, nelle piante, le porzioni del genoma a monte di regioni codificanti e tramite i programmi TSSP e CpGfinder, disponibili al sito Softberry (http://www.softberry.com), mostrata in Figura 3.12.
TAGCCTAGTGGAGATGGATATGATCGGGTGGTTCCGCTGGTGGCACGATGATACTCCAGT -870 GGTCCGTCAGTGATCCTAACTTGCCGTTCAAAAAGAACATCGTCTGTATATATAGCCTCT -780 GCACATGTAACAGTTGCGCCACTAACCAATGGTTGTCGTTGACACGTTTGATACGCGATA -690 TTAAAAAATGAGTTGAGAGATTATTTTACACAAATCAAGGAAATATGTGATAATTTTTAA -600 TGGTATAATTAACCTTGTCTTAAAATGGATTTTCATTATTATGAAATAATTTAGACATTC -510 TTGTGATATATGCATCATCCAAGGTCCTAAAGACATTTTCTTAATAAAAACAAAGATTAA -420 ATCATAAAAAAAACATTAAATTATGATTAAATCTGCCAACTAACTATTGATTGGATTAAG -330 AAATATAGAACGACACAGGAGGAGGCATGGACCATGTAGATCCAAAAGTCATAAATCAAA -240 GTAATTAAAGTGCATGCATTAGTCAAAGAGGGTGGTGGTCAAATCAAATGCACCTTAACC -150 AAAAACACTCAATGCTAACCAACTTTGGTAGCTCCGGCGAATACCCGGTTCTTGTTCACT -90 GAGCCACCATGCCACGTGTCCTCACCACCCCACCGGCCCACCACCATCAGCAGCAGCTCC -30 TTATATTATCTCCTCCTCACTTTGTTTTCAATTTCTAGAGAGAGACAATTCCCTCCCTTA +31 GAGAGAGAAACATATGGAACGTGGAGGAGGTTTCCATGGCTACCACAGGCTCCCCATCCA +91
CCCTACATCTGG +103
Fig. 3.12. Sequenza dell’estremità 5’ del gene HaL1L contenente il promotore prossimale. TTATAT: TATA Box;
CC: nelle piante l’elemento CC è posizionato immediatamente a monte del TATA box; AGCAG,GAGAGAGA: enhancer della trascrizione;
CA: putativo sito di inizio della trascrizione (TSS); A è il primo nucleotide trascritto; la numerazione delle basi della sequenza inizia da questo dimero: -1/+1;
ATTGG: CAAT-box nell’ orientamento revertito; ATG: codone di inizio della traduzione ;
In azzurro e in giallo sono evidenziate zone ricche in GC, i cara tteri in arancio indicano il 5’UTR presente sul cDNA.
Il promotore di HaL1L è risultato essere di tipo ‘TATA’ poiché a -29 pb dal codone di inizio della trascrizione è presente la sequenza TTATA, nel cui intorno sono localizzati motivi tipici della regione del promotore nelle piante. Sono infatti riconoscibili due residui di citosina immediatamente adiacenti al 5’ del TATA box ed il motivo Y-Patch (CTCCTC) presente in posizione -20 in H. annuus. Inoltre, essendo perfettamente rispettata la YR-rule (YYAN(T/A)YY, dove: Y=C\T; R = A\G; N = qualsiasi base) il putativo sito di inizio della trascrizione (TSS) è individuabile nel dimero CA la cui collocazione sposta di cinque nucleoditi verso il 5’ la reale lunghezza del 5’UTR del cDNA. La numerazione della sequenza di DNA ottenuta inizia da questo dimero (-1/+1), dove il residuo di adenina rappresenta il primo nucleotide trascritto. Altri elementi tipici del promotore sono riconoscibili nella sequenza CCAAT, che è presente nell’orientamento revertito a partire dal nucleotide 280, e nelle due zone ricche in CG posizionate rispettivamente da 700 a 500 e da -153 a -52.
3.3.2 Cis-acting sequence
In aggiunta all’analisi della regione del promotore prossimale descritta sopra, è stata eseguita una ricerca dei possibili siti di legame per trans-acting factor. A tale proposito le sequenze intergeniche a monte e a valle del gene, oltre alla sequenza dell’introne, sono state confrontate con diversi database presenti in rete. In particolare il software MatInspector (Cartharius et al., 2005), che utilizza il database PLACE per cis-element delle piante, permette di visualizzare graficamente le sequenze di interesse. I grafici ottenuti (Figura 3.13) sono stato rielaborati tenendo conto delle caratteristiche intrinseche del gene, evidenziando particolari fattori di trascrizione associabili.
Nel capitolo dedicato alla discussione dei risultati saranno presi in esame i siti di legame per fattori di trascrizione che sembrano essere più direttamente coinvolti nei processi di embriogenesi.
Posizione Nome
da - a
Strand Sequenza MYCATERD: sequenza MYC necessaria per l’espressione di erd1 (early responsive to dehydration) di Arabidopsis; la proteina NAC lega in modo
specifico il motivo CATGTG. 2 - 8 (-) CATGTGC
MYCATRD2: sito di legame MYC (rd22BP1) nel gene rd22 coinvolto nella risposta alla disidratazione di Arabidopsis thaliana; indotto da ABA;
localizzato a circa 200 pb a monte del gene. 3 - 9 (+) CACATGT
MYBATRD2: sito di legame MYB (ATMYB2) presente nel gene rd22 coinvolto nella risposta alla disidratazione di Arabidopsis thaliana, localizzato a
circa 141 pb a monte del gene. 23 - 29 (+) CTAACCA
MYB1AT: sito di riconoscimento MYB ritrovato nei promotori del gene rd22 in altri geni di Arabidopsis coinvolti nella risposta della pianta alla
disidratazione. 24 - 29 (+) TAACCA
WBOXATNPR1: "W-BOX" ritrovato nel promotore del gene NPR1 di Arabidopsis thaliana; localizzato in tandem tra +70 e +79; sono sequenze
riconosciuti specificatamente dall’acido salicilico; sito di legame delle proteine WRKY. 39 - 53 (+)
GTTGACA CGTTTG CAREOSREP1: CAREs (CAACTC regulatory elements) trovato nella regione promotrice di una cisteina proteinasi (REP-1) in riso. 71 - 76 (-) CAACTC
-300ELEMENT: presente a monte del promotore dei geni B-ordeina alfa-gliadina e gamma-gliadina in orzo e in geni coinvolti nella sintesi della
glutenine a basso peso molecolare di grano. 83 - 91 (-)
TGTAAAA TA WUSATA: sequenza target di WUS presente solitamente nell’introne del gene AGAMOUS di Arabidopsis; (Lohmann et al., Cell 105:793-803 (2001). 118 - 124 (+) TTAATGG CCAATBOX1: sequenza comune ritrovata nelle regioni 5’ non codificanti, di geni eucariotici. 291 - 295 (-) CCAAT
DPBF-CORE-DCDC3: classe di fattori di trascrizione bZIP, DPBF-1 e 2 (Dc3 promoter-binding factor-1 e 2) binding core sequence; trovati nel promotore del gene Dc3 di carota. Dc3 ha un’espressione embrione-specifica, e può essere anche indotto dall’ABA. Il gene di risposta all’acido
abscissico in Arabidopsis ABI5 codifica un fattore di trascrizione bZIP; mutanti difettivi abi5 mostrano difetti pleiotropici in risposta all’ABA. ABI5 314 - 320 (+)
ACACAG G DPBFcoreDc-Dc3 CCAAT box DPBFcoreDc-Dc3 WboxHV-iso1 WB-boxPC-wrky1 ELREcorePC-rp1 MYB 1 CTRMCAMV35S Wbox-npr1 -300ELEMENT CARE s MYB 1 WUS-Ag ARF MYC-rd2 MYB-rd2 MYB-rd2
MYC-erd CATATG-GM-SAUR
WB-BOX PC-WRKY1: “WB BOX" trovata in prezzemolo nei promotori del gene WRKY1; risulta anche conservato nei promotori del gene WRKY3; richiesto per la risposta agli elicitori; WB BOX e WC BOX (S000142) costituiscono un palindormo; il fattore di trascrizione WRKY gioca un ruolo importante nella regolazione dei geni early defence-response.
374 - 388 (-) TTTGACT AATGCA ELRE-CORE-PCRP1-ElRE (Elicitor Responsive Element) ritrovati in prezzemolo: geni PR1 con elemento sequenza di consenso W1 e W2; i Box W1
e W2 sono rispettivamente siti di legame di WRKY1 e WRKY2; "WA box"; uno dei box W trovato in prezzemolo. Richiesti per la risposta agli elicitori. 390 - 404 (-) TTTGACC ACCACC MYB1AT: sito di riconoscimento MYB ritrovato nei promotori del gene rd22 e in altri geni coinvolti nella risposta alla disidratazione di Arabidopsis. 417 - 422 (+) TAACCA
MYBATRD2: sito di legame per MYB (ATMYB2) nei geni rd22 coinvolti nella risposta alla disidratazione, localizzato a circa 141pb a monte del gene
rd22. 436 - 442 (+) CTAACCA
MYB1AT: sito di riconoscimento MYB ritrovato nei promotori del gene rd22, coinvolto nella risposta alla disidratazione, e in altri geni di Arabidopsis. 437 - 442 (+) TAACCA ABRETAEM: “ABRE, ABA responsive element" trovato in grano, nel gene Em; i transacting factor EMBP-1 legano il motivo CACGTGGC. 490 - 502 (-) GGACAC GTGGCAT DPBF-CORE-DCDC3: classe di fattori di trascrizione bZIP, DPBF-1 e 2 (Dc3 promoter-binding factor-1 e 2) binding core sequence; trovati nel
promotore del gene Dc3 di carota. Dc3 ha un’espressione embrione-specifica, e può essere anche indotto dall’ABA. Il gene di risposta all’acido abscissico in Arabidopsis ABI5 codifica un fattore di trascrizione bZIP; mutanti difettivi abi5 mostrano difetti pleiotropici in risposta all’ABA. ABI5 regola molti geni coinvolti nella tarda embriogenesi. GIA1 (growth-insensitivity to ABA) è identico a ABI5.
494 - 500 (-) ACACGT G CTRM-CAM-V35S: motivo ricco in CT (complementare: GAGA) trovato in una regione di 60 nucleotidi (S1) a monte del sito di inizio della trascrizione
del CaMV 35S RNA; può aumentare la trascrizione genica. 578 - 586 (-)
TCTCTCT CT ARFAT, ARF (fattore di risposta auxina): sito di legame ritrovato nei promotori di geni coinvolti nella risposta precoce e primaria all’auxina in
Arabidopsis thaliana; AuxRE, Binding site of Arabidopsis ARF1 (Auxin response factor1); sequenza ritrovata nell’elemento NDE in soia, SAUR (Small Auxin-Up RNA) promotore genico 15A, queste elemento è ricorrente nelle regioni fiancheggainti al 5' dei geni regolati entrambi da IAA e BL (Goda et al., 2004).
583 - 589 (-) TTGTCTC
CTRM-CAMV35S: motivo ricco in CT (complementare: GAGA) trovato in una regione di 60 nucleotidi (S1) a monte del sito di inizio della trascrizione
del CaMV 35S RNA; Può aumentare la trascrizione genica. 601 - 609 (-)
TCTCTCT CT CATAT-GGM-SAUR: sequenza ritrovata nell’elemento NDE in soia, SAUR (Small Auxin-Up RNA) promotore genico 15A; coinvolto nella risposta
B
CIS-ACTING SEQUENCE PRENSENTI NELLA REGIONE DELL’INTRONE
Nome Posizione
da-a Strand Sequenza
MYB: sito di riconoscimento MYB ritrovato in diversi geni di Zea mays coinvolti nella risposta della pianta alla disidratazione.
30 – 46; 33 – 49; 58 – 74; 144 – 160; 146 – 162; 147 – 163; 149 – 165;
153 – 169; 173 – 189; 674 – 690; 183 – 199; 179-195 (-) TAACCA MYBPH3: Myb-like proteina ritrovata in Petunia hybrida 62 – 78; 380 – 396; 576 – 592; 600 - 616 (+) TAGT
GA-MYB: regolatore delle GA, gene myb ritrovato in orzo 251 - 267 (+) GTTAT
SP8BF: Proteina di legame del DNA: lega le sequenze SP8a (ACTGTGTA) e
SP8b (TACTATT) geni della sporamina e beta-amilasi 384 – 396; 406 - 418 (+) TACT
AREF: Elemento di legame repressore trascrizionale 604 - 616 (-) TGTC AAC
TEIL: tabacco EIN3-like, insensibile all’etilene 183 - 191 (-) TGTAT
ABRE: Attivatore trascrizionale indotto da ABA 652 - 668 (+) AACAAGT
RY: Motivi conservati RY e Sph in promotori specifici dei semi 654 – 680; 657 – 683; 661 - 687 (-) CATGC A
AREF: Elemento di risposta alle auxine 692 - 704 (+) TGTCCC
AHBP: Arabidopsis thaliana homeo box protein 1 449 – 459; 763 - 773 (+) TAATTATT
AHBP Omeodominio di proteine a cerniera di leucina Hahb-4 di girasole 763 - 773 (-) ATAATTAA
WUS: Omeodominio della proteina WUSCHEL 800 - 810 (-) TTAATGA
SUCB: Motivo ritrovato in diversi geni legati alla risposta agli zuccheri 845 – 863; 825 – 843; 562 - 580 (-) AAGATCA
SURE: Elemento di risposta agli zuccheri 840 - 852 (-) TAAAAAAAA
HOCT: Motivo ottamerico trovato nei geni istonici H3 e H4 228 - 244 (-) CGATCAGCA
MYB MYB-like MYB-like MYB-like Motivo ottamerico SP8 MYB RY e Sph ARE SURE ARE AREF WUSHEL Proteina Homeobox
Nome Posizione
da -a Strand
Sequenza WRKY: fattore di tipo zinc-finger associato alla difesa da patogeni, W box 88 - 104 (-) GTTTGAC R2R3-type: myb-like fattore trascrizionale (sito di legame I-type) 114 - 130 (-) CAGTTT
SUCB: sequenza di diversi geni in risposta agli zuccheri 166 - 184 (-) AATTCA
MYB: Myb-like proteina di Petunia hybrida 194 - 210 (-) CTGTTAAA
NAC: Wheat NAC domain DNA binding factor 200 - 222 (-) ATAACGGA
MYB-GA: gene myb regolatore delle GA ritrovato in orzo 201 - 217 (+) CCGTTATT
Hahb-4: Omeodominio cerniera di leucine ritrovato in Helianthus Annuus 209 - 219 (+) TATTAG
WUS: Omeodominio proteina WUSCHEL 243 - 253 (+) TTAATGA
Hahb-4: Omeodominio cerniera di leucine ritrovato in Helianthus Annuus 247 - 257 (+) ATGATTA
WUS: Omeodominio proteina WUSCHEL 251 - 261 (-) TTTAATC A
BLR: repressore trascrizionale BELLRINGER 265 - 275 (-) ATATTA
MYB: Myb-like proteina di Petunia hyb rida 385 - 401 (+) GCTAGTTAC
MYB: Myb-like proteina di Petunia hybrida 389 - 405 (-) TTAGTAA
GT1-Box : fattore di legamen con un dominio tri-elica legante il DNA 391 - 407 (-) GGTTAGT
ABRE: elemento di risposta all’ABA 421 - 437 (+) ACGTCTC
MybSt1: (Myb 1) con una singola ripetizione myb ritrovato in Solanum tuberosum 462 - 478 (+) TATCCGA
Motivo GAAA: coinvolto nell’attivazione trascrizionale 482 - 498 (+) GAAATACAT
MYB-like MYB-like MYB-like SURE SUCB NAC BELLIRINGER Leucine zipper WUSHEL WUSHEL Leucine zipper ABRE MIB GAAA Leucine zipper
C
(A) Regione intergenica al 5’. La numerazione riportata nella tabella sottostante considera come posizione 1 il p rimo nucleotide della regione posizionata a (-1557;+47) considerando +1 il putativo sito di inizio della trascrizione.
(B) Regione dell’introne. La numerazione riportata nella tabella sottostante considera come posizione 1 il primo nucleotide della regione intronica posizionata a (+103; +966) considerando +1 il putativo sito di inizio della trascrizione.
(C) Regione del 3’UTR. La numerazione riportata nella tabella sottostante considera come posizione 1 il primo nucleotide della regione posizionata a (+1506;+1906) considerando +1 il putativo sito di inizio della trascrizione.
3.3.3 Analisi della regione al 3’ del gene
La ricerca dei segnali di poliadenilazione nucleare eseguita al sito http://genes.mit.edu/genomescan.html ha visualizzato due motivi GCAACAAATT e GTAAAAACT posizionati rispettivamente a –85 e –48 pb relativamente all’ultimo nucleotide presente sul 3’UTR dell’mRNA, a +24 dallo stesso nucleotide è invece posizionabile il segnale di terminazione della trascrizione con la sequenza AGTGTTTA. In particolare la sequenza GCAACAAATT è necessaria nel processo di taglio del trascritto, prima dell’attacco della coda poliA, che avviene in corrispondenza del dinucleotide TA evidenziato in Fig. 3.14. Infine a –127 dal sito di taglio è riconoscibile la sequenza GTTTGTTTTTT, definita CPE (Cytoplasmic
Polyadenylation Element), che rappresenta un segnale di poliadenilazione
citoplasmatica. AGTGCGGCTGGCCAGTCTGGGCCTGCTGGTTTCGAGCCGTATGCTCAGTGTAAAGACTAA 60 CTGGTTTCGATTTTCGAGCCGTGTGTTGGTTGGTTTGTTTTTTTTTCTTGTTTGTTTGTC 120 TTCTTGGTCTTGGAGCAACAAATTACATTGGGTCAAACTTTGGCAAGGCATGTAAAAACT 180 GACACTCTCCATCTACTGGTAGTAATCTGCTTTTCTTCCTAACTTTTATTTTATGAATTT 240 GGAGTGTTTAAGATTTAACAGTTCCGTTATTATTAGTATTTTTTTAACATTTAAAAATAT 300 CAATTAATGATTAAAAATGTTTAAATAATATTTTCCTAACATTTAACCCGGTATTATTGG 360 TGTTTTTTGAAACGATGCTTGCTTTCTCTTCCAAAATTTGTGCCATCGCCCTAGCCTGGA 420 TTTGTCGAGAAAGACAAAATGAATGGCTAGTTACTAACCTACTCGACGCGGGAAGGGATT 480 TACAACGTCTCTGATCTGAAGAACTACAGCTTTTTGTATTCAATATCCGATAAAAATGCT 540 CAAGAAATCATGATTAGTCCT 561
Figura 3.14. Sequenza dell’estremita 3’ del gene HaL1L.
GCAACAAATT, GTAAAAACT: segnali di poliadenilazione nucleare; AGTGTTTA: segnale di terminazione della trascrizione;
in violetto è indicata la porzione del CDS di HaL1L, TAA rappresenta il codone di fine della traduzione;
in arancio è indicata la regione 3’UTR dell’mRNA per HaL1L;
in verde è indicata la porzione di gene simile a AT5G47680.1 di A. thaliana.
CTA rappresenta il codone di fine della traduzione per il gene a valle di HaL1L (simile a AT5G47680.1 di A. thaliana) orientato inversamente;
3.3.4 Analisi delle citosine metilate nelle isole CnG del
promotore
Per valutare l’eventuale coinvolgimento di eventi di metilazione di residui di citosina nella regolazione della trascrizione di HaL1L è stato deciso di utilizzare il trattamento con sodio bisolfito per la selettiva modificazione di citosine non metilate in uracile.
La prima fase della procedura richiede l’identificazione nella sequenza di DNA di regioni a monte del gene HaL1L in cui siano ravvisabili le così dette “isole CG”. In realtà deve essere tenuto conto che nelle piante anche citosine presenti in siti CnG o CnnG o semplicemente CHH (dove H = A, C or T), possono essere soggette a metilazione differenziale, quindi una volta stabilito un protocollo sperimentale efficiente, dovrà essere valutata l’opportunità di estendere l’analisi a tutta la regione intergenica al 5’ di HaL1L.
Tramite il programma CpGfinder al sito Softberry (www.softberry.com) sono state individuate due zone che possono essere soggette a metilazione e che sono state etichettate come Is1 e Is2 (Fig. 3.12 e Tab 3.1).
Inizio Fine Strand CpG %CG CG/GC P(CpG)/exp P(CpG) Lunghezza Is1 -700 -500 + 12 44.5 1,200 1,222 (1,81) 0,060 200 Is2 -153 -52 + 5 53.0 1,000 0,853 (1,39) 0,051 101 Tab.3.1. Probabili isole sottoposte a metilazione del gene HaL1L.
Una volta individuate le due regioni Is1 e Is2 sono stati condotti gli esperimenti di modificazione con il sodio bisolfito.
L’efficacia del trattamento dipende strettamente dai seguenti parametri:
• Purezza e dimensioni del DNA.
• Concentrazione del sodio bisolfito e idrochinone. • Temperatura.
• Tempo di trattamento.
• Caratteristiche dei primers da utilizzare nelle PCR che seguono il trattamento con sodio bisolfito.
Per individuare il metodo che permettesse il maggior grado di conversione sono stati quindi effettuati 9 trattamenti diversi utilizzando o meno kit disponibili in commercio e variando i parametri sopraddetti (Tabella 2.1 in Materiali e Metodi). Nella fase di messa a punto del protocollo è stato utilizzato DNA di foglie di H.
annuus, poichè dalle analisi descritte nel paragrafo 3.1, in questo organo, l’HaL1L
risulta praticamente silente, condizione che presuppone uno stato di ipermetilazione delle citosine.
I DNA derivati da ciascun trattamento sono stati sottoposti a due successive PCR in cui la coppia di primers utilizzata nella seconda PCR è posizionata internamente alla coppia di primers della prima PCR. Il trattamento a qualunque temperatura, durata e concentrazione del sodio bisolfito ha un effetto distruttivo sul DNA, per questo e per il fatto che i filamenti + e – del DNA non sono più complementari, in taluni casi le PCR mostrano bande multiple di amplificati aspecifici o non producono affatto amplificati evidenziabili. Tuttavia, una volta stabilite le condizioni che permettessero di visulizzare, almeno nella seconda PCR, prodotti di amplificazione, questi sono stati clonati e sequenziati.
Per quanto riguarda l’Is1 in particolare una complicazione è derivata dal fatto che il prodotto di amplificazione ottenuto nelle prime prove effettuate, è risultato tossico
per le cellule TOP10F’ utilizzate nel clonaggio. Per la soluzione, almeno parziale, del problema è stato necessario scegliere coppie di primers che dessero un amplificato di minori dimensioni.
A causa della non complementarietà dei filamenti, è necessario analizzare un consistente numero di cloni sequenziati e confrontare tra loro solo quelli derivati da uno stesso filamento del DNA trattato.
Fig. 3.15. Amplificazione del DNA modificato con differenti trattamenti.
Corsia 1-5 Isola1: trattamento 8, trattamento 6, trattamento 7, trattamento 9 DNA foglie, trattamento 9 DNA embrioni. (Primers utilizzati: I PCR: Nero-Blu, II PCR Rosso-Viola)
Corsia 5-10 Isola2: trattamento 8, trattamento 9 DNA foglie, trattamento 9 DNA embrioni, trattamento 6, trattamento 7. (Primers utilizzati: I PCR: Giallo-Indaco, II PCR: Pippo-Is2Fnest) M: Marker
Il protocollo più efficiente è risultato essere il numero 9 utilizzante i reagenti del kit Chemicon, ma dove sono state introdotte fasi di denaturazione a 98 °C, alternate all’incubazione a 55 °C durante il trattamento con il sodio bisolfito come riportato nella Tabella 2.1 di Materiali e Metodi. Non sembrano invece esserci miglioramenti significativi nell’estendere a 180 minuti l’incubazione quando utilizzato il kit SIGMA,
M 1 2 3 4 5 6 7 8 9 10
Isola1: 271 pb Isola2: 452 pb
e neppure l’impiego di microbeads come nel protocollo 4. Sostanziale è invece risultata la scelta dei primers necessari per le amplificazioni successive al trattamento con il sodio bisolfito. Infatti le uniche coppie di primers utilizzabili sono risultate essere per Is1: Nero-Blu; Rosso-Viola; per l’Is2: Giallo-Indaco, Pippo-Is2Fnest. (Fig. 3.15)
Nella Fig. 3.16 sono confrontati alcuni cloni derivati dai trattamenti 7, 8 e 9 relativi alla regione Is1.
is1-1 TTTATTGAGTTTTTTTCTGAATTAGTGTATAGCCGTTATAGTTTAGTGGAGATGGATATGATCGGGTGGTTCCGCTGGTGGTACGATGATATTCCAGTGGTCCGTCAGTGAT is1-3 TTTATTGAGTTTTTTTCTGAATTAGTGTATAGTTGTTATAGTTTAGTGGAGATGGATATGATCGGGTGGTTTCGTTGGTGGTACGATGATATTTCAGTGGTTCGTCAGTGAT is1-4 TTTATTGAGTTTTTTCCTGAATTAGTGTATAGCCGTTATAGCTTAGTGGAGATGGATATGATCGGGTGGTTTCGTTGGTGGTACGATGATATTTCAGTGGTTCGTCAGTGAT is1-11 TTTATTGAGTTTTTTTCTGAATTAGTGTATAGTTGTTATAGTTTAGTGGAGATGGATATGATCGGGTGGTTCCGTTGGTGGCACGATGATACTCCAGTGGTCCGTCAGTGAT is1-12 TTTATTGAGTTTTTTTCTGAATTAGTGTATAGTCGTTATAGTTTAGTGGAGATGGATATGATCGGGTGGTTTCGTTGGTGGTACGATGATATTTCAGTGGTTCGTCAGTGAT is1-13 TTTATTGAGTTTTTCTCTGAATTAGTGTATAGTTGTTATAGTCTAGTGGAGATGGATATGATCGGGTGGTTTCGTTGGTGGTACGATGATACTTCAGTGGTCCGTCAGTGAT is1-16 TTTATTGAGTTTTTTTCTGAATTAGTGTATAGTTGTTATAGTTTAGTGGAGATGGATATGATCGGGTGGTTTCGTTGGTGGTACGATGATATTTCAGTGGTTCGTCAGTGAT is1-17 TTTATTGAGTTTTTTTCTGAATTAGTGTATAGTTGTTATAGTTTAGTGGAGATGGATATGATCGGGTGGTTTCGTTGGTGGTACGATGATATTTCAGTGGTTCGTCAGTGAT is1-18 TTTATTGAGTTTTTTTCTGAATTAGTGTATAGTTGTTATAGTTTAGTGGAGATGGATATGATCGGGTGGTTTCGTTGGTGGTACGATGATATTTCAGTGGTTCGTCAGTGAT ************ * **************** ******* **************************** ** ****** ********* * ******* ********** ISOLA1 CCTAACTTGCCGTTCAAAAAGAACATCGTCTGTATATATAGCCTCTGCACATGTAACAGTTGCGCCACTAACCAATGGTTGTCGTTGACACGTTTGATACGCGATATTAAAA is1-1 TTTAATTTGTCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA is1-3 TTTAATTTGCCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA is1-4 TTTAATTTGTCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA is1-11 CCTAACTTGTCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA is1-12 TTTAATTTGTCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATCAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA is1-13 TTTAATTTGCCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATCAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA is1-16 TTTAATTTGTCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA is1-17 TTTAATTTGTCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA is1-18 TTTAATTTGTCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGTTATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAA *** **** *** ******** ***************** **** * ************** * *** *************** * ********************* ISOLA1 AATGAGTTGAGAGATTATTTTACACAAATCAAGG Is1-1 AATGAGTTGAGAGGTTATTTTATATAAATCAAGG Is1-3 AATGAGTTGAGAGATTATTTTATATAAATTAAGG Is1-4 AATGAGTTGAGAGATTATTTTATATAAATCAAGG Is1-11 AATGAGTTGAGAGATTATTTTATATAAATCAAGG Is1-12 AATGAGTTGAGAGATTATTTTATACAAATCAAGG Is1-13 AATGAGTTGAGAGATTATTTTATATAAATCAAGG Is1-16 AATGAGTTGAGAGATTATTTTATATAAATTAAGG Is1-17 AATGAGTTGAGAGATTATTTTATATAAATTAAGG Is1-18 AATGAGTTGAGAGATTATTTTATATAAATTAAGG ********************** * **** ****
Fig. 3.16. Allineamento cloni isola1 ottenuti con differenti metodi di incubazione.
3.4 Indagine preliminare sullo stato di
metilazione di HaL1L in organi che esprimono
differenzialmente il gene
Le indagini condotte e descritte nei paragrafi 3.1 indicano che la quantità di trascritto del gene HaL1L è massima negli embrioni a cinque giorni di sviluppo, mentre sembra essere praticamente nulla nelle foglie. Un confronto preliminare dello stato di metilazione del DNA degli organi suddetti è stato quindi eseguito utilizzando il protocollo di trattamento con sodio bisolfito n. 9 (Tab.2.2 Materiali e Metodi) e le coppie di primers Nero-Blu, Rosso-Viola per l’Is1, e Giallo-Indaco, Pippo-Is2Fnest per l’Is2. L’analisi dei risultati è stata eseguita con il programma CyMATE (http://www.gmi.oeaw.ac.at/CyMATE), che permette di estrarre, da un allineamento di sequenze, dati quantitativi e qualitativi e di riconoscere pattern specifici di metilazione. Nella Tab. 3.2 ottenuta con CyMate sono visualizzate le tre classi principali di siti metilabili individuati nella regione dell Is1:
1° base 2° base 3° base Classe
C o T (Y) qualsiasi (N) qualsiasi (N) nessuna C o T (Y) G qualsiasi (N) YGN C o T (Y) qualsiasi tranne G (H) G YHG C o T (Y) qualsiasi tranne G (H) qualsiasi tranne G (H) YHH
§ Classe “CGN”: frequenza 12 siti su 49 (24.49%). Per le posizioni: [36, 65, 75, 86, 105, 125, 141, 177, 197, 205, 214, 216]
§ Classe “CHG”: frequenza 11 siti su 49 (22.45%). Per le posizioni [19, 35, 74, 77, 97, 104, 108, 124, 144, 159, 171]
§ Classe “CHH”: frequenza 26 siti su 49 (53.06%). Per le posizioni [15, 16, 18, 44, 45, 84, 94, 96, 115, 116, 120, 129, 138, 156, 157, 162, 164, 179, 180, 182, 186, 187, 203, 249, 251, 256],
Nelle Fig. 3.17 e 3.18 sono invece visualizzate le sequenze rappresentative rispettivamente del DNA delle foglie e degli embrioni a cinque giorni di sviluppo, confrontate tra loro e con il DNA non trattato nella porzione di DNA indicata come Is1.
Fig. 3.17. Allineamento dei cloni ottenuti da DNA di foglie ed embrioni.
Il grafico deriva dall’analisi realizzata con il programma CyMATE. Sono confrontate le sequenze rappresentative rispettivamente del DNA non trattato, del DNA delle foglie e degli embrioni a cinque giorni di sviluppo sottoposti a trattamento con bisolfito, relativamente all’Is1.
Fig. 3.18. Confronto delle sequenze rappresentative rispettivamente del DNA non trattato, DNA delle foglie e degli embrioni a cinque giorni di sviluppo sottoposti a trattamento con bisolfito. Le frecce indicano i siti differentemente metilati.
Con l’analisi preliminare effettuata sono stati identificati nell’Is1 residui di citosina che rappresentano potenziali siti di metilazione differenziale. Infatti alcune citosine, indicate con frecce nella Fig 3.18, nel DNA dell’embrione sono sostituite da timine dopo il trattamento con sodio bisolfito, mentre non lo sono nel DNA delle foglie. Ciò indica che solo in quest’ultimo organo le posizioni evidenziate hanno subito la modificazione biochimica dell’aggiunta di un gruppo metilico.
Foglie AGATTTATTGAGTTTTTTTCTGAATTAGTGTATAGTTGTTATAGTTTAGTGGAGATGGAT Embrioni AGATTTATTGAGTTTTTTTTTGAATTAGTGTATAGTTGTTATAGTTTAGTGGAGATGGAT 60 ATGATCGGGTGGTTCCGCTGGTGGCACGATGATACTCCAGTGGTCCGTCAGTGATCCTAA ATGATCGGGTGGTTTCGCTGGTGGTACGATGATATTTCAGTGGTTCGTCAGTGATTTTAA ATGATCGGGTGGTTTTGCTGGTGGTACGATGATATTTCAGTGGTTCGTTAGTGATTTTAA 120 CTTGCCGTTCAAAAAGAACATCGTCTGTATATATAGCCTCTGCACATGTAACAGTTGCGC TTTGTCGTTTAAAAAGAATATCGTCTGTATATATAGTTTCTGTATATGTAACAGTTGCGT TTTGTCGTTTAAAAAGAATATCGTTTGTATATATAGTTTTTGTATATGTAATAGTTGCGT 180 CACTAACCAATGGTTGTCGTTGACACGTTTGATACGCGATATTAAAAAATGAGTTGAGAG TATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAAAATGAGTTGAGAG TATTAATTAATGGTTGTCGTTGATATGTTTGATACGCGATATTAAAAAATGAGTTGAGAG 240 ATTATTTTACACAAATCAAGGAAATATGTAA ATTATTTTATATAAATTAAGGAAATATGTAA ATTATTTTATATAAATTAAGGAAATATGTAA 270